Генетика, 2021, T. 57, № 9, стр. 1021-1030
Компоненты внутриродовой динамики размера генома у растений и животных
А. М. Бутанаев *
Федеральный исследовательский центр “Пущинский научный центр биологических исследований”,
Институт фундаментальных проблем биологии Российской академии наук
142290 Московская область, Пущино, Россия
* E-mail: boutanaev@mail.ru
Поступила в редакцию 30.09.2020
После доработки 19.01.2021
Принята к публикации 04.02.2021
Аннотация
Настоящее исследование посвящено динамике размера генома в процессе эволюции видов растений и животных, принадлежащих к одному роду. В масштабе таксономических категорий царств растений и животных размер генома видов имеет тенденцию к снижению, т.е. виды с небольшим геномом существенно преобладают. Однако внутри родов, как растений, так и животных, размер генома может увеличиваться, уменьшаться или оставаться относительно стабильным на протяжении всего времени эволюции рода. Эти три варианта составляют базовые компоненты внутриродовой динамики размера генома. Помимо простой динамики, состоящей из одной компоненты, существует сложная составная динамика размера генома, включающая две или даже три базовые компоненты. Динамика диплоидов и полиплоидов внутри родов растений может существенно различаться, что отражает действие разных эволюционных факторов. Причины смены базовой компоненты в случае сложной динамики не вполне понятны, однако одной из них может быть глобальное похолодание, начавшееся три миллиона лет назад.
Размер генома (РГ) – фундаментальный признак, являющийся точкой приложения эволюционных факторов и играющий важную роль в эволюции и адаптации видов растений и животных. Основной вклад в увеличение количества ДНК вносят полиплоидия и мобильные генетические элементы [1–4], а ее потеря связана с механизмами рекомбинации и репарации (см, например, [5]). Возможный вклад в изменение РГ небольших вставок и делеций обсуждается в [6, 7]. В настоящее время известно достаточно много корреляций между размером генома и факторами внешней среды, а также биохимическими и структурными внутриклеточными параметрами у растенийи животных (см., например, [8–15]). На РГ могут влиять такие многофакторные признаки как уровень сложности индивидуального развития [16], возрастающая сложность организации в процессе эволюции [17] и интенсивность метаболизма [18] у животных, а также листовая стратегия (leaf strategy) [19]. Листовая стратегия подразумевает время жизни листа и отношение масса/поверхность. В смешанной группе покрытосеменных и голосеменных растений присутствует слабая корреляция между РГ и параметрами листовой стратегии, однако в отдельных группах тех и других корреляция отсутствует. Известна также ассоциация РГ и жизненного цикла у растений [14]. Снижение функциональной эффективности (интенсивность функций на 1 пг ДНК) у покрытосеменных с одновременным увеличением содержания ДНК может быть связано с изменением климата в сторону более жестких условий [20]. В отдельных случаях изменение РГ совпадает с классификацией таксоноврастений и может использоваться в их систематике [21, 22]. Тот факт, что селективное давление влияет на РГ вида, подтвержден в многочисленных работах и не вызывает сомнения. Однако в нашем недавнем исследовании внутриродовой динамики РГ у диплоидных семенных растений приведены доказательства того, что естественный отбор, по-видимому, играет значительно меньшую роль в эволюции этого признака, в отличие от того, как это принято считать [23]. Была показана линейность динамики РГ на протяжении миллионов лет эволюции многих родов растений. Такая линейность не предполагает существенного влияния отбора, так как маловероятно, что факторы внешней среды, действуя одно направленно в сторону уменьшения или увеличения РГ, оставались бы неизменными на протяжении всей эволюции рода. Существуют, по меньшей мере, две интерпретации такой внутриродовой динамики РГ [23]. Одна из них утверждает, что отбор действует только на его значительные изменения, в то время как небольшие вставки и делеции ДНК не влияют на его эволюцию, что приводит к преобладанию действия внутриклеточных факторов, таких как система ферментов метаболизма ДНК [24]. Другая интерпретация основана на концепции динамического хаоса в эволюции РГ. В этом случае постулируется эволюционная положительная обратная связь между изменением РГ в ту или иную сторону и действием отбора опять же на ферменты метаболизма ДНК, которые, в том числе, отвечают за рекомбинацию и дупликацию [25].
Механизмы эволюции РГ внутри родов растений и животных, определяющие линейную динамику, в значительной степени не известны, и требуется еще немало исследований как самой динамики РГ, так и определяющих ее факторов.
БАЗЫ ДАННЫХ И КОМПЬЮТЕРНОЕ ПРОГРАММИРОВАНИЕ
Величины размера генома растений (количество ДНК в пикограммах) получены из базы данных “C‑value database” (Royal Botanic Gardens, Kew, UK.; http://data.kew.org/cvalues) [26]. Данные по размеру генома видов млекопитающих, птиц и земноводных получены из базы данных “Animal genome size database” (http://www.genomesize.com) [27]. Компьютерная программа для обработки текстовой информации баз данных была создана ранее [23]. Программа извлекает необходимую информацию и приписывает РГ каждого вида соответствующему роду и семейству.
ВРЕМЕНА ДИВЕРГЕНЦИИ ВИДОВ И СТАТИСТИКА
Для вычисления времен дивергенции видов использовали опубликованные калиброванные филогенетические деревья для растений [28–34] и животных: млекопитающих [35–40], птиц [41–43] и земноводных [44–47]. В основном филогенетические деревья публикуются в виде графических образов без времен дивергенции, поэтому прямое измерение длины ветвей дерева – единственный способ их нахождения. Подробно вычисление времен дивергенции видов на основе графических образов дендрограмм описано в [23].
Корреляционный анализ, регрессионный анализ и статистические критерии были выполнены с использованием программы SPSS Statistics 17.0.
ДИНАМИКА РАЗМЕРА ГЕНОМА У ДИПЛОИДНЫХ РАСТЕНИЙ
В нашей предыдущей работе была обнаружена сильная корреляция Пирсона между временем дивергенции видов (эволюционным возрастом) и размером генома внутри родов диплоидных семенных растений. Линейность зависимости была подтверждена регрессионным анализом [23]. Обнаруженная прямая и обратная корреляция отражает, соответственно, линейное уменьшение или увеличение РГ в зависимости от времени дивергенции. Эта линейная зависимость сохранялась в течение миллионов лет, что подразумевает постепенное увеличение или уменьшение РГ в процессе эволюции видов, принадлежащих к одному и тому же роду. В некоторых родах корреляция отсутствовала, что предполагает относительно стабильный РГ. Эти три характеристики, т.е. относительно стабильное количество ДНК, ее накопление или потеря, представляют собой основные базовые компоненты внутриродовой динамики РГ (рис. 1; диплоиды родов Malus, Limonium и Pinus).
Рис. 1.
Примеры внутриродовой динамики размера генома у растений. Представлены рода Malus, Limonium, Pinus и Crataegus с простыми базовыми компонентами у диплоидов (круги синего цвета) и рода Hordeum и Oryza со сложной двухкомпонентной динамикой. Разные компоненты у диплоидов показаны синими и красными кругами. Представлены также примеры динамики РГ у тетраплоидов Hordeum и Oryza (коричневые квадраты).
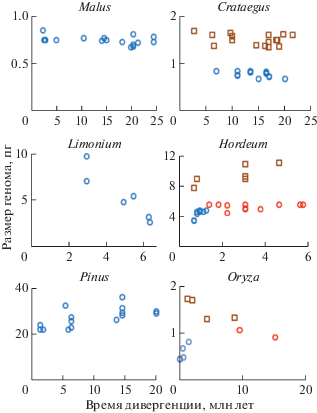
В настоящем исследовании применен такой же методологический подход с вовлечением других видов растений, а также видов животных с целью обобщения полученной информации. Результаты (коэффициенты корреляции и коэффициенты детерминации линейной регрессии), подтверждающие сильную корреляцию между РГ и временем дивергенции видов внутри родов, а также линейный характер этой зависимости, представлены в табл. 1. Из 17 базовых компонент динамики РГ со статистически значимой корреляцией (17 родов растений и животных) линейная зависимость не подтверждена лишь для трех. Это относится к родам Hordeum, Oryza (рис. 1, синий цвет) и Schiedea (рис. 2, зеленый).
Таблица 1.
Зависимость размера генома от времени дивергенции видов внутри родов растений и животных
| Семейство | Род | Плоидность | Компонента динамики РГ | r | p | R2 | p | Виды | |
|---|---|---|---|---|---|---|---|---|---|
| в анализе | исключенные | ||||||||
| Растения | |||||||||
| Rosaceae | Malus | 2n | рис. 1, син. | –0.40 | 0.05 | 0.16 | 0.06 | 17 | 0 |
| Rosaceae | Crataegus | 2n | рис. 1, син. | –0.56 | 0.02 | 0.31 | 0.04 | 13 | 0 |
| Rosaceae | Crataegus | 4n | рис. 1, кор. | –0.23 | 0.39 | 0.05 | 0.11 | 16 | 0 |
| Plumbaginaceae | Limonium | 2n | рис. 1, син. | –0.92 | 0.005 | 0.85 | 0.01 | 6 | 0 |
| Pinaceae | Pinus | 2n | рис. 1, син. | 0.65 | 0.004 | 0.42 | 0.01 | 15 | 0 |
| Poaceae | Hordeum | 2n | рис. 2, кр. | 0.40 | 0.22 | 0.16 | 0.30 | 11 | 0 |
| Poaceae | Hordeum | 2n | рис. 2, син. | 0.42 | 0.004 | 0.18 | 0.30 | 8 | 0 |
| Poaceae | Hordeum | 4n | рис. 1, кор. | 0.79 | 0.03 | 0.62 | 0.03 | 7 | 0 |
| Poaceae | Oryza | 2n | рис. 1, син. | 0.87 | 0.04 | 0.76 | 0.06 | 5 | 0 |
| Amaryllidaceae | Allium | 2n | рис. 2, кр. | 0.89 | 0.03 | 0.79 | 0.04 | 5 | 0 |
| Amaryllidaceae | Allium | 2n | рис. 2, син. | 0.15 | 0.64 | 0.02 | 0.62 | 12 | 0 |
| Amaryllidaceae | Allium | 2n | рис. 2, зел. | –0.25 | 0.46 | 0.06 | 0.44 | 11 | 0 |
| Caryophyllaceae | Schiedea | 2n | рис. 2, син. | 0.84 | 0.04 | 0.71 | 0.04 | 5 | 0 |
| Caryophyllaceae | Schiedea | 2n | рис. 2, зел. | 0.43 | 0.04 | 0.18 | 0.07 | 18 | 0 |
| Животные | |||||||||
| Млекопитающие | |||||||||
| Vespertilionidae | Myotis | 2n | рис. 3, кр. | –0.93 | 3 × 10–5 | 0.86 | 9 × 10–4 | 11 | 0 |
| Vespertilionidae | Myotis | 2n | рис. 3, син. | 0.77 | 2 × 10–4 | 0.59 | 0.001 | 17 | 0 |
| Macropodidae | Macropus | 2n | рис. 3, син. | –0.67 | 0.04 | 0.45 | 0.04 | 5 | 1 |
| Птицы | |||||||||
| Turdidae | Turdus | 2n | рис. 3, син. | –0.81 | 0.03 | 0.65 | 0.02 | 7 | 0 |
| Gruidae | Grus | 2n | рис. 3, син. | –0.03 | 0.93 | 10–5 | 0.06 | 10 | 0 |
| Cisticolidae | Cisticola | 2n | рис. 3, син. | –0.34 | 0.58 | 0.12 | 0.57 | 5 | 0 |
| Psittacidae | Amazona | 2n | рис. 3, син. | –0.31 | 0.38 | 0.10 | 0.60 | 10 | 0 |
| Земноводные | |||||||||
| Plethodontidae | Bolitoglossa | 2n | рис. 3, син. | –0.49 | 0.03 | 0.24 | 0.04 | 16 | 0 |
| Plethodontidae | Pseudoeurycea | 2n | рис. 3, син. | 0.95 | 0.01 | 0.91 | 0.01 | 5 | 1 |
| Plethodontidae | Plethodon | 2n | рис. 3, син. | 0.75 | 0.02 | 0.56 | 0.02 | 9 | 3 |
Примечание. Представлены результаты корреляционного и регрессионного анализа. Значения коэффициентов корреляции (r) и коэффициентов детерминации (R2) с их уровнями значимости (p) показаны для родов, у которых число видов с доступными данными РГ и времени дивергенции 5 и более, что позволяет достаточно надежную статистическую обработку. Столбец “Компонента динамики РГ” указывает на соответствующий график или часть графика на рис. 1–3. Сокращения кор., кр., зел. и син. соответсвуют коричневому (тетраплоиды), красному, зеленому и синему (все диплоиды) цвету точек на графиках.
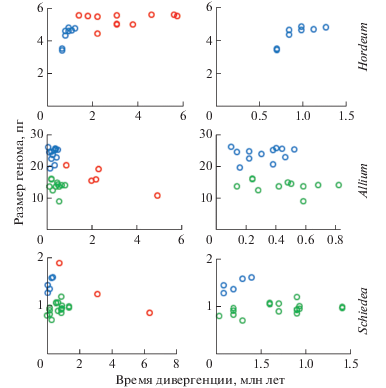
Рис. 2.
Сложная многокомпонентная динамика РГ у диплоидов родов Hordeum, Allium и Schiedea. Разные компоненты показаны красным, синим и зеленым цветом. В родах Allium и Schiedea РГ дивергирует.
Необходимо отметить, что не все рода имеют достаточное количество видов для статистической обработки данных (см., например, род Oryza на рис. 1, красная компонента РГ). Тем не менее статистически достоверные результаты нашего предыдущего исследования, касающегося динамики РГ многих родов диплоидных семенных растений [23], а также результаты настоящей работы (табл. 1) позволяют их экстраполировать и на большинство малочисленных родов как растений, так и животных.
Помимо основных базовых компонент РГ-динамики в родах растений существуют более сложные составные типы, как это показано на рис. 1. Например, у диплоидных видов Oryza (рис. 1) и Hordeum (рис. 1, 2) внутриродовая динамика РГ сложная, состоящая из двух базовых компонент: относительно стабильного и уменьшающегося РГ (соответственно красные и синие круги). Внутри рода Allium (Amaryllidaceae) динамика даже более сложная. Вслед за увеличением РГ у видов Allium в течение около 4.0 млн лет (рис. 2, средний ряд, красный цвет) последовала их дивергенция на две группы со средними значениями РГ 13.6 ± 1.9 и 23.6 ± 2.1 (средний ряд на рис. 2, зеленый и синий цвет). Согласно критерию Стьюдента, разница между средними статистически достоверна (р = = 1.9е–8). Впоследствии, хотя диапазон колебаний групповых значений РГ существенный, что составляет 5.0 и 10.4 пг (синие и зеленые круги), внутри обеих групп РГ не проявлял тенденции к увеличению или снижению на протяжении около 800 тыс. лет эволюции (корреляция отсутствует со значениями, соответственно, r = –0.25 и r = 0.15; табл. 1).
Другой интересный пример сложной динамики РГ представляет род Schiedea (Caryophyllaceae). У видов этого рода РГ возрастал на протяжении 6.0 млн (рис. 2, нижний ряд, красный цвет) и затем дивергировал, как и в случае Allium. Поскольку распределение РГ в обеих группах дивергировавших видов Schiedea отличается от нормального, вместо средних величин использовались медианы (1.43 ± 0.1 и 0.95 ± 0.02; синий и зеленый цвет соответственно). Согласно непараметрическому критерию Краскела–Уоллиса, различие между группами достоверно (p = 8е × 10–4). Впоследствии РГ уменьшался в обеих группах видов, дивергировавших по этому признаку, с коэффициентами корреляции 0.84 (синий цвет) и 0.43 (зеленый) (табл. 1).
Род Schiedea еще интересен тем, что его филогения, биология и экология интенсивно изучались, что предоставляет возможность связать переход динамики РГ от одного типа (компоненты) к другому с изменяющимися условиями окружающей среды. Виды этого рода являются эндемиками, обитающими на вулканических Гавайских островах, сформировавшихся последовательно в виде островной цепи. Монофилетическое происхождение 32 современных видов рода Schiedea, 26 из которых были включены в настоящее исследование, дает возможность полагать, что все они произошли в результате единственного события колонизации цепи вулканических островов. Местообитание большинства видов ограничено одним островом, однако пять из них встречаются более чем на одном острове [48]. Новые линии Schiedea, населяющие Гавайский архипелаг, могли появиться в результате переселения на более молодые острова до того, как более старые разрушались вследствие эрозии. Эволюция рода Schiedea включала межостровную и обратную колонизацию, а также внутриостровное видообразование [48, 49].
Дивергенция РГ в роде Schiedea, представленная на рис. 2, может быть связана с дивергенцией разных линий внутри рода, в частности с предпочитаемым местобитанием и системой размножения. Две группы с дивергировавшим РГ включают 5 (группа 1) и 18 (группа 2) видов, которые на рис. 2 показаны синим и зеленым цветом. Эти виды принадлежат к пяти секциям: Schiedea (S), Mononeura I (MI), Mononeura II (MII), Nothoschiedea (N), Alphaschiedea (Alp) и Alsinidendron (Als) [48]. Распределение видов по секциям представлено в табл. 2. В группе 1 четыре вида из пяти входят в секцию MI, в которой отсутствуют виды из группы 2, и один вид принадлежит к секции S. Напротив, половина членов группы 2 (9 из 18) принадлежит к этой секции (S). Виды из группы 1 также отсутствуют в секциях MII, N, Alp и Als, в то время как другая половина видов из группы 2 распределена по этим секциям.
Таблица 2.
Дивергенция размера генома и возможная ассоциация с филогенией, местообитанием и системой размножения рода Schiedea
| Виды группы 1 | Виды группы 2 | |
|---|---|---|
| Секция рода Schiedea | ||
| MI | 4 | 0 |
| S | 1 | 9 |
| MII | 0 | 4 |
| Als | 0 | 2 |
| Alp | 0 | 2 |
| N | 0 | 1 |
| Местообитание | ||
| Сухие склоны и скалы | 1 | 9 |
| Умеренно влажные леса | 2 | 7 |
| Влажные леса | 2 | 2 |
| Система размножения | ||
| Мономорфная | 4 | 10 |
| Диморфная | 1 | 8 |
Примечание. Виды группы 1 и группы 2 представлены на рис. 2 соответственно синим и зеленым цветом.
Разные виды Schiedea предпочитают три разных местообитания: сухие склоны и скалы, леса с умеренной влажностью и влажные леса [49]. Распределение видов двух групп по этим местообитаниям представлено в табл. 2. Лишь один вид из пяти в группе 1 населяет сухие местообитания. Остальные 4 вида предпочитают умеренно влажные и влажные леса. Напротив, половина видов из группы 2 занимают сухие склоны и скалы.
У видов рода Schiedea обнаружены две системы размножения: мономорфная и диморфная [50]. Две группы видов, дивергировавших по признаку РГ, также различаются в отношении системы размножения. Большинство видов из группы 1 (4 из пяти) имеют мономорфную систему размножения, тогда как в группе 2 мономорфизм характерен для 10 видов, а остальные 8 видов имеют диморфную систему размножения.
Таким образом, наблюдается явное смещение в распределении видов в группе 1 по трем факторам: филогенетическому родству, местообитанию и системе размножения. Единственный вид, отличающийся от остальных в этой группе по всем трем признакам, – S. mannii. Напротив, в группе 2 виды распределены примерно поровну между сухими и влажными местообитаниями, а также между двумя системами размножения (табл. 2). Возможная ассоциация между дивергенцией РГ и различиями в местообитании и системе размножения позволяет предположить, что внешние факторы играли существенную роль в его дивергенции. Можно полагать также, что вследствие изоляции островных популяций РГ основателей разных линий был разным и что дивергенция по признаку РГ происходила в разное эволюционное время: примерно 0.4 млн (группа 1) и 1.4 млн (группа 2) (рис. 2 и табл. 3).
ДИНАМИКА РАЗМЕРА ГЕНОМА У ПОЛИПЛОИДНЫХ РАСТЕНИЙ
Выше рассматривалась внутриродовая динамика РГ у диплоидных семенных растений, однако полиплоидные виды в этом отношении не исключение и следуют той же тенденции изменения РГ, т.е. его увеличения, уменьшения или относительной стабильности в процессе эволюции рода. На рис. 1 (коричневые квадраты) представлена динамика РГ тетраплоидов родов Crataegus, Hordeum и Oryza. Можно видеть, что в случае Crataegus РГ тетраплоидов существенно не изменяется на протяжении 18 млн, в то время как в роде Hordeum РГ снижается в течение 4 млн, а в роде Oryza – растет в течение 7 млн. Необходимо отметить, что динамика РГ у диплоидов и полиплоидов, относящихся к одному роду, может существенно различаться. Так, у диплоидов Crataegus РГ увеличивается с эволюционным временем, а у тетраплоидов он стабилен (рис. 1 и табл. 1). Диплоиды Hordeum и Oryza имеют двухкомпонентную динамику, тогда как тетраплоиды этих родов соответственно редуцируют и аккумулируют ДНК (рис. 1 и табл. 1).
ДИНАМИКА РАЗМЕРА ГЕНОМА У ЖИВОТНЫХ
Внутриродовая динамика РГ у животных включает те же базовые компоненты стабильности, увеличения и уменьшения, а также сложную динамику, состоящую из более чем одной компоненты. Особенность родов животных состоит в том, что большинство из них содержит небольшое число видов, что исключает статистический анализ ассоциации между переменными времени дивергенции и РГ. Тем не менее даже в малочисленных родах явно прослеживаются те же тенденции динамики РГ, что и в родах с большим числом видов. Например, в классе млекопитающих виды рода летучих мышей Artibeus накапливали ДНК в течение 1.8 млн лет. В широко известном роде Canis РГ оставался стабильным на протяжении 30 млн, начиная от серого волка (C. lupus; 30 млн, 2.8 пг), койота (C. latrans; 16 млн, 2.8 пг), красного волка (C. rufus; 0.39 млн, 3.0 пг) и заканчивая домашней собакой (C. familiaris; 0.03 млн, 3.1 пг).
Многочисленный род летучих мышей Myotis имеет сложную динамику РГ. Сначала РГ достаточно интенсивно растет в процессе эволюции от 1.9 до 3.3 пг в течение 4.3 млн, а затем, после резкого падения до 2.4 пг, постепенно незначительно снижается до 2.2 пг на протяжении 2.7 млн (рис. 3, красный и синий цвет; табл. 1). Времена дивергенции для рода Myotis вычислены на основе калиброванных дендрограмм из трех независимых исследований [35–37]. Максимальная разница между временами дивергенции повторяющихся видов этого рода была 0.06 млн в нисходящей компоненте динамики РГ (рис. 3, синий цвет) и 0.96 млн в восходящей компоненте (рис. 3, красный цвет), что свидетельствует о надежности подхода определения значений временной переменной (см. Материалы и методы и [23]).
Рис. 3.
Примеры внутриродовой динамики размера генома у животных. В роде Myotis динамика РГ двухкомпонентная. Выбросы из линейной зависимости между РГ и временем дивергенции обозначены знаком “×”.
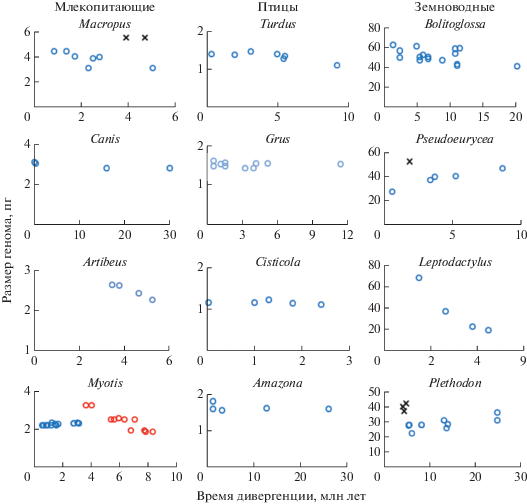
В классе земноводных у саламандр в роде Bolitoglossa РГ увеличивается на 20 пг за 20 млн, т.е. на 1 пг в миллион лет, в то время как в родах Pseudoeurecia и Plethodon РГ снижается на 20 пг за 8.1 млн и на 8 пг за 20 млн соответственно. В роде лягушек Leptodactylus РГ существенно возрастает на 48.3 пг в течение 3 млн (16 пг за 1 млн). В трех родах птиц, Grus, Cisticola и Amazona, РГ остается стабильным на протяжении соответственно 10.8, 2.4 и 24.5 млн, а в роде Turdus незначительно увеличивается на 0.28 пг за 8.8 млн (табл. 1 и рис. 3).
В родах Macropus, Pseudoeurycea и Plethodon некоторые виды не следуют общей тенденции, т.е. являются выбросами, и были исключены из анализа (рис. 3). В предыдущей работе мы подробно рассматривали причину таких выбросов в родах растений и пришли к выводу, что их появление не правомерно считать случайными ошибками определения как количества ДНК, так и времени дивергенции [23]. В настоящем исследовании, так же как и в предыдущем, стьюдентизированные остатки линейной регрессии видов-выбросов по переменной РГ находились в интервале 2 < σ < 3, тогда как за случайные отклонения принимаются значения, у которых остатки лежат в интервале σ > 3. Помимо результатов статистической обработки данных в поддержку неслучайного присутствия видов-выбросов, обращает внимание достаточная надежность современных методов изучения размера генома и филогении видов (подробнее см. в [23]), что также свидетельствует в пользу их закономерного появления в процессе эволюции РГ рода.
РГ видов-выбросов, в отличие от остальных, мог подвергаться сильному давлению отбора и быстрому эволюционному изменению в сторону увеличения или уменьшения. Подтверждением служит пример рода кенгуру Macropus с восходящей динамикой РГ в течение 4.1 млн лет. Для этого рода времена дивергенции взяты из двух независимых работ [38, 39]. Как видно из рис. 3, один вид из пяти (M. rufogriseus) является выбросом из линейной зависимости в обоих исследованиях филогении Macropus (знак “×” на рис. 3), что подтверждает закономерность присутствия таких видов во внутриродовой динамике РГ. Разница в значении времени дивергенции для этого вида составляет 0.7 млн. Возможно, что при большем числе доступных для анализа видов, принадлежащих к одному роду, некоторые из этих выбросов обратятся в дополнительную базовую компоненту сложной динамики РГ.
ПЕРЕХОД МЕЖДУ БАЗОВЫМИ КОМПОНЕНТАМИ СЛОЖНОЙ ДИНАМИКИ РГ И ГЛОБАЛЬНОЕ ИЗМЕНЕНИЕ КЛИМАТА
Интересно, что переход от одной базовой компоненты к другой у родов со сложной динамикой РГ происходил, примерно, в интервале 0.5–3.0 млн лет (табл. 3). У четырех родов растений этот переход имел место позднее (менее 2 млн), а у единственного представленного в настоящем исследовании рода животных с двухкомпонентной динамикой (Myotis) – ранее (3.2 млн). Точка 3 млн на шкале геологического времени хорошо известна в области исследования истории глобального климата. Известно, что последние три миллиона лет характеризуются циклами оледенений и межледниковых периодов с общим понижением глобальной температуры (см., например, [51–53]). Возможно, что смена тенденции в эволюции РГ видов внутри родов связана с понижением глобальной температуры 3 млн лет тому назад, однако только для немногих изученных родов характерна его сложная динамика. Например, у растений такая динамика наблюдается лишь у четырех родов из 26 (15%) (предыдущее исследование и настоящее), а у животных – это один род Myotis из 12 (8.3%). Отсюда следует, что глобальное похолодание было не единственным фактором, влияющим на внутриродовую динамику РГ как у растений, так и у животных, а также то, что в динамику простых компонент существенный вклад вносят скорее внутриклеточные факторы по сравнению с селективным влиянием внешней среды.
Таким образом, линейность динамики РГ внутри родов растений и животных является ее характерным признаком наряду с относительно стабильным РГ на протяжении всей эволюции рода. Такая линейность в течение многих миллионов лет предполагает менее существенную роль отбора по сравнению с видоспецифичными факторами, например, ферментной системой метаболизма ДНК, контролирующую такие функции, как репликация и рекомбинация. Это следует из того, что однонаправленное действие внешних факторов на протяжении длительного эволюционного времени маловероятно. Можно полагать, что действие отбора проявляется в полной мере при смене тенденции изменения РГ, в том числе его дивергенции, в процессе эволюции родов с многокомпонентной динамикой. По-видимому, существенное действие отбора также характерно для видов, представляющих собой выбросы из общей внутриродовой линейной тенденции.
В динамике РГ на более низком уровне таксономических категорий остается еще много неясного и накопление фактов в отношении как растений, так и животных представляет значительный интерес. В частности, это касается факторов, определяющих эволюцию РГ внутри родов.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Список литературы
Ågren J.A., Wright S.I. Co-evolution between transposable elements and their hosts: A major factor in genome size evolution? // Chromosome Res. 2011. V. 19. P. 777–786. https://doi.org/10.1007/s10577-011-9229-0
Zhao M., Ma J. Co-evolution of plant LTR-retrotransposons and their host genomes // Protein Cell. 2013. V. 4. P. 493–501. https://doi.org/10.1007/s13238-013-3037-6
Elliott T.A., Gregory T.R. Do larger genomes contain more diverse transposable elements? // BMC Evol. Biol. 2015. V. 5. P. 69.
Ahuja M.R., Neale D. Evolution of genome size in conifers // Silvae Genet. 2005. V. 54. № 3. P. 126–137.
Puchta H. The repair of double-strand breaks in plants: mechanisms and consequences for genome evolution // J. Exp. Bot. 2005. V. 56. P. 1–14.
Petrov D.A. Mutational equilibrium model of genome size evolution // Theor. Popul. Biol. 2002. V. 61. P. 531–544.
Vitte C., Panaud O. LTR retrotransposons and flowering plant genome size: Emergence of the increase/decrease model // Cytogenet. Genome Res. 2005. V. 110. P. 91–107.
Chénais B., Caruso A., Hiard S., Casse N. The impact of transposable elements on eukaryotic genomes: from genome size increase to genetic adaptation to stressful environments // Gene. 2012. V. 509. P. 7–15. https://doi.org/10.1016/j.gene.2012.07.042
Cavalier-Smith T. Skeletal DNA and the evolution of genome size // Annu. Rev. Biophys. Bioeng. 1982. V. 11. P. 273–302.
Cavalier-Smith T. Economy, speed and size matter: evolutionary forces driving nuclear genome miniaturization and expansion // Ann. Bot. 2005. V. 95. P. 147–175.
Gregory T.R. Coincidence, coevolution, or causation? DNA content, cell size, and the C-value enigma // Biol. Reviews. 2001. V. 7. P. 65–101.
Vinogradov A.E. Genome size and chromatin condensation in vertebrates // Chromosoma. 2005. V. 113. P. 362–369.
Alonso C., Pérez R., Bazaga P., Herrera C.M. Global DNA cytosine methylation as an evolving trait: Phylogenetic signal and correlated evolution with genome size in angiosperms // Front. Genet. 2015. V. 6. P. 4. https://doi.org/10.3389/fgene.2015.00004
Garnatje T., Vallès J., Garcia S. et al. Genome size in Echinops L. and related genera (Asteraceae, Cardueae): karyological, ecological and phylogenetic implications // Biol. Cell. 2004. V. 96. P. 117–124.
Grotkopp E., Rejmanek M., Sanderson M. et al. Evolution of genome size in pines (Pinus ssp.) and its life-history correlates: supertree analysis // Evolution. 2004. V. 58. № 8. P. 1705–1729.
Gregory T.R. Genome size and developmental complexity // Genetica. 2002. V. 115. № 1. P. 131–146.
Марков A.В., Анисимов В.А., Коротаев А.В. Взаимосвязь размера генома и сложности организма в эволюционном ряду от прокариот к млекопитающим // Палеонт. журн. 2010. Т. 4. С. 3–14.
Vinogradov A.E., Anatskaya O.V. Genome size and metabolic intensity in tetrapods: a tale of two lines // Proc. Biol. Sci. 2006. V. 273. P. 27–32.
Morgan H.D., Westoby M. The relationship between nuclear DNA content and leaf strategy in seed plants // Ann. Bot. 2005. V. 96. P. 1321–1330.
Шереметьев С.Н., Гамалей Ю.В., Слемнев Н.Н. Направления эволюции генома покрытосеменных // Цитология. 2011. Т. 53. № 4. С. 295–312.
Гриф В.Г. Количество ДНК на геном в биосистематике растений // Цитология. 1998. Т. 40. № 7. С. 690–705.
Ohri D. Genome size variation and plant systematics // Ann. Bot. 1998. V. 82. P. 75–83.
Бутанаев А.М., Немчинов Л.Г. Внутриродовая динамика размера генома у диплоидных семенных растений // Генетика. 2020. Т. 56. № 6. С. 667–676. https://doi.org/10.31857/S0016675820060041
Bennetzen J.L., Ma J., Devos K.M. Mechanisms of recent genome size variation in flowering plants // Ann. Bot. 2005. V. 95. P. 127–132.
Vinogradov A.E. Evolution of genome size: Multilevel selection, mutation bias or dynamical chaos? // Curr. Opin. Genet. Dev. 2004. V. 14. P. 620–626.
Pellicer J., Leitch I.J. The Plant DNA C-values database (release 7.1): An updated online repository of plant genome size data for comparative studies // New Phytol. 2020. V. 226. № 2. P. 301–305. https://doi.org/10.1111/nph.16261
Gregory T.R. Animal Genome Size Database. http://www.genomesize.com. 2020.
Willyard A., Wallace L.E., Wagner W.L. et al. Estimating the species tree for Hawaiian Schiedea (Caryophyllaceae) from multiple loci in the presence of reticulate evolution // Mol. Phylogenet. Evol. 2011. V. 60. № 1. P. 29–48. https://doi.org/10.1016/j.ympev.2011.04.001
Gurushidze M. Phylogenetic relationships and diversification processes in Allium subgenus Melanocrommyum: Dis. zur Erlangung des akademischen Grades doctorrerumnaturalium (Dr. rer. Nat.) Vorgelegt der Naturwissenschaftlichen Fakultät I – Biowissenschaften – der Martin-Luther Univ. Halle-Wittenberg. https://d-nb. info/1024874451/34.
Ammiraju J.S.S., Fan C., Yu Y. et al. Spatio-temporal patterns of genome evolution in allotetraploid species of the genus Oryza // Plant J. 2010. V. 6. № 3. P. 430–442. https://doi.org/10.1111/j.1365-313X.2010.04251.x
Brassac J., Blattner F.R. Species level phylogeny and polyploid relationships in Hordeum (Poaceae) inferred by next-generation sequencing and in silico cloning of multiple nuclear loci // Syst. Biol. 2015. V. 64. № 5. P. 792–808. https://doi.org/10.1093/sysbio/syv035
Lo E.Y.Y., Donoghue M.J. Expanded phylogenetic and dating analyses of the apples and their relatives (Pyreae, Rosaceae) // Mol. Phylogenet. Evol. 2012. V. 63. P. 230–243.
Wang B. Hybridization and evolution in the genus Pinus // Department of Ecology and Environmental Science, Umeå Univ., Umeå, Sweden. 2013. http://www.diva-portal.org/smash/get/diva2:652236/FULLTEXT01.pdf.
Lledó M.D. Molecular phylogenetics of Limonium and related genera (Plumbaginaceae): biogeographical and systematic implications // Am. J. Bot. 2005. V. 92. № 7. P. 1189–1198. https://doi.org/10.3732/ajb.92.7.1189
Stadelmann B., Jacobs D.S., Schoeman C., Ruedi M. Phylogeny of African Myotis bats (Chiroptera, Vespertilionidae) inferred from cytochrome b sequences // Acta Chiropterologica. 2004. V. 6. № 2. P. 177–192. https://doi.org/10.3161/001.006.0201
Stadelmann B., Lin L.K., Kunz T.H., Ruedi M. Molecular phylogeny of New World Myotis (Chiroptera, Vespertilionidae) inferred from mitochondrial and nuclear DNA genes // Mol. Phylogenet. Evol. 2007. V. 43. № 1. P. 32–48.
Ruedi M., Stadelmann B., Gager Y. et al. Molecular phylogenetic reconstructions identify East Asia as the cradle for the evolution of the cosmopolitan genus Myotis (Mammalia, Chiroptera) // Mol. Phylogenet. Evol. 2013. V. 69. № 3. P. 437–449.
Phillips M.J., Haouchar D., Pratt R.C. et al. Inferring kangaroo phylogeny from incongruent nuclear and mitochondrial genes // PLoS One. 2013. V. 8. № 2. P. e57745. https://doi.org/10.1371/journal.pone.0057745
Meredith R.W., Westerman M., Springer M. A phylogeny and timescale for the living genera of kangaroos and kin (Macropodiformes: Marsupialia) based on nuclear DNA sequences // Australian J. Zool. 2008. V. 56. P. 395–410.
Larsen P.A., Marchán-Rivadeneira M.R., Baker R.J. Speciation dynamics of the fruit-eating bats (genus Artibeus): With evidence of ecological divergence in Central American populations // Bat Evolution, Ecology, and Conservation / Eds Adams R., Pedersen S. N.Y.: Springer, 2013. P. 315–339. https://doi.org/10.1007/978-1-4614-7397-8_16
Nagy J., Végvári Z., Varga Z. Phylogeny, migration and life history: filling the gaps in the origin and biogeography of the Turdus thrushes // J. Ornithology. 2019. V. 160. № 2. P. 529–543.
Krajewski C., Sipiorski J.T., Anderson F.E. Complete mitochondrial genome sequences and the phylogeny of cranes (Gruiformes: Gruidae) // The Auk. 2010. V. 127. № 2. P. 440−452.
Lima N.C.B., Soares A.E.R., de Paula Almeida L.G. et al. Comparative mitogenomic analyses of Amazona parrots and Psittaciformes // Genet. Mol. Biol. 2018. V. 41. № 3. P. 593−604. https://doi.org/10.1590/1678-4685-GMB-2017-0023
Wiens J.J., Parra-Olea J., García-París M., Wake D.B. Phylogenetic history underlies elevational biodiversity patterns in tropical salamanders // Proc. Biol. Sci. 2007. V. 274. № 1612. P. 919−928.
Hedges S.B., Heinicke M.P. Molecular phylogeny and biogeography of West Indian frogs of the genus Leptodactylus (Anura, Leptodactylidae) // Mol. Phylogenet. Evol. 2007. V. 44. № 1. P. 308−314.
Kozak K.H., Weisrock D.W., Larson A. Rapid lineage accumulation in a non-adaptive radiation: phylogenetic analysis of diversification rates in eastern North American woodland salamanders (Plethodontidae: Plethodon) // Proc. R. Soc. B. 2006. № 273. P. 539–546. https://doi.org/10.1098/rspb.2005.3326
Vieites D.R., Min M.-S., Wake D.B. Rapid diversification and dispersal during periods of global warming by plethodontid salamanders // PNAS. 2007. V. 104. № 50. P. 19903–19907.
Wagner W.L., Funk V.A., Weller S.G. et al. Phylogeny and biogeography in Schiedea and Alsinidendron (Caryophyllaceae) // Hawaiian Biogeography: Evolution on a hot spot archipelago / Eds Wagner W.L., Funk V.A. 1995. P. 221–258. https://repository.si.edu/handle/10088/7620.
Willyard A., Wallace L.E., Wagner W.L. et al. Estimating the species tree for Hawaiian Schiedea (Caryophyllaceae) from multiple loci in the presence of reticulate evolution // Mol. Phylogenet. Evol. 2011. V. 60. P. 29–48.
Wagner W.L., Weller S.G., Sakai A. Monograph of Schiedea (Caryophyllaceae–Alsinoideae) // Syst. Bot. Monographs. 2005. V. 72. https://repository.si.edu/handle/10088/18515
Ravelo A.C., Andreasen D.H., Lyle M. et al. Regional climate shifts caused by gradual global cooling in the Pliocene epoch // Nature. 2004. V. 429(6989). P. 263–267. https://doi.org/10.1038/nature02567
Bartoli G., Sarnthein M., Weinelt M. et al. Final closure of Panama and the onset of northern hemisphere glaciation // Earth and Planetary Sci. Letters. 2005. V. 237. № 1–2. P. 33–44.
Zachos J., Pagani M., Sloan L. et al. Trends, rhythms, and aberrations in global climate 65 Ma to present // Science. 2001. V. 292 (5517). P. 686–693.
Дополнительные материалы отсутствуют.


