Генетика, 2021, T. 57, № 9, стр. 1039-1053
Филогенетические связи видов Азиатской России подродов Phacoxytropis и Tragacanthoxytropis рода Oxytropis на основе полиморфизма маркеров хлоропластного и ядерного геномов
А. Б. Холина 1, *, М. М. Козыренко 1, Е. В. Артюкова 1, М. Н. Колдаева 2, Д. В. Санданов 3, И. Ю. Селютина 4
1 Федеральный научный центр биоразнообразия наземной биоты Восточной Азии Дальневосточного отделения Российской академии наук
690022 Владивосток, Россия
2 Ботанический сад-институт Дальневосточного отделения Российской академии наук
690022 Владивосток, Россия
3 Институт общей и экспериментальной биологии Сибирского отделения
Российской академии наук
670047 Улан-Удэ, Россия
4 Центральный Сибирский ботанический сад Сибирского отделения
Российской академии наук
630090 Новосибирск, Россия
* E-mail: kholina@biosoil.ru
Поступила в редакцию 05.11.2020
После доработки 15.12.2020
Принята к публикации 18.01.2021
Аннотация
На основе анализа нуклеотидного полиморфизма межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хлоропластной ДНК видов Oxytropis Азиатской России (O. tragacanthoides секции Hystrix подрода Tragacanthoxytropis, O. coerulea, O. filiformis и O. mandshurica секции Janthina и O. deflexa и O. glabra секции Mesogaea подрода Phacoxytropis) установлено, что все популяции характеризуются высоким гаплотипическим разнообразием (h изменяется от 0.676 до 1.000), кроме видов секции Mesogaea (h изменяется от 0 до 0.333). Обнаружены видоспецифичные маркеры у O. tragacanthoides, O. deflexa, O. glabra и O. mandshurica, а также специфичные маркеры для секции Mesogaea. Реконструкция филогенетических связей хлоротипов видов подродов Phacoxytropis, Tragacanthoxytropis и Oxytropis показала, что виды секции Janthina с высокой достоверностью объединяются в одну кладу с видами подродов Tragacanthoxytropis и Oxytropis, однако взаимоотношения их остались неразрешенными. Анализ генеалогических связей риботипов ITS ядерной ДНК выявил общий риботип у видов O. tragacanthoides, O. coerulea, O. lanata, O. chankaensis, O. oxyphylla и O. triphylla, относящихся к трем подродам. Выявленная генетическая близость при четких морфологических различиях характерна для таксонов с общим происхождением, испытавших относительно недавнюю быструю адаптивную радиацию. Полученные данные полиморфизма маркеров ядерного и хлоропластного геномов подтверждают статус O. coerulea, O. filiformis и O. mandshurica как самостоятельных видов.
Род Oxytropis DC. (сем. Fabaceae) сформировался предположительно около 5.6 млн лет назад на границе миоцена–плиоцена [1, 2] в ходе эволюции древних видов рода Astragalus L. подрода Phaca (L.) Bunge [2, 3]. По морфологическим признакам и экологии к ним наиболее близки виды Oxytropis deflexa (Pall.) DC. и O. glabra (Lam.) DC. секции Mesogaea Bunge подрода Phacoxytropis Bunge [2–4]. Только эти два вида секции представлены во флоре Азиатской России [5]. O. deflexa – это восточносибирско-североамериканский вид, который обнаруживает значительный полиморфизм на протяжении обширного прерывистого ареала [4–6]; считается реликтом, наиболее древним видом секции [3]; занесен в региональные Красные книги, так как существует в изолированных малочисленных популяциях. O. glabra – полиморфный вид, распространенный в европейской части России (Башкирия, р. Урал), Средней и Центральной Азии, Монголии и Северо-Западном Китае [5–7].
Подрод Phacoxytropis включает также секцию Janthina Bunge, виды которой морфологически сходны с видами секции Mesogaea [5]. В Азиатской России секция Janthina представлена шестью видами: O. kaspensis Krasnob. et Pschen., O. ladyginii Kryl., O. saposhnikovii Kryl., O. coerulea (Pall.) DC., O. filiformis DC., O. mandshurica Bunge, отношения между тремя последними видами довольно сложные [5]. Малышев [5] отмечал, что O. filiformis плохо отличается от O. coerulea и интрогрессирует с ней в Предбайкалье и Забайкалье; O. filiformis и O. mandshurica – это самостоятельные виды; O. caerulea (Pall.) DC. и O. coerulea Turcz. являются синонимами O. coerulea (Pall.) DC. Авторы работы “Флора Китая” [6] для O. filiformis в качестве синонима приводят O. coerulea Turcz., а для O. caerulea (Pall.) DC. – O. mandshurica, и относят O. filiformis и O. caerulea к секции Eumorpha (Bge.) Abduss., которую вместе с секциями Mesogaea и Janthina помещают в подрод Oxytropis ex genere Oxytropis DC. В дальнейшем исследование нуклеотидного полиморфизма маркеров хлоропластного генома видов Азиатской России подродов Oxytropis и Phacoxytropis [8] показало, что подроду Phacoxytropis корреспондирует лишь секция Mesogaea, а секция Janthina этого же подрода объединяется с секциями Orobia Bunge, Verticillares DC. и Xerobia Bunge подрода Oxytropis. Кроме того, были выявлены молекулярные различия между O. coerulea и O. mandshurica, которые свидетельствуют о самостоятельности этих таксонов. Однако до сих пор существуют противоречия и другие нерешенные вопросы.
Подрод Tragacanthoxytropis Vass. является иной древней ветвью рода Oxytropis. Виды этого подрода представлены кустарничковыми формами и морфологически резко отличаются от видов других подродов [3, 9]. Одним из наиболее интересных видов подрода Tragacanthoxytropis является O. tragacanthoides Fisch. ex DC. секции Hystrix Bunge. Это горно-степной вид c прерывистым ареалом, встречается в Центральном и Юго-Восточном Алтае и Монголии, изредка отмечается в Тувинской котловине и Хакасии, где проходит северная граница ареала, а также в нескольких пунктах Прибайкалья [5, 9]. O. tragacanthoides считается реликтом миоцен-плиоценовой флоры [3, 9]; занесен в региональные Красные книги как вид уязвимый, находящийся под угрозой исчезновения, и как вид, возможно, исчезнувший.
Данная работа является продолжением популяционных исследований эндемичных видов Oxytropis [10–14] и филогенетических связей видов секций Verticillares [15], Orobia [16], Arctobia [17] и подродов рода Oxytropis [8] по данным секвенирования межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хлоропластной ДНК (хпДНК) и ITS ядерной ДНК (рДНК).
Цель исследования – изучение генетического разнообразия и популяционной структуры видов Oxytropis Азиатской России секций Mesogaea и Janthina подрода Phacoxytropis, секции Hystrix подрода Tragacanthoxytropis и реконструкция филогенетических связей видов подродов Phacoxytropis, Tragacanthoxytropis и Oxytropis по данным изменчивости нуклеотидных последовательностей межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хпДНК и ITS рДНК.
МАТЕРИАЛЫ И МЕТОДЫ
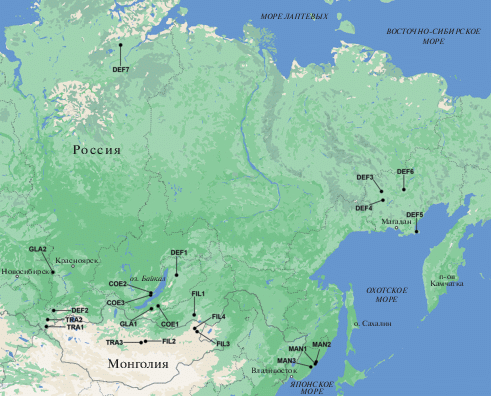
Материалом служили 124 растения, относящиеся к шести видам: O. tragacanthoides (20 образцов) секции Hystrix подрода Tragacanthoxytropis, O. coerulea (18), O. filiformis (20) и O. mandshurica (34) секции Janthina, O. deflexa (25) и O. glabra (7) секции Mesogaea подрода Phacoxytropis из 22 природных местонахождений (табл. 1, рис. 1). Названия видов, секций и подродов рода Oxytropis приведены согласно обработке Малышева [5].
Таблица 1.
Исследуемые популяции видов Oxytropis подродов Tragacanthoxytropis и Phacoxytropis и параметры генетического разнообразия по данным хпДНК
| Вид, местонахождение (число образцов) |
Координаты с.ш., в.д. |
Код | Гаплотип | Разнообразие | |
|---|---|---|---|---|---|
| h (SD) | π (SD) | ||||
| Подрод Tragacanthoxytropis | |||||
| Секция Hystrix | |||||
| O. tragacanthoides | |||||
| Республика Алтай, плато Укок, правобережье р. Жумалы, высота 2422 м (10) | 49.51°, 88.06° | TRA1 | P1–P7 | 0.867 (0.107) | 0.0048 (0.0027) |
| Республика Алтай, окр. с. Чаган-Узун, высота 1780 м (5) | 50.10°, 88.38° | TRA2 | P8–P10 | 0.700 (0.218) | 0.0008 (0.0006) |
| Монголия, Центральный аймак, окр. сомона Ундэрширээт, высота 1282 м (5) | 47.55°, 105.11° | TRA3 | P11–P13 | 0.800 (0.164) | 0.0005 (0.0004) |
| Подрод Phacoxytropis | |||||
| Секция Janthina | |||||
| O. coerulea | |||||
| Республика Бурятия, окр. с. Заиграево, высота 675 м (5) | 51.87°, 108.24° | COE1 | P14–P17 | 0.900 (0.161) | 0.0017 (0.0012) |
| Иркутская обл., окр. с. Сарма, высота 476 м (12) | 53.12°, 106.85° | COE2 | P18–P27 | 0.970 (0.044) | 0.0046 (0.0025) |
| Иркутская обл., западное побережье оз. Байкал, окр. п. Сахюрта (1) | 53.01°, 106.89° | COE3 | P28 | – | – |
| O. filiformis | |||||
| Забайкальский край, окр. оз. Ножий, высота 686 м (4) | 50.82°, 114.72° | FIL1 | P29–P32 | – | – |
| Монголия, Центральный аймак, окр. сомона Аргалант, высота 1488 м (5) | 47.77°, 105.90° | FIL2 | P29, P33–P36 |
1.000 (0.126) | 0.0048 (0.0031) |
| Монголия, Восточный аймак, окр. сомона Гурванзагал, высота 837 м (2) | 48.86°, 115.11° | FIL3 | P37, P38 | – | – |
| Монголия, Восточный аймак, окр. сомона Гурванзагал, высота 797 м (9) | 49.27°, 114.71° | FIL4 | P30, P37, P39–P41 |
0.722 (0.159) | 0.0012 (0.0008) |
| O. mandshurica | |||||
| Приморский край, Сихотэ-Алинский заповедник, бух. Удобная (15) | 44.95°, 136.55° | MAN1 | P42–P47 | 0.838 (0.061) | 0.0015 (0.0009) |
| Приморский край, Сихотэ-Алинский заповедник, кордон “Благодатное” (4) | 45.21°, 136.53° | MAN2 | P45 | – | – |
| Приморский край, окр. г. Дальнегорск, падь Барачная (15) | 44.57°, 135.62° | MAN3 | P42, P45, P48, P49 | 0.676 (0.101) | 0.0004 (0.0003) |
| Секция Mesogaea | |||||
| O. deflexa | |||||
| Республика Бурятия, Джергинский заповедник, урочище Биранкур (2) | 55.11°, 111.46° | DEF1 | P50, P51 | – | – |
| Республика Тыва, Саянский перевал (1) | 51.43°, 89.53° | DEF2 | P52 | – | – |
| Магаданская обл., окр. г. Сусуман (9) | 62.83°, 148.22° | DEF3 | P53, P54 | 0.222 (0.166) | 0.0098 (0.0054) |
| Магаданская обл., окр. с. Оротук, надпойменная терраса р. Колыма (9) | 62.10°, 148.49° | DEF4 | P54 | 0.000 (0.000) | 0.0000 (0.0000) |
| Магаданская обл., п-ов Пьягина, мыс Средний, у ручья (1) | 59.32°, 154.57° | DEF5 | P55 | – | – |
| Магаданская обл., пос. Сеймчан (1) | 62.93°, 152.39° | DEF6 | P55 | – | – |
| Восточный Таймыр, район слияния рек Большая Лесная Рассоха и Новая (2) | 72.60°, 101.26° | DEF7 | P55, P56 | – | – |
| O. glabra | |||||
| Республика Бурятия, окр. с. Оронгой, высота 528 м (6) | 51.55°, 107.03° | GLA1 | P57, P58 | 0.333 (0.215) | 0.0001 (0.0002) |
| Красноярский край, окр. с. Темра, берег оз. Гнилое (1) | 55.43°, 89.27° | GLA2 | P59 | – | – |
Рис. 1.
Карта-схема с указанием мест сбора растений видов Oxytropis tragacanthoides секции Hystrix подрода Tragacanthoxytropis, O. coerulea, O. filiformis и O. mandshurica секции Janthina, O. deflexa и O. glabra секции Mesogaea подрода Phacoxytropis из 22 природных местонахождений. Код популяции см. табл. 1.
ДНК экстрагировали из лиофильно высушенных листьев. Буфер для экстракции содержал 100 мM Трис-HCl (pH 8.0), 0.7 M NaCl, 40 мМ EDTA, 1% СTAB (hexadecyltrimethylammonium bromide) и 10 мл/л β-меркаптоэтанола. Экстракт инкубировали при 65°С в течение 40 мин. ДНК депротеинизировали смесью хлороформ : октанол (24 : 1) и осаждали равным объемом изопропанола в присутствии 0.3 M ацетата натрия. ДНК промывали 75%-ным этанолом и растворяли в буфере, содержащем 10 мМ Трис-HCl (рН 8.0) и 1 мМ EDTA. Количество ДНК в образце определяли путем сравнения с ДНК фага лямбда известной концентрации методом электрофореза в 1.4%-ном агарозном геле [10]. Методы амплификации межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG приведены в наших предыдущих работах [8, 12, 13]. ITS регион рДНК амплифицирован с праймерами ITS1 и ITS4 в реакционных условиях и температурном режиме, приведенных в работе [18]. Циклическое секвенирование обеих цепей фрагментов ДНК осуществляли с использованием набора флуоресцентно меченых нуклеотидов Big Dye Terminator v. 3.1 (Applied Biosystems, США). Нуклеотидные последовательности прямых и обратных цепей определяли на генетическом анализаторе ABI 3500 (Applied Biosystems), затем редактировали и собирали с помощью пакета программ Staden Package v. 1.5 [19]. Для каждого образца последовательности регионов выравнивали в программе SeaView v. 4.7 [20].
Матрицу объединенных последовательностей трех спейсеров хпДНК использовали для расчета гаплотипического (h) и нуклеотидного (π) разнообразия (для популяций с числом образцов пять и более), степени дивергенции (Dxy) между популяциями/видами на основе нуклеотидных замен, для анализа молекулярной дисперсии (AMOVA) и для идентификации гаплотипов с помощью пакетов программ Arlequin v. 3.5 [21] и DnaSP v. 5.0 [22]. Статистическую значимость (P) индексов фиксации (ΦST) оценивали на основе 1023 пермутаций. Филогенетический анализ проводили методами ML, MP и NJ с помощью пакета программ PAUP v. 4.0b10 [23]. Оптимальную модель эволюции нуклеотидных замен для ML-анализа выбирали в программе Modeltest v. 3.06 [24] с использованием иерархических тестов. Для ML и MP анализов применяли эвристический поиск оптимальной топологии с алгоритмом TBR (Tree Bisection-Reconnection). Статистическую достоверность порядка ветвления оценивали с помощью бутстреп-анализа 1000 альтернативных деревьев (BP, %). Кроме того, был использован баесовский подход (Bayesian Inference, BI) в программе MrBayes 3.1.6 [25] на портале CIPRES (http://www.phylo.org/) [26]. Для оценки достоверности определены апостериорные вероятности (Posterior Probabilities, РР). Значения BP < 50% и РР < 0.95 не рассматривались. В качестве внешней группы были использованы полученные нами ранее [8] нуклеотидные последовательности этих же спейсеров (LM653198, LM653161, LM653235) A. davuricus (Pall.) DC. Генеалогические связи риботипов ITS рДНК анализировали методом медианного связывания (MJ) в программе Network v. 5.0.1.1 [27], кодируя каждую делецию/вставку, независимо от ее размера, как единичное мутационное событие. В качестве внешней группы была использована полученная нами ранее последовательность ITS (LM653272) A. davuricus.
Работа проводилась с использованием оборудования ЦКП “Биотехнология и генетическая инженерия” ФНЦ Биоразнообразия ДВО РАН.
РЕЗУЛЬТАТЫ
Нуклеотидные последовательности каждого из регионов psbA–trnH, trnL–trnF и trnS–trnG хпДНК у 124 образцов исследованных видов характеризуются относительно низкой нуклеотидной изменчивостью и разной длиной вследствие присутствия коротких (1–8 пн) и длинных (52–168 пн) инсерций/делеций (инделей), моно- и динуклеотидных повторов. Длина объединенной матрицы трех регионов после выравнивания составила 2769 сайтов. Обнаружено 42 вариабельные нуклеотидные замены, из них 38 были информативны согласно методу максимальной экономии и 4 единичные. Выявлено 59 хлоротипов (P1–P59), из них 40 (67.8%) уникальные, общих хлоротипов у видов не обнаружено (табл. 1). Последовательности хлоротипов депонированы в GenBank/ENA/EMBL-EBI, номера доступа приведены в табл. 2. У всех изученных видов, кроме O. coerulea и O. filiformis, обнаружены видоспецифичные маркеры. Так, последовательности O. mandshurica и O. tragacanthoides имеют нуклеотид A в позициях 862 и 1658 соответственно. У O. deflexa выявлены четыре нуклеотидные замены (T в позициях 332 и 1406, C в позиции 2105, G в позиции 1410) и вставка 97–110 пн (позиции 684–793); у O. glabra – шесть нуклеотидных замен (G в позициях 261, 332, 1935, 2099, 2597 и T в позиции 2346) и делеция нуклеотида А в позиции 861. Кроме того, обнаружены маркерные нуклеотидные замены, специфичные для секции Mesogaea: все последовательности представителей этой секции имеют шесть замен (G в позиции 136, T в позиции 618, С в позициях 650 и 1087, A в позициях 1393 и 1394) и шесть инделей длиной от 1 до 6 пн, отсутствующих у других. Популяции O. tragacanthoides подрода Tragacanthoxytropis и видов O. coerulea, O. filiformis и O. mandshurica секции Janthina подрода Phacoxytropis характеризуются высоким гаплотипическим и низким/средним нуклеотидным разнообразием, а популяция O. glabra – низким гаплотипическим и нуклеотидным разнообразием. В популяции O. deflexa DEF3 при низком гаплотипическом разнообразии выявлен достаточно высокий уровень нуклеотидного разнообразия, а популяция DEF4 оказалась мономорфной (табл. 1).
Таблица 2.
Хлоротипы видов Oxytropis и номера доступа в GenBank/ENA/EMBL-EBI нуклеотидных последовательностей межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хпДНК
| Хлоротип | Номер доступа | ||
|---|---|---|---|
| psbA–trnH | trnL–trnF | trnS–trnG | |
| O. tragacanthoides | |||
| P1 | MW172222 | MW177548 | MW177535 |
| P2 | MW172223 | MW177549 | MW177536 |
| P3 | MW172224 | MW177550 | MW177537 |
| P4 | MW172225 | MW177551 | MW177538 |
| P5 | MW172226 | MW177552 | MW177539 |
| P6 | MW172227 | MW177553 | MW177540 |
| P7 | MW172228 | MW177554 | MW177541 |
| P8 | MW172229 | MW177555 | MW177542 |
| P9 | MW172230 | MW177556 | MW177543 |
| P10 | MW172231 | MW177557 | MW177544 |
| P11 | MW172232 | MW177558 | MW177545 |
| P12 | MW172233 | MW177559 | MW177546 |
| P13 | MW172234 | MW177560 | MW177547 |
| O. coerulea | |||
| P14 | LR898256 | LR898302 | LR898413 |
| P15 | LR898257 | LR898303 | LR898414 |
| P16 | LR898258 | LR898304 | LR898415 |
| P17 | LR898259 | LR898305 | LR898416 |
| P18 | LR898260 | LR898306 | LR898417 |
| P19 | LR898261 | LR898307 | LR898418 |
| P20 | LR898262 | LR898308 | LR898419 |
| P21 | LR898263 | LR898309 | LR898420 |
| P22 | LR898264 | LR898310 | LR898421 |
| P23 | LR898265 | LR898311 | LR898422 |
| P24 | LR898266 | LR898312 | LR898423 |
| P25 | LR898267 | LR898313 | LR898424 |
| P26 | LR898268 | LR898314 | LR898425 |
| P27 | LR898269 | LR898315 | LR898426 |
| P28 | LR898270 | LR898316 | LR898427 |
| O. filiformis | |||
| P29 | LR898271 | LR898317 | LR898428 |
| P30 | LR898272 | LR898318 | LR898429 |
| P31 | LR898273 | LR898319 | LR898430 |
| P32 | LR898274 | LR898320 | LR898431 |
| P33 | LR898275 | LR898321 | LR898432 |
| P34 | LR898276 | LR898322 | LR898433 |
| P35 | LR898277 | LR898323 | LR898434 |
| P36 | LR898278 | LR898324 | LR898435 |
| P37 | LR898278 | LR898325 | LR898436 |
| P38 | LR898280 | LR898326 | LR898437 |
| P39 | LR898281 | LR898327 | LR898438 |
| P40 | LR898282 | LR898328 | LR898439 |
| P41 | LR898283 | LR898329 | LR898440 |
| O. mandshurica | |||
| P42 | LR898284 | LR898330 | LR898441 |
| P43 | LR898285 | LR898331 | LR898442 |
| P44 | LR898286 | LR898332 | LR898443 |
| P45 | LR898287 | LR898333 | LR898444 |
| P46 | LR898288 | LR898334 | LR898445 |
| P47 | LR898289 | LR898335 | LR898446 |
| P48 | LR898290 | LR898336 | LR898447 |
| P49 | LR898291 | LR898337 | LR898448 |
| O. deflexa | |||
| P50 | LR898292 | LR898338 | LR898449 |
| P51 | LR898293 | LR898339 | LR898450 |
| P52 | LR898294 | LR898340 | LR898451 |
| P53 | LR898295 | LR898341 | LR898452 |
| P54 | LR898296 | LR898342 | LR898453 |
| P55 | LR898297 | LR898343 | LR898454 |
| P56 | LR898298 | LR898344 | LR898455 |
| O. glabra | |||
| P57 | LR898299 | LR898345 | LR898456 |
| P58 | LR898300 | LR898346 | LR898457 |
| P59 | LR898301 | LR898347 | LR898458 |
| O. triphylla* | |||
| H1 | LT856461 | LT856494 | LT856527 |
| H2 | LT856462 | LT856495 | LT856528 |
| H3 | LT856463 | LT856496 | LT856529 |
| H4 | LT856464 | LT856497 | LT856530 |
| H5 | LT856465 | LT856498 | LT856531 |
| H6 | LT856466 | LT856499 | LT856532 |
| H7 | LT856467 | LT856500 | LT856533 |
| H10 | LT856472 | LT856505 | LT856538 |
| O. lanata** | |||
| V1 | LT994841 | LT994895 | LT994949 |
| V3 | LT994843 | LT994897 | LT994951 |
| V4 | LT994844 | LT994898 | LT994952 |
| V5 | LT994845 | LT994899 | LT994953 |
| V7 | LT994847 | LT994901 | LT994955 |
| V9 | LT994849 | LT994903 | LT994957 |
| V13 | LT994853 | LT994907 | LT994961 |
| V14 | LT994854 | LT994908 | LT994962 |
| V16 | LT994856 | LT994910 | LT994964 |
| V18 | LT994858 | LT994912 | LT994966 |
| O. gracillima** | |||
| V58 | MH174938 | LT996062 | LT996067 |
| V59 | LT996058 | LT996063 | LT996068 |
| V60 | LT996059 | LT996064 | LT996069 |
| V61 | LT996060 | LT996065 | LT996070 |
| V62 | LT996061 | LT996066 | LT996071 |
| O. sordida*** | |||
| H1 | LS991870 | LS991896 | LS991922 |
| H2 | LS991871 | LS991897 | LS991923 |
| H3 | LS991872 | LS991898 | LS991924 |
| H4 | LS991873 | LS991899 | LS991925 |
| H5 | LS991874 | LS991900 | LS991926 |
| O. ochotensis**** | |||
| H1 | MK806162 | MK806201 | MK806240 |
| H2 | MK806163 | MK806202 | MK806241 |
| H3 | MK806164 | MK806203 | MK806242 |
| H4 | MK806165 | MK806204 | MK806243 |
| H5 | MK806166 | MK806205 | MK806244 |
| H7 | MK806168 | MK806207 | MK806246 |
| H9 | MK806170 | MK806209 | MK806248 |
| H11 | MK806172 | MK806211 | MK806250 |
| H12 | MK806173 | MK806212 | MK806251 |
| H14 | MK806175 | MK806214 | MK806253 |
Согласно результатам AMOVA (табл. 3), самые большие и значимые межпопуляционные различия у O. deflexa и у O. tragacanthoides (ΦST = 0.81602 и ΦST = 0.75975 соответственно, P < 0.0001), около 35% от общей генетической изменчивости приходится на межпопуляционную компоненту у O. coerulea и у O. mandshurica (ΦST = 0.34055, P < 0.05 и ΦST = 0.33889, P < 0.0001 соответственно). Статистически незначимая высокая дифференциация определена между популяциями O. glabra (ΦST = 0.71429, P > 0.05), а у O. filiformis дифференциация между популяциями отсутствует (ΦST = = –0.08489, P > 0.05). Другим показателем степени генетической разобщенности между популяциями является дивергенция нуклеотидных последовательностей (Dxy). У O. tragacanthoides межпопуляционные значения Dxy (среднее число нуклеотидных замен на один сайт и среднее число нуклеотидных различий (число фиксированных различий)) изменяются от 0.00142 до 0.00226 и от 3.400 (3) до 5.400 (2) соответственно. Наименьшие значения Dxy определены между популяциями у O. mandshurica и у O. filiformis (табл. 4), наибольшие – между популяциями O. deflexa, между двумя популяциями O. glabra дивергенция отсутствует (табл. 5).
Таблица 3.
Распределение генетической изменчивости (AMOVA) между группами Oxytropis
| Источник дисперсии | Генетические различия (%) между | ||
|---|---|---|---|
| группами | популяциями внутри групп | особями в популяции | |
| Популяции видов Oxytropis | |||
| Одна группа: (популяции O. tragacanthoides) | – | 75.97* | 24.03 |
| Одна группа: (популяции O. coerulea) | – | 34.05** | 65.95 |
| Одна группа: (популяции O. filiformis) | – | –8.49 ns | 108.49 |
| Одна группа: (популяции O. mandshurica) | – | 33.89* | 66.11 |
| Одна группа: (популяции O. deflexa) | – | 81.60* | 18.4 |
| Одна группа: (популяции O. glabra) | – | 71.43 ns | 28.57 |
| Две группы: (популяции O. coerulea) и (популяции O. filiformis) | 19.72 ns | 16.51** | 32.98* |
| Три группы: (популяции O. coerulea), (популяции O. filiformis) и (популяции O. mandshurica) | 57.41* | 10.03** | 32.56* |
| Две группы: (популяции O. deflexa) и (популяции O. glabra) | 74.74** | 21.17* | 4.09* |
| Две группы: (популяции видов секции Janthina) и (популяции видов секции Mesogaea) | 89.75* | 8.91* | 1.33* |
| Две группы: (популяции O. tragacanthoides) и (популяции видов секции Janthina) | 32.23** | 44.68* | 23.09* |
| Две группы: (популяции O. tragacanthoides) и (популяции видов секции Mesogaea) | 79.94** | 18.32* | 1.74* |
Таблица 4.
Нуклеотидная дивергенция между популяциями видов Oxytropis coerulea, O. filiformis и O. mandshurica секции Janthina подрода Phacoxytropis
| Популяция | COE1 | COE2 | COE3 | FIL1 | FIL2 | FIL3 | FIL4 | MAN1 | MAN2 | MAN3 |
|---|---|---|---|---|---|---|---|---|---|---|
| COE1 | – | 3.917 (2) | 4.000 (4) | 0.000 (0) | 0.200 (0) | 0.000 (0) | 0.000 (0) | 4.067 (4) | 4.000 (4) | 4.000 (4) |
| COE2 | 0.00164 | – | 4.250 (2) | 3.917 (2) | 4.117 (2) | 3.917 (2) | 3.917 (2) | 6.317 (4) | 6.250 (4) | 6.250 (4) |
| COE3 | 0.00168 | 0.00178 | – | 4.000 (4) | 4.200 (4) | 4.000 (4) | 4.000 (4) | 4.067 (4) | 4.000 (4) | 4.000 (4) |
| FIL1 | 0.00000 | 0.00164 | 0.00168 | – | 0.200 (0) | 0.000 (0) | 0.000 (0) | 4.067 (4) | 4.000 (4) | 4.000 (4) |
| FIL2 | 0.00008 | 0.00173 | 0.00176 | 0.00008 | – | 0.200 (0) | 0.200 (0) | 4.267 (4) | 4.200 (4) | 4.200 (4) |
| FIL3 | 0.00000 | 0.00164 | 0.00168 | 0.00000 | 0.00008 | – | 0.000 (0) | 4.067 (4) | 4.000 (4) | 4.000 (4) |
| FIL4 | 0.00000 | 0.00164 | 0.00168 | 0.00000 | 0.00008 | 0.00000 | – | 4.067 (4) | 4.000 (4) | 4.000 (4) |
| MAN1 | 0.00170 | 0.00265 | 0.00170 | 0.00170 | 0.00179 | 0.00170 | 0.00170 | – | 0.067 (0) | 0.067 (0) |
| MAN2 | 0.00167 | 0.00262 | 0.00167 | 0.00167 | 0.00176 | 0.00167 | 0.00167 | 0.00003 | – | 0.000 (0) |
| MAN3 | 0.00167 | 0.00262 | 0.00167 | 0.00167 | 0.00176 | 0.00167 | 0.00167 | 0.00003 | 0.00000 | – |
Примечание. Выше диагонали – среднее число нуклеотидных различий (число фиксированных различий), ниже диагонали – среднее число нуклеотидных замен на один сайт. Код популяции см. табл. 1.
Таблица 5.
Нуклеотидная дивергенция между популяциями видов Oxytropis deflexa и O. glabra секции Mesogaea подрода Phacoxytropis
| Популяция | DEF1 | DEF2 | DEF3 | DEF4 | DEF5 | DEF6 | DEF7 | GLA1 | GLA2 |
|---|---|---|---|---|---|---|---|---|---|
| DEF1 | – | 4.000 (2) | 4.000 (2) | 4.000 (2) | 6.000 (4) | 6.000 (4) | 6.000 (4) | 15.000 (13) | 15.000 (13) |
| DEF2 | 0.00175 | – | 0.000 (0) | 0.000 (0) | 3.000 (3) | 3.000 (3) | 3.000 (3) | 14.000 (14) | 14.000 (14) |
| DEF3 | 0.00167 | 0.00000 | – | 0.000 (0) | 3.000 (3) | 3.000 (3) | 3.000 (3) | 16.000 (16) | 16.000 (16) |
| DEF4 | 0.00167 | 0.00000 | 0.00000 | – | 3.000 (3) | 3.000 (3) | 3.000 (3) | 16.000 (16) | 16.000 (16) |
| DEF5 | 0.00263 | 0.00131 | 0.00131 | 0.00131 | – | 0.000 (0) | 0.000 (0) | 12.000 (12) | 12.000 (12) |
| DEF6 | 0.00263 | 0.00131 | 0.00131 | 0.00131 | 0.00000 | – | 0.000 (0) | 12.000 (12) | 12.000 (12) |
| DEF7 | 0.00263 | 0.00131 | 0.00131 | 0.00131 | 0.00000 | 0.00000 | – | 12.000 (12) | 12.000 (12) |
| GLA1 | 0.00661 | 0.00639 | 0.00705 | 0.00705 | 0.00547 | 0.00547 | 0.00547 | – | 0.000 (0) |
| GLA2 | 0.00661 | 0.00638 | 0.00705 | 0.00705 | 0.00547 | 0.00547 | 0.00547 | 0.00000 | – |
Примечание. См. примечание к табл. 4.
В табл. 6 приведены значения Dxy между видами. Низкая дивергенция определена между O. coerulea и O. filiformis, значение которой ниже таковых между популяциями O. coerulea (табл. 4). Дивергенция между O. filiformis и O. mandshurica соответствует межпопуляционным значениям, а дивергенция O. coerulea от O. mandshurica в ≈ 1.4 раза превышает межпопуляционную. Нуклеотидная дивергенция O. tragacanthoides от видов секций Janthina в ≈ 1.8 раз выше межпопуляционных значений, а от видов секции Mesogaea в ≈ 5 раз выше (табл. 6). По данным иерархического AMOVA (табл. 3), дифференциация между O. coerulea и O. filiformis низка и незначима (ΦCT = 0.19727, P > 0.075), а дифференциация между тремя видами секций Janthina составляет более 57% (ΦCT = 0.57412, P < 0.0001). Наибольшие различия выявлены между секциями Mesogaea и Janthina (ΦCT = 0.89754, P < 0.0001), чуть меньшие – между O. tragacanthoides и секцией Mesogaea (ΦCT = 0.79942, P < 0.004), а генетические различия между O. tragacanthoides и секцией Janthina оказались ниже популяционных значений (ΦCT = 0.32227, P < 0.009).
Таблица 6.
Нуклеотидная дивергенция между видами Oxytropis подродов Tragacanthoxytropis и Phacoxytropis
| Вид | O. tragacanthoides | O. coerulea | O. filiformis | O. mandshurica | O. deflexa | O. glabra |
|---|---|---|---|---|---|---|
| O. tragacanthoides | – | 7.939 (2) | 7.600 (5) | 7.579 (5) | 22.910 (14) | 22.800 (20) |
| O. coerulea | 0.00334 | – | 2.883 (0) | 5.529 (2) | 24.829 (16) | 24.389 (21) |
| O. filiformis | 0.00320 | 0.00121 | – | 4.079 (4) | 23.490 (18) | 23.050 (23) |
| O. mandshurica | 0.00318 | 0.00232 | 0.00171 | – | 21.549 (17) | 22.029 (22) |
| O. deflexa | 0.01097 | 0.01193 | 0.01128 | 0.01034 | – | 13.600 (10) |
| O. glabra | 0.01031 | 0.01106 | 0.01044 | 0.00994 | 0.00620 | – |
Примечание. См. примечание к табл. 4.
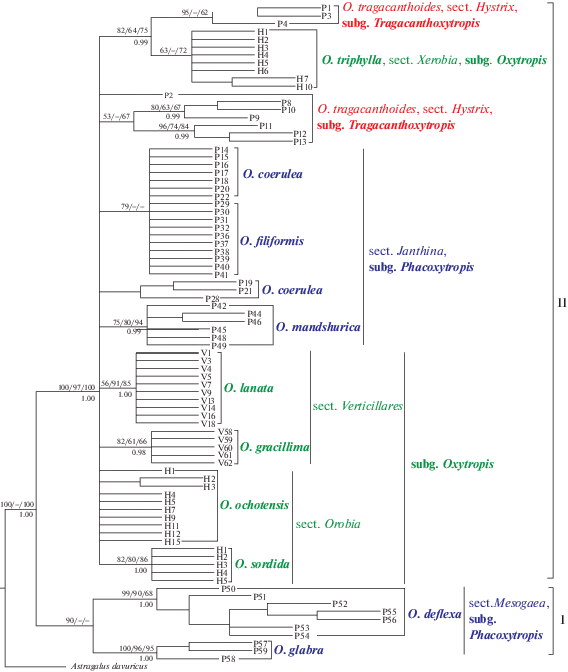
Для реконструкции филогенетических связей в матрицу, включающую 46 из 59 выявленных хлоротипов видов двух подродов (некоторые уникальные хлоротипы были исключены в связи с большой представленностью), добавлены полученные нами ранее последовательности хлоротипов пяти видов подрода Oxytropis: O. triphylla (Pall.) Pers. (8 хлоротипов) секции Xerobia Bunge, O. lanata (Pall.) DC. (10) и O. gracillima Bunge (5) секции Verticillares DC., O. sordida (Willd.) Pers. (5) и O. ochotensis Bunge (10) секции Orobia Bunge (табл. 2). Длина объединенной матрицы 84 последовательностей хлоротипов трех регионов после выравнивания составила 2790 сайтов. Обнаружено 48 вариабельных нуклеотидных замен, из них 43 были информативны согласно методу максимальной экономии и 5 единичные. Общих хлоротипов у видов не обнаружено. Филогенетический анализ методами MP, NJ, ML дал деревья сходной топологии. На рис. 2 представлено MP-дерево (консенсус 10 000 деревьев: длина 413 шагов, CI = = 0.5617, HI = 0.4383, RI = 0.8002), в котором хлоротипы видов Oxytropis формируют две сестринские клады. Клада I с высокой степенью поддержки только в MP-анализе объединяет хлоротипы O. deflexa и O. glabra секции Mesogaea подрода Phacoxytropis, при этом хлоротипы каждого из видов формируют высокоподдержанные группы (рис. 2). Клада II с высокой степенью поддержки во всех анализах объединяет хлоротипы видов подродов Tragacanthoxytropis, Oxytropis и секции Janthina подрода Phacoxytropis, но взаимоотношения между ними остались неразрешенными. Хорошо поддержанные группы образуют хлоротипы видов O. mandshurica, O. sordida, O. gracillima, O. lanata. Следует отметить, что три хлоротипа O. tragacanthoides (P1, P3 и P4) подрода Tragacanthoxytropis объединились в одну группу со средней поддержкой в MP, ML, NJ и высокой в BI анализах с хлоротипами O. triphylla секции Xerobia подрода Oxytropis (рис. 2).
Рис. 2.
MP-дерево филогенетических связей хлоротипов хпДНК видов Oxytropis подродов Tragacanthoxytropis, Phacoxytropis и Oxytropis. Числами над ветвью обозначены значения индекса бутстрепа, рассчитанные для MP/NJ/ML методов (>50%), под ветвью – значения апостериорной вероятности, рассчитанные для BI анализа (>0.95). Названия видов, секций и подродов рода Oxytropis приведены согласно обработке Малышева [5].
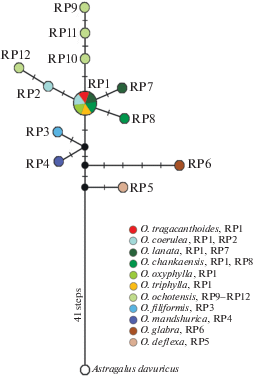
Регион ITS рДНК отсеквенирован у 49 образцов: O. tragacanthoides (13), O. coerulea (11), O. filiformis (7), O. mandshurica (9), O. deflexa (6) и O. glabra (3), представляющих большинство выявленных в данной работе хлоротипов хпДНК. ITS регион характеризуется одинаковой длиной (603 пн) и низкой нуклеотидной изменчивостью: 592 сайта были мономорфными и 11 вариабельными и информативными согласно методу максимальной экономии. Шесть замен (позиции 57, 68, 90, 122, 200, 201) обнаружено в спейсере ITS1 и пять (позиции 422, 427, 513, 548, 555) – в ITS2. Выявлено шесть риботипов: O. coerulea принадлежат два риботипа (RP1 и RP2), у всех других видов по одному: O. filiformis – RP3, O. mandshurica – RP4, O. deflexa – RP5, O. glabra – RP6, O. tragacanthoides – RP1, который является общим с O. coerulea. Последовательности риботипов видов депонированы в GenBank/ENA/ EMBL-EBI под номерами доступа MW186811, LR898459–LR898464. Для выявления генеалогических связей в матрицу риботипов видов двух подродов были добавлены полученные нами ранее последовательности ITS видов подрода Oxytropis: O. triphylla (MW015143) секции Xerobia, O. lanata (LM653259, LM653260), O. chankaensis Jurtz. (FR839001, FR839010) и O. oxyphylla (Pall.) DC. (FR839000) секции Verticillares и O. ochotensis (MK795939, MK795941–MK795943) секции Orobia, а также A. davuricus в качестве внешней группы. Построенная медианная сеть представлена на рис. 3. Наиболее близким видом к Astragalus является O. deflexa, риботип которого через 42 мутационных шага и гипотетический риботип (вымерший или не выявленный в данном исследовании) связан с риботипом A. davuricus. Пять мутационных шагов и гипотетический риботип разделяют виды O. deflexa и O. glabra секции Mesogaea подрода Phacoxytropis. К ним близки риботипы O. mandshurica и O. filiformis секции Janthina этого же подрода. Наиболее распространенный и общий для шести видов, относящихся к трем подродам, риботип RP1 занимает центральное положение в сети и образует звездчатую структуру с другими риботипами, связанными одномутационными переходами (рис. 3).
Рис. 3.
Генеалогическая сеть риботипов ITS рДНК видов Oxytropis подродов Tragacanthoxytropis (RP1), Phacoxytropis (RP1–RP6) и Oxytropis (RP1, RP7–RP12), построенная с помощью MJ-метода. Размер окружностей отражает частоту встречаемости риботипов, маленькие черные кружки – гипотетические гаплотипы, поперечные тонкие штрихи на ветвях – мутационные события.
ОБСУЖДЕНИЕ
Генетическое разнообразие исследованных популяций видов O. tragacanthoides подрода Tragacanthoxytropis и O. coerulea, O. filiformis и O. mandshurica секции Janthina подрода Phacoxytropis характеризуется сочетанием высоких значений гаплотипического и низких/средних значений нуклеотидного разнообразия (табл. 1), которое было отмечено для ряда эндемичных видов Oxytopis [12], некоторых популяций O. glandulosa [13] и O. ruthenica [16], а также других представителей сем. Fabaceae – Astragalus onobrychis L. [28], видов рода Sophora L. [29]. Это указывает на быстрый рост популяции после падения численности, сопровождающийся восстановлением гаплотипической изменчивости за счет мутационного процесса [28, 30]. Низкое гаплотипическое разнообразие в популяциях видов O. deflexa и O. glabra секции Mesogaea подрода Phacoxytropis (табл. 1) может быть следствием дрейфа генов в изолированных малочисленных популяциях и влияния отбора в суровых условиях существования. Высокий уровень межпопуляционной дифференциации у O. deflexa (табл. 3) объясняется, в первую очередь, значительной географической удаленностью местонахождений (Южная Сибирь, Магаданская обл., Таймыр, рис. 1). Отсутствие нуклеотидной дивергенции между удаленными популяциями O. glabra (табл. 5) может быть проявлением анцестрального полиморфизма широко распространенного вида. У O. deflexa и O. glabra выявлены видоспецифичные маркерные нуклеотидные замены хпДНК, а также маркерные замены для секции Mesogaea. Виды секции Mesogaea значительно дивергированы от видов секции Janthina этого же подрода и O. tragacanthoides подрода Tragacanthoxytropis (табл. 6). Базальная позиция риботипов ITS рДНК O. deflexa и O. glabra в генеалогической сети (рис. 3) свидетельствует об их более древнем происхождении. Обособленное положение было показано ранее для O. deflexa [31] по данным полиморфизма маркеров TRPT ядерного и matK хлоропластного геномов, а также для O. deflexa и O. glabra по изменчивости ITS рДНК [32]. Кроме этого, изучение полиморфизма межгенного спейсера trnL–trnF и интрона trnL хпДНК видов Oxytropis, обитающих в Турции, показало, что O. kotschyana секции Mesogaea значительно дивергирована как от видов секции Janthina, так и от видов других подродов [33].
В данном исследовании секция Janthina представлена тремя видами. У O. filiformis отсутствует межпопуляционная дифференциация (табл. 3) и выявлен очень низкий уровень нуклеотидной дивергенции (табл. 4), что обусловлено, вероятно, активным обменом генами между близко расположенными популяциями. У O. coerulea популяции значительно дивергированы друг от друга (табл. 4), при этом такой же уровень нуклеотидной дивергенции выявлен между популяциями O. coerulea и O. filiformis, а между популяцией СОЕ1 O. coerulea, расположенной восточнее оз. Байкал (Бурятия), и популяциями FIL1, FIL3 и FIL4 O. filiformis из Забайкалья и Монголии нуклеотидная дивергенция отсутствует (табл. 4). Результаты анализа полиморфизма межгенных спейсеров хпДНК (отсутствие видоспецифичных маркеров хпДНК и значимой генетической дифференциации у O. coerulea и O. filiformis и объединение хлоротипов в одну, хотя и слабо поддержанную, филогруппу (рис. 2)), указывают на генетическую близость этих двух видов, тем не менее отсутствие общих хлоротипов и наличие разных риботипов ITS рДНК подтверждают самостоятельность O. coerulea и O. filiformis. У O. mandshurica выявлен видоспецифичный маркер в спейсере trnL–trnF хпДНК, хлоротипы образуют высокоподдержанную филогруппу (рис. 2), отмечен индивидуальный риботип. Эти результаты, а также высокий уровень дифференциации между видами секции Janthina (табл. 3), все в целом подтверждает статус O. mandshurica как самостоятельного вида.
Между популяциями O. tragacanthoides выявлены значительная нуклеотидная дивергенция и высокая межпопуляционная дифференциация, что может быть связано с изоляцией местонахождений и приуроченностью вида к специфическим экотопам, а также с наличием кариологических рас. Так, у O. tragacanthoides обнаружены как диплоидные (2n = 16), так и тетраплоидные (2n = 32) представители [5, 9, 34]. Выявленные хлоротипы O. tragacanthoides в MP-дереве разделились на две группы, в одной из них со средней в MP, ML, NJ и высокой в BI анализах поддержками (рис. 2) три хлоротипа объединены с хлоротипами O. triphylla секции Xerobia подрода Oxytropis, который является диплоидом с 2n = 16 [35]. Кроме этого, эти виды имеют один общий риботип ITS (рис. 3). Однако морфологически O. tragacanthoides и O. triphylla резко отличаются как друг от друга, так и от других видов Oxytropis. Представители подрода Tragacanthoxytropis – это подушковидные колючие кустарнички, а характерной особенностью O. triphylla являются листья с одной, редко двумя парами листочков, в целом по морфологическим признакам вид выделяют в особый олиготипный ряд Triphyllae внутри секции Xerobia [5]. Можно предположить, что генетическая близость этих реликтовых видов, обитающих в Южной Сибири, обусловлена широким распространением предковых форм Oxytropis на данной территории [9].
Филогенетические связи хлоротипов видов Oxytropis Азиатской России подродов Tragacanthoxytropis, Oxytropis и секции Janthina подрода Phacoxytropis, образующих высокоподдержанную кладу II (рис. 2), остались неразрешенными. Кроме того, анализ генеалогических связей риботипов ITS рДНК выявил у видов O. tragacanthoides, O. coerulea, O. lanata, O. chankaensis, O. oxyphylla и O. triphylla, относящихся к этим трем подродам, общий риботип RP1 (рис. 3). Все это может быть проявлением сетчатой эволюции, отмеченной для видов рода Oxytropis [5]. Выявленная генетическая близость при четких морфологических различиях характерна для таксонов с общим происхождением, испытавших относительно недавнюю быструю адаптивную радиацию, что отмечено для ряда родов сем. Fabaceae [1]. Учитывая обособление группы бобовых, в которую входят Astragalus и Oxytropis, около 39 млн лет назад [36], можно сказать, что дивергенция рода Oxytropis, появившегося на границе миоцена–плиоцена около 5.6 млн лет назад, была относительно недавней. Быстрая адаптивная радиация показана для видов Oxytropis [1] и других родов сем. Fabaceae: Pultenaea [37], Astragalus [38], Sophora [29] и др. Определенный вклад в полученную картину взаимосвязей видов Oxytropis трех подродов может вносить и происходившая на ранних этапах эволюции рода гибридизация между не полностью специализированными таксонами после быстрой радиации, но перед расхождением генеалогических линий, как это было отмечено для видов рода Pultenaea [37].
Таким образом, проведенный анализ нуклеотидного полиморфизма межгенных спейсеров psbA‒trnH, trnL–trnF и trnS–trnG хпДНК и ITS рДНК расширенной выборки образцов из разных популяций видов O. coerulea, O. filiformis и O. mandshurica секции Janthina подрода Phacoxytropis подтвердил, что три этих таксона являются самостоятельными видами. Результаты реконструкции филогенетических связей хлоротипов и анализа генеалогических связей риботипов ITS видов подродов Phacoxytropis, Tragacanthoxytropis и Oxytropis согласуются с мнением Zhu с соавт. [6] о принадлежности видов O. coerulea, O. filiformis и O. mandshurica подроду Oxytropis, однако необходимы дальнейшие исследования с привлечением других видов этой секции.
Авторы выражают благодарность И.Н. Поспелову и Е.Б. Поспеловой за предоставление образцов O. deflexa с п-ова Таймыр.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Shavvon R.S., Kazempour-Osaloo S., Maassoumi A.A. et al. Increasing phylogenetic support for explosively radiating taxa: The promise of high-throughput sequencing for Oxytropis (Fabaceae) // J. Syst. Evol. 2017. V. 55. № 4. P. 385–404. https://doi.org/10.1111/jse.12269
Положий А.В. К вопросу о происхождении и эволюции рода Oxytropis (Fabaceae) // Бот. журн. 2003. Т. 88. № 10. С. 55–59.
Положий А.В. Флорогенетический анализ остролодочников Средней Сибири // Уч. зап. Томского гос. ун-та. Биология и почвоведение. 1965. № 51. С. 18–38.
Юрцев Б.А. Oxytropis DC. // Арктическая флора СССР. Л.: Наука, 1986. Вып. 9. Ч. 2. С. 61–146.
Малышев Л.И. Разнообразие рода Остролодка (Oxytropis) в Азиатской России // Turczaninowia. 2008. Т. 11. № 4. С. 5–141.
Zhu X., Welsh S.L., Ohashi H. Oxytropis // Flora of China. 2010. V. 10. P. 453–500. http://www.efloras.org.
Положий А.В. Oxytropis DC. – Остролодочник // Флора Сибири. Новосибирск: ВО “Наука”, 1994. Т. 9. С. 74–151.
Холина А.Б., Козыренко М.М., Артюкова Е.В. и др. Филогенетические взаимоотношения видов Oxytropis DC. subg. Oxytropis и Phacoxytropis (Fabaceae) Азиатской России на основе анализа нуклеотидных последовательностей межгенных спейсеров хлоропластного генома // Генетика. 2016. Т. 52. № 8. С. 895–909.
Пешкова Г.А. Флорогенетический анализ степной флоры гор Южной Сибири. Новосибирск, 2001. 192 с.
Артюкова Е.В., Холина А.Б., Козыренко М.М., Журавлев Ю.Н. Анализ генетической изменчивости редкого эндемичного вида Oxytropis chankaensis Jurtz. (Fabaceae) на основе RAPD маркеров // Генетика. 2004. Т. 40. № 7. С. 877–884.
Artyukova E.V., Kozyrenko M.M., Kholina A.B., Zhuravlev Yu.N. High chloroplast haplotype diversity in the endemic legume Oxytropis chankaensis may result from independent polyploidization events // Genetica. 2011. V. 139. № 2. P. 221–232. https://doi.org/10.1007/s10709-010-9539-8
Холина А.Б., Козыренко М.М., Артюкова Е.В., Санданов Д.В. Современное состояние популяций эндемичных видов Oxytropis Байкальской Сибири и их филогенетические связи по данным секвенирования маркеров хлоропластной ДНК // Генетика. 2018. Т. 54. № 7. С. 795–806.
Kholina A., Kozyrenko M., Artyukova E. et al. Plastid DNA variation of the endemic species Oxytropis glandulosa Turcz. (Fabaceae) // Turk. J. Bot. 2018. V. 42. P. 38–50. https://doi.org/10.3906/bot-1706-11
Холина А.Б., Козыренко М.М., Артюкова Е.В. и др. Генетическое разнообразие и филогенетические связи Oxytropis evenorum (Fabaceae) по данным секвенирования межгенных спейсеров хлоропластной ДНК // Вестник СВНЦ ДВО РАН. 2019. № 2. С. 117–125. https://doi.org/10.1134/S0016675819060055
Холина А.Б., Козыренко М.М., Артюкова Е.В., Санданов Д.В. Дивергенция видов Oxytropis секции Verticillares (Fabaceae) степной флоры Байкальской Сибири на основе анализа хлоропластной ДНК // Генетика. 2019. Т. 55. № 6. С. 665–674.
Козыренко М.М., Холина А.Б., Артюкова Е.В. и др. Молекулярно-филогенетическая характеристика эндемичных дальневосточных близкородственных видов секции Orobia рода Oxytropis (Fabaceae) // Генетика. 2020. Т. 56. № 4. С. 421–432.
Холина А.Б., Козыренко М.М., Артюкова Е.В. и др. Филогенетические отношения видов Oxytropis секции Arctobia северо-востока Азии по данным секвенирования межгенных спейсеров хлоропластного и ITS ядерного геномов // Генетика. 2020. Т. 56. № 12. С. 1386–1397.
Mir B.A., Koul S., Kumar A. et al. Intraspecific variation in the internal transcribed spacer (ITS) regions of rDNA in Withania somnifera (Linn.) Dunal // Indian J. Biotechnol. 2010. V. 9. P. 325–328.
Bonfeld J.K., Smith K.F., Staden R. A new DNA sequence assembly program // Nucl. Acids Res. 1995. V. 23. P. 4992–4999.
Gouy M., Guindon S., Gascuel O. SeaView version 4: A multiplatform graphical user interface for sequence alignment and phylogenetic tree building // Mol. Biol. Evol. 2010. V. 27. P. 221–224. https://doi.org/10.1093/molbev/msp259
Excoffier L., Lischer H.E.L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Resour. 2010. V. 10. P. 564–567.
Librado P., Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. № 11. P. 1451–1452.
Swofford D.L. PAUP*: Phylogenetic analysis using parsimony (*and other methods): version 4.04. Sunderland, MA, USA: Sinauer Associates Inc., 2003.
Posada D., Crandall K.A. Modeltest: testing the model of DNA substitution // Bioinformatics. 1998. V. 14. P. 817–818. https://doi.org/10.1093/bioinformatics/14.9.817
Ronquist F., Huelsenbeck J.P. MrBAYES3: Bayesian phylogenetic inference under mixed models // Bioinformatics. 2003. V. 19. P. 1572–1574. https://doi.org/10.1093/bioinformatics/btg180
Miller M.A., Pfeiffer W., Schwartz T. Creating the CIPRES Science Gateway for inference of large phylogenetic trees // Proc. Gateway Computing Environments Workshop (GCE). LA: New Orleans, 2010. https://doi.org/10.1109/GCE.2010.5676129
Bandelt H.-J., Forster P., Röhl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. № 1. P. 37–48.
Plenk K., Willner W., Demina O.N. et al. Phylogeographic evidence for long-term persistence of the Eurasian steppe plant Astragalus onobrychis in the Pannonian region (eastern Central Europe) // Flora. 2020. V. 264. 151555. 10 p. https://doi.org/10.1016/j.flora.2020.151555
Shepherd L.D., Lange P.J., Perrie L.R., Heenan P.B. Chloroplast phylogeography of New Zealand Sophora trees (Fabaceae): Extensive hybridization and widespread Last Glacial Maximum survival // J. Biogeogr. 2017. V. 44. P. 1640–1651. https://doi.org/10.1111/jbi.12963
Абрамсон Н.И. Филогеография: итоги, проблемы, перспективы // Информ. вестник ВОГиС. 2007. Т. 11. № 2. С. 307–331.
Meyers Z.J. A contribution to the taxonomy and phylogeny of Oxytropis section Arctobia (Fabaceae) in North America: Master Diss. Fairbanks. Alaska: Univ. Alaska Fairbanks, 2012. 155 p.
Archambault A., Strömvik M.V. Evolutionary relationships in Oxytropis species, as estimated from the nuclear ribosomal internal transcribed spacer (ITS) sequences point to multiple expansions into the Arctic // Botany. 2012. V. 90. № 8. P. 770–779. https://doi.org/10.1139/B2012-023
Tekpinar A., Karaman Erkul S., Aytac Z., Kaya Z. Phylogenetic relationships among native Oxytropis species in Turkey using trnL intron, trnL–F IGS, and trnV intron cpDNA regions // Turk. J. Bot. 2016. V. 40. P. 472–479. https://doi.org/10.3906/bot-1506-45
Krivenko D.A., Kotseruba V.V., Kazanovsky S.G. et al. Fabaceae // IAPT/IOPB chromosome data 11 / Ed. Marhold K. // Taxon. 2011. V. 60. № 4. P. 1222; E12, E13.
Konichenko E.S., Selyutina I.Yu., Dorogina O.V. Oxytropis triphylla // IAPT/IOPB chromosome data 14 / Ed. Marhold K. // Taxon. 2012. V. 61. № 6. P. 1339; E13.
Lavin M., Herendeen P.S., Wojciechowski M.F. Evolutionary rates analysis of Leguminosae implicates a rapid diversification of lineages during the Tertiary // Syst.Biol. 2005. V. 54. P. 530–549. https://doi.org/10.1080/10635150590947131
Orthia L.A., Crisp M.D., Cook L.G., de Kok R.P.J. Bush pea: A rapid radiation with no support for monophyly of Pultenaea (Fabaceae: Mirbelieae) // Aust. Syst. Bot. 2005. V. 18. P. 133–147. https://doi.org/10.1071/SB04028
Bagheri A., Maassoumi A.A., Rahiminejad M.R. et al. Molecular phylogeny and divergence times of Astragalus section Hymenostegis: An analysis of a rapidly diversifying species group in Fabaceae // Sci. Rep. 2017. №7: 14033. 9 pages. https://doi.org/10.1038/s41598-017-14614-3
Дополнительные материалы отсутствуют.


