Генетика, 2022, T. 58, № 11, стр. 1261-1272
Популяционно-генетическая структура степного волка России и Казахстана по микросателлитным локусам
П. А. Казимиров 1, *, С. В. Леонтьев 2, 3, А. В. Нечаева 1, М. М. Белоконь 1, Ю. С. Белоконь 1, А. Я. Бондарев 4, А. В. Давыдов 4, Д. В. Политов 1, **
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Казахский агротехнический университет им. С. Сейфуллина
010011 Нур-Султан, Республика Казахстан
3 Новосибирский государственный аграрный университет
Новосибирск, 630039Россия
4 Федеральный центр развития охотничьего хозяйства
105118 Москва, Россия
* E-mail: farenklaw@gmail.com
** E-mail: dmitri_p@inbox.ru
Поступила в редакцию 23.06.2022
После доработки 30.06.2022
Принята к публикации 01.07.2022
- EDN: MVPHRK
- DOI: 10.31857/S0016675822110042
Аннотация
Проведено популяционно-генетическое изучение волка, Canis lupus Linnaeus, 1758, на части ареала в России и Казахстане с фокусом на происхождение, состояние генофонда и статус степного волка (номинативный подвид C. l. campestris). С помощью анализа 18 ядерных микросателлитных локусов показано, что уровень внутрипопуляционного генетического разнообразия степного волка Казахстана достаточно высок, аллельное и генное разнообразие сравнимо с популяциями степного волка других регионов (степи европейского юга России) и лесного и горно-лесного экотипов, населяющих Кавказ, Приуралье и Западную Сибирь. Анализ дифференциации изученных выборок в программе STRUCTURE позволяет сделать заключение о наличии в генофонде степного волка как местного кластера генотипов, так и иммигрантов из соседних, прилегающих к степи с севера, территорий. Обсуждается возможное влияние антропогенного пресса в виде регулирования популяций в советское и постсоветское время и последующая реколонизация из окружающих донорских популяций.
Вопрос о подвидах волка Canis lupus Linnaeus, 1758 (Mammalia: Carnivora: Canidae), обоснованности их выделения, статусе и генетической основе наблюдаемых морфоэкологических различий широко дискутировался в течение всей истории изучения этого несомненно самого важного для человека крупного хищника [1–3]. Несмотря на способность к распространению на большие расстояния, основную роль в которых играет натальная миграция [4], что должно вести к перемешиванию генетического материала, волк отдельных регионов и природных зон имеет своеобразие морфологических признаков, экологии и поведения, что явилось основанием для выделения многочисленных подвидов. Однако, поскольку подвиды волка обычно связаны не с выраженной дифференциацией формы тела, черепа или иных структур, а в основном с размерами тела и особенностями мехового покрова и его окраски, часто, особенно в англоязычной литературе, они называются экотипами, что подчеркивает слабую степень изоляции между ними и неопределенность генетической основы, лежащей за видимыми внешними различиями. Наиболее принятая в России и странах бывшего СССР система подвидов [2, 3, 5] для части ареала волка, охватывающего Россию, Казахстан и государства Средней Азии, включает тундряного волка C. l. albus Kerr, 1792, лесного C. l. lupus (Linnaeus, 1758), кавказского C. l. cubanensis Ognev, 1923, степного C. l. campestris Dwigubski, 1804, монгольского/тибетского C. l. chanco Gray, 1863 и пустынного C. l. desertorum Bogdanov, 1882. Иногда выделяется также сибирский лесной подвид C. l. altaicus [1, 5, 6] и некоторые другие [7]. В то же время при тщательном анализе серьезных оснований для подтверждения подвидового статуса, например алтайского волка, выявлено не было [8].
Степной волк не был исключением в ряду дискуссий о правомерности выделения подвидов на основании достаточно слабо выраженных морфологических различий у волков бореальной и умеренной зон Северной Евразии, касающихся главным образом размеров и окраски, т.е. признаков, подверженных отбору в конкретных условиях среды на фоне потенциально интенсивного обмена генами между локальными популяциями. Волк, населяющий открытые пространства западной и центральной части Палеарктики к югу от лесной зоны, еще в начале XIX в. был выделен в отдельный подвид C. l. campestris Dwigubski, 1804. По сравнению с лесным степной волк мельче, окраска более светлая, в восточной части ареала с примесью рыжих тонов. Существенных морфологических и анатомических различий с другими подвидами не выявлено, скорее это были признаки морфотипа/экотипа. Ареал степного волка включает степи Предкавказья, Нижнего Поволжья, Прикаспия, Приуралья, Западной Сибири, Казахстана и Средней Азии [2]. На востоке ареал степного волка охватывает север Монголии и Внутреннюю Монголию (Китай). Считается, что с севера степной волк контактирует с лесным, или обыкновенным, волком (C. l. lupus s.l.), а с юго-запада – с кавказским (прискаспийским) C. l. cubanensis (подвид, ныне признаваемый в основном лишь некоторыми териологами стран бывшего СССР). В Средней Азии и Казахстане степной волк контактирует на юге с признаваемым далеко не всеми специалистами пустынным волком C. l. desetorum, а на юго-востоке (и юге, если не выделять волка пустынного в подвид) – с монгольским (тибетским), C. l. chanco. Насколько глубоки генетические различия между этими подвидами – вопрос крайне сложный, как спорно и то, могут ли данные по отдельным генетическим маркерам являться основанием для поддержки или отрицания подвидового статуса. Так, под сомнение было поставлено и выделение сразу нескольких подвидов палеарктических волков – C. l. chanco, C. l. desertorum, C. l. cubanensis и собственно степного волка C. l. campestris, которые было предложено ввести в синонимию обыкновенного (лесного, евразийского) C. lupus lupus [9, 10]. В настоящее время доминирует точка зрения, согласно которой все эти подвиды являются слабо дивергировавшими подгруппами бореальной ветви евразийского волка, хотя в отношении тибетского/монгольского волка, распространенного от Каспия до Тибета и на восток до тихоокеанского побережья Китая, подвидовой статус обычно сохраняется. В то же время на юге Азии, где ареал волка в XX в. оказался фрагментированным, выделяются не только подвиды (азиатский, или индийский волк C. l. pallipes, аравийский C. l. arabs [11]), но и, в частности на индийском субконтиненте, новые виды, например гималайский (C. himalayaensis) и индийский (C. indica) волки, которые считаются одними из наиболее древних в Евразии на основании изменчивости их мтДНК [12, 13]. В то же время более поздние исследования с помощью мтДНК [14] показали близость северо- и восточноазиатских волков, а не гималайских, к плейстоценовым, и в целом крайне сложную и запутанную филогеографическую картину, по крайней мере по последовательностям контрольного региона мтДНК. Тем не менее примеры “дробительского” подхода к внутривидовой систематике палеарктического с выделением не только множества подвидов у внутриподвидовых рас волка сохранились до недавнего времени [7]. Очевидно, что вопрос далек от разрешения, и пути дальнейших исследований лежат в области использования молекулярно-генетических маркеров и геномных подходов [15–17]. Крайне интересны и важны исследования ископаемых и субфоссильных волков и собак [18], при изучении современного генетического разнообразия волка много внимания уделяется событиям плейстоцена и их влиянию на происхождение рецентных форм, в том числе и в Сибири [19].
Однако при анализе дифференциации степного волка нужно учесть, что не меньший, чем палеогеография, след в генетической структуре популяций должен был оставить антропогенный пресс. В советский период истории волк открытых пространств как на севере, в тундре, так и на юге от лесной зоны, в степи, оказался более уязвим, чем лесной волк, поскольку с развитием технических средств, применяемых для борьбы с ним, таких как автотранспорт, вертолеты, а также яды, в степях стало возможным добывать тысячи и даже десятки тысяч особей хищника в год [2, 20, 21]. Кроме того, с освоением целинных земель степной зоны юга РСФСР и Казахстана, особенно в послевоенное время, задача регулирования популяций волка была признана важнейшей в государственном масштабе для достижения целей снижения прямого ущерба от хищничества волка для сельского и охотничьего хозяйства [22].
Наиболее обширная часть ареала степного волка сохранилась до настоящего времени на территории Республики Казахстан [22, 23], где после периода тотальной борьбы с волком в Казахской ССР численность волка стабилизировалась на более низком, но в целом достаточно высоком, уровне. Одной из проблем, которая требует исследования популяционно-генетическими методами, является выявление автохтонного генетического субстрата и возможных мигрантов из потенциальных донорских популяций, которыми для волка Казахстана можно признать как группировки горно-лесного волка Алтая, так и степных и пустынных волков Средней Азии, Китая и Монголии.
Оценка современного состояния генофонда степного волка может не только дать ответ на вопрос о его статусе, происхождении, но и практических путях регулирования его численности и снижения накала конфликта с человеком. Понимание того факта, что экономический вред от волка не абсолютен, а его воздействие на популяции диких животных и степные экосистемы в целом имеет и обратную сторону – повышение их адаптивной устойчивости, в целом пришло [2, 20–29]. Тем не менее популяционно-генетических исследований, основанных на массовом материале из типовых местообитаний степного волка в России и Казахстане, до настоящего времени не проводилось. В наших предыдущих работах по волку Алтая [26] и Сибири [30] на основе изменчивости шести микросателлитных локусов мы показали в целом достаточно слабо выраженную дифференциацию на изученной части ареала, однако основные популяционные группировки волка в Сибири имеют некоторую географическую локализацию, в которой степной волк из номинативных местообитаний юга Сибири (Алтайский край, Бурятия) оказался в значительной степени перемешан с лесным.
Целью настоящей работы было изучение уровней генетического разнообразия и паттернов дифференциации по расширенной панели ядерных микросателлитных локусов популяционных группировок волка, обитающего в степной зоне Российской Федерации и Республики Казахстан в сравнении с соседними регионами лесной и горно-лесной зон европейской части России, Приуралья, Сибири и Казахстана.
МАТЕРИАЛЫ И МЕТОДЫ
Характеристика биологического материала
Материал для популяционно-генетического анализа представлен высушенными фрагментами шкур или фиксированными тканями мышц или внутренних органов волка, сбор которых проводился в ходе государственных мероприятий по регулированию численности вида в России и Казахстане. Образцы из Республики Казахстан собраны автором (С.В. Леонтьев), биоматериал из регионов России прислан по запросу из региональных охотуправлений и предоставлен для настоящего исследования ФГБУ “Центрохотконтроль”/ФГБУ “Федеральный центр развития охотничьего хозяйства” (А.Я. Бондарев, А.В. Давыдов, П.М. Павлов). Всего было исследовано 326 особей из 16 регионов России и Казахстана. Для определенных видов анализа образцы из выборок также были отнесены к “макрорегиону” на основе места происхождения образца (табл. 1).
Таблица 1.
Географическое происхождение материала и распределение по группам
| Регион | Изначальное количество образцов | Количество образцов, вошедших в анализ |
|---|---|---|
| Степи и лесостепи европейской части РФ (EurStRu) | ||
| Республика Калмыкия | 12 | 8 |
| Ростовская область | 3 | 3 |
| Ставропольский край | 69 | 43 |
| Кавказский регион РФ (Cau) | ||
| Республика Адыгея | 10 | 8 |
| Кабардино-Балкарская Республика | 5 | 4 |
| Республика Чечня | 12 | 8 |
| Леса Приуралья, РФ (ForUr) | ||
| Пермский край | 43 | 35 |
| Запад Казахстана (KzW) | ||
| Актюбинская область | 16 | 16 |
| Западно-Казахстанская область | 9 | 7 |
| Атырауская область | 12 | 11 |
| Костанайская область | 13 | 8 |
| Тургайский регион* | 13 | 13 |
| Восток Казахстана (KzE) | ||
| Восточно-Казахстанская область | 6 | 6 |
| Карагандинская область | 30 | 24 |
| Леса Западной Сибири, РФ (ForSib) | ||
| Тюменская область | 12 | 10 |
| Республика Алтай | 61 | 52 |
Выделение ДНК и проведение ПЦР
Выделение тотальной ДНК от волков с территории РФ проводили наборами DNeasy Blood & Tissue Kit компании “QiaGen” или ДНК-Экстран 2 компании “Синтол” в соответствии с протоколами производителя. При выделении ДНК наборами ДНК-Экстран 2 после проведения лизиса дополнительно удаляли из супернатанта липиды четыреххлористым углеродом. К лизату добавляли 300 мкл CCl4, перемешивали на вортексе, центрифугировали одну минуту при 3000 об./мин, после чего полученный супернатант, содержащий раствор ДНК, переносили в чистые пробирки. Дальнейшее выделение проводили в соответствии с протоколом проводителя. ДНК от волков, добытых охотниками по лицензиям на территории Казахстана, выделялась на базе Казахского агротехнического университета им. С. Сейфуллина с помощью наборов для выделения Qiagen Blood and Tissue DNA Extraction Kit (Qiagen, США). Для проверки качества выделенных препаратов ДНК использовали электрофорез в агарозном геле с визуализацией в УФ-свете после окраски бромистым этидием.
Генотипирование изначально проводилось по 31 аутосомному микросателлитному локусу [31] (табл. 2), сборку ПЦР-реакций проводили отдельно для каждой из четырех мультиплексных смесей праймеров (1a,b; 2a,b; 3a,b; 4a,b), однако в отличие от оригинального протокола для амплификации использовали мастер-миксы GenPak™ PCRCore (OOO “Лаборатория Изоген”, Москва, Россия). Режимы амплификации не отличались от рекомендованных в статьях, где описаны соответствующие праймеры [32–34, 36, 39]. После амплификации ПЦР-продукты хранили при –20°C (кратковременно при 2–8°C). Для проверки амплификации специфического ПЦР-продукта и его качества использовали электрофорез в полиакриламидном геле.
Таблица 2.
Характеристики микросателлитных маркеров, используемых в исследовании
| Локус | Число аллелей (по [31]) | Источник |
|---|---|---|
| 2010 | 5 | [32] |
| 2137 | 14 | [32] |
| AHT002 | 8 | [33] |
| AHT121 | 13 | [34] |
| vWf | 9 | [35] |
| CXX.253 | 8 | [36] |
| 2079 | 5 | [32] |
| MS41B | 9 | [37] |
| PEZ03 | 15 | [38] |
| PEZ06 | 12 | [38] |
| CXX.225 | 4 | [36] |
| 2096 | 5 | [32] |
| 2201 | 23 | [32] |
| CXX.250 | 6 | [36] |
| 2006 | 5 | [32] |
| 2140 | 14 | [32] |
| AHT119 | 9 | [34] |
| 2054 | 8 | [32] |
| 2088 | 17 | [32] |
| 2168 | 14 | [32] |
| AHT004 | 4 | [34] |
| CXX.20 | 5 | [36] |
| 2159 | 16 | [32] |
| CXX.123 | 8 | [36] |
| CXX.204 | 6 | [36] |
| AHT103 | 7 | [34] |
| AHT138 | 8 | [34] |
| 2001 | 8 | [32] |
| AHT101 | 12 | [33] |
| AHT106 | 15 | [33] |
| AHT126 | 11 | [39] |
Перед фрагментным анализом образцы объединялись в четыре смеси (меченные разными цветовыми метками) в соотношении по 1 мкл мультиплексов a и b каждой реакции и 13 мкл воды milli-Q. Фрагментный анализ проводился c помощью капиллярного электрофореза на ДНК-анализаторах компании “Синтол” (Москва, Россия).
Считывание и обработка результатов
Считывание результатов фрагментного анализа и получение первичных матриц генотипов проводили в программе STRand v2.4.110 [40]. В качестве дополнительного программного обеспечения использовали пакет Fragman v1.0.9 для среды R [41]. Матрицы многолокусных генотипов предварительно обрабатывали с помощью макроса GenAlEx v6.503 [42], работающего на базе электронной таблицы MS Excel. Так как фрагментный анализ исследуемых образцов проводился в разных партиях и на разных приборах, для установления соответствия аллелей была проведена полокусная компенсация погрешности прибора на основании данных о частотах аллелей. Кроме того, некоторые образцы из предыдущих партий фрагментного анализа переставлялись в следующие эксперименты в качестве реперных. Локусы, для которых изменчивость, связанная с погрешностью прибора, осталась выше, чем изменчивость, обусловленная популяционными процессами, были исключены из анализа. Также были исключены локусы и особи с большим количеством отсутствующих данных.
В связи с особенностями получения материала – от особей, добытых охотниками, в коллекции потенциально присутствовало большое число близкородственных особей (так как волков часто добывают семьями). Родство не всегда указано в сопроводительной информации, поэтому мы проверили его сами, чтобы исключить потенциальных близких родственников из анализа. Так как большое количество близкородственных особей может внести погрешность в популяционный анализ, было принято решение исключить близкородственных особей. Для этого использовался пакет Demerelate для среды R [43]. Определение индекса родства проводили по [44]. Далее из пар волков с индексом родства >0.5 удаляли либо особь с большим количеством отсутствующих данных, либо случайную особь. Если одна из особей в паре уже была удалена, то вторая особь автоматически оставалась в анализе.
Также проводилась проверка на присутствие в локусах нуль-аллелей. Для этого использовали три независимых теста: проверка присутствия отклонения от равновесия Харди–Вайнберга для каждого локуса в каждом макрорегионе; проверка наличия отклонения индекса фиксации от нуля (рассчитано по χ2) для каждого локуса как по макрорегионам, так и по совокупным данным; проверка присутствия нуль-аллеля для каждого локуса по макрорегионам. Первые два теста проводились в GenAlEx, для выявления и оценки частот нуль-аллелей использовалась программа Micro-Checker v2.2.3 [45]. Локусы, для которых все три теста дали положительный результат, были исключены из анализа.
Анализ популяционной структуры методами байесовского анализа проводили в программе Structure v2.3.4 [46–48] со следующими параметрами: длина периода “разогрева” (“burn-in”) – 10 000; количество MCMC (Markov chain Monte Carlo) повторов после “разогрева” – 100 000; модель наследования – “admixture”; число кластеров K – 1–12, число повторов для каждого K – 5, флаг UsePopInfo=0. На основе анализа генетической структуры проводился расчет наиболее вероятного числа исходных кластеров (K) при помощи метода Эванно [49] с использованием конвейерного веб-сервиса СLUMPAK [50]. Этот же веб-сервис использовали для получения совокупных результатов для каждого K. Для интерпретации выбиралось наиболее вероятное значение K, а также несколько соседних значений с близкими оценками вероятности по Эванно.
Для выделенных популяций GenAlEx были рассчитаны основные популяционно-генетические параметры: среднее число аллелей на локус – NA; число эффективных аллелей – NE; средняя наблюдаемая гетерозиготность – HO; средняя ожидаемая гетерозиготность – HE; внутрипопуляционный индекс фиксации – F; показатель межпопуляционной дифференциации – FST; теоретическое число мигрантов между популяциями на поколение при наблюдаемом значении FST – Nm; среднее число приватных (специфичных для популяции) аллелей – Np.
РЕЗУЛЬТАТЫ
Девять локусов были исключены из анализа из-за значительной погрешности прибора или большого количества пропущенных данных. Четыре локуса (2006, 2159, CXX.123 и AHT126) исключены из-за того, что в них выявлены нуль-аллели, отклонения от равновесия Харди–Вайнберга и отклонения индекса фиксации от нуля. Для двух локусов (AHT119 и 2201) наблюдалось присутствие нуль-аллеля во всех кластерах с частотами от 0.09 до 0.24, однако отклонение от Харди–Вайнберга и отклонение индекса фиксации от нуля наблюдали лишь в отдельных кластерах, в связи с чем было принято решение оставить их в анализе. Всего в итоговый набор анализируемых микросателлитов вошло 18 локусов: 2137, 2010, CXX.253, vWf, 2079, PEZ03, AHT119, 2201, 2096, CXX.250, CXX.225, 2140, 2054, 2168, AHT138, CXX.204, AHT106, 2001.
В результате исключения близкородственных индивидуумов из анализа было удалено 70 особей. В итоговый анализ вошло 256 образцов. Изначальное число особей из разных регионов и итоговое число образцов, которые вошли в анализ, представлены в табл. 1.
Анализ внутрипопуляционной изменчивости
Для анализа популяционно-генетических параметров выборки волка юга степной и лесостепной зон Европейской части России были объединены с кавказским регионом в единый макрорегион, так как анализ популяционной структуры показал, что они представляют собой единый кластер, а малое число особей в кавказском макрорегионе препятствует адекватному сравнению популяционно-генетических параметров с другими регионами.
Среднее число аллелей на локус по популяциям варьирует от 7.72 ± 0.57 на востоке Казахстана до 9.11 ± 0.63 на западе Казахстана (табл. 3). В этих же выборках наблюдаются крайние значения количества эффективных аллелей – 4.66 ± 0.34 и 5.47 ± 0.39 соответственно. Наибольшее число приватных аллелей выявлено в выборках лесного волка Сибири. В целом выявленный уровень аллельного разнообразия по микросателлитным локусам был характерным для популяций волка, не прошедших “бутылочное горлышко”.
Таблица 3.
Оценки популяционно-генетических параметров в объединенных по регионам выборках волка по 18 микросателлитным локусам
| Выборка | N | NA | NE | I | HO | HE | uHE | F | Np |
|---|---|---|---|---|---|---|---|---|---|
| EurStRu + Cau | 73.11 | 8.77 | 4.88 | 1.73 | 0.68 | 0.77 | 0.78 | 0.114 | 6 |
| 0.26 | 0.63 | 0.33 | 0.07 | 0.02 | 0.01 | 0.01 | 0.02 | ||
| ForUr | 33.38 | 8.00 | 5.29 | 1.74 | 0.66 | 0.78 | 0.79 | 0.14 | 6 |
| 0.44 | 0.67 | 0.48 | 0.08 | 0.02 | 0.02 | 0.02 | 0.03 | ||
| KzW | 53.11 | 9.111 | 5.467 | 1.83 | 0.70 | 0.80 | 0.81 | 0.12 | 7 |
| 0.51 | 0.63 | 0.39 | 0.07 | 0.01 | 0.01 | 0.01 | 0.02 | ||
| KzE | 29.33 | 7.72 | 4.66 | 1.67 | 0.68 | 0.76 | 0.77 | 0.10 | 3 |
| 0.25 | 0.57 | 0.34 | 0.07 | 0.03 | 0.01 | 0.01 | 0.03 | ||
| ForSib | 60.88 | 8.94 | 4.97 | 1.73 | 0.69 | 0.77 | 0.77 | 0.09 | 12 |
| 0.30 | 0.71 | 0.43 | 0.08 | 0.02 | 0.02 | 0.02 | 0.02 |
Примечание. Верхняя строка – среднее, нижняя – ошибка. N – объем выборки; NA – общее число аллелей; NE – число эффективных аллелей; I – индекс Шеннона; HO – наблюдаемая гетерозиготность; HE – ожидаемая гетерозиготность; uHE – ожидаемая гетерозиготность с поправкой на размер выборки; F – внутрипопуляционный индекс фиксации; Np – среднее число приватных (специфичных для популяции) аллелей.
Наблюдаемая гетерозиготность варьирует между регионами лишь в небольшой степени – почти все значения находятся в рамках стандартной ошибки друг от друга с максимальной разницей между средними значениями по регионам, равной 0.04. Минимальные значения наблюдаются в лесах Приуралья (0.66 ± 0.02), максимальные – на западе Казахстана (0.70 ± 0.01). Разница между ожидаемой и наблюдаемой гетерозиготностью во всех регионах составляет ~0.1 ± стандартная ошибка для данного региона. При этом минимальные значения ожидаемой гетерозиготности наблюдаются на востоке Казахстана – 0.76 ± 0.01; максимальные – на западе Казахстана – 0.80 ± 0.01. Для популяции лесов востока России не наблюдается различий между значениями ожидаемой гетерозиготности, как без поправки на размер популяции, так и с ее учетом. В остальных популяциях средние значения с поправкой и без нее различаются на 0.01, что находится в рамках стандартной ошибки. Минимальные значения индекса фиксации наблюдаются в популяции лесов востока России – 0.09 ± 0.02, максимальные – в лесах Приуралья – 0.14 ± 0.03.
Число приватных аллелей в популяциях варьирует от трех на востоке Казахстана до 12 в лесах Сибири. При этом в остальных регионах наблюдается практически идентичное число приватных аллелей – 6 в лесах запада России и в лесах Приуралья и 7 на западе Казахстана.
Таким образом, можно сделать вывод о том, что ни в одном из исследуемых районов не наблюдается какого-либо значительного снижения уровня внутрипопуляционной изменчивости.
Анализ межпопуляционной подразделенности
Степень генетической подразделенности популяций волка в изученной части ареала на территории России и Казахстана оказалась невысокой (FST = 0.029). Анализ матрицы генетических дистанций между географическими популяциями (рис. 1) показал значительное расхождение друг от друга популяций трех регионов (Западного, лесного Приуральского и Восточного). Популяции Казахстана занимают промежуточное положение между выборками лесного волка Уральского и Восточного регионов и наиболее удалены от выборок запада России.
Рис. 1.
Результаты многомерного анализа межпопуляционной дифференциации волка методом главных координат по матрице попарных генетических дистанций.

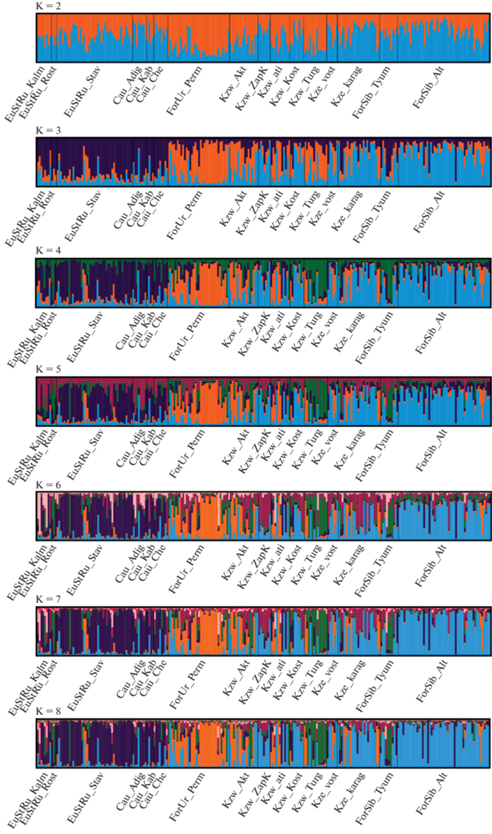
Методом Эванно было выявлено оптимальное число исходных кластеров K = 6. При рассмотрении меньших К можно выделить три крупных генетических кластера, внутри которых при больших значениях К выделяются более мелкие популяции (рис. 2). Эти три “макрокластера” имеют следующий состав:
1. Западный: степи и лесостепи Европейской части России, включая Кавказ, а также часть Тюменской области.
2. Уральский (данный кластер начинает выявляться уже при K = 2): леса Приуралья, а также Тургайская, Актюбинская и частично Атырауская области Казахстана.
3. Восточный: леса Алтая, часть Тюменской области, Западно-Казахстанская, Костанайская и Карагандинская области.
Стоит отметить, что степные регионы, представленные в данном исследовании в основном территорией Казахстана, при К = 3 не обособляются.
При К = 4 происходит выделение нового кластера, присутствующего в основном в Тургайском регионе Казахстана и Тюменской области. При К = 5 происходит дробление Западного кластера – от него отделяется популяция Республики Калмыкия. При К = 6 происходит выделение нового кластера на территории Казахстана, присутствующего в основном в Западно-Казахстанской и Карагандинской областях.
При К > 6 выделения новых обособленных кластеров не происходит – наблюдается дробление кластера, ассоциированного с популяцией Калмыкии, на более мелкие фрагменты без значительного влияния на общую популяционную структуру. Начиная с К = 10, видимых изменений генетической структуры с увеличением К не наблюдается. Таким образом, можно говорить о том, что К = 6 в полной мере описывает межпопуляционную изменчивость в рамках данной работы.
При рассмотрении К = 6 с точки зрения экотипов можно отметить, в первую очередь, ярко выраженную генетическую идентичность лесных и европейских степных регионов – три “макрокластера”, выделившихся на малых значениях К, все еще занимают доминирующее положение в популяционной структуре. Со степными регионами ассоциированы два кластера. Первый, приуроченный преимущественно к Тургайскому региону, выделяется уже на К = 4. Особи, принадлежащие к этому кластеру, присутствуют также в Актюбинской и Атырауской областях Казахстана, Тюменской, Ростовской областях и Ставропольском крае России. Второй степной кластер, ассоциированный в первую очередь с Западно-Казахстанской и Карагандинской областями Казахстана, выделяется только на К = 6, что может свидетельствовать о его меньшем уровне генетической обособленности. Особи, принадлежащие к данному кластеру, присутствуют также в Костанайской и Восточно-Казахстанской областях Казахстана, а также в Республике Алтай и Пермском крае.
Стоит отметить относительно высокую долю волков, относящихся к “лесным” кластерам, в выборках с территории Казахстана. Эти особи, вероятно, ассоциированы с регионами лесов и лесостепей или являются потомками мигрантов из лесных и горно-лесных ландшафтов.
ОБСУЖДЕНИЕ
Уровни внутрипопуляционного генетического разнообразия в популяциях степного волка оказались высокими и близкими к наблюдаемым в соседних частях ареала и природных зонах. Таким образом можно сделать вывод о том, что если сокращение численности и повлияло на параметры внутрипопуляционного разнообразия, то не самым драматическим образом, или что генетическое разнообразие было восстановлено за счет миграции из соседних регионов, в пользу чего свидетельствует и наличие общих исходных кластеров многолокусных генотипов у казахстанского, южнорусского и сибирского волков.
Анализ популяционной структуры демонстрирует значительное влияние окружающих регионов на популяции волка в Казахстане. Так, в западных областях, в частности в Актюбинской, наблюдается большое количество особей, относящихся к уральскому кластеру. В восточных и центральных регионах присутствует большое количество особей, относящихся к алтайскому кластеру. В обоих случаях наблюдаются как особи, практически целиком относящиеся к уральскому или алтайскому кластеру, так и их потомки с меньшей долей принадлежности к кластеру.
Влияние кавказского кластера на популяционную структуру Казахстана выражено значительно в меньшей степени – он представлен лишь отдельными особями. Тем не менее все еще наблюдается разнообразие доли принадлежности к кавказскому кластеру, что свидетельствует о присутствии как свежих мигрантов, так и их потомков.
Несмотря на такое существенное влияние со стороны российских популяций, в Казахстане присутствуют два генетических кластера, ассоциированных в первую очередь именно с территорией Республики, что свидетельствует о наличии на ее территории генетически обособленных популяционных группировок. При этом присутствие особей, относящихся к этим кластерам за пределами Казахстана, служит свидетельством двухстороннего генетического обмена между популяциями Казахстана и сопредельных территорий. Об активном генетическом потоке между популяциями свидетельствуют также и высокие, сравнимые с популяциями РФ, популяционно-генетические параметры.
Численность и плотность популяции волка на открытых пространствах, в том числе в степи, потенциально выше, чем у таежного (лесного, горно-лесного) волка, что объясняется трудностями, которые он должен преодолевать в условиях большой высоты снежного покрова в лесу, затрудняющего передвижение и охоту [51]. Однако большая уязвимость волка перед человеком в тех же открытых биотопах приводит к некоторому балансу. При усилении пресса истребительных мероприятий волка в рамках программ регулирования его численности средняя плотность населения волка в степи снижается, однако в случае Казахстана, по-видимому, предпринятые усилия не привели к “бутылочному горлышку”, т.е. не достигли эффекта коллапса, ведущего к генетическому обеднению. При том, что на пиках добычи волка, в 1953–1955 гг., в год уничтожалось до 13–21 тыс. особей, на спаде, во время и после Великой Отечественной войны (например, 1942 г. – 2000 особей), в 1966 г. – 7875 волков – при том, что отстрел велся не так интенсивно, никогда не было падения до значений ниже 7 тыс. добытых зверей [22]. В 1971–1978 гг. на территории Казахской ССР добывалось от 7.4 до 9.9 тыс. особей, и эти оценки были ниже рассчитанной величины приплода. Для сравнения во всей Российской Федерации в настоящее время добывается в среднем около 10–11 тыс. волков в год [52]. Таким образом, демографические причины не могли вызвать генетического обеднения и, скорее всего, не вызвали.
Помимо репродукции автохтонного степного волка, другим важным фактором, который нужно рассмотреть в качестве поддерживающего и повышающего генетическое разнообразие популяции, является иммиграция из соседних регионов. Донорскими популяциями для степного волка Южной Сибири и Казахстана могли быть: с северо-запада – волк лесов и лесостепей Приуралья и Зауралья (C. l. lupus), с севера и северо-востока – горно-таежный волк Алтая и Саян C. l. lupus (или C. l. altaicus), с юга – пустынный волк Средней Азии C. l. desertorum, с востока – монгольский/тибетский подвид C. l. chanco. Мы можем отметить реальный вклад северных, сибирских кластеров в генетическую конституцию степного волка. Представительство у сибирско-казахстанского волка кластеров, характерных для степного волка популяций степей юга Европейской части РФ, невелико. Материал от пустынного и монгольского волка в нашем исследовании отсутствовал, и в связи с этим трудно судить о связях этих подвидов/экотипов со степным. Можно лишь предполагать, что один из “автохтонных” кластеров, а именно выявленный в Карагандинской области, каким-то образом связан с этими подвидами. Что касается генетического субстрата местного степного волка, наши данные свидетельствуют о том, что, несмотря на иммиграцию, он сохранился и является одной из основ генофонда популяции волка Казахстана.
Дальнейшие исследования с привлечением данных по другим классам генетических маркеров и расширение географии выборок требуются для более детального анализа популяционно-генетической структуры степного волка.
Этические нормы обращения с животными не нарушались, ни одно животное не было добыто специально для данного исследования. Все образцы взяты от законно добытых особей.
Работа выполнена при финансовой поддержке гранта Российского фонда фундаментальных исследований 18-04-01300, а также частично в рамках темы госзадания (ИОГен РАН) 0112-2019-0001.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Pocock R.I. The races of Canis lupus // Proc. Zool. Society London. 1935. V. 105. № 3. P. 647–686.
Волк. Происхождение, систематика, морфология, экология / Под ред. Бибикова Д.И. М.: Наука, 1985. 606 с.
Огнев С.И. Звери Восточной Европы и Северной Азии. М.: Изд-во АН СССР, 1931. 776 с.
Wabakken P., Sand H., Kojola I. et al. Multistage, long-range natal dispersal by a global positioning system–collared Scandinavian wolf // J. Wildlife Management. 2007. V. 71. № 5. P. 1631–1634. https://doi.org/10.2193/2006-222
Гептнер В.Г., Наумов Н.П., Юргенсон Т.Б. и др. Млекопитающие Советского Союза. Т. 2. Ч. 1. Морские коровы и хищные. М.: Высш. шк., 1967. С. 123–193.
Суворов А.П., Смирнов М.Н. О подвидовом положении алтайского горно-таежного волка (Canis lupus altaicus) // Тр. Тигирекского заповедника. 2005. № 1. С. 247–350.
Суворов А.П., Кириенко Н.Н. К вопросу о внутривидовом полиморфизме волка (Canis lupus), обитающего на территории России // Вестник Красноярского гос. аграрного ун-та. 2008. № 3. С. 6.
Бондарев А.Я. Морфологическая характеристика волка (Canis lupus) Западной и Средней Сибири // Вестник Алтайского гос. аграрного ун-та. 2012. Т. 87. № 1. С. 30–34.
Bush R.H. The Wolf Almanac. N.Y.: Lyons & Burford, 1993. 256 p.
Pilot M., Greco C., vonHoldt B.M. et al. Genome-wide signatures of population bottlenecks and diversifying selection in European wolves // Heredity. 2014. V. 112. № 4. P. 428–442. https://doi.org/10.1038/hdy.2013.122
Bray T.C., Mohammed O.B., Butynski T.M. et al. Genetic variation and subspecific status of the grey wolf (Canis lupus) in Saudi Arabia // Mamm. Biology. 2014. V. 79. № 6. P. 409–413. https://doi.org/10.1016/j.mambio.2014.06.005
Aggarwal R.K., Kivisild T., Ramadevi J., Singh L. Mitochondrial DNA coding region sequences support the phylogenetic distinction of two Indian wolf species // J. Zool. Syst. and Evol. Research. 2007. V. 45. № 2. P. 163–172. https://doi.org/10.1111/j.1439-0469.2006.00400.x
Sharma D.K., Maldonado J.E., Jhala Y.V., Fleischer R.C. Ancient wolf lineages in India // Proc. Royal Soc. London. Series B–Biol. Sciences. 2004. V. 271. P. S1–S4.
Ersmark E., Klutsch C.F.C., Chan Y.L. et al. From the past to the present: Wolf phylogeography and demographic history based on the mitochondrial control region. // Frontiers Ecol. Evol. 2016. V. 4. P. 1–12 https://doi.org/10.3389/fevo.2016.00134
Hindrikson M., Remm J., Pilot M. et al. Wolf population genetics in Europe: A systematic review, meta-analysis and suggestions for conservation and management // Biol. Reviews. 2017. V. 92. № 3. P. 1601–1629. https://doi.org/10.1111/brv.12298
vonHoldt B.M., Pollinger J.P., Earl D.A. et al. A genome-wide perspective on the evolutionary history of enigmatic wolf-like canids // Genome Res. 2011. V. 21. № 8. P. 1294–1305. https://doi.org/10.1101/gr.116301.110
vonHoldt B.M., Stahler D.R., Bangs E.E. et al. A novel assessment of population structure and gene flow in grey wolf populations of the Northern Rocky Mountains of the United States // Mol. Ecol. 2010. V. 19. № 20. P. 4412–4427. https://doi.org/10.1111/j.1365-294X.2010.04769.x
Ovodov N.D., Crockford S.J., Kuzmin Y.V. et al. A 33,000-year-old incipient dog from the Altai Mountains of Siberia: Evidence of the earliest domestication disrupted by the last glacial maximum // PLoS One. 2011. V. 6. № 7. P. 1–6. https://doi.org/10.1371/journal.pone.0022821
Ramos-Madrigal J., Sinding M.-H.S., Carøe C. Genomes of Pleistocene Siberian wolves uncover multiple extinct wolf lineages // Curr. Biology. 2021. V. 31. № 1. P. 198–206. https://doi.org/10.1016/j.cub.2020.10.002
Леонтьев С.С. Состояние волка как биологического ресурса в Казахстане // Вестник НГАУ. 2018. № 1. С. 80–87.
Леонтьев С.В., Бербер А.П. Оценка состояния биологического ресурса волка в Казахстане // Вестник охотоведения. 2018. Т. 15. № 2. С. 90–98.
Федосенко А.К., Слудский А.А. Волк – Canis lupus Linnaeus, 1758 // Млекопитающие Казахстана. Т. 3. Ч. 1. Алма-Ата: Наука, 1981. С. 8–56.
Федосенко А.К. Волки. Алма-Ата: Кайнар, 1986. 95 с.
Бондарев А.Я. О принципах регулирования численности волка // Вестник Алтайского гос. аграрного ун-та. 2012. № 9(95). С. 70–71.
Бондарев А.Я. Волки Западной Сибири: численность, распределение и загрязненность. М.: ФГБУ “Центрохотконтроль”, 2013. 248 с.
Бондарев А.Я., Воробьевская Е.А., Политов Д.В. О генетическиой дифференциации волка Сибири // Вестник АГАУ. 2013. Т. 9. № 107. С. 49–57.
Бондарев А.Я., Дёжкин А.В., Павлов П.М. Регулирование популяций волка: проблемы и решения // Териофауна России и сопредельных территорий. X съезд Териологического общества при РАН. М.: Тов-во науч. изд. КМК, 2016. С. 52.
Леонтьев С.В. Об изменении плотности населения волков на степной территории Казахстана // Современные проблемы охотничьего хозяйства Казахстана и сопредельных стран: Материалы Междунар. научно-практ. конф. Алматы, 2014. С. 174–177.
Леонтьев С.В. Питание и роль волка в природе и сельском хозяйстве Казахстана // Инновации в продовольственной безопасности. 2017. Т. 4(18). С. 107–118.
Талала М.С., Бондарев А.Я., Захаров Е.C., Политов Д.В. Генетическая дифференциация популяций волка Canis lupus L. Сибири по микросателлитным локусам // Генетика. 2020. Т. 56. № 1. С. 67–77.https://doi.org/10.1134/S0016675820010129
Åkesson M., Liberg O., Sand H. et al. Genetic rescue in a severely inbred wolf population // Mol. Ecol. 2016. V. 25. № 19. P. 4745–4756. https://doi.org/10.1111/mec.13797
Francisco L.V., Langston A.A., Mellersh C.S. et al. A class of highly polymorphic tetranucleotide repeats for canine genetic mapping // Mammal. Genome. 1996. V. 7. № 5. P. 359–362. https://doi.org/10.1007/s003359900104
Holmes N.G., Dickens H.F., Parker H.L. et al. Eighteen canine microsatellites. Two polymorphic bovine microsatellite loci // Animal Genetics. 1995. V. 26. № 2. P. 132–133. https://doi.org/10.1111/j.1365-2052.1995.tb02659.x
Holmes N.G., Mellersh C.S., Humphreys S.J. et al. Isolation and characterization of microsatellites from the canine genome // Animal Genetics. 1993. V. 24. № 4. P. 289–292. https://doi.org/10.1111/j.1365-2052.1993.tb00313.x
Shibuya H., Collins B.K., Huang T.H.M., Johnson G.S. A polymorphic (AGGAAT), tandem repeat in an intron of the canine von Willebrand factor gene // Animal Genetics. 1994. V. 25. № 2. P. 122. https://doi.org/10.1111/j.1365-2052.1994.tb00094.x
Ostrander E.A., Sprague G.F., Rine J. Identification and characterization of dinucleotide repeat (CA)n markers for genetic mapping in dog // Genomics. 1993. V. 16. № 1. P. 207–213. https://doi.org/10.1006/geno.1993.1160
Sundqvist A.K., Ellegren H., Olivier M., Vilà C. Y chromosome haplotyping in Scandinavian wolves (Canis lupus) based on microsatellite markers // Mol. Ecol. 2001. V. 10. № 8. P. 1959–1966. https://doi.org/10.1046/j.1365-294x.2001.01326.x
Neff M.W., Broman K.W., Mellersh C.S. et al. A second-generation genetic linkage map of the domestic dog, Canis familiaris // Genetics. 1999. V. 151. № 2. P. 803–820. https://doi.org/10.1093/genetics/151.2.803
Holmes N.G., Strange N.J., Binns M.M. et al. Three polymorphic canine microsatellites // Animal Genetics. 1994. V. 25. № 3. P. 200. https://doi.org/10.1111/j.1365-2052.1994.tb00122.x
Toonen R.J., Hughes S. Increased throughput for fragment analysis on an ABI PRISM 377 automated sequencer using a membrane comb and STRand software // BioTechniques. 2001. V. 31. № 6. P. 1320–1324.
Covarrubias-Pazaran G., Diaz-Garcia L., Schlautman B. et al. Fragman: An R package for fragment analysis // BMC Genetics. 2016. V. 17. № 1. P. 62. https://doi.org/10.1186/s12863-016-0365-6
Peakall R., Smouse P.E. GENALEX 6: Genetic analysis in Excel. Population genetic software for teaching and research // Mol. Ecol. Notes. 2006. V. 6. № 1. P. 288–295. https://doi.org/10.1093/bioinformatics/bts460
Kraemer P., Gerlach G. Demerelate: Calculating interindividual relatedness for kinship analysis based on codominant diploid genetic markers using R // Mol. Ecol. Resour. 2017. V. 17. P. 1371–1377. https://doi.org/10.1111/1755-0998.12666
Blouin M.S., Parsons M., Lacaille V., Lotz S. Use of microsatellite loci to classify individuals by relatedness // Mol. Ecol. 1996. V. 5. № 3. P. 393–401. https://doi.org/10.1111/j.1365-294x.1996.tb00329.x
Van Oosterhout C., Hutchinson W.F., Wills D.P.M., Shipley P. MICRO-CHECKER: Software for identifying and correcting genotyping errors in microsatellite data // Mol. Ecol. Notes. 2004. V. 4. № 3. P. 535–538. https://doi.org/10.1111/j.1471-8286.2004.00684.x
Falush D., Stephens M., Pritchard J.K. Inference of population structure using multilocus genotype data: Linked loci and correlated allele frequencies // Genetics. 2003. V. 164. № 4. P. 1567–1587. https://doi.org/10.1093/genetics/164.4.1567
Hubisz M.J., Falush D., Stephens M., Pritchard J.K. Inferring weak population structure with the assistance of sample group information // Mol. Ecol. Resour. 2009. V. 9. № 5. https://doi.org/10.1111/j.1755-0998.2009.02591.x
Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data // Genetics. 2000. V. 155. Issue 2. P. 945–959. https://doi.org/10.1093/genetics/155.2.945
Evanno G., Regnaut S., Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE: A simulation study // Mol. Ecol. 2005. V. 14. № 8. P. 2611–2620. https://doi.org/10.1111/j.1365-294X.2005.02553.x
Kopelman N.M., Mayzel J., Jakobsson M. et al. Clumpak: A program for identifying clustering modes and packaging population structure inferences across K // Mol. Ecol. Resour. 2015. V. 15. № 5. P. 1179–1191. https://doi.org/10.1111/1755-0998.12387
Формозов А.Н. Роль снежного покрова в жизни млекопитающих и птиц // Материалы к познанию фауны и флоры СССР. М.: МОИП, 1946. С. 47–49.
Дёжкин А.В., Данилкин А.А., Кузякин В.А. Концептуальные основы рационального ресурсопользования в охотничьем хозяйстве России // Государственное управление ресурсами. Спец. выпуск “Охота и охотничьи ресурсы Российской Федерации”. 2011. С. 332–339.
Дополнительные материалы отсутствуют.