Генетика, 2022, T. 58, № 12, стр. 1459-1464
Использование технологии KASP для изучения ассоциаций однонуклеотидных вариантов в генах GPAD4, CCL3, DGKG, PPARGC1A, STAT1, TLR4 с молочной продуктивностью крупного рогатого скота
М. В. Модоров 1, 2, *, А. А. Клещева 1, К. Р. Осинцева 1, И. В. Ткаченко 1, М. Ю. Севостьянов 1, Н. Н. Зезин 1
1 Уральский федеральный аграрный научно-исследовательский центр
Уральского отделения Российской академии наук
620142 Екатеринбург, Россия
2 Институт экологии растений и животных Уральского отделения
Российской академии наук
620144 Екатеринбург, Россия
* E-mail: mmodorov@gmail.com
Поступила в редакцию 04.02.2022
После доработки 07.04.2022
Принята к публикации 12.04.2022
- EDN: QKDTXA
- DOI: 10.31857/S0016675822120086
Аннотация
С использованием метода KASP-генотипирования определены частоты аллелей шести однонуклеотидных полиморфизмов (SNP) в выборке племенного голштинизированного черно-пестрого скота Свердловской области. SNP8: g.107062990G>A гена TLR4 (rs8193048) не проявил изменчивости в анализируемой выборке крупного рогатого скота, остальные SNP были изменчивы. Проведен ассоциативный тест между генотипами коров и удоем за 305 дней первой лактации, процентным содержанием и количеством жира и белка молока. Для SNP CCL3 rs109686238, DGKG rs41608610, PPARGC1A rs133669403 и STAT1 rs43705173 – ассоциации не выявлены. При проведении маркер-ориентированной селекции, направленной на увеличение молочной продуктивности голштинизированного черно-пестрого скота Свердловской обл., использовать эти локусы не рекомендуется. Для SNP27: g.36531442T>C гена GPAD4 (rs109913786) показана ассоциация с процентным содержанием жира в молоке. Дана рекомендация по использованию этого SNP в программах маркер-ориентированной селекции уральской популяции голштинизированного черно-пестрого скота.
Дикий бык (Bos taurus L. 1758) является одним из наиболее важных объектов животноводства, генетическим исследованиям которого уделяется много внимания. К настоящему времени расшифрован геном и получены тысячи прочтений полного генома представителей многих пород [1, 2]. Разработаны и активно используются SNP-чипы, включающие от нескольких десятков до сотен тысяч маркеров [3–5]. Для тысяч геновариантов показаны ассоциации с хозяйственно ценными признаками крупного рогатого скота [6–8].
Основной массив данных, способствующих современному прогрессу генетических исследований крупного рогатого скота, получен с использованием SNP-чипов и прочтений полного генома. В то же время существует ряд научных и прикладных задач, которые могут быть эффективно (дешевле и быстрее) решены с использованием более простых методов генетических исследований. К таким задачам можно отнести:
1. Подтверждение достоверности происхождения породы крупного рогатого скота с использованием STR-маркеров [9, 10].
2. Определение геновариантов менделирующих признаков (фенов, проявление которых определяется одним геном). В качестве примера можно привести наследственные заболевания крупного рогатого скота [11–13], одну из мутаций гена CSN3 (однонуклеотидный полиморфизм SNP – 6: g.85656736T>C, rs43703015), ассоциированную с сыродельческими качествами молока, а также одну из мутаций в гене CSN2 (SNP 6: g.85451132C>G, rs43703013), определяющую так называемое А2‑молоко [14].
3. Поиск казуальной мутации для какого-либо признака [15, 16]. Довольно часто скрининг выборки, проведенный с использованием SNP-чипов и полногеномного секвенирования, указывает на наличие нескольких кандидатных геновариантов, ассоциированных с исследуемым признаком [15, 17–23]. Для выбора казуальной мутации необходимо оценить распространение кандидатных геновариантов в популяции и их связь с фенотипом.
4. Различного рода научные проекты, в задачи которых входит генотипирование образцов по небольшому числу маркеров [24, 25]. В эту же группу можно включить задачи, связанные с проведением маркер-ориентированной селекции по локусам, вносящим существенный вклад в проявление количественных хозяйственно ценных признаков животных.
Если есть необходимость определить генотипы небольшой по размеру выборки животных, процедура постановки и оптимизации ПЦР может являться наиболее долгим, трудоемким и непредсказуемым этапом работы. В связи с этим интерес представляют методы ПЦР, не требующие значительных ресурсозатрат на этапе постановки реакции и определении геновариантов животных. В качестве подобного метода в данной работе мы рассмотрим KASP (competitive allele specific PCR, конкурентная аллель-специфическая ПЦР).
KASP позволяет проводить генотипирование образцов по принципу “один локус в одной лунке” (uniplex) с использованием реагентов LGC Genomics (http://www.lgcgenomics.com, https://www.biosearchtech.com/). Состав реагентов реакции, как и последовательность уникальных праймеров для исследования целевого полиморфизма, является коммерческой тайной производителя, детекция продукта проводится по конечной точке. В качестве локусов могут быть выбраны биаллельные SNP, инсерции и делеции [26, 27]. Удобство использования KASP определяется простотой постановки реакции (необходимо смешать универсальный мастер-микс, праймеры и ДНК). Кроме этого, метод подразумевает использование единой для всех маркеров последовательности температур и времени термоциклирования. В данной работе с использованием метода KASP-генотипирования будет проанализирована связь шести SNP с молочной продуктивностью голштинизированного черно-пестрого скота Свердловской обл.
МАТЕРИАЛЫ И МЕТОДЫ
Объектом исследования был племенной голштинизированный черно-пестрый скот 2016–2018 гг. рождения, содержащийся в сельскохозяйственной организации, расположенной на территории Свердловской обл. Данные о продуктивности животных (удой за 305 дней первой лактации, процентном содержании и массе белка и жира в молоке) брали из ИАС “Селэкс – Молочный скот”. Размеры проанализированных выборок коров для разных маркеров составили от 91 до 182 голов.
ДНК выделяли из крови, законсервированной в ЭДТА, с использованием набора “ДНК-Экстран-1” (“Синтол”, Россия). Концентрацию ДНК измеряли с использованием Nabi Spectrophotometer (MicroDigital, Респубика Корея). В качестве генетических маркеров были выбраны шесть SNP, для которых ранее в работе E. Viale с соавт. [3] была показана ассоциация с молочной продуктивностью, либо количеством жира в молоке голштинской породы крупного рогатого скота, разводимой на территории Италии. При заполнении формы заказа реагентов использовали UCSC GenomeBrowser (https://genome-euro.ucsc.edu/), из которого брали последовательность нуклеотидов в районе расположения мутации. Названия SNP и последовательности ДНК с их локализацией приведены в табл. 1. Реагенты были изготовлены LGC Genomics (США), заказ осуществляли через официального дистрибьютора LGC Genomics в России ООО “Максим Медикал”. KASP-генотипирование проводили с использованием Applied Biosystems Real-Time PCR System 7500 согласно протоколу производителя реагентов. Для амплификации использовали 96-луночные прозрачные планшеты Kirgen (KG2561) и пленку для планшетов (KG2571). Расшифровку генотипов проводили после 42 циклов ПЦР.
Таблица 1.
Локализация исследуемых SNP в геноме Bos taurus с указанием последовательностей, используемых при заказе реагентов для KASP-генотипирования
| SNP (ген) | Положение SNP* | Последовательность | Присутствие локуса на SNP-чипах** |
|---|---|---|---|
| rs109913786 (AGPAT6, syn. GPAD4) |
chr27: 36220692 36531442 |
AGCGGGAGCAGTAAGGCCCTGGACAACAC TCCCGAGTTTGAGCTCTCGGA[T/C]ATTTTCT ATTTCTGCCGGAAAKGAATGGAGACCATCA TGGACGATGAGGT | Illumina BovineHDBead Chip |
| rs109686238 (CCL3) |
chr19: 14673538 14359777 |
CACACGAATAGGGCAGGCGCGATGTTGCCC GGGACRGTGGCCCAAGCCTC[T/C]TTTGGAT GGAAGGAACTGAATTAAGGAATTCCCGCCA GAGGACCAGGAAG | Illumina BovineSNP50 BeadChip |
| rs41608610 (DGKG) | chr1: 81589478 80999135 |
AGTATACAATAGCCACATGTGGCTAATGGC TAAGTCACTTTGGCAACTTA[C/T]GGAGATA AAAAAACAGAAAGGAGACAGTAAGACAGA TTATTTGGTATTTG | Illumina BovineHDBead Chip, Illumina BovineSNP50 BeadChip |
| rs133669403 (PPARGC1A) |
chr6: 44875315 43401661 |
ATTACCTGGGCCGCCGGCTATGGGGCGATC TTGAACGTGACGCRCACAGG[A/G]GCGAATT TCGGTGTGTGCGGTGTCTGCAGTGGCCTGA CTCATAGTAGTAG | |
| rs43705173 (STAT1) |
chr2: 79888611 79518124 |
GCTTTATGATGCTGGCTAATATCAATAGAA GGAAGTAAACTTTACAAATT[C/T]ATGAGTA GTATCTTCCATTTCAGCTTTAATACCAAAGT TGAATATATTCTG | |
| rs8193048 (TLR4) |
chr8: 108834063 107062990 |
CGATCATCAGTGTGTCGGTGGTCACTGTGC TCCTGGTGTCTGTGGTAGGR[G/A]TCCTAGT CTACAAGTTCTATTTCCACCTGATGCTTCTT GCTGGCTGCAAA |
Примечание. * – сверху позиция в сборке генома Bos_taurus_UMD_3.1.1/bosTau8 (https://genome-euro.ucsc.edu/), внизу – позиция в сборке генома ARS-UCD1.2 (106 annotation release, https://www.ncbi.nlm.nih.gov/), ** – данные взяты с сайта https://www.ensembl.org/.
Статистическую обработку данных проводили в R-project [28]. Выполняли ассоциативный тест между генотипом коров и их молочной продуктивностью, а именно: удоем за 305 дней первой лактации, процентном содержанием и массой белка и жира в молоке. При проведении ассоциативного теста использовали дисперсионный анализ (ANOVA), множественные сравнения групповых средних выполняли с использованием критерия Тьюки. При проверке статистических гипотез использовали уровень статистической значимости p < 0.05. Вводили поправку Бонферрони на множественное сравнение p < 0.05/6 = 0.0083 (шесть генетических маркеров).
РЕЗУЛЬТАТЫ
Частоты аллелей и генотипов шести исследуемых SNP представлены в табл. 2. SNP8: g.107062990G>A гена TLR4 не проявил изменчивости, все проанализированные особи имели гомозиготные генотипы GG. Остальные пять локусов были изменчивы. Для трех SNP частоты минорных аллелей не более чем на 3% отличались от частот, отмеченных для популяции голштинской породы скота, разводимой на территории Италии [3], они составили 22% для DGKG rs41608610, 20% для PPARGC1A rs133669403 и 16% для CCL3 rs109686238. Частота минорного аллеля STAT1 rs43705173 в выборке уральского скота составляет 38%, что на восемь процентов больше, чем в популяции Италии. Для GPAD4 rs109913786 обнаруженная нами частота минорного аллеля составляет 36%, что на 17% больше, чем в выборке итальянских голштинов. Во всех случаях наблюдаемые частоты генотипов статистически значимо не отличались от частот, соответствующих равновесию Харди–Вайнберга (p < 0.05).
Таблица 2.
Частота встречаемости аллелей и генотипов в выборке голштинизированного черно-пестрого скота Свердловской обл.
| SNP (ген) | N | Генотип | Частота генотипа, % | Частота аллеля, % | |
|---|---|---|---|---|---|
| rs109913786 (AGPAT6, syn. GPAD4) |
180 | TT | 15.0 | T | 36.1 |
| TC | 42.2 | C | 63.9 | ||
| CC | 42.8 | ||||
| rs109686238 (CCL3) |
182 | AA | 72.8 | A | 84.3 |
| AG | 23.9 | G | 15.7 | ||
| GG | 3.3 | ||||
| rs41608610 (DGKG) | 171 | AA | 61.4 | A | 77.8 |
| AG | 32.8 | G | 22.2 | ||
| GG | 5.8 | ||||
| rs133669403 (PPARGC1A) |
89 | GG | 64 | G | 80.3 |
| GA | 32.6 | A | 19.7 | ||
| AA | 3.4 | ||||
| rs43705173 (STAT1) |
92 | GG | 35.9 | G | 62.5 |
| GA | 53.2 | A | 37.5 | ||
| AA | 10.9 | ||||
| rs8193048 (TLR4) |
91 | GG | 100 | G | 100 |
| GA | 0 | A | 0 | ||
| AA | 0 | ||||
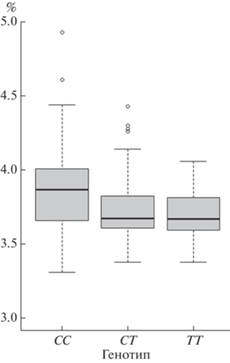
Ассоциативный тест выявил связь генотипов SNP27: g.36531442T>C гена GPAD4 с процентным содержанием жира в молоке коров за первую лактацию (р < 0.00029). Особи с генотипом CC статистически значимо превосходили особей с генотипом TC (p = 0.0018) и особей с генотипом TT (p = 0.0031), рис. 1. Особи с генотипом CC имели средний процент жира в молоке – 3.87%, особи с генотипом TC – 3.73%, с генотипом TT – 3.69%. Ассоциаций других генотипов с молочной продуктивностью коров выявлено не было.
Рис. 1.
Жирность молока у коров с разными генотипами по замене 27: g.36531442T>C (rs109913786) в гене GPAD4.
Минимальное достаточное количество ДНК для корректного определения генотипа составило не менее 50 нг/реакцию.
ОБСУЖДЕНИЕ
Во многих исследованиях полногеномного поиска ассоциаций (GWAS) было показано, что в районе локализации гена GPAD4 находится локус количественного признака (QTL), связанного с процентным содержанием жира молока. Связь различных геновариантов GPAD4 с процентным содержанием жира в молоке была показана для нескольких пород крупного рогатого скота [2, 3, 29–31].
Исследуемый нами SNP27: g.36531442T>C гена GPAD4 (rs109913786) локализован в третьем из 12 экзонов, мутация является синонимичной. Ранее ассоциативный тест для данного локуса был проведен только в двух работах. Для швицкой породы коров ассоциация геновариантов rs109913786 с молочной продуктивностью обнаружена не была [32 ] , тогда как для скота голштинской породы, разводимой на территории Италии, локус был ассоциирован как с процентным содержанием, так и с количеством жира в молоке [3]. Согласно результатам нашего исследования, геновариант CCSNP27: g.36531442T>C гена GPAD4 ассоциирован с высоким процентным содержанием жира в молоке голштинизированного черно-пестрого скота Свердловской обл.
Таким образом, метод KASP-генотипирования показал свою эффективность при проведении популяционно-генетических исследований крупного рогатого скота. Метод позволил расшифровать генотипы животных для всех SNP, попавших в сферу наших интересов, при этом затраты времени на дизайн реагентов и отработку протоколов амплификации были минимальными. SNP GPAD4 rs109913786 можно рекомендовать для использования в программах маркер-ориентированной селекции уральской популяции голштинизированного черно-пестрого скота. Для пяти SNP, а именно: DGKG rs41608610, PPARGC1A rs133669403, CCL3 rs109686238, STAT1 rs43705173, TLR4 rs8193048, ассоциации с удоем, процентным содержанием жира и белка в молоке в выборке голштинизированного черно-пестрого скота обнаружены не были. При проведении маркер-ориентированной селекции, направленной на увеличение молочной продуктивности голштинизированного черно-пестрого скота Свердловской обл., использовать эти локусы не рекомендуется.
Исследование выполнено при финансовой поддержке РФФИ в рамках научного проекта № 20-016-00239.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Elsik C.G., Tellam R.L., Worley K.C. The genome sequence of taurine cattle: A window to ruminant biology and evolution // Science. 2009. V. 324. № 5926. P. 522–528. https://doi.org/10.1126/science.1169588
Hayes B.J., Daetwyler H.D. 1000 bull genomes project to map simple and complex genetic traits in cattle: Applications and outcomes // Ann. Rev. Animal Biosciences. 2019. V. 7. P. 89–102. https://doi.org/10.1146/annurev-animal-020518-115024
Viale E., Tiezzi F., Maretto F. et al. Association of candidate gene polymorphisms with milk technological traits, yield, composition, and somatic cell score in Italian Holstein-Friesian sires // J. Dairy Sci. 2017. V. 100. № 9. P. 7271–7281. https://doi.org/10.3168/jds.2017-12666
Neumann G.B., Korkuć P., Arends D. et al. Design and performance of a bovine 200k SNP chip developed for endangered German Black Pied cattle (DSN) // BMC Genomics. 2021. V. 22. Article number 905. https://doi.org/10.1186/s12864-021-08237-2
Zaalberg R.M., Poulsen N.A., Bovenhuis H. et al. Genetic analysis on infrared-predicted milk minerals for Danish dairy cattle // J. Dairy Sci. 2021. V. 104. № 8. P. 8947–8958. https://doi.org/10.3168/jds.2020-19638
Jiang J., Ma L., Prakapenka D. et al. A large-scale genome-wide association study in US Holstein cattle // Frontiers in Genet. 2019. V. 10. https://doi.org/10.3389/fgene.2019.00412
Liu L., Zhou J., Chen C.J. et al. GWAS – based identification of new loci for milk yield, fat, and protein in Holstein cattle // Animals. 2020. V. 10. № 11. https://doi.org/10.3390/ani10112048
Hoze C., Escouflaire C., Fritz S., Capitan A. A splice site mutation in CENPU is associated with recessive embryonic lethality in Holstein cattle // J. of Dairy Sci. 2020. V. 103. № 1. P. 607–612. https://doi.org/10.3168/jds.2019-17056
Модоров М.В., Ткаченко И.В., Грин А.А. и др. Генетическая структура популяции голштинизированного черно-пестрого скота на территории Урала // Генетика. 2021. Т. 57. № 4. С. 437–444. https://doi.org/10.31857/S001667582104010X
Модоров М.В., Ткаченко И.В., Грин А.А. Индивидуальная идентификация особей и контроль родословной в популяции голштинизированного черно-пестрого скота Урала // Вопр. нормативно-правового регулирования в ветеринарии. 2019. № 4. С. 119–121. https://doi.org/10.17238/issn2072-6023.2019.4.119
Khan M.Y.A., Omar A.I., He Y. et al. Prevalence of nine genetic defects in Chinese Holstein cattle // Veter. Med. and Sci. 2021. V. 7. № 5. P. 1728–1735. https://doi.org/10.1002/vms3.525
Zhang Y., Liang D., Huang H. et al. Development and application of KASP assays for rapid screening of 8 genetic defects in Holstein cattle // J. Dairy Sci. 2020. V. 103. № 1. P. 619–624. https://doi.org/10.3168/jds.2019-16345
Модоров М.В., Мартынов Н.А., Шкуратова И.А. и др. Распространение рецессивных генетических нарушений в уральской популяции крупного рогатого скота // Генетика. 2022. Т. 58. № 4. С. 429–437. https://doi.org/10.31857/S0016675822040105
Caroli A.M., Chessa S., Erhardt G.J. Invited review: Milk protein polymorphisms in cattle: Effect on animal breeding and human nutrition // J. Dairy Sci. 2009. V. 92. № 11. P. 5335–5352. https://doi.org/10.3168/jds.2009-2461
Lehner S., Zerbin I., Doll K. et al. A genome-wide association study for left-sided displacement of the abomasum using a high-density single nucleotide polymorphism array // J. Dairy Sci. 2018. V. 101. № 2. P. 1258–1266. https://doi.org/10.3168/jds.2017-13216
Sieck R.L., Fuller A.M, Bedwell P.S. et al. Mandibulofacial dysostosis attributed to a recessive mutation of CYP26C1 in Hereford cattle // Genes. 2020. V. 11. № 11. https://doi.org/10.3390/genes11111246
Adams H.A., Sonstegard T.S., VanRaden P.M. et al. Identification of a nonsense mutation in APAF1 that is likely causal for a decrease in reproductive efficiency in Holstein dairy cattle // J. Dairy Sci. 2016. V. 99. № 8. P. 6693–6701. https://doi.org/10.3168/jds.2015-10517
Kunz E., Rothammer S., Pausch H. et al. Confirmation of a non-synonymous SNP in PNPLA8 as a candidate causal mutation for Weaver syndrome in Brown Swiss cattle // Genet. Select. Evol. 2016. V. 48. Article number 21. https://doi.org/10.1186/s12711-016-0201-5
Pausch H., Schwarzenbacher H., Burgstaller J. et al. Homozygous haplotype deficiency reveals deleterious mutations compromising reproductive and rearing success in cattle // BMC Genomics. 2015. V. 16. Article number 312. https://doi.org/10.1186/s12864-015-1483-7
Rothammer S., Kunz E., Seichter D. et al. Detection of two non-synonymous SNPs in SLC45A2 on BTA20 as candidate causal mutations for oculocutaneous albinism in Braunvieh cattle // Genet. Select. Evol. 2017. V. 49. Article number 73. https://doi.org/10.1186/s12711-017-0349-7
Schwarzenbacher H., Burgstaller J., Seefried F.R. et al. A missense mutation in TUBD1 is associated with high juvenile mortality in Braunvieh and Fleckvieh cattle // BMC Genomics. 2016. V. 17. Article number 400. https://doi.org/10.1186/s12864-016-2742-y
Schwarzenbacher H., Wurmser C., Flisikowski K. et al. A frameshift mutation in GON4L is associated with proportionate dwarfism in Fleckvieh cattle // Genet. Select. Evol. 2016. V. 48. Article number 25. https://doi.org/10.1186/s12711-016-0207-z
Venhoranta H., Pausch H., Flisikowski K. et al. In frame exon skipping in UBE3B is associated with developmental disorders and increased mortality in cattle // BMC Genomics. 2014. V. 15. 890. https://doi.org/10.1186/1471-2164-15-890
Neary M.T., Breckenridge R.A., Neary J.M. et al. A comparison of DNA collection methods in cattle and yaks // J. Anim. Sci. 2014. V. 92. № 9. P. 3811–3815. https://doi.org/10.2527/jas.2013-7445
Pareek C.S., Błaszczyk P., Dziuba P. et al. Single nucleotide polymorphism discovery in bovine liver using RNA-seq technology // PLoS One. 2017. V. 12. № 2. https://doi.org/10.1371/journal.pone.0172687
Semagn K., Babu R., Hearne S., Olsen M. Single nucleotide polymorphism genotyping using Kompetitive Allele Specific PCR (KASP): Overview of the technology and its application in crop improvement // Mol. Breeding. 2014. V. 33. № 1. P. 1–14. https://doi.org/10.1007/s11032-013-9917-x
Rasheed A., Wen W., Gao F. et al. Development and validation of KASP assays for genes underpinning key economic traits in bread wheat // Theor. Appl. Genet. 2016. V. 129. № 10. P. 1843–1860. https://doi.org/10.1007/s00122-016-2743-x
R Core Team. R: A language and environment for statistical computing. R Foundation for Statistical Computing. Vienna, Austria, 2020. https://www.R-project.org
Littlejohn M.D., Tiplady K., Lopdell T. et al. Expression variants of the lipogenic AGPAT6 gene affect diverse milk composition phenotypes in Bos taurus // PLoS One. 2014. V. 9. № 1. https://doi.org/10.1371/journal.pone.0085757
Bouwman A.C., Bovenhuis H., Visker M.H.P.W., van Arendonk J.A.M. Genome-wide association of milk fatty acids in Dutch dairy cattle // BMC Genetics. 2011. V. 12. https://doi.org/10.1186/1471-2156-12-43
Wang X., Wurmser C., Pausch H. et al. Identification and dissection of four major QTL affecting milk fat content in the German Holstein-Friesian population // PLoS One. 2012. V. 7. № 7. https://doi.org/10.1371/journal.pone.0040711
Cecchinato A., Ribeca C., Chessa S. et al. Candidate gene association analysis for milk yield, composition, urea nitrogen and somatic cell scores in Brown Swiss cows // Animal. 2014. V. 8. № 7. P. 1062–1070. https://doi.org/10.1017/S1751731114001098
Дополнительные материалы отсутствуют.


