Генетика, 2023, T. 59, № 5, стр. 584-600
Разработка тест-системы для ДНК-идентификации особей вида енотовидная собака
А. Е. Гребенчук 1, *, О. Н. Лукашкова 2, С. А. Котова 2, И. С. Цыбовский 3
1 Государственный комитет судебных экспертиз Республики Беларусь
220073 Минск, Республика Беларусь
2 Научно-практический центр Государственного комитета судебных экспертиз
Республики Беларусь
220114 Минск, Республика Беларусь
3 Республиканское унитарное предприятие “БелЮрОбеспечение”
220069 Минск, Республика Беларусь
* E-mail: iamsanya94@mail.ru
Поступила в редакцию 04.08.2022
После доработки 23.08.2022
Принята к публикации 09.09.2022
- EDN: FNOVHT
- DOI: 10.31857/S0016675823050065
Аннотация
По результатам исследования полиморфизма 39 микросателлитных (STR) локусов и трех локусов половой принадлежности, специфичных к различным видам семейства псовые, с целью ДНК-идентификации биологических образцов животных вида енотовидная собака (Nyctereutes procyonoides) предложена тест-система NPlex, включающая 14 аутосомных STRs и два половых локуса. Тест-система валидирована для решения экспертных задач по установлению принадлежности биологических следов, обнаруженных на местах правонарушений (следов крови и выделений, фрагментов мышечных или костных тканей, дермы и волос и т.п.), конкретной особи животного вида енотовидная собака, а также для установления биологического родства животных данного вида. Валидационные мероприятия проведены в соответствии с протоколом Scientific Working Group on DNA Analysis Methods.
Вид енотовидная собака (Nyctereutes procyonoides) является самым неизученным в семействе псовые. Доступна информация о нескольких группах исследователей [1–4], которые занимаются выявлением индивидуальных генетических характеристик этого вида животных. Поскольку данный вид псовых лидирующий распространитель таких заболеваний как бешенство и чесотка, основное внимание ученых направлено именно на исследования зоонозных инфекций у вида [5–8].
Вид енотовидная собака в начале XX в. обитал только на Дальнем Востоке и включал пять подвидов: Nyctereutes procyonoides procyonoides (обитал в Китае и Северном Вьетнаме); N. p. orestes (населял горную область Китая); N. p. ussuriensis (был распространен в большинстве регионов Сибири и Восточного Китая); N. p. koreensis и N. p. viverrinus (обитали на территории Кореи и Японии соответственно) [9]. В Беларуси енотовидная собака была интродуцирована в 1936 г. (Гомельская, Минская и Витебская области) [10]. Но уже до этого времени на территории республики встречались единичные особи енотовидной собаки, мигрировавшие из соседних областей России, в которых их интродукция была проведена раньше [11]. Енотовидная собака в Беларуси является ненормируемым охотничьим видом, охота на которого разрешена весь год. Однако отстрел данного животного без охотничьей путевки или в заповедниках классифицируется как факт незаконной охоты, что приводит к возбуждению уголовных дел и, как следствие, к назначению судебных экспертиз. Численность енотовидной собаки на территории страны в 2020 г. составляла около 15 000 особей, из которых 5500 особей (около 40% численности) были добыты в ходе плановых охотничьих мероприятий [12].
МАТЕРИАЛЫ И МЕТОДЫ
Объекты исследования и выделение ДНК
Для изучения STR-полиморфизма вида енотовидная собака было исследовано 165 образцов мышечной и хрящевой ткани и образцов шерсти животных (73 биологических образца самцов и 92 самок). Коллекция образцов диких животных была законно сформирована при содействии ГУ “Полесский государственный радиационно-экологический заповедник”, а также в сотрудничестве с РГОО “Белорусское общество охотников и рыболовов”.
ДНК из биологических образцов выделяли по методике, основанной на высвобождении ДНК в ходе инкубации образцов биологического материала в лизирующем буфере 20 мM Tris-HCl, pH 8.0, содержащем 2% SDS, 100 мM NaCl, 20 мM EDTA, с протеиназой К и 0.01 мМ дитиотрейтолом, при 37–56°С и периодическом встряхивании. Лизат мышечной и хрящевой ткани подвергали общепринятой процедуре очистки на силикагеле [13]. Определение количества выделенной ДНК проводили методом спектрофотометрии с использованием прибора для измерения концентрации общей фракции ДНК для микрообъемов объектов исследования DS-11 (DeNovix, США) согласно рекомендациям производителя.
Дизайн тест-системы, амплификация микросателлитных локусов и генотипирование
С целью разработки тест-системы для криминалистической ДНК-идентификации животных биологического вида енотовидная собака проведено исследование полиморфизма 39 STR-локусов и трех локусов половой принадлежности. Подробный перечень локусов указан в табл. S1 Приложения.
В процессе оптимизации условий протекания амплификации исследовали специфичность и интенсивность выявления аллелей в зависимости от значений концентрации ионов Mg2+, температуры отжига и концентрации праймеров, количества и качества ДНК, с использованием различных стабилизаторов полимеразы (Triton X-100, Tween-20, BSA, TMGNa, DMSO) в изменяющихся количествах, а также с ДНК-полимеразами различных типов и производителей. В результате конечный состав реакционной смеси общим объемом 10 мкл составил: 10 мМ Tris-HCl, pH 8.6; 25 мМ KCl; 2.0 мМ MgCl2; 0.2 мМ каждого из dNTP; 0.2–1.0 мкМ каждого из пары праймеров; 0.15 ед. активности ДНК-полимеразы; 1.5 нг/мкл БСА (бычий сывороточный альбумин); 0.02% Triton X-100 и 1–20 нг анализируемой ДНК.
При разработке тест-системы NPlex амплификацию проводили на программируемых приборах термоциклического типа “C1000” (BioRad, США) в следующих условиях: 10 мин инициальной инкубации при 95°С; 30× [30 с при 95°С, 40 с при 60°С и 60 с при 72°С] с финальным этапом элонгации в течение 30 мин при 72°С.
Характерное для каждого из образцов сочетание аллелей выявляли путем электрофоретического разделения продуктов ПЦР в генетическом анализаторе 3500 Genetic Analyzer (Applied Biosystems, США). Определение размеров выявленных аллелей (в пн) и соответствующих генотипов ДНК в исследуемых локусах проводили с использованием внутренних стандартов размера Orange 500 bp (NimaGen®, Нидерланды) и GeneScan-600 LIZТМ SizeStandard v2.0 (ThermoFisher Scientific, США), а также программного пакета GeneMapper ID-X v1.6.
Секвенирование аллелей локусов
Для выявления возможных изоаллелей и микровариантов последовательности, а также для тандемного исчисления аллелей тест-системы проводили определение первичной структуры аллелей путем секвенирования методом обрыва цепи. Нуклеотидные последовательности аллелей каждого из STR-локусов и локусов половой принадлежности особей вида енотовидная собака определяли в прямом и обратном направлении. Секвенирование проводили на приборе 3500 Genetic Аnalyzer с использованием набора для секвенирования BigDye® Tеrminator v.3.1 Cycle Sequencing Kit (Applied Biosystems) и BrilliantDye™ Terminator kit v.3.1 (NimaGen®). Полученные результаты анализировали с помощью пакета программ Sequencing Analysis Software v.5.4 (Applied Biosystems). Сравнительный анализ последовательностей аллелей исследованных локусов у целевых видов и видов-источников проводили на основе ресурсов базы данных нуклеотидных последовательностей GenBank [14] и программного продукта BioEdit v7.0.5.3 [15]. Последовательности каждого локуса тест-системы NPlex, имеющие минимальный и максимальный молекулярный размер аллелей, были внесены в базу данных GenBank с присвоением соответствующих номеров доступа, которые указаны в табл. S2 Приложения.
Статистическая обработка результатов
Основной анализ генетического разнообразия, а именно выявление уровня полиморфизма, расчет частот встречаемости аллелей, значения наблюдаемой (НO) и ожидаемой (НЕ) гетерозиготности, оценку соответствия равновесию Харди–Вайнберга (HWE) и оценку сцепления локусов проводили с использованием программных пакетов GenAlEx v6.5 [16] и Arlequin v3.5.1.3 [17]. Установление возможных ошибок в интерпретации генетических профилей локусов с интенсивными статтер-фрагментами, при выпадении аллелей, при наличии “нуль”-аллелей или других возможных артефактов ПЦР проводили с помощью программ Micro-Checker v2.2.1 [18] и Cervus v3.0.7 [19].
Кластерный анализ массива генотипов выборки енотовидной собаки, полученного с разработанной тест-системой, оценивали с использованием программы Structure v2.3.4 [20, 21].
Криминалистические параметры отобранных микросателлитных локусов (расчет PIC, вероятностей случайного совпадения генотипов для неродственных особей и для сибсов, частоты встречаемости определенного генотипа) были рассчитаны с помощью программных продуктов GenAlEx v6.5 и Cervus v3.0.7. Конвертацию массивов генотипов енотовидной собаки в генетические файлы вышеперечисленных программ проводили с использованием вспомогательного компонента PGDSpider [22].
Валидационные мероприятия
Процесс апробации и оптимизации условий использования тест-системы был направлен на достижение высокого уровня специфичности амплификации, оптимальной интенсивности выявления флуорофоров маркера и заключался в подборе температуры отжига праймеров, концентрации праймеров и их соотношения в системе, а также количества ДНК в пробе.
Валидационные мероприятия проведены в соответствии с протоколом Scientific Working Group on DNA Analysis Methods (SWGDAM) и включали в себя: исследование работоспособности тест-системы как с коммерческими наборами реагентов различных производителей (ThermoFisher Scientific, США, ОДО “Праймтех”, РБ; ООО “Синтол”, РФ; ЗАО “Евроген”, РФ; ООО “АртБиоТех”, РБ; BioRad, США), так и с реакционными смесями лабораторного приготовления, а также тестирование разработанной системы на повторяемость, воспроизводимость, специфичность, анализ смесей, чувствительность и устойчивость. Анализ результатов валидационных мероприятий проводили с помощью программного обспечения GeneMapper ID-X v1.4 (ThermoFisher Scientific).
Повторяемость. С целью оценки повторяемости получения результатов с использованием тест-системы NPlex контрольные образцы ДНК были амплифицированы 16 раз с последующим анализом результатов генотипирования.
Воспроизводимость. Оценена вариабельность получения результатов с использованием тест-системы NPlex между операторами, временными интервалами (сутки) и используемыми приборами. Каждый оператор независимо соблюдал протокол в отдельные дни и с использованием разного оборудования.
Специфичность. В рамках анализа специфичности оценена перекрестная применимость данной системы на матрицах ДНК других представителей семейства псовые. Также для валидации тест-системы NPlex по аналитической специфичности проведена проверка возможности отжига различных комбинаций праймеров, использованных в тест-системе, на матрицах ДНК других представителей класса млекопитающие. Анализ проводили с использованием базы нуклеотидных последовательностей GenBank и программного продукта Primer-BLAST.
Анализ смесей. С использованием предлагаемой тест-системы были проанализированы образцы, содержащие смесь биологического материала различных особей енотовидной собаки, а также образцы, содержащие смесь ДНК енотовидной собаки и ДНК другого представителя семейства псовые, в различных соотношениях компонентов смеси. Соотношения смесей были следующими: 1 : 1, 1 : 2, 1 : 5, 1 : 15 и 1 : 30, причем мужской образец был второстепенным компонентом.
Чувствительность. Во время валидационных и контрольных исследований тест-система NPlex была проанализирована с использованием мартиц ДНК в разном количестве (0.1, 0.5, 1, 2, 3, 5, 10, 20 нг) и различного качества (ДНК с различным индексом деградации).
Устойчивость. В рамках валидационных мероприятий продемонстрирована устойчивость результатов, полученных с помощью тест-системы NPlex, с использованием образца контрольной ДНК енотовидной собаки. Амплификацию контрольного образца ДНК с ипользованием тест-системы NPlex проводили при 20, 25, 30, 35 и 40 циклах ПЦР в трех повторностях.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Конструирование мультиплексной тест-системы
Уже на первоначальном этапе исследования пять микросателлитных локусов были исключены из последующего анализа по причине отсутствия специфической амплификации (табл. S1 Приложения). На втором этапе оценивался уровень полиморфизма отобранных 34 локусов и их способность к амплификации в мультиплексной системе. Локусы с мономорфным проявлением признака, высокой интенсивностью статтер-фрагментов или препятствующие мультиплексированию были удалены из последующего анализа.
Исключения составили два локуса vWF.x и Nyct10, у которых были выявлены один и два аллеля соответственно. Аллели локуса vWF.x содержат в своей структуре гексануклеотидные тандемные повторы. У енотовидной собаки, лисицы обыкновенной и песца обыкновенного локус имеет мономорфное выявление, у собак и волков – полиморфное. Локус Nyct10 у лисицы обыкновенной проявляется мономорфно, у енотовидной собаки – полиморфно, а у волка, собаки и песца локус не амплифицируется.
Поскольку в Беларуси объектами незаконной охоты могут быть все вышеперечисленные дикие виды животных, а охоту на енотовидную собаку нередко проводят с использованием охотничьих собак, существует высокая вероятность наличия смешанных криминалистических образцов. В таком случае локусы с выраженным межвидовым дифференцирующим потенциалом могут быть полезными компонентами тест-системы в качестве внутреннего молекулярно-генетического контроля видовой принадлежности исследуемого образца.
В результате проведенного секвенирования аллелей отобранных локусов была установлена выраженная размерная гомоплазия с типом тандема (AG)n(GGAA)n в локусе FH3771 и было выявлено большое количество микровариантов в динуклеотидном локусе CPH4. Данные локусы также были исключены из дальнейшей работы. Причины исключения остальных исследованных маркеров более подробно указаны в табл. S1 Приложения.
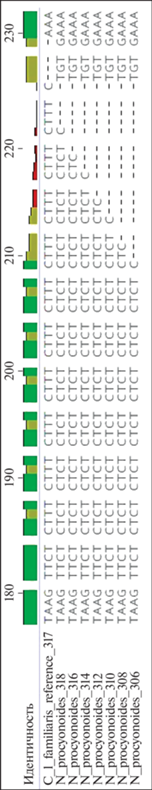
Неожиданный результат был получен при секвенировании аллелей локуса FH2361. При переносе данного маркера на ДНК енотовидной собаки аллели с исходно тетрануклеотидным тандемом (CTTT)n у собаки домашней визуализируются при электрофорезе как динуклеотидные и имеют тандемное строение (CT)n (рис. 1).
Рис. 1.
Участок последовательности локуса FH2361 у собаки домашней (на рисунке C _l_familiaris_reference_317) и у енотовидной собаки (на рисунке N _procyonoides_318, N_procyonoides_316, N_procyonoides_314, N_procyonoides_312, N_procyonoides_310, N_procyonoides_308, N_procyonoides_306).
Таким образом, в результате изучения полиморфизма 39 STR-локусов и трех локусов половой принадлежности было отобрано 14 аутосомных микросателлитных локусов (гексануклеотид vWF.x [23]; три локуса с тетрануклеотидным повтором – FH2096 [24], Nyct10 [2], PEZ17 [25]; два тринуклеотидных локуса – Nyct9, Nyct11 [2]; восемь динуклеотидных – FH2361 [25], NPPM30, NPPM609, NPPM965 [1], Nyct3, Nyct4, Nyct6 [2] и V602 [26]) и два половых (DBX6, DBY7 [27]). Из них девять локусов разработаны для генотипирования енотовидной собаки (Nyct10, Nyct9, Nyct11, NPPM30, NPPM609, NPPM965, Nyct3, Nyct4, Nyct6), шесть локусов – для собаки домашней (vWF.x, FH2096, PEZ17, FH2361, DBX6, DBY7) и один локус – для лисицы обыкновенной (V602).
Типичная электрофореграмма разработанной тест-системы для идентификации енотовидной собаки приведена на рис. 2.
Статистический анализ результатов использования тест-системы
При статистическом анализе результатов генотипирования установлено, что распределение частот гетерозиготных профилей в исследуемой выборке соответствует нормальному (рис. 3).
Рис. 3.
Распределение частот гетерозиготных профилей в выборке енотовидной собаки с использованием тест-системы NPlex (n = 165). а – диапазон значения одного стандартного отклонения (1σ); а + б – двух стандартных отклонений (2σ); а + б + в – трех стандартных отклонений (3σ).
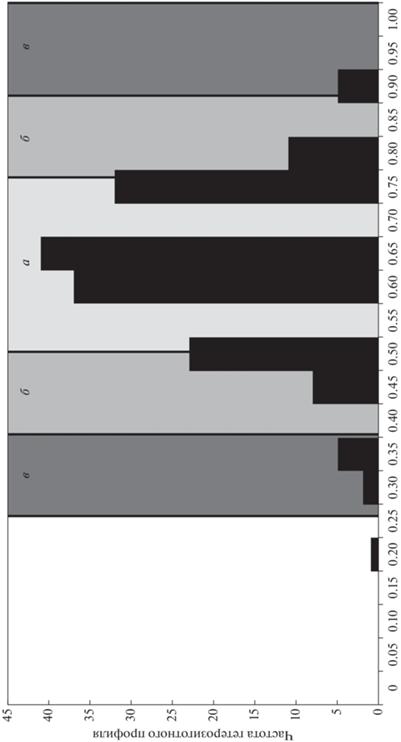
При этом распределение частот гетерозиготных профилей выборки подчиняется правилу трех сигм (σ). На расстоянии одного стандартного отклонения (1σ) от среднего значения 0.658 находятся 66.7% значений из данного распределения (диапазон значений 0.530–0.786, стандартное отклонение σ = 0.127); 95.2% значений лежат на расстоянии двух стандартных отклонений (диапазон 0.402–0.914); 99.4% – не более трех σ (диапазон значений 0.274–1.041). Результаты свидетельствуют, что выборка животных репрезентативна и панель локусов тест-системы NPlex выявляет полиморфизм образцов адекватно.
Одновременно был проведен анализ результатов генотипирования с помощью программ Micro-Checker v2.2.1 и Cervus v3.0.7 на предмет выпадения аллелей, неверной интерпретации результатов генотипирования по причине интенсивных статтер-фрагментов, артефактов ПЦР, “нуль”-аллелей и т.п. Анализ показал отсутствие “нуль”-аллелей или других отклонений у всех исследованных локусов.
Оценка сцепления локусов с использованием программных пакетов GENALEX v6.5 и Arlequin v3.5.1.3 показала отсутствие сцепленного наследования признаков, что свидетельствует о независимом распределении аллелей локусов тест-системы.
Кластерный анализ выборки, проведенный с использованием программы Structure v2.3.4, показал отсутствие подразделения на популяции и признал выборку образцов генетически однородной.
Аллельное распределение в суммарной выборке енотовидных собак для всех исследованных полиморфных локусов соответствовало распределению Харди–Вайнберга (P > 0.05).
Средняя ожидаемая гетерозиготность (НЕ) для микросателлитных локусов, входящих в тест-систему, составила 0.655. Средняя наблюдаемая гетерозиготность (НO) составила 0.657. Детальная информация об исследованных локусах представлена в табл. 1.
Таблица 1.
Характеристика микросателлитных локусов тест-системы NPlex для идентификации особей енотовидной собаки
| Локус | Выявленный размерный диапазон в тандемах | Выявленный истинный диапазон, пн | Na | HO | HE | HWЕ | PIC |
|---|---|---|---|---|---|---|---|
| vWF.X | 2/2 | 121/121 | 1 | Мономорфный локус | |||
| Nyct10 | 8–9 | 188–191 | 2 | 0.503 | 0.500 | ns | 0.375 |
| Nyct11 | 5–8 | 103–112 | 3 | 0.570 | 0.585 | ns | 0.495 |
| NPPM609 | 12–19 | 183–197 | 5 | 0.612 | 0.581 | ns | 0.547 |
| NPPM965 | 9–13 | 220–228 | 5 | 0.412 | 0.389 | ns | 0.357 |
| NPPM30 | 17–23 | 226–238 | 6 | 0.648 | 0.741 | ns | 0.695 |
| Nyct9 | 6–14 | 162–186 | 6 | 0.776 | 0.785 | ns | 0.752 |
| Nyct3 | 16–22 | 237–249 | 7 | 0.636 | 0.691 | ns | 0.652 |
| FH2361 | 14–20 | 306–318 | 7 | 0.463 | 0.436 | ns | 0.409 |
| Nyct4 | 10–23 | 215–241 | 7 | 0.780 | 0.795 | ns | 0.765 |
| FH2096 | 4–10 | 84–108 | 7 | 0.661 | 0.635 | ns | 0.595 |
| Nyct6 | 11–25 | 143–171 | 8 | 0.812 | 0.799 | ns | 0.771 |
| PEZ17 | 9–17 | 192–224 | 9 | 0.818 | 0.768 | ns | 0.737 |
| V602 | 3–14 | 124–146 | 9 | 0.855 | 0.811 | ns | 0.785 |
| DBX6 | X | 246 | 1 | Мономорфный локус | |||
| DBY7 | Y | 117 | 1 | Мономорфный локус | |||
Примечание. * Na – число выявленных аллелей в локусе; HO – наблюдаемая гетерозиготность; HE – ожидаемая гетерозиготность; HWЕ – значимость отклонения от равновесия Харди–Вайнберга; ns – статистически незначимое отклонение от равновесия HWЕ; PIC – показатель информативности генетического полиморфизма.
Согласно D. Botstein et al. [28] PIC для высокоинформативного локуса должен превышать значение 0.5, для достаточно информативного может быть менее 0.5, но обязательно превышать значение 0.25. Показатель PIC для малоинформативного локуса имеет значение менее 0.25. Как следует из табл. 1, минимальные значения PIC выявлены у локусов Nyct10 (0.375), NPPM965 (0.357). Такое значение PIC для локуса Nyct10 обусловлено существованием только двух аллелей, а для локуса NPPM965 (пять аллелей) – сильной выраженностью мажорного аллеля. Максимальное значение PIC выявлено у локуса V602 (0.785), что объясняется большим количеством выявленных аллелей с относительно равномерным распределением в популяции. Среднее значение PIC составило 0.629. Таким образом, полученные нами данные следует считать значимыми для интерпретации результатов и оценки уровня изменчивости.
Суммарно в выборке с использованием тест-системы NPlex были идентифицированы 82 аллеля, частоты встречаемости которых приведены в табл. 2. Наиболее полиморфными оказались локусы V602 и PEZ17, у них было выявлено по девять аллелей. Наименьший уровень полиморфизма показали локусы Nyct10 (два аллеля), Nyct11 (три аллеля). Наиболее выраженные мажорные аллели были обнаружены в локусах NPPM965 – аллель 11 (частота 0.764), FH2361 – 14 (0.735), NPPM609 – 13 (0.612), FH2096 – 5 (0.545) и Nyct3 – 17 (0.482). В локусах Nyct11, NPPM30, Nyct6 и Nyct4 выявлено два и более доминантных аллеля.
Таблица 2.
Частоты встречаемости аллелей локусов тест-системы NPlex в популяции енотовидной собаки
| Аллель | NYCT11 | V602 | NYCT10 | NPPM609 | NPPM965 | NPPM30 | FH2096 | NYCT6 | NYCT3 | PEZ17 | FH2361 | NYCT9 | NYCT4 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 3 | 0.130 | ||||||||||||
| 4 | 0.091 | 0.006 | |||||||||||
| 5 | 0.464 | 0.161 | 0.545 | ||||||||||
| 6 | 0.009 | 0.155 | 0.097 | ||||||||||
| 7 | 0.436 | 0.073 | |||||||||||
| 8 | 0.100 | 0.300 | 0.494 | 0.003 | 0.012 | 0.197 | |||||||
| 9 | 0.097 | 0.506 | 0.094 | 0.015 | 0.003 | ||||||||
| 10 | 0.197 | 0.136 | 0.194 | 0.036 | 0.176 | 0.015 | |||||||
| 11 | 0.003 | 0.764 | 0.209 | 0.091 | |||||||||
| 12 | 0.164 | 0.170 | 0.024 | ||||||||||
| 13 | 0.612 | 0.003 | 0.379 | 0.191 | 0.284 | ||||||||
| 14 | 0.012 | 0.094 | 0.191 | 0.735 | 0.315 | 0.235 | |||||||
| 15 | 0.067 | 0.118 | 0.018 | ||||||||||
| 16 | 0.012 | 0.009 | 0.024 | ||||||||||
| 17 | 0.055 | 0.009 | 0.482 | 0.003 | 0.012 | 0.216 | |||||||
| 18 | 0.282 | 0.300 | 0.152 | 0.073 | |||||||||
| 19 | 0.064 | 0.321 | 0.152 | 0.079 | 0.134 | 0.116 | |||||||
| 20 | 0.024 | 0.209 | 0.003 | ||||||||||
| 21 | 0.064 | 0.061 | 0.073 | ||||||||||
| 22 | 0.264 | 0.070 | 0.006 | ||||||||||
| 23 | 0.015 | 0.045 | 0.061 | ||||||||||
| 25 | 0.191 |
Криминалистическую применимость локусов тест-системы NPlex для идентификационных целей оценивали по таким критериям как вероятность случайного совпадения генотипов двух индивидуумов (P(ID) – для неродственных особей и P(ID)sib – для родственных животных) и частота встречаемости определенного генотипа. Поскольку в исследованной выборке отсутствует подразделение на популяции, расчет показателей проводили на основе полученных данных совокупной выборки для 13 несцепленных локусов. Результаты приведены в табл. 3, при этом перечень локусов отражает убывание их значений P(ID).
Таблица 3.
Значения вероятности случайного совпадения генотипов двух неродственных особей и сибсов для отдельных локусов и их комбинаций
| Локус | P(ID) для одного локуса | P(ID)sib для одного локуса | P(ID) с включением последующего локуса | P(ID)sib с включением последующего локуса |
|---|---|---|---|---|
| NPPM965 | 4.1 × 10–1 | 6.6 × 10–1 | 4.1 × 10–1 | 6.6 × 10–1 |
| NYCT10 | 3.8 × 10–1 | 5.9 × 10–1 | 1.5 × 10–1* | 3.9 × 10–1** |
| FH2361 | 3.5 × 10–1 | 6.2 × 10–1 | 5.2 × 10–2 | 2.4 × 10–1 |
| NYCT11 | 2.6 × 10–1 | 5.2 × 10–1 | 1.4 × 10–2 | 1.3 × 10–1 |
| NPPM609 | 2.1 × 10–1 | 5.1 × 10–1 | 2.9 × 10–3 | 6.5 × 10–2 |
| FH2096 | 1.8 × 10–1 | 4.8 × 10–1 | 5.1 × 10–4 | 3.1 × 10–2 |
| NYCT3 | 1.3 × 10–1 | 4.4 × 10–1 | 6.8 × 10–5 | 1.3 × 10–2 |
| NPPM30 | 1.1 × 10–1 | 4.1 × 10–1 | 7.7 × 10–6 | 5.5 × 10–3 |
| PEZ17 | 8.5 × 10–2 | 3.9 × 10–1 | 6.5 × 10–7 | 2.1 × 10–3 |
| NYCT9 | 7.9 × 10–2 | 3.8 × 10–1 | 5.2 × 10–8 | 8.0 × 10–4 |
| NYCT4 | 7.2 × 10–2 | 3.7 × 10–1 | 3.7 × 10–9 | 3.0 × 10–4 |
| NYCT6 | 6.9 × 10–2 | 3.7 × 10–1 | 2.6 × 10–10 | 1.1 × 10–4 |
| V602 | 6.1 × 10–2 | 3.6 × 10–1 | 1.6 × 10–11 | 3.9 × 10–5 |
| LR | 6.4 × 1010 | 2.5 × 104 |
Примечание. P(ID) – вероятность случайного совпадения генотипов двух индивидуумов для неродственных особей; P(ID)sib – вероятность случайного совпадения генотипов двух индивидуумов для родственных животных; * – здесь и далее произведение P(ID) предыдущих и данного локусов; ** – здесь и далее произведение P(ID)sib предыдущих и данного локусов.
Значение вероятности случайного совпадения исследуемого образца по заданному генотипу было максимальным для локуса NPPM965, а минимальным для локуса V602. Высокий уровень значения P(ID) для локуса NPPM965 (пять аллелей) объясняется сильно выраженным мажорным аллелем 11 (частотой встречаемости 0.764). Минимальное значение случайного совпадения для локуса V602 обусловлено максимальным числом аллелей (9) с относительно равномерным распределением частот в популяции.
Частота встречаемости генотипа в выборке варьировала от 6.3 × 10–17 до 2.5 × 10–10 со средним значением 1.1 × 10–11. Значения P(ID) и P(ID)sib для тест-системы NPlex составляли 1.6 × 10–11 и 3.9 × 10–5 соответственно. Значения LR для неродственных особей и сибсов составили 6.4 × 1010 и 2.5 × 104 соответственно. Учитывая, что численность популяции енотовидной собаки в Республике Беларусь по состоянию на 2020 г. оценивалась в 15 000 особей, сила исключения, обеспечиваемая STR-локусами тест-системы NPlex, позволяет достигать надежного уровня доказательств в криминалистическом ДНК-анализе.
При этом необходимо учитывать, что исследованная выборка была однородной и, как следствие, при расчете вероятности случайного совпадения не были учтены значения FST (коэффициента инбридинга) для данной конкретной популяции енотовидной собаки. В случае использования NPlex для других регионов и мест обитаний енотовидной собаки следует создать соответствующий массив генотипов и оценить уровень субструктурирования популяции. При наличии субструктурирования значения P(ID) и P(ID)sib будут различные для каждой субпопуляции.
Валидация тест-системы NPlex
При разработке тест-системы NPlex синтез праймеров, входящих в тест-систему, проводился в АО “Гентерра” (Российская Федерация).
В результате валидационных мероприятий по исследованию работоспособности тест-системы с коммерческими наборами реагентов различных производителей (ThermoFisher Scientific, США; ОДО “Праймтех”, РБ; ООО “Синтол”, РФ; ЗАО “Евроген”, РФ; ООО “АртБиоТех”, РБ; BioRad, США) установлено, что использование различных реагентов данных производителей на выявляемые параметры системы не влияет.
Повторяемость. При проведении исследований в одинаковых условиях продемонстрирована повторяемость результатов, полученных с помощью тест-системы NPlex.
В каждом параллельном анализе для каждого образца ДНК при использовании тест-системы NPlex выявлены все ожидаемые аллели во всех локусах. Полученные данные указывают на то, что тест-система NPlex стабильно дает результат с высокой повторяемостью.
Воспроизводимость. Для оценки вариабельности исследования тест-системы NPlex было получено 88 результатов. Прецизионность каждого образца, генотипированного тест-системой NPlex, составила 100%.
Специфичность. Тест-система NPlex предназначена для ДНК-идентификации и установления родства животных вида енотовидная собака, обитающих на территории Беларуси. Перекрестная активность тест-системы NPlex при генотипирования ДНК других видов семейства псовые (в части совпадения диапазона выявляемых аллелей других видов семейства псовые с диапазоном енотовидной собаки) представлена в табл. 4.
Таблица 4.
Результаты перекрестной амплификации микросателлитных локусов тест-системы NPlex на матрицах ДНК других видов семейства псовые
| Локус | Vulpes vulpes (лисица обыкновенная) |
Vulpes lagopus (песец обыкновенный) |
Canis lupus lupus (волк обыкновенный) и Canis lupus familiaris (собака домашняя) |
|---|---|---|---|
| vWF.X | В аллельном диапазоне | Вне аллельного диапазона | |
| DBX | Вне аллельного диапазона | В аллельном диапазоне | |
| NPPM609 | n/a | » | |
| Nyct6 | n/a | » | |
| NPPM965 | Вне аллельного диапазона | n/a | |
| Nyct10 | » | n/a | |
| Nyct4 | n/a | В аллельном диапазоне | |
| Nyct11 | Общий аллельный диапазон для всех видов | ||
| V602 | » | ||
| NPPM30 | » | ||
| FH2096 | » | ||
| PEZ17 | » | ||
| FH2361 | » | ||
| Nyct9 | » | ||
| DBY | » | ||
| Nyct3 | n/a | ||
Из приведенных результатов следует, что локусы тест-системы NPlex проявляют значительную перекрестную активность с матрицами ДНК других видов из семейства псовых. Последнее не кажется удивительным, поскольку семейство объединяет виды, связанные общим происхождением в силу филогенетического родства. С учетом перекрестной активности локусов включение в тест-систему NPlex локусов с низкой полиморфностью (vWF.x, Nyct10), но с выраженным межвидовым дифференцирующим потенциалом является оправданным.
При использовании данной тест-системы на матрицах ДНК представителей семейства кошачьи (кошка домашняя, рысь европейская), семейства куньи (хорь лесной, выдра речная, куница лесная, ласка, норка американская, барсук), семейства бобровые (бобр речной), семейства заячьи (заяц-русак, заяц-беляк), отряда парнокопытные (косуля европейская, олень благородный, олень пятнистый, лось, лань европейская, бык домашний, зубр европейский, кабан европейский), отряда непарнокопытные (лошадь домашняя), класса птицы (курица домашняя, гусь серый, индейка) и отряда приматы (человек) продукты ПЦР в используемых локусах не выявляются.
Также с использованием базы нуклеотидных последовательностей GenBank и программного продукта Primer-BLAST было проанализировано суммарно 289 возможных комбинаций праймеров тест-системы NPlex. Установлено, что только одна комбинация потенциально может приводить к неспецифической амплификации: праймер “DBY7 F” дает неспецифическую амплификацию (фрагмент в 173 пн) с праймером “Nyct10 R” на матрице ДНК кабана европейского (Sus scrofa). Полученный результат свидетельствует о высокой аналитической специфичности тест-системы.
Анализ смесей. Образцы, содержащие смесь биологического материала различных особей енотовидной собаки, а также енотовидной собаки и различных представителей семейства псовые могут быть проанализированы с использованием тест-системы. В данном случае интерпретация результатов генотипирования будет в значительной мере зависеть от качественных и количественных характеристик смеси. Тем не менее необходимо отметить, что наличие в ПЦР-смеси примеси ДНК другого псового легко диагностируется ввиду различий в аллельном диапазоне локусов, обладающих дифференцирующим потенциалом.
Чувствительность. Для тест-системы NPlex оптимальное количество ДНК, вносимой в ПЦР, составляет 2 нг. Во время валидационных и контрольных исследований тест-система показала полное выявление генотипа животного в диапазоне от 0.5 до 15 нг ДНК.
Минимальное вносимое количество ДНК, позволяющее получить полный генетический профиль животного, составляет 0.5 нг и является пределом определения тест-системы.
Устойчивость. Тест-система NPlex оптимизирована при использовании 30 циклов амплификации. В рамках валидационных мероприятий продемонстрирована устойчивость результатов, полученных с помощью тест-системы NPlex на образце конторольной ДНК енотовидной собаки. Для тест-системы NPlex получено 15 результатов, демонстрирующих устойчивость исследования. Электрофореграмма результатов амплификации контрольного образца ДНК енотовидной собаки при различном количестве циклов аплификации представлена на рис. 4.
Рис. 4.
Электрофореграмма результатов амплификации контрольного образца ДНК енотовидной собаки при различных количествах циклов амплификации. 40с, 35с, 30с, 25с, 20с – число циклов амплификации. По вертикали – интенсивность флуоресценции.
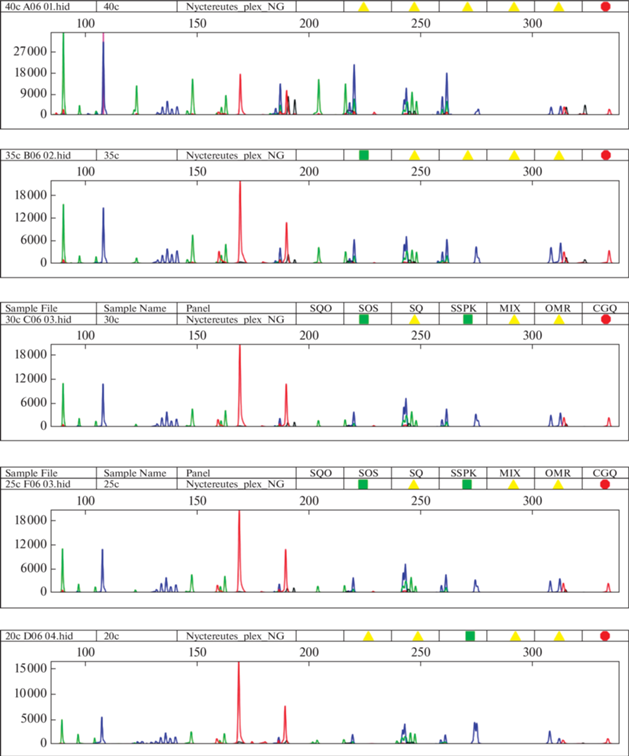
Минимальное количество циклов амплификации, при которых возможно полное выявление генотипа животного с использованием тест-системы NPlex, составляет 20. Оптимальное количество циклов амплификации с использованием тест-системы NPleх составляет 30. При этом увеличение циклов амплификации до 40 не приводит к появлению неспецифических продуктов амплификации, а уменьшение количества циклов до 20 может приводить к выпадению аллелей только в локусах DBX и Nyct9. Остальные локусы NPleх амплифицируются эффективно.
В ходе настоящего исследования был определен спектр STR-локусов, которые обладают достаточным полиморфизмом при работе с ДНК енотовидной собаки, и была разработана мультиплексная тест-система NPlex для идентификации особей енотовидной собаки в криминалистических исследованиях. В ходе экспериментов была проведена оценка полиморфизма, криминалистических параметров локусов, включенных в тест-систему, а также подтверждена тандемная структура аллелей локусов. Разработанная тест-система NPlex обладает высокой повторяемостью и прецизионностью в 100%, имеет высокую аналитическую специфичность и низкий предел обнаружения генетического материала. По результатам генетико-статистического анализа массива генотипов образцов енотовидной собаки все локусы наследуются независимо и соответствуют равновесию Харди–Вайнберга. Разработанная тест-система NPlex может применяться при исследовании вещественных доказательств по делам о незаконной охоте, о нападении диких животных и по иным делам, связанным с участием енотовидных собак.
Итогом разработки тест-системы явилась методика ДНК-идентификации биологических образцов животных вида енотовидная собака (Nyctereutes procyonoides), предназначенная для практикующих судебных экспертов Государственного комитета судебных экспертиз Республики Беларусь и научных сотрудников по профилю исследования. Методика включена в Реестр судебно-экспертных методик и иных методических материалов Государственного комитета судебных экспертиз Республики Беларусь, что соответствует имплементации разработки в национальную правовую систему.
Авторы выражают благодарность Республиканскому государственому общественному объединению “Белорусское общество охотников и рыболовов” и персонально И.С. Юрченко, заведующему отдела экологии фауны Государственного природоохранного научно-исследовательского учреждения “Полесский государственный радиационно-экологический заповедник” за помощь в формировании коллекции биологических образцов.
Подробная информация об используемых последовательностях праймеров и особенностях исследованных локусов, а также номера доступа к последовательностям аллелей в GenBank приведены в табл. S1 и S2 Приложения.
Все авторы участвовали в исследовании и анализе результатов.
Данная работа выполнялась на базе государственного учреждения “Научно-практический центр Государственного комитета судебных экспертиз Республики Беларусь” и финансировалась Национальной Академией наук Республики Беларусь (номер государственной регистрации НИОК(Т)Р: 20190195).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Таблица S1.
Микросателлитные и половые локусы, задействованные в исследовании
| № п/п | Маркер | Вид-источник | Тандем | Результат исследования | Ссылка на источник литературы |
|---|---|---|---|---|---|
| 1 | NPPM30 | N. procyonoides | (TG)n | Включен в тест-систему | Yan S.Q. et al., 2013 |
| 2 | NPPM609 | N. procyonoides | (GT)n | Включен в тест-систему | Yan S.Q. et al., 2013 |
| 3 | NPPM905 | N. procyonoides | (TG)n | Высокое содержание нуль-аллелей | Yan S.Q. et al., 2013 |
| 4 | NPPM941 | N. procyonoides | (TG)n | Нестабильная амплификация в мультиплексной системе | Yan S.Q. et al., 2013 |
| 5 | NPPM965 | N. procyonoides | (GT)n | Включен в тест-систему | Yan S.Q. et al., 2013 |
| 6 | Nyct1 | N. procyonoides | (GT)n | Высокая интенсивность статтеров | Hong Y. et al., 2013 |
| 7 | Nyct3 | N. procyonoides | (TG)n | Включен в тест-систему | Hong Y. et al., 2013 |
| 8 | Nyct4 | N. procyonoides | (TG)n | Включен в тест-систему | Hong Y. et al., 2013 |
| 9 | Nyct6 | N. procyonoides | (GA)n | Включен в тест-систему | Hong Y. et al., 2013 |
| 10 | Nyct7 | N. procyonoides | (CT)n | Нестабильная амплификация в мультиплексной системе | Hong Y. et al., 2013 |
| 11 | Nyct8 | N. procyonoides | (TG)n | Несоответствие распределению Харди–Вайнберга | Hong Y. et al., 2013 |
| 12 | Nyct9 | N. procyonoides | (CTG)n | Включен в тест-систему | Hong Y. et al., 2013 |
| 13 | Nyct10 | N. procyonoides | (GCT)n | Включен в тест-систему | Hong Y. et al., 2013 |
| 14 | Nyct11 | N. procyonoides | (TGC)n | Включен в тест-систему | Hong Y. et al., 2013 |
| 15 | Nyct12 | N. procyonoides | (GCA)n | Нестабильная амплификация в мультиплексной системе | Hong Y. et al., 2013 |
| 16 | CPH4 | C. lupus familiaris | (CA)n | Динуклеотидный повтор с микровариантами | Caniglia R. et al., 2010 |
| 17 | CPH12 | C. lupus familiaris | (CA)n | Мономорфное проявление признака | Caniglia R. et al., 2010 |
| 18 | Ren112I02 | C. lupus familiaris | (TG)n | Нестабильная амплификация в мультиплексной системе | Breen M. et al., 2001 |
| 19 | vWF.x | C. lupus familiaris | (AGGAAT)n | Включен в тест-систему | DeNise S. et al., 2004 |
| 20 | VGL3438 | C. lupus familiaris | (AAAG)n | Сцепленное наследование признака с локусом FH2010 | Magory Cohen T. et al., 2013 |
| 21 | V602 | V.vulpes | (CT)n(CA)16 | Включен в тест-систему | Whiteside H.M. et al., 2011 |
| 22 | PEZ08 | C. lupus familiaris | (GA)n | Нестабильная амплификация в мультиплексной системе | Halverson J. et al., 2005 |
| 23 | PEZ10 | C. lupus familiaris | (GAAA)n | Отсутствие специфической амплификации | Halverson J. et al., 2005 |
| 24 | PEZ12 | C. lupus familiaris | (AAAG)n | Мономорфное проявление признака | Padar Z. et al., 2004 |
| 25 | PEZ16 | C. lupus familiaris | (GAAA)n | Несоответствие распределению Харди–Вайнберга | Halverson J. et al., 2005 |
| 26 | PEZ17 | C. lupus familiaris | (TTTC)n | Включен в тест-систему | Dayton M. et al., 2009 |
| 27 | FH2001 | C. lupus familiaris | (ATCT)n | Выявлено 2 аллельных варианта | Francisco L.V. et al., 1996 |
| 28 | FH2004 | C. lupus familiaris | (TTCT)n | Отсутствие специфической амплификации | Caniglia R. et al., 2010 |
| 29 | FH2010 | C. lupus familiaris | (GAAT)n | Сцепленное наследование признака с локусом VGL3438 | Halverson J. et al., 2005 |
| 30 | FH2016 | C. lupus familiaris | (CTTT)n | Мономорфное проявление признака | Halverson J. et al., 2005 |
| 31 | FH2054 | C. lupus familiaris | (ATCT)n | Мономорфное проявление признака | Halverson J. et al., 2005 |
| 32 | FH2079 | C. lupus familiaris | (GGAT)n | Отсутствие специфической амплификации | Francisco L.V. et al., 1996 |
| 33 | FH2096 | C. lupus familiaris | (AATG)n | Включен в тест-систему | Caniglia R. et al., 2010 |
| 34 | FH2274 | C. lupus familiaris | (CTTT)n | Высокое содержание нуль-аллелей | Verardi A. et al., 2006 |
| 35 | FH2309 | C. lupus familiaris | (GAAA)n | Высокое содержание нуль-аллелей | Dayton M. et al., 2009 |
| 36 | FH2328 | C. lupus familiaris | (AAAG)n | Отсутствие специфической амплификации | Mellersh C.S. et al., 1997 |
| 37 | FH2361 | C. lupus familiaris | (TTTC)n | Включен в тест-систему | Dayton M. et al., 2009 |
| 38 | FH3241 | C. lupus familiaris | (TTCT)n | Несоответствие распределению Харди–Вайнберга | van Asch et al., 2009 |
| 39 | FH3771 | C. lupus familiaris | (AG)n(GGAA)n | Наличие размерной гомоплазии | Zatoń-Dobrowolska M. et al., 2014 |
| 40 | DBX | Hyaena hyaena | – | Включен в тест-систему | Seddon J.M. et al., 2005 |
| 41 | DBY | Hyaena hyaena | – | Включен в тест-систему | Seddon J.M. et al., 2005 |
| 42 | SRY | C. lupus familiaris | – | Перекрытие размерных диапазонов с локусом тест-системы | DeCandia A. et al., 2016 |
Таблица S2.
Номер доступа GenBank, последовательности праймеров и локусов, входящих в тест-систему NPlex
| Локус | Флуорофор | Последовательности праймеров | Номер доступа GenBank |
|---|---|---|---|
| vWF.X | VIC | CTCCCCTTCTCTACCTCCACCTCTAA | OM371048 |
| CAGAGGTCAGCAAGGGTACTATTGTG | |||
| Nyct10 | NED | CTTGCTGCAAATCTCCCATT | OM716956 OM716957 |
| CAAGGAGAGGAGCTGTTTGC | |||
| Nyct11 | FAM | CCAGTCATCCTGCCTTTGTT | OM716958 OM716959 |
| GTGCCCTTGTGGGTTTCTTA | |||
| NPPM609 | FAM | TTTGGGGTCACTCAGATAGGAAG | OM716960 OM716961 |
| TTTTCCAGAAGGGAGAACAGGT | |||
| NPPM965 | FAM | AGAGCAAAGAAACAGGGCTATAG | OM716962 OM716963 |
| GCTGATTTTGTGTTCTGCTCTGT | |||
| NPPM30 | FAM | AGGACTATTTCACGCCTTGTTG | OM716964 OM716965 |
| ATTCCCACCTCAGTGATTACAG | |||
| Nyct9 | PET | CCCTCAATGGTCTTATCCCC | OM716966 OM716967 |
| ACGACCCCTTCATCTGACTG | |||
| Nyct3 | VIC | TGGACAAGGTCACACAGGAA | OM716968 OM716969 |
| ACCCTCCAAGTGTTCACGAC | |||
| FH2361 | NED | GCTTGGAAGGTGAGACTGAATG | OM716970 OM716971 |
| AGCACTTAGAATGTACCAGGCA | |||
| Nyct4 | PET | TGCTTCTGTCTCCCCTGTCT | OM716972 OM716973 |
| AGTTCAGCCGGGTTGTAATG | |||
| FH2096 | VIC | CCGTCTAAGAGCCTCCC | OM716974 OM716975 |
| GACAAGGTTTCCTGGTTCCA | |||
| Nyct6 | VIC | GATCCAGCTGTCACTGCTTT | OM716976 OM716977 |
| GTCTGCTTCTCCCTCTCCCT | |||
| PEZ17 | VIC | CTAAGGGACTGAACTTCTCC | OM716978 OM716979 |
| GTGGAACCTGCTTAAGATTC | |||
| V602 | FAM | CAGCCTGGACTACAATTCTCTTT | OM716980 OM716981 |
| CCCCAAGTCTTTTGTCCAGA | |||
| DBX | FAM | ATGCTGCAGTTTTTCCAGA | OM630073 |
| TACGCTGGGTCTTAGTT | |||
| DBY | NED | GGTCCAGGAGAGGCTTTGAA | OM630074 |
| TGCCATTGTTTAAAAGGAAG |
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Yan S.Q., Li Y.M., Bai C.Y. et al. Development and characterization of polymorphic microsatellite markers for Chinese raccoon dog (Nyctereutes procyonoides procyonoides) // Genet. Mol. Res. 2013. V. 12. P. 6351–6355. https://doi.org/10.4238/2013.December.6.2
Hong Y., Kim K.S., Lee H., Min M.S. Population genetic study of the raccoon dog (Nyctereutes procyonoides) in South Korea using newly developed 12 microsatellite markers // Genes & Genet. Systems. 2013. V. 88. № 1. P. 69–76. https://doi.org/10.1266/ggs.88.69
Paulauskas A., Griciuvienė L., Juknelyte S. et al. Genetic diversity and population structure of raccoon dog (Nyctereutes procyonoides) in invaded areas // XMAT. 2014. P. 78. Antalya, Turkey. https://www.neobiota.eu/wp/wp-content/uploads/NEOBIOTA-2014-Abstract-Book.pdf
Nørgaard L.S., Mikkelsen D.M.G., Elmeros M. et al. Population genomics of the raccoon dog (Nyctereutes procyonoides) in Denmark: Insights into invasion history and population development // Biol. Invasions. 2017. V. 19. № 5. P. 1637–1652. https://doi.org/10.1007/s10530-017-1385-5
Oh S.Y., Kim S.A., Kim J.Y. et al. Detection of antibodies against the rabies virus in Korean raccoon dogs (Nyctereutes procyonoides koreensis) // J. Zoo and Wildlife Med. 2012. V. 43. № 1. P. 174–176. https://doi.org/10.1638/2011-0063.1
Kido N., Itabashi M., Takahashi M., Futami M. Epidemiology of sarcoptic mange in free-ranging raccoon dogs (Nyctereutes procyonoides) in Yokohama // Jap. Veter. Parasitology. 2013. V. 191. № 1–2. P. 102–107. https://doi.org/10.1016/j.vetpar.2012.07.026
Bagrade G., Deksne G., Ozoliņa Z. et al. Echinococcus multilocularis in foxes and raccoon dogs: an increasing concern for Baltic countries // Parasites & Vectors. 2016. V. 9. № 1. P. 1–9. https://doi.org/10.1186/s13071-016-1891-9
Griciuvienė L., Paulauskas A., Radzijevskaja J. et al. Impact of anthropogenic pressure on the formation of population structure and genetic diversity of raccoon dog Nyctereutes procyonoides // Curr. Zool. 2016. V. 62. № 5. P. 413–420. https://doi.org/10.1093/cz/zow038
Kauhala K., Kowalczyk R. Invasion of the raccoon dog Nyctereutes procyonoides in Europe: history of colonization, features behind its success, and threats to native fauna // Curr. Zool. 2011. V. 57. № 5. P. 584–598. https://doi.org/10.1093/czoolo/57.5.584
Lever C. Naturalized Mammals of the World. London: Longman, 1985. Available from: https://www.cabi.org/isc/abstract/19860537058
Deinet S., Ieronymidou C., McRae L. et al. Wildlife comeback in Europe: The recovery of selected mammal and bird species // Final Rep. to Rewilding Europe by ZSL, BirdLife International and the European Bird Census Council. London, UK, 2013. Available from: https://rewildingeurope.com/wp-content/uploads/20-13/11/Wildlife-Comeback-in-Europe-the-recovery-of-selected-mammal-and-bird-species.pdf
Sillero-Zubiri C., Hoffmann M., Macdonald D.W. Canids: Foxes, wolves, jackals, and dogs // Status Survey and Conservation Action Plan. 2004. V. 95. Gland, Switzerland: IUCN, available from: https://www.carnivoreconservation.org/files/actionplans/canids.pdf
Boom R.C.J.A., Sol C.J., Salimans M.M. et al. Rapid and simple method for purification of nucleic acids // J. Clin. Microbiol. 1990. V. 28. № 3. P. 495–503. https://doi.org/10.1128/jcm.28.3.495-503.1990
Benson D.A., Karsch-Mizrachi I., Lipman D.J. et al. GenBank // Nucl. Acids Res. 2005. V. 34. P. D16–D20. https://doi.org/10.1093/nar/gkj157
Hall T., Biosciences I., Carlsbad C. BioEdit: An important software for molecular biology // GERF Bull. Biosci. 2011. V. 2. № 1. P. 60–61. https://www.researchgate.net/profile/Ahmed-Alzohairy/publication/2585-65830_BioEdit_An_important_software_for_molecular_biology/links/0deec528a87d3f2ee0000000/BioEdit-An-important-software-for-molecular-biology.pdf
Peakall R.O.D., Smouse P.E. GENALEX 6: Genetic analysis in Excel. Population genetic software for teaching and research // Mol. Ecol. Notes. 2006. V. 6. № 1. P. 288–295. https://doi.org/10.1093/bioinformatics/bts460
Excoffier L., Lischer H. An integrated software package for population genetics data analysis // Comput. and Mol. Popul. Genet. Lab (CMPG). 2006. Berne, Switzerland: Institute Zool., Univ. Berne, PMID 19325852.
Van Oosterhout C., Hutchinson W.F., Wills D.P., Shipley P. MICRO-CHECKER: Software for identifying and correcting genotyping errors in microsatellite data // Mol. Ecol. Notes. 2004. V. 4. № 3. P. 535–538. https://doi.org/10.1111/j.1471-8286.2004.00684.x
Marshall T.C., Slate J.B.K.E., Kruuk L.E.B., Pemberton J.M. Statistical confidence for likelihood – based paternity inference in natural populations // Mol. Ecol. 1998. V. 7. № 5. P. 639–655. https://doi.org/10.1046/j.1365-294x.1998.00374.x
Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data // Genetics. 2000. V. 155. № 2. P. 945–959. https://doi.org/10.1534/genetics.116.195164
Earl D.A., VonHoldt B.M. STRUCTURE HARVESTER: A website and program for visualizing STRUCTURE output and implementing the Evanno method // Conservation Genet. Res. 2012. V. 4. № 2. P. 359–361. https://doi.org/10.1007/s12686-011-9548-7
Lischer H.E., Excoffier L. PGDSpider: An automated data conversion tool for connecting population genetics and genomics programs // Bioinformatics. 2012. V. 28. № 2. P. 298–299. https://doi.org/10.1093/bioinformatics/btr642
DeNise S., Johnston E., Halverson J. et al. Power of exclusion for parentage verification and probability of match for identity in American kennel club breeds using 17 canine microsatellite markers // Animal Genet. 2004. V. 35. № 1. P. 14–17. https://doi.org/10.1046/j.1365-2052.2003.01074.x
Caniglia R., Fabbri E., Greco C. et al. Forensic DNA against wildlife poaching: Identification of a serial wolf killing in Italy // Forensic Sci. Int. Genet. 2010. V. 4. № 5. P. 334–338. https://doi.org/10.1016/j.fsigen.2009.10.012
Dayton M., Koskinen M.T., Tom B.K. et al. Developmental validation of short tandem repeat reagent kit for forensic DNA profiling of canine biological material // Croatian Med. J. 2009. V. 50. № 3. P. 268–285. https://doi.org/10.3325/cmj.2009.50.268
Whiteside H.M., Dawson D.A., Soulsbury C.D., Harris S. Mother knows best: Dominant females determine offspring dispersal in red foxes (Vulpes vulpes) // PLoS One. 2011. V. 6. № 7. P. e22145. https://doi.org/10.1371/journal.pone.0022145
Seddon J.M. Canid-specific primers for molecular sexing using tissue or non-invasive samples // Conservation Genet. 2005. V. 6. № 1. P. 147–149. https://doi.org/10.1007/s10592-004-7734-9
Botstein D., White R.L., Skolnick M., Davis R.W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms // Am. J. Hum. Genet. 1980. V. 32. № 3. P. 314. PMID 6247908.
Дополнительные материалы отсутствуют.



