Генетика, 2023, T. 59, № 5, стр. 493-506
Эуполиплоидия как способ видообразования у растений
А. В. Родионов *
Ботанический институт им. В.Л. Комарова Российской академии наук
197376 Санкт-Петербург, Россия
* E-mail: avrodionov@mail.ru
Поступила в редакцию 17.07.2022
После доработки 01.09.2022
Принята к публикации 12.09.2022
- EDN: DRWNGX
- DOI: 10.31857/S0016675823050119
Аннотация
При описании явлений, связанных с событиями полногеномной дупликации геномов (WGD), используют термины неополиплоид, мезополиплоид и палеополиплоид в их современной “пост-геномной” интерпретации. По нашему мнению, в потоке меняющихся состояний генома между неополиплоидами и палеополиплоидами имеет смысл выделить стадию эуполиплоида – такое состояние полиплоида, когда его полиплоидная природа не вызывает никаких сомнений, но геном (кариотип) эуполиплоида в отличие от неополиплоида уже относительно стабилен. В состоянии эуполиплоида находится большинство геномов/кариотипов многочисленных полиплоидных видов растений, полиплоидная природа кариотипа которых не вызывает сомнений у исследователей – генетиков, кариологов и флористов. Факультативно эуполиплоиды могут вступать в новые раунды межвидовой гибридизации с сохранением у гибрида уровня плоидности родителей или с возникновением аллополиплоида более высокого уровня плоидности. Эуполиплоидизация генома – это радикальный и быстрый способ видо- и родообразования у растений. Таким путем возникли десятки тысяч видов современных растений. Удачные сочетания аллелей субгеномов эуполиплоида, характерные для высоких полиплоидов крупные размеры, частый переход к неполовому размножению могут способствовать успешному освоению эуполиплоидами новых ареалов, адаптации к экстремальным условиям существования на краю ареалов, но не обретению новых ароморфозов – это видообразование, но видообразование на уже освоенном уровне эволюционной сложности, шаг, не ведущий сам по себе к прогрессивной эволюции.
По оценкам кариосистематиков около половины видов цветковых растений полиплоиды [1]. Только в родах, где есть и диплоиды и полиплоиды и потому определение плоидности не вызывает сомнений, полиплоидных видов не менее 15%. Причем в родах с низким основным числом хромосом х = 2–7 доля полиплоидов существенно больше – до 50% [2]. Результаты сравнительного исследования групп сцепления представителей всех основных филогенетических ветвей наземных растений показали, что следы одного или нескольких актов полногеномной дупликации (WGD) есть в геномах всех исследованных в этом отношении голосеменных и покрытосеменных растений [3–5]. Поэтому удивляет не то, что полиплоидов много, а то, что есть много – около 50–85% – растений, у которых кариотип выглядит так, словно они диплоиды.
То, что один или несколько актов WGD имели место в эволюции геномов всех голосеменных и покрытосеменных растений, не кажется тривиальным. У представителей других филогенетических ветвей, сестринских ветви семенных растений (Spermatophyta), WGD выявляется не всегда. Не видно следов WGD в геномах предков многоклеточных наземных растений – харовых водорослей [6]. Дупликаций – последствий WGD не обнаружено в геномах плаунка Selaginélla [7], антоцеротового мха Anthoceros angustus [8], печеночного мха Marchantia polymorpha [9]. Напротив, предки настоящих мхов (Bryophyta), папоротников и гомоспоровых плауновидных, так же как цветковые растения, пережили по крайней мере один акт WGD в своей истории [10]. Примечательно, что около 70% зарегистрированных актов WGD у предков современных растений непосредственно предшествовали периоду экологической катастрофы на границе мелового периода и есть основания думать, что какие-то особенности появившихся тогда неополиплоидных геномов помогли их обладателям адаптироваться к новым экологическим условиям [3, 11].
WGD – АВТО- ИЛИ АЛЛОПОЛИПЛОИДИЗАЦИЯ?
Событие WGD может произойти двумя различными способами – может произойти удвоение генома вида, такой полиплоид с тремя и более идентичными геномами (AAA, AAAA, …) называют автополиплоидом, в то время как полиплоид, возникший после межвидовой гибридизации в результате удвоения близких, но неодинаковых, гомеологичных геномов (AABB, AABBCC и т.п.), именуют аллополиплоидом. Как часто встречаются авто- и аллополиплоды в природе – неизвестно. Оценки разных авторов, даже когда речь идет о хорошо изученных семействах, значительно различаются (сравни: [12, 13]). Казалось бы, различия между автополиплоидами и аллополиплоидами очевидны – мультипликация генома одного вида в первом случае и мультипликация геномов как следствие объединения в одном ядре (геноме) субгеномов нескольких разных видов – во втором. Есть два основных методических подхода для дифференциации авто- и аллополиплоидов: таксономический и кариологический. Полиплоид относят к автополиплоидам, если опытный систематик считает, что диплоидные предки полиплоида были растениями одного вида, – это таксономический метод дифференциации. Не менее основательный вывод может быть сделан при кариологическом исследовании: если в мейозе I при конъюгации хромосом образуются мультиваленты – это автополиплоид, если хромосомы конъюгируют попарно, мультивалентов нет – аллополиплоид.
Проблема в том, что следует считать разными видами в мире растений. В ходе дискуссий о границах видов предложено различать виды “биологические” – репродуктивно изолированные друг от друга [1, 14, 15] и виды “морфологические” или “таксономические” – природные популяции, обладающие большими или меньшими морфологическими особенностями (морфологическим гиатусом) и определенным ареалом [1, 16–18]. Сравнительно часто морфологически хорошо различаемые виды растений способны при скрещивании давать жизнеспособное, полностью или частично фертильное потомство. Показано, что возможность давать жизнеспособные гибриды может сохраняться у географически разобщенных видов растений миллионы и десятки миллионов лет [18]. Когда вследствие изменений климата или деятельности человека ареалы “таксономических” видов смыкаются, эти виды достаточно часто вполне успешно могут скрещиваться, давая фертильное, часто полиплоидное потомство. Этот путь появления полиплоидных видов Дж.Л. Стеббинс назвал “the secondary contact hypothesis” [19, 20]. “Таксономические” виды в последнее время чаще всего выделяют на основе сочетания их морфологических особенностей и ДНК-штрихкода, подразумевая, что само по себе наличие своеобразного набора признаков и различий в ДНК-штрихкодах есть основание для предположения о наличии репродуктивного барьера, дополнительные доказательства существования которого в этом случае не требуются [21–23].
Поскольку вопрос, “что перед нами – авто- или аллополиплоид?” априорно означает, что мы, возможно, имеем дело с результатом межвидового скрещивания, следовательно “родителей” предполагаемого аллополиплоида нельзя отнести к “биологическим” видам на основе бесспорного критерия биологического вида – его репродуктивной изоляции. Остается предположить, что события межвидовой гибридизации, ведущие к появлению аллополиплоидов, происходят при участии “таксономических” или “морфологических” видов. Однако границы “таксономических” видов тоже вызывают дискуссию. Проблема в том, что некоторые таксономически значимые признаки растений определяются моногенно. Например, виды Avena clauda и A. eriantha хорошо различаются по морфологии цветка и колоска, но различия эти определяются аллелями одного гена. Оба вида можно видеть в смешанных популяциях и известно, что гибриды между ними образуются легко и они фертильны. Это пример двух “хороших” морфологических видов, однако с генетической точки зрения их видовой статус необоснован [15]. Противоположная ситуация с видами A. damascena и A. prostrata – по морфологии их трудно различить, однако они репродуктивно изолированы друг от друга [15]. По подсчетам Л. Райсберга и соавт. [24] только 70% “таксономических” видов репродуктивно изолированы. Частоту встречаемости репродуктивно изолированных, но морфологически неразличимых, криптических видов растений в настоящее время подсчитать невозможно [25, 26].
К нерешенным вопросам, имеющим отношение к таксономическому критерию авто- или аллополиплодии, относится проблема таксономического статуса циторас – растений, близких друг к другу по морфологии, но имеющих разные уровни плоидности. Это один вид или несколько разных видов? Как правило, циторасы автополиплоидного происхождения не имеют существенных морфологических различий, кроме, может быть, размеров клеток и размеров самого растения. Их лишь в редких случаях, когда ареалы циторас различаются, относят к разным видам [26, 27]. Напротив, циторасы аллополиплоидного происхождения часто относят к разным видам и даже родам, даже тогда, когда морфологические различия между природными популяциями с разной композицией генома/кариотипа не очевидны [25–28].
Кариологический критерий гибридного происхождения полиплоида независим от экспертной оценки таксономистами-морфологами видового ранга его предков. Согласно ему вид считается аллополиплоидом, если у него нет аллосиндеза (конъюгации хромосом в мейозе I с образованием мультивалентов) и дисомное наследование аллелей. Напротив, для автополиплоидов характерны мультиваленты в мейозе I и полисомное наследование аллелей [13, 29]. Но получение и выращивание на экспериментальном поле межвидовых гибридов и исследование их хромосом в мейозе – трудоемкое, долговременное, плохо укладывающееся в современную практику планирования и финансирования научных проектов занятие. Крайне редко подобное исследование затрагивает объекты вне узкого круга модельных объектов генетики растений. Среди представителей 47 семейств растений Ж. Ли с соавт. [30] выявили только 208 полиплоидных видов растений, у которых хотя бы однажды было исследовано поведение хромосом в мейозе. Среди них, учитывая интегрированные филогенетические, цитологические и генетические данные, 118 видов были классифицированы авторами как аллополиплоиды, а 90 – как автополиплоиды [12, 30]. Однако в этой выборке только 92 вида демонстрировали строго бивалентное спаривание хромосом в мейозе I, в то время как у 116 видов в профазе мейоза формировались мультиваленты (по крайней мере один мультивалент). Что важно: среди полиплоидов, которые рассматривались авторами исследования как аллополиплоиды, 48% видов демонстрировали строго бивалентное спаривание, а у 52% видов наблюдались один или много мультивалентов. Среди автополиплоидов соответствующие частоты были равны 39 и 61% [30]. Как и ожидалось, частота строго бивалентного спаривания выше у аллополиплоидов, чем у автополиплоидов, однако различия между сравниваемыми типами полиплоидов незначительны. Высокая частота встречаемости мультивалентов у видов, которые считались аллополиплоидными, и частота строго бивалентного спаривания у автополиплоидов заставляют или отказаться от критерия “строго бивалентное спаривание” как индикатора аллополиплоидной природы полиплоида, или признать, что интегрированные “phylogenetic, cytological, and genetic data”, использованные авторами для дифференциации авто- и аллополиплоидов [12, 30], не позволяют корректно различить алло- или автополиплоиды.
Как результат, на сегодняшний день роль автополиплоидии в процессах видообразования у растений остается неясной или не вполне обоснованной [26, 27, 29–31]. Как правило, автополиплоид значительно в меньшей степени, чем аллополиплоид, отличается от своих диплоидных предков по морфологии [26, 27], по транскриптому [32, 33] и протеому [34]. Напротив, есть основания предполагать, что активное видообразование, появление таксономически значимых новаций в морфологии и физиологии предков современных дикорастущих растений, возникновение и/или целенаправленное создание многочисленных сельскохозяйственных культур были связаны с аллополиплоидами – полиплоидизацией геномов, сопровождавшей отдаленную гибридизацию [3, 11–13, 29, 35, 36].
НЕОПОЛИПЛОИДЫ, МЕЗОПОЛИПЛОИДЫ, ЭУПОЛИПЛОИДЫ, ПАЛЕОПОЛИПЛОИДЫ
При описании явлений, связанных с появлением нового полиплоида и его последующими трансформациями, используют предложенные С. Фаварже [37] термины неополиплоид, мезополиплоид и палеополиплоид в их современной “пост-геномной” интерпретации [38]. При этом неополиплоидом называют недавно возникшие полиплоиды с кариотипом (геномом), число хромосом в котором есть результат сочетания полных наборов хромосом (субгеномов) предковых диплоидных видов. Термин палеополиплоид используется при описании генома (кариотипа), у которого морфология хромосом и поведение хромосом в мейозе как у диплоида, а полиплоидное прошлое палеополиплоида становится очевидным только после анализа последовательностей ядерного генома. Мезополиплоидные виды демонстрируют диплоидоподобный мейоз, дисомное наследование аллелей, имеют уменьшенное в сравнении неополиплоидным предком, часто очень низкое “квазидиплоидное” число хромосом, однако родительские субгеномы в геноме мезополиплоида все еще можно различить цитогенетически или молекулярно-филогенетически [38]. По нашему мнению, в этом потоке меняющихся состояний генома между неополиплоидами и палеополиплоидами имеет смысл выделить стадию эуполиплоида (от эу, др.-греч. εὖ – “хорошо”, хороший, добротный; настоящий, подлинный, истинный) – такое состояние полиплоида, когда его полиплоидная природа не вызывает никаких сомнений (кариотип представляет собой сочетание кариотипов родителей), но геном (кариотип) эуполиплоида в отличие от неополиплоида уже относительно стабилен [39, 40].
ЭУПОЛИПЛОИДЫ: САЛЬТАЦИОННОЕ ВИДООБРАЗОВАНИЕ ПУТЕМ МОРФОЛОГИЧЕСКОЙ ДЕСПЕЦИАЛИЗАЦИИ И ЧАСТИЧНОЙ РЕПРОДУКТИВНОЙ ИЗОЛЯЦИИ
В состоянии эуполиплоида находится большинство геномов/кариотипов многочисленных полиплоидных видов растений, полиплоидная природа кариотипа которых не вызывает сомнений у исследователей флоры. Среди цветковых растений полиплоидные геномы чаще появлялись в сем. Poaceae; тоже часто, но в 3 раза реже, чем у злаков, в семействах Montiaceae и Brassicaceae. Очень редко акты полиплоидизации имели место в порядке Arecales [41]. К типичным эуполиплоидам относятся многочисленные аллотетраплоидные виды с упорядоченным, диплоидного типа расхождением хромосом в мейозе. Такие полиплоиды называют амфидиплоидами (термин введен М.С. Навашиным [42: с. 136]).
Проблемы с расхождением хромосом в мейозе и митозе у неополиплоидов связаны с важной особенностью центромеров эукариот – правильно работающий кинетохор формируется как многокомпонентный нуклеино-протеиновый комплекс, в основе которого лежит взаимодействие быстро изменяющейся в ходе дивергенции таксонов центромерной ДНК и тех доменов центромерных белков CenH3 и CENP-C, которые взаимодействуют с центромерной ДНК и тоже быстро изменяются в эволюции [43, 44]. Способность связываться с центромерной ДНК другого вида у центромерных белков может сохраняться долго. Показано, например, что CenH3 Lepidium и Zea могут взаимодействовать с центромерной ДНК Arabidopsis, образуя работающие центромеры, разводящие хромосомы Arabidopsis в дочерние клетки, но в условиях конкуренции, когда в геноме присутствуют и CenH3 Lepidium и CenH3 Arabidopsis, хромосомы с “гетерогенными” кинетохорами расходятся хуже, часто попадают в микроядра и элиминируются [45]. Стабилизация расхождения хромосом у эуполиплоида может быть достигнута утратой центромерной ДНК одного из родителей, как это наблюдается у аллотетраплоидной Zingeria trichopoda [46], и/или гомогенизацией генов CenH3 в сторону одного из родителей [47].
Потери хромосом в мейозе нео- и мезополиплоидов связаны не только с несовершенством “бастардных” центромер, но и с аберрантным спариванием хромосом в мейозе. Мультиваленты у автополиплоидов и аллополиплоидов повышают вероятность неравного расхождения хромосом в мейозе I, появления зигот и эндосперма с несбалансированными хромосомными наборами [29]. Отбор к повышению фертильности полиплоида должен идти в направлении хромосомных наборов, в которых гомологичные и гомеологичные хромосомы преимущественно или исключительно конъюгировали бы с образованием бивалентов [12, 48, 49]. Несколько разных механизмов работают для достижения этого результата. Прежде всего предпочтительному спариванию гомологов, а не гомеологов, способствуют предусловия мейоза. В интерфазе в соматических клетках [46, 50, 51] и в премейотической интерфазе у полиплоидов [52] субгеномы разного происхождения в большей или меньшей степени пространственно разделены.
Преимущественному спариванию гомологов перед гомеологами способствует и то, что при дивергенции предковых видов аллополиплоида в хромосомах каждого вида накопились микроинверсии, такие, как найдены в геноме Triticum [53] и Oryza [54]. Не менее важно то, что за время дивергенции предков аллополиплоидов в их геномах происходили взрывная амплификация транспозонов одних семейств (новых или ранее малочисленных) и исчезновение всех или большей части последовательностей из семейств транспозонов общего предка. Такой сценарий изменений паттерна умеренных повторов в геноме называют моделью “рождение–и–смерть” [55]. Например, из 33 семейств ретротранспозонов в геноме вида Oryza punctata шесть семейств, занимающих 25 млн пн (6.3% генома), не найдены в геномах других видов риса [54]. Отслеживание судьбы 75 ортологичных локусов LTR-содержащих ретротранспозонов у восьми наиболее близкородственных видов из группы родства O. sativa показало, что эти последовательности теряются из генома со средней скоростью 3620 пн/млн лет на транспозон. Экстраполируя эти данные на весь геном, можно предсказать, что если не будет следующего взрыва транспозиций, то геном O. sativa ssp. japonica уменьшится на четверть своего размера в течение следующих 3–4 млн лет [54].
Процессы преимущественного спаривания гомологичных хромосом в мейозе аллополиплоидов находятся под генетическим контролем [49, 56]. Аллели, супрессоры спаривания гомеологичных хромосом, обнаружены у диплоидных предков полиплоидов в разных подсемействах растений, что неудивительно, поскольку мы знаем, что все они прошли через несколько актов WGD. По-видимому, неополиплоид в большинстве случаев получает их от своих диплоидных (палеополиплоидных) предков, но известны случаи, когда бивалентное спаривание хромосом объяснялось новыми мутациями [56–58]. Иногда переход от мультивалентного к бивалентному спариванию у неополиплоидов происходит настолько быстро, что заставляет предполагать эпигенетическую регуляцию процесса [56].
Второй этап, на котором исправляются ошибки спаривания гомологов, относится ко времени формирования синаптонемного комплекса (СК), когда образование СК между гомологами идет эффективно, а локальные участки СК, появившиеся было между гомеологами, разбираются [49, 59, 60].
Помимо проблем с расхождением хромосом, у неополиплоидов и мезополиплоидов в соматических клетках наблюдается повышенная частота фрагментов хромосом и межгеномных транслокаций [61, 62]. Существует точка зрения, что межгеномные транслокации являются одним из механизмов преодоления ядерно-цитоплазматической несовместимости неополиплоидов [63, 64]. Вероятность межгеномных транслокаций затруднена тем, что субгеномы разного происхождения в интерфазном ядре у аллополиплоидов находятся в разных компартментах [46, 50, 51]. Можно предположить, что это же обстоятельство в какой-то степени ограничивает межгеномную миграцию транспозонов и иных рассеянных повторов. В существовании такого ограничения нас убеждает феномен GISH-гибридизации, при которой общегеномная ДНК одного предполагаемого “предка” гибридизируется с хромосомами только одного субгенома, но не с другими (см., например: 46, 65]). Как долго сохраняются различия в наборах рассеянных повторов между субгеномами у аллополиплоида в разных семействах – неизвестно. У тетра- и гексаплоидных овсов, сформировавшихся 8–10 млн лет назад [66], выявляемое с помощью GISH различие между A- и C-субгеномами по рассеянным повторам сохранилось [67], а у Nicotiana nesophila, возникшего около 4.5 млн лет назад, предками которого, как полагают, являлись N. sylvestris и N. obtusifolia, GISH уже не выявляет различия между субгеномами [68].
Факультативно эуполиплоиды могут вступать в новые раунды межвидовой гибридизации с сохранением у гибрида уровня плоидности или с появлением аллополиплоида более высокого уровня плоидности [69, 70]. Показано, что у полиплоидов высоких порядков постзиготическая изоляция циторас часто выражена слабее, чем при гибридизации диплоидов и тетраплоидов [71]. Среди полиплоидов высоких порядков часто встречаются разные варианты анеуплоидий, не коррелирующие с морфологическими гиатусами и интерпретируемые как проявление внутривидового разнообразия циторас. Неполная постзиготическая изоляция имеет следствием непрерывный поток аллелей между циторасами среди полиплоидов высоких порядков, что замедляет дивергенцию морфотипов у многохромосомных полиплоидов [71]. Таким путем возникают целые комплексы факультативно перекрестно-опыляющихся природных рас, отчасти апомиктических или самоопыляющихся “микровидов”, которые невозможно разделить на какие-либо “крупные” (“морфологические”) виды [17, 72]. Такая ситуация имеет место во многих родах злаков, например у Poa, Elymus, Leymus, Thinopyrum [73].
Появление вследствие межвидовой гибридизации эуполиплоидов с разными уровнями полиплоидии — это радикальный и быстрый способ видообразования. Таким путем возникли десятки тысяч видов современных растений [41, 74]. Так, в результате гибридизации между тетраплоидным североамериканским видом (2n = 4x = 32) Symphyotrichum boreale (Asteraceae) и гексаплоидом (2n = = 6x = 48) S. novi-belgii появился редкий эндемичный вид – декаплоид (2n = 10x = 80) S. anticostense [75]. Наивысшие известные уровни полиплоидизации среди цветковых – 16× у Artemisia (Asteraceae) [76] и Primula (Primulaceae) [77], 24× у Tradescantia (Commelinaceae), 28× у Eleocharis и 32× у Cyperus (оба Cyperaceae) [78]. Относительно часто встречаются высокие полиплоиды у злаков: 18× у Avenula adsurgens [79], Arundo donax [80] и Anthoxanthum amarum [81], 20× у Helictochloa lusitanica [69], 36× у Andropogon barbinodis [82], 38× у Poa litorosa [83].
Анализ чисел хромосом в кариотипах около 30 тыс. видов цветковых растений, относящихся к 147 семействам 46 порядков, показал, что недавно возникшие полиплоиды (эуполиплоиды) есть во всех порядках и в 139 из 147 семейств11 [41].
Tипичный пример эуполиплоида – кариотип и геном сформировавшегося около 9 тыс. лет назад гексаплоидного вида Triticum aestivum (2n = 42, x = 7, BBAADD), возникшего в результате объединения геномов тетраплоидной полбы (эммера) T. turgidum (2n = 28, ВВАА) и генома диплоидного (палеополиплоидного) вида Aegilops tauschii (2n = 14, DD). Это был только последний из раундов полиплоидизации, приведший к появлению вида T. aestivum. Примерно 500–800 тыс. лет назад ему предшествовало появление тетраплоидного генома эммера в результате гибридизации двух диплоидных (палеополиплоидных) видов: со стороны растения-опылителя, донора субгена А, это был вид, похожий на T. urartu, но дивергировавший от T. urartu на ~1.3 млн лет ранее, и материнского растения-донора субгенома B – вида, родственного Ae. speltoides, но дивергировавшего от общего предка с Ae. speltoides за ~4.5 млн лет до этого [84–87]. Подчеркнем, что геном и кариотип современных линий и сортов T. aestivum выглядят стабильными, но кариотип ресинтезированных аллополиплоидов с геномной конституцией BBAADD в первых поколениях в той или иной степени (в зависимости от линейной принадлежности родительских видов) нестабилен – в этот момент идет потеря части дуплицированных последовательностей протеин-кодирующих генов, реорганизация повторов (репитома). Однако, как правило, через несколько поколений геном стабилизируется, спаривание хромосом в мейозе идет без образования мультивалентов [85, 86].
Сходство групп сцепления трех субгеномов современных T. aestivum между собой и с группами сцепления диплоидных предков (макросинтения) высокое, однако в среднем через каждые 75 млн пн в геноме мягкой пшеницы встречаются микроинверсии со средней длиной 10.5 млн пн, преимущественно лежащие в обедненных генами районах [53]. По числу генов субгеномы A, B и D примерно равны (35 345, 35 643 и 34 212 генов соответственно). Не различаются они существенно и по числу копий транспозонов разных классов. После полиплоидизации в геноме T. aestivum экспансии транспозонов не происходило [53]. Однако в геноме пшеницы шли и идут процессы постепенной геномной диплоидизации (“фракционирования генома” [38, 48]), но идут они равномерно, без дискриминации одного из субгеномов – в полиплоидном геноме T. aestivum каждый из субгеномов уже потерял примерно по 10% предковых генов [53]. Возможно это, как раз, и есть показатель квазистабильного состояния генома эуполиплоида. Несколько иная ситуация в аллополиплоидном геноме Avena sativa (кариотип AACCDD) – у овса в субгеноме С на 12% генов меньше, чем в субгеномах А и D, однако гены эти не утрачены, а транслоцированы из богатого генами участка С-субгенома длиной не менее 226 млн пн в хромосомы A и D-субгеномов – т.е. геном гексаплоидного овса также находится в квазистабильном состоянии [87].
Детально изучавшие кариотипы и процессы видообразования Пшеницевых А. Лёве [88, 89] и Д.Р. Дьюи [90] пришли к выводу, что новые рода и виды у Triticeae образуются путем межвидовой гибридизации и полиплоидизации через появление все новых и новых комбинаций субгеномов. А. Лёве [88, 89] предложил считать родом группу близкородственных видов, имеющих или специфический диплоидный геном, или особую, только для этого рода характерную комбинацию субгеномов. При этом автополиплоидное умножение генома или субгенома не считалось достаточным для того, чтобы относить автополиплоидные комбинации в разные рода (например: кариотипы JJHH, JJJJHH, JJJJHHHH – кариотипы видов одного рода). Привлекательная с генетической точки зрения идея близка к предложению Л.Н. Делоне [91] различать незначительные, микроэволюционные, идущие в пределах рода изменения размеров и морфологии хромосом и макроэволюционные, существенные реорганизации хромосомных наборов. “Кариотип Делоне” – это таксономическая единица, группа видов, отличающаяся по морфологии хромосомного набора от другой группы видов, другого “кариотипа Делоне” [91].
А. Лёве [89] выделил у Пшеницевых 24 варианта диплоидных геномов, обозначенных латинскими буквами от А до Z, и 15 вариантов полиплоидных геномов, в формировании которых принимали участие 13 из 24 возможных первичных диплоидных геномов. В предлагаемой А. Лёве системе количество родов в трибе Пшеницевые возросло, при этом, руководствуясь геномным критерием рода, пришлось раздробить некоторые общеизвестные традиционные рода, такие как Triticum, на несколько: T. monococcum и T. urartu с кариотипом АА вошли в род Crithodium, T. durum (AABB) отнесли в род Gigachilon, T. aestivum (BBAADD) – род Triticum. Геном Hordeum vulgare и H. bulbosum был обозначен как I, геном остальных представителей рода Hordeum, диплоидов и полиплоидов, был обозначен как H [89, 92]. Со временем букв английского алфавита для обозначения вариантов геномов стало не хватать, в частности оказалось, что у видов H. murinum другой первичный (диплоидный) геном – он был обозначен как Xu, геном видов H. gussoneanum и H. marinum назван Xa-геномом [92, 93]. Некоторые иные модификации обозначения геномов можно найти в статье [92].
В качестве примера можно привести три акта видо/родообразования у пшеницевых [90] (рис. 1).
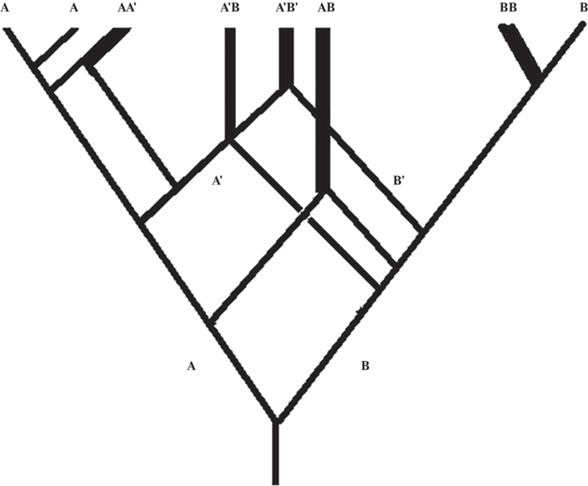
Обсуждая особенности видо- и родообразования у растений, следует обратить внимание на то, что одни и те же комбинации субгеномов (один и тот же род) могут возникать неоднократно в разное время и в разных точках ареалов (рис. 2). Так, дизъюнкция ареалов эуполиплоидных видов в некоторых случаях может быть следствием независимого происхождения полиплоидных природных рас со схожей комбинацией субгеномов на разных территориях, на разных континентах.
Рис. 2.
Один и тот же эуполиплоидный вид/род может возникать неоднократно в разное время и в разных точках своего ареала.
Последовательное применение в таксономии геномной концепции рода часто не совпадало с дискретностью морфологических признаков, считавшихся таксономически значимыми в систематике Злаков, не поддерживалось или поддерживалось, но с оговорками и исключениями, опытными систематиками растений [28, 94–97]. Однако неслучайно закон гомологических рядов Н.И. Вавилова [98, 99] был открыт, прежде всего, в работе с Пшеницевыми – при углубленном анализе морфологических вариаций у каждого вида злаков можно выявить едва ли не весь комплекс признаков, наблюдавшихся у других видов данной трибы. Лишь некоторые из признаков видоспецифичны и только их можно рассматривать как таксономически значимые радикалы [98, 99]. Особенно это заметно, когда анализируются морфологические признаки полиплоидов, как правило отличающихся удивительным морфологическим разнообразием [28, 100].
Р.В. Камелин [96: c. 98, 100] справедливо заметил, что установление геномного состава видов путем попарного скрещивания и трудоемкого анализа поведения хромосом в мейозе 1 [90, 97, 101] — задача трудно разрешимая, для многих природных видов едва ли возможная. Поэтому в первом десятилетии XXI в. предложенная Лёве концепция разграничения родов Пшеницевых (и не только Пшеницевых) по геномному критерию родов не казалась Р.В. Камелину и другим ботаникам-систематикам убедительной, поскольку геномы полиплоидных злаков сложны по составу, а в 2005 г. не было уверенности, что первичные геномы диплоидов и субгеномы полиплоидов определены однозначно правильно и что эволюция современных известных нам диплоидов и полиплоидов шла от диплоидов к полиплоидам, а не наоборот. Не зная результатов полногеномного секвенирования, Р.В. Камелин [96] вполне резонно допускал то, чему сейчас есть сравнительно-геномные доказательства: геномы, которые представляются нам первичными диплоидами, могут в действительности являться недавними дериватами полиплоидных геномов.
Но, с другой стороны, прав был и выдающийся агростолог Н.Н. Цвелев [28], 30 лет назад писавший о возможностях практического применения геномного критерия: “в настоящее время они [возможности], конечно, очень ограничены, так как геномный анализ [методами Кихары и Дьюи] требует проведения очень трудоемких исследований. Однако то, что невозможно теперь, со временем может стать возможным. Вероятно будет разработана новая методика, значительно облегчающая генетический анализ, который, несомненно, очень перспективен. Не исключено, что в будущем станет возможным использование содержащейся в геномах избыточной информации, что откроет широкие возможности для создания новых таксонов, а может быть и для воссоздания уже вымерших таксонов, геномы которых сохранились в хромосомных наборах ныне живущих видов, но не используются ими в онтогенезе”. Н.Н. Цвелев полагал [28, 100], что геномный критерий родов заслуживает внимания хотя бы потому, что в настоящее время однозначных синапоморфий для построения системы родов Пшеницевых на основании таксономически убедительных морфологических гиатусов или репродуктивной изоляции все равно не найдено, поэтому кажется вполне возможным, что новые и будущие методики исследования геномов вполне могут сделать геномный критерий родов инструментом в руках систематика-практика [28].
На важное обстоятельство, во многом определяющее особенности морфологии диплоидных и полиплоидных злаков, обратил внимание Н.Н. Цвелев [100, 102]. Один из базисных принципов теории эволюции гласит, что в пределах конкретной филогенетической ветви характеристики любого общего предка являются более примитивными, неспециализированными, чем характеристики потомков на различных ветвях его древа, которые всегда более или менее специализированы пропорционально времени их появления [103]. Традиционный взгляд на видообразование и эволюцию морфологических признаков у растений предполагал, что эволюционными потенциями обладают неспециализированные формы, в каждой из филогенетических ветвей идут разнонаправленные процессы специализации; специализированные формы не способны к диверсификации [100, 102]. Однако у злаков виды с диплоидными кариотипами, как правило, представляют собой высокоспециализированные формы; часто это виды с узкими или разорванными ареалами, в то время как возникшие в результате аллополиплоидизации их эуполиплоидные потомки с их сложной комбинацией субгеномов выглядят деспециализированными. Это проявляется в том, что у них появляются более древние, а потому и более примитивные в эволюционно-морфологическом смысле признаки, как это наблюдается, например, у полиплоидных Elymus в сравнении с их диплоидными предками из родов Hordeum и Pseudoroegneria [73, 97]. Многочисленные полиплоидные Calamagrostis sensu lato менее специализированы в сравнении с их вероятными диплоидными предками из родов Agrostis, Trisetum [73, 102, 104]. PacBio-секвенирование четырех протяженных (около 1 тпн) низкокопийных районов ядерного генома папоротников из рода Botrychium (сем. Ophioglossаceae) показало, что многочисленные полиплоиды в этом роде возникли недавно, не более 5 млн лет назад, в результате недавней быстрой “неадаптивной радиации”, когда относительно небольшое количество диплоидных видов (12 из 20 исследованных) в результате независимых актов межвидовой гибридизации дали 19 тетраплоидов и один гексаплоидный вид, причем геномы трех диплоидных видов участвовали в формировании субгеномов тетраплоидов только один раз, а субгеном, полученый от B. pallidum, найден в геномах семи тетраплоидов и одного гексаплоида [105]. Таким образом, мы видим, что при сальтационном видообразовании путем аллополиплоидизации и интрогрессии эволюция словно идет вспять: высокоспециализированные эволюционно стазисные диплоидные (в действительности палеополиплоидные!) “предки” в результате гибридизации дают начало нескольким или многим эуполиплоидным родам, которые по совокупности признаков выглядят малоспециализированными.
Переход неополиплоида к эуполиплоидному, квазистабильному состоянию часто происходит в сочетании с переходом к многолетней форме жизни, самоопылению, апомиксису и вегетативному размножению, что важно при ограниченных возможностях найти полового партнера [74, 106–109]. Само по себе полиплоидное состояние генома не повышает устойчивости к низким температурам [19, 20], однако низкие температуры, соленость и иные неблагоприятные для данного вида экологические факторы, с которыми растениям приходится сталкиваться на границах их естественных ареалов, на нарушенных землях и во вновь осваиваемых экологических нишах, повышают вероятность появления гамет с нередуцированным числом хромосом у растений с облигатным или факультативным половым размножением [110–112]. Вероятно поэтому эуполиплоидов много среди инвазионных видов [113] и среди видов-эндемиков островной флоры [108]. В тундре Северного полушария 51% видов полиплоиды, в расположенной южнее тайге их 47% [74]. В западной Канаде, Гренландии и европейской Арктике 87% эндемиков полиплоиды, в Берингии таких эндемиков 69% [114, 115], на Аляске 55% [20].
В экстремальных условиях существования вероятность межвидовой гибридизации и появления аллополиплоидов увеличивается. Интересный и поучительный факт приводит Н.Н. Цвелев [102]: виды двух богатых видами секций рода Мятлик – Poa и Stenopoa – в умеренно-теплой зоне часто растут вместе не скрещиваясь. Гибриды между ними и гибридогенные, от них произошедшие, виды встречаются только в Арктике и в высокогорьях.
Влияя на частоту появления нередуцированных гамет, экстремальные климатические условия способствуют относительной многочисленности полиплоидов в Арктике, однако большого видового разнообразия и многочисленных популяций здесь нет. Эуполиплоидное стабильное состояние генома в сочетании с бесполым размножением, фиксированной гетерозиготностью, защитой от инбридинга и непредсказуемыми результатами генетического дрейфа позволяет арктическим полиплоидам длительное время успешно сосуществовать и успешно конкурировать с предварительно адаптированными и тоже немногочисленными диплоидными (палеополиплоидными) родственниками [74, 114].
Ахиллесова пята этих прекрасно адаптированных к условиям среды полиплоидов, часто клонально размножающихся и потому устойчиво передающих потомству весь комплекс прошедших отбор адаптивных признаков экстремалов, – низкий уровень внутривидовой изменчивости. Но нет уверенности в том, что следующие поколения этого вида не столкнутся с новыми радикальными вызовами со стороны видов-конкурентов или среды обитания. На этот случай природой создан механизм множественной изменчивости и генетического дрейфа на основе мейотической нестабильности и дисплоидии. Яркий пример явлений подобного рода приводит Йенс Клаусен [116]: в прериях северо-запада США растет крупный и многочисленный мятлик Poa ampla (сейчас: Poa juncifolia). В его кариотипе 2n = 63 хромосомы. От 90 до 95 процентов жизнеспособных семян производятся видом апомиктически, в результате чего все растения в потомстве имеют 63 хромосомы, фенотипически они воспроизводят родителей. Однако небольшая часть потомства, от 5 до 10%, появляется на свет в результате оплодотворения. Как правило, это небольшие, слабые растения, различающиеся по числу хромосом. У них насчитывали 2n = 56, 60–63, 66, 70, 82–84, 90–93, 98–102, 126, до 2n = 147 хромосом. Очевидно, перед нами своеобразный резерв, целенаправленное продуцирование все новых и новых вариантов комбинаций аллелей, некоторые из которых могут оказаться более успешными, чем родительское поколение, при будущем непредсказуемом, новом по сочетанию экологических факторов, но, увы, неизбежном экологическом кризисе.
Прошедшие отбор удачные сочетания аллелей субгеномов эуполиплоида, характерные для высоких полиплоидов крупные размеры, переход к неполовому размножению – все эти факторы способствуют успешному освоению эуполиплоидами новых ареалов, адаптации к экстремальным условиям существования на краю ареалов. Но, в то же время, мутации в геномах полиплоидов забуферены, а экспериментально полученные полиплоиды, как правило, от своих диплоидных предков ничем принципиально новым не отличаются [19, 20, 35, 117]. Складывается впечатление, что, легко формируя многочисленные новые, в той или иной степени репродуктивно изолированные полиплоидные виды, видовая и родовая самостоятельность которых определяется оригинальным сочетанием субгеномов, сам по себе акт WGD ничего принципиально нового не создает, что большая часть многочисленных полиплоидных видов – “слепые” ветви на филогенетическом древе цветковых растений (“dead-end”) [19, 20, 48, 118]. Переход неополиплоида к состоянию эуполиплоида – это путь к созданию нового вида, но это видообразование на уже освоенном уровне эволюционной сложности, шаг, не ведущий сам по себе к прогрессивной эволюции.
Сравнительная геномика показывает, что в долговременной перспективе, через миллион–другой лет после WGD, носители палеополиплоидных геномов имеют шанс вступить в состояние “взрывного” видообразования (диверсификации) и дать начало новым крупным надвидовым таксонам [3, 48, 119, 120]. Этому, по-видимому, должны предшествовать радикальные перестройки генома полиплоида – должна произойти диплоидизация генома, его “фракционирование”, уменьшение числа хромосом в геноме за счет серии транслокаций (дисплоидия) [48, 119, 120]. Иначе говоря, видообразование путем создания новых сочетаний субгеномов и стабилизации генома у эуплоидов, разобранное в нашей статье, и создание новых форм, дающих начало новым ароморфозам – это два разных направления эволюции растений.
Работа выполнена в рамках проекта РНФ 22-24-01117.
Настоящая статья не содержит результатов каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит результатов каких-либо исследований с участием в качестве объекта людей.
Список литературы
Grant V. Plant Speciation. N.Y.; London: Columbia Univ. Press, 1971. 233 p.
Wood T.E., Takebayashi N., Barker M.S. et al. The frequency of polyploid speciation in vascular plants // Proc. Natl Acad. Sci. USA. 2009. V. 106. P. 13875–13879. https://doi.org/10.1073/pnas.0811575106
Van de Peer Y., Mizrachi E., Marchal K. The evolutionary significance of polyploidy // Nature Rev. Genet. 2017. V. 18. P. 411–424. https://doi.org/10.1038/nrg.2017.26
Liu H., Wang X., Wang G. et al. The nearly complete genome of Ginkgo biloba illuminates gymnosperm evolution // Nature Plants. 2021. V. 7. P. 748–756. https://doi.org/10.1038/s41477-021-00933-x
Liu Y., Wang S., Li L. et al. The Cycas genome and the early evolution of seed plants // Nature Plants. 2022. V. 8. P. 389–401. https://doi.org/10.1038/s41477-022-01129-7
Nishiyama T., Sakayama H., De Vries J. et al. The Chara genome: secondary complexity and implications for plant terrestrialization // Cell. 2018. V. 174. P. 448–464. https://doi.org/10.1016/j.cell.2018.06.033
Banks J.A., Nishiyama T., Hasebe M. et al. The Selaginella genome identifies genetic changes associated with the evolution of vascular plants // Science. 2011. V. 332. P. 960–963 .https://doi.org/10.1126/science.1203810
Zhang J., Fu X.X., Li R.Q. et al. The hornwort genome and early land plant evolution // Nature Plants. 2020. V. 6. P. 107–118. https://doi.org/10.1038/s41477-019-0588-4
Bowman J.L., Kohchi T., Yamato K.T. et al. Insights into land plant evolution garnered from the Marchantia polymorpha genome // Cell. 2017. V. 171. P. 287–304. https://doi.org/10.1016/j.cell.2017.09.030
Szövényi P., Gunadi A., Li F.W. Charting the genomic landscape of seed-free plants // Nature Plants. 2021. V. 7. P. 554–565. https://doi.org/10.1038/s41477-021-00888-z
Benton M.J., Wilf P., Sauquet H. The Angiosperm terrestrial revolution and the origins of modern biodiversity // New Phytologist. 2022. V. 233. P. 2017–2035. https://doi.org/10.1111/nph.17822
Barker M.S., Arrigo N., Baniaga A.E. et al. On the relative abundance of autopolyploids and allopolyploids // New Phytologist. 2016. V. 210. P. 391–398.
Doyle J.J., Sherman-Broyles S. Double trouble: taxonomy and definitions of polyploidy // New Phytologist. 2017. V. 213. P. 487–493. https://doi.org/10.1111/nph.14276
Mayr E. The biological species concept // Species Concepts and Phylogenetic Theory: A Debate / Eds Wheeler Q.D., Meier R. N.Y. Chichester: Columbia Univ. Press, 2000. P. 17–29.
Ladizinsky G. Studies in Oat Evolution. Berlin; Heidelberg: Springer, 2012. P. 1–18.
Комаров В.Л. Учение о виде у растений (страница из истории биологии). М.; Л.: Изд-во Акад. наук СССР, 1940. 212 с.
Цвелев Н.Н. Вид как один из таксонов // Бюлл. МОИП. Отд. Биол. 1995. Т. 100. Вып. 5. С. 62–68.
Levin D.A. The long wait for hybrid sterility in flowering plants // New Phytologist. 2012. V. 196. P. 666–670.
Stebbins G.L. Polyploidy and the distribution of the arctic-alpine flora: New evidence and a new approach // Bot. Helvetica. 1984. V. 94. P. 1–13.
Stebbins G.L. The origin and success of polyploids in the boreal circumpolar flora: A new analysis // Trans. Bot. Sci. Edinburgh. 1986. V. 45. P. 17–31.
Liu J., Moeller M., Gao L.-M. et al. DNA barcoding for the discrimination of Eurasian yews (Taxus L., Taxaceae) and the discovery of cryptic species // Mol. Ecol. Resour. 2011. V. 11. P. 89–100. https://doi.org/10.1111/j.1755-0998.2010.02907.x
Bell D., Long D.G., Forrest A.D. et al. DNA barcoding of European Herbertus (Marchantiopsida, Herbertaceae) and the discovery and description of a new species // Mol. Ecol. Resources. 2012. V. 12. P. 36–47. https://doi.org/10.1111/j.1755-0998.2011.03053.x
Gill F.B. Species taxonomy of birds: Which null hypothesis? // The Auk: Ornithological Advances. 2014. V. 131. P. 150–161. https://doi.org/10.1642/AUK-13-206.1
Riesberg L.H., Wood T.E., Baack E.J. The nature of plant species // Nature. 2006. V. 440. P. 524–527. https://doi.org/10.1038/nature04402
Шнеер В.С., Коцеруба В.В. Криптические виды растений и их выявление по генетической дифференциации популяций // Экол. генетика. 2014. Т. 12. № 3. С. 12–26.
Soltis D.E., Soltis P.S., Schemske D.W. et al. Autopolyploidy in angiosperms: have we grossly underestimated the number of species? // Taxon. 2007. V. 56. P. 13–30. https://doi.org/10.2307/25065732
Шнеер В.С., Пунина Е.О., Родионов А.В. Внутривидовые различия в плоидности у покрытосеменных и их таксономическая интерпретация // Ботан. журн. 2018. Т. 103. № 5. С. 555–585. https://doi.org/10.1134/S0006813618050010
Цвелев Н.Н. О геномном критерии родов у высших растений // Ботан. журн. 1991. Т. 76. № 5. С. 669–676.
Ramsey J., Schemske D.W. Neopolyploidy in flowering plants // Annu. Rev. Ecol. Syst. 2002. V. 33. P. 589–639.
Li Z., McKibben M.T., Finch G.S. et al. Patterns and processes of diploidization in land plants // Annu. Rev. Plant Biol. 2021. V. 72. P. 387–410. https://doi.org/10.1146/annurev-arplant-050718-100344
Levin D.A. Plant speciation in the age of climate change // Annals Botany. 2019. V. 124. P. 769–775. https://doi.org/10.1093/aob/mcz108
Yu Z., Haberer G., Matthes M. et al. Impact of natural genetic variation on the transcriptome of autotetraploid Arabidopsis thaliana // Proc. Natl Acad. Sci. USA. 2010. V. 107. P. 17809–17814. https://doi.org/10.1073/pnas.1000852107
Liu B., Sun, G. Transcriptome and miRNAs analyses enhance our understanding of the evolutionary advantages of polyploidy // Critical Rev. Biotechnology. 2019. V. 39. P. 173–180. https://doi.org/10.1080/07388551.2018.1524824
Soltis D.E., Misra B.B., Shan S. et al. Polyploidy and the proteome // Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics. 2016. V. 1864. P. 896–907. https://doi.org/10.1016/j.bbapap.2016.03.010
Soltis D.E., Visger C.J., Marchant D.B., Soltis P.S. Polyploidy: pitfalls and paths to a paradigm // Am. J. Botany. 2016. V. 103. P. 1146–1166. https://doi.org/10.3732/ajb.1500501
Родионов А.В., Шнеер В.С., Гнутиков А.А. и др. Диалектика видов: от исходного единообразия, через максимально возможное разнообразие к конечному единообразию // Ботан. журн. 2020. Т. 105. № 9. С. 835–853. https://doi.org/10.31857/S0006813620070091
Favarger C. Sur l’emploi des nombres chromosomiques en géographie botanique historique // Ber. Geobot. Inst. Rübel. 1961. T. 32. S. 119–146.
Mandáková T., Joly S., Krzywinski M. et al. Fast diploidization in close mesopolyploid relatives of Arabidopsis // The Plant Cell. 2010. V. 22. P. 2277–2290.
Родионов А.В., Носов Н.Н., Ким Е.С. и др. Происхождение полиплоидных геномов мятликов (Poa L.) и феномен потока генов между Северной Пацификой и субантарктическими островами // Генетика. 2010. Т. 46. № 12. С. 1598–1608.
Родионов А.В., Амосова А.В., Беляков Е.А. и др. Генетические последствия межвидовой гибридизации, ее роль в видообразовании и фенотипическом разнообразии растений // Генетика. 2019. Т. 55. № 3. С. 255–272.
Zhan S.H., Otto S.P., Barker M.S. Broad variation in rates of polyploidy and dysploidy across flowering plants is correlated with lineage diversification // bioRxiv. 2021. this version posted March 31, 2021.https://doi.org/10.1101/2021.03.30.436382
Навашин М.С. Об изменении числа и морфологических признаков хромосом у межвидовых гибридов // Тр. по прикладной ботанике, генетике и селекции. 1927. Т. 17. Вып. 3. С. 121–150.
Talbert P.B., Bryson T.D., Henikoff S. Adaptive evolution of centromere proteins in plants and animals // J. Biology. 2004. V. 3(4). P. 1–17. https://doi.org/10.1186/jbiol11
Melters D.P., Bradnam K.R., Young H.A. et al. Comparative analysis of tandem repeats from hundreds of species reveals unique insights into centromere evolution // Genome Biol. 2013. V. 14(1). P. 1–20. https://doi.org/10.1186/gb-2013-14-1-r10
Maheshwari S., Tan E.H., West A. et al. Naturally occurring differences in CENH3 affect chromosome segregation in zygotic mitosis of hybrids // PLoS Genetics. 2015. V. 11(1). P. e1004970. https://doi.org/10.1371/journal.pgen.1004970
Kotseruba V., Gernand D., Meister A., Houben, A. Uniparental loss of ribosomal DNA in the allotetraploid grass Zingeria trichopoda (2n = 8) // Genome. 2003. V. 46. P. 156–163. https://doi.org/10.1139/g02-104
Marimuthu M.P., Maruthachalam R., Bondada R. et al. Epigenetically mismatched parental centromeres trigger genome elimination in hybrids // Sci. Advances. 2021. V. 7(47). eabk1151. https://doi.org/10.1126/sciadv.abk1151
Mandáková T., Lysak M.A. Post-polyploid diploidization and diversification through dysploid changes // Curr. Opinion Plant Biol. 2018. V. 42. P. 55–65. https://doi.org/10.1016/j.pbi.2018.03.001
Soares N.R., Mollinari M., Oliveira G.K. et al. Meiosis in polyploids and implications for genetic mapping: A review // Genes. 2021. V. 12. P. 1517. https://doi.org/10.3390/genes12101517
Wang M., Wang P., Lin M. et al. Evolutionary dynamics of 3D genome architecture following polyploidization in cotton // Nat. Plants. 2018. V. 4. P. 90–97. https://doi.org/10.1038/s41477-017-0096-3
Concia L., Veluchamy A., Ramirez-Prado J.S. et al. Wheat chromatin architecture is organized in genome territories and transcription factories // Genome Biol. 2020. V. 21(1). https://doi.org/10.1186/s13059-020-01998-1
Barea L., Redondo-Río Á., Lucena-Marín R. et al. Homologous chromosome associations in domains before meiosis could facilitate chromosome recognition and pairing in wheat // Scientific Reports. 2022. V. 12(1). P. 1–11. https://doi.org/10.1038/s41598-022-14843-1
International Wheat Genome Sequencing Consortium. Shifting the limits in wheat research and breeding using a fully annotated reference genome // Science. 2018. V. 361. eaar7191.
Stein J.C., Yu Y., Copetti D. et al. Genomes of 13 domesticated and wild rice relatives highlight genetic conservation, turnover and innovation across the genus Oryza // Nat. Genetics. 2018. V. 50(2). P. 285–296. https://doi.org/10.1038/s41588-018-0040-0
Nei M., Rooney A.P. Concerted and Birth–and–Death evolution of multigene families // Annu. Rev. Genet. 2005. V. 39. P. 121–152. https://doi.org/0.1146/annurev.genet.39.073003.112240
Gonzalo A. All ways lead to Rome–Meiotic stabilization can take many routes in nascent polyploid plants // Genes. 2022. V. 13(1). P. 147. https://doi.org/10.3390/genes13010147
Viegas W.S., Mello-Sampayo T., Feldman M., Avivi L. Reduction of chromosome pairing by a spontaneous mutation on chromosomal arm 5DL of Triticum aestivum // Can. J. Genet. Cytol. 1980. V. 22. P. 569–575. https://doi.org/10.1139/g80-062
Svačina R., Sourdille P., Kopecký D., Bartoš J. Chromosome pairing in polyploid grasses // Front. Plant Sci. 2020. V. 11. P. 1056. https://doi.org/10.3389/fpls.2020.01056
Jenczewski E., Alix K. From diploids to allopolyploids: The emergence of efficient pairing control genes in plants // Crit. Rev. Plant Sci. 2004. V. 23. P. 21–45. https://doi.org/10.1080/07352680490273239
Cuñado N., Blazquez S., Melchor L. et al. Understanding the cytological diploidization mechanism of polyploid wild wheats // Cytogenet. Genome Res. 2005. V. 109. P. 205–209. https://doi.org/10.1159/000082401
Tang Z.X., Fu S.L., Yan B.J. et al. Unequal chromosome division and inter-genomic translocation occurred in somatic cells of wheat–rye allopolyploid // J. Plant. Res. 2012. V. 125. P. 283–290. https://doi.org/10.1007/s10265-011-0432-z
Luo J., Zhao L., Zheng J. et al. Karyotype mosaicism in early generation synthetic hexaploid wheats // Genome. 2020. V. 63. P. 329–336. https://doi.org/10.1139/gen-2019-0148
Gill B.S., Chen P.D. Role of cytoplasm-specific introgression in the evolution of the polyploid wheats // Proc. Natl Acad. Sci. USA. 1987. V. 84. № 19. P. 6800–6804. https://doi.org/10.1073/pnas.84.19.6800
Бадаева Е.Д., Шелухина О.Ю., Дедкова О.С. и др. Сравнительное цитогенетическое исследование гексаплоидных видов Avena L. // Генетика. 2011. Т. 47. № 6. С. 783–795.
Amosova A.V., Badaeva E.D., Muravenko O.V., Zelenin A.V. An improved method of genomic in situ hybridization (GISH) for distinguishing closely related genomes of tetraploid and hexaploid wheat species // Rus. J. Developm. Biol. 2009. V. 40. № 2. P. 90–94.
Liu Q., Lin L., Zhou X. et al. Unraveling the evolutionary dynamics of ancient and recent polyploidization events in Avena (Poaceae) // Scientific Reports. 2017. V. 7(1). P. 1–13. https://doi.org/10.1038/srep41944
Jellen E.N., Gill B.S., Cox T.S. Genomic in situ hybridization differentiates between A/D- and C-genome chromatin and detects intergenomic translocations in polyploid oat species (genus Avena) // Genome. 1994. V. 37. P. 613–618.
Lim K.Y., Kovarik A., Matyasek R. et al. Sequence of events leading to near-complete genome turnover in allopolyploid Nicotiana within five million years // New Phytologist. 2007. V. 175. P. 756–763. https://doi.org/10.1111/j.1469-8137.2007.02121.x
Winterfeld G., Schneider J., Perner K., Röser M. Polyploidy and hybridization as main factors of speciation: complex reticulate evolution within the grass genus Helictochloa // Cytogen. Genome Res. 2014. V. 142. P. 204–225. https://doi.org/10.1159/000361002
Suissa J.S., Kinosian S.P., Schafran P.W. et al. Homoploid hybrids, allopolyploids, and high ploidy levels characterize the evolutionary history of a western North American quillwort (Isoëtes) complex // Mol. Phylogen. Evol. 2022. V. 166. P. 107332. https://doi.org/10.1016/j.ympev.2021
Sutherland B.L., Galloway L.F. Postzygotic isolation varies by ploidy level within a polyploid complex // New Phytologist. 2017. V. 213. P. 404–412.
Камелин Р.В. Особенности видообразования у цветковых растений // Тр. Зоол. ин-та РАН. Приложение № 1. 2009. С. 141–149.
Цвелев Н.Н., Пробатова Н.С. Злаки России. М.: Тов-во науч. изд. КМК, 2019. 646 с.
Rice A., Šmarda P., Novosolov M. et al. The global biogeography of polyploid plants // Nature Ecol. Evol. 2019. V. 3. P .265–273. https://doi.org/10.1038/s41559-018-0787-9
Vaezi J., Brouillet L. Origin of Symphyotrichum anticostense (Asteraceae: Astereae), an endemic, high polyploid species of the Gulf of St. Lawrence region, based on morphological and nrDNA evidence // Botany. 2022. https://doi.org/10.1139/cjb-2021-0145
Pellicer J., Garcia S., Garnatje T. et al. Chromosome counts in Asian Artemisia L. (Asteraceae) species: From diploids to the first report of the highest polyploid in the genus // Bot. J. Linn. Soc. 2007. V. 153. P. 301–310. https://doi.org/10.1111/j.1095-8339.2007.00611.x
Guggisberg A., Mansion G., Kelso S., Conti E. Evolution of biogeographic patterns, ploidy levels, and breeding systems in a diploid–polyploid species complex of Primula // New Phytologist. 2006. V. 171. P. 617–632. https://doi.org/10.1111/j.1469-8137.2006.01722.x
Kaur H., Mubarik N., Kumari S., Gupta R.C. Chromosome numbers and basic chromosome numbers in monocotyledonous genera of the Western Himalayas (India) // Acta Biologica Cracoviensia. Ser. Botanica. 2014. V. 56. № 2. P. 9–19. https://doi.org/10.2478/abcsb-2014-0016
Winterfeld G., Schneider J., Perner K., Röser M. Origin of highly polyploid species: Different pathways of auto and allopolyploidy in 12–18× species of Avenula (Poaceae) // Int. J. Plant Sci. 2012. V. 173. P. 399–411. https://doi.org/10.1086/664710
Hardion L., Verlaque R., Rosato M. et al. Impact of polyploidy on fertility variation of Mediterranean Arundo L. (Poaceae) // Comptes Rendus Biologies. 2015. V. 338. P. 298–306. https://doi.org/10.1016/j.crvi.2015.03.013
Chumová Z., Krejčíková J., Mandáková T. et al. Evolutionary and taxonomic implications of variation in nuclear genome size: Lesson from the grass genus Anthoxanthum (Poaceae) // PLoS One. 2015. V. 10(7). e0133748. https://doi.org/10.1371/journal.pone.0133748
Gould F.W. Pollen size as related to polyploidy and speciation in the Andropogon saccharoides–A. barbinodis complex // Brittonia. 1957. V. 9. № 2. P. 71–75.
Hair J.B., Beuzenberg E.J. High polyploidy in a New Zealand Poa // Nature. 1961. V. 189. P. 160.
Li L.F., Zhang Z.B., Wang Z.H. et al. Genome sequences of the five Sitopsis species of Aegilops and the origin of polyploid wheat B subgenome // Mol. Plant. 2022. V. 15. P. 488–503. https://doi.org/10.1016/j.molp.2021.12.019
Levy A.A., Feldman M. Evolution and origin of bread wheat // The Plant Cell. 2022. koac130. https://doi.org/10.1093/plcell/koac130
Eilam T., Anikster Y., Millet E. et al. Nuclear DNA amount and genome downsizing in natural and synthetic allopolyploids of the genera Aegilops and Triticum // Genome. 2008. V. 51. P. 616–627. https://doi.org/10.1139/G08-043
Kamal N., Tsardakas Renhuldt N., Bentzer J. et al. The mosaic oat genome gives insights into a uniquely healthy cereal crop // Nature. 2022. V. 606. P. 113–119. https://doi.org/0.1038/s41586-022-04732-y
Löve Á. Generic evolution of the wheat grasses // BioI. Zentralbl. 1982. T. 101. S. 199–212.
Löve Á. Conspectus of the Triticeae // Feddes Repert. 1984. T. 95. S. 425–521.
Dewey D.R. The genomic system of classification as a guide to intergeneric hybridization with the perennial Triticeae // Gene Manipulation in Plant Improvement. Boston, MA: Springer, 1984. P. 209–279.
Делоне Л.Н. Сравнительное кариологическое исследование видов Muscari Mill. и Bellevalia Lapeyr. // Вестн. Тифлисского бот. сада. 1922. Т. 2. Вып. 1. С. 1–32.
Wang R.R.-C., von Bothmer R., Dvorak J. et al. Genome symbols in the Triticeae (Poaceae) // Herbarium Publications. Logan, Uta: Uta State Publ., 1994. Paper 20. P. 1–19. https://digitalcommons.usu.edu/herbarium_pubs/20
Blattner F.R. Taxonomy of the genus Hordeum and barley (Hordeum vulgare) // The Barley Genome. Cham.: Springer, 2018. P. 11–23.
Baum B.R., Estes J.R., Gupta P.K. Assessment of the genomic system of classification in the Triticeae // Am. J. Botany. 1987. V. 74. P. 1388–1395.
Barkworth M.E. Taxonomy of the Triticeae: A historical perspective // Hereditas. 1992. V. 116. P. 1–14.
Камелин Р.В. Лекции по систематике растений. Главы теоретической систематики растений. Барнаул: Азбука, 2004. 226 с.
Yen C., Yang J.-L., Yen Y. Hitoshi Kihara, Áskell Löve and the modern genetic concept of the genera in the tribe Triticeae (Poaceae) // J. Syst. Evol. 2005. V. 43. P. 82–93.
Vavilov N.I. The law of homologous series in variation // J. Genet. 1922. V. 12. P. 47–89.
Вавилов Н.И. Закон гомологических рядов в наследственной изменчивости. М.; Л.: ОГИЗ-Сельхозгиз, 1935. 56 с.
Цвелев Н.Н. О возможности деспециализации путем гибридогенеза на примере эволюции трибы Triticeae семейства Злаков (Poaceae) // Журн. общей биологии. 1975. Т. 36. № 1. С. 90–99.
Kihara H. Genom analyse bei Triticum und Aegilops // Cytologia. 1930. V. 1. № 3. P. 263–284.
Цвелев Н.Н. Гибридизация как один из факторов увеличения биологического разнообразия и геномный критерий родов у высших растений // Биологическое разнообразие: подходы к изучению и сохранению. СПб., 1992. С. 193–201.
Simpson G.G. Tempo and Mode in Evolution. N.Y.: Columbia Univ. Press, 1944. 237 p.
Saarela J.M., Bull R.D., Paradis M.J. et al. Molecular phylogenetics of cool-season grasses in the subtribes Agrostidinae, Anthoxanthinae, Aveninae, Brizinae, Calothecinae, Koeleriinae and Phalaridinae (Poaceae, Pooideae, Poeae, Poeae chloroplast group 1) // PhytoKeys. 2017. V. 87. 1–139. https://doi.org/10.3897/phytokeys.87.12774
Dauphin B., Grant J.R., Farrar D.R., Rothfels C.J. Rapid allopolyploid radiation of moonwort ferns (Botrychium; Ophioglossaceae) revealed by PacBio sequencing of homologous and homeologous nuclear regions // Mol. Phylogenet. Evol. 2018. V. 120. P. 342–353. https://doi.org/10.1016/j.ympev.2017.11.025
Schinkel C.C., Kirchheimer B., Dellinger A.S. et al. Correlations of polyploidy and apomixis with elevation and associated environmental gradients in an alpine plant // AoB Plants. 2016. V. 8. P. 1–16. https://doi.org/10.1093/aobpla/plw064
Herben T., Suda J., Klimešová J. Polyploid species rely on vegetative reproduction more than diploids: a re-examination of the old hypothesis // Annals Botany. 2017. V. 120. P. 341–349. https://doi.org/10.1093/aob/mcx009
Meudt H.M., Albach D.C., Tanentzap A.J. et al. Polyploidy on islands: its emergence and importance for diversification // Front. Plant Sci. 2021. V. 12:637214. https://doi.org/10.3389/fpls.2021.637214
Villa S., Montagna M., Pierce S. Endemism in recently diverged angiosperms is associated with polyploidy // Plant Ecology. 2022. V. 223. P. 479–492. https://doi.org/10.1007/s11258-022-01223-y
Pécrix Y., Rallo G., Folzer H. et al. Polyploidization mechanisms: temperature environment can induce diploid gamete formation in Rosa sp. // J. Exp. Bot. 2011. V. 62. P. 3587–3597. https://doi.org/10.1093/jxb/err052
Klatt S., Schinkel C.C., Kirchheimer B. et al. Effects of cold treatments on fitness and mode of reproduction in the diploid and polyploid alpine plant Ranunculus kuepferi (Ranunculaceae) // Annals Botany. 2018. V. 121. P. 1287–1298.
Fox D.T., Soltis D.E., Soltis, P.S. et al. Polyploidy: A biological force from cells to ecosystems // Trends Cell Biol. 2020. V. 30. P. 688–694. https://doi.org/10.1016/j.tcb.2020.06.006
Prentis P.J., Wilson J.R., Dormontt E.E. et al. Adaptive evolution in invasive species // Trends Plant Sci. 2008. V. 13. P. 288–294.
Brochmann C., Brysting A.K., Alsos I.G. et al. Polyploidy in arctic plants // Biol. J. Linn. Soc. 2004. V. 82. P. 521–536. https://doi.org/10.1111/j.1095-8312.2004.00337.x
Пробатова Н.С. Хромосомные числа в семействе Poaceae и их значение для систематики, филогении и фитогеографии (на примере злаков Дальнего Востока России) // Комаровские чтения. 2007. Вып. 55. С. 9–103.
Clausen J. Introgression facilitated by apomixis in polyploid Poas // Euphytica. 1961. V. 10. P. 87–94. https://doi.org/10.1007/BF00037208
Soltis D.E., Visger C.J., Soltis P.S. The polyploidy revolution then… and now: Stebbins revisited // Am. J. Botany. 2014. 101. P. 1057–1078. https://doi.org/10.3732/ajb.1400178
Mayrose I., Zhan S.H., Rothfels C.J. et al. Recently formed polyploid plants diversify at lower rates // Science. 2011. V. 333. P. 1257. https://doi.org/10.1126/science.1207205
Schranz M.E., Mohammadin S., Edger P.P. Ancient whole genome duplications, novelty and diversification: The WGD Radiation Lag-Time Model // Curr. Opinion Plant Biol. 2012. V. 15. P. 147–153. https://doi.org/10.1016/j.pbi.2012.03.011
Wang X., Morton J.A., Pellicer J. et al. Genome downsizing after polyploidy: Mechanisms, rates and selection pressures // The Plant J. 2021. V. 107. P. 1003–1015. https://doi.org/10.1111/tpj.15363
Дополнительные материалы отсутствуют.



