Вопросы ихтиологии, 2019, T. 59, № 5, стр. 615-619
Способ фиксации личинок рыб для морфологических и генетических исследований
Н. В. Гордеева 1, 2, *, С. Г. Кобылянский 2, С. А. Евсеенко 2
1 Институт общей генетики РАН – ИОГЕН
Москва, Россия
2 Институт океанологии РАН – ИО РАН
Москва, Россия
* E-mail: ribka04@mail.ru
Поступила в редакцию 18.12.2018
После доработки 28.12.2018
Принята к публикации 16.01.2019
Аннотация
Для фиксации и хранения личинок морских глубоководных рыб опробованы три водных буферных раствора, позволяющие стабилизировать ДНК, а также сохранить исходную форму тела и морфологические структуры. По результатам оценки качества ДНК и внешнего вида личинок спустя 6 мес. после фиксации к использованию рекомендован содержащий диметилсульфоксид буфер. Этот фиксатор, в отличие от этанола или формалина, не токсичен, не горюч, не требует хранения образцов в холодильнике, а также совместим со стандартными методиками выделения ДНК или протоколами коммерческих наборов.
Техническое развитие и удешевление методов молекулярной генетики позволили в последние десятилетия существенно продвинуться в разрешении вопросов эволюции, систематики и филогении рыб (Johnson et al., 2009; Betancur et al., 2017). В таксономических и популяционных исследованиях становится общепринятым комплексный подход, в рамках которого анализируются разнообразные морфологические, экологические и генетические признаки, поэтому правильный выбор способа фиксации материала приобретает особую важность. Зоологические, в том числе ихтиологические, коллекции традиционно фиксируют в формалине, как правило, с последующим помещением в 70%-ный этанол для длительного хранения на протяжении многих десятилетий (Pisani, 1973). Фиксированные таким способом образцы оказываются практически непригодными для молекулярно-генетического анализа из-за сильной фрагментации ДНК, модификации нуклеотидов, образования комплексов ДНК–формальдегид и связей между цепями ДНК (Serth et al., 2000; Quach et al., 2004; Campos, Gilbert, 2012; Do, Dobrovic, 2012; Wong et al., 2014). Как показывает практика, анализ повреждённой формалином ДНК требует дополнительных усилий и финансовых затрат, что, однако, не гарантирует получение положительного результата (Shedlock et al., 1997; Schander, Halanych, 2003; Chakraborty et al., 2006; Klopfleisch et al., 2011). В лучшем случае с использованием стандартных методик секвенирования по Сэнгеру удастся прочитать недлинные последовательности обычно митохондриальной ДНК, которые иногда приходится складывать из совсем коротких фрагментов, каждый из которых получают с помощью индивидуально подобранных праймеров. C появлением технологий высокопроизводительного секвенирования (или секвенирования следующего поколения – NGS), позволяющих одновременно считывать несколько сотен миллионов коротких фрагментов (50–150 пар нуклеотидов, п.н.), появилась возможность получить намного больше информации из повреждённой формалином ДНК (Hykin et al., 2015), однако этот анализ пока ещё слишком дорог для большинства лабораторий. Необходимо иметь в виду, что на сохранность генетического материала в фиксированных формалином образцах могут повлиять многие факторы. Во-первых, от длительности нахождения тканей в формалине прямо зависит количество и качество содержащейся в них ДНК (Schander, Halanych, 2003; Bucklin, Allen, 2004). Недавно было показано, что кратковременная экспозиция (до 1 недели) в формалине при низкой температуре (+4°С) может и не иметь существенных последствий для целостности молекул ДНК, во всяком случае позволяет амплифицировать из таких образцов фрагменты значительной длины (Vivien et al., 2016). Также отмечалось, что использование для фиксации формалина с добавлением NaCl или формальдегида, приготовленного разведением в морской воде, замедляет фрагментацию ДНК в образцах (Seutin et al., 1991; Paireder et al., 2013). Следует учитывать и другие обстоятельства сбора и хранения образцов: например, в условиях высокой температуры и повышенного солнечного излучения деградация ДНК происходит интенсивнее. Таким образом, вероятность успешного извлечения генетической информации из фиксированных формалином образцов, особенно в старых коллекциях, невозможно предсказать.
Этанол (в абсолютной или 94–96%-ной концентрации) – наиболее широко применяемый фиксатор для молекулярно-генетического анализа. Для долговременного сохранения ДНК образцы различных тканей или мелкие организмы целиком рекомендуется фиксировать в объёмном соотношении не менее 1 : 3 и хранить при –20°С (Steinke, Hanner, 2011). Из-за сильной дегидратации и денатурации белков (Seutin et al., 1991) фиксированные в этаноле ткани могут существенно деформироваться, что затрудняет анализ внешней морфологии и таксономическую идентификацию, особенно у животных с мягким обводнённым телом (Castro, Thomason, 1973). Этот способ фиксации, в частности, совершенно не подходит для личинок слабо изученных глубоководных рыб, для которых таксономическое описание и определители разработаны в основном на основе внешней морфологии взрослых особей. Поскольку во избежание посмертной деградации ДНК личинки должны быть зафиксированы как можно быстрее после вылова, проведение полноценного морфологического исследования уже не представляется возможным.
Альтернативой классическим фиксаторам – формалину и этанолу – могут служить многокомпонентные водные растворы, содержащие различные вещества (растворители, детергенты, соли), которые позволяют сохранять исходную форму и морфологические структуры, а также инактивировать нуклеазы и стабилизировать высокомолекулярную ДНК (Post et al., 1993; Quicke et al., 1999; Srinivasan et al., 2002; Nagy, 2010). В сравнении с этанолом и формалином к их преимуществам можно отнести нетоксичность, негорючесть и вследствие этого лёгкость транспортировки, а по сравнению с коммерческими буферами, предназначенными для стабилизации нуклеиновых кислот (например, RNAlater (“Ambion”, США) или его отечественный аналог IntactRNA (ЗАО “Евроген”)), – невысокую стоимость и доступность компонентов. Один из буферных растворов, который можно приготовить самостоятельно, содержащий диметилсульфоксид (ДМСО), уже хорошо зарекомендовал себя в морфологических и генетических исследованиях различных морских беспозвоночных – нематод (Dawson et al., 1998; Yoder et al., 2006), моногеней (Strona et al., 2009) и моллюсков (Williams, 2007), а также применялся для фиксации различных тканей позвоночных животных (Seutin et al., 1991; Kilpatrick, 2002), причём анализируемые пробы хранились до 5 лет при температуре 20–65°С. Апротонный растворитель ДМСО сам по себе не консервирует ДНК, но облегчает проникновение в клетки стабилизирующих ДНК веществ (Kilpatrick, 2002), а также благодаря своей высокой проникающей способности может быстро восстанавливать и поддерживать их первоначальную форму (Yoder et al., 2006). Помимо упомянутых фиксаторов при сборе образцов для молекулярно-генетического анализа в полевых условиях предлагалось использовать разнообразные лизирующие растворы (Seutin et al., 1991; Longmire et al., 1997; Nagy, 2010), которые позволяют обойтись без заморозки или охлаждения образцов, а также подготавливают ткани непосредственно к выделению ДНК. Однако качество выделяемой из таких образцов ДНК может оказаться несколько худшим, чем из фиксированных с помощью содержащих ДМСО растворов (Kilpatrick, 2002).
Цель работы – протестировать три буферных раствора для фиксации и хранения личинок морских рыб. Качество ДНК в фиксированных образцах оценивали с помощью амплификации и секвенирования фрагмента митохондриального гена 1 субъединицы цитохром-оксидазы (cox 1 мтДНК), который используется для ДНК-баркодинга.
МАТЕРИАЛ И МЕТОДИКА
Личинок отлавливали в ходе 39-го рейса НИС “Профессор Логачёв” (ИО РАН) в тропической части Северной Атлантики разноглубинным тралом Айзекса–Кидда в модификации Самышева–Асеева. По возможности сразу же после разбора траловых уловов личинок помещали в один из трёх фиксирующих растворов – содержащий диметилсульфоксид буфер (DESS), лизирующий буфер TENT и буфер Лонгмира (таблица).
Положение станций 39-го рейса НИС “Профессор Логачев”, на которых разными способами были зафиксированы личинки морских рыб
| Станция | Дата лова | Координаты | Горизонт лова, м | Фиксирующий раствор (число личинок, экз.) | |
|---|---|---|---|---|---|
| с.ш. | з.д. | ||||
| 39L182rt | 27.02.2018 | 14°22′46′′ | 44°33′46′′ | 1500–0 | DESS (4), TENT (2), Лонгмира (3) |
| 39L183rt | 27.02.2018 | 14°26′27′′ | 44°33′12′′ | 700–0 | DESS (1), TENT (1), Лонгмира (1) |
| 39L189rt | 01.03.2018 | 13°31′42′′ | 45°0′22′′ | 700–0 | DESS (1), Лонгмира (1) |
| 39L192rt | 02.03.2018 | 13°28′6′′ | 44°34′51′′ | 2500–0 | DESS (1), Лонгмира (1) |
Раствор DESS, содержащий 20% ДМСО, 0.25 M этилендиаминтетраацетата натрия (ЭДТA) и насыщенный NaCl (pH 8.0), приготовляли упрощённым способом, предложенным Йодер с соавторами (Yoder et al., 2006) для фиксации нематод. Вначале приготовляли 0.5 М раствор ЭДТA, для чего 372.24 г динатриевой соли ЭДТА растворяли в 500 мл воды. Затем pH раствора доводили до отметки 8 с помощью 5 M раствора NaOH (200 г кристаллического гидросксида натрия на 1 л воды), которого понадобится ~500 мл. Значение pH проверили с помощью индикаторной бумаги; по достижении нужной величины добавляли воду до конечного объёма 2 л. Для приготовления 2 л буфера DESS компоненты смешивали в следующем порядке: 1 л 0.5 М ЭДТА, 400 мл ДМСО, 600 мл воды и ~300–400 г NaCl до полного насыщения раствора, о чём свидетельствует появление осадка.
Для приготовления лизирующего буфера TENT (10 мM Трис-(гидроксиметил)аминометана (Трис-основание), 10 мM ЭДТА, 100 мM NaCl и 2% детергента Tween 20 (“Sigma”, США)) смешивали 10 мл 1 M буфера Трис-HCl (pH 8.0), 20 мл 0.5 M ЭДТA (pH 8.0), 20 мл 5 M NaCl и 20 мл Tween 20; доводили водой до 1 л.
Для приготовления лизирующего буфера Лонгмира (0.1 М Трис-основания, 0.1 М ЭДТА, 0.01 М NaCl и 0.5%-ного додецилсульфата натрия (SDS)) смешивали 100 мл 1 M буфера Трис-HCl (pH 8.0), 200 мл 0.5 M ЭДТA (pH 8.0), 2 мл 5 M NaCl и 25 мл 20%-ного SDS; объём раствора доводили до 1 л (Longmire et al., 1997).
Рекомендуется использовать воду высокой степени очистки (бидистиллированную или деионизованную), однако подойдет и дистиллированная вода. Все буферы можно приготовить заранее и хранить при комнатной температуре.
Личинок заливали фиксатором в соотношении 1 : 3 (DESS) или 1 : 10 (TENT и Лонгмира) в индивидуальной 2-миллилитровой пробирке с завинчивающейся крышкой и матовой площадкой для маркировки. До выделения ДНК зафиксированные образцы хранили ~6 мес. при комнатной температуре (20–30°С).
ДНК экстрагировали из фрагмента хвостовой части личинки (достаточно 5–10 мг), который перед тем дважды промывали в бидистиллированной/деионизованной воде, поскольку ДМСО и ЭДТА из фиксирующего раствора потенциально могут ингибировать амплификацию ДНК (ПЦР). Затем использовали стандартную методику экстракции фенолом и хлороформом (Маниатис и др., 1984) или коммерческий набор DiatomTM DNA Prep100 (ООО “Лаборатория ИЗОГЕН”) согласно протоколу; в обоих случаях лизис проводили 12–24 ч при 56°С.
Для амплификации фрагмента сox 1 размером около 650 п.н. использовали пару универсальных праймеров FishF1 и FishR1 и условия, описанные в работе Уорда с соавторами (Ward et al., 2005). Количество и качество ПЦР-продуктов визуально оценивали с помощью электрофореза в 1%-ном агарозном геле. Затем их очищали от компонентов реакции с помощью процедуры переосаждения этанолом в присутствии ацетата аммония и секвенировали с использованием набора BigDye Terminator v3.1 Cycle Sequencing Kit (“Applied Biosystems”, США) на автоматическом секвенаторе ABI PRISM 3100 Genetic Analyzer. Для редактирования полученных последовательностей, оценки их качества и поиска максимального соответствия среди имеющихся в базе данных NCBI с помощью компьютерных программ BLAST (NCBI. 2018) использовали программу Geneious (Kearse et al., 2012).
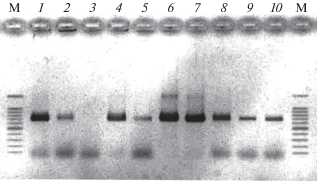
Рис. 1.
Результаты электрофореза в 1%-ном агарозном геле продуктов амплификации фрагмента гена cox1 мтДНК у личинок, фиксированных в буферных растворах TENT (дорожки 1–3) и DESS (4–10). На крайних дорожках (М) – маркер 100 bp Ladder (“Promega”, США) (длины фрагментов сверху вниз: 1500 п.н., 1000–100 с шагом 100 п.н.).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Спустя несколько месяцев после фиксации в буфере Лонгмира личинки подверглись полному лизису, в буфере TENT лизис затронул только верхние покровы, а у хранящихся в DESS личинок внешний вид и форма не изменились. Выделение ДНК, амплификация и секвенирование фрагмента cox 1 мтДНК из личинок, зафиксированных в буферах DESS и TENT, оказались в целом успешными. О качестве ампликонов можно судить по результату их электрофоретического разделения в агарозном геле (рисунок). Полученные последовательности cox 1 позволили легко идентифицировать видовую принадлежность личинок путём поиска с помощью программ BLAST максимального соответствия последовательностям морских рыб, хранящимся в NCBI/Genbank. В результате установлено, что личинки принадлежат видам: Nemichthys scolopaceus (Nemichthyidae), Lampanyctus alatus, Hygophum taaningi, Ceratoscopelus warmingii (Myctophidae), Bothus ocellatus (Bothidae), Eurypharynx pelecanoides (Eurypharyngidae), Valenciennellus tripuntulatus (Sternoptychidae) и Diretmichthys parini (Diretmidae). Большинство последовательностей были высокого качества (87–92%) и длиной 631–658 п.н., а три из них – более короткие (порядка 400 п.н.) и невысокого качества (5–10%), этого, однако, оказалось вполне достаточно для их видовой идентификации. Скорее всего, неудовлетворительный результат объясняется относительно более поздним помещением образцов в фиксирующий раствор, когда процессы посмертной деградации ДНК в клетках уже развились. На это указывает то обстоятельство, что короткие последовательности получились у личинок L. alatus (TENT и DESS) и V. tripunctulatus, собранных на одной станции 39L 182rt. Полноразмерные последовательности cox1 мтДНК личинок шести видов были помещены в NCBI/Genbank под номерами MK050650–MK050655.
Таким образом, буфер DESS можно рекомендовать как эффективный, недорогой и нетоксичный фиксатор личинок рыб, которых в дальнейшем планируется использовать как для молекулярно-генетического, так и для морфологического анализа. Фиксировать личинок в DESS следует как можно быстрее после вылова для сохранения наилучшего качества ДНК в объёмном соотношении 1 : 3 и хранить при комнатной температуре.
Список литературы
Маниатис Т., Фрич Э., Сэмбрук Дж. 1984. Методы генетической инженерии. Молекулярное клонирование. М.: Мир, 480 с.
Betancur-R. R., Wiley E.O., Arratia G. et al. 2017. Phylogenetic classification of bony fishes // BMC Evol. Biol. V. 17. № 1. P. 162. https://doi.org/10.1186/s12862-017-0958-3
Bucklin A., Allen L.D. 2004. MtDNA sequencing from zooplankton after long-term preservation in buffered formalin // Mol. Phylogen. Evol. V. 30. P. 879–882.
Campos P., Gilbert T.P. 2012. DNA extraction from formalin-fixed material // Methods in molecular biology. V. 840 / Eds. Shapiro B., Hofreiter M. N.Y.: Humana Press. P. 81–85. https://doi.org/10.1007/978-1-61779-516-9_11
Castro C.E., Thomason I.J. 1973. Permeation dynamics and osmoregulation in Aphelenchus avenae // Nematologica. V. 19. P. 100–108. https://doi.org/10.1163/187529273X00150
Chakraborty A., Sakai M., Iwatsuki Y. 2006. Museum fish specimens and molecular taxonomy: a comparative study on DNA extraction protocols and preservation techniques // J. Appl. Ichthyol. V. 22. № 2. P. 160–166. https://doi.org/10.1111/j.1439-0426.2006.00718.x
Dawson M.N., Raskoff K.A., Jacobs D.K. 1998. Field preservation of marine invertebrate tissue for DNA analyses // Mol. Mar. Biol. Biotechnol. V. 7. № 2. P. 145–152.
Do H., Dobrovic A. 2012. Dramatic reduction of sequence artefacts from DNA isolated from formalin fixed cancer biopsies by treatment with uracil-DNA glycosylase // Oncotarget. V. 3. P. 546–558.
Hykin S.M., Bi K., McGuire J.A. 2015. Fixing formalin: a method to recover genomic-scale DNA sequence data from formalin-fixed museum specimens using high-throughput sequencing // PLoS ONE. V. 10. № 10. P. e0141579. https://doi.org/10.1371/journal.pone.0141579
Johnson G.D., Paxton J.R., Sutton T.T. et al. 2009. Deep-sea mystery solved: astonishing larval transformations and extreme sexual dimorphism unite three fish families // Biol. Lett. V. 5. P. 235–239. https://doi.org/10.1098/rsbl.2008.0722
Kearse M., Moir R., Wilson A. et al. 2012. Geneious basic: an integrated and extendable desktop software platform for the organization and analysis of sequence data // Bioinformatics. V. 28. № 12. P. 1647–1649.
Kilpatrick C.W. 2002. Noncryogenic preservation of mammalian tissues for DNA extraction: an assessment of storage methods // Biochem. Genet. V. 40. № 1–2. P. 53–62. https://doi.org/10.1023/A:1014541222816
Klopfleisch R., Weiss A.T.A., Gruber A.D. 2011. Excavation of a buried treasure – DNA, mRNA, miRNA and protein analysis in formalin fixed, paraffin embedded tissues // Histol. Histopathol. V. 26. P. 797–810.
Longmire J.L., Maltbie M., Baker R.J. 1997. Use of “lysis buffer” in DNA isolation and its implications for museum collections // Occas. Pap. Mus. Texas Tech. Univ. V. 163. P. 1–3.
Nagy Z.T. 2010. A hands-on overview of tissue preservation methods for molecular genetic analyses // Org. Divers. Evol. V. 10. № 1. P. 91–105. https://doi.org/10.1007/s13127-010-0012-4
NCBI. 2018. National Center for Biotechnology Information. (https://www.ncbi.nlm.nih.gov/Blast.cgi)
Quach N., Goodman M.F., Shibata D. 2004. In vitro mutation artifacts after formalin fixation and error prone translesion synthesis during PCR // BMC Clin. Pathol. V. 4. № 1. 5 p. https://doi.org/10.1186/1472-6890-4-1
Quicke D.L.J., Lopez-Vaamonde C., Belshaw R. 1999. Preservation of hymenopteran specimens for subsequent molecular and morphological study // Zool. Scripta. V. 28. № 1–2. P. 261–267. https://doi.org/10.1046/j.1463-6409.1999.00004.x
Paireder S., Werner B., Bailer J. et al. 2013. Comparison of protocols for DNA extraction from long-term preserved formalin fixed tissues // Analyt. Biochem. V. 439. P. 152–160. https://doi.org/10.1016/j.ab.2013.04.006
Pisani G.R. 1973. A guide to preservation techniques for amphibians and reptiles. Lawrence: Soc. Study Amphibians and Reptiles, 26 p.
Post R.J., Flook P.K., Millest A.L. 1993. Methods for the preservation of insects for DNA studies // Biochem. Syst. Ecol. V. 21. № 1. P. 85–92.
Schander C., Halanych K.M. 2003. DNA, PCR and formalinized animal tissue – a short review and protocols // Org. Divers. Evol. V. 3. P. 195–205.
Serth J., Kuczyk M.A., Paeslack U. et al. 2000. Quantitation of DNA extracted after micropreparation of cells from frozen and formalin-fixed tissue sections // Amer. J. Pathol. V. 156. P. 1189–1196. https://doi.org/10.1016/S0002-9440(10)64989-9
Seutin G., White B.N., Boag P.T. 1991. Preservation of avian blood and tissue samples for DNA analysis // Can. J. Zool. V. 69. P. 82–90.
Shedlock A.M., Haygood M.G., Pietsch T.W., Bentzen P. 1997. Enhanced DNA extraction and PCR amplification of mitochondrial genes from formalin-fixed museum specimens // BioTechniques. V. 22. P. 394–400.
Srinivasan M., Sedmak D., Jewell S. 2002. Effect of fixatives and tissue processing on the content and integrity of nucleic acids // Amer. J. Pathol. V. 161. № 6. P. 1961–1971.
Steinke D., Hanner R. 2011. The FISH-BOL collaborators' protocol // Mitochondrial DNA. V. 22. P. 10–14. https://doi.org/10.3109/19401736.2010.536538
Strona G., Stefani F., Galli P. 2009. Field preservation of monogenean parasites for molecular and morphological analyses // Parasitol. Int. V. 58. P. 51–54. https://doi.org/10.1016/j.parint.2008.10.001
Vivien R., Ferrari B.J.D., Pawlowski J. 2016. DNA barcoding of formalin‑fixed aquatic oligochaetes for biomonitoring // BMC Res. Notes. V. 9. P. 342–345. https://doi.org/10.1186/s13104-016-2140-1
Ward R.D., Zemlak T.S., Innes B.H. et al. 2005. DNA barcoding Australia’s fish species // Philos. Trans. Roy. Soc. Biol. Sci. V. 360B. P. 1847–1857.
Williams S.T. 2007. Safe and legal shipment of tissues samples: does it affect DNA quality? // J. Molluscan Stud. V. 73. P. 416–418.
Wong S.Q., Li J., Tan A.Y.-C. et al. 2014. Sequence artefacts in a prospective series of formalin-fixed tumours tested for mutations in hotspot regions by massively parallel sequencing // BMC Med. Genomics. V. 7. P. 1–10. https://doi.org/10.1186/1755-8794-7-23
Yoder M., Tandingan De Ley I., King I. et al. 2006. DESS: a versatile solution for preserving morphology and extractable DNA of nematodes // Nematology. V. 8. № 3. P. 367–376.
Дополнительные материалы отсутствуют.
Инструменты
Вопросы ихтиологии


