Вопросы ихтиологии, 2023, T. 63, № 3, стр. 251-264
Новый вид китовидковой рыбы рода Cetomimus (Cetomimidae) из тропической части Центральной Атлантики
С. Г. Кобылянский 1, *, Н. В. Гордеева 1, 2, А. В. Мишин 1
1 Институт океанологии РАН – ИО РАН
Москва, Россия
2 Институт общей генетики РАН – ИОГен РАН
Москва, Россия
* E-mail: kobylianskysg@gmail.com
Поступила в редакцию 20.06.2022
После доработки 31.07.2022
Принята к публикации 16.08.2022
- EDN: BYIWGT
- DOI: 10.31857/S0042875223030098
Аннотация
Приводится описание нового для науки вида батипелагической китовидковой рыбы рода Cetomimus, два экземпляра которого были пойманы над центральной тропической частью Срединно-Атлантического хребта на горизонтах 1500–0 и 2680–0 м. Для вида характерны 3½ жаберные дуги (за третьей жаберной дугой имеется маленькое отверстие), развитая только вокруг ануса и над основаниями первого–шестого лучей анального плавника кавернозная ткань; большие поры канала боковой линии, приблизительно равные ширине этого канала; отсутствие крупных, клапановидных треугольных выростов по краю перемычек между порами в задней части боковой линии; 42–48 позвонков; 20, 17–18 и 18 лучей соответственно в грудном, спинном и анальном плавниках; 17–18 пор в канале боковой линии между верхним краем жаберной крышки и концом хвостового стебля, а также некоторые иные диагностические признаки. Приведены первые молекулярно-генетические данные для описываемого вида (последовательности COX-1 мтДНК, или ДНК-баркоды), а также анализ межвидовой дивергенции в роде Cetomimus.
Род Cetomimus Goode et Bean, 1895 (Cetomimidae), описанный лишь по самкам, насчитывает в настоящее время семь номинальных видов и морфологически подразделён согласно имеющейся классификации (Harry, 1952) на два подрода – монотипический Psapharocetus Harry, 1952 (с единственным видом C. kerdops Parr, 1934) и Cetomimus s. str., к которому относятся шесть уже известных видов, а также описываемый в настоящей статье новый вид. Особую группу среди Cetomimus s. str. образует один ещё не описанный вид китовидок Cetomimus sp. (Paxton et al., 2016), обладающий сравнительно укороченным телом и малым числом пор в боковой линии (11–14), окаймлённых в задней части тела крупными мягкими клапановидными выростами, основания которых шире участка канала боковой линии между порами (Paxton et al., 2016). Вторая группа объединяет прочие виды подрода с заметно вытянутым в длину телом и относительно большим числом пор боковой линии (15–28), которые в задней её части или полностью лишены каких-либо окаймляющих выростов, или эти выросты (у большинства видов, за исключением C. teevani Harry, 1952), сравнительно небольших размеров, а их основания ýже канала боковой линии между порами. К этой группе относят виды с тремя целиком сформированными жаберными дугами и полностью редуцированной четвёртой жаберной дугой (отверстие за третьей дугой отсутствует) – C. gillii Goode et Bean, 1895; C. craneae Harry, 1952 и C. teevani, а также виды, у которых к верхней части третьей жаберной дуги примыкает сильно редуцированная и укороченная четвёртая жаберная дуга, несущая на себе лишь несколько укороченных жаберных лепестков (имеется очень маленькое отверстие между третьей и четвёртой дугами) – C. picklei (Gilchrist, 1922), C. hempeli, Maul, 1969, C. compunctus Abe, Marumo et Kawaguchi, 1965 и описанный в настоящей работе новый вид (Harry, 1952; Abe et al., 1965; Maul, 1969; Paxton, 1986; Paxton, Bray, 1986; Angulo, 2015; Paxton et al., 2016). Таксономическая ревизия рода Cetomimus до сих пор не проведена, поэтому представления о его объёме и видовом составе остаются до конца не определёнными. Помимо уже известных видов имеются также данные о по крайней мере пяти ещё не описанных видовых формах рода Cetomimus (Paxton, Bray, 1986; Paxton, 1989). Все представители Cetomimus – чрезвычайно редко встречающиеся в уловах мезо- и батипелагические рыбы, которые нигде не достигают значительной численности. За всё время исследований во всех трёх основных океанах было поймано лишь немногим более 180 экз. этого рода, причём их подавляющее большинство было собрано в Атлантике (Brauer, 1906; Craddock, Mead, 1970; Paxton, 1989; Tolley et al., 1989; Angulo, 2015). Число станций, на которых были пойманы разные виды Cetomimus, очень невелико, поэтому каждое новое нахождение поставляет дополнительную информацию об их географическом распространении и батиметрическом распределении.
В последние годы для описания новых таксонов глубоководных рыб и анализа их положения в общей системе костных рыб наряду с применением традиционных методик сравнительной анатомии и морфологии нашли широкое применение методы молекулярной генетики, позволяющие оценить как генетическое разнообразие различных таксономических групп, так и их филогенетические связи. ДНК-баркодинг является одним из наиболее распространённых и эффективных инструментов идентификации, классификации и анализа видов на основе короткого (~650 пар нуклеотидов) стандартного фрагмента ДНК митохондриального гена первой субъединицы цитохромоксидазы c (COX-1) (Hebert et al., 2003). Применение этого инструментария подходит не только для идентификации видов с помощью поиска референсной последовательности COX-1 в имеющихся базах генетических данных (BoldSystems11 и NCBI22), но и для предварительного выделения групп конспецифичных последовательностей нуклеотидов и определения на их основе межвидовых границ в общем массиве полученной информации. ДНК-баркодинг также пригоден для выявления и анализа сложных или незавершённых эволюционных процессов внутри видов и/или их групп, идентификации криптических видов, определения интрогрессии и гибридизации, неполной дивергенции филогенетических линий и так далее. У рыб результативность ДНК-баркодинга для идентификации различных морских и пресноводных видов, как правило, превышает 90% (Ward et al., 2005; Hubert et al., 2008; Pereira et al., 2013). Обычно в качестве стандартного порога межвидовой дивергенции принимается величина 2%, которая была рассчитана эмпирически из распределения внутри- и межвидовых оценок генетических дистанций (Ward, 2009). Однако нередки случаи более слабых межвидовых различий, тем не менее позволяющие корректно идентифицировать виды по ДНК-баркоду (Ward et al., 2005).
Некоторые вопросы классической морфологической таксономии глубоководных рыб могут быть разрешены лишь при применении методов молекулярной генетики. Например, анализ митохондриального (мт) генома помог установить, что представители семейств Mirapinnidae (Eutaeniophorus festivus) и Megalomycteridae (Ataxolepis apus), сильно различающиеся морфологически, на самом деле являются соответственно личинками и самцами китовидок рода Cetomimus, типовые экземпляры которых в коллекциях, напомним, до этого были представлены исключительно самками (Johnson et al., 2009). Установление родовой синонимии указанных представителей трёх семейств обнаруживает уникальный случай сочетания радикальной онтогенетической трансформации и полового диморфизма у позвоночных животных, сопряжённых с существенными преобразованиями скелета.
Необходимость привлечения дополнительных методов при исследовании двух экземпляров описанного в настоящей работе нового вида Cetomimus (помимо анализа морфологии) также была обусловлена и тем, что при общей схожести пропорций тела и значений счётных признаков у них наблюдаются значительные различия в числе позвонков (48 у голотипа и 42 у паратипа). Поскольку все китовидковые рыбы встречаются в уловах лишь спорадически, сложно дать экспертную оценку пределов варьирования пластических и меристических признаков в популяции каждого конкретного вида. Поэтому для исключения ошибочных определений и подтверждения конспецифичности имеющихся у нас экземпляров был осуществлён анализ их мтДНК.
Таким образом цель настоящей работы – привести морфологическое описание нового вида рода Cetomimus, а также проанализировать межвидовые границы в роде Cetomimus с использованием полученных авторами ДНК-баркодов и имеющихся данных из баз генетической информации с применением нескольких методов, основанных на анализе парных дистанций (ABGD) и филогении (GMYC и bPTP).
МАТЕРИАЛ И МЕТОДИКА
Материалом послужили 2 экз. (самки) рыб, пойманных в 37-м и 39-м рейсах научно-исследовательского судна (НИС) “Профессор Логачев” (2015 и 2018 гг.) над центральной тропической частью Срединно-Атлантического хребта (САХ). Рыбы пойманы незамыкающимся разноглубинным тралом Айзекса–Кидда в модификации Самышева–Асеева (РТАКСА) во время двух ночных тралений в пелагиали над ложем рифтовой долины САХ в олиготрофных периферических водах Центрального Северо-Атлантического антициклонического круговорота. Материал хранится в коллекции Зоологического музея Московского государственного университета (ЗММУ). При обработке материала за основу приняли схему измерений и подсчётов пластических и меристических признаков, ранее применявшуюся для других видов Cetomimus (Harry, 1952; Maul, 1969; Angulo, 2015). В работе приняты следующие обозначения: SL – стандартная длина тела рыбы, c – длина головы, ao – длина рыла, о – горизонтальный диаметр глаза, po – заглазничное расстояние, hc – высота головы на вертикали середины орбиты, io – ширина межглазничного промежутка, lmx – длина верхней челюсти, lmd – длина нижней челюсти, imd – поперечное расстояние между задними окончаниями нижних челюстей правой и левой сторон тела, H – высота тела на вертикали основания грудного плавника, h – высота хвостового стебля, lpc – длина хвостового стебля; aD, aP, aA – антедорсальное, антепекторальное и антеанальное расстояния; lD, lA – длина оснований спинного и анального плавников; lP – длина грудного плавника; pD1, pA1 – расстояния от начала оснований спинного и анального плавников до конца хвостового стебля; pD2, pA2 – расстояния от конца оснований спинного и анального плавников до хвостового стебля; lC – длина хвостового плавника; D, A, P – число лучей в спинном, анальном и грудном плавниках; C – суммарное число сегментированных лучей в хвостовом плавнике; Cd, Cv – число сегментированных лучей в дорсальной и вентральной лопастях хвостового плавника; ll – число пор боковой линии тела между задневерхним краем жаберной крышки и окончанием хвостового стебля, vert. – суммарное число туловищных и хвостовых позвонков, включая уростиль.
Для молекулярно-генетического анализа помимо двух указанных типовых экземпляров использовали также самца (ст. 2172-1, 07.11–08.11.2009 г., 04°40′ ю.ш., 12°16′ з.д. , горизонт лова 1800–0 м) и личинку (ст. 2184-3, 19.11.2009 г., 29°28′ ю.ш., 00°11′ в.д., горизонт лова 2000–0 м) Cetomimus sp., собранных авторами ранее в Южной Атлантике в 21-м рейсе НИС “Академик Иоффе”. Экстракцию геномной ДНК из фиксированных в 96%-м этаноле образцов мышечной ткани проводили с использованием коммерческого набора DiatomTM DNA Prep100 (“Лаборатория ИЗОГЕН”, Россия). Для амплификации и секвенирования фрагмента COX-1 мтДНК использовали универсальные праймеры FishF1 и FishR2 (Ward et al., 2005). Редактирование хроматограмм и сведение их в консенсусные последовательности проводили в программе Geneious33. Полученные последовательности длиной 622–647 пар нуклеотидов (п.н.) были депонированы в GenBank/NCBI (таблица).
Для реконструкции филогенетических связей в дополнение к собственным данным использовали последовательности COX-1 мтДНК представителей Cetomimidae и близкородственного вида Barbourisia rufa (семейство Barbourisiidae) из NCBI и BOLD (таблица). В перечне заимствованных последовательностей из генетических баз данных в таблице видовые названия приведены так, как они в них были зарегистрированы, в том числе и утратившие валидность видовые названия Ataxolepis apus (самцы Cetomimus spp.) и Eutaeniophorus festivus (личинки Cetomimus spp.). После выравнивания всех 28 последовательностей их конечная длина составила 523 п.н., что превышает минимальную длину (486 п.н.) стандартного ДНК-баркода (Hanner, 2009).
Список последовательностей COX-1 мтДНК, представленных в реестре генетических баз данных (NCBI и BOLD) и использованных для анализа филогении и межвидовой дивергенции представителей Cetomimidae и близкородственного вида Barbourisia rufa (Barbourisiidae)
| Гаплотип | Идентификация в реестре баз данных | Источник информации | |
|---|---|---|---|
| номер (ID) | вид | ||
| Cetomimus paxtoni | |||
| (♂)* | AP010887 | Ataxolepis apus | Johnson et al., 2009 |
| (♀)* | ON810776 | Cetomimus paxtoni (голотип) | Наши данные |
| (♂) | SCAFB1317-09 | Ataxolepis apus | BOLD |
| (♀) | UKFBJ1157-08 | Cetomimus sp. | То же |
| (♀)* | ON810777 | Cetomimus paxtoni (паратип) | Наши данные |
| Cetomimus | |||
| sp. 3 (♂)* | ON810779 | Ataxolepis apus | Наши данные |
| sp. 1 (♀)* | GLF133-14 | Cetomimus sp. | BOLD |
| sp. 4 (♀)* | AP010881 | Cetomimus Atl. sp. 1 | Johnson et al., 2009 |
| sp. 1 (Lrv.)* | AP010885 | Eutaeniophorus festivus | То же |
| sp. 2 (Lrv.)* | ON810778 | То же | Наши данные |
| Gyrinomimus grahami | |||
| * | FJ164637 | Gyrinomimus grahami | NCBI |
| FJ164638 | То же | То же | |
| AP010883 | Gyrinomimus sp. nov. B2 | Johnson et al., 2009 | |
| Gyrinomimus myersi* | AP010884 | Gyrinomimus myersi | То же |
| Ditropichthys storeri* | FNZ198-06 | Ditropichthys storeri | BOLD |
| Danacetichthys galathenus* | AP002936 | Danacetichthys galathenus | Johnson et al., 2009 |
| Cetostoma regani | |||
| 1* | GU071727 | Cetostoma regani | Bucklin et al., 2010 |
| 1 | AP010886 | Parataeniophorus gulosus | Johnson et al., 2009 |
| 2* | AP004423 | Cetostoma regani | То же |
| 2 | AP004424 | Parataeniophorus gulosus | » |
| 3* | KY033585 | Cetostoma regani | Kenchington et al., 2017 |
| 3 | KY033586 | То же | То же |
| 3 | AP010882 | » | Johnson et al., 2009 |
| 3 | MAECO412-09 | » | BOLD |
| Procetichthys kreffti* | AP010880 | Procetichthys kreffti | Johnson et al., 2009 |
| Barbourisia rufa | |||
| 1* | JQ354000 | Barbourisia rufa | Elz et al., 2012 |
| 2* | AP010879 | То же | Johnson et al., 2009 |
| 2 | FMVIC153-08 | » | BOLD |
Примечание. * Гаплотип представлен на рис. 6; ♂ – самец, ♀ – самка, Lrv. – личинка; BOLD – Barcode of Life Data System V. 4 (https://www.boldsystems.org. Version 06/2022); NCBI – NIH genetic sequence database (https://www.ncbi.nlm.nih.gov. Version 06/2022).
Поиск межвидовых границ среди анализируемых последовательностей COX-1 мтДНК и определение гипотетических групп (видов) в роде Cetomimus и у других представителей Cetomimidae осуществляли с применением нескольких методов. Первый метод – ABGD, или автоматический поиск “разрыва”, так называемого “barcoding gap”, в оценках внутри- и межвидовых генетических дистанций (Puillandre et al., 2012). В рамках этого метода подразумевается, что внутривидовая дивергенция много меньше межвидовой; стандартом для видов животных считаются их десятикратное различие (Hebert et al., 2004). Матрицу генетических расстояний на основе двухпараметрической модели Кимуры (K2P) (Kimura, 1980) рассчитывали в программе Mega v. 7 и затем анализировали в онлайн-версии программы ABGD (http://www.abi.snv.jussieu.fr/public/abgd/abgdweb.html. Version 05/2022) с заданными значениями “разрыва” (X) 1.0 и внутривидовой дивергенции (P) между 0.001 (0.1%) и 0.1 (10%).
Для разделения видов также использовали построение филогенетических деревьев, применяя два метода – GMYC (Generalized Mixed Yule Coalescent method, Sahu et al., 2016) и bPTP (Bayesian implementation of the Poisson tree Processes, Zhang et al., 2013).
Для анализа GMYC исключили повторяющиеся последовательности в онлайн-сервисе FaBox (Villesen, 2007), затем с оставшимися гаплотипами (таблица) построили ультраметрическую дендрограмму в программе BEAST v1.8.0 (Drummond, Rambaut, 2007). Выбор оптимальной модели эволюции проводили в программе jModeltest (Posada, 2008) на платформе CIPRES Science Gateway version 3.3 web service (Miller et al., 2010). Согласно байесовскому информационному критерию (BIC), наилучшее соответствие показала модель: HKY + G (G = 0.14). Использовали стандартную оценку скорости мутации мтДНК у рыб − 1% в млн лет (Bermingham et al., 1997). Реконструкцию филогении на основании алгоритма Байеса проводили с помощью 10 млн итераций метода MCMC (Markov chain Monte Carlo) с шагом 1000. Точность анализа проверяли в программе Tracer v. 1.7.1 (Rambaut et al., 2018) на основании показателя эффективного размера выборки (ESS) > 200. Сформированный в программе TreeAnotator (Drummond et al., 2012) файл с филогенетическим деревом в формате Newick затем использовали для анализа GMYC в онлайн-сервисе http://species.h-its.org/gmyc. Для графического представления филогении реконструкцию BEAST визуализировали в программе FigTree44. Анализ в программе bPTP проводили на основании филогении, реконструированной с использованием метода максимального правдоподобия (maximum-likelihood, ML) в программе RAxML 8.0 (Kozlov et al., 2019) с помощью 1000 “быстрых” бутстреп-повторов. Для выделения кластеров (видов) на основании K2P-дистанций при заданных значениях межвидовой дивергенции (1 и 2%) использовали программу TaxonDNA/Species Identifier v. 1.8 (Meier et al., 2006).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Изучение внешней морфологии двух имеющихся в нашем распоряжении экземпляров рода Cetomimus, пойманных в Центральной Атлантике, показало, что они в должной мере отличаются от других известных представителей рода, чтобы считаться новым для науки видом, описание которого дано ниже.
Cetomimus paxtoni sp. nova
(рис. 1)
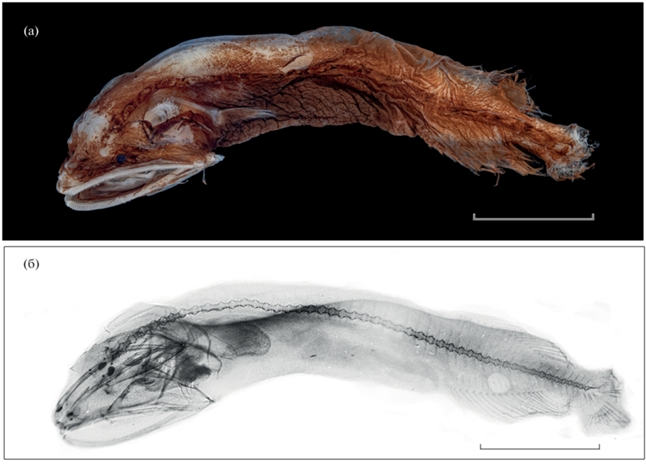
Рис. 1.
Внешний вид (а) и рентгенограмма (б) голотипа Cetomimus paxtoni sp. nov. ЗММУ P-24520 SL 50.5 мм. Масштаб: 10 мм.
Ataxolepis apus (non Myers et Freihofer, 1966) Johnson et al., 2009 – самец, GenBank ID SCAFB1317-09, 43°48′32′′ с.ш., 58°55′19′′ з.д.; самец, GenBank ID AP010887, 39°50′46′′ с.ш., 67°19′59′′ з.д.
Cetomimus sp. – самка, BOLD Systems ID UKFBJ1157-08, 39°57′32′′ с.ш., 67°19′48′′ з.д.
Голотип. Самка SL 50.5 мм, НИС “Профессор Логачев”, 39-й рейс, станция 39L188rt, 28.02–01.03.2018 г., 13°47′45′′–13°52′44′′ с.ш., 44°58′34′′–45°00′49′′ з.д., глубина места 3532 м, РТАКСА, горизонт лова 1500–0 м, ЗММУ P-24520 (гаплотип GenBank ID ON810776).
Паратип. Самка SL 52.0 мм, НИС “Профессор Логачев”, 37-й рейс, станция 37L112rt, 08.02.2015 г., 17°08′13′′ с.ш., 46°30′05′′ з.д., глубина места 4000 м, РТАКСА, горизонт лова 2680–0 м, ЗММУ P-24521 (гаплотип GenBank ID ON810777).
Диагноз. Вид Cetomimus c 3½ жаберными дугами (за третьей жаберной дугой имеется маленькое отверстие), с кавернозной тканью вокруг ануса и над основаниями первого–шестого лучей анального плавника и отсутствующей в других частях тела; с большими порами канала боковой линии, приблизительно равными ширине этого канала; без крупных клапановидных треугольных выростов по краю перемычек между порами в задней части боковой линии; с 42–48 позвонками; с 20, 17–18 и 18 лучами соответственно в грудном, спинном и анальном плавниках; с 17–18 порами в канале боковой линии между вертикалью верхнего края жаберной крышки и концом хвостового стебля; с невысоким телом, максимальная высота которого составляет 15–16% SL; с относительно узким и длинным хвостовым стеблем, высота которого содержится более двух раз в его длине; с головой средних размеров, составляющей 30–31% SL; с широко закруглённым в дорсальной проекции рылом, с диаметром глаза и длиной верхней челюсти, составляющими соответственно 6 и 84–87% длины головы.
Описание. D 18 (17)55, A 18 (18), P 20 (20), C 17 (17), Cd 8 (9), Cv 9 (8), ll 17 (18), vert. 48 (42).
Тело невысокое, заметно вытянуто в длину, сжато с боков; на вертикали оснований грудных плавников в поперечном сечении имеет форму вытянутого овала; отчётливо сужается от головы к хвосту; наибольшей высоты достигает примерно на вертикали начала основания грудных плавников; наибольшая высота тела содержится 6.3 (6.5) раза в SL. Грудные плавники слабые, относительно короткие, их основания смещены ближе к вентральному краю тела. Основание первого луча грудного плавника расположено несколько выше горизонтали нижнего края орбиты; все лучи соединены между собой плавниковой мембраной. Брюшные плавники отсутствуют. Кавернозная ткань на основании спинного плавника и перед ним, а также на хвостовом стебле и истмусе отсутствует (рис. 2а). Анальное отверстие располагается непосредственно перед началом основания анального плавника и окружено участком кавернозной ткани, которая далее простирается вдоль основания анального плавника вплоть до вертикали пятого (шестого) луча (рис. 2б). Хвостовой стебель относительно длинный, низкий; его высота укладывается 2.1 (2.1) раза в собственной длине. Основания спинного и анального плавников смещены далеко назад и имеют почти супротивное расположение. Начало основания первого луча анального плавника расположено слегка перед вертикалью начала основания спинного. Длина основания анального плавника в 1.1 (1.1) раза превосходит длину основания спинного. Все парные и непарные плавники лишены шипов. Чешуя на теле и голове отсутствует; кожа очень мягкая, легко собирается в складки. Замкнутый сейсмосенсорный канал боковой линии на теле имеется; при этом он прободён очень крупными, вытянутыми в длину вдоль горизонтальной оси тела овальными порами. Их ширина примерно равна ширине канала боковой линии. Передний участок канала боковой линии также частично заходит на дорсолатеральную поверхность головы, где он простирается вперёд от вертикали верхнего угла жаберной крышки приблизительно до вертикали переднего края орбиты и открывается наружу четырьмя крупными порами. Целиком замкнутые участки канала боковой линии, располагающиеся между порами боковой линии, имеют вид узких перемычек из кожи. Задний край некоторых перемычек между порами в хвостовой части тела несёт очень маленькие, треугольной формы выросты, частично прикрывающие сверху каждую последующую пору; ширина их основания заметно меньше ширины канала боковой линии. На наружной поверхности каждого замкнутого участка задней части канала боковой линии зачастую располагается пара маленьких, супротивно сидящих, коричневых папилл, разделённых между собой килевидной продольной складкой кожи (рис. 3а). Иногда за этой парой имеются одна или две дополнительные папиллы. В ряде работ такие образования идентифицируют как слизевые трубки (“mucus-tubes”) (Harry, 1952) или их причисляют к светящимся органам (Maul, 1969). Мы, однако, более склонны рассматривать их в качестве невромастов. Свободно сидящие папиллы располагаются также на коже дорсальной поверхности передней части спины за головой (рис. 3б), на передней части рыла, на дорсальной поверхности головы в межглазничном пространстве в виде поперечной полосы (рис. 3 в), а также на коже вдоль нижнечелюстного сейсмосенсорного канала. Вершины некоторых из них имеют белёсый цвет, что производит впечатление наличия здесь выходного отверстия. Все каналы сейсмосенсорной системы головы замкнутые, сверху покрыты кожей и открываются наружу немногочисленными, очень крупными порами.
Рис. 2.
Основания спинного (а) и анального (б) плавников голотипа Cetomimus paxtoni sp. nov.: lep – первые плавниковые лучи, kav – кавернозная ткань, an – анальное отверстие. Масштаб: 1 мм.

Рис. 3.
Особенности строения папилл (→) на различных частях тела голотипа Cetomimus paxtoni sp.nov.: а – участок боковой линии на хвостовом стебле, б – спина, в – теменная часть головы; pl – пора боковой линии. Масштаб: 1 мм.

Голова средних размеров, её дорсальный край заметно выгнут; длина головы содержится 3.3 (3.3) раза в SL. Глаз очень маленький, 16.9 (15.9) раза в длине головы, смещён ближе к краю верхней челюсти, сильно редуцирован, зрачок имеется, хрусталик отсутствует. Межглазничный промежуток в 6.3 (6.0) раза превышает горизонтальный диаметр глаза. Рыло относительно длинное, содержится 2.6 (2.7) раза в длине головы, его передний конец в дорсальной проекции широко закруглён. Обонятельная капсула открывается наружу двумя ноздрями, расположенными в переднебоковой части рыла; передняя ноздря округлая, имеет заметно приподнятый и утолщённый передний край, задняя слегка вытянута вдоль горизонтальной оси; клапаны на краях ноздрей отсутствуют. Рот очень большой, конечный. Верхняя челюсть несколько вогнута внутрь в своей средней части, её задний конец слегка расширен, простирается далеко за вертикаль заднего края орбиты. Длина верхней челюсти содержится 1.2 (1.1) раза в длине головы. Нижняя челюсть, напротив, слегка выгнута наружу так, что при смыкании челюстей заметной щели между ними не образуется. Длина нижней челюсти примерно равна длине головы. В задней части нижней челюсти сразу за вертикалью окончания praemaxillare имеется короткий, латерально направленный шип; задняя часть anguloarticulare имеет вид уплощённого треугольного отростка, доходящего до оснований лучей грудного плавника. Окончания отростков anguloarticularia правой и левой сторон тела в вентральной проекции широко разделены между собой; расстояние между ними содержится 3.4 (3.4) раза в длине нижней челюсти. Зубы на челюстях расположены в неровных, поперечных диагональных рядах, очень мелкие, с относительно широкими основаниями, увеличиваются в размерах от внешнего края челюсти к внутреннему; вершины наиболее крупных внутренних зубов заметно загнуты в каудальном направлении (рис. 4а); в передней части premaxilaria диагональные поперечные ряды содержат по пять зубов, тогда как в диагональных рядах в передней части dentalia – шесть зубов. На головке сошника (vomer) имеется куполообразное округлое зубное пятно, содержащее у голотипа 48 зубов, вершины которых обращены по направлению к глотке (рис. 4б). Зубы на palatinum и ectopterygoideum организованы в вытянутые зубные пятна. В жаберной полости имеются три полностью сформированные жаберные дуги; к верхней части третьей жаберной дуги также примыкает очень короткая, сильно редуцированная четвёртая жаберная дуга. Между третьей и четвёртой дугами имеется очень маленькое отверстие. В литературных источниках подобное строение жаберного аппарата обозначается как 3½ или 3.5 (Maul, 1969; Angulo, 2015). Жаберные тычинки на всех жаберных дугах отсутствуют. Первые три жаберные дуги несут на себе элементы озубления в виде накладных костных пластинок, покрытых зубами; озубление в наибольшей степени развито на первой жаберной дуге. Все basibranchialia жаберных дуг также покрыты единой длинной и узкой зубной пластинкой (рис. 4в), имеющей форму гантели; в передней и задней своей частях она немного расширена, а в центральной несколько сужена. Ширина этой пластинки в центральной части составляет лишь ~10% её длины. На hypobranchiale-1 имеется одна относительно крупная зубная пластинка веретеновидной формы. На ceratobranchiale-1 и epibranchiale-1 зубные пластинки занимают почти целиком всю длину и ширину этих костей; симфизис между ними также несёт маленькую, покрытую зубами костную пластинку. К передней и задней частям pharyngobranchiale-1 примыкает по одной выпуклой зубной пластинке (рис 4б). Многочисленные, загнутые в каудальном направлении зубы на зубных пластинках жаберных дуг имеют форму, схожую с таковой зубов на верхней и нижней челюстях, однако имеют заметно меньшие размеры. Псевдобранхия отсутствует.
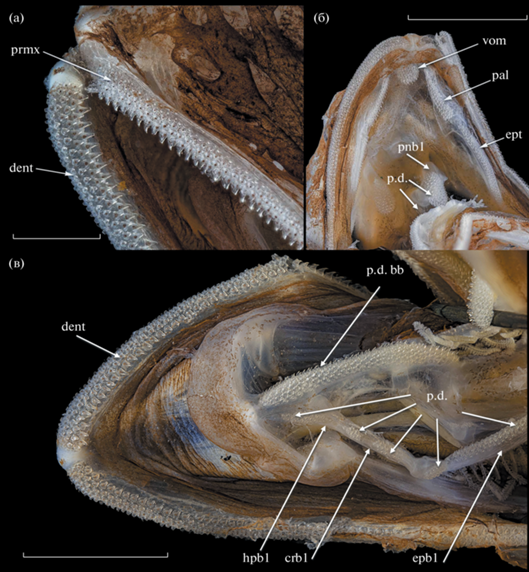
Рис. 4.
Особенности озубления челюстей, некоторых элементов нейрокраниума, нёбно-крыловидной, гиоидной и жаберной дуг голотипа Cetomimus paxtoni sp.nov.: а – верхняя и нижняя челюсти, б – нёбо и элементы верхней части скелета жаберных дуг, в – нижняя челюсть и элементы скелета жаберных дуг. prmx – praemaxillare, dent – dentale, vom – vomer, pal – palatinum, ept – ectopterygoideum; phb1, hpb1, crb1 и epb1 – соответственно pharyngobranchiale, hypobranchiale, ceratobranchiale и epibranchiale первой жаберной дуги; bb – basibranchiale, p.d. – plattum dentale. Масштаб, мм: а – 0.5, б – 5, в – 2.
Измерения голотипа и паратипа. SL 50.5 и 52.0 мм. В % SL: c 30.1 (30.6), ao 11.5 (11.2), o 1.8 (1.9), po 16.6 (17.7), hc 12.3 (13.8), io 11.3 (11.5), lmx 25.3 (26.7), lmd 29.1 (30.4), imd 8.5 (9.0), H 15.8 (15.4), h 5.9 (5.8), lpc 12.7 (11.9), aD 69.7 (72.5), aP 31.1 (31.4), aA 67.7 (72.1), lD 14.7 (15.6), lP 9.5 (8.3), lA 16.6 (17.5), lC – (8.8), pD1 26.7 (28.1), pD2 12.3 (12.1), pA1 27.3 (26.5), pA2 11.1 (11.0). В % c: ao 38.2 (36.5), o 5.9 (6.3), po 55.3 (57.9), hc 40.3 (45.8), io 37.5 (37.7), lmx 84.2 (87.4), lmd 96.7 (99.4), imd 28.3 (29.6).
Окраска тела и головы фиксированных этанолом рыб однотонная, варьирует от коричневой (голотип) до тёмно-коричневой (паратип), что, возможно, обусловлено особенностями их состояния перед фиксацией. Жаберно-ротовая полость коричневая. В толще основания каждого зуба на обеих челюстях у голотипа имеется крупный, округлый меланофор коричневого цвета.
Этимология. Вид назван в честь Д.Р. Пакстона (J.R. Paxton) – замечательного австралийского ихтиолога, внёсшего выдающийся вклад в изучение рыб семейства Cetomimidae.
Сравнительные замечания. C. paxtoni вместе с C. picklei, C. compunctus и C. hempeli принадлежит к группе видов, обладающих 3½ жаберными дугами. Согласно описанию C. picklei, известному лишь по голотипу, основным диагностическим признаком, отличающим этот вид от других представителей рода, является очень малое число пор в канале боковой линии (10 против 15–28) (Gilchrist, 1922). Эту же информацию далее тиражировали и в более поздних работах, посвящённых описанию новых видов и составу фауны китовидковых рыб (Harry, 1952; Maul, 1969; Angulo, 2015). Между тем, необходимо иметь в виду и публикацию Пакстона и Брэя (Paxton, Bray, 1986), в которой приводится фотография голотипа C. picklei, а также ряд важных диагностических признаков этого вида, отсутствующих в работе Гилхриста (Gilchrist, 1922). Из переописания и фотографии C. picklei следует, что этот вид обладает вытянутым и относительно узким телом, а в канале его боковой линии насчитывается 21 пора, а не 10, как было указано в первоописании. Этот вид имеет маленькое отверстие за третьей жаберной дугой, что свидетельствует о наличии укороченной рудиментарной четвёртой жаберной дуги (общее число дуг 3½), 56 позвонков, а также участок кавернозной ткани, простирающейся вдоль основания анального плавника – с первого по шестой его луч. Кавернозная ткань на других частях тела C. picklei отсутствует. Таким образом, согласно вышеприведённым данным, C. paxtoni хорошо отличается от C. picklei заметно меньшим числом позвонков (42–48 против 56), меньшим числом пор в туловищном канале боковой линии (17–18 против 21), а также несколько бо́льшим числом лучей в спинном, грудном и анальном плавниках (соответственно 17–18 против 16; 20 против 18 и 18 против 16). У C. paxtoni по сравнению с C. picklei больше длина верхней челюсти (25.3–26.7 против 12.3% SL) и максимальная высота тела (15.4–15.8 против 14.3), а также меньше антедорсальное расстояние (69.7–72.5 против 76.9% SL).
C. paxtoni отличается от C. compunctus отсутствием кавернозной ткани перед и на передней части основания спинного плавника, а также на дорсальной и вентральной частях хвостового стебля; у него меньше позвонков (42–48 против 52) и пор в туловищном канале боковой линии (17–18 против 26), меньше число лучей в грудном плавнике (20 против 23–24), меньше максимальная высота тела (15.4–15.8 против 25.4% SL), длина головы (30.1–30.6 против 35.2% SL), а также длина основания спинного и анального плавников (соответственно 14.7–15.6 против 20.4 и 16.6–17.5 против 19.7% SL) и, напротив, больше длина верхней челюсти (25.3–26.7 против 20.8% SL), диаметр глаза (1.8–1.9 против 0.5% SL), антедорсальное и антеанальное расстояния (соответственно 69.7–72.5 против 59.9 и 67.7–72.1 против 59.5% SL).
C. paxtoni отличается от типовых экземпляров C. hempeli отсутствием кавернозной ткани перед и на начале основания спинного плавника, а также некоторыми меристическими и пластическими признаками – числом пор в туловищном канале боковой линии (17–18 против 20), числом лучей в грудном плавнике (20 против 22–23), максимальной высотой тела (15.4–15.8 против 17.9–18.6% SL), длиной головы (30.1–30.6 против 34.3–35.3% SL), длиной верхней челюсти (25.3–26.7 против 27.9–32.7% SL), антедорсальным расстоянием (69.7–72.5 против 75.0–78.2% SL) и длиной основания анального плавника (16.6–17.5 против 14.3–15.4% SL). Данные о C. hempeli также приведены в обзоре китовидковых рыб из вод Мексиканского залива (Tolley et al., 1989). Однако сведения о внешнем виде четырёх пойманных экземпляров, приведённые в этой работе, очень скудны, а подсчёты некоторых меристических признаков имеют довольно большой разброс значений, что, возможно, свидетельствует о причислении авторами к C. hempeli разных видовых форм. Поэтому вопрос о конспецифичности упомянутых рыб типовым экземплярам в значительной мере остаётся открытым, в связи с чем мы предпочли не ссылаться на эти материалы, как на 100% адекватно идентифицированные.
Основным признаком, отличающим C. paxtoni от C. craneae, C. teevani и C. gillii, составляющих отдельную видовую группу, является отсутствие у трёх последних видов рудиментарной четвёртой жаберной дуги и, как следствие, отверстия за третьей жаберной дугой.
Помимо этого важного, на наш взгляд, признака C. paxtoni отличается от C. craneae отсутствием кавернозной ткани перед и на основании спинного плавника, меньшим числом пор в канале боковой линии (17–18 против 22) и лучей в грудном плавнике (20 против 23), а также рядом пропорций тела и головы – меньшими максимальной высотой тела (15.4–15.8 против 20.9% SL), антедорсальным расстоянием (69.7–72.5 против 76.7% SL), длиной головы (30.1–30.6 против 36.7% SL), длиной верхней челюсти (25.3–26.7 против 28.9% SL), шириной межглазничного промежутка (11.3–11.5 против 14.9% SL) и бо́льшим диаметром глаза (1.80–1.90 против 0.85% SL).
C. paxtoni отличается от C. teevani отсутствием пятна кавернозной ткани перед и на основании спинного плавника, очень крупными порами канала боковой линии, ширина которых равна ширине канала (у C. teevani поры относительно маленькие, их ширина не превышает половину ширины канала боковой линии); отсутствием крупных клапановидных выростов по заднему краю перемычек между порами в каудальной части боковой линии, вдвое превышающих по длине горизонтальный диаметр пор. C. paxtoni также отличается от C. teevani меньшим числом позвонков (42–48 против 50), меньшими максимальной высотой тела (15.4–15.8 против 21.5% SL), длиной головы (30.1–30.6 против 37.8% SL) и верхней челюсти (25.3–26.7 против 30.1% SL), а также длиной основания спинного плавника и антедорсальным расстоянием (соответственно 14.7–15.6 против 19.9 и 69.7–72.5 против 77.6% SL).
C. paxtoni внешне весьма похож на C. gillii, однако помимо отмеченных выше различий в строении жаберного аппарата новый вид отличается от голотипа C. gillii несколько бо́льшим числом лучей в грудном, спинном и анальном плавниках (соответственно 20 против 16, 17–18 против 16 и 18 против 16), меньшими длиной головы (30.1–30.6 против 36.4% SL) и антедорсальным расстоянием (69.7–72.5 против 75.0% SL), а также максимальной высотой тела (15.4–15.8 против 22.8% SL). Из данных литературы, в которых приведены некоторые сведения о морфологических признаках других экземпляров C. gillii, пойманных в Атлантическом (9 экз.) (Parr, 1928; Harry, 1952; Tolley et al., 1989) и Тихом (1 экз.) океанах (Angulo, 2015), следует, что число лучей в грудном плавнике (16, редко 17) у этого вида, по-видимому, является довольно консервативным, мало изменчивым признаком, пригодным для его идентификации.
Экземпляр рода Cetomimus SL 41.0 мм из Индийского океана, описанный Брауером (Brauer, 1906) как C. gillii и позднее переописанный Молем (Maul, 1969), который указал на его явные отличия от голотипа C. gillii, на наш взгляд, довольно схож с C. paxtoni по наличию 3½ жаберных дуг и отверстия за третьей жаберной дугой; отсутствию кавернозной ткани на других участках тела, кроме начала основания анального плавника; наличию близких значений числа пор в туловищном канале боковой линии (около 19 против 17–18) и лучей в грудном, спинном и анальном плавниках (соответственно 21 против 20, 19 против 17–18 и 19 против 18). При этом указанный экземпляр при несколько меньшей длине тела отчётливо отличается от C. paxtoni по ряду пластических признаков – у него больше максимальная высота тела (25.6 против 15.4–15.8% SL), антедорсальное расстояние (76.8 против 69.7–72.5% SL), длина основания спинного и анального плавников (22.4 против 14.7–15.6 и 22.0 против 16.6–17.5% SL), а также длина головы и верхней челюсти (соответственно 35.4 против 30.1–30.6 и 31.7 против 25.3–26.7% SL). Исходя из различий в пропорциях тела резонно возникают сомнения в конспецифичности C. paxtoni и этого экземпляра. Возможно, мы имеем дело с ещё одним не описанным видом, относящимся к той же видовой группе.
C. paxtoni хорошо отличается от C. kerdops (подрод Psapharocetus) широко закруглённым (против приострённого) в дорсальной проекции рылом и далеко расставленными (против сходящимися) друг от друга в вентральной проекции задними окончаниями правого и левого anguloarticularе нижней челюсти (Harry, 1952).
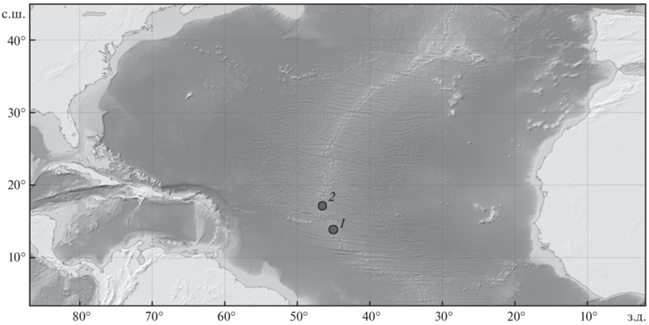
Распространение. Голотип и паратип C. paxtoni пойманы в тропических водах Центральной Атлантики, в пелагиали над рифтовой долиной САХ (рис. 5), на горизонтах соответственно 1500–0 и 2680–0 м.
Рис. 5.
Места поимки голотипа (1) и паратипа (2) Cetomimus paxtoni sp. nov. в Центральной Атлантике.
Молекулярно-генетический анализ. Филогенетическая реконструкция положения основных родов (Cetostoma, Gyrinomimus, Danacetichthys, Ditropichthys, Procetichthys) семейства Cetomimidae (рис. 6) по результатам анализа COX-1 мтДНК в целом согласуется с опубликованными ранее данными (Johnson et al., 2009; Кобылянский и др., 2020). Что же касается видового состава рода Cetomimus, необходимо сразу оговориться, что в силу малого числа собранных образцов, имеющихся в базах генетических данных, эти молекулярно-генетические исследования находятся в начальной стадии. Поэтому число номинальных видов Cetomimus, описанных по морфологическим признакам, значительно превосходит таковое, зарегистрированное в базах NCBI и BOLD, при том что зачастую они даже не определены до видового уровня. Тем не менее, если оперировать известными генетическими данными, то число кластеров (потенциальных видов) в роде Cetomimus варьирует от одного до пяти в зависимости от использованного подхода. С применением автоматической оценки уровня межвидовых различий (ABGD), равно как и с использованием стандартного для большинства видов рыб уровня межвидовой дивергенции 2% в настоящее время удаётся выделить только три группы последовательностей. Отдельными видами оказываются самец Cetomimus sp. 3 из тропической Южной Атлантики (ON810779) и самка Cetomimus sp. 4 (AP010881, = Cetomimus Atl. sp. 1 из работы: Johnson et al., 2009), а также совокупность всех прочих известных гаплотипов Cetomimus, включая C. paxtoni.
Рис. 6.
Определение межвидовых границ в семействе Cetomimidae на основании изменчивости COX-1 мтДНК (таблица). Столбики обозначают число групп последовательностей COX-1 мтДНК, выделяемых с использованием разных методов: ABGD, GMYC, bPTP и выделение кластеров
(видов) на основании K2P- дистанций при заданных значениях (1 и 2%) межвидовой дивергенции.
Филогенетические связи отображены в виде байесовской ультраметрической дендрограммы.
Поддержка узлов ветвления (постериорная вероятность): ⚫ ≥ 0.95, 0.95 >  ≥ 0.75, ⚪ < 0.75. Для представителей рода Cetomimus указана жизненная форма (♂ – самец, ♀ – самка, Lrv. – личинка), * голотип, ** паратип.
Масштаб длины ветвей указан слева внизу в абсолютных величинах числа замен на сайт.
≥ 0.75, ⚪ < 0.75. Для представителей рода Cetomimus указана жизненная форма (♂ – самец, ♀ – самка, Lrv. – личинка), * голотип, ** паратип.
Масштаб длины ветвей указан слева внизу в абсолютных величинах числа замен на сайт.
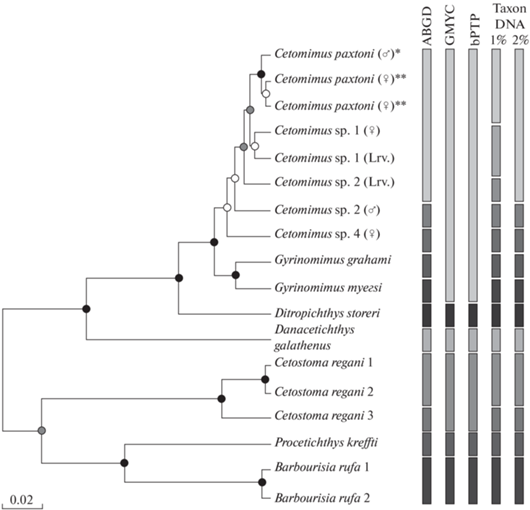
При более расширенном подходе и 1%-ном уровне межвидовой дивергенции в роде Cetomimus на кладограмме выделяются пять кластеров, составляющих максимальное их число. В одном кластере вместе с новым видом C. paxtoni оказываются североатлантические образцы: два самца – SCAFB1317-09, 43°48′32′′ с.ш., 58°55′19′′ з.д. и AP010887, 39°50′46′ с.ш., 67°19′59′′ з.д., а также одна самка UKFBJ1157-08, 39°57′32′′ с.ш., 67°19′48′′ з.д. (на рис. 6 не обозначены). Все они, по-видимому, также могут быть причислены к виду C. paxtoni, однако мы не включили их в типовой материал, поскольку не имели возможности ознакомиться с ваучерными экземплярами. Кроме того, в дополнение к уже упомянутым выделяются ещё два потенциально валидных вида – Cetomimus sp. 1, самка которого была поймана у берегов Гренландии (GLF133-14, 62°01′12′′ с.ш., 40°10′48′′ з.д.), а личинка – вблизи побережья Флориды (AP010885, 25°30′ с.ш., 79°19′ з.д.), а также Cetomimus sp. 2, известный лишь по поимке его личинки в Южной Атлантике в районе Китового хребта (ON810778). Оценить принадлежность упомянутых здесь гаплотипов Cetomimus к какому-либо из уже описанных номинальных видов пока не представляется возможным, поскольку для этого необходимо детальное сравнение ваучерных экземпляров (если они имеются в музейных коллекциях) с типовыми материалами, по которым были произведены первоописания данных видов. Проведённый анализ лишь позволяет сделать предварительный вывод о том, что у глубоководных китовидковых рыб рода Cetomimus межвидовая дивергенция может быть неглубокой, ниже стандартного порогового значения 2%. Обычно это связывают с недавними процессами дивергенции (радиации) и небольшим эволюционным возрастом видов (Pereira et al., 2013). Этот вопрос требует дальнейшего изучения с привлечением новых материалов для морфологических и молекулярно-генетических исследований этой группы рыб. Тем не менее, полученные результаты свидетельствуют о большом значении данных ДНК-баркодинга для идентификации видов китовидковых рыб и дальнейшего развития молекулярной систематики Cetomimidae.
Список литературы
Кобылянский С.Г., Гордеева Н.В., Котляр А.Н. 2020. Новые нахождения редкого вида Rondeletia bicolor (Stephanoberycoidei) над Срединно-Атлантическим хребтом и некоторые вопросы филогении семейства Rondeletiidae // Вопр. ихтиологии. Т. 60. № 1. С. 16–25. https://doi.org/10.31857/S0042875220010099
Abe T., Marumo R., Kawaguchi K. 1965. Description of a new cetomimid fish from Suruga Bay // Jpn. J. Ichthyol. V. 12. № 3–6. P. 57–63. https://doi.org/10.11369/jji1950.12.57
Angulo A. 2015. Cetomimus gillii Goode & Bean, 1895 (Cetomimiformes: Cetomimidae): range extension and first record in the Tropical Eastern Pacific // Mar. Biodivers. Rec. V. 8. Article e22. https://doi.org/10.1017/S1755267215000019
Bermingham E., McCafferty S., Martin A.P. 1997. Fish biogeography and molecular clocks: perspectives from the Panamanian isthmus // Molecular systematics of fishes. San Diego et al.: Acad. Press. P. 113–128. https://doi.org/10.1016/B978-012417540-2/50009-9
Brauer A. 1906. Die Tiefsee-Fische. L. Systematischer Teil. Jena: G. Fischer, 432 p.
Bucklin A., Ortman B.D., Jennings R.M. et al. 2010. A “Rosetta Stone” for metazoan zooplankton: DNA barcode analysis of species diversity of the Sargasso Sea (Northwest Atlantic Ocean) // Deep Sea Res. Part II. Top. Stud. Oceanogr. V. 57. № 24–26. P. 2234–2247. https://doi.org/10.1016/j.dsr2.2010.09.025
Craddock J.E., Mead G.W. 1970. Midwater fishes from the Eastern South Pacific Ocean // Sci. Res. Southeast Pac. Exped. Contribut. Anton Bruun Rept. № 3. P. 1–46.
Drummond A., Rambaut A. 2007. BEAST: Bayesian evolutionary analysis by sampling trees // BMC Evol. Biol. V. 7. Article 214. https://doi.org/10.1186/1471-2148-7-214
Drummond A.J., Suchard M.A., Xie D. et al. 2012. Bayesian Phylogenetics with BEAUti and the BEAST v.1.7 // Mol. Biol. Evol. V. 29. № 8. P. 1969–1973. https://doi.org/10.1093/molbev/mss075
Elz A., Schwenke P., Park L. 2012. NWFSC forensic marinefish voucher collection (https://www.ncbi.nlm.nih.gov/nuccore/JQ354000. Version 08/2022).
Gilchrist J.D.F. 1922. Deep-sea fishes procured by the S.S. “Pickle” (Part I) // Report of the fisheries and marine biological survey, Union of South Africa. № 2. P. 41–79.
Hanner R. 2009. Data Standards for BARCODE Records in INSDC (BRIs) // BARCODE Data Standards v.2.4. P. 1–4. (https://repository.si.edu/bitstream/handle/10088/96-518/BARCODE%20Data%20Standards%20v2.4.pdf?-sequence=1&isAllowed=y. Version 08/2022).
Harry R.R. 1952. Deep-sea fishes of the Bermuda Oceanographic Expeditions, families Cetomimidae and Rondeletiidae // Zoologica. V. 37. Pt. 1. P. 55–72. https://doi.org/10.5962/p.184690
Hebert P.D.N., Cywinska A., Ball S.L. et al. 2003. Biological identifications through DNA barcodes // Proc. R. Soc. Lond. B. V. 270. № 1512. P. 313–321. https://doi.org/10.1098/rspb.2002.2218
Hebert P.D.N., Stoeckle M.Y.,Tyler S. et al. 2004. Identification of Birds through DNA Barcodes // PLoS Biol. V. 2. № 10. Article e312. P. 1657–1663. https://doi.org/10.1371/journal.pbio.0020312
Hubert N., Hanner R., Holm E. et al. 2008. Identifying Canadian freshwater fishes through DNA barcodes // PLoS One. V. 3. № 6. Article e2490. https://doi.org/10.1371/journal.pone.0002490
Johnson G.D., Paxton J.R., Sutton T.T. et al. 2009. Deep-sea mystery solved: astonishing larval transformations and extreme sexual dimorphism unite three fish families // Biol. Lett. V. 5. № 2. P. 235–239. https://doi.org/10.1098/rsbl.2008.0722
Kenchington E.L., Baillie S.M., Kenchington T.J. et al. 2017. Barcoding Atlantic Canada’s mesopelagic and upper bathypelagic marine fishes // PLoS One. V. 12. № 9. Article e0185173. https://doi.org/10.1371/journal.pone.0185173
Kimura M. 1980. A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences // J. Mol. Evol. V. 16. № 2. P. 111–120. https://doi.org/10.1007/BF01731581
Kozlov A.M., Darriba D., Flouri T. et al. 2019. RAxML-NG: A fast, scalable, and user-friendly tool for maximum likelihood phylogenetic inference // Bioinformatics. V. 35. № 21. P. 4453–4455. https://doi.org/10.1093/bioinformatics/btz305
Maul G.E. 1969. On the genus Cetomimus (Cetomimidae) with the description of a new species // Bocagiana. № 18. P. 1–12
Meier R., Shiyang K., Vaidya G. et al. 2006. DNA barcoding and taxonomy in Diptera: a tale of high intraspecific variability and low identification success // Syst. Biol. V. 55. № 5. P. 715–728. https://doi.org/10.1080/10635150600969864
Miller M.A., Pfeiffer W., Schwartz T. 2010. Creating the CIPRES Science Gateway for inference of large phylogenetic trees // Gateway Computing Environments Workshop (GCE). New Orleans: IEEE. P. 1–8. https://doi.org/10.1109/GCE.2010.5676129
Parr A.E. 1928. Deep-sea fishes of the order Iniomi from the waters around the Bahama and Bermuda Islands // Bull. Bingham Oceanogr. Coll. V. 3. № 3. P. 1–193.
Paxton J.R. 1986. Family Cetomimidae // Fishes of the North-Eastern Atlantic and the Mediterranean. Paris: UNESCO. V. 2. P. 524–525.
Paxton J.R. 1989. Synopsis of the whalefishes (family Cetomimidae) with descriptions of four new genera // Rec. Aust. Mus. V. 41. № 2. P. 135–206. https://doi.org/10.3853/j.0067-1975.41.1989.141
Paxton J.R., Bray D.J. 1986. Order Cetomimiformes // Smiths’ Sea fishes. Berlin: Springer. P. 433–434.
Paxton J.R., Trnski T., Johnson G.D. 2016. Cetomimidae // The living marine resources of the Eastern Central Atlantic. Rome: FAO. V. 3. Pt. 1. P. 2174–2182.
Pereira L.H., Hanner R., Foresti F. et al. 2013. Can DNA barcoding accurately discriminate megadiverse Neotropical freshwater fish fauna? // BMC Genet. V. 14. Article 20. https://doi.org/10.1186/1471-2156-14-20
Posada D. 2008. jModelTest: phylogenetic model averaging // Mol. Biol. Evol. V. 25. № 7. P. 1253–1256. https://doi.org/10.1093/molbev/msn083
Puillandre N., Lambert A., S. Brouillet S. et al. 2012. ABGD, Automatic Barcode Gap Discovery for primaryspecies delimitation // Mol. Ecol. V. 21. № 8. P. 1864–1877. https://doi.org/10.1111/j.1365-294X.2011.05239.x
Rambaut A., Drummond A.J., Xie D. et al. 2018. Posterior summarization in Bayesian phylogenetics using Tracer 1.7 // Systematic Biol. V. 67. № 5. P. 907–904. https://doi.org/10.1093/sysbio/syy032
Sahu S.K., Singh R., Kathiresan K. 2016. Multi-gene phylogenetic analysis reveals the multiple origin and evolution of mangrove physiological traits through exaptation // Estuar. Coast. Shelf. Sci. V. 183. Pt. A. P. 41–51. https://doi.org/10.1016/j.ecss.2016.10.021
Tolley S.G., Gartner J.V., Lancraft T.M. 1989. Whalefishes (Beryciformes: Cetomimoidei) of the gulf of Mexico // Bull. Mar. Sci. V. 45. № 3. P. 671–677.
Villesen P. 2007. FaBox: an online toolbox for FASTA sequences // Mol. Ecol. Notes. V. 7. № 6. P. 965–968. https://doi.org/10.1111/j.1471-8286.2007.01821.x
Ward R.D. 2009. DNA barcode divergence among species and genera of birds and fishes // Mol. Ecol. Res. V. 9. № 4. P. 1077–1085. https://doi.org/10.1111/j.1755-0998.2009.02541.x
Ward R.D., Zemlak T.S., Innes B.H. et al. 2005. DNA barcoding Australia’s fish species // Philos. Trans. R. Soc. B. V. 360. № 1462. P. 1847–1857. https://doi.org/10.1098/rstb.2005.1716
Zhang J., Kapli P., Pavlidis P. et al. 2013. A General species delimitation method with applications to phylogenetic placements // Bioinformatics. V. 29. № 22. P. 2869–2876. https://doi.org/10.1093/bioinformatics/btt499
Дополнительные материалы отсутствуют.
Инструменты
Вопросы ихтиологии


