Известия РАН. Серия биологическая, 2019, № 2, стр. 144-153
Дифференциация камчатских популяций микижи Parasalmo (Oncorhynchus) mykiss по локусам микросателлитной ДНК
С. Д. Павлов , *, А. В. Семенова , М. Н. Мельникова
Московский государственный университет им. М.В. Ломоносова, биологический ф-т
119234 Москва, Ленинские горы, 1, стр. 12, Россия
* E-mail: serge_pavlov@mail.ru
Поступила в редакцию 26.02.2018
После доработки 23.10.2018
Принята к публикации 23.10.2018
Аннотация
Уточнены имеющиеся сведения о дифференциации камчатских популяций микижи Parasalmo (Oncorhynchus) mykiss по локусам микросателлитной ДНК. С использованием как традиционных оценок генетической дифференциации (общие и попарные FST-статистики), так и методов Байесовской кластеризации определены популяционно-генетические отношения вида в камчатском регионе. Подтверждено, что обособленность камчатских популяций микижи приурочена к крупным речным бассейнам Западной и Восточной Камчатки. Отмечено, что наиболее обособлены по микросателлитным локусам микижа р. Жупанова (Восточная Камчатка), а также выборки из рек Квачина и Утхолок (Западная Камчатка).
Камчатская микижа Parasalmo (Oncorhynchus) mykiss, или микижа, которая относится к семейству лососевых рыб (Salmonidae) – объект промысла и аквакультуры мирового значения. Несмотря на широкую рыбохозяйственную интродукцию в обоих земных полушариях, природное распространение этого вида ограничено северной бореалью Пацифики. Нативные популяции микижи встречаются по североамериканскому и азиатскому (преимущественно п-ов Камчатка) побережьям Тихого океана. Вид отличается высокой морфоэкологической пластичностью и сложной популяционной структурой. Высокий уровень адаптивности к различным условиям обитания проявляется в существовании у вида различных жизненных стратегий, которые могут быть представлены разными экоформами в пределах одной популяции (Павлов и др., 2001). Исследование таких популяционно-сложных, или “комплексных”, видов требует особенно пристального изучения природных генофондов, использования существующей изменчивости митохондриального и ядерного генома для понимания популяционных отношений и особенностей существования каждого конкретного вида. Полиморфизм локусов микросателлитной (ядерной) ДНК широко используется для подобных задач.
Современный уровень исследований полиморфизма локусов микросателлитной ДНК (STR-локусов) у микижи определен авторами многочисленных работ, изучавших интродуцированные и природные популяции Северной и Южной Америки, Европы (McCuscer et al., 2000; Beacham et al., 2004; Narum et al., 2004; Lulla et al., 2005; Olsen et al., 2006; Taylor et al., 2007; Abadia-Cardoso et al., 2016; Leitwein et al., 2017 и др.). Множество этих работ контрастирует с единичными популяционно-генетическими исследованиями микижи по микросателлитам в азиатской части ареала. Первое изучение полиморфизма STR-локусов, проведенное на собранном нами материале (Williams et al., 2002), выявило пространственно-генетическую дифференциацию у камчатских популяций вида. В дальнейшем совместно с американскими коллегами (McPhee et al., 2007) было указано на малоэффективность прямого копирования локусов микросателлитной ДНК, высокополиморфных у североамериканских форелей, для камчатских популяций микижи. Последующие работы в этой области (Семенова и др., 2010; Павлов и др., 2011) подтвердили географическую уникальность камчатской микижи, ее региональную различимость и дифференциацию.
Цель работы – уточнение генетических отношений между популяциями микижи внутри азиатского региона.
МАТЕРИАЛЫ И МЕТОДЫ
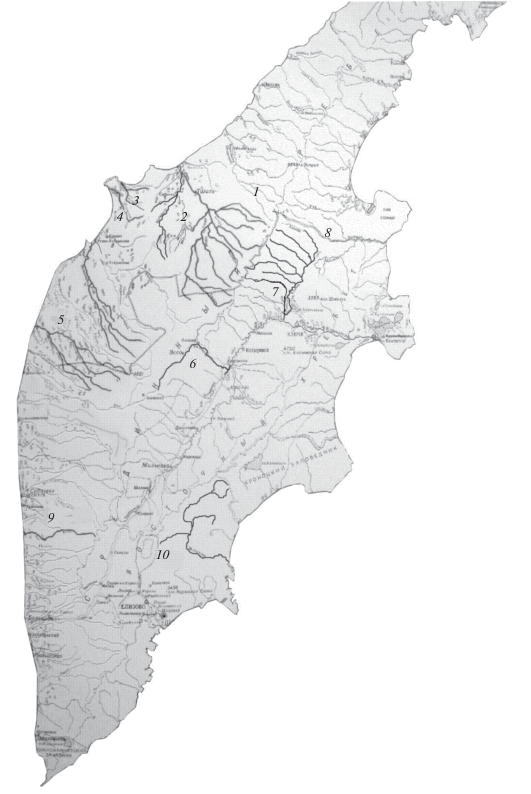
Материалом для исследования полиморфизма микросателлитных локусов послужили выборки микижи из речных бассейнов западного и восточного побережий Камчатки, собранные в 2002–2013 гг. (табл. 1, рис. 1). В работе использованы коллекции кафедры ихтиологии МГУ, собранные авторами статьи, а также К.В. Кузищиным, М.А. Груздевой, А.Л. Сенчуковой. Данные о генетической изменчивости некоторой части выборок, а также методики выделения и амплификации ДНК и характеристики 10 STR-локусов были опубликованы ранее (Павлов и др., 2011).
Таблица 1.
Характеристика исследованного материала
| Номер выборки* |
Река (район сбора) |
Годы сбора | Объем выборки | Обозначение выборки |
|---|---|---|---|---|
| 1 | Тигиль | 2002 | 35 | TIG |
| 2 | Седанка | 2002 | 33 | SED |
| 3 | Квачина | 2003, 2010 | 57 | KVA |
| 4 | Утхолок | 2004 | 53 | UTH |
| 5 | Сопочная | 2002 | 25 | SOP |
| 6 | Быстрая | 2005 | 42 | BST |
| 7 | Двухюрточная | 2004 | 45 | DVU |
| 8 | Коль | 2004 | 26 | KOL |
| 9 | Еловка | 2006 | 17 | ELV |
| 10 | Жупанова | 2013 | 54 | ZHP |
* Нумерация выборок соответствует их локальностям на рис. 1.
Данные по всем локусам были проанализированы в целях обнаружения возможных ошибок генотипирования, а также присутствия нуль-аллелей с помощью программы MICROCHECKER (Van Oosterhout et al., 2004). Проверку влияния предполагаемых нуль-аллелей на FST-оценки генетической дифференциации проводили с использованием рекомендаций и программы FREENA (Chapuis, Estoup, 2007).
Программа GDA (http://lewis.eeb.unconn.edu/lewishome/software.html) была использована для оценки частот аллелей, аллельного разнообразия (А), ожидаемой и наблюдаемой гетерозиготности (HE и HO соответственно), cтепени дифференциации популяций (θ). Отклонения от равновесия Харди–Вайнберга были тестированы с использованием коэффициента инбридинга FIS в программе GENEPOP on the Web (http://genepop.curtin.edu.au), их достоверность была оценена с использованием точных тестов Фишера. Оценки аллельного разнообразия (AR), скорректированные по минимальному размеру выборки, были получены в программе FSTAT 2.9.3 (http://www2.unil.ch/popgen/softwares/fstat.htm).
Однофакторный дисперсионный анализ (ANOVA) был проведен в программе Excel для оценки значимости различий AR и HE между выборками. Программа GENEPOP была использована для оценки неравновесности по сцеплению, а также попарной дифференциации популяций на основании коэффициентов FST (Weir, Cockerham, 1984).
Для того чтобы выявить, проходили ли исследуемые популяции микижи периоды уменьшения размера популяций или “горлышка бутылки” в недавнем прошлом, мы использовали программу BOTTLENECK 1.2.02 (Piry et al., 1999). Односторонний тест Уилкоксона был использован для трех различных моделей: бесконечного числа аллелей (IAM), пошаговой мутационной (SMM) и двухфазной (TPM). Достоверный избыток гетерозиготности указывает на недавнюю редукцию популяционного размера (Сornuet, Luikart, 1996). Для каждой популяции учитывались только полиморфные локусы в равновесии Харди–Вайнберга.
Анализ популяционной структуры проводился методом Байеса в программе STRUCTURE 2.3.4 (Pritchard et al., 2000) с использованием модели, допускающей генетическое смешение и корреляцию аллельных частот среди кластеров с 400 000 burn-in- и 800 000 MCMC-итерациями для К (гипотетического числа популяций) от 1 до 10, по три анализа для каждого значения К.
Построение UPGMA-дендрограмм и оценку бутстреп-поддержки осуществляли с помощью программы Phylip 3.65 (http://evolution.gs.washington.edu/phylip/html).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Все исследованные нами микросателлитные локусы, за исключением One104, были полиморфны, число аллелей в локусе варьировало от 2 (One103) до 20 (Oki10), в среднем 9.7, всего было обнаружено 97 различных аллелей (A). Показатели генетической изменчивости в выборках приведены в табл. 2.
Таблица 2.
Генетическая изменчивость популяций микижи по полиморфным микросателлитным локусам
| Локус | Показатель | Выборка | Среднее на локус | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| KVA | UTH | SOP | TIG | SED | KOL | ELV | DVU | BST | ZHP | |||
| Ssa197 | n | 57 | 53 | 25 | 35 | 33 | 26 | 17 | 45 | 42 | 52 | 38.5 |
| A/AR | 2/1.25 | 2/1.85 | 2/2 | 2/2 | 2/2 | 2/2 | 2/2 | 2/1.99 | 3/2.33 | 2/2 | 3/2.03 | |
| HE | 0.017 | 0.107 | 0.428 | 0.506 | 0.505 | 0.448 | 0.427 | 0.295 | 0.331 | 0.504 | 0.381 | |
| HO | 0.017 | 0.113 | 0.44 | 0.571 | 0.515 | 0.576 | 0.235 | 0.222 | 0.285 | 0.615 | 0.332 | |
| Ssa20.19 | n | 55 | 53 | 25 | 35 | 33 | 26 | 17 | 45 | 42 | 54 | 38.5 |
| A/AR | 4/3.22 | 3/2.79 | 4/3.52 | 3/2.97 | 3/2.98 | 3/2.99 | 2/2 | 3/2.99 | 3/2.95 | 3/2.98 | 5/3.02 | |
| HE | 0.597 | 0.538 | 0.595 | 0.59 | 0.598 | 0.619 | 0.427 | 0.574 | 0.567 | 0.573 | 0.582 | |
| HO | 0.6 | 0.49 | 0.52 | 0.628 | 0.666 | 0.384 | 0.117 | 0.533 | 0.5 | 0.463 | 0.514 | |
| One103 | n | 57 | 53 | 25 | 34 | 33 | 26 | 17 | 45 | 42 | 53 | 38.5 |
| A/AR | 2/2 | 2/2 | 2/1.98 | 2/2 | 2/1.89 | 2/2 | 2/2 | 2/2 | 2/2 | 2/1.97 | 2/2 | |
| HE | 0.399 | 0.353 | 0.183 | 0.313 | 0.116 | 0.291 | 0.451 | 0.475 | 0.501 | 0.187 | 0.358 | |
| HO | 0.368 | 0.377 | 0.2 | 0.264 | 0.061 | 0.346 | 0.647 | 0.533 | 0.381 | 0.132 | 0.322 | |
| One108 | n | 57 | 53 | 25 | 35 | 32 | 26 | 17 | 45 | 40 | 53 | 38.3 |
| A/AR | 14/10.7 | 15/8.65 | 10/8.77 | 11/8.23 | 12/8.9 | 9/7.92 | 8/7.44 | 11/8.26 | 7/6.12 | 12/8.04 | 19/9.72 | |
| HE | 0.885 | 0.816 | 0.848 | 0.845 | 0.864 | 0.85 | 0.784 | 0.801 | 0.804 | 0.795 | 0.869 | |
| HO | 0.964 | 0.867 | 0.76 | 0.914 | 0.843 | 0.846 | 0.705 | 0.822 | 0.75 | 0.83 | 0.845 | |
| One111 | n | 57 | 48 | 21 | 34 | 32 | 26 | 14 | 44 | 39 | 53 | 36.8 |
| A/AR | 8/5.42 | 7/4.53 | 4/3.89 | 7/5.44 | 7/5.75 | 4/3.53 | 4/4 | 7/5.51 | 5/4.4 | 8/6.5 | 12/5.89 | |
| HE | 0.47 | 0.303 | 0.67 | 0.727 | 0.681 | 0.656 | 0.753 | 0.737 | 0.572 | 0.721 | 0.693 | |
| HO | 0.456 | 0.208 | 0.285 | 0.294 | 0.437 | 0.153 | 0.357 | 0.204 | 0.128 | 0.396 | 0.298 | |
| One112 | n | 57 | 53 | 25 | 35 | 33 | 26 | 17 | 45 | 40 | 53 | 38.4 |
| A/AR | 11/7.67 | 12/8.29 | 9/7.72 | 9/7.32 | 8/6.45 | 11/9.88 | 5/4.82 | 8/5.46 | 8/6.02 | 9/7.77 | 15/8.09 | |
| HE | 0.77 | 0.845 | 0.704 | 0.831 | 0.765 | 0.896 | 0.689 | 0.53 | 0.768 | 0.859 | 0.812 | |
| HO | 0.701 | 0.943 | 0.76 | 0.828 | 0.757 | 0.807 | 0.647 | 0.488 | 0.725 | 0.792 | 0.75 | |
| Ots3 | n | 57 | 53 | 25 | 35 | 33 | 26 | 17 | 45 | 41 | 49 | 38.1 |
| A/AR | 3/2.34 | 2/1.26 | 3/2.48 | 3/2.84 | 3/2.78 | 3/2.87 | 2/2 | 2/1.85 | 3/1.91 | 3/2.64 | 3/2.53 | |
| HE | 0.133 | 0.018 | 0.152 | 0.258 | 0.247 | 0.246 | 0.213 | 0.106 | 0.072 | 0.397 | 0.184 | |
| HO | 0.14 | 0.018 | 0.12 | 0.287 | 0.272 | 0.269 | 0.235 | 0.111 | 0.073 | 0.326 | 0.173 | |
| Oki10 | n | 57 | 53 | 25 | 35 | 33 | 26 | 17 | 45 | 41 | 53 | 38.5 |
| A/AR | 11/8.13 | 9/6.38 | 10/8.39 | 9/7.32 | 9/6.82 | 12/9.66 | 9/8.38 | 16/11.3 | 10/7.84 | 12/9.19 | 20/10.3 | |
| HE | 0.833 | 0.791 | 0.791 | 0.693 | 0.693 | 0.808 | 0.786 | 0.891 | 0.857 | 0.809 | 0.839 | |
| HO | 0.789 | 0.716 | 0.76 | 0.6 | 0.787 | 0.807 | 0.764 | 0.755 | 0.66 | 0.634 | 0.722 | |
| Omy1011 | n | 53 | 53 | 25 | 35 | 33 | 26 | 17 | 45 | 41 | 51 | 37.9 |
| A/AR | 5/4.64 | 8/6.05 | 7/6.57 | 7/6.46 | 9/5.53 | 7/5.61 | 7/6.47 | 7/5.84 | 8/6 | 9/7.07 | 17/6.73 | |
| HE | 0.722 | 0.721 | 0.789 | 0.65 | 0.641 | 0.666 | 0.711 | 0.612 | 0.627 | 0.808 | 0.731 | |
| HO | 0.622 | 0.792 | 0.76 | 0.571 | 0.696 | 0.73 | 0.706 | 0.577 | 0.658 | 0.882 | 0.701 | |
| Среднее ввыборке | n | 56.4 | 52.5 | 26.5 | 34.8 | 32.8 | 26 | 16.7 | 44.9 | 41 | 52.5 | |
| AAR | 6.1/4.64 | 6.1/4.28 | 5.2/4.63 | 5.4/4.56 | 5.6/4.41 | 5.4/4.74 | 4.2/4.01 | 5.9/4.62 | 5/4.06 | 6.1/4.91 | ||
| HE | 0.483 | 0.449 | 0.516 | 0.541 | 0.511 | 0.548 | 0.524 | 0.502 | 0.505 | 0.57 | ||
| HO | 0.466 | 0.452 | 0.46 | 0.495 | 0.503 | 0.492 | 0.441 | 0.425 | 0.413 | 0.509 | ||
Программа MICROCHECKER не обнаружила возможных ошибок генотипирования ни в одном из изученных локусов. В 8 из 10 выборок показана возможность присутствия нуль-аллелей в локусе One111. После проверки влияния возможных нуль-аллелей на оценки генетической дифференциации оказалось, что с учетом их влияния значения FST изменяются на 1–5.6%.
Достоверно значимые отклонения от равновесия Харди–Вайнберга (р < 0.05) в распределении генотипов наблюдаются в 8 из 100 оценок после проведения коррекции Бонферрони. Все они связаны с дефицитом гетерозигот вследствие присутствия нуль-аллелей в локусе One111 (табл. 2). В связи с возможным влиянием нуль-аллелей на статистические оценки информация по данному локусу была исключена из дальнейшего сравнительного анализа.
Оценки корреляции между генотипами по всем выборкам не обнаружили достоверных значений ни по одной паре локусов. Наибольшая изменчивость микижи наблюдалась по локусам Oki10, One108, One112. Наименее полиморфным оказался локус One103 (табл. 2).
Средние значения HE у микижи из разных рек были приблизительно сходными и варьировали от 0.449 (р. Утхолок) до 0.57 (р. Жупанова). Также довольно близкими были показатели аллельного разнообразия, среднее число аллелей в выборках, скорректированное по минимальному объему (AR), варьировало от 4.01 (р. Еловка) до 4.91 (р. Жупанова). Микижа из р. Жупанова характеризуется наибольшими значениями HE и AR среди исследованных выборок. Однофакторный дисперсионный анализ ANOVA не выявил достоверных различий между выборками ни по HE (F = 0.204, P = 0.99), ни по AR (F = 0.207, P = 0.98).
Анализ в программе BOTTLENECK не показал уменьшения размеров популяций ни в одной из исследованных выборок микижи (IAM: P > 0.32, TPM: P > 0.628, SMM: P > 0.843).
Генетическая гетерогенность всей совокупности выборок хорошо выражена. Показана достоверная дифференциация микижи по частотам аллелей и генотипов (G-тест, Р = 0–0.004) по каждому локусу, а также по всем локусам суммарно (Р = 0).
Попарные оценки межвыборочной дифференциации FST приведены в табл. 3. Статистически достоверна дифференциация FST в 42 случаях из 45 сравнений (FST = 0.001–0.142). Недостоверны сравнительные оценки в парах рек Тигиль–Седанка, Тигиль–Коль, Сопочная–Коль.
Таблица 3.
Оценки показателей попарной генетической дифференциации FST
| KVA | UTH | SOP | TIG | SED | KOL | ELV | DVU | BST | ZHP |
|---|---|---|---|---|---|---|---|---|---|
| KVA | |||||||||
| UTH | 0.023 | ||||||||
| SOP | 0.056 | 0.065 | |||||||
| TIG | 0.091 | 0.085 | 0.016 | ||||||
| SED | 0.106 | 0.103 | 0.021 | 0.003 | |||||
| KOL | 0.061 | 0.052 | 0.012 | 0.001 | 0.023 | ||||
| ELV | 0.074 | 0.088 | 0.024 | 0.023 | 0.078 | 0.02 | |||
| DVU | 0.065 | 0.1 | 0.039 | 0.059 | 0.073 | 0.05 | 0.013 | ||
| BST | 0.112 | 0.142 | 0.073 | 0.079 | 0.117 | 0.061 | 0.035 | 0.037 | |
| ZHP | 0.088 | 0.091 | 0.047 | 0.040 | 0.051 | 0.028 | 0.061 | 0.095 | 0.109 |
Степень генетической дифференциации среди всех исследованных выборок микижи была достоверно значима, θ = 5.97% с 95%-ным доверительным бутстреп-интервалом (4.26–8.85%). Наибольший вклад в дифференциацию вносят локусы Ssa197, One103 и Ots3 (θ = 17.2, 7.92 и 5.56% соответственно).
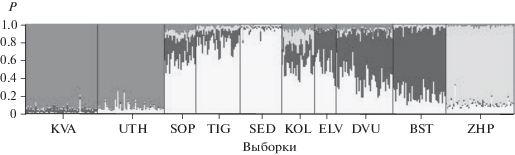
Результаты кластеризации в программе STRUCTURE показывают, что максимальная вероятность, соответствующая минимальной оценке лог-правдоподобия lnPr(X/K), показана для K = 4, т.е. принадлежности особей суммарной выборки к четырем кластерам (рис. 2). В первый кластер с долями вероятностей 0.936 и 0.918 обособляются выборки из рек Утхолок и Квачина соответственно. Второй кластер сформирован выборками из рек Сопочная, Тигиль, Седанка, Коль и Еловка со средними вероятностями 0.574, 0.758, 0.931, 0.5 и 0.541 соответственно. Третий кластер образуют выборки рек Двухюрточной и Быстрой, средние вероятности принадлежности к кластеру для этих выборок составляют 0.523 и 0.757 соответственно. Четвертый кластер представлен выборкой из р. Жупанова, средняя вероятность для этой выборки составляет 0.88.
Рис. 2.
Кластерный анализ микижи Камчатки в программе STRUCTURE. Обозначения выборок соответствуют приведенным в табл. 1; для рис. 2 и 3.
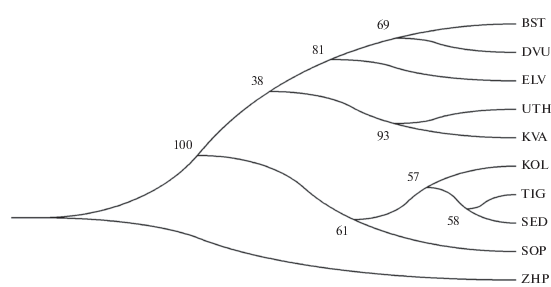
UPGMA-дендрограмма, построенная с использованием наиболее отличающейся от остальных выборки из р. Жупанова в качестве аут-группы, визуально отражает дифференциацию камчатской микижи на группы выборок, в основном соответствующих их географической локализации (рис. 3). Достаточно высокий уровень бутстреп-поддержки (>70%) имеет совместная кластеризация выборок из рек Быстрая–Двухюрточная, Утхолок–Квачина и Коль–Тигиль–Седанка–Сопочная.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В предыдущем исследовании (Павлов и др., 2011) было показано, что большинство камчатских популяций микижи дифференцируется по их принадлежности к крупным речным бассейнам. При этом на имеющемся в то время материале можно было выделить северо-западный, юго-западный и восточный кластеры пространственной дифференциации. Увеличение объемов выборок и применение новых методик статистической обработки результатов при использовании той же панели STR-локусов позволило уточнить эту картину (рис. 2).
Все исследованные выборки достаточно обособлены, большинство попарных оценок генетической дифференциации FST достоверны (табл. 3). Можно предполагать, что в отсутствие других факторов это связано с высокой степенью хоминга, устойчивой пространственной изоляцией и локальными адаптациями камчатской микижи. Это неудивительно, поскольку значительный уровень межпопуляционной генетической дифференциации характерен также и для популяций микижи из других частей ареала (Nielsen et al., 1997; Beacham et al., 1999, 2000; Heath et al., 2001; Silverstein et al., 2004; Taylor et al., 2007).
Кластеризация выборок с использованием UPGMA-дендрограммы демонстрирует для камчатских популяций микижи наиболее четкое филогеографическое дерево, коррелирующее с принадлежностью выборок к различным речным бассейнам и побережьям камчатского региона (рис. 3).
Значительно дифференцирована от других популяций микижа р. Жупанова. По использованным микросателлитным локусам для нее характерны наиболее высокие в азиатском регионе значения HE и АR. Ее отличия от остальной камчатской микижи подтверждаются не только результатами STR-анализа, но и другими участками ядерного и митохондриального генома, а также биологическими особенностями (Павлов, 2000; Павлов и др., 2001, 2004, 2011; Кузищин и др., 2002; Pavlov et al., 2010). Причины таких отличий и особенности происхождения микижи из этой реки требуют отдельного исследования, которое проводится в настоящее время.
Достаточно сильно обособлены выборки микижи из рек Утхолок и Квачина. Высокие отличия от других выборок микижи Камчатки выявляются как с помощью традиционных оценок популяционной дифференциации (FST), генетических дистанций, так и с использованием байесовского метода анализа (табл. 3, рис. 2, 3). Возможно, это связано с морфоэкологическими особенностями микижи из этих рек. В отличие от других исследованных популяций здесь обитает преимущественно проходная форма, достигающая наиболее крупных размеров на камчатском побережье (Павлов и др., 2001). Сами реки короткие, относятся к классическим тундровым водотокам, имеют торфяной или “коричневый” состав воды. Не исключено, что формирование структуры популяций микижи определялось сходными экологическими условиями в этих реках и нашло отражение в полученных нами микросателлитных профилях.
Относительная близость восточно-камчатской микижи из бассейна р. Камчатка (выборка из р. Еловка) и крупных западно-камчатских бассейнов (выборки из рек Сопочная, Тигиль, Седанка) может иметь похожее объяснение. Бассейны рек, где обитают указанные популяции, крупные, отличаются близкой геоморфологией верховий. Они берут начало в предгорьях Срединного хребта Камчатки, имеют горное или тундрово-горное наполнение, по характеру воды “светлые”. Популяции в этих реках или их значительная часть (для западных рек) представлены резидентной формой микижи (форелью). Предполагая в эволюционном прошлом распространение большинства популяций камчатской микижи от одной предковой формы и их последующую изоляцию по крупным речным бассейнам, возможно, мы наблюдаем сходное формирование популяционной структуры вида в сходных условиях среды. Интересно, что похожая картина была недавно описана для сахалинской симы Oncorhynchus masou, когда выборки из разнесенных крупных речных бассейнов обнаруживали больше сходства, чем выборки симы из близлежащих малых рек (Животовский и др., 2017). Объяснением этому могут быть процессы случайного дрейфа генов, наиболее интенсивные в популяциях симы малых рек, что приводит к их значительной дивергенции. Следует отметить, что рассматриваемые нами выборки микижи (из рек Еловка, Сопочная, Тигиль) также относятся к популяциям из крупных речных бассейнов Камчатского п-ова.
Генетическое разнообразие камчатской микижи по микросателлитным локусам для всей совокупности выборок характеризуется следующими показателями: HE = 0.515, АR = 4.46. Эти оценки хорошо согласуются с результатами исследований предыдущих лет (Willams et al., 2002; McPhee et al., 2007; Павлов и др., 2011).
Полученные оценки генетического разнообразия несколько меньше показателей гетерозиготности по STR-локусам, чем у американских популяций микижи. Например, HE = 0.55–0.59 на Аляске (Olsen et al., 2006), 0.51–0.87 в Британской Колумбии (Heath et al., 2001; Hendry et al., 2002; Heggenes et al., 2006), 0.68–0.86 в шт. Вашингтон (Аrdren, Kapuscinski, 2003; Narum et al., 2006), 0.62–0.79 в Калифорнии (Aguilar, Garza, 2006), 0.42–0.62 в Канаде (Taylor et al., 2007). Хотя уменьшение средних оценок гетерозиготности может быть связано с использованием различного набора локусов, наиболее вероятно, что снижение генетического разнообразия может быть следствием эффекта основателя (явление снижения разнообразия при заселении малым числом представителей вида новой географической территории), а также относительно недавней колонизации микижей этого региона.
Таким образом, показано, что обособленность камчатских популяций микижи приурочена к крупным речным бассейнам Западной и Восточной Камчатки. Наиболее дифференцированы по микросателлитным локусам микижа из р. Жупанова (Восточная Камчатка), а также микижа из рек Квачина и Утхолок (Западная Камчатка). Популяции микижи, обитающие в ряде крупных камчатских речных бассейнов, обладают близкими микросателлитными профилями. Очевидно, что на примере камчатской микижи полиморфизм микросателлитных локусов еще раз доказывает свое микроэволюционное и филогеографическое приложение на популяционном уровне.
Исследование выполнено при финансовой поддержке Российского научного фонда (грант “Научные основы создания национального банка-депозитария живых систем” № 14-50-00029).
Список литературы
Животовский Л.А., Рубцова Г.А., Никитин В.Д., Прохоров А.В., Шайхаев Е.Г., Коткин К.С., Гво Д.Ч., Афанасьев К.И. Генетическая дифференциация и вопросы сохранения популяций симы Oncorhynchus masou Brevoort 1856 (Pisces: Salmonidae) // Биология моря. 2017. Т. 43. № 1. С. 70–78.
Кузищин К.В., Павлов С.Д., Груздева М.А., Павлов Д.С. Особенности нерестовой популяции и экология размножения пресноводной микижи Parasalmo mykiss в бассейне реки Жупановой (восточная Камчатка) // Вопр. ихтиологии. 2002. Т. 42. № 5. С. 626–638.
Павлов Д.С., Савваитова К.А., Кузищин К.В., Груздева М.А., Павлов С.Д., Медников Б.М., Максимов С.В. Тихоокеанские благородные лососи и форели Азии. М.: Научный мир, 2001. 200 с.
Павлов С.Д. Аллозимная изменчивость и генетическая дивергенция тихоокеанских форелей (род Parasalmo) западной Камчатки // Генетика. 2000. Т. 36. № 9. С. 1251–1261.
Павлов С.Д., Колесников А.А., Мельникова М.Н., Ушакова М.В. Генетическая дивергенция камчатской микижи (Parasalmo (Oncorhynchus) mykiss) на ареале по результатам рестрикционного анализа и секвенирования гена цитохрома b мтДНК // Генетика. 2004. Т. 40. № 12. С. 1695–1701.
Павлов С.Д., Семенова А.В., Рубцова Г.А., Афанасьев К.И. Анализ изменчивости микросателлитных локусов у камчатской микижи (Parasalmo (Oncorhynchus) mykiss) // Генетика. 2011. Т. 47. № 10. С. 1346–1356.
Семенова А.В., Рубцова Г.А., Афанасьев К.И., Павлов С.Д. Анализ микросателлитной ДНК у камчатской микижи (Parasalmo (Oncorhynchus) mykiss). Подбор локусов и оптимизация методики // Генетика. 2010. Т. 46. № 7. С. 1004–1008.
Abadía-Cardoso A., Pearse D.E., Jacobson S., Marshall J., Dalrymle D., Kawasaki F., Ruiz-Campos G., Garza G.C. Population genetic structure and ancestry of steelhead/rainbow trout (Oncorhynchus mykiss) at the extreme southern edge of their range in North America // Conserv. Genet. 2016. V. 17. № 3. P. 675–689.
Aguilar A., Garza J.C. A comparison of variability and population structure for major histocompatability complex and microsatellite loci in California coastal steelhead (Oncorhynchus mykiss Walbaum) // Mol. Ecol. 2006. V. 15. P. 923–937.
Ardren W.R., Kapuscinski A.R. Demographic and genetic estimates of effective population size (Ne) reveals genetic compensation in steelhead trout // Mol. Ecol. 2003. V. 12. P. 35–49.
Beacham T.D., Khai D.L., Candy J.R. Population structure and stock identification of steelhead trout (Oncorhynchus mykiss) in British Columbia and the Columbia River based on microsatellite variation // Env. Biol. Fish. 2004. V. 69. P. 95–109.
Beacham T.D., Pollard S., Le K.D. Population structure and stock identification of steelhead in southern British Columbia, Washington, and the Columbia River based on microsatellite DNA variation // Trans. Amer. Fish. Soc. 1999. V. 128. № 6. P. 1068–1084.
Beacham T.D., Pollard S., Le K.D. Microsatellite DNA population structure and stock identification of steelhead trout (Oncorhynchus mykiss) in the Nass and Skeena rivers in northern British Columbia // Mar. Biotech. 2000. V. 2. № 6. P. 587–600.
Chapuis M.P., Estoup A. Microsatellite null alleles and estimation of population differentiation // Mol. Biol. Evol. 2007. V. 24. P. 621–631.
Cornuet J.M., Luikart G. Description and power analysis of two tests for detecting recent population bottlenecks from allele frequency data // Genetics. 1996. V. 144. № 4. P. 2001–2014.
Heath D.D., Pollard S., Herbinger C. Genetic structure and relationships among steelhead trout (Oncorhynchus mykiss) populations in British Columbia // Heredity. 2001. V. 86. P. 618–627.
Heggenes J., Beere M., Tamkee P., Taylor E.B. Genetic diversity in steelhead before and after conservation hatchery operation in a coastal, boreal river // Trans. Amer. Fish. Soc. 2006. V. 35. P. 251–267.
Hendry M.A., Wenburg J.K., Myers K.W., Hendry A.P. Genetic and phenotypic variation through the migratory season provides evidence for multiple populations of wild steelhead in the Dean River, British Columbia // Trans. Amer. Fish. Soc. 2002. V. 131. P. 418–434.
Leitwein M., Garza J.C., Pearse D.E. Ancestry and adaptive evolution of anadromous, resident, and adfluvial rainbow trout (Oncorhynchus mykiss) in the San Francisco bay area: application of adaptive genomic variation to conservation in a highly impacted landscape // Evol. Appl. 2017. V. 10. № 1. P. 56–67.
Lulla P., Gross R., Paaver T. Genetic diversity and differentiation of imported into Estonia rainbow trout (Oncorhynchus mykiss) strains based on microsatellite DNA variation // J. Agricul. Sci. 2005. V. 16. P. 37–42.
McCuscer M., Parkinson E., Taylor E. Mitochondrial DNA variation in rainbow trout (Onchorhynchus mykiss) across its native range: testing biogeographical hypotheses and their relevance to conservation // Mol. Ecol. 2000. V. 9. P. 2089–2108.
McPhee M.V., Utter F., Stanford J.A, Kuzishin K.V., Savvaitova K.A., Pavlov D.S., Allendorf F.W. Population structure and partial anadromy in Oncorhynchus mykiss from Kamchatka: relevance for conservation around the Pacific Rim // Ecol. Fresh. Fish. 2007. V. 16. № 4. P. 539–547.
Narum S.R., Contor C., Talbot A., Powell M.S. Genetic divergence of sympatric resident and anadromous forms of Oncorhynchus mykiss in the Walla Walla River, USA // J. Fish Biol. 2004. V. 65. P. 471–488.
Narum S.R., Powell M.S., Evenson R., Sharp B., Talbot A.J. Microsatellites reveal population substructure of Klickitat River native steelhead and genetic divergence from an introduced stock // North Amer. J. Fish. Manag. 2006. V. 26. P. 147–155.
Nielsen J.L., Carpanzano C., Fountain M.C., Gan C.A. Mitochondrial DNA and nuclear microsatellite diversity in hatchery and wild Oncorhynchus mykiss from freshwater habitats in southern California // Trans. Amer. Fish. Soc. 1997. V. 126. № 3. P. 397–417.
Olsen J.B., Wuttig K., Fleming D., Kretschmer E.J., Wenburg J.K. Evidens of partial anadromy and resident-form dispersal bias on a fine scale in populations of Oncorhynchus mykiss // Conserv. Genet. 2006. V. 7. P. 613–619.
Pavlov S.D., Melnikova M.N., Senchukova A.L., Pivovarov E.A. Development of a new system of genetic markers for Kamchatkan mykiss (Parasalmo (O.) mykiss) as approach to study salmonic fish populations // 2nd Moscow Inter. Conf. “Molecular Phylogenetics”. M.: Torus Press, 2010. P. 63.
Piry S., Luikart G., Cornuet J.M. Computer note. BOTTLENECK: a computer program for detecting recent reductions in the effective size using allele frequency data // J. Heredity. 1999. V. 90. № 4. P. 502–503.
Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data // Genetics. 2000. V. 155. P. 945–959.
Silverstein J.T., Rexroad C.E., King T.L. Genetic variation measured by microsatellites among three strains of domesticated rainbow trout (Oncorhynchus mykiss, Walbaum) // Aquac. Res. 2004. V. 35. № 1. P. 40–48.
Taylor E., Tamkee P., Sterling G., Hughson W. Microsatellite DNA analysis of rainbow trout (Oncorhynchus mykiss) from western Alberta, Canada: native status and evolutionary distinctiveness of “Athabasca” rainbow trout // Conserv. Genet. 2007. V. 8. P. 1–15.
Van Oosterhout C., Hutchinson W.F., Wills D.P.M., Shipley P. MICRO-CHECKER: software for identifying and correcting genotyping errors in microsatellite data // Mol. Ecol. Notes. 2004. V. 4. P. 535–538.
Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure // Evolution. 1984. V. 38. P. 1358–1370.
Williams R.N., Powell M.S., Pavlov S.D., Proebstel D.S. Forthcoming. molecular genetic variation among rainbow trout Oncorhynchus mykiss Walbaum (Salmonidae, Salmoniformes) from the Kamchatka Peninsula // Summary of the Eighth Pacific Coast Steelhead Management Meeting. Port Townsend: Washington Press, 2002. P. 27–28.
Дополнительные материалы отсутствуют.
Инструменты
Известия РАН. Серия биологическая