Известия РАН. Серия биологическая, 2022, № 1, стр. 15-25
Принципы идентификации нуклеотидных последовательностей в составе спектров ISSR-маркеров
Н. В. Бардуков 1, *, В. С. Артамонова 1
1 ФГБУН Институт проблем экологии и эволюции им. А.Н. Северцова РАН
119071 Москва, Ленинский проспект, д. 33, Россия
* E-mail: bardukv-nikolajj@mail.ru
Поступила в редакцию 27.03.2020
После доработки 26.02.2021
Принята к публикации 26.02.2021
- EDN: XDNYNK
- DOI: 10.31857/S102634702201005X
Аннотация
Разработан алгоритм для теоретического прогнозирования нуклеотидных последовательностей, амплифицирующихся в результате ISSR-PCR, на примере секвенированного генома лошади. Экспериментально показано, что теоретически предсказанные последовательности присутствуют в реальном электрофоретическом спектре ампликонов. Обсуждаются перспективы применения разработанного подхода для расшифровки спектров анонимной амплификации.
Системы геномного сканирования, т.е. одновременного анализа множества локусов, очень востребованы. В основном их создают на основе SNP-чипов (Kulbrock et al., 2013; Porto-Neto et al., 2013), хотя более простыми и доступными являются системы, основанные на принципе анонимной амплификации, в частности, ISSR-анализ (Inter Simple Sequence Repeats) (Zietkiewicz et al., 1994). При использовании в ПЦР праймера, комплементарного какому-либо микросателлитному мотиву и имеющего “якорные” нуклеотиды на 3'‑конце, на тестируемой ДНК-матрице синтезируется набор ампликонов, который различается у представителей разных видов, пород (сортов), и даже у отдельных животных (растений). Это позволяет сравнивать между собой виды, популяции (породы, сорта), отдельных особей (Basha, Sujatha, 2007; Воронкова и др., 2011; Столповский, 2013; Al-Ameri et al., 2016; Kalia et al., 2017).
Однако многие исследователи скептически относятся к работе с такими системами, поскольку остается неизвестным, из каких участков генома происходят ампликоны, составляющие ISSR-спектр, и чем обусловлен выявляемый полиморфизм (Банникова, 2004).
С другой стороны, несмотря на наличие объективных недостатков, данный тип ДНК-маркеров востребован и в настоящее время. Наибольшее количество исследований, выполняемых с помощью ISSR-анализа, лежит в области изучения генетики растений. Они востребованы при селекции, а также для оценки разнообразия в генофондах диких видов (Дорогина и др., 2019; Степанов и др., 2019; Kim et al., 2019; Chen et al., 2020). Продолжаются, хотя и с меньшей интенсивностью, исследования генетики животных. В частности, в последнее время ISSR-маркеры применяли при изучении азиатских слонов (Kriangwanich et al., 2018), псовых (Глазко и др., 2020), искусственно выведенных пород и природных популяций рыб (Пелеева и др., 2018). Имеются работы, объектами которых становились лошади. Так, с помощью ISSR-анализа изучали особенности генетики чистокровных лошадей арабской породы (Sheikh et al., 2018), а в работе (Донт и др., 2018) с помощью ISSR-маркеров были найдены локусы, характерные для лошадей тракененской, тяжеловозных и рысистых пород. Авторы последней работы рекомендуют использовать разработанную ими систему для определения породной принадлежности и при создании генетических паспортов животных. Таким образом, можно утверждать, что ISSR-анализ до сих пор имеет свою нишу и активно используется исследователями в разных странах.
Однако лишь в настоящее время, когда стали известны полные последовательности геномов животных тех видов, которых изучают методами анонимной амплификации, стало возможно с помощью in silico PCR (моделирование результатов ПЦР по секвенированным последовательностям) рассчитать ISSR-спектры ампликонов и сравнить теоретически ожидаемые спектры с экспериментальными. Такой способ позволяет понять, какие именно нуклеотидные последовательности формируют ISSR-спектры. Для решения подобных задач были созданы специальные компьютерные программы (Kalendar et al., 2017), но, судя по работам, опубликованным в настоящее время, несмотря на принципиальную возможность их использования для моделирования спектров анонимных ДНК-маркеров, с этой целью они не применяются.
В настоящей работе представлен пример использования подхода in silico PCR для расшифровки состава спектров ISSR-маркеров.
МАТЕРИАЛЫ И МЕТОДЫ
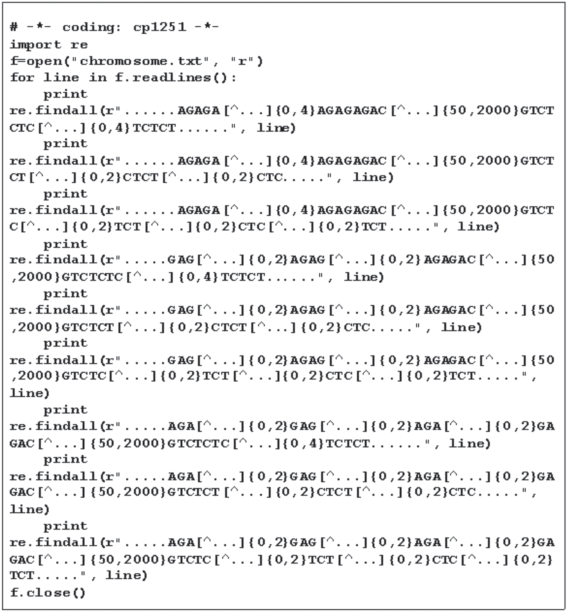
В данном исследовании в качестве примера был создан алгоритм для теоретического прогнозирования нуклеотидных последовательностей, получаемых в результате ISSR-PCR при использовании праймеров (GA)9C или (AG)9C, записанный на языке Python 2.6. (рис. 1). Расчеты выполняли в программе IDLE (Python GUI) (http://www.python.org/). Предварительный вариант алгоритма был опубликован в работе (Бардуков и др., 2014). Преимуществом данного алгоритма является большая гибкость и прозрачность настройки поискового запроса (например, имеется возможность учесть различные варианты неполной комплементарности ДНК-матрицы заданному праймеру) по сравнению с поиском с помощью программ in silico PCR. Это условие особенно важно в случае анонимной амплификации, при которой существует высокая вероятность отжига праймера на не полностью комплементарной ему матрице. Важной особенностью алгоритма является то, что первые пять нуклеотидов матрицы должны точно соответствовать 3'-концу праймера, поскольку было экспериментально показано, что именно эта область оказывает определяющее влияние на эффективность синтеза ампликонов (Rejiali, 2018).
Рис. 1.
Алгоритм поиска теоретически возможных ампликонов, которые могут присутствовать в ISSR-спектре при использовании в ПЦР праймера (GA)9C. Алгоритм для праймера (AG)9C аналогичен.
При поиске теоретически возможных ампликонов были использованы последовательности секвенированного генома домашней лошади (Equus caballus), представленные в GenBank (EquCab2.0.; NC_009144.2 – NC_009175.2).
Поскольку программного обеспечения, позволяющего адекватно оценивать температуру отжига праймера на не полностью комплементарной матрице, в настоящее время не существует, с целью сегрегации найденных последовательностей по степени вероятности их появления в составе спектра, для каждого возможного сайта отжига праймера оценивали температуру плавления дуплексов, образуемых ими с праймером, по формуле:
Теоретически рассчитанный спектр ампликонов сравнивали с экспериментальным, для чего производили генотипирование лошадей алтайской и карачаевской пород, образцы крови которых имелись в коллекции Центра нанобиотехнологий РГАУ-МСХА им. К.А. Тимирязева. Образцы крови лошадей алтайской породы (48 образцов из хозяйства СПК “Энчи”, СПК зарегистрирован в селе Чибиля Улаганского района Республики Алтай, а также 48 образцов из СПК “Чингиз”, СПК зарегистрирован в селе Кара-Кудюр Улаганского района Республики Алтай) были собраны в ходе экспедиции сотрудников Лаборатории сравнительной генетики животных Института общей генетики им. Н.И. Вавилова РАН в Республику Алтай, а 34 образца крови лошадей карачаевской породы были собраны сотрудниками Центра нанобиотехнологий РГАУ-МСХА им. К.А. Тимирязева в частных хозяйствах Карачаево-Черкесской Республики и Ставропольского Края.
ДНК выделяли из образцов периферической крови лошадей с помощью набора реагентов “ДНК-Экстран-1” (ООО “Синтол”, Москва) согласно инструкции фирмы-производителя.
Для выполнения ПЦР использовали амплификатор Терцик (“ДНК-технология”, Россия) и наборы реагентов ПЦР-РВ (ООО “Синтол”). Концентрацию MgCl2 в ПЦР-смеси доводили до 2.5 мМ. Условия ПЦР были следующими: первичная денатурация ДНК (t = 94°С, 2 мин); 35 циклов синтеза ПЦР-продукта, включающих три этапа (денатурация (t = 94°С, 30 с), отжиг праймера (t = = 55°С, 30 с), элонгация (t = 72°С, 2 мин)); финальная элонгация цепей ДНК (t = 72°С, 10 мин). В качестве ISSR-праймеров использовали олигонуклеотиды с последовательностями (GA)9C и (AG)9C.
Разделение ПЦР-продуктов проводили методом горизонтального электрофореза в 1.5%-ном агарозном геле. Для уточнения размеров ампликонов при сопоставлении длины теоретически рассчитанного амплифицируемого фрагмента с локусами экспериментального ISSR-спектра выполняли электрофорез в 5%-ном полиакриламидном геле (ПААГ). Во всех случаях для электрофореза использовали Tris-боратный буфер (1xTBE: 89 мМ Тris, 89 мМ борная кислота, 2 мМ ЭДТА, pH 8.3) (Маниатис и др., 1984).
Окрашивание ПЦР-продуктов производили раствором бромистого этидия (0.5 мкг/мл), визуализацию результатов – на трансиллюминаторе УВТ-1 (“Биоком”, Россия) и системе гель-документирования INGENIUS (“SYNGENE”, Великобритания) при длине волны 312 нм. Для определения длин ампликонов использовали маркеры молекулярных масс “M 27” (“СибЭнзим”, Россия) и “50 bp DNA Step Ladder” (“Promega”, США).
Поиск последовательностей, гомологичных заданным, в геномах животных разных видов, представленных в GenBank, выполняли с использованием встроенной программы BLAST (https:// blast.ncbi.nlm.nih.gov/Blast.cgi).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
С помощью разработанного алгоритма было выявлено 185 теоретически возможных ампликонов длиной не более 2000 п.о. Следует отметить однако, что, несмотря на их большое количество, ни один из этих ампликонов не имел сайтов отжига, полностью комплементарных праймеру (GA)9C.
Теоретическая оценка температуры плавления (Tm) дуплексов показала, что, даже при полной комплементарности праймера и матрицы, она не превышает 57°С, а для теоретически предсказанных сайтов, образуемых праймером и флангами расчетных ампликонов, не превышает 55°С. Между тем, принято считать, что температура отжига праймера TA теор. ≈ Tm – 5, т.е., примерно на пять градусов ниже температуры плавления дуплексов, образуемых праймером и матрицей (ПЦР “в реальном времени”, 2009).
Это означает, в свою очередь, что при температуре отжига праймера, равной 55°С, получение богатого ампликонами ISSR-спектра на матрице, не имеющей участков, полностью комплементарных матрице, теоретически невозможно (TA теор. ≈ ≈ 50°С для теоретически предсказанных сайтов отжига, наиболее комплементарных праймеру). Однако известно, что при использовании праймера (GA)9C и температуре отжига 55°С разные исследователи неоднократно получали на ДНК лошадей ISSR-спектры, насыщенные ампликонами (Glazko et al., 2016; Голик и др., 2018).
В связи с этим, мы поставили серию экспериментов и определили, что в реальных условиях амплификация большинства ампликонов для выбранной нами системы матрица-праймер полностью прекращается только при температуре отжига праймера ≥65°С, т.е. TА max эксп. ≈ TA теор. +15°С.
Далее, мы ранжировали теоретически предсказанные ампликоны по степени вероятности их появления в экспериментальном спектре, исходя из теоретической оценки температуры плавления дуплексов на обоих концах каждого из ампликонов. Для дальнейшего анализа были отобраны только те ампликоны, которые имели расчетную температуру плавления ДНК-дуплексов, образуемых матрицей и праймером, не ниже 40°С (табл. 1). В результате для последующего анализа было отобрано 25 последовательностей.
Таблица 1.
Соответствие между теоретически рассчитанными и экспериментально полученными спектрами ампликонов
| Название теоретически предсказанного ампликона | Последовательности ДНК на сайтах посадки праймера с расчетной температурой плавления дуплексов и длина теоретически предсказанного ампликона | Локализация теоретически предсказанного ампликона на хромосоме | Соответствие спектру |
|---|---|---|---|
| 171–1326 | GGAAGAGAGAGAGAGAGAC2 (52°C); GTCTCTCTCTCTCTTTCTC3 (54°C); 1326 п.о. | NC_009160.3 (48 971 749–48 973 074) | Мажорный кластер около 1250–1350 п.о. |
| 1–1319 | ATCCTAGAGAGAGAGAGAC (41°C); GTCTCTCTGGCTCTGTCTT (44°C); 1319 п.о. | NC_009144.3 (163 465 329–163 466 647) | |
| 2–1302 | GAAAGAGAGAGAGAGAGAC (54°C); GTCTCTCTCCCTCTGTCTG (48°C); 1302 п.о. | NC_009145.3 (28 343 941–28 345 242) | |
| 6–1301 | TAAAGAGAATGAGAGAGAC (44°C); GTCTCTCTCTCAGTCTTCT (41°C); 1301 п.о. | NC_009149.3 (51 069 282–51 070 582) | |
| 25–1281 | GAGGGAGAGAGACAGAGAC (52°C); GTCTCTCAGCATCTCTCTG (41°C); 1281 п.о. | NC_009168.3 (29 106 855–29 108 135) | |
| 3–1214 | GCCTGAGAGAGAAAGAGAC (46°C); GTCTCTCTGTCTCTGTCTG (46°C); 1214 п.о. | NC_009146.3 (30 151 710–30 152 923) | |
| 4–1148 | TCTCGAGAGACAGAGAGAC (41°C); GTCTCTCTCTCTCTCTTCC (52°C); 1148 п.о. | NC_009147.3 (17 092 678–17 093 825) | Полоса около 1100–1150 п.о. |
| 25–1281 | ATGAGAGAGAGAGAGAGAC (52°C); GTCTCTCAGCATCTCTCTG (41°C); 1140 п.о. | NC_009168.3 (29 106 996–29 108 135) | |
| 20–1115 | GAGGGGCAGAGAGAGAGAC (51°C); GTCTCTCTCTCTCGCTGGG (46°C); 1115 п.о. | NC_009163.3 (7 278 613–7 279 727) | |
| 10–1102 | AACTGAGAGACAGAGAGAC (44°C); GTCTCTGTCTCTTCCTCGG (40°C); 1102 п.о. | NC_009153.3 (20 333 365–20 334 465) | |
| 21–1060 | GAGAGAGGATGAGAGAGAC (51°C); GTCTCTCTCTTGCTCTGAC (46°C); 1060 п.о. | NC_009164.3 (57 129 665–57 130 724) | Нет типичных четких полос |
| 15–922 | GGAAGAGAGAGAGAGAGAC (52°C); GTCTCTCGCTGTGTCTCTCTCCCTCTCTATG4 (48°C); 910–922 п.о. | NC_009158.3 (88 931 543–88 932 464) | Нет типичных четких полос |
| 24–900 | TAGAAAGAGAGAGAGAGAC (50°C); GTCTCTCTCTCTAACATGC (44°C); 900 п.о. | NC_009167.3 (24 755 030–24 755 929) | |
| X–876 | TTTATAGAGAGAGAGAGAC (44°C); GTCTCTCTCTCTCACTCAC (55°C); 876 п.о. | NC_009175.3 (76 760 564–76 761 439) | Мажорный кластер около 820–880 п.о. |
| 18–871 | AAGACAGAGAGAGAGAGACAGAGAGAC4 (50–54°C); GTCTCTCTTTCTCTCTCTC (54°C); 863-871 п.о. | NC_009161.3 (27 521 579–27 522 449) | |
| 15–922 | GGAAGAGAGAGAGAGAGAC (52°C); GTCTCTCTCTGTCTCTCTCTATTTCTTTC4 (46–54°C); 855–865 п.о. | NC_009158.3 (88 931 543–88 932 407) | |
| 18–871 | GAGAGAGAGGGAGAGAGAC (55°C); GTCTCTCTTTCTCTCTCTC (54°C); 835 п.о. | NC_009161.3 (27 521 615–27 522 449) | |
| 10–833 | CTGAATGAGAGAGAGAGAC (46°C); GTCTCTCTGTCTCCATCTCTCTCTCTC4 (48–52°C); 825–833 п.о. | NC_009153.3 (60 737 537–60 738 369) | |
| 19–823 | TAGAGAGAGAGAGAGAGAC (54°C); GTCTCTCTGTGTCTCTATG (41°C); 823 п.о. | NC_009162.3 (43 532 121–43 532 942) | |
| 8–805 | GGGAGACAGAGACAGAGAC (48°C); GTCTCTCTTTCTCTCTGTC (50°C); 805 п.о. | NC_009151.3 (8 521 975–8 522 779) | Нет типичных четких полос |
| X–637 | CAAAACGAGAGAGAGAGAC (44°C); GTCTCTCCATCTCTCTCTC (52°C); 637 п.о. | NC_009175.3 (1 358 676–1 359 312) | Минорные полосы 610–690 п.о. |
| 10–572 | AAGAAAGAGGGAGAGAGAC (48°C); GTCTCTCTCTCTTTCTCTT (50°C); 572 п.о. | NC_009153.3 (29 331 545–29 332 116) | Полосы около 540–580 п.о. |
| 18–871 | TAGAGAGAGACAGAGAGAC (50°C); GTCTCTCTTTCTCTCTCTC (54°C); 563 п.о. | NC_009161.3 (27 521 887–27 522 449) | |
| X–637 | CAAAACGAGAGAGAGAGAC (44°C); GTCTCTCTATCTCTCTCCA (48°C); 552 п.о. | NC_009175.3 (1 358 676–1 359 227) | |
| 10–572 | AAGAAAGAGGGAGAGAGAC (48°C); GTCTCTCTCTGTTTCTATC (46°C); 551 п.о. | NC_009153.3 (29 331 545–29 332 094) | |
| 10–546 | GGGAGGAAGAGAGAGAGAC (51°C); GTCTCTCTGTGTCTCTTTC (46°C); 546 п.о. | NC_009153.3 (23 373 675–23 374 221) | |
| 11–513 | GAGGGAGAGAGAGAGAGAC (55°C); GTCTCTCTGGTTCTCTCCT (41°C); 513 п.о. | NC_009154.3 (121 598–122 110) | Нет типичных четких полос |
| 11–513 | AGGAGACAGAGAGAGAGAC (48°C); GTCTCTCTGGTTCTCTCCT (41°C); 491 п.о. | NC_009154.3 (121 620–122 110) | Мажорная полоса около 490 п.о. |
| 28–489 | GAGAGAAAGAGAGAGAGAC (54°C); GTCTCTCTCTCTCACACAC (53°C); 489 п.о. | NC_009171.3 (34 185 907–3 4186 395) | |
| X–637 | CAAAACGAGAGAGAGAGAC (44°C); GTCTCTCCATCTCTCTCCA (46°C); 450 п.о. | NC_009175.3 (1 358 676–1 359 125) | Нет типичных четких полос |
| X–637 | CAAAACGAGAGAGAGAGAC (44°C); GTCTCTCCATCTCTCTCCA (46°C); 418 п.о. | NC_009175.3 (1 358 676–1 359 093) | |
| 21–377 | TGAGGAGAGGGAGAGAGAC (44°C); GTCTCTCTATCTCTCTCTA (50°C); 377 п.о. | NC_009164.3 (41 224 113–41 224 489) | Мажорная полоса около 380 п.о. |
| X–637 | CAAAACGAGAGAGAGAGAC (44°C); GTCTCTCTCTCCATCTTTT (44°C); 366 п.о. | NC_009175.3 (1 358 676–1 359 041) | |
| 10–394 | ACAGCAAGAGAGAGAGACAGAGAGAAAGAGAG AGACTGAGTGAGTGAGAGACAGAGAGAGAC4 (44–52°C); GTCTCTCTCTCTCTCTCCA (52°C); 354–394 п.о. | NC_009153.3 (58 797 548–58 797 941) | |
| 7–349 | GAGAGATGGAGAGAGAGAC (52°C); GTCTCTTCCTCTCTCTTCC (46°C); 349 п.о. | NC_009150.3 (85 378 714–85 379 062) | Нет типичных четких полос |
Примечание. 1Номер хромосомы, в которой локализован предполагаемый ампликон; 2последовательность прямого сайта отжига; 3 последовательность обратного сайта отжига; 4 расчетные сайты посадки праймера (GA)9C с возможностью отжига праймера на разных участках сайта, подчеркиванием выделены возможные положения нуклеотида, связывающегося с 3'‑концевым нуклеотидом праймера.
Теоретически рассчитанный спектр сравнивали с экспериментальным, полученным в ПЦР с использованием праймера (GA)9C (Бардуков и др., 2014) при температуре отжига праймера равной 55°С.
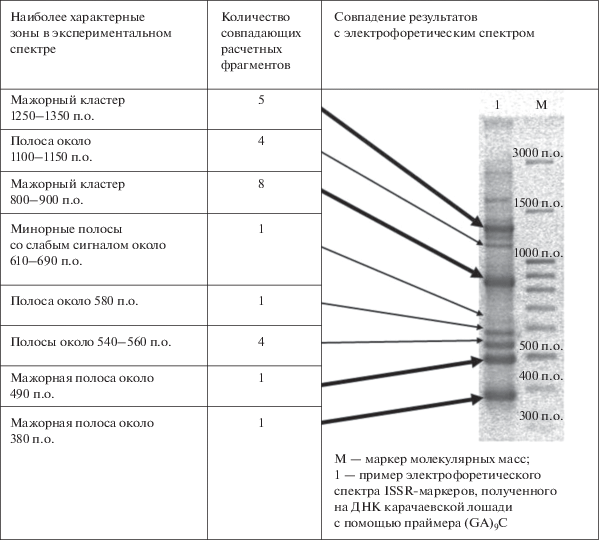
Сопоставление показало хорошее соответствие между теоретически ожидаемым и экспериментальным спектрами: для всех мажорных и большинства минорных локусов экспериментального ISSR-спектра были найдены расчетные ампликоны, соответствующие им по длине.
Наибольшее число расчетных ампликонов было найдено для широких мажорных полос, состоящих, по всей видимости, из ампликонов сходной длины, но происходящих из разных частей генома – т.н. гетерогенных кластеров (рис. 2). Теоретически предсказанные ампликоны, совпадающие по длине с мажорными полосами и полосами в составе гетерогенных кластеров, имели более высокие расчетные температуры плавления дуплексов (от 52 до 54°С), по сравнению с другими предполагаемыми ампликонами, найденными в геноме (рис. 2). Это хорошо согласовывалось с особенностями реального электрофоретического спектра, однако являлось лишь косвенным подтверждением того, что были найдены ампликоны, действительно входящие в состав ISSR-спектра.
Рис. 2.
Соответствие между теоретически предсказанными (Tm ≥ 40°С) и экспериментально полученными ампликонами. Жирные стрелки указывают на мажорные полосы, для которых теоретически предсказаны ампликоны с высокой температурой плавления (от 52 до 54°С) на расчетных сайтах посадки праймера (GA)9C.
Дальнейшую проверку возможностей выявления в ISSR-спектрах теоретически предсказанных ампликонов выполняли двумя способами.
На первом этапе был рассмотрен клонированный и секвенированный ранее полиморфный ампликон длиной ~420 п.о. из ISSR-спектра, полученного на геномной ДНК лошадей при использовании в ПЦР праймера (AG)9C (Глазко и др., 2013). На основе секвенированной последовательности (длина ампликона составляла, как оказалось, 416 п.о.) были спланированы праймеры F‑416 (5'-GAGAGAGAGAGAGAGAGCCCATGG-3') и R-416 (5'-GAGAGAGAGAGAGAGCGAAAGAGC-3') для избирательной амплификации данного локуса. Массовое тестирование лошадей показало, что амплификация этого локуса проходит успешно только у жеребцов, что говорит о его локализации в Y-хромосоме, которая в геноме лошади, представленном в GenBank, отсутствует (в GenBank представлен геном кобылы). В связи с этим, как и ожидалось, найти данный фрагмент в геноме домашней лошади с помощью алгоритма, разработанного в настоящей работе, не удалось.
При поиске в GenBank последовательностей, гомологичных клонированному ампликону выяснилось, что участок Y-хромосомы, найденный в ISSR-спектре лошади, секвенирован у лошади Пржевальского (Equus przewalskii, NW_007676839.1) и осла (Equus asinus, NW_014638651.1). Эти последовательности обозначены как “unplaced genomic scaffold”, т.е. их локализацию в геноме авторам установить не удалось. Однако наше исследование доказывает, что это участки Y-хромосомы.
Применение разработанного алгоритма к участкам хромосом E. przewalskii и E. asinus показало, что внутри последовательностей NW_007676839.1 и NW_014638651.1 действительно удается выявить ампликоны, гомологичные последовательности полиморфного ампликона домашней лошади. Таким образом, с помощью разработанного алгоритма поиска в последовательностях Y-хромосом близких видов удалось найти ампликон, который ранее был выделен из ISSR-спектра, полученного с использованием в качестве матрицы ДНК лошади, и затем клонирован.
Сравнение трех последовательностей показало, что участок Y-хромосомы лошади, амплифицирующийся при использовании ISSR-праймера (AG)9C, имеет не один, а два возможных сайта посадки праймера на 3'-конце (рис. 3), но отжиг ISSR-праймера (AG)9C, судя по клонированному ПЦР-продукту, происходит на внешнем сайте. В то же время для лошади Пржевальского и осла отжиг на этом сайте невозможен из-за точечной замены, и затравка синтеза ПЦР-продукта может иметь место только с внутреннего сайта (рис. 3). Таким образом, в ISSR-спектрах осла и лошади Пржевальского будет отсутствовать ампликон, точно совпадающий по длине с соответствующим ампликоном из ISSR-спектра домашней лошади. Это показывает, что единичные точечные мутации могут менять температуру отжига праймера в том или ином его положении, давая преимущества в связывании праймера одному из двух-трех возможных сайтов, с чем, видимо, связана, по крайней мере, часть полиморфизма, который демонстрируют ISSR-маркеры.
Рис. 3.
Гомологичные последовательности домашней лошади ((а), выявлена при клонировании полиморфного ISSR-ампликона), лошади Пржевальского (б) и осла (в), представляющие собой участок Y-хромосомы. Светло-серым цветом выделены сайты посадки праймера (AG)9C, темно-серым – нуклеотиды, некомплементарные участкам последовательности этого праймера. Температура плавления ДНК-дуплексов между праймером и сайтами посадки в случае (а) не может быть рассчитана из-за отсутствия этой последовательности в секвенированном геноме домашней лошади (последовательности флангов ампликона остались неизвестны); в случаях (б) и (в) предполагаемые температуры плавления для прямых сайтов отжига праймера сложно рассчитать из-за наличия в последовательностях однонуклеотидных вставок. Рамкой в последовательности домашней лошади выделен участок, гомологичный сайту отжига праймера (AG)9C на ДНК лошади Пржевальского и осла.

На втором этапе проверки возможности выявления в ISSR-спектрах теоретически предсказанных ампликонов была поставлена обратная задача. Предстояло экспериментально подтвердить возможность амплификации теоретически предсказанного ампликона в ISSR-спектре, полученном с применением праймера (GA)9C при стандартных условиях ПЦР. С этой целью в экспериментальном ISSR-спектре, полученном при использовании в качестве матрицы ДНК лошади карачаевской породы, был выбран ампликон, который имел длину порядка 570 п.о., не входил в состав крупных мажорных кластеров и отличался минорным характером проявления (рис. 4). С помощью алгоритма, разработанного в данном исследовании, в 10-й хромосоме лошади был найден схожий по длине ампликон (572 п.о.), фланкированный последовательностями, частично комплементарными праймеру (GA)9C.
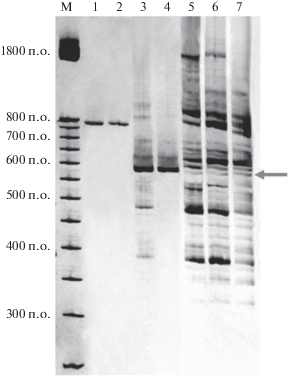
Рис. 4.
Электрофорез в 5%-ом ПААГ. М – маркер молекулярных масс; 1, 2 – ПЦР-продукт, полученный с праймеров F-10-740 и R-10-740 на геномной ДНК лошади; 3, 4 – ПЦР-продукт, полученный с использованием праймера (GA)9C, когда матрицей служил ПЦР-продукт, представленный на дорожках 1 и 2 (синтез дополнительных минорных ампликонов происходил на геномной ДНК, остатки которой присутствовали в пробе); 5–7 – электрофоретический спектр, полученный при использовании в ПЦР праймера (GA)9C и геномной ДНК лошадей карачаевской породы в качестве матрицы. Стрелка указывает на теоретически предсказанный ампликон длиной 572 п.о.
Для экспериментальной проверки соответствия расчетного ампликона ампликону, присутствующему в экспериментальном спектре, были подобраны праймеры F-10-740 (5'-GGCTCTC- TTTAAGGATCTAGAAC-3') и R-10-740 (5'-GTAATTCTTTCACTCATTCTGCTAC-3'), комплементарные участкам, находящимся от последовательности расчетного ампликона на расстоянии 67 нуклеотидов слева и 70 справа.
Электрофорез продуктов амплификации, выполненной с этих праймеров, продемонстрировал наличие фрагмента ожидаемой длины и отсутствие других ПЦР-продуктов (рис. 4). Этот ПЦР-продукт использовали в качестве ДНК-матрицы (после разведения в 2000 раз) в последующей ПЦР с праймером (GA)9C. ПЦР с праймером (GA)9C проводили при тех же условиях, что и при использовании в качестве матрицы геномной ДНК лошадей (TА эксп. = 55°С). В этой реакции был получен ПЦР-продукт длиной 572 п.о., что говорило об успешном отжиге праймера на матрице и доказывало, что расчетный ампликон эффективно синтезируется и формирует полосу в ISSR-спектре (рис. 4).
Чтобы исключить возможность случайного совпадения длины фрагментов, ПЦР-продукт, полученный с помощью праймеров F-10-740 и R‑10-740 был секвенирован у двух лошадей карачаевской породы. Анализ последовательностей показал, что внутренняя часть ампликона, находящаяся между теоретически рассчитанными сайтами отжига праймера (GA)9C идентична у этих двух образцов и полностью соответствует последовательности из GenBank.
Интересно, однако, что внутри секвенированных последовательностей, но вне зоны, ограниченной сайтами отжига праймера (GA)9C, был найден полиморфный микросателлитный локус с коровым мотивом (AGAT)n. Он располагался между теоретически рассчитанным сайтом посадки праймера (GA)9C и сайтом посадки праймера R-10-740, его аллельные варианты представлены на рис. 5.
Рис. 5.
Аллельные варианты микросателлитного локуса с коровым мотивом (AGAT)n, находящегося между теоретически рассчитанным сайтом отжига праймера (GA)9C и сайтом отжига праймера R-10-740. 1 – участок нуклеотидной последовательности из 10-й хромосомы лошади, представленной в GenBank; 2, 3 – аналогичные участки последовательности у лошади карачаевской породы, являющейся гетерозиготной по микросателлитному локусу с коровым мотивом (AGAT)n; 4 – соответствующий участок последовательности лошади карачаевской породы, гомозиготной по данному микросателлитному локусу.

Пример ампликона, происходящего из десятой хромосомы, с прилегающим к нему полиморфным микросателлитным локусом (AGAT)n представляет большой интерес, поскольку внутри 25-ти теоретически рассчитанных ампликонов было найдено множество микросателлитных мотивов (в табл. 1 наряду с фланговыми последовательностями ампликонов даны ссылки на локализацию этих ампликонов в секвенированных хромосомах лошади, что позволяет убедиться в наличии микросателлитных мотивов внутри данных ампликонов). Таким образом, представляется весьма вероятным, что в ряде случаев полиморфизм ISSR-спектров наблюдается из-за различий в длине микросателлитов, расположенных внутри ампликонов.
ЗАКЛЮЧЕНИЕ
Полученные результаты показывают, что моделирование ISSR-спектров с помощью анализа последовательностей секвенированных хромосом позволяет расшифровать значительную часть анонимного спектра, а также определить типичные для конкретного спектра причины полиморфизма его ампликонов. В настоящем исследовании установлено, что основными причинами полиморфизма ISSR-спектров, полученных с использованием праймеров (GA)9C и (AG)9C являются, по всей видимости, точечные нуклеотидные замены в т. н. якорных нуклеотидах и полиморфизм микросателлитов, расположенных внутри ампликонов.
Результаты работы показывают, что комплексный подход, включающий методы биоинформатики и экспериментальные приемы работы с последовательностями, способными воспроизводиться при анонимной амплификации, перспективен для расшифровки последовательностей, составляющих наблюдаемый спектр. В будущем он позволит выявлять общие закономерности формирования спектров ампликонов, что значительно упростит интерпретацию получаемых результатов. Не менее важно и то, что данный подход позволяет значительно снизить трудозатраты при создании на основе ISSR-ампликонов узкоспециализированных локус-специфичных маркеров. Упрощение разработки таких ДНК-маркеров позволит исключить целый каскад биотехнологических процедур, а значит, появится возможность быстро создавать их под необходимые задачи – будь то определение принадлежности животного или растения к конкретной популяции, породе, сорту, выявление гибридов, поиск надежных маркеров для выявления филогенетических связей.
Благодарности. Авторы благодарят сотрудников Центра нанобиотехнологий ФГБОУ ВО РГАУ-МСХА им. К.А. Тимирязева за предоставленные пробы ДНК лошадей.
Финансирование. Работа выполнена в рамках госзадания (тема 6: Экология и биоразнообразие водных сообществ 0109-2018-0076 АААА-А18-118042490059-5).
Список литературы
Банникова А.А. Молекулярные маркеры и современная филогенетика млекопитающих // Журн. общей биологии. 2004. Т. 65. № 4. С. 278–305.
Бардуков Н.В., Феофилов А.В., Глазко Т.Т., Глазко В.И. ISSR-PCR маркеры и мобильные генетические элементы в геноме домашней лошади Equus caballus // Сельскохозяйственная биология. 2014. № 4. С. 42–57.
Воронкова В.Н., Цэндсурэн Цэдэв, Сулимова Г.Е. Сравнительный анализ информативности ISSR-маркеров для оценки генетического разнообразия пород лошадей // Генетика. 2011. Т. 47. № 8. С. 1131–1134.
Глазко В.И., Гладырь Е.А., Феофилов А.В., Бардуков Н.В., Глазко Т.Т. ISSR-PCR маркеры и мобильные генетические элементы в геномах сельскохозяйственных видов млекопитающих // Сельскохозяйственная биология. 2013. № 2. С. 71–76.
Глазко В.И., Косовский Г.Ю., Блохина Т.В., Есин А.Г., Глазко Т.Т. Геномный “портрет” некоторых видов псовых, полученный с помощью ISSR-PCR и IRAP-PCR маркеров // Кролиководство и звероводство. 2020. № 1. С. 28–39.
Голик Т.В., Эркенов Т.А., Глазко Т.Т., Глазко В.И. Спектры ISSR-PCR маркеров в оценках популяционно-генетической дифференциации карачаевской лошади в хозяйствах Карачаево-Черкесской Республики // Известия ТСХА. 2018. № 3. С. 61–77.
Донт Ю.У., Тимарова А.В., Комарова Л.В., Боронникова С.В. Генетический полиморфизм трех пород лошади домашней // Вестник Пермского университета. Сер.: Биология. 2018. Вып. 1. С. 50–56.
Дорогина О.В., Васильева О.Ю., Нуждина Н.С., Буглова Л.В., Жмудь Е.В., Зуева Г.А., Комина О.В., Кубан И.С., Гусар А.С., Дудкин Р.В. Формирование и изучение коллекционного генофонда ресурсных видов рода Miscanthus Anderss. в условиях лесостепи Западной Сибири // Вавиловский журн. генетики и селекции. 2019. Т. 23. № 7. С. 926–932.
Маниатис Т., Фрич Э., Сэмбрук Дж. Методы генетической инженерии. Молекулярное клонирование. М.: Мир, 1984. 480 с.
Пелеева А.Р., Комарова Л.В., Васильева Ю.С. Анализ генетического разнообразия естественных популяций и ремонтно-маточных стад стерляди на основании полиморфизма межмикросателлитных маркеров // Бюллетень науки и практики. 2018. Т. 4. № 4. С. 20–29.
Ребриков Д.В., Саматов Г.А., Трофимоа Д.Ю., Семенов П.А., Савилова А.М., Кофиади И.А., Абрамов Д.Д. ПЦР “в реальном времени” / Под ред. Ребрикова Д.В. М.: БИНОМ. Лаборатория знаний, 2009. 215 с.
Степанов И.В., Супрун И.И., Токмаков С.В., Лободина Е.В. Поиск эффективных IRAP и ISSR маркеров для генетического анализа подвоев яблони // Плодоводство и виноградарство Юга России. 2019. № 60. С. 11–20.
Столповский Ю.А. Популяционно-генетические основы сохранения генофондов доместицированных видов животных // Вавиловский журн. генетики и селекции. 2013. Т. 17. № 4/2. С. 900–915.
Al-Ameri A.A., Al-Qurainy F., Gaafar A.-R.Z., Khan S., Nadeem M. Molecular identification of sex in Phoenix dactylifera using inter simple sequence repeat markers // BioMed Res. Intern. V. 2016. № 4530846.
Basha S.D., Sujatha M. Inter and intra-population variability of Jatropha curcas (L.) characterized by RAPD and ISSR markers and development of population-specific SCAR markers // Euphytica. 2007. V. 156. P. 375–386.
Chen X., Feng H., Du Y., Luo S., Li W., Yu L., Feng Z., Cui T., Zhou L. Genetic polymorphisms in mutagenesis progeny of Arabidopsis thaliana irradiated by carbon-ion beams and γ-rays irradiations // Int J Radiat Biol. 2020. V. 96(2). P. 267–275.
Glazko V.I., Erkenov T.A., Glazko T.T., Dzatoev K.M. Genetic structure of Karachai horses on ISSR-PCR markers // Biogeosystem Technique. 2016. V. 9. № 3. P. 195–204.
Kalendar R., Khassenov B., Ramankulov Y., Samuilova O., Ivanov K.I. FastPCR: An in silico tool for fast primer and probe design and advanced sequence analysis // Genomics. 2017. V. 109(3–4). P. 312–319.
Kalia P., Saha P., Ray S. Development of RAPD and ISSR derived SCAR markers linked to Xca1Bo gene conferring resistance to black rot disease in cauliflower (Brassica oleracea var. botrytis L.) // Euphytica. 2017. V. 213. № 232.
Kim J.Y., Adhikari P.B., Ahn C.H., Kim D.H., Kim Y.C., Han J.Y., Kondeti S., Yong Choi Y.E. High frequency somatic embryogenesis and plant regeneration of interspecific ginseng hybrid between Panax ginseng and Panax quinquefolius // J. Ginseng Res. 2019 V. 43(1). P 38–48.
Kriangwanich W., Nganvongpanit K., Buddhachat K., Brown J.L., Siengdee P., Chomdej S., Bansiddhi P., Thitaram C. Genetic diversity and variation in captive Asian elephants (Elephas maximus) in Thailand // Trop. Conser. Science. 2018. V. 11.
Kulbrock M., Lehner S., Metzger J., Ohnesorge B., Distl O. A genome-wide association study identifies risk loci to equine recurrent uveitis in German warmblood horses // PLoS One. 2013. V. 8(8). e71619.
Porto-Neto L.R., Sonstegard T.S., Liu G.E., Bickhart D.M., Da Silva M.V., Machado M.A., Utsunomiya Y.T., Garcia J.F., Gondro C., Van Tassell C.P. Genomic divergence of zebu and taurine cattle identified through high-density SNP genotyping // BMC Genomics. 2013. V. 14. P. 876.
Rejali N.A., Moric E., Wittwer C.T. The effect of single mismatches on primer extension // Clin Chem. 2018. V. 64. № 5. P. 801–809.
Sheikh A., Ahmed M.M.M., Mutwakil M.H.Z., Saini K.S., Alsulaimany F.A.S., EL Hanafy A.A., Sabir J.S.M. Comparative molecular analysis of ISSR markers in Arabian horse breeds // J. Anim. Plant Sciences. 2018. V. 28(1). P. 332–336.
Zietkiewicz E., Rafalski A., Labuda D. Genome fingerprinting by seguence repeat (SSR) anchored polymerase chain reaction amplification // Genomics. 1994. V. 20. P.176–183.
Дополнительные материалы отсутствуют.
Инструменты
Известия РАН. Серия биологическая


