Химическая физика, 2021, T. 40, № 2, стр. 44-48
Модель комплекса РНК-полимеразы вируса SARS-CoV-2 с фавипиравиром
И. В. Поляков 1, 2, Б. Л. Григоренко 1, 2, А. В. Немухин 1, 2, *
1 Институт биохимической физики им. Н.М. Эмануэля Российской академии наук
Москва, Россия
2 Московский государственный университет им. М.В. Ломоносова
Москва, Россия
* E-mail: anemukhin@yahoo.com
Поступила в редакцию 02.07.2020
После доработки 27.07.2020
Принята к публикации 21.09.2020
Аннотация
Методами молекулярного моделирования построена модель многодоменного комплекса, объясняющая механизм ингибирующего действия фавипиравира на РНК-зависимую РНК-полимеразу (RdRp) коронавируса SARS-CoV-2. В качестве начальных атомных координат использованы данные криоэлектронной микроскопии для апо-формы RdRp вируса SARS-CoV-2 и данные по структуре RdRp вируса гепатита C. После соответствующих замен сконструирован комплекс RdRp, содержащий цепочки РНК, и потенциальный ингибитор фермента – фавипиравир в форме рибозатрифосфата. Методами молекулярной динамики оптимизирована структура комплекса в водных оболочках, включающая более ста тысяч атомов. Анализ активного центра с включенной молекулой фавипиравира позволяет объяснить химическую реакцию фермента с ингибитором.
ВВЕДЕНИЕ
Пандемия коронавируса COVID-19 стимулировала активное изучение молекулярных процессов, протекающих во всех компонентах вируса SARS-CoV-2, включая важнейший его участок – РНК-зависимую РНК-полимеразу (RdRp), ответственную за репликацию и транскрипцию генетической информации [1]. Потенциальные ингибиторы RdRp рассматриваются как перспективные лекарственные препараты для лечения болезни, и одним из наиболее интересных кандидатов является фавипиравир [2]. Следует отметить, что на момент написания статьи соответствующий препарат одобрен Минздравом РФ для лечения COVID-19.
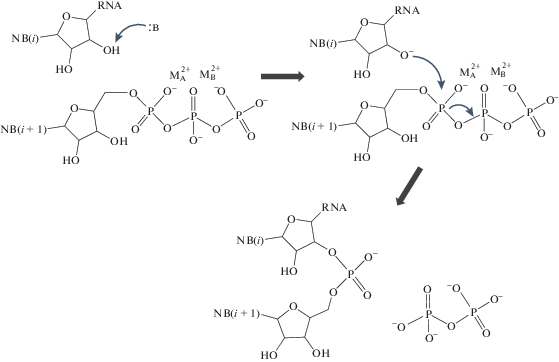
На рис. 1 схематично показан механизм химических превращений в активном центре РНК-полимеразы. При депротонировании атома O3' рибозы концевого нуклеотида в цепи NB(i) (левая часть рис. 1) происходит нуклеофильная атака O3' на атом PA свободного нуклеотида NB(i + 1) (центральная часть рис. 1), что приводит к разрыву связи PA–O3A, отщеплению пирофосфата и наращиванию цепи РНК (правая часть рис. 1). В качестве обобщенного основания (В) на стадии депротонирования может выступать аминокислотный остаток аспарагиновой кислоты. В активном центре присутствуют ионы металла ${\text{M}}_{{\text{A}}}^{{{\text{2}} + }},$ ${\text{M}}_{{\text{B}}}^{{{\text{2}} + }},$ где M – магний или марганец, координирующие отрицательно заряженные группы реактантов.
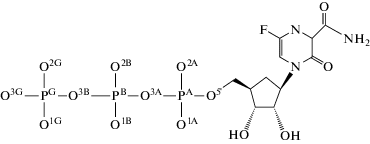
Нарушение функционирования этой молекулярной машины приводит к блокированию размножения вируса. На рис. 2 показана молекула фавипировира (в соединении с рибозатрифосфатом – FRTP), которая может конкурировать с нативными субстратами фермента в активном центре и остановить нежелательный синтез РНК-вируса.
Данная работа направлена на исследование физико-химических аспектов биомолекулярных процессов, связанных с ингибированием важнейшего фермента RdRp вируса SARS-CoV-2. Мы используем средства молекулярного моделирования для построения и анализа активного центра РНК-полимеразы с включенной молекулой фавипиравира. Полученные в 2020 г. данные криоэлектронной микроскопии для апо-формы RdRp [3], а также известные результаты по кристаллической структуре RdRp вируса гепатита C [4] предоставляют начальную структурную информацию, на основе которой возможно довести моделирование до анализа молекулярных групп в конфигурации фермента с кофактором (фавипиравиром в форме рибозатрифосфата), имитирующей фермент-субстратный комплекс. Сведения о механизме химической реакции в РНК-полимеразе [5], а также опыт моделирования катализируемых ферментами реакций преобразования нуклеозидтрифосфатов методами квантовой механики/молекулярной механики [6, 7] позволяют объяснить ингибирующее действие фавипиравира.
МОДЕЛИ И МЕТОДЫ
Как и все коронавирусы, SARS-CoV-2 использует многодоменную архитектуру молекулярной машины для репликации и транскрипции своей РНК. После расщепления вирусных полипротеинов образуется набор неструктурированных белков (nsp), три из которых входят в минимальную конфигурацию, обеспечивающую функционирование комплекса, а именно, nsp12 (RdRp), nsp7 и nsp8. В работе [3] сообщается о структуре комплекса nsp12–nsp7–nsp8 без цепей РНК и кофакторов с разрешением в 2.9 Å (PDB ID: 6M71). В качестве дополнительного источника структурных данных мы использовали результаты рентгеноструктурного анализа [4] для фермента RdRp вируса гепатита C. Соответствующая структура PDB ID: 4WTG наряду с комплексом nsp12–nsp7–nsp8 содержит цепочки РНК, кофакторы (ионы Mn2+), а также соединение софосбувир (в форме рибозадифосфата). Использование процедуры выравнивания позволило провести сравнение начальной структуры PDB ID: 6M71 и активного центра фермента в структуре PDB ID: 4WTG. Далее ионы Mn2+ были заменены на Mg2+, а софосбувир – на FRTP (см. рис. 2); боковые цепи ряда остатков активного сайта были повернуты в соответствии с положением их аналогов в структуре PDB ID: 4WTG.
Полученная модельная система была полностью сольватирована молекулами воды; были добавлены ионы натрия и хлора для нейтрализации заряда и обеспечения физиологической концентрации NaCl: 0.15 моль/л. Молекулярно-динамические расчеты проводились с помощью программы NAMD [8], анализ траекторий – в программе VMD [9]. Использовались условия ансамбля NTP с периодическими граничными условиями; шаг интегрирования составлял 2 фс, при этом все связи тяжелых атомов с атомами водорода были фиксированы (алгоритмы SHAKE/SETTLE). Потенциалы взаимодействия соответствовали силовому полю CHARMM36 [10]. Нестандартная топология и параметры остатка FRTP были описаны параметрами CGenFF [11]. Длина траекторий составляла не менее 40 нс, кадры траектории записывались через 200 пс.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
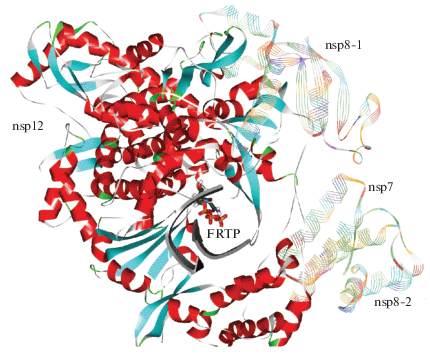
На рис. 3 показан комплекс без сольватирующих оболочек молекул воды. Белки nsp7 и nsp8 важны для поддержания конструкции, но не участвуют в химических преобразованиях.
В центре рис. 3 видны показанные стержнями фосфатные группы молекулы FRTP. Стрелками обозначены цепочки РНК, содержащие гуаниновый (Gua), адениновый (Ade) и урациловый (Ura) нуклеотиды.
Рисунок 4 показывает, насколько стабильна модельная система вдоль молекулярно-динамической траектории. После начального участка (примерно 10 нс) вся конструкция довольно устойчива (рис. 4а). Анализ показывает, что наиболее подвижные участки в nsp12 относятся к петлям вдали от активного центра фермента.
Рис. 4.
Среднеквадратичные отклонения координат (RMSD) тяжелых атомов белка (а) и молекулы FRTP (б).
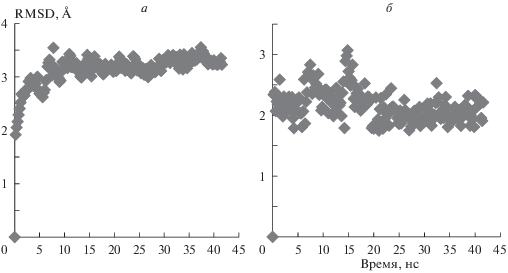
Для уравновешивания положения FRTP требуется больше времени – примерно 20 нс. Это объясняется тем, что молекула FRTP не является нативным субстратом фермента, а также особенностями начального этапа конструирования модельной системы, а именно: группы FRTP замещали другое соединение в кристаллической структуре PDB ID: 4WTG.
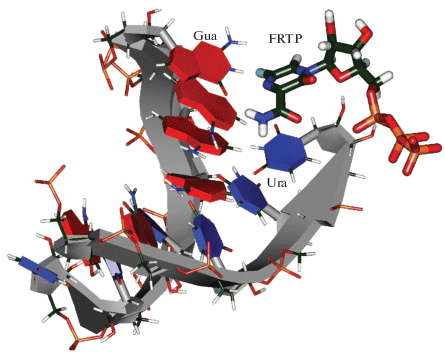
На рис. 5 показано, что молекула фавипиравира достаточно удачно входит в комплекс. “Голова” FRTP структурно похожа на аденин и образует пару с ближайшим основанием Gua. На рис. 6 представлена структура активного центра фермента RdRp.
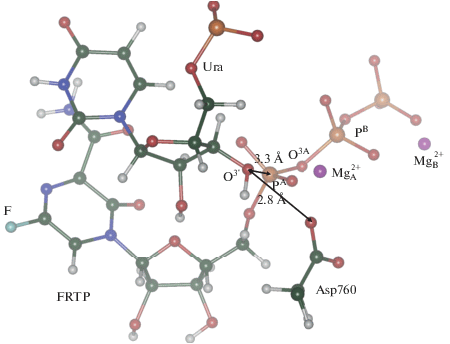
Присоединение фавипиравира к концевому нуклеотиду Ura происходит по химической реакции, показанной на рис. 1. На первом этапе депротонируется группа O3'H3' рибозы урацила, причем очевидным обобщенным основанием является боковая цепь Asp760 – расстояние между атомом кислорода O3' и атомом кислорода карбоксильной группы аспарагиновой кислоты составляет 2.8 Å, обеспечивая эффективный перенос протона. Расстояние от O3' до атома фосфора PA, равное 3.3 Å, также благоприятствует нуклеофильной атаке с образованием связи O3'–PA и разрывом связи PA–O3A. На рис. 6 не показаны боковые цепи аминокислотных остатков Asp761 и Asp618, входящих в координационные сферы ионов магния, а также положительно заряженные остатки Arg553, Arg555 и Lys551, которые связывают и ориентируют трифосфатный участок молекулы FRTP.
Настоящая работа подтверждает современные достижения компьютерного моделирования биомолекулярных процессов [12, 13]. В данном случае построенный активный центр фермента полностью согласуется с представлениями о каталитических процессах с участием нуклеозидтрифосфатов. Похожий активный центр формируется в случае фермента аденилатциклазы, катализирующего реакцию преобразования аденозинтрифосфата в циклический аденозинмонофосфат. Элементарные стадии этого процесса также включают депротонирование O3'H3'-группы рибозы, нуклеофильную атаку O3' на атом фосфора PA α-фосфатной группы нуклеозидтрифосфата и разрыв связи PA–O3A. Недавние расчеты комбинированным методом квантовой механики/молекулярной механики [6] показали, что энергетический профиль этой реакции имеет барьер для лимитирующей стадии порядка 16 ккал/моль. Мы ожидаем, что в случае реакции РНК-полимеразы SARS-CoV-2 с FRTP энергетический профиль будет аналогичным, что согласуется с эффективным встраиванием фавипиравира в цепочку РНК. Блокада синтеза РНК после захвата этого соединения, по всей видимости, связана с нарушением структуры формирующейся цепочки.
ЗАКЛЮЧЕНИЕ
Построенная методами молекулярного моделирования модель многодоменного комплекса РНК-полимеразы коронавируса SARS-CoV-2 объясняет механизм воздействия фавипиравира на процесс репликации. Анализ полученной структуры и активного центра фермента показывает, что фавипиравир в форме рибозатрифосфата может встраиваться в комплекс nsp12–nsp7–nsp8 с цепочками РНК и конкурировать с природными нуклеотидами в реакции наращивания цепи.
Работа выполнена при финансовой поддержке Российским фондом фундаментальных исследований (проект № 19-03-00076) с использованием оборудования Межведомственного суперкомпьютерного центра РАН и Центра коллективного пользования сверхвысокопроизводительными вычислительными ресурсами МГУ им. М.В. Ломоносова.
Список литературы
Venkataraman S., Prasad B.V.L.S., Selvarajan R. // Viruses. 2018. V. 10. P. 76;https://doi.org/10.3390/v10020076
Furuta Y., Gowen B. B., Takanashi K. et al. // Antiviral Res. 2013. V. 100. P. 446; https://doi.org/10.1016/j.antiviral.2013.09.015
Gao Y., Yan L., Huang Y. et al. // Science. 2020. V. 368. P. 779; https://doi.org/10.1126/science.abb7498
Appleby T.C., Perry J.K., Murakami E. et al. // Ibid. 2013. V. 347. P. 771; https://doi.org/10.1126/science.1259210
Wang B., Feig M., Cukier R.I. et al. // Chem. Rev. 2013. V. 113. P. 8546; https://doi.org/10.1021/cr400046x
Grigorenko B., Polyakov I., Nemukhin A. // J. Phys. Chem. B. 2019. V. 124. P. 451;https://doi.org/1021/acs.jpcb.9b07349
Grigorenko B.L., Kots E.D., Nemukhin A.V. // Org. Biol. Chem. 2019. V. 17. P.4879; https://doi.org/10.1039/c9ob00463g
Phillips J.C., Braun R., Wang W. et al. // J. Comp. Chem. 2005. V. 26. P. 1781; https://doi.org/10.1002/jcc.20289
Humphrey W., Dalke A., Schulten K. // J. Molec. Graphics. 1996. V. 14. P. 33; https://doi.org/1016/0263-7855(96)00018-5
Best R.B., Zhu X., Shim J. et al. // J. Chem. Theory Comput. 2012. V. 8. P. 3257; https://doi.org/10.1021/ct300400x
Vanommeslaeghe K., Hatcher E., Acharya C. et al. // J. Comput. Chem. 2010. V. 31. P. 671; https://doi.org/10.1002/jcc.21367
Григоренко Б.Л., Хренова М.Г., Кулакова А.М. и др. // Хим. физика. 2020. Т. 39. № 6. С. 13; https://doi.org/10.31857/S0207401X20060023
Метелешко Ю.И., Немухин А.В., Хренова М.Г. // Хим. физика. 2019. Т. 38. № 6. С. 3; https://doi.org/10.1134/S0207401X19060074
Дополнительные материалы отсутствуют.
Инструменты
Химическая физика