Кристаллография, 2019, T. 64, № 1, стр. 81-86
О перемещениях ДНК под действием ферментов разных типов. II
С. А. Пикин 1, *, Е. С. Пикина 2
1 Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника” РАН
Москва, Россия
2 Институт проблем нефти и газа РАН
Москва, Россия
* E-mail: pikin@crys.ras.ru
Поступила в редакцию 10.04.2018
После доработки 10.04.2018
Принята к публикации 25.05.2018
Аннотация
В первой части работы было показано, как структура и функционирование ферментов могут быть описаны в рамках формализма обобщенных сил и токов. Во второй части рассмотрены управляющие силы в энзимах II типа, транслокация ДНК в хеликазах.
УПРАВЛЯЮЩИЕ СИЛЫ В ЭНЗИМАХ II ТИПА
С помощью обобщенных сил и токов в [1–23] описаны структура и функционирование ферментов, влияющих на линейные движения и вращения молекул ДНК. Для всех типов ферментов применимы следующие кинетические уравнения:
(2)
${{{\mathbf{P}}}_{\gamma }} = \mu {\mathbf{\xi }},\quad {{\xi }_{x}} = {{n}_{z}}{{n}_{y}},\quad {{\xi }_{y}} = --{{n}_{z}}{{n}_{x}},$В первой части работы основное внимание было уделено ферментам рестрикции-модификации типа I и III [24], в которых каталитические реакции происходят главным образом с участием “топливных” молекул ATФ. Многочисленные эксперименты с ферментами II типа [25–30] указывают на большой вклад электростатических взаимодействий между белком энзима и фосфатными группами ДНК. Во всех процессах весьма важна роль ионов Mg2+, так как эти ионы, возможно, необходимы для специфического связывания ДНК и существенны для катализа, управляемого электростатическими взаимодействиями. Характеристики этих ферментов отражают следующие особенности: изгиб [28] и, возможно, закрутку (твист) основной цепи ДНК, т.е. ориентационную деформацию хиральной цепи ДНК; каталитические процессы [25–27], индуцирующие конформационные изменения в хиральном комплексе энзим–ДНК; трение между движущимся ферментом и ДНК, остающейся в покое [29, 30].
Эндонуклеазы рестрикции II типа действуют без использования источников энергии типа гидролиза молекул АТФ. После остановки энзима наблюдается явление дробления цепи ДНК. Можно ожидать, что поперечное электрическое поле E⊥ (перпендикулярное оси цепи ДНК) и полярный вектор р$_{0}$ в энзимах типа II индуцируются, например, благодаря остаткам ароматических и гидрофобных аминокислот, которые имеются в активных местах энзимов и у оснований ДНК, а также благодаря упоминавшемуся присутствию ионов Mg2+ и Mn2+.
Здесь разница ∆ есть мера изменения свободной энергии комплекса энзим–ДНК в ходе каталитического процесса, управляемого электростатическими взаимодействиями. Этот процесс аналогичен ферментным реакциям, управляемым AТФ в ходе последовательных циклов в субъединицах β ионных насосов F0F1-ATФ. Таким образом, можно полагать, что величины ∆ и E⊥ взаимосвязаны.
Полость энзима, в которой помещается ДНК, подвергается различным конформационным изменениям благодаря каталитическим реакциям, индуцирующим указанные деформации ДНК. Это явление позволяет вектору ξ, модуль которого пропорционален θ, возникать кооперативно, и такие отклонения закручиваются благодаря хиральным свойствам комплекса белок–ДНК. Взаимодействие поляризации электрического диполя Pγ с поперечным полем E⊥ приводит к движению так называемого кинка [31] вдоль оси z, если нет каких-либо препятствий. Кинк (перегиб) имеет определенную ширину h в пространственном распределении азимутального угла отклонения поляризации μξ – угла поворота φ(z, t) вектора μξ в плоскости xy. Эта закрутка, показанная на рис. 1, описывается уравнением [31]:
(3)
$\varphi (z,t) = \arctan \left[ {{\text{s}}{{{\text{h}}}^{{ - 1}}}\left( { \pm \frac{z}{h} + \frac{{{\mu }E{\theta }}}{{\gamma }}t} \right)} \right].$(5)
$\begin{gathered} \gamma {{\Omega }_{z}} \approx \nu {\kern 1pt} {\text{'}}{{F}_{z}}{\text{/}}V + a\nu {\kern 1pt} {\text{''}}\theta {{n}_{z}}\Delta {\text{/}}V, \\ {{\text{v}}_{z}} \approx {\eta }{{F}_{z}} + a{{n}_{z}}{\theta }\Delta , \\ \end{gathered} $(9)
$\Xi = - \frac{{{{\text{v}}_{z}}F}}{{u\Delta }} \approx \frac{{F{\text{ }}\left( {\left| c \right|\theta \Delta - \eta {\text{ }}F} \right)}}{{\Delta \left( {\lambda \Delta + \left| c \right|F\theta } \right)}}.$Рис. 1.
Движение кинка в сильном поперечном электрическом поле. Показаны ширина кинка h и его скорость ${{\text{v}}_{z}}$; геликоидальные повороты псевдовектора ξ, который в плоскости xy ведет себя как истинный вектор.
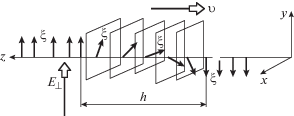
Экспериментально наблюдаемая линейная скорость составляет величину ${{\text{v}}_{z}}$ ~ 107 пар оснований в секунду [32]. Так как типичный шаг в геликоиде ДНК порядка 10 пар оснований или 3.5 нм, здесь предполагается, что ширина кинка h того же порядка, т.е. 3.5 нм, то после подстановки значений |μ| ~ 10–3 Кл м$^{{ - 2}}$ и γ ~ 10–3 Па с, которые типичны для сегнетоэлектрических жидких кристаллов, а также значений θ ~ 0.1 рад и |E⊥| ~ 107 В/м, находим, что скорость энзима-мотора с наибольшим КПД (формула (9)) равна
(10)
${{\text{v}}_{z}}\sim h\mu {{E}_{ \bot }}\theta {\text{/}}\gamma \sim {{10}^{6}}\;{\text{н м /c}}.$Эта оценка линейной скорости близка к экспериментально наблюдаемой по порядку величины [24].
При согласовании скоростей ${{\Omega }_{z}}$ и ${{\text{v}}_{z}}$, например, при $\Omega \approx 2{\pi }{{h}^{{ - 1}}}{{\text{v}}_{z}}$, как видно из (5), получаем
(11)
${{\text{v}}_{z}} \approx \frac{{{\nu }{\kern 1pt} {\text{''}} - \frac{{{\nu }{\kern 1pt} {\text{'}}}}{{\eta }}}}{{\frac{{2{\pi \gamma }V}}{h} - \frac{{{\nu '}}}{{\eta }}}}\left( {a{\theta }\Delta {{n}_{z}}} \right).$Согласно сделанным оценкам критическая сила ${{F}_{{cr}}}\sim - a{\theta }\Delta {/\eta }\sim {{10}^{{ - 4}}}$ пН может быть очень малой. Если энзим встречает некоторое препятствие, создающее силу F, большую, чем ${{F}_{{cr}}}$, то комплекс энзим–ДНК останавливается.
При F = ${{F}_{{cr}}}$ линейная скорость ${{\text{v}}_{z}}$ обращается в нуль, а угловая скорость, согласно (1), оказывается конечной:
(12)
${{\Omega }_{z}} \approx \frac{{a{\theta }\Delta {{n}_{z}}}}{{{ \gamma }V}}\left( {\nu {\kern 1pt} {\text{'/}}\eta - \nu {\kern 1pt} {\text{''}}} \right)\quad {\text{п р и }}\quad \theta \ne 0.$(13)
${\mathbf{M}} = [{\mathbf{gn}}],\quad {{M}_{{x,y}}} \approx a\Delta \theta {{V}^{{--1}}}(\nu {\kern 1pt} {\text{''}}--\nu {\kern 1pt} {\text{'/}}\eta ){{n}_{x}}_{{,y}}.$Для дробления ДНК необходима гораздо большая сила по сравнению с ${{F}_{{cr}}}$. Такое большое значение силы может быть локально связано с продольным электрическим полем (вплоть до значений порядка 109 В/м), а соответствующее взаимодействие может существовать между заряженными остатками и основаниями ДНК, которые находятся в окружении с очень низким значением диэлектрической постоянной. Такое окружение возникает, когда молекулы растворителя и ионы выталкиваются с поверхности раздела белок–ДНК во время процесса распознавания, сопровождаемого значительной кооперативной перестройкой поверхности раздела и взаимно индуцируемой подгонкой энзима и ДНК [20]. При ${{F}_{z}} \gg {{F}_{{cr}}}$ момент сил (13) равен
(14)
${\mathbf{M}} = [{\mathbf{gn}}],\quad {{M}_{{x,y}}} \approx \nu {\kern 1pt} {\text{'}}{{V}^{{--1}}}{{F}_{z}}{{n}_{z}}{{n}_{x}}_{{,y}}.$Для разрезания ДНК в полости более инертных энзимов I и III типов требуется значительно бóльшая сила Fz (в 10–100 раз больше) по сравнению с таковой в энзиме типа II. Это объясняет, почему эффективное разрезание ДНК энзимами типа I требует такого мощного фактора, как столкновение двух и большего числа комплексов транслокации, связанных с одной и той же молекулой ДНК: как правило, энзимы I типа расщепляют линейную ДНК примерно на половине расстояния между двумя последовательными сайтами-мишенями (иногда на расстоянии в несколько тысяч оснований между ними) [17].
ТРАНСЛОКАЦИЯ ДНК В ХЕЛИКАЗАХ
Белки хеликазы образуют гексамерные кольца с центральными каналами, имеющими определенную полярность, и способны окружать и разматывать дуплекс ДНК. Они имеют сходные черты с известными F0F1-ATФ-синтазами [1, 33–39], где энзиматические реакции протекают в субъединицах, подвергающихся циклическим конформационным изменениям. По каналам этих гексамеров может проходить лишь одна нить ДНК, поскольку канал диаметром ∼13 Å заметно меньше диаметра дуплекса ДНК (∼20 Å) [3]. Проталкивание нити ДНК через канал гексамера подобно транслокации ДНК энзимами рестрикции-модификации с помощью движущей силы kΔ, гдe k вычисляется согласно уравнению [24]:
Размерность коэффициента а в феноменологическом выражении ${{\text{v}}_{z}} \approx a{\theta }\Delta $ для эффективно работающего мотора есть [1/(сила⋅время)]. Примем, что эта микроскопическая сила, действующая на нуклеотид, есть f ∼ U/l. Для грубой оценки а можно представить как обратное произведение величины, имеющей размерность силы f, на характерное время протекания соответствующей каталитической реакции $\tau \sim 1{\text{/}}{{u}_{0}}$, где ${{u}_{0}}$ – скорость реакции, т.е. $a\sim {{u}_{0}}{\text{/}}f$, что соответствует вероятностному характеру процессов [24]. Величина ${{u}_{0}}$ соразмерна с экспериментально наблюдаемыми скоростями каталитических реакций u, фигурирующими в кинетических уравнениях [21, 22]. Эксперименты дают значения ${{u}_{0}}\sim u\sim {{10}^{{ - 3}}}{\kern 1pt} - {\kern 1pt} 1$ с–1, т.е. τ ~ 1–103 с для разных энзимов, в которых такие реакции идут с разной скоростью. По-видимому, коэффициенты $f\sim {{10}^{{ - 17}}}{\kern 1pt} - {\kern 1pt} {{10}^{{ - 16}}}$ Н и величины θ и Δ у рассматриваемых ферментов не различаются на несколько порядков, поэтому значение а в основном зависит от τ. Значения а были получены в [21] с помощью экспериментальных данных по измерению скорости транслокации ДНК для разных энзимов, причем для RM I (${{\text{v}}_{z}}$ ∼ 102–10–3 нм с–1 [2]) этот коэффициент в 1000 раз меньше, чем для RM II (${{\text{v}}_{z}}\sim {{10}^{6}}$ нм·с–1 [20]), поскольку соответственно были меньше скорость реакций u и скорость транслокации ДНК. В хеликазах скорость транслокации (порядка скорости репликации ДНК) составляет величину ∼102 нм с–1 при $u\sim {{10}^{{ - 3}}}$ с–1 [40]. Согласно (7) критическая останавливающая сила у этих ферментов ${{F}_{{cr}}}\sim {{10}^{{ - 18}}}$ Н при $\eta \sim {{10}^{{11}}}$ м$^{2}$ Дж–1 с–1, $\Delta \sim 10{{k}_{{\text{B}}}}T\sim {{10}^{{ - 19}}}$ Дж и $\theta \sim {{10}^{{ - 1}}}$. У ферментов RM I и RM II сила ${{F}_{{cr}}}\sim {{10}^{{ - 17}}}{\kern 1pt} - {\kern 1pt} {{10}^{{ - 16}}}$ Н по оценке [21].Закручивание одной нити ДНК в гексамерной хеликазе обусловлено специфической винтовой лестницей, которую образуют в канале хеликазы так называемые петли (рис. 2), имеющиеся у каждой из шести субъединиц гексамера, обладающего осью симметрии ${{C}_{6}}$. Кристалл хеликазы E1 содержит два гексамера из шести субъединиц каждый (рис. 2a) [3]. Эти петли содержат аминокислотные остатки (например, лизин в хеликазе бактериофага Т7 [3]), связывающие нуклеотиды ДНК с внутренней поверхностью канала. Нуклеотиды контактируют с субъединицами гексамера, и нить ДНК вынуждена образовать аналогичную спираль – правосторонний геликоид в вирусном инициаторе – белке Е1 (рис. 2б). Когда цепь ДНК изгибается и смещается, ее нуклеотиды сближаются с внутренней поверхностью гексамера и получают энергию порядка энергии связывания в области петель, т.е. несколько ккал на моль или около 10–20 Дж. Рисунок 2 сделан на основе данных [3, 40]. Таким образом, механизм закрутки одной нити ДНК в хеликазах напоминает кручение ДНК в энзимах типа I RM [41, 42]11.
Рис. 2.
Условное изображение гексамера хеликазы c внутренним каналом: а – вид сверху вдоль винтовой оси симметрии шестого порядка С6: в канале субъединицы гексамера имеют петли, связывающие нуклеотиды ДНК; б – вид сбоку: петли образуют спираль, с которой связана спираль нити ДНК; тонкие пунктирные линии обозначают границы субъединиц в плоскости xy гексамера; показаны фрагменты аминокислотных остатков, связанные с петлями. Рисунок сделан по мотивам [3, 40].
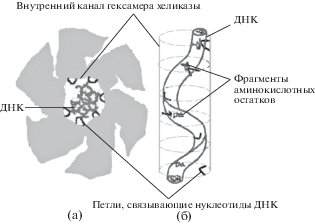
Хеликазы сходны с F0F1-ATP-синтазами, так как у тех и других существуют три характерных сайта, которые отличаются разной степенью вовлеченности молекул АТФ в координацию и гидролиз [3, 40]. В таких сайтах наблюдаются три конформации: слабо занятая (типа аденозиндифосфата – AДФ-типа), плотно занятая (АТФ-типа) и пустая. Сайты с молекулами АТФ, АДФ и без таковых содержат указанные формы петель ДНК-связывания. Они имеют кристаллографическую ось второго порядка [40]. У разных гексамеров может быть разное количество таких состояний, например у комплекса Е1–ДНК в первом гексамере конформации АТФ-типа имеются на трех поверхностях раздела, а во втором – только на двух. Такие петли у субъединиц АТФ-типа находятся на вершине гексамера, петли у субъединиц AДФ-типа занимают промежуточные позиции по высоте гексамера, петли у пустых субъединиц занимают дно гексамера [40].
В гексамере у Е1–ДНК, по модели [40], петли ДНК-связывания каждой субъединицы коллективно формируют спиральную лестницу. Каждая петля ДНК-связывания перемещается вместе с единственным нуклеотидом вниз от вершины благодаря гидролизу молекул АТФ и высвобождению молекул AДФ, причем гидролиз осуществляется ближе к вершине гексамера, а высвобождение AДФ – ближе к его дну. Нуклеотиды ДНК, осуществляющие линейную транслокацию в этом гексамере, присоединяются к данной субъединице по одному и совершают движение по винтовой лестнице, состоящей из петель ДНК-связывания (рис. 2). Вблизи дна такая петля освобождает присоединенный фосфат, и его движение по каналу гексамера заканчивается. При циклическом движении каждая субъединица гидролизует одну АТФ-молекулу, освобождает одну AДФ-молекулу и перемещает по каналу один нуклеотид. Таким образом, полный цикл включает в себя транслокацию шести нуклеотидов, обусловленную гидролизом шести молекул АТФ и высвобождением шести молекул AДФ.
Некоторые основания ДНК обладают дипольными моментами порядка 10 Д. Это содействует изгибам и скручиванию оснований, особенно при контакте с донорами или акцепторами электрических зарядов, располагающимися выше или ниже плоскости оснований [43]. Вообще, квадруполь-квадрупольные или ван-дер-ваальсовы силы могут вносить существенный вклад в рассматриваемые процессы благодаря заметным величинам квадрупольных моментов ДНК и оболочек энзимов [24]. Заметим, что это имеет место в случае жидких кристаллов, молекулы которых имеют размеры, сравнимые с размерами фрагментов нуклеотидов.
Для предотвращения захвата ДНК петлей энзима необходимы микроскопические силы f, действующие на нуклеотид, которые больше, чем величина U/l ~ æ/l ∼ 10–11 Н (cp. с [24], U – полная энергия Ван-дер-Ваальса, æ ∼ 10–20 Дж – энергия межмолекулярного взаимодействия), где размер нуклеотида l ∼ 1 нм. Во время элементарного акта движения ДНК между субъединицами гексамера и поворота нуклеотида от одной петли к другой возникает момент сил порядка М ∼ fl ∼ 10–20 Н·м, а работа этой силы есть A ∼ fl ∼ 10–20 Дж. Такие микроскопические акты совершаются импульсами за время более короткое, чем время, необходимое для конформационных изменений сайтов, где происходят сдвиги нуклеотидов и молекул ATФ и AДФ [42].
ЗАКЛЮЧЕНИЕ
Приведенные оценки являются качественными. Для согласованной работы рассмотренных моторов необходим тонкий баланс многих параметров энзима. Отметим, что механизмы транслокации ДНК энзимами RM I и хеликазами одинаковы. Они имеют сравнимые характеристики: ДНК в полостях этих энзимов обладают высокой вязкостью, низкой подвижностью и малыми хиральными кинетическими коэффициентами по сравнению с энзимами типа II RM. Проведены вычисления для движения комплекса энзим II типа–ДНК вдоль цепи ДНК в рамках двух механистических моделей: модели хирального кинка, двигающегося под действием поперечного электрического поля, и модельной системы кинетических уравнений. Энергия, вырабатываемая в ходе каталитических реакций, используется энзимами для совершения изгибания и закрутки цепей ДНК, углового и линейного движения энзимов RM II по цепи ДНК. Направления движения комплекса белок RM II–ДНК случайны, и это явление напоминает линейную диффузию. Когда молекулы воды и ионы выталкиваются с поверхности раздела белок–ДНК, в моторе появляются достаточно большие продольные силы, стремящиеся повернуть деформированный участок цепи ДНК вокруг ее поперечной оси, что приводит к ее разрезанию.
Сходство поведения энзимов RM I и хеликаз имеет, по-видимому, простой физический смысл: относительно массивные фрагменты ДНК (нуклеотиды) занимают энергетически выгодные позиции по отношению к aминокислотным остаткам и другим структурным элементам энзимов после гидролиза ATФ. Благодаря упомянутым взаимодействиям, активированным энзиматическими реакциями, эти фрагменты смещаются и переориентируются вдоль оси спиральной лестницы, образованной петлями энзимов. Все эти события связаны и происходят без дробления ДНК.
Рассмотрение макроскопических моделей этих моторов весьма полезно, хотя их микроскопическая природа очевидна, поскольку такие модели позволяют отразить некоторые основные черты подобных “машин”.
Очевидно, движения в полостях энзимов являются стохастическими. Конформационные изменения характерных сайтов являются своего рода триггерами для быстрых прерывистых движений и вращений нуклеотидов ДНК, обусловленных полярными силами.
Авторы выражают благодарность В. Хаазе и E. Демихову за совместную плодотворную работу, интересные дискуссии и полезную информацию по данной теме.
Работа поддержана Министерством науки и высшего образования в рамках Государственного задания ФНИЦ “Кристаллография и фотоника” РАН в части “теоретическое моделирование структуры и свойств ферментов в биологических материалах”, Министерством Науки и Высшего образования в рамках Государственного задания № 0139-2015-0022 (№ гос. задания АААА-А16-116030110081-2) в части “теория обобщенных сил и токов в движущейся ДНК – (E.С. Пикина).
Список литературы
Boyer P.D. // Biophim. Biophys. Acta. 1993. V. 1140. № 3. P. 215.
Seidel R., van Noort J., van der Scheer C. et al. // Nat., Struct. Mol. Biol. 2004. V. 11. № 9. P. 838.
Enemark E.J., JoshuaTor L. // Nature. 2006. V. 442. P. 270.
Abrahams P., Leslie A.G., Lutter R. et al. // Nature. 1994. V. 370. P. 621.
Boyer P.D. // Annu. Rev. Biochem. 1997. V. 66. P. 717.
Junge W., Sabbert D., Engelbrecht S. // Ber. Bunsen-Ges. Phys. Chem. 1966. V. 100. P. 2014.
Schulenberg F., Wellmer H., Lill A. et al. // Eur. J. Biochem. 1997. V. 249. № 1. P. 134.
Dimroth P., Wang H., Grabe M. et al. // Proc. Natl. Acad. Sci. USA. 1999. V. 96. № 9. P. 4924.
Junge W. // Proc. Natl. Acad. Sci. USA. PNAS 1999. V. 96. № 9. P. 4735.
Noji H., Yasuda R., Yoshida M. et al. // Nature. 1997. V. 386. P. 299.
де Жен P. Физика жидких кристаллов / Пер. с англ. M.: Мир, 1977. 400 с.
Pikin S.A. Structural Transformations in Liquid Crystals. N.Y.: Gordon and Breach, 1991. 423 p.
Weber V., Senior A.E. // Biochim. Biophys. Acta. 1997. V. 1319. № 1. P. 19.
Julicher F., Ajdari A., Prost J. // Rev. Mod. Phys. 1997. V. 69. P. 1269.
Pikin S., Haase W. // Zh. Éksp. Teor. Fiz. 2001. V.119. № 1. P. 199.
Gabler R. Electric Interactions in Molecular Biophysics. N.Y.: Academic Press, 1978. 363 p.
Dryden D.T.F., Murray N.E., Rao D.N. // Nucl. Acids. Res. 2001. V. 29. № 18. P. 3728.
White J.H. // Am. J. Math.1969. V. 91. № 3. P. 693.
Pikin S.A., Beresnev L.A., Haase W. et al. // Proc. EUROPTO Series, Imaging and Technologies. 1997. V. 2949. P. 11.
Pingoud A. Jeltsch A. // Eur. J. Biochem. 1997. V. 246(1). P. 1.
Pikin S.A. // Crystallography Reports. 2008. V. 53. P. 858.
Pikin S.A. // Crystallography Reports. 2007. V. 52. P. 1094.
Loginov E.B., Pikin S.A. // Crystallography Reports. 2006. V. 51. P. 308.
Пикин С.А., Пикина Е.С. // Кристаллография. 2018. Т. 63. № 5. С. 734.
Sidorova N.Y. // Proc. Natl. Acad. Sci. USA. 1996. V. 93. P. 12272.
Kostrewa D., Winkler F.K. // Biochemistry. 1995. V. 34. № 2. P. 683.
Newman M., Strzelecka T., Dorner L.F. et al. // Science. 1995. V. 269. P. 656.
Thompson J.F., Landy A. // Nucl. Acids Res. 1988. V. 16. № 20. P. 9687.
Record M.T., Anderson C.F., Lohman T.M. // Q. Rev. Biophys. 1979. V. 11. № 2. P. 103.
Jeltsch A., Wolfes H., Maass G. et al. // Proc. Natl. Acad. Sci. USA. 1993. V. 90. P. 8499.
Demikhov E., Pikin S.A., Pikina E.S. // Phys. Rev. E. 1995. V. 52. № 6. P. 6250.
Ehbrecht H.-J., Pingoud A., Urbanke C. et al. // Biol. Chem. 1985. V. 260. P. 6160.
Sawaya M.R., Guo S., Tabor S. et al. // Cell. 1999. V. 99. P. 167.
Lohman T.M., Bjornson K.P. // Annu. Rev. Biochem. 1996. V. 65. P. 169.
Patel S.S., Picha K.M. // Annu. Rev. Biochem. 2000. V. 69. P. 651.
Matson S.W., Kaiser Rogers K.A. // Annu. Rev. Biochem. 1990. V. 59. P. 289.
Hingorani M.M., Washington M.T., Moore K.C. et al. // Proc. Natl. Acad. Sci. USA. 1997. V. 94. P. 5012.
Washington M.T., Rosenberg A.H., Griffin K. et al. // J. Biol. Chem. 1996. V. 271. P. 26825.
Singleton M.R., Sawaya M.R., Ellenberger T. et al. // Cell. 2000. V. 101. № 6. P. 589.
Crampton D.J., Mukherjee S., Richardson C.C. // Mol. Cell. 2006. V. 21. № 2. P.165.
Kim J.S., DeGiovanni A., Jancarik J. et al. // Proc. Natl. Acad. Sci. USA. 2005. V. 102. P. 3248.
Pikin S.A. // Crystallography Reports. 2009. V. 54. P. 929.
Sponer J., Hobza P. // Chem. Rev. 1999. V. 99. № 11. P. 3247.
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография


