Кристаллография, 2019, T. 64, № 1, стр. 95-98
Моделирование комплекса фосфорибозилпирофосфатсинтетазы из Thermus thermophilus С АТФ и рибозо-5-фосфатом
Д. Д. Подшивалов 1, 2, 3, Д. Д. Сидоров-Бирюков 1, 2, В. И. Тимофеев 1, 2, 3, *, А. А. Литунов 1, М. А. Костромина 1, К. В. Синицына 1, Т. И. Муравьева 1, И. П. Куранова 1, 2, 3, Р. С. Есипов 1
1 Институт биоорганической химии РАН
Москва, Россия
2 Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника” РАН
Москва, Россия
3 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
* E-mail: tostars@mail.ru
Поступила в редакцию 23.11.2017
После доработки 23.11.2017
Принята к публикации 21.02.2018
Аннотация
Методом молекулярной динамики получено положение субстратов фосфорибозилпирофосфатсинтетазы из Thermus thermophilus (АТФ и рибозо-5-фосфата). Показано, что система пришла в равновесие и положения гигантов в активном центре стабильны. На основе полученного комплекса проанализировано окружение субстратов, показаны аминокислотные остатки фермента, имеющие полярные взаимодействия с субстратами. На основании данного анализа получены возможные точки мутагенеза, мутации в которых способны расширить субстратную специфичность фермента по рибосом-5-фосфату.
ВВЕДЕНИЕ
Ферменты семейства фосфорибозилпирофосфатсинтетаз (PRPP-синтетазы, КФ 2.7.6.1) катализируют образование 5-фосфорибозилпирофосфата (5-PRPP) из аденозинтрифосфата и рибозо-5-фосфата (Р5Ф) [1]. Продукт катализируемой реакции является промежуточным соединением в ряде важных метаболических процессов, приводящих к синтезу пуриновых и пиримидиновых нуклеотидов, коферментов, а также аминокислот гистидина и триптофана [2–6]. В некоторых бактериях 5-PRPP является субстратом более чем десяти ферментов. Поэтому 5-PRPP является важнейшим клеточным метаболитом, а PRPP-синтетазы относятся к ключевым ферментам, необходимым для жизни организма [7].
Некоторые ферменты, участвующие в нуклеотидном обмене, широко применяются в биотехнологии для химико-ферментативного синтеза аналогов природных нуклеотидов, многие из которых используются как противовирусные и противоопухолевые препараты [8]. Показано, что ферменты нуклеотидного обмена представляют интерес как кандидаты для использования в новой стратегии биосинтеза биологически важных нуклеотидов, так называемом каскадном методе биосинтеза [8]. Новая стратегия состоит в мультиферментном каскадном превращении D-пентоз в пуриновые нуклеотиды c использованием определенных ферментов нуклеинового обмена. Каскадный синтез включает в себя последовательное превращение в “одной колбе” D-пентоз сначала в 5-фосфопентозы под действием рибокиназы, затем под действием PRPP-cинтетазы в 5-фосфо-α-D-пентофуранозо-1-пирофосфаты (5-PRPP); присоединение 5-фосфорибозильной группы к гетероциклическому пуриновому основанию в присутствии аденинфосфорибозилтрансферазы (APR-трансферазы) дает конечные нуклеотиды. PRPP-синтетаза катализирует вторую стадию каскада.
Для биотехнологического применения предпочтительны ферменты, имеющие высокий температурный оптимум активности. Таким требованиям отвечают ферменты из термофильных организмов. Не менее важна и субстратная специфичность используемых ферментов. Меньшая степень селективности по отношению к пуриновому основанию и к углеводному субстрату дает возможность расширить круг синтезируемых соединений. Поэтому весьма актуален поиск мутантных форм ферментов, имеющих более широкую специфичность.
Показано, что в геноме бактерии Thermus thermophilus, принадлежащей к группе термофилов, белки с PRPP-синтетазной активностью кодируются двумя генами [9]. Оба гена были клонированы и экспрессированы, обе PRPP-синтетазы, Th. thermophilus PRPP-синтетазы I и II, выделены и охарактеризованы [9]. Оба фермента несколько различаются по длине полипептидной цепи. PRPP-синтетаза I содержит 307, а PRPP-синтетаза II 310 аминокислотных остатков, степень сходства между ними достигает 89%. Высокая степень сходства обеих аминокислотных последовательностей с PRPP-синтетазой из E. coli, более 75%, говорит об их принадлежности к первому классу PRPP-синтетаз. Оба фермента имеют максимальную активность при высокой температуре (75 и 85°С) и в этом отношении подходят для биотехнологического применения. В связи с перспективой биотехнологического применения, особый интерес представляет изучение особенностей структуры данных ферментов в комплексе с функциональными лигандами.
Отметим, что для PRPP-синтетазы из Th. thermophilus имеется пространственная структура фермента в комплексе с аденозиндифосфатом (АДФ) [10], что значительно упрощает построение стартовой модели для молекулярно-динамического эксперимента.
В настоящей работе методом молекулярной динамики получен комплекс PRPP c АТФ и 5-PRPP. На основе полученных данных выбраны аминокислотные остатки, замена которых может привести к изменению субстратной специфичности фермента.
МАТЕРИАЛЫ И МЕТОДЫ
Для проведения молекулярно-динамических экспериментов в качестве исходной модели использованы координаты PRPPS из Th. thermophilus в комплексе с АДФ, взятые из банка данных RCSD PDB(PDB ID:5T3O). Данный комплекс получен в [10] с разрешением в 2.2 Å методом РСА (Р5Ф).
Для добавления в систему необходимых лигандов (АТФ и Р5Ф) при помощи программы Coot [11] получены координаты АТФ путем наложения его на АДФ. Стартовые координаты Р5Ф получены при наложении координат исследуемой структуры на координаты структуры PRPP-синтетазы из Thermoplasma volcanium в комплексе с Р5Ф(PDBID: 3MBI) по Cα-атомам основной цепи. Структура, содержащая Р5Ф, получена методом РСА при разрешении в 1.8 Å.
Для моделирования молекулярной динамики атомы белка и лигандов были параметризованы в силовом поле Charmm36 [12]. Построение топологии белка проведено при помощи встроенной в программу GROMACS [13] утилиты pdb2gmx, построение топологии лигандов – при помощи веб-сервиса Charmmgui [14].
В качестве ячейки для моделируемой системы выбран куб, минимальное расстояние до ребер которого составляло 1 Å. Затем ячейка была заполнена водой типа TIP3P и добавлено 12 ионов Na для компенсации заряда в системе.
Затем была проведена минимизация энергии для релаксации возможных напряжений, возникших при сольватации. После минимизации энергии на атомы системы были наложены ограничения и установлены давление в 1 атмосферу и температура в 330 K. После установления всех необходимых параметров ограничения были сняты и проведен молекулярно-динамический эксперимент, в результате которого получены траектории движения всех атомов системы на временном интервале в 50 нс. Протокол молекулярной динамики описан в таблице.
Таблица 1.
Параметры расчетов
| Система в термостате | |
| Термостат | Модифицированный Berendsen |
| Т, K | 330 |
| Система в баростате | |
| Баростат | Parrinello-Rahman |
| Давление, атм | 1.00 |
| Продолжительность NVT и NPT динамики, пс | 100 |
| Молекулярная динамика | |
| Шаг временной сетки, пс | 0.002 |
| Количество шагов | 25 000 000 |
| Интервал времени, нс | 50 |
| Частота записи координат, пс | 40 |
| Всего кадров траектории | 1200 |
| Интегратор | Leap-frog |
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
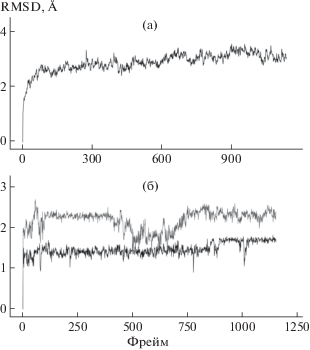
Полученная траектория проанализирована при помощи наложения атомов углерода основной цепи системы на их исходное положение и построен график среднеквадратичного отклонения молекулы белка и лигандов от своего положения (рис. 1). Полученные данные показывают, что система пришла в состояние равновесия.
Положение лигандов уточнено методом молекулярной динамики. После симуляции молекулярной динамики получены и проанализированы траектории движения атомов системы. Определены аминокислотные остатки, участвующие в связывании лигандов в активном центре HisB-Mt. Данные об окружении каждого из лигандов получены и проанализированы с помощью программы LigPlot.
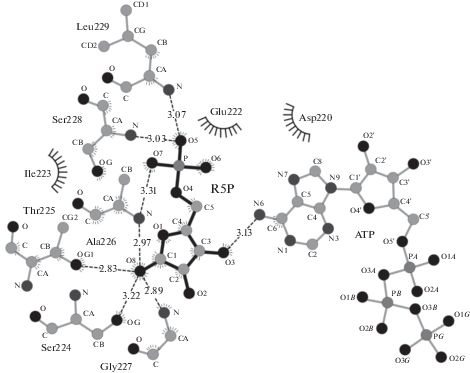
Окружение R5P представлено на рис. 2. Фосфат образует водородные связи с карбоксильной группой Asp220 и Ala226, Ser228, Leu229. Аминокислотные остатки Ser228 и Gly227 связаны с атомом кислорода углеводного цикла. Гидроксильная группа С1 атома цикла образует водородные связи с атомами азота и кислорода гидроксильной группы Thr225 и атомами азота Ala226 и Gly227.
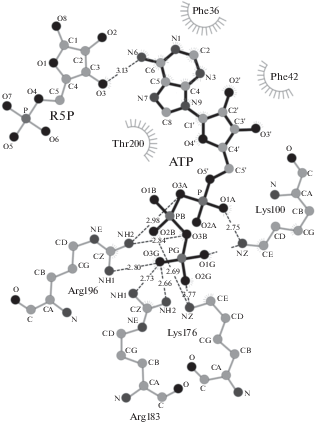
На рис. 3 показано, что АТФ образует водородные связи с белковыми цепями B и F, а также с R5P. Первые выражены связыванием трифосфата с аминокислотными остатками Arg196 и Lys100 цепи B и Arg183 и Lys176 цепи F. С R5P связывание осуществляется посредством образования водородной связи между гидроксильной группой С3-рибозы и NH2-группой аденозина.
Временная модель комплекса PRPPS + R5P + + АТФ проанализирована с использованием программ VMD и GROMACS. Лиганды имеют стабильное положение в активном центре фермента, следовательно, модель может позволить судить об окружении лигандов. Далее с использованием программ VMD и GROMACS был получен файл с координатами модели на второй наносекунде молекулярно-динамической траектории. Файл преобразован в формат .pdb и проанализирован с использованием программы PyMol. За положение рибозного кольца R5P могут отвечать боковые группы аминокислотных остатков Ser-228 и Thr225 как наиболее близких к нему. Следующим этапом работы предполагается экспериментально проверить следующие мутанты: Ser228-Ala и Thr225-Ala, чтобы определить влияние данных аминокислотных остатков на активность и специфичность фермента.
Работа выполнена при поддержке Российского научного фонда (проект № 14-50-00131) в части молекулярного моделирования и Министерства науки и высшего образования в рамках выполнения работ по Государственному заданию ФНИЦ “Кристаллография и фотоника” РАН в части анализа данных молекулярного моделирования.
Список литературы
Khorana H.G., Fernandes J.F., Kornberg A. // J. Biol. Chem. 1958. V. 230. P. 941.
Switzer R.L. // J. Biol. Chem. 1969. V. 244. P. 2854.
Becker M.A., Raivio K.O., Bakay B. et al. // J. Clin. Invest. 1980. V. 65. P. 109.
Hove-Jensen B., Nygaard P. // Eur. J. Biochem. 1982. V. 126. P. 327.
Hove-Jensen B. // Mol. Microbiol. 1983. V. 3. P. 1487.
Hove-Jensen B. // J. Bacteriol. 1988. V. 170. P. 1148.
Eriksen T.A., Kadziola A., Bentsen A.K. et al. // Nat. Struct. Biol. 2000. V. 7. P. 303.
Mikhailopulo A.I., Miroshnikov A.I. // Acta Nature. 2010. V. 10. P. 36.
Cherney M.M., Cherney L.T., Garen C.R. et al. // J. Mol. Biol. 2011. V. 413. P. 844.
Timofeev V.I., Sinitsyna E.V., Kostromina M.A. et al. // Acta Cryst. F. 2017. V. 73. P. 369.
Emsley P., Lohkamp B., Scottc W.G. et al. // Acta Cryst. D. 2010. V. 66. P. 486.
Huang J., Rauscher S., Nawrocki G. et al. // Nature Methods. 2016. V. 14. P. 71.
Hess B., Kutzner C., van der Spoel D. et al. // Chem. Theory Comput. 2008 V. 4. P. 435.
Jo S., Kim T., Iyer V.G. et al. // J. Comput. Chem. 2008. V. 29. P. 1859.
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография