Кристаллография, 2020, T. 65, № 4, стр. 605-607
Кристаллизация и предварительное рентгеноструктурное исследование ZAD-домена белка Serendipity-d из Drosophila melanogaster
К. М. Бойко 1, *, А. Ю. Николаева 2, А. Н. Бончук 3, П. Г. Георгиев 3, В. О. Попов 1, 2
1 Федеральный исследовательский центр “Фундаментальные основы биотехнологии”
Москва, Россия
2 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
3 Институт биологии гена РАН
Москва, Россия
* E-mail: kmb@inbi.ras.ru
Поступила в редакцию 03.03.2020
После доработки 03.03.2020
Принята к публикации 04.03.2020
Аннотация
Пространственная организация генома контролируется особым классом архитектурных белков, к числу которых относятся белки, содержащие гомо- и гетеро-димеризующиеся ZAD-домены. Такие домены являются уникальными для членистоногих и присутствуют на N-конце многих белков, имеющих в своем составе цинковые пальцы типа C2H2. Несмотря на постоянно растущий интерес к исследованию архитектурных белков, имеется очень мало данных об их структуре (структуре их функциональных доменов), которые могли бы пролить свет на механизмы регуляции процессов установления и поддержания пространственной организации хроматиновых доменов. Так, к настоящему моменту установлены лишь две структуры ZAD-доменов. Проведена кристаллизация не описанного ранее ZAD-домена белка Serendipity-d из Drosophila melanogaster и предварительное рентгеноструктурное исследование полученных кристаллов.
ВВЕДЕНИЕ
Пространственная организация генома имеет критическое значение в регуляции процессов, связанных с реализацией генетической информации. Известно, что в процесс регуляции транс-крипции у эукариот вовлечены многочисленные элементы генома, зачастую расположенные на значительном расстоянии друг от друга [1]. В последние несколько лет была сформирована концепция, согласно которой существует особый класс архитектурных белков, участвующих в организации общей архитектуры хромосом, а также в локальном контроле энхансер-промоторных взаимодействий [2–5].
Предполагается, что данные белки способны устанавливать дистанционные взаимодействия, а также привлекать различные белковые многокомпонентные комплексы, которые, в частности, могут менять статус окружающего хроматина, однако соответствующие механизмы действия архитектурных белков до сих пор не выяснены [6, 7].
Исходя из этого, особый интерес для изучения представляют архитектурные белки, способные напрямую или путем привлечения других белков-партнеров формировать белковые комплексы. За счет такого свойства возможно специфичное и эффективное сближение дистанционно разделенных регуляторных элементов генома либо выпетливание хроматиновых доменов.
К числу таких архитектурных белков относятся, в частности, широко распространенные у членистоногих мультидоменные транскрипционные факторы, содержащие на N-конце ZAD-домены (Zinc-finger Associated Domain). Несмотря на широкое распространение ZAD-доменов в составе транскрипционных факторов (в геноме дрозофилы аннотированы 95 различных ZAD-доменов), их функциональная роль остается почти неизученной, хотя и показано, что они играют ключевую роль в организации специфических дистанционных взаимодействий в трансгенной модельной системе [8]. Несмотря на достаточно большой объем молекулярно-биологических данных в этой области, к настоящему моменту имеется существенный недостаток структурных данных, которые могли бы способствовать идентификации структурных мотивов, обеспечивающих некоторым представителям ZAD-доменов способность образовывать как гомо-, так и гетеродимеры. Так, к настоящему моменту известны лишь две пространственные структуры ZAD-домена белков Grauzone (код PDB – 1PZW) и CG2712 (6FP5). Показано, что эти домены имеют атипичный фолд, содержащий две альфа-спирали и подвижные петли, координированные атомом цинка.
В настоящей работе получены кристаллы не описанного ранее ZAD-домена белка Serendipity-d из Drosophila melanogaster. Проведено предварительное рентгеноструктурное исследование полученных кристаллов.
МАТЕРИАЛЫ И МЕТОДЫ
Очистка и выделение ZAD-домена белка Serendipity-d из D. melanogaster, получение кристаллов. ZAD-домен белка Serendipity-d (1–90 ак), слитый с GST, экспрессировали в клетках E. coli штамма BL21 (DE3) в течение ночи при +18°C. Клетки E. coli, содержащие рекомбинантный ZAD-домен белка Serendipity-d, разрушали ультразвуком в буфере: 20 мМ Tris, pH 7.5, 150 мМ NaCl, 20 мM KCl, 5 мМ MgCl2, 0.1% NP40, 0.1 мM ZnCl2, 1 мМ DTT. Лизат центрифугировали при 20 000 g в течение 1 ч при +4°C и наносили на предварительно уравновешенную 2 мл колонку с глутатион-агарозой. Затем колонку промывали 20 мл буфера: 20 мМ Tris, pH 7.5, 500 мМ NaCl, 20 мМ KCl, 5 мМ MgCl2, 0.1% NP40, 0.1 мМ ZnCl2, 1 мМ DTT, а затем 20 мл буфера: 20 мМ Tris, pH 7.5, 200 мМ NaCl, 0.1 мМ ZnCl2, 5 мМ цитрата натрия, 1 мМ DTT. Белок элюировали с колонки, отрезая от GST, для этого добавляли 0.5 мг TEV-протеазы в 10 мл буфера: 20 мМ Tris, pH 7.5, 200 мМ NaCl, 0.1 мМ ZnCl2, 5 мМ цитрата натрия, 1 мМ DTT, колонку закрывали и инкубировали на ротаторе в течение ночи при +4°C. Фракцию проскока собирали и переводили в предварительно дегазированный буфер: 20мМ Tris, pH 7.4, 0.1 мМ ZnCl2, 1 мМ DTT, при помощи колонки HiPrep Desalting (GE Healthcare). Полученный препарат наносили на предварительно уравновешенную тем же буфером колонку Source 15Q (GE Healthcare) и собирали проскок, содержащий белок, концентрировали до 10 мг/мл при помощи концентраторов Millipore для последующей кристаллизации.
Предварительный скрининг условий кристаллизации проводили с использованием роботизированной системы кристаллизации Rigaku (США) [9] и стандартных наборов для кристаллизации глобулярных белков компании Hampton Research (США): Crystal Screen HT, Crystal Screen Cryo HT, Index HT, PEG/Ion HT, PEGRx HT и SaltRx HT. Кристаллизацию проводили в 96-луночных пластиковых планшетах Art Robbins Instruments (США) при температуре 15°С. Использовали белковый препарат с концентрацией 10 мг/мл. Кристаллы белка появились в ряде условий, а для последующей оптимизации были выбраны условия, в которых кристаллы имели наилучший внешний вид (отдельные кристаллы призматической формы) и наибольшие размеры – порядка 50 × 50 × 50 мкм. В этих условиях кристаллы росли в течение 5–7 дней. Оптимизацию кристаллов ZAD-домена проводили методом диффузии в парах (вариант “висячая капля”) с использованием ранее найденных предварительных условий кристаллизации. В процессе оптимизации варьировались концентрации осадителя и белка, а также температура кристаллизации. Финальные условия кристаллизации содержали: 0.1 M хлорида натрия, 0.1 M ХЕПЕС, pH 7.5, 1.5 M сульфата лития. Кристаллы максимального размера росли в течение 7 дней.
Сбор и обработка дифракционных данных. Решение структуры. Перед сбором рентгенодифракционных данных кристаллы ZAD-домена вылавливали петлей и переносили в криораствор, содержащий кроме компонентов, входящих в противораствор, 25% глицерина, после чего кристалл в петле замораживали в парах азота. Дифракционные наборы собирали при температуре 100 K на станции BL41XU синхротронного источника SPring-8 (Япония). Дифракционную картину фиксировали детектором Pilatus6M-F. Для расчета стратегии сбора данных использована программа HKL2000 [10], предложившая следующие параметры: длина волны 1.0 Å, угол вращения 720°, угол качания 0.5°, расстояние кристалл–детектор 700 мм. Набор дифракционных данных обработан с использованием программы iMosflm [11]. Характеристики кристалла и статистика набора данных приведены в табл. 1.
Таблица 1.
Кристаллографические данные и параметры съемки кристалла ZAD-домена белка Serendipity-d
| Пр. гр. | P41212 |
| a, b, c, Å; | 97.79, 97.79, 92.69 |
| α = β = γ, град | 90 |
| Т, K | 100 |
| λ, Å | 1.0 |
| Разрешение, Å | 97.79–3.67 (4.02–3.67)* |
| Число независимых рефлексов | 5202 (1206) |
| Повторяемость | 5.2 (5.0) |
| Полнота набора, % | 98.90 (99.0) |
| I/σ (I) | 5.4 (2.3) |
| Rmeas, % | 17.0 (50.8) |
| CC1/2,% | 99.2 (94.5) |
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Использованные в рентгеноструктурном эксперименте кристаллы ZAD-домена белка Serendipity-d из D. melanogaster имели форму призмы (рис. 1) и размеры порядка 200 × 200 × 300 мкм.
Рис. 1.
Кристаллы ZAD-домена белка Serendipity-d из D. melanogaster, выросшие в оптимизированных условиях.

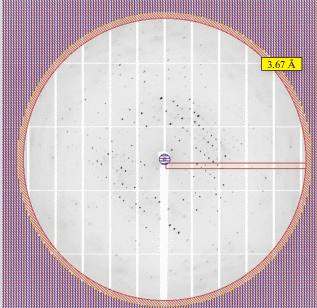
Кристаллы дифрагировали до разрешения 3.67 Å (рис. 2) и принадлежали пр. гр. P41212 c параметрами ячейки a = b = 97.79, с = 92.69 Å. Оценка содержания растворителя в элементарной ячейке по методу Мэттьюса [12], проведенная с помощью программы из пакета CCP4 [13], показала, что в независимой части содержатся две субъединицы белка.
Рис. 2.
Дифракционная картина от кристаллов ZAD-домена белка Serendipity-d из D. melanogaster, визуализированная в iMosflm.
Решение структуры методом молекулярного замещения (программа MOLREP [14]) с использованием в качестве модели известных структур ZAD-доменов (коды PDB – 6FP5, 1PZW), а также их фрагментов, не дало результата. Возможной причиной является относительно низкое разрешение полученных структурных данных. В дальнейшем планируется провести оптимизацию других условий кристаллизации, полученных для данного белка, с целью улучшения качества кристаллов и снять набор данных для аномального рассеяния на атоме цинка, входящем в состав данного белка, для решения структуры методом аномального рассеяния.
Работы по экспрессии и выделению белка выполнены при финансовой поддержке Российского научного фонда (грант № 19-74-10099), кристаллизация и рентгеноструктурный эксперимент выполнены при поддержке Федерального космического агентства (проект КЭ (ЦР) “Кристаллизатор”), работы по анализу полученных данных выполнены при поддержке Министерства науки и высшего образования РФ.
Список литературы
Szabo Q., Bantignies F., Cavalli G. // Sci. Adv. 2019. V. 5. https://doi.org/10.1126/sciadv.aaw1668
Dekker J., Mirny L. // Cell. 2016. V. 164. P. 1110.
Maksimenko O., Georgiev P. // Front Genet. 2014. V. 5. P. 28.
Maksimenko O., Kyrchanova O., Klimenko N. et al. // Biochim Biophys Acta Gene Regul Mech. 2020. V. 1863. P. 194446.
Zolotarev N., Maksimenko O., Kyrchanova O. et al. // Nucleic Acids Research. 2017. V. 45. P. 12285.
Fedotova A.A., Bonchuk A.N., Mogila V.A. et al. // Acta Naturae. 2017. V. 9. P. 47.
Zheng H., Xie W. // Nat. Rev. Mol. Cell. Biol. 2019. V. 20. P. 535.
Zolotarev N., Fedotova A., Kyrchanova O. et al. // Nucleic Acids Res. 2016. V. 44. P. 7228.
Boyko K.M., Lipkin A.V., Popov V.O. et al. // Crystallography Reports. 2013. V. 58. P. 442.
Otwinowski Z., Minor W. // Macromolecular Crystallography. Pt. A. 1997. V. 276. P. 307.
Battye T.G.G., Kontogiannis L., Johnson O. et al. // Acta Cryst. D. 2011. V. 67. P. 271.
Matthews B.W. // J. Mol. Biol. 1968. V. 33. P. 491.
Winn M.D., Ballard C.C., Cowtan K.D. et al. // Acta Cryst. D. 2011. V. 67. P. 235.
Vagin A.A., Isupov M.N. // Acta Cryst. D. 2001. V. 57. P. 1451.
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография


