Кристаллография, 2020, T. 65, № 6, стр. 995-1008
Потенциал современных информационных технологий для анализа данных экспериментов на установках Cryo-EM и XFEL
С. А. Бобков 1, *, А. Б. Теслюк 1, 2, Т. Н. Баймухаметов 1, Е. Б. Пичкур 1, Ю. М. Чесноков 1, Д. Ж. Ассалауова 3, А. А. Пойда 1, 2, А. М. Новиков 1, С. И. Золотарев 3, К. А. Иконникова 1, В. Е. Велихов 1, И. А. Вартаньянц 3, 5, А. Л. Васильев 1, 2, 4, В. А. Ильин 1, 2
1 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
2 Московский физико-технический институт
Долгопрудный, Россия
3 Немецкий центр по синхротнонным исследованиям (DESY)
Гамбург, Германия
4 Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника” РАН
Москва, Россия
5 Национальный исследовательский ядерный университет “МИФИ”
Москва, Россия
* E-mail: s.bobkov@grid.kiae.ru
Поступила в редакцию 14.06.2020
После доработки 14.06.2020
Принята к публикации 22.06.2020
Аннотация
Представлен новый подход к организации конвейерной обработки данных экспериментов крио-электронной микроскопии (Cryo-EM) и на рентгеновских лазерах на свободных электронах (XFEL) с использованием современных информационно-технологических (ИТ) достижений, связанных с развитием технологий контейнеризации. Такой подход позволяет отделить работу пользователя на прикладном уровне от разработок ИТ-специалистов на системном и промежуточном уровнях. Пользователю остается только выполнить две несложные подготовительные операции: поместить прикладные пакеты в контейнеры и записать прикладную логику конвейерной обработки данных в формате прикладного композитного задания. Обсуждаются примеры контейнеризированных конвейеров для Cryo-EM- и XFEL-экспериментов по изучению пространственной структуры одиночных наноразмерных биологических объектов (вирусов, макромолекул и др.). На сайте проекта по разработке контейнеризированного конвейера представлены примеры программных кодов помещения прикладных пакетов в Docker-контейнеры, а также примеры прикладных композитных заданий, написанных на языке высокого уровня CWL. Примеры снабжены комментариями, что поможет неискушенному в ИТ исследователю получить представление, как организовывать Docker-контейнеры и формировать CWL-композитные задания для Cryo-EM- и XFEL-конвейерной обработки данных.
ВВЕДЕНИЕ
Прогресс последних десятилетий в области информационных технологий (ИТ) и вычислительных средств позволил радикально расширить спектр решаемых задач в самых разных научных дисциплинах. Одно из направлений ИТ, получивших системное развитие в последние десять лет, возможно, способно кардинально изменить прикладную парадигму компьютинга для обработки больших наборов данных в естественно-научных экспериментах. Это направление связано с развитием технологий контейнеризации, которые позволяют эффективно использовать в приложениях программные модули, созданные для работы на разных вычислительных платформах.
Как правило, для проведения таких экспериментов организуется международное научное сообщество, которое разрабатывает различные вычислительные модули для работы с данными на разных этапах их анализа. Такие прикладные разработки часто ориентированы на выполнение на различных компьютерных платформах и вычислительных средах, вплоть до разных операционных систем. Высокая степень такой гетерогенности представляет большую проблему для неискушенных пользователей, которым приходится “вручную” объединять отдельные этапы обработки данных, начиная с “сырых” данных до получения конечных результатов. Серьезные проблемы также могут возникать и при попытках использования внешних пакетов на высокопроизводительных вычислительных ресурсах – на суперкомпьютерах и с использованием графических ускорителей. Для организации комплексной работы пользователям приходится привлекать ИТ-специалистов, что представляет дополнительные проблемы, как минимум, научно-организационного характера.
В настоящее время в центре внимания научного сообщества находятся исследования пространственных структур биологических макромолекул (белков, вирусов, макромолекулярных комплексов). Все больший интерес структурных биологов привлекают такие экспериментальные подходы, как криогенная просвечивающая электронная микроскопия (Cryo-EM) [1], и эксперименты с использованием лазеров на свободных электронах (XFEL) [2], которые позволяют восстанавливать пространственную структуру биологических макромолекул с разрешением вплоть до межатомных расстояний. Отметим, что в 2017 г. за развитие Cryo-EM была присуждена Нобелевская премия И. Франку, Р. Хендерсону и Ж. Дюбоше. Исходные данные в таких экспериментах представляют собой набор двумерных изображений исследуемых объектов – двумерных (2D) проекций трехмерных структур в прямом пространстве в случае Cryo-EM-экспериментов и 2D-дифракционных картин в обратном пространстве в случае XFEL-экспериментов.
В представленной работе описаны перспективы современных ИТ-достижений для преодоления перечисленных выше проблем при организации конвейерной обработки больших наборов данных, получаемых в экспериментах по изучению пространственной структуры наноразмерных частиц методом, который в Cryo-EM получил название Single Particle Analysis (SPA) [3]. В XFEL-экспериментах принято использовать несколько другую терминологию – Single Particle Imaging (SPI) [4, 5]. Интерес к таким экспериментам связан в основном с тем, что традиционно используемый в структурной биологии метод рентгеноструктурного анализа подразумевает необходимость кристаллизации объектов исследования, при этом во многих случаях кристаллизация либо невозможна, либо существенно затруднена.
Процесс обработки больших наборов 2D-изображений использует математический аппарат, во многом аналогичный задачам распознавания трехмерных образов по их 2D-проекциям в технологиях искусственного интеллекта, бурно и успешно развивающихся в последние годы [6, 7]. Современные достижения в области искусственного интеллекта стали активно применяться в обработке данных экспериментов в структурной биологии, в том числе в SPA Cryo-EM- и SPI XFEL-экспериментах. В настоящее время в этих приложениях большое внимание привлекают сверточные нейронные сети [8, 9], показавшие высокую эффективность в задачах по распознаванию образов. Поэтому в различных исследовательских группах в мире разрабатываются модули с использованием технологий искусственного интеллекта для отдельных этапов обработки данных SPA/SPI экспериментов, в том числе в виде свободного программного обеспечения (ПО). Их использование другими исследовательскими группами в составе своих вычислительных конвейеров часто затруднено в связи с несовместимостью доступных компьютерных ресурсов с программным окружением, для которого такой пакет разрабатывался.
В данной работе обсуждается организация конвейерной обработки данных SPA Cryo-EM- и SPI XFEL-экспериментов с использованием технологии контейнеризации. Эта технология позволяет встраивать в конвейер внешние вычислительные модули и дает возможность выполнения полного цикла обработки данных на гетерогенных вычислителях, включая суперкомпьютеры с использованием графических ускорителей. Предварительный анализ рабочих потоков конвейерной обработки данных SPA Cryo-EM- и SPI XFEL-экспериментов был опубликован в [10, 11].
В первом разделе настоящей работы описаны прикладные аспекты SPA Cryo-EM- и SPI XFEL-экспериментов с акцентом на актуальное состояние компьютерной обработки и анализ данных в этих экспериментах.
Во втором разделе описана технология контейнеризации Docker [12] – помещение в контейнер прикладного вычислительного модуля вместе с программной средой, необходимой для его работы. Также в этом разделе описана технология оркестрации Kubernetes [13] – организация совместной работы контейнеров в Docker-среде. Здесь же дано описание технологии микросервисов [14], используемой для универсальной организации взаимодействия между контейнерами. Для управления данными и запусками контейнеров на выполнение в среде Docker использована система REANA [15]. Наконец, в этом разделе будет описан язык высокого уровня CWL [16], с помощью которого ИТ-неискушенный пользователь может организовать конвейерную обработку данных с использованием перечисленных выше технологий. Детальное изложение технологических аспектов разработки контейнеризированной платформы с обсуждением примера ее использования для организации конвейерной обработки модельных данных SPI XFEL-эксперимента и восстановления трехмерной структуры белка KLH1 [17] опубликовано в [18].
В третьем разделе прикладные аспекты разработанной авторами контейнеризированной платформы обсуждаются на примере тестовой обработки данных SPA эксперимента TvNiR [19], выполненного в 2017 г. на просвечивающем крио-электронном микроскопе Titan Krios в НИЦ “Курчатовский институт”.
В четвертом разделе обсуждаются прикладные аспекты реализации контейнеризированной платформы для конвейерной обработки SPI XFEL-экспериментов.
В Заключении представлены планы по дальнейшему развитию прикладных аспектов разработанной контейнеризированной платформы для организации конвейерной обработки данных SPA Cryo-EM- и SPI XFEL-экспериментов.
1. ПРИКЛАДНЫЕ АСПЕКТЫ SPA/SPI-ЭКСПЕРИМЕНТОВ ПО ИССЛЕДОВАНИЮ ПРОСТРАНСТВЕННОЙ СТРУКТУРЫ ОТДЕЛЬНЫХ БИОЛОГИЧЕСКИ ОБЪЕКТОВ
Крио-электронная микроскопия. Бурное развитие метода Cryo-EM в последние годы связано в основном с двумя факторами:
– развитие приборно-инструментальной базы, прежде всего появление высокоэффективных детекторов электронов [20];
– совершенствование методов обработки данных Cryo-EM [21, 22].
Группу методов Cryo-EM c использованием просвечивающей электронной микроскопии прежде всего представляют два метода – анализ (проекций) одиночных частиц (SPA) и крио-электронная томография [23]. Оба метода имеют свою специфику. Так как в настоящей работе представлены результаты автоматизации эксперимента в области анализа одиночных частиц, будем говорить преимущественно об этом методе, хотя и крио-электронная томография вписывается в рамки предлагаемых решений обработки результатов. Статистические данные, представленные на сайте базы данных белков (Protein data bank, [24]), свидетельствуют, что к середине 2020 г. с пространственным разрешением лучше 4 Å определено около двух тысяч структур, а с разрешением хуже 4 Å – более пяти тысяч. Метод SPA позволяет определить с высоким разрешением структуру белков различного типа, включая мембранные белки. Результаты работ представлены в ряде статей и обзоров [25–27].
В SPA Cryo-EM-эксперименте образец, замороженный (витрифицированный) при температуре жидкого этана, представляет собой электронно-микроскопическую сетку с тонким (как правило, 30–80 нм) слоем аморфного льда, содержащим исследуемые частицы (макромолекулы, вирусы, белки). Образец, соблюдая криогенный температурный режим, помещается в крио-электронный микроскоп. При получении одного изображения удается зафиксировать около ста частиц в случайных пространственных ориентациях. Количество частиц определяется концентрацией подготовленного раствора, их размерами и варьируется в достаточно широких пределах. Из этого изображения необходимо извлечь проекции исследуемых объектов, которые фактически представляют собой 2D-проекции трехмерного рассеивающего электростатического потенциала частиц. На этом этапе в получившемся наборе могут оказаться изображения различных загрязнений: частицы кристаллического льда, края углеродной сетки и прочие ложно-положительные изображения – шум, который не подлежит дальнейшей обработке. Необходимо провести отсев примесных изображений. Для 2D-проекций исследуемого объекта необходимо определить соответствующие пространственные ориентации, затем их можно объединять в трехмерную структуру. Таким образом, эту весьма упрощенную схему обработки можно разделить на пять базовых этапов:
1) подготовка и витрификация образцов;
2) получение экспериментальных данных – наборов экспозиций (стеков), состоящих из ряда отдельных изображений, полученных при средней дозе 1–3 э/Å2;
3) первичная обработка стеков:
a) коррекция влияния дрейфа образца на изображение отдельных частиц. Дрейф возникает из-за неоднородного распределения температуры в электронно-микроскопическом образце, а также напряжений в материалах сеток, создаваемых при сверхбыстрой заморозке;
б) оценка параметров приборной функции передачи контраста [28]. Из-за крайне низкого контраста органических частиц, состоящих в основном из сравнительно легких атомов (углерод, азот, кислород, водород), изображения приходится получать с дефокусировкой в диапазоне 0.5–3 мкм, что приводит к искажению “сырых” изображений частиц вследствие модуляции сигнала функцией передачи контраста;
4) отбор 2D-изображений исследуемых частиц по морфологическим критериям близости изображений к предполагаемой структуре (четвертичной в случае белка или макромолекулы) и их 2D-классификация [29];
5) реконструкция 3D-карты электростатического потенциала. На этом этапе (в начале или в процессе) проводится и 3D-классификация полученных данных с целью обнаружения конформаций объекта исследования, которые могут присутствовать в растворе в момент заморозки. 3D-классификация и 3D-реконструкция могут проводиться итерационно, а также “фокусироваться” на отдельных областях макромолекул, вызывающих интерес исследователя.
В SPA Cryo-EM-экспериментах по определению структуры белков (стандартно) используются следующие прикладные программные пакеты:
− на этапе 2: EPU (Thermo Fisher Scientific, США), SerialEM [30];
− на этапе 3: Motioncor2 [31], CTFFIND [32], GCTF [33];
− на этапах 4, 5: Gautomatch [34], Relion [35], CisTEM [36] и cryoSPARC [22].
В разд. 4 обсуждаются прикладные аспекты организации контейнеризированного конвейера обработки SPA Cryo-EM-данных на примере эксперимента TvNiR [19]. В этом эксперименте исследовалась трехмерная структура фермента цитохром c нитритредуктазой из бактерии Thioalkalivibrio Nitratireducens. Она функционирует в форме стабильного гексамера с характерным размером около 120 Å, молекулярной массой порядка 370 кДа, симметрией D3 и катализирует реакцию окисления нитрита до аммония. В [19] была восстановлена карта электронной плотности фермента с разрешением 2.56 Å и построена соответствующая пространственная модель. “Сырые” данные (стеки) были получены в эксперименте на просвечивающем крио-электронном микроскопе Titan Krios 60-300 (Thermo Fisher Scientific, США), оснащенном источником полевой эмиссии электронов типа Шоттки XFEG, корректором сферических аберраций (CEOS GmbH, Германия) и КМОП (CMOS) – устройством прямого детектирования электронов Falcon II (Thermo Fisher Scientific, США) при ускоряющем напряжении 300 кВ.
В эксперименте было получено 3 055 двумерных стеков изображений белка TvNiR, состоящих из 30 отдельных изображений, с общим временем детектирования 1.5 с, с использованием программы EPU (версия 1.9.1.16 REL, Thermo Fisher Scientific, США). Экспериментальные данные обрабатывали в несколько последовательных этапов на базе высокопроизводительного вычислительного кластера Курчатовского центра обработки данных, оснащенного графическими ускорителями Nvidia Tesla K80. Для основных процедур восстановления пространственной структуры использовали следующие прикладные программные пакеты: Motioncor2 (версия 1.0.5), GCTF (версия 1.18), Gautomatch (версия 0.56) и Relion. Подробности использования ПО, оценки пространственного разрешения и восстановления трехмерных карт электронной плотности приведены в [19].
Сбор “сырых” данных – стеков изображений образца TvNiR, занимал около 72 ч (с учетом настройки прибора и сессии EPU). Полный набор “сырых” изображений объемом ∼3 ТБ состоял из 3055 стеков. Весь процесс обработки полученных “сырых” экспериментальных данных с учетом выполнения процедур, требующих контроля и манипуляций человека, занимал 75 ч.
Рентгеновские лазеры на свободных электронах (XFEL). SPI-эксперименты, использующие интенсивные фемтосекундные рентгеновские импульсы от лазеров на свободных электронах (XFEL), рассматриваются как перспективный инструмент для изучения структуры биологических объектов в их естественном состоянии при комнатной температуре без кристаллизации образцов [5]. В начале 2000-х годов было высказано предположение, что ультракороткие импульсы от XFEL могут давать дифракционные картины до развития радиационных повреждений и поэтому могут быть использованы для восстановления пространственной структуры изолированных макромолекул с атомарным разрешением [4]. Сразу после начала работы первых XFEL-установок [37, 38] стало ясно, что прогресс в получении трехмерных изображений электронной плотности некристаллизованных биологических частиц (макромолекул) с высоким разрешением в рамках SPI-подхода оказался не таким быстрым по сравнению с методом SFX – серийной фемтосекундной кристаллографией [39–42]. Существует несколько причин более низкого разрешения, получаемого в экспериментах SPI по сравнению с методом SFX. Наиболее важными из них являются: слабый сигнал от одиночной частицы по сравнению с фоном прибора, ограниченное число изображений исследуемого объекта, собираемых в ходе эксперимента, и неоднородность образца. Чтобы преодолеть ограничения SPI-методики, несколько лет назад был создан консорциум SPI [43]. В последние годы достигнут определенный прогресс в совершенствовании этой методики. Помимо технических ограничений существует проблема, с которой сталкивается SPI-метод – большой объем данных, которые собираются и должны быть проанализированы в ходе и сразу после эксперимента желательно в режиме квази-онлайн.
Как было отмечено ранее [5], процесс восстановления пространственной структуры одиночных частиц разбивается на несколько этапов: начальный анализ и предварительный отбор собранных данных, классификация 2D-дифракционных картин, определение относительной ориентации дифракционных картин в обратном пространстве, восстановление комплексных фаз (теряемых при фиксации интенсивности рентгеновских волн на детекторе) и восстановление трехмерной структуры.
Этот рабочий процесс был разработан в [44–47], однако сейчас полный анализ занимает от нескольких месяцев до нескольких лет. В сегодняшних вариантах рабочего потока по обработке SPI-данных экспериментов на XFEL все еще много ручной работы и неоптимальных процедур. В настоящее время возникла необходимость в разработке полностью автоматизированной конвейерной обработки SPI XFEL-данных, начиная с «сырых» данных и заканчивая восстановлением пространственной структуры изучаемых биологических объектов, в режиме квази-онлайн. Решение этой задачи позволит получать первый вариант 3D-структуры исследуемых объектов уже в ходе эксперимента, что особенно важно для отладки процесса накопления данных. Кроме того, конвейерная обработка данных в режиме квази-онлайн даст возможность оперативно управлять ходом эксперимента, например досрочно прекращать эксперимент, если в процессе конвейерной обработки выявятся какие-то критические отклонения от ожидаемых характеристик. Создание конвейерной квази-онлайн обработки SPI-данных особенно актуально в связи с началом работы лазеров на свободных электронах с мегагерцовой частотой генерации рентгеновских фемтосекундных импульсов на Европейском XFEL [48].
2. ТЕХНОЛОГИИ КОНТЕЙНЕРИЗАЦИИ
Часто используемый подход для построения сложных и в то же время гибких систем обработки данных основан на микросервисной архитектуре [14]. Ключевая идея данной архитектуры – разбиение на системном уровне используемого (прикладного) ПО на небольшие самодостаточные компоненты, каждая из которых обособленно выполняет свою часть работы. Каждая из этих программных компонент работает в своем системном окружении, что чаще всего реализуется с помощью технологий контейнеризации, таких как Docker [12], LCX [49] или CRI-O [50]. Это позволяет легко объединять разнообразные программные продукты с различными требованиями к программному окружению на системном или прикладном уровнях для работы в рамках конкретной инфраструктуры. Разделение программных средств на слабо зависимые друг от друга компоненты (микросервисы) и стандартизация взаимодействия микросервисов между собой позволяют гибко модифицировать отдельные из них, не затрагивая остальные элементы разрабатываемой (конвейерной) системы.
Использование технологии контейнеризации обеспечивает переносимость контейнеров между различными вычислительными инфраструктурами. Контейнеры Docker соответствуют стандартному формату для описания процедуры создания образа контейнера [51], а также стандартному открытому формату для хранения образов контейнеров [52]. Это обеспечивает переносимость контейнеров между различными вычислительными инфраструктурами без конвертации образов. Кроме того, стандартный формат хранения образов контейнеров позволяет легко организовать репозитории контейнеризированного программного обеспечения, такие как Docker Hub [53]. Дальнейшая установка и запуск программных продуктов любой сложности могут быть выполнены неподготовленным пользователем с помощью одной команды с указанием адреса репозитория и идентификатора приложения.
Необходимым компонентом системы для запуска контейнеризированных приложений в больших вычислительных инфраструктурах является оркестратор – система управления вычислительными ресурсами и контейнерами. Система Docker обеспечивает запуск и управление контейнеров на одном узле, при этом отдельной задачей является распределение приложений по разным узлам и обеспечение взаимодействия между контейнерами на разных узлах. Данную задачу решают такие системы оркестрации контейнеров, как Docker Swarm [54], Rancher [55] и Kubernetes [13]. В задачу оркестратора входят управление вычислительными ресурсами, организация виртуальной сетевой инфраструктуры для взаимодействия контейнеров и обмена данными, управление системами хранения, управление вычислительными задачами и их масштабирование, разграничение полномочий и обеспечение безопасности. В данной работе использовали оркестратор Kubernetes, который предоставляет разработчикам приложений большие возможности для управления контейнерами. Эта система оркестрации доступна в большинстве вычислительных центров как научных, так и в коммерческих, облачных провайдерах, таких как Google Cloud или Amazon AWS. Применение стандартной платформы Kubernetes для развертывания приложений обеспечивает легкую переносимость контейнеризированных конвейеров между различными вычислительными центрами.
Еще одним необходимым компонентом организации конвейера по обработке данных является система управления заданиями, которая обеспечивает запуск, доступ к входным, промежуточным и выходным данным, а также управляет ходом выполнения конвейера. В данной работе использована система REANA [15], созданная в ЦЕРН для создания воспроизводимых процедур анализа данных. Выбор REANA был обусловлен двумя факторами: совместимость с платформой Kubernetes и контейнеризированными приложениями; поддержка языка CWL для высокоуровневого описания прикладных композитных заданий. Одна из целей данной работы – обеспечение пользователей удобным инструментом для самостоятельной подготовки эффективного выполнения на различных ИТ-инфраструктурах анализа экспериментальных данных. Использование стандартного языка описания прикладных заданий позволяет пользователю задать логику обработки данных на высоком уровне абстракции, в то время как система REANA и платформа Kubernetes обеспечат эффективную организацию и масштабирование вычислений на системном уровне.
Для запуска заданий на языке CWL существует базовая программная реализация в виде пакета cwl-runner [16]. Однако она позволяет выполнять композитные задания только в пределах одного узла. В разрабатываемом подходе с использованием технологий контейнеризации выполнение конвейерной обработки на суперкомпьютерах в распределенном режиме обеспечивается системой оркестрации Kubernetes и системой управления работой конвейеров REANA.
Использование стандартных компонент промежуточного программного уровня, таких как Docker, Kubernetes и REANA, позволяет избавить пользователя от работы по сборке и настройке приложений, а также специальных программных реализаций для запуска на выполнение с учетом особенностей доступных вычислительных ресурсов, включая параллелизацию вычислений. Такой подход дает возможность конечному пользователю создавать переносимые программные конвейеры, которые могут быть легко развернуты в любом вычислительном центре, где поддерживается платформа Kubernetes.
Предлагаемая схема организации конвейера обработки экспериментальных данных позволяет отделить часть, связанную с низким (системным/промежуточным) программным уровнем, которая обеспечивается системными программистами независимо от конкретики прикладной задачи, от прикладного уровня, где задается логика обработки данных, что только и является задачей для пользователей. На системном/промежуточном уровне обеспечивается технологическая программная инфраструктура (в составе технологий, описанных выше), связанная с управлением ресурсами и организацией масштабирования вычислений.
На прикладном уровне остаются только два типа разработок, непосредственно связанных с особенностями прикладной работы: помещение прикладных пакетов/модулей в контейнеры и написание на языке высокого уровня CWL логики конвейерной обработки данных в формате композитного задания. Примеры упаковки прикладного пакета в Docker-контейнер и примеры композитного задания для SPA Cryo-EM- и SPI XFEL-приложений можно найти по ссылке [56], в которую включены комментарии и пояснения используемых CWL языковых конструкций.
3. РЕАЛИЗАЦИЯ КОНТЕЙНЕРИЗИРОВАННОГО КОНВЕЙЕРА ДЛЯ ОБРАБОТКИ ДАННЫХ SPA CRYO-EM-ЭКСПЕРИМЕНТОВ НА ПРИКЛАДНОМ УРОВНЕ
Полная обработка SPA Cryo-EM-данных может быть проведена с помощью программного пакета Relion [35]. Пакет разработан в 2012 г., актуальная версия 3.0 выпущена в 2019 г. Он предоставляет возможность автоматического выполнения всех этапов обработки, от “сырых” данных до восстановления 3D-структуры.
Опишем реализацию контейнеризированного конвейера для обработки SPA Cryo-EM-данных на примере эксперимента с белком TvNiR [19]. Обработка SPA Cryo-EM-данных представляла собой выполнение следующей последовательности операций:
1) коррекция дрейфа;
2) оценка параметров функции передачи контраста (CTF);
3) поиск координат (проекций) частиц;
4) извлечение проекций найденных частиц со сжатием в 4 раза по каждой оси (binning 4);
5) 2D-кластеризация проекций найденных частиц на 30 групп, каждая группа объединяет изображения со схожей структурой;
6) ручная выборка групп изображений частиц, соответствующих исследуемому объекту;
7) сохранение отобранных проекций частиц со сжатием в 2 раза по каждой оси (binning 2);
8) повторная 2D-кластеризация сжатых проекций частиц на 15 групп, выбранных после первого этапа кластеризации;
9) повторная ручная выборка классов изображений частиц, соответствующих исследуемому объекту;
10) построение начальной 3D-карты пространственного распределения электростатического потенциала (ЭП) на основе выбранных на предыдущих этапах групп изображений;
11) уточнение начальной 3D-карты пространственного распределения ЭП с использованием полноразмерных изображений отдельных частиц.
Все этапы реализованы в пакете Relion, для их выполнения требуются либо встроенные в пакет модули, либо внешние программы. Для обработки данных эксперимента TvNiR использовали внешние программы Motioncor2 [31] и CTFFIND [32] на первом и втором этапах соответственно. Поэтому в отличие от реализации контейнеризированного конвейера для обработки XFEL-данных, в которой в разные контейнеры помещали разные прикладные пакеты, в данном случае все пакеты (Relion, Motioncor2 и CTFFIND) вкладывали в один контейнер, который обозначили как Cryo-EM-контейнер. Полученный Cryo-EM-контейнер использовали для выполнения всех этапов обработки данных эксперимента TvNiR, при этом разные этапы обработки выполняли с помощью вызова на исполнение этого контейнера с разными входными параметрами.
Одним из ключевых аспектов формирования полного рабочего потока и его (автоматического) исполнения в вычислительной среде Docker является запись порядка вызовов Cryo-EM-контейнера на выполнение различных этапов обработки с описанием соответствующих этим этапам параметров запуска и стеков входных и выходных данных. В ИТ-литературе такая запись называется “прикладным композитным заданием” [57]. Для вычислительной среды Docker запись композитных заданий выполняется с использованием специального языка высокого уровня CWL.
Опишем пример композитного задания для выполнения первого этапа “Коррекция дрейфа”. В этом случае во входных параметрах необходимо указать ссылку, по которой размещены файлы входных данных, поступивших после он-лайн-обработки данных на выходе из Cryo-EM. Также для корректировки дрейфа необходимо указать параметры проведения эксперимента – угловое разрешение, напряжение и доза облучения для одного изображения. К выходным данным первого этапа относятся ссылки на директорию с данными (2D-изображениями), в которых дрейф скорректирован, а также на файл, содержащий таблицу параметров корректировки дрейфа, которые были определены на этом этапе.
В программной среде пакета Relion вычисления проводятся в предположении, что все необходимые для вычислений файлы находятся в одной директории, доступной для пакета Relion в любой момент времени. Однако при выполнении композитного задания в среде Docker есть возможность выполнять отдельные этапы задания на любых доступных вычислительных ресурсах, что является потенциально важным для повышения эффективности выполнения полного цикла обработки данных. При выполнении композитных заданий доступ обеспечивается только к файлам и директориям, ссылки на которые были явно указаны во входных параметрах при вызове контейнера. Например, при выполнении этапов “Сохранение изображений частиц” Relion неявно использует данные, которые создаются на этапе “Учет CTF”. Соответственно в CWL-композитном задании все необходимые ссылки должны быть явно прописаны.
В результате при выполнении CWL-композитного задания отдельные этапы конвейерной обработки данных выполняются автоматически при наличии свободных вычислительных ресурсов и доступности входных данных. Пользователю не требуется следить за выполнением отдельных этапов и вручную запускать последующие.
Заметим, что при обработке данных SPA-экспериментов есть возможность “естественной” параллелизации вычислений – весь набор “сырых” данных разбивается на несколько групп, которые обрабатываются параллельно (независимо друг от друга). В случае обработки SPA Cryo-EM-данных такой “естественный” параллелизм возможен на первых четырех этапах. В стандартной практике выполнения вычислений в пакете Relion для реализации “естественной” параллелизации пользователи должны вручную писать скрипты запуска заданий и адаптировать их к доступной вычислительной инфраструктуре. При выполнении CWL-композитного задания в среде Docker решение этой задачи существенно облегчено благодаря встроенной возможности разделить список входных файлов на соответствующее число ветвей для параллельной обработки.
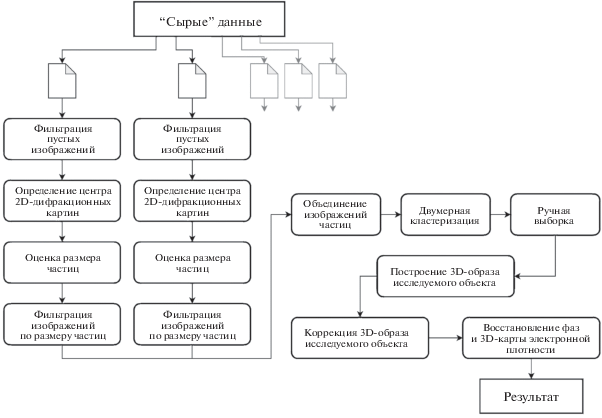
На рис. 1 представлена схема разработанного CWL-композитного задания для обработки SPA Cryo-EM-данных на примере обработки данных TvNiR-эксперимента. На схеме показано разбиение “сырых” данных на несколько поднаборов, которые обрабатываются параллельно. При этом две параллельные ветви показаны с расшифровкой выполняемых операций (корректировка дрейфа, определение параметров CTF, поиск и сохранение изображений отдельных частиц) с последующим сбором выходных наборов обработанных 2D-изображений в объединенный стек для выполнения дальнейших этапов обработки, начиная с этапа 2D-кластеризации.
Рис. 1.
Рабочий поток обработки данных эксперимента TvNiR. Слева представлены две параллельные ветви рабочего потока, каждая из которых выполняется для одного входного файла. Результаты всех ветвей объединяются перед этапом 2D-кластеризации.

В настоящее время в пакете Relion остался один этап, который выполняется пользователем вручную. Это этап ручной выборки классов (кластеров), полученных на предыдущем этапе 2D-кластеризации. Пользователь анализирует усредненные изображения для каждого кластера и, отбрасывая изображения примесей и артефактов, выбирает те, которые, по мнению пользователя, относятся к исследуемому объекту. Например, при обработке ста стеков из эксперимента TvNiR на первом этапе 2D-кластеризации обрабатывалось 27 650 изображений одиночных частиц, и все эти изображения были разбросаны на 30 групп (изменяемый параметр). Наиболее крупный кластер включал в себя 4 256 изображений, в некоторых кластерах содержалось всего одно изображение. Через графический интерфейс Relion вручную было выбрано пять кластеров, которые в сумме содержали 19 084 изображений.
Наиболее часто для графического интерфейса внутри контейнеров используют внешнюю программу Xpra [58]. Поэтому и эта программа была вложена в тот же Cryo-EM-контейнер. Когда CWL-композитное задание выполнит этап “Двумерная кластеризация” для перехода на этап “Ручная выборка”, внутри контейнера запускается веб-сервер Xpra. К нему подключается пользователь и проводит отбор получившихся кластеров через графический интерфейс Relion на веб-браузере (рис. 2). Пользователю необходимо отобрать и сохранить кластеры изображений, относящихся к исследуемому объекту. По окончании этапа пользователь закрывает окно графического интерфейса Relion. После этого возобновляется автоматическое выполнение CWL-композитного задания и начинается выполнение следующих этапов.
Рис. 2.
Графический интерфейс Relion для отбора 2D-классов (кластеров изображений), соответствующих структуре исследуемого объекта.

В дальнейшем планируется автоматизировать этап ручного отбора кластеров изображений, соответствующих пространственной структуре исследуемого объекта, за счет встраивания пакета Cinderella [59] в Cryo-EM-контейнер. В пакете Cinderella для отбора используется многослойная сверточная нейронная сеть. Она обучена отличать изображения биологических частиц от примесей и артефактов. В этом пакете обучение проводилось на 20 размеченных наборах данных из различных SPA Cryo-EM-экспериментов. Для адаптации нейронной сети к особенностям SPI Cryo-EM-эксперимента, данные которого обрабатываются, можно провести обучение (или дообучение) и на других наборах данных. Это предмет дальнейших исследований.
Обработка данных эксперимента TvNiR была повторно проведена через запуск CWL-композитного задания с использованием созданного Cryo-EM-контейнера в среде Docker. Результаты восстановления пространственной структуры совпадают с результатами оригинальной обработки, опубликованными в [19]. Некоторые различия присутствуют только в результатах этапа кластеризации, что обусловлено вероятностной природой используемого метода кластеризации на основе EM-алгоритма [29].
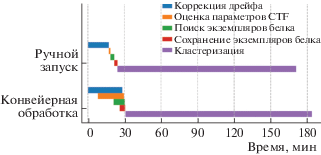
Отметим, что общее время выполнения обработки данных TvNiR в ручном режиме и с помощью разработанного контейнеризированного конвейера практически совпадает. Это подтверждает, что накладные расходы (временны́е) на работу контейнеризированных технологий незначительны.
Тестирование временных параметров проводили для части рабочего потока, которая включает в себя первые пять этапов, до первого этапа “Ручная выборка”. При конвейерной обработке разные стеки изображений из “сырых” данных обрабатываются независимо в параллельном режиме – всего таких стеков в проведенном тесте было сто. Выбор пяти первых этапов является наиболее репрезентативным с точки зрения оценки накладных расходов на работу промежуточного программного стека, реализующего описанные выше технологии контейнеризации. Временны́е затраты на выполнение этих пяти этапов обработки данных сравнивали для двух вариантов: ручной запуск каждого последующего этапа вычислений через аргументы командной строки, автоматическое выполнение CWL-композитного задания с помощью программного инструмента cwltool [16]. Последний инструмент запуска является эталонным для выполнения композитных заданий на языке CWL и используется в REANA для обработки CWL-сценариев. Так как измерения времени расчета по двум (описанным выше) вариантам расчетов проводили на одном сервере, использование cwltool позволило избежать затрат на передачу данных между управляющими и вычислительными серверами, которые присутствуют в REANA.
Измерения времени расчета для обоих вариантов проводили на сервере с двумя процессорами Intel Xeon E5-2680 v3, всего 24 ядра и 48 потоков, 128 Гб оперативной памяти, а также c тремя графическими ускорителями Nvidia Tesla K80. Использовали сетевую систему хранения Ceph 13.2.8, сетевое подключение основано на технологии Infiniband FDR. Были установлены библиотеки для параллельных вычислений MPI и OpenMP, которые автоматически использовались как в варианте CWL-композитного задания, так и при ручном запуске вычислений в Relion, что позволило максимально сократить время расчета. Измерения проводили по 5 раз для каждого случая, в результате получено среднее время вычислений. Расчет с использованием GPU применяли на этапах кластеризации изображений частицы в обоих вариантах тестирования.
В результате при ручном запуске время расчета составило 2 ч 51 мин, тогда как при конвейерной обработке потребовалось 3 ч 3 мин. На рис. 3 представлен временной график для отдельных этапов рабочего потока. При конвейерной обработке начальные этапы выполняются с перекрытием, разные ветви рабочего потока находятся на разных этапах обработки. Это приводит к частичной потере производительности, так как конвейер ждет завершения последней ветви. Суммарное время расчета при конвейерной обработке больше на 7% по сравнению с ручным запуском задач, основная потеря времени происходит при завершении ветвей конвейера перед этапом кластеризации.
4. РЕАЛИЗАЦИЯ КОНТЕЙНЕРИЗИРОВАННОГО КОНВЕЙЕРА ДЛЯ ОБРАБОТКИ ДАННЫХ SPI XFEL-ЭКСПЕРИМЕНТОВ НА ПРИКЛАДНОМ УРОВНЕ
В данном разделе описана реализация контейнеризированного конвейера для обработки данных SPI XFEL-экспериментов. Результаты представлены на примере данных эксперимента [60], проведенного на станции AMO [61, 62] лазера на свободных электронах LCLS в 2015 г. Данные размещены в открытом доступе в базе CXIDB [63] под номером 58.
В SPI-экспериментах на XFEL собираются 2D-изображения, полученные на детекторе после дифракции фемтосекундных рентгеновских импульсов на одиночных образцах исследуемого объекта, которые в момент попадания в них рентгеновского импульса находятся в разных ориентациях. Поэтому “сырые” данные в этих экспериментах представляют собой набор 2D-дифракционных изображений (дифракционных картин), являющихся сечениями 3D-образа исследуемого объекта в обратном пространстве. По сути 3D-образ является фурье-преобразованием пространственного распределения электронной плотности в исследуемом объекте. В определенном отношении восстановление 3D-образа по 2D-дифракционным изображениям является аналогом восстановления пространственной структуры в SPA Cryo-EM-экспериментах по множеству 2D-проекций идентичных образцов исследуемого объекта, находящихся в различных ориентациях при прохождении через них электронного пучка.
Обработка SPI XFEL-данных представляет собой последовательность следующих этапов:
1) фильтрация пустых изображений, т.е. без характерных признаков дифракционной картины;
2) определение центра 2D-дифракционных картин на изображениях;
3) оценка размера частиц на 2D-дифракционных изображениях;
4) фильтрация 2D-дифракционных изображений по размеру частиц и точности оценки их размера;
5) кластеризация отфильтрованных 2D-дифракционных изображений на заданное число групп (кластеров), каждая группа объединяет изображения со схожей дифракционной картиной;
6) ручная выборка групп изображений частиц с дифракционной картиной, относящейся к исследуемому объекту;
7) построение 3D-образа исследуемого объекта в обратном пространстве на основе выбранных групп (кластеров) 2D-изображений;
8) коррекция 3D-образа исследуемого объекта в обратном пространстве;
9) восстановления фаз и восстановление 3D-карты распределения электронной плотности в исследуемом объекте.
Для обработки SPI XFEL-данных использовали оригинальные программные пакеты SPI-Preprocess и SPI-EM-Classifier, разработанные авторами статьи. Этапы 1–4 реализованы в программе SPI-Preprocess, этап 2D-кластеризации и ручной выборки реализован в программе SPI-EM-Classifier. Для построения 3D-образа в обратном пространстве на основе дифракционных изображений использована программа Dragonfly [64]. Последующие этапы реализованы в виде рабочих скриптов на языках Python и MATLAB, разработанных авторами статьи. Их встраивали в разработанную контейнеризированную платформу (помещение в контейнеры и включение в соответствующее CWL-композитное задание) в процессе разработки.
На текущий момент CWL-композитное задание для контейнеризированной конвейерной обработки SPI XFEL-данных реализовано до шестого этапа “Ручная выборка”. При этом использовали два контейнера: контейнер с программой SPI-Preprocess и контейнер с программой SPI-EM-Classifier.
Так же, как в случае конвейерной обработки SPA Cryo-EM-данных, для SPI-экспериментов на XFEL существует возможность “естественной” параллелизации вычислений. “Сырые” данные можно разделить на несколько наборов 2D-дифракционных изображений, которые на первых шести этапах (до кластеризации отфильтрованных 2D-дифракционных изображений) обрабатываются независимо друг от друга. При выполнении CWL-композитного задания в среде Docker для каждого файла работает независимая ветвь конвейера.
На рис. 4 представлен рабочий поток выполнения отдельных этапов обработки SPI XFEL-данных в рамках разрабатываемого CWL-композитного задания. На схеме показано разбиение “сырых” данных на несколько поднаборов, которые обрабатываются параллельно. При этом две параллельные ветви показаны с расшифровкой выполняемых операций и с последующим сбором выходных наборов обработанных 2D-изображений в объединенный стек для выполнения дальнейших этапов обработки, начиная с этапа 2D-кластеризации.
ЗАКЛЮЧЕНИЕ
Представлен подход для организации конвейерной обработки экспериментальных данных Cryo-EM и XFEL с использованием современных ИТ-решений: технологий контейнеризации, оркестрации контейнеров и высокоуровневого языка описаний вычислительных заданий CWL. Такой подход представляет для пользователей ряд существенных преимуществ:
– технологии контейнеризации и микросервисная архитектура позволяют организовать работу прикладных программных пакетов, используемых для обработки данных, в виде независимых самодостаточных модулей с универсальными входными и выходными интерфейсами. Такой подход позволяет строить гибкую конвейерную систему обработки, в которой можно легко заменять часть модулей, не затрагивая остальные;
– технология оркестрации контейнеров обеспечивает эффективное использование различных суперкомпьютерных и облачных вычислительных инфраструктур, снимая с пользователя неспецифичные для него задачи по организации распределенных вычислений между узлами гетерогенных высокопроизводительных систем;
– прикладная логика обработки данных задается в виде компактного описания на языке высокого уровня CWL в формате прикладного композитного задания.
Предлагаемый контейнеризированный подход к организации конвейерной обработки экспериментальных данных позволяет отделить работу пользователя на прикладном уровне от разработок ИТ-специалистов на системном и промежуточном уровнях. Работа, связанная с обеспечением запуска прикладного ПО на доступных пользователю компьютерных ресурсах, в том числе с учетом конкретностей установленного на них системного и промежуточного ПО, остается на стороне ИТ-специалистов. Эту работу они выполняют на универсальном уровне без учета конкретностей прикладного ПО. Для этого они должны установить на компьютерных ресурсах ПО промежуточного уровня – Docker, Kubernetes, REANA и CWL. А (прикладному) пользователю остается только выполнить две подготовительные работы:
– поместить прикладные пакеты, используемые для обработки данных, в Docker контейнеры;
– записать прикладную логику конвейерной обработки данных в формате прикладного композитного задания на языке высокого уровня CW.
На сайте [56] приведены примеры разработок как для SPA Cryo-EM-, так и для SPI XFEL-приложений. В этих примерах приведены соответствующие программные коды и даны комментарии, которые помогут пользователям, не имеющим ИТ-компетенций.
Оставшиеся этапы в контейнеризированных конвейерах для обработки данных SPA/SPI-экспериментов, требующие ручного труда, будут автоматизированы за счет помещения в Docker-контейнеры соответствующих прикладных пакетов. Примером является пакет Cinderella [59], использующий сверточную нейронную сеть, который планируется поместить в Cryo-EM-контейнер, что позволит автоматизировать этап ручного отбора кластеров изображений, соответствующих пространственной структуре исследуемого объекта.
В дальнейшем будут проведены исследования в направлении использования естественного параллелизма при обработке больших наборов данных.
Важной частью работы будет развитие сайта проекта по развитию контейнеризированной платформы для конвейерной обработки данных SPA/SPI-экспериментов. На сайте будут размещены инсталляционные версии платформы для развертывания на различных компьютерных ресурсах по лицензиям свободно распространяемого ПО. Созданные Docker-контейнеры для SPA/SPI-прикладных модулей, а также прикладные CWL-композитные задания, разработанные для конкретных экспериментов, будут дополняться. Необходимость дополнений и новых разработок связана как с появлением новых, более эффективных прикладных пакетов, так и с активным развитием аппаратных составляющих Cryo-EM- и XFEL-экспериментов.
Работы по созданию контейнеризированной платформы для организации конвейерной обработки данных SPA/SPI-экспериментов на Cryo-EM и XFEL проведены при поддержке совместного Российского научного фонда (грант № 18-41-06001) и Helmholtz Associations Initiative Networking Fund (HRSF-0002).
Разработка и развертывание инфраструктуры и программных сервисов системного уровня для работы платформы и хранения данных проводились согласно тематическому плану НИОКР НИЦ “Курчатовский институт”.
Работа выполнена с использованием вычислительных ресурсов, предоставленных в рамках проекта № 1571 НИЦ Курчатовский институт “Создание цифровой платформы распределенного хранения, обработки и анализа научных данных на базе суперкомпьютерных и грид-технологий”.
Список литературы
Callaway E. // Nature News. 2015. V. 525. № 7568. P. 172. https://doi.org/10.1038/525172a
Altarelli M., Mancuso A.P. // Philos. Trans. R. Soc. B. 2014. V. 369. № 1647. P. 20130311. https://doi.org/10.1098/rstb.2013.0311
Bharat T.A.M., Russo C.J., Löwe J. et al. // Structure. 2015. V. 23. № 9. P. 1743. https://doi.org/10.1016/j.str.2015.06.026
Neutze R., Wouts R., van der Spoel D. et al. // Nature. 2000. V. 406. № 6797. P. 752. https://doi.org/10.1038/35021099
Gaffney K.J., Chapman H.N. // Science. 2007. V. 316. № 5830. P. 1444. https://doi.org/10.1126/science.1135923
Saxena A., Sun M., Ng A.Y. // IEEE Trans. Pattern Analysis Machine Intelligence. 2008. V. 31. № 5. P. 824. https://doi.org/10.1109/TPAMI.2008.132
Rezende D.J., Eslami S.M. Ali, Mohamed S. et al. // Advances in neural information processing systems. Curran Associates, Inc, 2016. P. 4996.
LeCun Y., Bengio Y. // Convolutional Networks for Images, Speech, and Time-Series. MIT Press, 1998. P. 255.
Krizhevsky A., Sutskever I., Hinton G.E. // Advances in Neural Information Processing Systems, Curran Associates, Inc, 2012. P. 1097.
Pichkur E., Baimukhametov T., Teslyuk A. et al. // J. Phys.: Conf. Ser. 2018. V. 955. P. 012005. https://doi.org/10.1088/1742-6596/955/1/012005
Bobkov S.A., Teslyuk A.B., Zolotarev S.I. et al. // Lobachevskii Journal of Mathematics. 2018. V. 39. № 9. P. 1170. https://doi.org/10.1134/S1995080218090093
Merkel D. // Linux J. 2014. № 239. P. 2.
https://kubernetes.io/
Nadareishvili I., Mitra R., McLarty M. et al. Microservice architecture: aligning principles, practices, and culture. O’Reilly Media, Inc, 2016.
Šimko T., Heinrich L., Hirvonsalo H. et al. // EPJ Web of Conferences. EDP Sciences, 2019. V. 214. P. 06034. https://doi.org/10.1051/epjconf/201921406034
Amstutz P., Crusoe M. R., Tijanić N. et al. // Common Workflow Language working group. 2016. https://doi.org/10.6084/m9.figshare.3115156.v2
Gatsogiannis C., Markl J. // J. Mol. Biol. 2009. V. 385. № 3. P. 963. https://doi.org/10.1016/j.jmb.2008.10.080
Tesliuk A., Bobkov S., Ilyin V. et al. // 2019 Ivannikov ISPRAS Open Conference (ISPRAS). IEEE Xplore. 2019. P. 67. https://doi.org/10.1109/ISPRAS47671.2019.00016
Баймухаметов T.Н., Чесноков Ю.M., Пичкур E.Б. и др. // Acta Naturae. 2018. Т. 10. № 3. С. 48. https://doi.org/10.32607/20758251-2018-10-3-48-56
Cheng Y. // Cell. 2015. V. 161. № 3. P. 450. https://doi.org/10.1016/j.cell.2015.03.049
Scheres S.H.W. // J. Structur. Biol. 2015. V. 189. № 2. P. 114. https://doi.org/10.1016/j.jsb.2014.11.010
Punjani A., Rubinstein J.L., Fleet D.J. et al. // Nature Methods. 2017. V. 14. № 3. P. 290. https://doi.org/10.1038/nmeth.4169
Plitzko J., Baumeister W.P. // Springer Handbook of Microscopy. Springer, Cham, 2019. P. 2-2. https://doi.org/10.1007/978-3-030-00069-1_4
https://www.ebi.ac.uk/pdbe/emdb/statistics_num_res.html/
Nwanochie E., Uversky V.N. // Int. J. Mol. Sci. 2019. V. 20. № 17. P. 4186. https://doi.org/10.3390/ijms20174186
Bai X.C., McMullan G., Scheres S.H.W. // Trends Biochem. Sci. 2015. V. 40. № 1. P. 49. https://doi.org/10.1016/j.tibs.2014.10.005
Mitra A.K., van Raaij M. // Acta Cryst. F. 2019. V. 75. № 1. P. 1. https://doi.org/10.1107/S2053230X18017806
Williams D.B., Carter C.B. Transmission Electron Microscopy. Springer, 2009. 768 p. https://doi.org/10.1007/978-0-387-76501-3_10
Scheres S.H.W., Valle M., Nuñez R. et al. // J. Mol. Biol. 2005. V. 348. № 1. P. 139. https://doi.org/10.1016/j.jmb.2005.02.031
Mastronarde D.N. // Microscopy Microanalysis. 2018. V. 24. № S1. P. 864. https://doi.org/10.1017/S1431927618004816
Zheng S.Q., Palovcak E., Armache J.-P. et al. // Nature Methods. 2017. V. 14. № 4. P. 331. https://doi.org/10.1038/nmeth.4193
Rohou A., Grigorieff N. // J. Struct. Biol. 2015. V. 192. № 2. P. 216. https://doi.org/10.1016/j.jsb.2015.08.008
Zhang K. // J. Struct. Biol. 2016. V. 193. № 1. P. 1. https://doi.org/10.1016/j.jsb.2015.11.003
Zhang K., Li M., Sun F. Gautomatch: an efficient and convenient gpu-based automatic particle selection program. https://www.mrc-lmb.cam.ac.uk/kzhang/ 2011.
Scheres S.H.W. // J. Struct. Biol. 2012. V. 180. № 3. P. 519. https://doi.org/10.1016/j.jsb.2012.09.006
Grant T., Rohou A., Grigorieff N. // Elife. 2018. V. 7. P. e35383. https://doi.org/10.7554/eLife.35383
Emma P., Akre R., Bionta A.R. et al. // Nature Photonics. 2010. V. 4. № 9. P. 641. https://doi.org/10.1038/nphoton.2010.176
Ishikawa T., Aoyagi H., Asaka T. et al. // Nature Photonics. 2012. V. 6. № 8. P. 540. https://doi.org/10.1038/nphoton.2012.141
Seibert M.M., Ekeberg T., Maia F.R.N.C. et al. // Nature. 2011. V. 470. № 7332. P. 78. https://doi.org/10.1038/nature09748
Hantke M.F., Haase D., Maia F.R.N. C et al. // Nature Photonics. 2014. V. 8. № 12. P. 943. https://doi.org/10.1038/nphoton.2014.270
van der Schot G., Svenda M., Maia F.R.N.C. et al. // Nature Commun. 2015. V. 6. № 1. P. 1. https://doi.org/10.1038/ncomms6704
Ekeberg T., Svenda M., Abergel C. et al. // Phys. Rev. Lett. 2015. V. 114. № 9. P. 098102. https://doi.org/10.1103/PhysRevLett.114.098102
Aquila A., Barty A., Bostedt C. et al. // Struct. Dynamics. 2015. V. 2. № 4. P. 041701. https://doi.org/10.1063/1.4918726
Kurta R.P., Donatelli J.J., Yoon C.H. et al. // Phys. Rev. Lett. 2017. V. 119. № 15. P. 158102. https://doi.org/10.1103/PhysRevLett.119.158102
Rose M., Bobkov S., Ayyer K. et al. // IUCrJ. 2018. V. 5. №. 6. P. 727. https://doi.org/10.1107/S205225251801120X
Shi Y., Yin K., Tai X. et al. // IUCrJ. 2019. V. 6. № 2. P. 331. https://doi.org/10.1107/S2052252519001854
Assalauova D., Kim Y.Y., Bobkov S. et al. // IUCrJ. 2020. V. 7. № in press
Sobolev E., Zolotarev S., Giewekemeyer K. et al. // Commun. Phys. 2020. V. 3. Article number 97. https://doi.org/10.1038/s42005-020-0362-y
https://linuxcontainers.org/lxc/introduction/
https://cri-o.io/
https://docs.docker.com/engine/reference/builder/
https://opencontainers.org/about/overview/
https://hub.docker.com/
https://docs.docker.com/get-started/swarm-deploy/
https://rancher.com/
https://bio1.grid.kiae.ru/sw/workflows/
Yu J., Buyya R. // ACM Sigmod Record. 2005. V. 34. № 3. P. 44. https://doi.org/10.1007/s10723-005-9010-8
https://xpra.org/
Wagner T., Lusnig L., Pospich S. et al. // bioRxiv. 2020. https://doi.org/10.1101/2020.02.28.969196
Reddy H.K.N., Yoon C.H., Aquila A. et al. // Scientific Data. 2017. V. 4. P. 170079. https://doi.org/10.1038/sdata.2017.79
Bozek J.D. // Europ. Phys. J. Special Topics. 2009. V. 169. № 1. P. 129. https://doi.org/10.1140/epjst/e2009-00982-y
Ferguson K.R., Bucher M., Bozek J.D. et al. // J. Synchrotron Radiation. 2015. V. 22. № 3. P. 492. https://doi.org/10.1107/S1600577515004646
Maia F.R.N.C. // Nature Methods. 2012. V. 9. № 9. P. 854. https://doi.org/10.1038/nmeth.2110
Ayyer K., Lan T.-Y., Elser V. et al. // J. Appl. Cryst. 2016. V. 49. № 4. P. 1320. https://doi.org/10.1107/S1600576716008165
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография