Кристаллография, 2020, T. 65, № 6, стр. 886-895
Влияние состава буфера на конформационную подвижность N-концевых фрагментов DPS и характер взаимодействия с ДНК. Исследование методом малоуглового рентгеновского рассеяния
Е. Ю. Сошинская 1, Л. А. Дадинова 1, А. А. Можаев 1, 2, Э. В. Штыкова 1, *
1 Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника” РАН
Москва, Россия
2 Институт биоорганической химии РАН им. М.М. Шемякина и Ю.А. Овчинникова РАН
Москва, Россия
* E-mail: shtykova@crys.ras.ru
Поступила в редакцию 27.04.2020
После доработки 27.04.2020
Принята к публикации 20.05.2020
Аннотация
ДНК-связывающий белок Dps играет ключевую роль в формировании в живых бактериальных клетках кристаллических комплексов Dps–ДНК, что позволяет бактериям выживать в условиях стресса, при воздействии различных неблагоприятных факторов. Такие геномзащитные механизмы способны приводить к возникновению резистентности бактерий к антибиотикам и другим лекарственным препаратам. Выяснение фундаментальных биохимических, генетических и структурных основ резистентности имеет первостепенное значение для разработки стратегий, направленных на ограничение возникновения и распространения устойчивости бактерий, а также разработки инновационных терапевтических подходов. С помощью метода малоуглового рентгеновского рассеяния исследованы конформационные характеристики Dps и его N-концевых фрагментов, определяющие характер взаимодействия этого белка с ДНК в растворе.
ВВЕДЕНИЕ
Способность адаптироваться к различным видам стресса у живых организмов проявляется прежде всего на молекулярном уровне. Хорошо известно, что в процессе голодания или при других неблагоприятных условиях бактерии продуцируют большое количество белка Dps (DNA-binding Protein of Starvation), который, связываясь с ДНК, эффективно защищает ее от повреждения как in vitro, так и in vivo [1, 2]. При этом образуется устойчивый кристаллический комплекс Dps–ДНК [3]. Важным следствием такой защитной реакции является возникновение бактериальной резистентности к антибиотикам и другим лекарственным средствам. Преодоление устойчивости различных патогенов к соответствующим медицинским препаратам чрезвычайно важно для современной медицины, поэтому исследованию структуры и условиям формирования биокристаллов Dps–ДНК уделяется большое внимание [4–7].
Поскольку Dps играет ключевую роль в процессе биокристаллизации, его структура, в том числе на атомном уровне, хорошо изучена [8–11]. Известно, что Dps представляет собой сферический додекамер, гомологичный по структуре ферритину. Существует гипотеза, что защитная функция Dps связана с его способностью связывать ионы железа [8], но молекулярные и структурные механизмы таких биохимических реакций все еще полностью не выяснены.
Первичная структура мономера Dps представляет собой полипептидную цепь из 167 аминокислотных остатков с молекулярной массой 18.7 кДа и содержит высококонсервативную последовательность, состоящую из четырех α-спиралей [8]. Нативная молекула Dps собрана из 12 идентичных субъединиц с суммарной молекулярной массой 224.4 кДа, обладает симметрией типа 23, может накапливать до 450 ионов железа во внутренней полости и выполняет ряд важных биологических функций в бактериальной клетке [9]. Было показано, что in vitro Dps также может существовать в виде стабильного тримера [10, 11], который, однако, трансформируется в додекамер в процессе инкубации в течение 12 ч при 37°С. Эти две олигомерные формы Dps различаются по своим свойствам взаимодействия с ДНК. Dps в виде додекамера способен связывать ДНК и образовывать с ней большие кристаллические массивы, тогда как тример не может этого сделать [11].
Додекамер Dps, экспрессированный из Mycobacterium smegmatis, обладает способностью обратимо диссоциировать на димеры при рН выше 7.5 и ниже 6.0 [12]. На основании структурного анализа этого белка был сделан вывод о том, что различные стадии диссоциации резко изменяют способность Dps к связыванию с ДНК: димеры имеют низкую эффективность формирования защитного комплекса из-за разрушения внутренней полости додекамера, где железо осаждается и минерализуется после окисления [12].
Помимо олигомерного состояния важное значение для взаимодействия Dps с ДНК имеют его N- или C-концевые последовательности. Так, у Dps из Mycobacterium smegmatis разупорядоченные С-терминалы содержат 26 аминокислотных остатков с тремя положительно заряженными остатками лизина (Lys172, Lys177 и Lys181), которые, предположительно, участвуют в связывании с ДНК [13]. Два типа Dps из Lactococcus lactis имеют спиральные N-концевые последовательности, состоящие из 26 аминокислотных остатков, удаление которых ухудшает способность образования кристаллического комплекса Dps–ДНК [14].
Экспрессированные из бактерии Deinococcus radiodurans два вида Dps (DrDps1 и DrDps2) имеют более длинные N-концевые домены по сравнению с любыми другими членами семейства Dps, изученными до сих пор. Они содержат 54 и 41 остаток соответственно. Отсечение первых 50 аминокислотных остатков N-концевой области DrDps1 практически полностью подавляет способность этого белка взаимодействовать с ДНК, указывая на то, что эти области играют решающую роль в образовании со-кристаллов Dps–ДНК [15–17]. При этом N-терминал DrDps1 можно условно разделить на два региона: положительно заряженную область (остатки 1–29), которая содержит шесть остатков лизина (Lys3, Lys4, Lys7, Lys13, Lys15 и Lys16) и один аргинин (Arg28), и область (остатки 30–55), в которой находится сайт связывания с металлами с мотивом Asp36x2His39x10His50x4Glu55, расположенным на внешней поверхности додекамера. Разрушение этого сайта влияет на самосборку додекамера, а также снижает способность связывания белка с ДНК [17].
Исследование методом атомно-силовой микроскопии структуры Dps из Escherichia coli также показало важность N-концевых областей при взаимодействии белка с ДНК. N-терминал Dps из этой бактерии содержит 20 аминокислотных остатков, четыре из которых имеют положительный заряд: три лизина (Lys5, Lys8 и Lys10) и один аргинин (Arg18), и усечение первых 18 аминокислот предотвращает комплексообразование с ДНК [18].
Малоизученным аспектом формирования кристаллических комплексов Dps–ДНК остается влияние металлических бивалентных ионов. В [5] было сделано предположение, что додекамеры Dps не могут связываться с ДНК напрямую и комплексообразование с ДНК происходит опосредовано через ионные мостики, образованные Mg2+ в определенном диапазоне концентраций этих катионов, и их присутствие имеет решающее значение для образования со-кристаллов Dps–ДНК. В работе также показано влияние ЭДТА (этилендиаминтетрауксусной кислоты) на комплексообразование. Аналогичные выводы сделаны в [18], где представлены доказательства, указывающие на зависимость формирования со-кристаллов Dps–ДНК от наличия в растворе Mg2+, Fe2+, хелатирующих агентов, а также от рН и ионной силы раствора [18].
В настоящее время основной гипотезой является предположение, что главную роль в формировании комплекса играют именно N-концевые области, богатые лизином. В соответствии с этой гипотезой члены семейства Dps, которые не обладают положительно заряженным N-терминалом, не могут связываться с ДНК. Это относится, например, к так называемым Listeria innocua ferritin [9], Dlp-1 иDlp-2 из Bacillus anthracis [19] и HP-NAP из Helicobacter pylori [20].
Несмотря на интенсивные исследования Dps из разных организмов, кристаллическая структура была определена только для додекамерного ядра этого белка. Структура атомного разрешения N-концевых областей остается неизвестной, предположительно, из-за гибкости и разупорядочения этих фрагментов и, следовательно, невозможности их кристаллизации. Исследования в растворе методом малоуглового рентгеновского рассеяния (МУРР) проводились только для Dps, выделенного из Porphyromonas gingivalis, с целью изучения формы сферического додекамера [18] и для двух видов Dps из Deinococcus radiodurans [15], где были подробно изучены конформации N-концевых областей белка и показано их разупорядочение и роль во взаимодействии с ДНК.
В [6, 7] с помощью МУРР и крио-электронной микроскопии были определены типы кристаллической упаковки со-кристаллов Dps-ДНК в растворе в зависимости от состава буфера.
В данной работе поставлена задача с помощью МУРР рассмотреть взаимосвязь основных факторов, определяющих формирование со-кристаллов Dps–ДНК в растворе. Таким образом, исследование посвящено изучению влияния конформационной подвижности N-концевых областей Dps из Escherichia coli на характер взаимодействия белка с ДНК в присутствии бивалентных ионов металлов при различных значениях рН и ионной силы раствора, а также воздействия ЭДТА в качестве хелатирующего агента. Выбор ЭДТА связан со способностью этого вещества образовывать растворимые комплексы с широким спектром металлов, предотвращая их взаимодействие с металлсвязывающими сайтами белка [21]. Полученные данные будут полезны при дальнейшем изучении структуры и свойств защитного комплекса Dps–ДНК как in vitro, так и в живых клетках.
МАТЕРИАЛЫ И МЕТОДЫ
Подготовка образца ДНК. В качестве образца ДНК использовали кольцевой вектор pcDNA-hIRR-GFP 9900 пар оснований [7, 22]. Выделение вектора проводили с использованием диоксида кремния S5631 (Sigma-Aldrich, США) в соответствии с протоколом, описанным в [23]. После выделения вектор осаждали изопропанолом, промывали 70%-ным этанолом, сушили на воздухе и растворяли в воде milliQ (Merck Millipore, Франция). Концентрацию ДНК определяли с использованием спектрофотометра ND-1000 (NanoDrop Technologies Inc., США).
Подготовка образца DPS. Экспрессию и очистку Dps проводили по ранее разработанной методике [6]. Фрагмент ДНК, кодирующий ген Dps MSTAKLVKSKATNLLYTRNDVSDSEKKATVELLNRQVIQFIDLSLITKQAHWNMRGANFIAVHEMLDGFRTALIDHLDTMAERAVQLGGVALGTTQVINSKTPLKSYPLDIHNVQDHLKELADRYAIVANDVRKAIGEAKDDDTADILTAASRDLDKFLWFIESNIE (UniProtKB – P0ABT2 (DPS_ECOLI)), был получен с помощью ПЦР (полимеразной цепной реакции) амплификации ДНК штамма E. coli K12 MG1655 с использованием 5'-GATATGAACATATGAGTACCGCTAAATTAG-3'- и 5'-TATAAGCTTATTCGATGTTAGACTCGATAAAC-3'-олигонуклеотидов. Ген Dps E. coli был клонирован в вектор pET-22b(+) по сайтам рестрикции NdeI и HindIII. Экспрессию проводили в клетках E. coli BL21-Gold (DE3) при выращивании на среде LB (Lysogeny Broth – культуральная жидкость) в присутствии ампициллина (150 мкг/мл) при 37°С. Транскрипцию рекомбинантного гена индуцировали 0.5 мМ IPTG (изопропил-β-D-1-тиогалактопиранозид) при OD600 0.8, наработку белка проводили в течение четырех часов после индукции.
Затем белок чистили с использованием ионообменной хроматографии на колонке с DEAE-сефарозой FF (GE Healthcare, США), уравновешенной 20 мМ Tris-HCl, 100 мМ NaCl, pH 7.5. Фракции в проскоке с колонки, не связавшиеся с сорбентом, собирали, так как они содержат большую часть белка Dps, свободного от связанной ДНК [24]. Собранные фракции тестировали на наличие примесей ДНК с помощью измерения соотношения OD260/OD280, оно обычно составляло около 0.7. Далее белок концентрировали на центрифужной пробирке Amicon с порогом отсечения молекулярной массы 10 кДа и диализовали в буфере для хранения, содержащем 10 мМ Tris-HCl, рН 7.5, 100 мМ NaCl, 0.5 мМ ЭДТА (буфер I). Чистоту белка контролировали электрофорезом в полиакриламидном геле. Очищенный Dps разделяли на аликвоты и хранили при –20°С. Непосредственно перед измерениями белок размораживали и использовали для дальнейшего изучения либо в исходном буфере (буфер I), либо диализовали в двух разных буферах: 50 мМ Tris-HCl, 50 мМ NaCl, 0.5 мМ EDTA, pH 8.0 (буфер II) и 20 мМ Tris-HCl, 100 мМ NaCl, pH 7.5 (буфер III).
Эксперимент и анализ данных МУРР. Исследования структуры белка с помощью МУРР проводили на синхротроне PetraIII (DESY, Гамбург) на линии P12, оснащенной оборудованием для автоматической смены образцов и двумерным детектором Pilatus2M (фирма DECTRIS, Швейцария). Интенсивность рассеяния I(s) измеряли в области значений волновых векторов 0.08 < s < 7 нм–1, где s = (4πsin θ)/λ – вектор рассеяния, 2θ – угол рассеяния, λ = 0.124 нм – длина волны рассеяния [25]. Для анализа и модельных расчетов использовали наиболее информативный интервал волновых векторов 0.08 < s < 2.4 нм–1.
Для каждого образца было снято по 50 экспериментальных кривых рассеяния для контроля возможных радиационных повреждений. Радиационного повреждения обнаружено не было.
Растворы белка Dps и смесей белка с ДНК в разных буферах измеряли при температуре 10°C.
Обработку полученных данных проводили с использованием стандартных процедур и программного пакета ATSAS [26]. Первичная обработка кривых была проведена с использованием программы PRIMUS [27]. C ее помощью проводили усреднение экспериментальных кривых рассеяния от образцов и соответствующего буфера с последующим вычитанием рассеяния буфера из кривых МУРР образцов.
Радиусы инерции (Rg) определяли по начальному участку кривой рассеяния в области самых малых значений s с помощью формулы
справедливой в области (sRg) < 1.3. Из наклона прямолинейного участка графика Гинье – ln I(s) от s2 – определяли интенсивность рассеяния в нулевой угол I(0), которая пропорциональна квадрату молекулярной массы рассеивающей частицы, и радиусы инерции.Молекулярные массы (ММ) рассчитывали по данным МУРР двумя методами: с помощью подхода Байеса (MMBayesian) [28] и из значения объема Порода VP (ММP) [28], используя эмпирическое соотношение между VP и ММP, которое для белков равно в среднем 1.65 [26].
Для построения функций парных расстояний p(r), которые необходимы для восстановления формы белка в растворе по данным МУРР, использовали компьютерную программу GNOM [30]. Функции парных расстояний определяли с помощью косвенного фурье-преобразования интенсивности рассеяния в соответствии с интегральным уравнением
(2)
$p(r) = \frac{1}{{2{{{\pi }}^{2}}}}\mathop \smallint \limits_0^\infty \,srI(s){\text{sin}}(sr)ds,$Ab initio-метод восстановления формы низкого разрешения белка Dps был реализован с помощью программы DAMMIN [31], которая использует алгоритм имитации отжига путем минимизации невязки χ2 с экспериментальными данными
(3)
${{{\chi }}^{2}} = \frac{1}{{N - 1}}\mathop \sum \limits_j {{\left[ {\frac{{{{I}_{{{\text{exp}}}}}({{s}_{j}}) - c{{I}_{{{\text{calc}}}}}({{s}_{j}})}}{{{\sigma }({{s}_{j}})}}} \right]}^{2}},$Для восстановления структуры белка и его гибких N-терминалов, которые отсутствуют в кристаллографической структуре, применяли гибридный метод (программа CORAL [32]), сочетающий в себе ab initio-алгоритм и моделирование жесткими телами. Программа использует имеющиеся в Protein Data Bank структуры высокого разрешения, теоретическая интенсивность которых рассчитывается программой CRYSOL [33], и достраивает недостающие фрагменты макромолекул, используя ab initio-процедуру. При этом недостающие аминокислоты моделируются с помощью виртуальных остатков, свернутых в белковоподобную цепь так, чтобы рассеяние от цельной структуры соответствовало экспериментальному рассеянию, минимизируя невязку χ2 (уравнение (3)). Для гибридного моделирования использовали структуру атомного разрешения белка Dps PDB ID: 1DPS.
Неоднозначность полученных моделей была проанализирована с помощью программы AMBIMETER [34].
Вспомогательная программа GEN_DAM_body [V.V. Volkov, не опубликовано] программного комплекса ATSAS [26] позволяет строить простые тела (шар, сфера, цилиндр) различных размеров и плотностей, состоящие из сферических элементов – так называемые “шариковые модели”.
Для анализа объемной доли свободных Dps и ДНК в растворе использовали программу OLIGOMER [27]. Для определения объемных фракций ${{{v}}_{k}}$ каждой компоненты смеси программа находит линейную комбинацию интенсивностей рассеяния от каждой компоненты Ik(s). В этом случае интенсивность рассеяния I(s) описывается уравнением
OLIGOMER использует алгоритм неотрицательных линейных коэффициентов для минимизации расхождения χ2 между предсказанной кривой рассеяния от смеси и экспериментальными данными МУРР.Модели белков, полученные различными способами, проанализированы с помощью программы SUPCOMB [35], чтобы определить разницу в их пространственной организации и указать нормализованное пространственное отклонение (NSD). Как правило, значение NSD, близкое к 1.0, показывает, что модели схожи.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Влияние состава буфера и pH на тип кристаллической упаковки комплекса Dps–ДНК показано в [6, 7]. Как исходный буфер, используемый при получении белка Dps (здесь буфер I), так и буфер с более щелочным рН (здесь буфер II) имели в своем составе ЭДТА в качестве хелатирующего агента. В результате в растворе сформировались со-кристаллы Dps–ДНК, что отразилось в появлении на кривых малоуглового рассеяния серии Брэгговских пиков (рис. 1а, кривые 1 и 2). Расчеты показали, что при использовании буфера I с pH 7.5 образовалась триклинная решетка (рис. 1а, кривая 1), а в буфере II с pH 8.0 и более высокой ионной силой (рис. 1а, кривая 2) – кубическая. Несмотря на обнаруженный полиморфизм комплекса, основным элементом, определяющим структуру со-кристаллов, являлся белок Dps. Именно он формировал 3D-матрицу, в которой ДНК благодаря своей гибкости располагалась в свободном пространстве между молекулами белка. Роль ЭДТА состояла в создании прочных комплексов с двухвалентными катионами металлов, т.е. в свободном состоянии эти катионы в растворе отсутствовали. Поэтому в настоящей работе исследована возможность формирования кристаллического комплекса Dps–ДНК без ЭДТА, а также влияние на стабильность и существование этого комплекса при наличии в растворе свободных катионов Fe2+ и Mg2+. Выбор именно этих катионов обусловлен предположением, что, во-первых, защитная активность Dps связана с его способностью связывать ионы железа [8], а во-вторых, тем, что, вероятно, в процессе формирования комплекса додекамеры Dps связываются с ДНК не напрямую, а опосредованно через ионные мостики, образованные Mg2+ [5]. Отметим, что исходные растворы Dps и ДНК не содержали эти металлы, как не содержали их и использованные в данной работе буферы I и II. Тем не менее анализ кривых МУРР объективно демонстрирует формирование кристаллических структур (рис. 1а, кривые 1, 2), что противоречит упомянутым выше литературным данным.
Рис. 1.
Экспериментальные кривые рассеяния от смеси Dps с ДНК в исходных буферах I (1), II (2), III (3), буфере II + + FeSO4 (4) и буфере II + MgCl2 (5), кривая рассеяния от раствора Dps в буфере II с добавлением FeSO4 (6) (а). Сравнение экспериментальных кривых рассеяния от белка Dps при трех концентрациях в буферах II (1) и III (2) (б). Графики Кратки для растворов белка в буферах II (красные точки) и III (зеленые точки) соответственно (в). Графики Гинье для растворов белка в буферах II (1, красные точки) и III (2, зеленые точки) (г). Цветные рисунки представлены в электронной версии журнала.
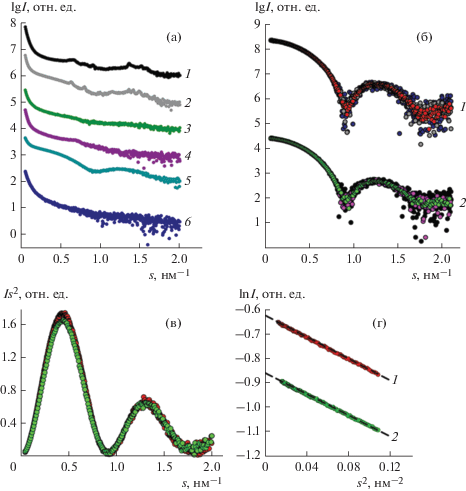
Как видно из рис. 1а (кривая 3), при использовании буфера III (20 мМ Tris-HCl, 100 мМ NaCl, pH 7.5), в составе которого отсутствует ЭДТА, кристаллический комплекс Dps–ДНК не образуется: на кривой рассеяния отсутствуют Брэгговские пики. Однако это не означает, что не образуется некристаллический комплекс. В отсутствие ЭДТА при сливании растворов белка и ДНК в соотношении одна молекула белка на 60 пар оснований ДНК, найденном в [6], происходит помутнение раствора, что явно свидетельствует о взаимодействии этих двух компонентов. Кривая рассеяния в данном случае (рис. 1а, кривая 3) характерна для неупорядоченных соединений с высокой степенью полидисперсности. То есть без ЭДТА возможно формирование некристаллического (неупорядоченного) комплекса Dps–ДНК и/или агрегация компонентов.
Поскольку предполагается, что катионы Fe2+ и Mg2+ играют существенную роль при формировании со-кристаллов Dps–ДНК, следующим шагом работы было изучение их влияния на уже сформированные кристаллические комплексы. На рис. 1а (кривые 4, 5) представлены кривые МУРР после добавления к кристаллическим комплексам Dps–ДНК в растворе (как в буфере I, так и в буфере II) бивалентных Fe2+- и Mg2+-катионов в виде 10 мМ солей FeSO4 или MgCl2 соответственно. Как видно из рисунка, вне зависимости от исходной упаковки кристаллических комплексов характеристические Брэгговские пики исчезают, т.е. наличие в растворе свободных бивалентных ионов металлов приводит к разрушению кристаллических решеток. Однако разрушение со-кристаллов происходит по-разному. В случае с Fe2+ на кривой рассеяния наблюдается единственный широкий максимум на s = 9.3–9.6 нм–1, что примерно соответствует первому максимуму от триклинной упаковки и размеру Dps, т.е. кристаллическая решетка практически полностью разрушается и имеется небольшое количество упорядоченных молекул Dps в сочетании с неупорядоченными агрегатами. Хотя свободные катионы Mg2+ также практически полностью разрушают кристаллический комплекс Dps–ДНК, в растворе отчетливо прослеживается форм-фактор белка, существующего независимо от ДНК. В этом случае логично предположить, что кривая 5 на рис. 1а может представлять собой сумму рассеяния невзаимодействующих друг с другом Dps и ДНК. С помощью программы OLIGOMER [27] показано, что это предположение верно: в растворе, содержащем свободные катионы Mg2+, присутствуют около 47 об. % ДНК, 52 об. % Dps и незначительное количество их комплексов.
Таким образом, исследование показывает, что со-кристаллизация Dps–ДНК не только не требует присутствия в растворе Fe2+- и Mg2+-катионов, но и образование кристаллических комплексов может происходить исключительно в их отсутствие, гарантией чего является наличие в растворе ЭДТА в нужной концентрации, которая прочно связывает положительно заряженные ионы металлов. Избыток катионов приводит к разрушению комплексов.
Поскольку белок Dps играет ключевую роль в формировании в живых бактериальных клетках кристаллических комплексов Dps–ДНК, необходимо выяснить особенности его структуры, при которых возможно формирование таких со-кристаллов.
Кривые МУРР белка Dps были измерены в растворе в присутствии ЭДТА (буферы I и II) и в отсутствие этого компонента (в буфере III). Для исключения влияния межчастичной интерференции на интерпретацию данных МУРР измерения были проведены при трех разных концентрациях белка в каждом буфере в диапазоне от 0.9 до 3.5 мг/мл (рис. 1б). Поскольку в указанном интервале не наблюдалось концентрационной зависимости, то для дальнейшей обработки и интерпретации данных МУРР были выбраны кривые с наибольшей концентрацией, т.е. 3.5 мг/мл в буферах с ЭДТА и 3.4 мг/мл в буфере без него, так как эти кривые являются наименее шумящими. Профили МУРР для Dps в разных буферах очень похожи (рис. 2а, 2г). Графики Кратки, показанные на рис. 1в, также демонстрируют почти полное совпадение. Они имеют характерный колоколообразный вид, что указывает на компактность и структурированность белка с небольшим количеством разупорядоченных, гибких областей. Поскольку предполагаемое дополнительное разупорядочение поверхностных областей Dps при добавлении в раствор ЭДТА может относиться только к N-концевым фрагментам белка, электронная плотность такого разупорядоченного поверхностного слоя будет незначительна и чувствительность графиков Кратки недостаточна для обнаружения этого явления.
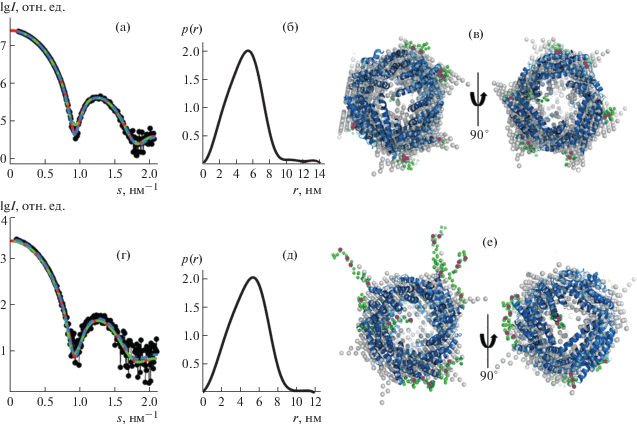
Рис. 2.
Сравнение экспериментальных кривых рассеяния от белка Dps в буферах II (а) и III (г) (черные точки) и теоретических кривых, рассчитанных по функции парных расстояний (сплошные красные линии), DAMMIN-модели (сплошные зеленые линии), CORAL-модели (пунктирные голубые линии). Функции парных расстояний для белка Dps в буферах II и III (б, д). Наложение моделей DAMMIN (серые шарики) и CORAL (голубые спирали и зеленые шарики) белка в буфере II и III (в, е) в двух ориентациях. Красными шариками на моделях CORAL отмечены Lys5, Lys8, Lys10 и Arg18.
Однако анализ графиков в координатах Гинье показывает небольшое, но детектируемое увеличение Rg при добавлении в буфер ЭДТА (рис. 1г, табл. 1). То есть общий объем макромолекулы несколько увеличивается и вероятнее всего за счет развертывания находящихся на поверхности белка N-концевых доменов.
Таблица 1.
Макромолекулярные характеристики белка Dps во II и III буферах
| Образец | Rg, нм | VP, нм 3 | Dmax, нм | MMPorod, кДа | MMBayesian, кДа |
|---|---|---|---|---|---|
| Dps в буфере II | 3.89 ± 0.1 | 317 ± 31 | 14 | 224 ± 20 | 208 |
| Dps в буфере III | 3.81 ± 0.1 | 292 ± 30 | 12 | 192 ± 20 | 186 |
Значения ММ, определенные по экспериментальным кривым рассеяния, исходя из значения Породовского объема [29] и обобщенного Байесовского подхода [28], совпадают для двух растворов в пределах погрешности. Их среднее значение составляет 203 кДа. Это соответствует ММ додекамера (табл. 1) и в пределах погрешности совпадает с теоретическим значением ММ для Dps, рассчитанной по аминокислотной последовательности мономера, умноженной на 12, т.е. равной 224 кДа.
Для анализа различий в структуре белка в разных буферах были построены функции парных расстояний с помощью программы GNOM [30]. Как видно из рис. 2б, 2д, максимальные размеры белка Dmax, которые определяются из условия p(r) = 0 при r > Dmax, различны и составляют 14 нм в буфере I или II и 12 нм в буфере III в отсутствие ЭДТА. Причем функция парных расстояний в области, близкой к максимальному размеру, демонстрирует низкую интенсивность, что соответствует низкой электронной плотности на периферии белка и объясняет невозможность с помощью графика Кратки выявить различия в структуре Dps с предположительно более развернутыми N-концевыми доменами.
Для восстановления формы низкого разрешения образца была использована программа DAMMIN [31], основанная на алгоритме имитации отжига, в пределах сферы, диаметр которой равен Dmax, найденному по функции парных расстояний. Поскольку образец в растворе существует в виде додекамера, то для ab initio-восстановления формы была применена симметрия P23. Результаты восстановления представлены на рис. 2в, 2е серыми шариками. Коэффициенты невязки χ2 с экспериментальными данными, равные 1.9 для белка в буфере I или II и 2.4 для белка в буфере III, свидетельствуют о хорошем соответствии экспериментальных кривых и кривых рассеяния от полученных форм низкого разрешения (рис. 2а, 2г).
Как видно из рис. 2в, 2е, образцы содержат полость в центре додекамера, что соответствует известной кристаллической структуре Dps, а также выступающие части, которые, предположительно, относятся к N-концевым областям белка. В буфере без ЭДТА эти выступающие части заметно меньшей длины, чем в присутствии хелатирующего агента.
Поскольку решение обратной задачи по восстановлению трехмерных форм по одномерной кривой рассеяния является неоднозначным, был проведен анализ неоднозначности полученных моделей с помощью программы AMBIMETER [34]. Анализ показал, что найденные модели имеют индекс неоднозначности меньше единицы. Данный результат указывает на практически единственное решение задачи восстановления.
Для определения структуры N-концевых фрагментов Dps в зависимости от наличия в растворе хелатирующего агента использовали гибридный подход, реализованный в программе CORAL [16]. Поскольку предполагается, что взаимодействие с отрицательно заряженными основаниями ДНК происходит через расположенные в N-концевых областях положительно заряженные лизины (Lys5, Lys8 и Lys10) и аргинин (Arg18), пространственная локализация и доступность этих аминокислот для контакта с ДНК очень важна. Для моделирования структуры полноразмерного белка использовали известные атомные координаты аминокислот додекамера Dps (PDB ID: 1DPS), а отсутствующие в кристалле первые 22 аминокислотных остатка, относящиеся к N-терминалам белка, были достроены с помощью ab initio-алгоритма. На рис. 2в, 2е представлены полученные модели, которые показывают, что в буфере II, т.е. в присутствии ЭДТА, N-терминалы белка свободно простираются в растворе и Lys5, Lys8, Lys10 и Arg18, отмеченные красными шариками, являются доступными для взаимодействия с ДНК. В то же время в буфере III, не содержащем хелатирующий агент, эти аминокислоты плотно прилегают к поверхности белка. Невязка χ2 для полученных решений равна 1.1 для Dps в буфере II и 1.6 в буфере III без ЭДТА, что свидетельствует о хорошем соответствии рассеяния от моделей с экспериментальными данными.
Таким образом, два независимых подхода к моделированию структуры Dps демонстрируют схожие результаты: наличие хелатирующего агента в растворе приводит к развертыванию N-концевых терминалов и доступности ключевых положительно заряженных аминокислот, трех лизинов и аргинина, необходимых для контакта с ДНК, которая несет множество отрицательных зарядов, так как каждый фосфатный остаток этой макромолекулы содержит ионизированную гидроксильную группу. Количественно взаимное соответствие результатов моделирования структуры двумя методами было определено с помощью программы SUPCOMB [35]. Нормализованное пространственное отклонение NSD составило 1.6 для моделей белка в буфере II и 1.8 в буфере III. То есть полученные разными способами модели хорошо совпадают, что видно из рис. 2в, 2е.
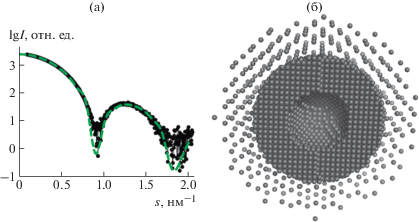
Еще один метод, который может подтвердить наличие разупорядоченных N-концов Dps, включает в себя использование программы GEN_DAM_body, позволяющей моделировать объекты различной формы и плотности с помощью простых геометрических тел. Для этого были построены два полых, вложенных друг в друга шара различных радиусов, соответствующих реальным размерам Dps и условные электронные плотности которых различаются в 10 раз (рис. 3б). Предполагается, что менее плотный полый шар моделирует поверхностный слой белка с разупорядоченными и редко расположенными в пространстве N-концевыми доменами. От данной геометрической модели с помощью программы CRYSOL [26] была рассчитана теоретическая кривая рассеяния и сравнена с экспериментальной кривой на рис. 3а. Как видно из рисунка, геометрическая модель хорошо описывает экспериментальные данные для раствора Dps, содержащего ЭДТА. Таким образом, еще один независимый метод моделирования структуры подтвердил вывод о разупорядочении N-концевых фрагментов Dps в присутствии ЭДТА.
Рис. 3.
Сравнение экспериментальной кривой рассеяния от белка Dps в буфере II (черные точки) и теоретической кривой, рассчитанной от GEN_DAM_body модели (пунктирная зеленая кривая) (а). Модель белка, построенная с помощью программы GEN_DAM_body (б).
Однако природа этого явления ясна не полностью. Известно, что очищенный Dps содержит только несколько атомов железа, предположительно связанных с отрицательно заряженными аминокислотами Asp и Glu в N-концевой области белка. Кроме того, железо относится к переходным металлам и образует прочные комплексы, например, с азотом (N), входящим в состав аминогрупп аминокислот. Так, в гемоглобине Fe2+ связывается с шестью лигандами, четыре из которых представлены атомами азота порфиринового кольца гема. Таким же образом в Dps двухвалентные атомы железа могут образовывать связи с соседними белковыми цепями, притягивая их друг к другу и стабилизируя поверхностные участки додекамера. ЭДТА, будучи сильным хелатирующим агентом, может разрушить эти связи железа с аминокислотами и тем самым способствовать разворачиванию N-концевой области белка и взаимодействию с ДНК.
Свойством железа образовывать комплексы и стягивать соседние белковые цепи можно также объяснить разрушение уже сформированных со-кристаллов Dps–ДНК и наблюдаемую тотальную агрегацию (рис. 1а, кривая 4). В то же время Mg2+ в отличие от железа действует только как противоион и просто нейтрализует отрицательные заряды ДНК: со-кристаллы Dps–ДНК при этом распадаются, но не агрегируют (рис. 1а, кривая 4).
Для проверки предположения о воздействии свободных катионов железа на агрегационное состояние белка в раствор Dps в буфере II была добавлена соль железа FeSO4 в концентрации 10 мМ. Кривая рассеяния 6 на рис. 1а характерна для неупорядоченных полидисперсных образцов. То есть катионы железа, взаимодействуя одновременно с аминогруппами соседних молекул белка, действительно способствуют его агрегации.
ЗАКЛЮЧЕНИЕ
В работе была поставлена задача с помощью малоуглового рентгеновского рассеяния рассмотреть взаимосвязь основных факторов, определяющих формирование со-кристаллов Dps–ДНК в растворе. При решении задачи получена новая информация о структуре белка Dps и особенностях его взаимодействия с ДНК при формировании защитного кристаллического комплекса. Полученные данные необходимы для дальнейших исследований этого интересного и важного феномена, поскольку вопросов осталось довольно много.
Авторы выражают благодарность В.В. Волкову (ФНИЦ “Кристаллография и фотоника” РАН) за полезную дискуссию и ценные советы.
Работа выполнена при поддержке Российского научного фонда (проект № 18-74-0071).
Список литературы
Almiron M., Link A.J., Furlong D. et al. // Genes Dev. 1992. V. 6. P. 2646.
Martinez A., Kolter R. // J. Bacteriol. 1997. V. 179. P. 5188.
Wolf S.G., Frenkiel D., Arad T. et al. // Nature 1999. V. 400. P. 83.
Frenkiel-Krispin D., Ben-Avraham I., Englander J. et al. // Mol. Microbiol. 2004. V. 51. P. 395.
Frenkiel-Krispin D., Levin-Zaidman S., Shimoni E. et al. // EMBO J. 2001. V. 20. P. 1184.
Dadinova L.A., Chesnokov Yu.M., Kamyshinsky R.A. et al. // FEBS Lett. 2019. V. 593. P. 1360.
Kamyshinsky R., Chesnokov Yu., Dadinova L. et al. // Biomolecules. 2020. V. 10. P. 39.
Grant R.A., Filman D.J., Finkel S.E. et al. // Nat. Struct. Mol. Biol. 1998. V. 5. P. 294.
Bozzi M., Mignogna G., Stefanini S. et al. // J. Biol. Chem. 1997. V. 272. P. 3259.
Chowdhury R.P., Vijayabaskar M.S., Vishveshwara S. et al. // Biochem. 2008. V. 47. P. 11110.
Gupta S., Chatterji D. // J. Biol. Chem. 2003. V. 278. P. 5235.
Ceci P., Ilari A., Falvo E. et al. // J. Biol. Chem. 2005. V. 28. P. 34776.
Roy S., Saraswathi R., Gupta S. et al. // J. Mol. Biol. 2007. V. 370. P. 752.
Stillman T.J., Upadhyay M., Norte V.A. et al. // Mol. Microbiol. 2005. V. 57. P. 1101.
Santos S.P., Cuypers M.G., Round A. et al. // J. Mol. Biol. 2017. V. 429. P. 667.
Bhattacharyya G., Grove A. // J. Biol. Chem. 2007. V. 282. P. 921.
Nguyen K.H., Grove A. // Biochem. 2012. V. 51. P. 6679.
Ceci P., Cellai S., Falvo E. et al. // Nucl. Acids Res. 2004. V. 32. P. 5935.
Papinutto E., Dundon W.G., Pitulis N. et al. // J. Biol. Chem. 2002. V. 277. P. 15093.
Zanotti G., Papinutto E., Dundon W.G. et al. // J. Mol. Biol. 2002. V. 323. P. 125.
Vintiloiu A., Boxriker M., Lemmer A. et al. // Chem. Eng. J. 2013. V. 223. P. 436.
Mozhaev A.A., Serova O.V., Orsa A.N. et al. // Russ. J. Bioorg. Chem. 2019. V. 45. № 2. P. 179.
Marko M.A., Chipperfield R., Birnboim H.C. // Anal. Biochem. 1982. V. 121. P. 382.
Karas V.O., Westerlaken I., Meyer A.S. // J. Visualized Experiments. 2013. V. 75. P. e50390.
Blanchet C.E., Spilotros A., Schwemmer F. et al. // J. Appl. Cryst. 2015. V. 48. P. 431.
Franke D., Petoukhov M.V., Konarev P.V. et al. // J. Appl. Cryst. 2017. V. 50. P. 1212.
Konarev P.V., Volkov V.V., Sokolova A.V. et al. // J. Appl. Cryst. 2003. V. 36. P. 1277.
Hajizadeh N.R., Franke D., Jeffries C.M. et al. // Sci. Rep. 2018. V. 8. P. 7204.
Porod G., Glatter O., Kratky O. et al. Small angle X-ray Scattering. L.: Academic Press, 1982. 51.
Svergun D.I. // J. Appl. Cryst. 1992. V. 25. P. 495.
Svergun D.I. // Biophys. J. 1999. V. 76. P. 2879.
Petoukhov M.V., Franke D., Shkumatov A.V. et al. // J. Appl. Cryst. 2012. V. 45. P. 342.
Svergun D.I., Barberato C., Koch M.H.J. // J. Appl. Cryst. 1995. V. 28. P. 768.
Petoukhov M.V., Svergun D.I. // Acta Cryst. D. 2015. V. 71. P. 1051.
Kozin M., Svergun D. // J. Appl. Cryst. 2001. V. 34. P. 33.
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография


