Кристаллография, 2021, T. 66, № 5, стр. 785-788
Пространственная структура точечного мутанта эстеразы PMGL2
Д. А. Горяйнова 1, А. Ю. Николаева 2, М. В. Крюкова 2, Л. Е. Петровская 3, Д. А. Корженевский 2, Е. А. Крюкова 3, Д. А. Долгих 3, 4, К. М. Бойко 1, *
1 Институт биохимии им. А.Н. Баха РАН, Федеральный исследовательский центр “Фундаментальные основы биотехнологии” РАН
Москва, Россия
2 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
3 Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова РАН
Москва, Россия
4 Московский государственный университет им. М.В. Ломоносова
Москва, Россия
* E-mail: boiko_konstantin@inbi.ras.ru
Поступила в редакцию 17.07.2020
После доработки 17.07.2020
Принята к публикации 03.08.2020
Аннотация
Ферменты из экстремофильных организмов представляют повышенный интерес для задач биотехнологии, поскольку обладают природной адаптацией к экстремальным условиям, имеющим место в технологических процессах. Липазы являются обширным классом гидролитических ферментов, которые катализируют расщепление сложноэфирных связей в триацилглицеридах, а также находят многочисленные применения в биотехнологии. Исследована кристаллическая структура точечного мутанта эстеразы PMGL2, ген которой обнаружен в результате скрининга метагеномной библиотеки ДНК из вечномерзлого грунта Сибири. Структура получена с разрешением 1.5 Å. Проведено сравнение структур мутантной формы и дикого типа PMGL2 на уровне строения субъединицы, функционального димера и активного центра фермента.
ВВЕДЕНИЕ
В настоящее время одним из важных направлений научных исследований в биотехнологии является изучение возможностей практического использования микроорганизмов, обитающих в экстремальных условиях (экстремофилов). Такие микроорганизмы представляют особый интерес для биотехнологического применения, поскольку являются природными продуцентами ферментов, способных функционировать в нестандартных и часто экстремальных условиях: при повышенной или пониженной температуре, в присутствии высокой солености или органических растворителей и т.д. [1, 2].
Примером практически значимых ферментов являются липазы, являющиеся обширным классом гидролитических ферментов, которые катализируют расщепление сложноэфирных связей в триацилглицеридах, а также находят многочисленные применения в различных биотехнологических процессах [3, 4]. Липолитические ферменты имеют схожую пространственную структуру – двудоменный α/β-гидролазный фолд, включающий в себя каноническую каталитическую триаду, состоящую из остатков Ser-His-Asp/Glu.
Согласно существующей классификации, основанной на наличии консервативных мотивов, липазы делятся на несколько семейств – I–VIII [5–7], пространственные структуры представителей каждого из которых к настоящему моменту установлены. Бактериальные ферменты, относящиеся к семейству IV, по своей структуре и свойствам схожи с гормон-чувствительными липазами млекопитающих и потому были названы бактериальными гормон-чувствительными липазами (bHSL). Ферменты этого семейства в свою очередь подразделяются на два класса на основании последовательности мотива активного центра – GD(E)SAG и GTSAG [8–10]. В последнем случае допускается вариабельность остатка, предшествующего каталитическому серину. В отличие от хорошо изученных ферментов класса GDSAG ферменты второго класса изучены значительно меньше – установлены структуры всего трех ферментов. Одним из них является эстераза PMGL2, ген которой обнаружен в результате скрининга метагеномной библиотеки ДНК из вечномерзлого грунта. Эстераза относится к семейству HSL [11] и характеризуется наличием неканонического аминокислотного остатка (а.о.) в положении, предшествующем каталитическому S174, в консервативном мотиве активного центра – 172GXSAG176. В случае PMGL2 в этом положении находится остаток цистеина – C173. С целью выяснения функциональной роли данного остатка цистеина был сконструирован ряд мутантных вариантов PMGL2, содержащих в этом положении остатки треонина, аспарагиновой кислоты и серина, встречающиеся у гомологичных ферментов [12].
В настоящей работе исследована кристаллическая структура точечного мутанта C173S эстеразы PMGL2 (m3PMGL2) с разрешением 1.5 Å. Проведено сравнение структур мутантной формы и дикого типа PMGL2 на уровне строения субъединицы, функционального димера и активного центра фермента.
МАТЕРИАЛЫ И МЕТОДЫ
Очистка и выделение белка, кристаллизация. Очистку и выделение мутантной формы m3PMGL2 проводили согласно [10]. Очищенный белок концентрировали до 10 мг/мл при помощи концентраторов Millipore для последующей кристаллизации.
Кристаллизацию m3PMGL2 проводили при температуре +20°С методом диффузии в парах (висячая капля) [10]. Финальные условия кристаллизации следующие: 250 мМ MgCl2, 100 мМ HEPES 12% PEG 3350, pH 7.5.
Сбор и обработка дифракционных данных. Решение и уточнение структуры. Непосредственно перед сбором данных кристаллы m3PMGL2 кратковременно вымачивали в кристаллизационном растворе, содержащем дополнительно 25% глицерина в качестве криопротектора, после чего кристаллы вылавливали при помощи петли и помещали под струю жидкого азота. Дифракционные данные собраны на станции синхротронного источника SPring-8 (Harima Science Garden, Япония) при температуре 100 K. Дифракционная картина фиксировалась детектором Pilatus6M. Экспериментальные данные проиндексированы и обработаны с использованием программы iMOSFLM [13]. С помощью программы Pointless [14] установлена пр. гр. P21. Статистика набора данных приведена в табл. 1.
Таблица 1.
Кристаллографические данные и параметры съемки кристалла эстеразы PMGL2
| Пр. гр. | P21 |
| a, b, c, Å | 47.15, 92.53, 74.41 |
| α, β, γ, град | 90.00, 106.61, 90.00 |
| Т, K | 100 |
| λ, Å | 1.0 |
| Разрешение, Å | 56.48–1.50 (1.53–1.50)* |
| Число независимых рефлексов | 95216 (4656) |
| Повторяемость | 6.3 (6.3) |
| Полнота набора, % | 97.6 (95.9) |
| I/σ (I) | 16.1 (3.1) |
| Rmeas, % | 7.3 (61.2) |
| CC1/2, % | 99.9 (86.5) |
Структура m3PMGL2 получена с разрешением 1.50 Å при помощи метода молекулярного замещения с применением программы MOLREP [15]. В качестве стартовой модели использовали структуру PMGL2 дикого типа (код PDB6QIN). Уточнение структуры проводили с помощью программы REFMAC5 из набора CCP4 [16]. Визуальный осмотр карт электронной плотности и уточнение модели проводили с использованием программы COOT [17]. Уточнение структуры выполняли до достижения R-факторами следующих значений: Rwork = 16.8%, Rfree = 19.5% (табл. 2).
Таблица 2.
Данные уточнения эстеразы PMGL2
| Rfact, % | 16.8 |
| Rfree, % | 19.5 |
| Общий средний B-фактор | 18.5 |
| Средний B-фактор по белку | 17.9 |
| Средний B-фактор по лигандам | 27.2 |
| Средний B-фактор по растворителю | 25.8 |
| Число неводородных атомов | |
| Белок | 4833 |
| Лиганды | 24 |
| Растворитель | 414 |
| Всего | 5286 |
| Среднеквадратичные отклонения | |
| Длины связей, Å | 0.012 |
| Валентные углы, град | 1.891 |
| График Рамачандрана | |
| Наиболее благоприятные, % | 96.7 |
| Допустимые, % | 2.8 |
| Код PDB | 6ZL7 |
В случае m3PMGL2 в независимой части элементарной ячейки кристалла находятся две белковые цепи (в цепи A – 317 остатков, в цепи B – 320 остатков), один ион магния, две молекулы PEG 3350 и 453 молекулы растворителя. Электронная плотность отсутствует для 16 и 13 N-концевых аминокислот для цепей А и B соответственно, что может быть связано с их относительной подвижностью. Также в обеих цепях молекулы для а.о. 221–226 электронная плотность имела низкий уровень, позволивший, однако, достроить эти участки.
Анализ структуры проводили с использованием программы COOT и системы молекулярной графики PyMOL, Version 1.9.0.0 (Schrodinger, USA). Сравнение и наложение структур проводили с помощью сервиса PDBeFold [18], а контакты анализировали с использованием сервиса PDBePISA [19].
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Кристаллы m3PMGL2 принадлежат пр. гр. P21. В независимой части элементарной ячейки кристалла находятся две субъединицы белка, образующие функциональный димер.
Структура m3PMGL2 имеет типичный α/β-гидролазный фолд, состоящий из двух канонических доменов – CAP-домена и каталитического домена. Домен CAP содержит две α-спирали (α1 и α2). Каталитический домен состоит из восьми β-тяжей (β1–β8), окруженных девятью α-спиралями (α3–α11) [10], и содержит в активном центре каталитический остаток серина – S174.
Анализ контактов между субъединицами из независимой части показал, что m3PMGL2 аналогично многим другим эстеразам является димером в кристалле. Скрытая при формировании димера поверхность (buried surface area) составляет 1508 Å2 (11.3% поверхности субъединицы). Димер стабилизирован 10 солевыми мостиками и 20 водородными связями. Оценка гидрофобных межмолекулярных взаимодействий в димере, сделанная по выигрышу свободной энергии при формировании межмолекулярного контакта, дает величину –7.5 ккал/моль. Полученные значения схожи с таковыми для дикого типа PMGL2 [10].
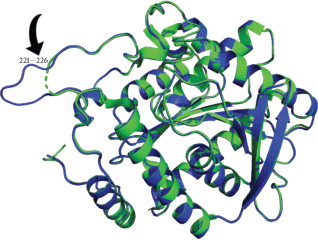
Проведенный анализ показал, что структуры субъединиц m3PMGL2 и дикого типа фермента также весьма схожи (RMSD = 0.181 Å2). Наложение субъединиц продемонстрировало, что элементы вторичной структуры расположены идентично, что говорит об отсутствии влияния точечной замены на общую структуру фермента (рис. 1). Основные различия между структурами обнаружены в петлях 221–226. В молекуле PMGL2 дикого типа а.о. 221–226 не имели электронной плотности [10] в отличие от молекулы m3PMGL2, где эта область имеет электронную плотность, хотя и низкого уровня.
Рис. 1.
Наложение структуры m3PMGL2 (темный) на структуру PMGL2 дикого типа (светлый). Расположение петли 221–226 обозначено стрелкой.
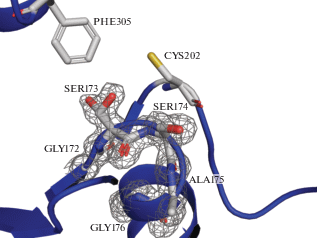
Полость активного центра m3PMGL2 сформирована остатками G172, S173, A175, G176. В активном центре m3PMGL2 на месте цистеина, предшествующего каталитическому S174, находится серин, что подтверждает проведенную мутацию (рис. 2). Электронная плотность для S173 демонстрирует, что он имеет двойное положение: в одном положении его боковая группа ориентирована по направлению к C202, а в другом – к боковой группе F305. В последнем положении S173 способен образовывать водородную связь с боковой группой E120 через молекулу растворителя.
ЗАКЛЮЧЕНИЕ
Исследована структура мутантной формы эстеразы PMGL2, проведено ее сравнение со структурой белка дикого типа и показано, что на уровне структуры димера, субъединицы и активного центра различий в строении двух форм PMGL2 не обнаружено. Полученные результаты позволяют утверждать, что замена цистеина на серин в положении 173 не влияет на структуру фермента, при этом биохимические характеристики мутантной формы близки к таковым для дикого типа [12].
Работы по экспрессии и выделению белка выполнены при финансовой поддержке Российского фонда фундаментальных исследований (грант № 18-04-00491), работы по кристаллографическому уточнению и анализу полученной структуры выполнены при поддержке Министерства науки и высшего образования РФ. Статья подготовлена по материалам кристаллизационных исследований, выполненных в ЦР (КЭ) “Кристаллизатор” (Шифр ЦР (КЭ) “МКС (Наука)” ФНИЦ-1) на российском сегменте Международной космической станции.
Список литературы
Kumar L.A.G., Singh B. // Biotechnology. 2011. V. 10. P. 121. https://doi.org/10.3923/biotech.2011.121.135
Karan R., Capes M.D., Dassarma S. // Aquat. Biosyst. 2012. V. 8. P. 4. https://doi.org/10.1186/2046-9063-8-4
Gaur R., Hemamalini R., Khare S.K. // Current Developments in Biotechnology and Bioengineering: Production, Isolation and Purification of Industrial Products. 2017. P. 175. https://doi.org/10.1016/B978-0-444-63662-1.00008-7
Casas-Godoy L., Duquesne S., Bordes F. et al. // Methods Mol. Biol. 2012. V. 861. P. 3. https://doi.org/10.1007/978-1-61779-600-5_1
Ollis D.L., Cheah E., Cygler M. et al. // Protein Eng. 1992. V. 5. P. 197. https://doi.org/10.1093/protein/5.3.197
Arpigny J.L., Jaeger K.E. // Biochem. J. 1999. V. 343. P. 177.
Nardini M., Dijkstra B.W. // Curr. Opin. Struct. Biol. 1999. V. 9. P. 732. https://doi.org/10.1016/s0959-440x(99)00037-8
Jeon J.H., Lee H.S., Kim J.T. et al. // Appl. Microbiol. Biotechnol. 2012. V. 93. P. 623. https://doi.org/10.1007/s00253-011-3433-x
Li P.Y., Ji P., Li C.Y. et al. // J. Biol. Chem. 2014. V. 289. P. 19031. https://doi.org/10.1074/jbc.M114.574913
Boyko K.M., Kryukova M.V., Petrovskaya L.E. et al. // PLOS ONE. 2020. V. 15 (1). P. e0226838. https://doi.org/10.1371/journal.pone.0226838
Petrovskaya L.E., Novototskaya-Vlasova K.A., Spirina E.V. et al. // FEMS Microbiol. Ecol. 2016. V. 92. P. fiw046. https://doi.org/10.1093/femsec/fiw046
Kryukova M.V., Petrovskaya L.E., Novototskaya-Vlasova K.A. et al. // Biochemistry. 2020. V. 85. P. 831. https://doi.org/10.1134/S0006297920060085
Battye T.G., Kontogiannis L., Johnson O. et al. // Acta Cryst. D. 2011. V. 67. P. 271. https://doi.org/10.1107/S0907444910048675
Evans P. // Acta Cryst. D. 2006. V. 62. P. 72. https://doi.org/10.1107/S0907444905036693
Vagin A., Teplyakov A. // J. Appl. Cryst. 1997. V. 30. P. 1022. https://doi.org/10.1107/S0021889897006766
Collaborative Computational Project N. // Acta Cryst. D. 1994. V. 50. P. 760. https://doi.org/10.1107/S0907444994003112
Emsley P., Cowtan K. // Acta Cryst. D. 2004. V. 60. P. 2126. https://doi.org/10.1107/S0907444904019158
Krissinel E., Henrick K. // Acta Cryst. D. 2004. V. 60. P. 2256. https://doi.org/10.1107/S0907444904026460
Krissinel E., Henrick K. // J. Mol. Biol. 2007. V. 372. P. 774. https://doi.org/10.1016/j.jmb.2007.05.022
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография