Кристаллография, 2021, T. 66, № 5, стр. 759-764
Роль конформационных изменений гексамерной молекулы бактериальных уридинфосфорилаз в связывании субстратов
К. М. Поляков 1, *, Н. Н. Мордкович 2, Т. Н. Сафонова 2, А. Н. Антипов 2, Н. А. Окорокова 2, П. В. Дороватовский 3, В. П. Вейко 2, **
1 Институт молекулярной биологии им. В.А. Энгельгардта РАН
Москва, Россия
2 Институт биохимии им. А.Н. Баха, Федеральный исследовательский центр “Фундаментальные основы биотехнологии” РАН
Москва, Россия
3 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
* E-mail: kmpolyakov@gmail.com
** E-mail: vladveiko@yahoo.com
Поступила в редакцию 17.09.2020
После доработки 19.11.2020
Принята к публикации 19.11.2020
Аннотация
Получены кристаллы и определены трехмерные структуры мутантных форм уридинфосфорилазы из Shewanella oneidensis MR-1 по остатку треонина активного центра. Показано, что петля 161–179, ответственная за узнавание нуклеозида, участвует в стабилизации гексамерной структуры белка, а ее разупорядочение существенно облегчает проникновение молекулы нуклеозида в активный центр фермента. Обсуждается роль конформационных изменений в функционировании фермента.
ВВЕДЕНИЕ
Уридинфосфорилаза (УФ, КФ 2.4.2.3) − ключевой фермент пиримидинового обмена [1–3]. УФ катализирует расщепление гликозидной связи C–N в уридине с образованием урацила и рибозо-1′-фосфата. Этот фермент участвует в катаболизме пиримидиновых нуклеозидов, играет важную роль в гомеостатической регуляции концентрации уридина в плазме, он обнаружен в большинстве тканей и опухолей практически всех живых организмов. Интерес к исследованию молекулярных основ действия УФ базируется на данных о том, что в клетках злокачественных новообразований у человека наблюдается повышенный уровень активности УФ [4]. Показано, что экспрессия УФ в некоторых опухолях в 2–4 раза выше по сравнению с соседними нормальными тканями [5]. Изучение структурно-функциональной взаимосвязи в УФ может существенно упростить дизайн высокоэффективных ингибиторов данного фермента, позволяющих подавить развитие и метастазирование опухоли.
К настоящему времени решены кристаллические структуры семи представителей УФ бактерий. Все эти ферменты формируют гексамерные молекулы с точечной симметрией 32. На основании структурных исследований для УФ было сделано предположение, что связывание нуклеозида происходит в открытой конформации фермента [6]. Доступ в активный центр мономера в открытой конформации обеспечивается за счет смещения петли, предшествующей C-концевой α-спирали, и частичного разупорядочения этих участков цепи. Ранее аналогичные конформационные изменения структуры мономеров были установлены для пуриновых нуклеозидфосфорилаз [7].
Несмотря на активное исследование УФ из разных источников, молекулярный механизм реакции, катализируемой этим белком, остается не до конца выясненным. Особый интерес представляет остаток треонина активного центра, инвариантный в структурах УФ [8], который по данным рентгеноструктурного анализа (РСА) осуществляет непосредственный контакт с обоими субстратами − уридином и ионом фосфата [6]. Для более детального изучения роли этого остатка в функционировании УФ ранее были получены две мутантные формы УФ из Shewanella oneidensis MR-1 (SoУФ) по остатку треонина 91 активного центра, замененного на аланин и серин (T91A и T91S соответственно), и исследованы их ферментативные характеристики [9]. В настоящей работе получены кристаллы этих мутантов и определены их пространственные структуры методом РСА. В результате исследования выявлены конформационные изменения для мутантной формы T91A, проявляющиеся как разупорядочение функциональной петли 161–179, которая включает в себя остатки, участвующие в связывании основания нуклеозида. Такие конформационные изменения приводят к повышению доступности активного центра молекулы белка для нуклеозида даже по сравнению с открытой формой фермента. В работе обсуждается роль этих изменений в связывании субстратов.
МАТЕРИАЛЫ И МЕТОДЫ
Получение мутантных форм уридинфосфорилазы. Мутантные формы УФ из Sh. oneidensis MR-1 по остатку треонина активного центра (T91S и T91A) были получены ранее, их ферментативные характеристики приведены в [9]. Рекомбинантные белки выделены в гомогенном состоянии по методике, описанной в [8], их первичные структуры подтверждены методом MALDI-TOF/TOF-масс-спектрометрического анализа их триптических гидролизатов.
Концентрацию белка определяли по методу Бредфорда [10] с окраской реагентом “Bio-Rad Protein Assay” (“Bio-Rad”, США). В качестве стандарта использовали раствор бычьего сывороточного альбумина (“Sigma”, США). Ферментативную активность рекомбинантных УФ и их мутантных форм определяли в калий-фосфатном буфере по методике [11].
Сохранение мутантными формами SoУФ четвертичной структуры подтверждали методом аналитической гель-фильтрации на колонке Tricorn 10/300 c сорбентом Superdex 200 с использованием прибора AKTA FPLC (“GE Healthcare”, Великобритания) в 10 мM натрий-фосфатном буфере, pH 7.4, содержащем 150 мM NaCl. В качестве белков-маркеров использовали набор “Gel Filtration Calibration Kits” (GE Healthcare Life Sciences, Великобритания), а также рекомбинантную SoУФ из Sh. oneidensis MR-1.
Кристаллизацию мутантных форм SoУФ проводили методом “висячей капли” посредством диффузии в парах при комнатной температуре в термостатированной комнате с использованием стандартных наборов для кристаллизации (скрининга) глобулярных белков компании Hampton Research. Для кристаллизации использовали лиофильно высушенный препарат, растворенный в деионизированной воде (MilliQ) с концентрацией 20 мг/мл. Раствор белка центрифугировали непосредственно перед кристаллизацией на микроцентрифуге в течение 10 мин со скоростью 18 000 об./мин для удаления инородных частиц и агрегатов. Для приготовления каждой капли использовалось 2.0 мкл раствора белка, который смешивался с противораствором в соотношении 1:1. Объем противораствора в резервуаре составлял 400 мкл. Кристаллизацию проводили в пластиковых планшетах для висячих капель “Linbro” фирмы Hampton Research с использованием силиконированных стекол диаметром 22 мм фирмы Hampton Reseаrch. Кристаллы мутантной формы T91S были получены с использованием буфера 0.1 М Bis-Tris (рН 6.5), содержащего 0.2 М сульфата аммония и полиэтиленгликоль (ПЭГ) 3350 (25 мас. %), в качестве противораствора. Кристаллы мутантной формы T91A получены с использованием раствора 1.0 М моногидрата сульфата лития с добавлением ПЭГ 8000 (2 мас. %). Кристаллы, пригодные для РСА, вырастали в течение одной–двух недель.
Сбор и обработка дифракционных данных. Наборы дифракционных данных собраны до разрешения 1.65 и 1.90 Å для T91S и T91A соответственно на станции белковой кристаллографии “Белок” синхротрона Национального исследовательского центра “Курчатовский институт“ при температуре 100 K. Перед сбором данных кристаллы помещали в крио-растворы, приготовленные из кристаллизационных растворов с добавлением 25% глицерина. Обработку дифракционных данных проводили по программам XDS и XSCALE [12].
Решение и уточнение структур мутантов. Структуры решены методом молекулярного замещения по программе MOLREP [13] с использованием в качестве стартовой модели нативной УФ из Shewanella oneidensis MR-1 [6], из которой предварительно были удалены молекулы воды. Кристаллографическое уточнение структуры проводили по программе REFMAC5 [14]. Исправление моделей структур с визуальным контролем параметров стереохимии проводили по программе COOT [15]. Структура T91S решена с разрешением 1.65 Å и уточнена до значения Rwork = 19.4%. Уточнение структуры мутантной формы T91S проводили с учетом двойникования. Структура T91A решена с разрешением 1.90 Å и уточнена до значения Rwork = 15.6%. Рисунки приготовлены по программе CCP4MG [16]. Кристаллографические расчеты были проведены с использованием комплекса программ CCP4 [17]. Межмолекулярные контакты в олигомерах анализировали по программе PISA [18]. В табл. 1 приведены статистические характеристики наборов рентгеноструктурных данных и уточненных структур.
Таблица 1.
Кристаллографические данные и статистические характеристики наборов рентгеноструктурных данных и уточнения структур
| Структура | Мутант T91S | Мутант T91A |
|---|---|---|
| Станция сбора данных | Станция Белок, Курчатовский институт | Станция Белок, Курчатовский институт |
| Пространственная группа | P21 | P3 |
| a, b, c, Å; α, β, γ, град | 91.40, 95.49, 91.41; 90, 120.1, 90 | 91.66, 91.66, 47.56; 90, 90, 120 |
| Разрешение, Å | 1.65 (1.75–1.65)* | 1.90 (2.02–1.90) |
| Полнота набора, % | 97.4 (88.9) | 99.4 (98.5) |
| Повторяемость | 4.9 | 6.4 |
| I/σ(I) | 8.8 (3.08) | 15.6 (3.24) |
| Rmeas, % | 13.9 (54.9) | 12.3 (68.8) |
| CC(1/2), % | 99.3 (88.3) | 99.8 (86.9) |
| Среднее значение B-фактора для всех атомов, Å2 | 14.3 | 19.6 |
| Число двойниковых доменов кристалла | 6** | 1 |
| Rwork, % | 19.4 | 15.6 |
| Rfree, % | 23.3 | 22.1 |
| Число мономеров в независимой части ячейки | 6 | 2 |
| Число уточняемых атомов белка | 10946 | 3590 |
| Число уточняемых молекул воды | 839 | 295 |
| R.m.s.d. от идеальной геометрии, по длинам валентных связей Å | 0.020 | 0.016 |
| R.m.s.d. от идеальной геометрии по валентным углам, град | 2.1 | 2.3 |
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Кристаллы мутантных форм T91A и T91S выращены, как указано выше, в различных условиях. Для всех кристаллографически независимых мономеров обеих структур электронная плотность подтверждает замены аминокислотного остатка в положении 91. В активном центре структуры T91S локализованы молекула урацила в одном мономере, сульфат-ионы в четырех мономерах и молекула глицерина в двух мономерах. Кроме того, в этой структуре локализованы два сульфат-иона, расположенные на оси третьего порядка гексамерной молекулы фермента. В структуре T91A надежно идентифицированы четыре молекулы глицерина, шесть ионов сульфата и ион лития, расположенный на некристаллографической оси симметрии, связывающей независимые мономеры. Однако в этой структуре не удается найти ионы сульфата, расположенные на оси третьего порядка. Несмотря на то что в независимой части элементарной ячейки структуры мутанта T91S содержится шесть кристаллографически независимых мономеров, а в структуре мутанта T91A – два мономера, функциональными олигомерами в обеих структурах являются сходные гексамерные структуры.
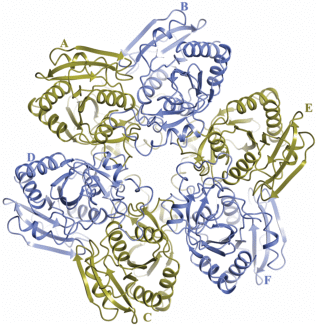
Гексамер Т91S сформирован шестью кристаллографически независимыми мономерами и имеет некристаллографическую симметрию 32 (рис. 1). Гексамер T91S состоит из трех димеров, связанных некристаллографической осью третьего порядка. Мономеры в димере связаны некристаллографическими осями симметрии второго порядка. Все мономеры находятся в закрытой конформации. Петля, предшествующая концевой α-спирали (остатки 220–235), частично разупорядочена во всех мономерах. На оси некристаллографической симметрии третьего порядка с двух сторон гексамера локализованы ионы сульфата, координированные остатками аргининов 175 трех мономеров.
Рис. 1.
Ход полипептидной цепи в гексамерной молекуле T91S. Гексамер образован шестью кристаллографически независимыми мономерами.
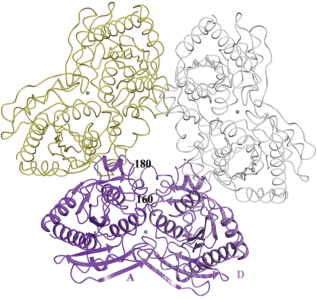
Гексамер T91A состоит из несимметричных димеров, связанных кристаллографической осью третьего порядка (рис. 2). Оба независимых мономера (А и D) находятся в открытой конформации. Отметим, что остатки петли 161–179 в мономере A не локализованы. Эта петля содержит глутамин 163 и аргинин 165, участвующие в связывании основания нуклеозида, а также аргинин 175, связывающий ион сульфата/фосфата на оси третьего порядка. Во втором мономере (мономере D) эта петля локализована на картах электронной плотности, но атомы соответствующих остатков имеют более высокие температурные факторы (около 40 Å2) по сравнению со средним температурным фактором по молекуле (19.6 Å2). Такое структурное различие мономеров и приводит к несимметричности гексамерной молекулы. Подобно мономерам в структуре T91S три мономера в T91А (мономер D и его симметричные копии) образуют контакты в области оси третьего порядка (рис. 2).
Рис. 2.
Ход полипептидной цепи в гексамерной молекуле T91A. Кристаллографически независимые мономеры A и D показаны как модели с β-слоями и α-спиралями. Димеры, связанные с исходным димером кристаллографической осью третьего порядка, показаны в виде ленточной модели с разными оттенками. В мономере A обозначены номера остатков, между которыми не локализована петля (160–180).
По оценкам программы PISA [18] гексамеры мутантных форм УФ стабильны. Энергии диссоциации гексамеров равны 70.6 и 36.0 ккал/моль для T91S и T91A соответственно. Такая разница в энергии диссоциации объясняется вкладом связывания двух сульфат-ионов, присутствующих на оси третьего порядка в структуре T91S (около 26.0 ккал/моль), и упорядочением в этой структуре петли 161–179.
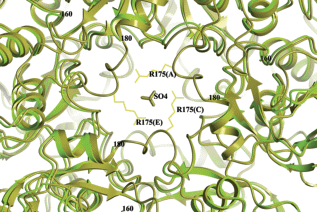
В структурах обоих мутантов остаток аргинина 175 в мономерах с упорядоченной петлей 161–179 принимает участие в стабилизации гексамеров за счет водородных связей с карбонильными атомами кислорода соседних мономеров (рис. 3). Кроме того, аргинин 175 образует солевые мостики с атомами ионов сульфата, расположенными на оси третьего порядка. Отметим, что в структуре T91A ионы сульфата на кристаллографической оси не выявлены. Сравнение структур T91A и T91S в области связывания этого сульфат-иона показано на рис. 3. Связывание одного иона сульфата на оси третьего порядка приводит к увеличению свободной энергии диссоциации гексамера на 13 ккал/моль.
Рис. 3.
Сравнение структур T91A (мономеры A, E и C; темный цвет) и T91S (мономеры A; светлый цвет) для области связывания сульфат-иона на оси третьего порядка гексамера.
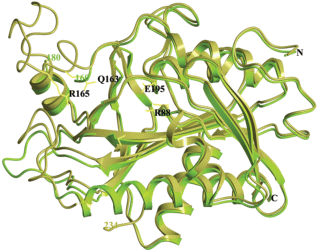
Сравнение мономера D структуры T91S в закрытой конформации и мономера А структуры T91A в открытой конформации с разупорядоченной петлей 161–179 приведено на рис. 4. Смещение петли в районе остатка 224 от активного центра, наблюдаемое в открытой конформации, может обеспечивать доступ молекулы нуклеозида в активный центр фермента. Дополнительное разупорядочение петли 161–179 в структуре мономера в открытой конформации значительно облегчает встраивание молекулы нуклеозида в активный центр фермента.
Рис. 4.
Сравнение мономера A структуры T91A (темный цвет) и мономера D структуры T91S (светлый цвет). Мономер А структуры T91A находится в открытой конформации (остаток 224 смещен в сторону от остатка 165, N-концевой фрагмент последней α-спирали упорядочен). В этой структуре не локализована петля 160–180. Мономер D структуры T91S находится в закрытой конформации (остаток 224 смещен в сторону остатка 165, N-концевой фрагмент последней α-спирали упорядочен). В этой структуре не локализована петля 232–234. Для структуры T91A показаны остатки активного центра.
Анализ структур УФ из PDB, решенных с разрешением выше 2 Å, показал, что все УФ из бактерий образуют гексамеры. В большинстве структур гексамеры имеют некристаллографическую ось симметрии третьего порядка. В таких структурах гексамеры симметричны и аналогичны структуре гексамера T91A. Однако для четырех бактериальных УФ из семи исследованных были получены кристаллические модификации, содержащие в структуре гексамеры с кристаллографической осью третьего порядка. При этом кристаллографически независимые мономеры в димерах могут быть как симметричными (E. coli, PDB_ID 1rxy; Yersinia pseudotuberculosis, PDB_ID 4ny1), так и несимметричными (структуры мутантной формы T91А из Shewanella oneidensis и УФ из Salmonela typhimurium (PDB_ID oxf, 3hsw, 1y1t)). В случае формирования несимметричного димера одна из молекул димера частично разупорядочена (отсутствуют остатки в районе петли 161–179).
Считается, что субстраты способны проникать в активный центр фермента, находящегося в открытой конформации, и связываться там. Однако сравнение структур мономеров в закрытой конформации из симметричных димеров и структур мономеров несимметричных димеров с разупорядоченной петлей 161–179 (на примере структур мономеров мутантных форм T91S и T91А) показывает, что проникновение молекулы нуклеозида в активный центр существенно облегчается в случае разупорядоченной петли 161–179 в несимметричных гексамерах (рис. 4).
Можно предположить, что встраивание субстрата в активный центр УФ происходит именно в мономеры с разупорядоченной петлей в районе остатков 161–179. При этом объемная молекула нуклеозида встраивается в активный центр первой и стабилизирует петлю в районе остатков 161–179 (два остатка из этой петли, глутамин и аргинин, образуют связи с атомами основания). Связывание нуклеозида приводит к стабилизации мономера УФ в открытой конформации. Далее происходит связывание фосфат-иона в активном центре фермента, приводящее к изменению конформации мономера на закрытую. Одновременно с этим гексамерная молекула стабилизируется за счет связывания сульфат/фосфат-ионов на оси третьего порядка. Ферментативная реакция происходит в мономерах в закрытой конформации. Благодаря меньшему размеру (по сравнению с нуклеозидом) продуктов реакции выход основания нуклеотида и рибозофосфата из активного центра не требует разупорядочения петли в районе остатков 161–179.
Присутствие ионов фосфата/сульфата на оси третьего порядка в структуре гексамера (рис. 4) может стабилизировать структуру за счет взаимодействия этих ионов с остатками аргинина 175 трех мономеров. По-видимому, ионы фосфата/сульфата принимают участие в разупорядочении петли в районе 186 остатка.
Таким образом, на основании анализа трехмерных структур мутантов T91A и T91S показано, что конформационные изменения, связанные с разупорядочением гибкой петли в районе остатков 161–179 бактериальной УФ, играют важную роль в связывании субстрата и осуществлении ферментативной реакции.
Работы в части кристаллизации мутантных форм УФ, сбора дифракционных наборов данных проведены в рамках выполнения работ по Государственному заданию Института биохимии им. А.Н. Баха, ФНИЦ “Фундаментальные основы биотехнологии”, в части получения мутантных форм УФ, определения каталитической активности белка, решения, уточнения и анализа трехмерных структур мутантов – при поддержке Российского фонда фундаментальных исследований (грант № 18-04-00784а). При проведении исследований использовали оборудование Центра коллективного пользования “Промышленные биотехнологии” Федерального исследовательского центра “Фундаментальные основы биотехнологии” РАН.
Список литературы
Cao D., Pizzorno G. // Drugs Today (Barc). 2004. V. 40. P. 431. https://doi.org/10.1358/dot.2004.40.5.850491
Cappiello M., Mascia L., Scolozzi C. et al. // Biochim. Biophys. Acta. 1998. V. 1425. P. 273. https://doi.org/10.1016/s0304-4165(98)00071-3
Tozzi M.G., Camici M., Mascia L. et al. // FEBS J. 2006. V. 273. P. 1089. https://doi.org/10.1111/j.1742-4658.2006.05155.x
Luccioni C., Beaumatin J., Bardot V. et al. // Int. J. Cancer. 1994. V. 58. P. 517. https://doi.org/10.1002/ijc.2910580411
Watanabe S., Uchida T. // Biochem. Biophys. Res. Commun. 1995. V. 216. P. 265. https://doi.org/10.1006/bbrc.1995.2619
Safonova T.N., Mikhailov S.N., Veiko V.P. et al. // Acta Cryst. D. 2014. V. 70. P. 3310. https://doi.org/10.1107/S1399004714024079
Koellner G., Bzowska A., Wielgus-Kutrowska B. et al. // J. Mol. Biol. 2002. V. 315. P. 351. https://doi.org/10.1006/jmbi.2001.5211
Мордкович Н.Н., Манувера В.А., Вейко В.П. и др. // Биотехнология. 2012. № 1. С. 21.
Мордкович Н.Н., Сафонова Т.Н., Антипов А.Н. и др. // Прикладная биохимия и микробиология. 2018. Т. 54. № 1. С. 16. https://doi.org/10.7868/S0555109918010026
Bradford M.M. //Anal. Biochem. 1976. V. 2. P. 248. https://doi.org/10.1006/abio.1976.9999
Leer J.C., Hammer-Jespersen K., Schwartz M. // Eur. J. Biochem. 1977. V. 75. P. 217. https://doi.org/10.1111/j.1432-1033.1977.tb11520.x
Kabsch W. // Acta. Cryst. D. 2010. V. 66. P. 125. https://doi.org/10.1107/S0907444909047337
Vagin A.A., Teplyakov A.V. // J. Appl. Cryst. 1997. V. 30. P. 1022. https://doi.org/10.1107/S0021889897006766
Murshudov G.N., Skubák P., Lebedev A.A. et al. // Acta. Cryst. D. 2011. V. 67. P. 355. https://doi.org/10.1107/S0907444911001314
Emsley P., Lohkamp B., Scott W. et al. // Acta. Cryst. D. 2010. V. 66. P. 486. https://doi.org/10.1107/S0907444910007493
McNicholas S., Potterton E., Wilson K.S. et al. // Acta. Cryst. D. 2011. V. 67. P. 386. https://doi.org/10.1107/S0907444911007281
Winn M.D., Ballard C.C., Cowtan K.D. et al. // Acta. Cryst. D. 2011. V. 67. P. 235. https://doi.org/10.1107/S0907444910045749
Krissinel E., Henrick K. // J. Mol. Biol. 2007. V. 372. P. 774. https://doi.org/10.1016/j.jmb.2007.05.022
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография


