Кристаллография, 2021, T. 66, № 5, стр. 742-749
Пространственная структура рекомбинантной термофильной рибокиназы thermus speсies 2.9 в комплексе с аденозиндифосфатом
В. И. Тимофеев 1, 2, Ю. А. Абрамчик 3, Т. И. Муравьева 3, Н. Е. Жухлистова 1, Р. С. Есипов 3, И. П. Куранова 1, 2, *
1 Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника” РАН
Москва, Россия
2 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
3 Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова РАН
Москва, Россия
* E-mail: inna@crys.ras.ru
Поступила в редакцию 08.09.2020
После доработки 22.09.2020
Принята к публикации 24.09.2020
Аннотация
Пространственная структура рекомбинантной термофильной рибокиназы Thermus speсies 2.9 установлена при разрешении 2.4 Å с использованием кристаллов, выращенных в невесомости методом встречной диффузии, и дифракционного набора, полученного на источнике синхротронного излучения SPring-8 (Япония). В активном центре фермента локализована молекула аденозиндифосфата и описано ее окружение. Показано, что пространственная структура рибокиназы Thermus species 2.9 весьма сходна со структурами других представителей ферментов этого семейства.
ВВЕДЕНИЕ
Рибокиназа из термофильного штамма бактерий Thermus species 2.9 (Th. sp. 2.9) относится к семейству углеводных рибокиназ (EC 2.7.1.15). Белки этого семейства катализируют фосфорилирование рибозы посредством переноса γ-фосфата аденозинтрифосфата (АТФ) на 5'-гидроксиметильную группу сахара [1, 2]. Реакция протекает в присутствии АТФ и ионов магния [3, 4]. D-рибоза является широко распространенным углеводом, вовлечена в производство энергии, является компонентом РНК и ДНК, входит в состав кофакторов многих ферментов. Через внешнюю клеточную мембрану рибоза диффундирует в периплазму, где связывается с рибозасвязывающими белками, транспортирующими ее через внутреннюю мембрану [5]. Катализируемое рибокиназой фосфорилирование 5'-гидроксиметильной группы является первой ступенью, вовлекающей рибозу в дальнейшие метаболические превращения [6–8]. Продукт реакции D-рибоза–5-фосфат используется для синтеза нуклеотидов, триптофана и гистидина или вовлекается в пентозафосфатный путь. Рибоза-5-фосфат инкорпорируется в АТФ или другие высокоэнергетические соединения. Фосфорибозилпирофосфатсинтетаза катализирует превращение рибоза-5-фосфата в 5-фосфорибозил-1-пирофосфат (5-PRPP), который вовлечен в биосинтетические реакции, приводящие к биосинтезу пуриновых и пиримидиновых оснований, кофакторов ферментов и аминокислот гистидина и триптофана [9, 10]. Рибокиназа участвует также в биохимических процессах, обеспечивающих повторное использование освобождающейся при расщеплении нуклеотидов рибозы [11].
Интерес к структуре и свойствам рибокиназ возрос в связи с применением этих белков в каскадной стратегии энзиматического синтеза биологически активных производных нуклеозидов, который протекает при последовательном действии рекомбинантной рибокиназы, фосфопентамутазы, нуклеозидфосфорилазы [12–16]. Многие производные нуклеозидов, модифицированные по гетероциклическому основанию и углеводной группе, являются важными терапевтическими препаратами против вирусных инфекций и злокачественных опухолей [17, 18]. Широкое использование каскадной стратегии ограничено высокой субстратной специфичностью используемых ферментов и их недостаточной стабильностью. Часть этих ограничений можно обойти, используя ферменты из термофильных организмов. Ферменты термофильных микроорганизмов менее чувствительны к структуре субстрата, активны при температуре 70–80°C и существенно повышают скорость реакции, увеличивая растворимость гетероциклических субстратов [19–21]. В [22] было показано, что рекомбинантная рибокиназа из термофильного штамма Th. sp. 2.9 как катализатор первой ступени реакции каскадного синтеза имеет ряд преимуществ. Фермент имеет максимум активности при 85°С, углеводным субстратом данной рибокиназы кроме D-рибозы может служить 2-дезокси-D-рибоза. Знание пространственной структуры дает возможность рационального конструирования новых мутантных форм фермента с более широкой селективностью, наиболее удобных для биотехнологического применения. В настоящей работе при разрешении 2.4 Å установлена пространственная структура рекомбинантной термофильной рибокиназы Th. sp. 2.9 в комплексе с аденозиндифосфатом (АДФ). Структура решена с использованием дифракционного набора, полученного на синхротроне SPring-8 (Япония) для кристаллов, выращенных в невесомости методом встречной диффузии. Показано, что по типу укладки полипептидной цепи фермент сходен с другими белками семейства рибокиназ.
МАТЕРИАЛЫ И МЕТОДЫ
Кристаллизация и сбор дифракционных данных. Для кристаллизации использовали препарат рекомбинантной рибокиназы, полученной и очищенной по методике [21]. Кристаллы были выращены в невесомости методом встречной диффузии из растворов белка с концентрацией 11.7 мг/мл в 0.25 М Трис-буфере, рН 8.0, содержащем 50 мМ КCl, 1 мМ АТФ, 5 мМ MgCl2, 5% глицерина, 0.04% NaN3 [23]. Концентрация сульфата аммония в осадителе (Na-цитратный буфер, рН 5.5, содержащий 45 мМ KCl, 0.1 М Na-цитрата, pH 5.5, 5мМ MgCl2, 5 мМ АТФ, 0.04% NaN3) была увеличена до 15.5% в отличие от концентрации, используемой в [23].
Дифракционный набор для кристалла, предварительно замороженного в токе жидкого азота, собран при 100 K на станции BL41XU синхротрона SPring-8 (Япония), оснащенной детектором EIGER. Для сбора данных использовали метод вращения, длина волны составляла 0.8 Å, угол вращения 360°, угол качания 0.1°, расстояние кристалл–детектор 400 мм. Набор был обработан с использованием программы iMosflm [24]. Кристаллы фермента описываются пр. гр. P21, содержат четыре молекулы в независимой части ячейки, a = 44.210, b = 155.550, c = 83.040 Å, β = 98.20°. Статистика набора представлена в табл. 1.
Таблица 1.
Статистические характеристики экспериментального набора рентгеновских данных и результаты уточнения структуры рибокиназы Th. sp. 2.9
| Обработка набора | |
| Пространственная группа | P21 |
| a, b, c, Å | 44.2, 155.6, 83.0 |
| β, град | 98.2 |
| Разрешение, Å | 30.0–2.40 (2.53–2.40)* |
| Количество независимых рефлексов | 41 928 |
| Полнота набора, % | 96.9 (97.7) |
| I/σ(I) | 5.29 (4.47) |
| Rmrgd–F, % | 7.7 (14.0) |
| Уточнение | |
| PDB_ID | 6ZNX |
| Разрешение, Å | 30.00–2.40 (2.46–2.40) |
| Количество рефлексов | 39758 |
| Rcryst, % | 23.0 |
| Rfree,% | 27.1 |
| Количество атомов | 8553 |
| B-фактор, Å2 | |
| Средний | 36.21 |
| RMS | |
| По длинам связей, Å | 0.007 |
| По углам, град | 1.635 |
| Карта Рамачандрана | |
| Наиболее благоприятные области | 97.1% |
| Допустимые области | 2.9% |
| Запрещенные области | 0.0% |
Решение и уточнение структуры. Структура рибокиназы Th. sp. 2.9 в комплексе с АДФ определена методом молекулярного замещения с помощью программы BALBES [25]. В качестве стартовой модели использованы координаты рибокиназы Vibrio cholerae O395 в комплексе с рибозой, АДФ и ионом натрия (PDB_ID: 4XDA) [26].
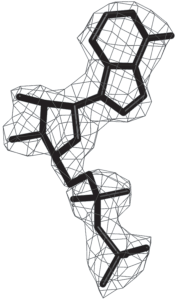
Уточнение структуры проведено по программе REFMAC [27]. Ручную правку модели осуществляли с помощью программы Coot [28], используя карты электронной плотности, рассчитанные с коэффициентами 2|Fo| – |Fc| и |Fo| – |Fc|. Также с помощью программы Coot с использованием карт электронной плотности были локализованы молекулы воды и АДФ. Молекула АДФ, вписанная в карту электронной плотности, приведена на рис. 1.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Кристаллы для определения пространственной структуры рекомбинантной рибокиназы из термофильного штамма бактерий Th. sp. 2.9 были выращены в невесомости методом встречной диффузии по технологии [29] из растворов белка, содержащих ионы магния и АТФ после оптимизации концентрации осадителя в условиях [23]. В результате разрешение дифракционного набора выращенных кристаллов было повышено с 2.87 до 2.4 Å.
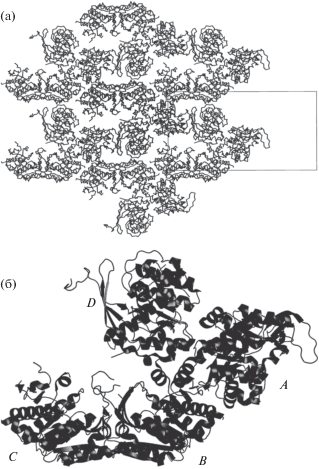
Пространственная структура рибокиназы Th. sp. 2.9 решена методом молекулярного замещения с использованием в качестве стартовой модели координат рибокиназы Vibrio cholerae (PDB_ID: 4XDA) и уточнена при разрешении 2.4 Å. Статистические параметры набора и результаты уточнения приведены в табл. 1. Кристаллы фермента (пр. гр. Р21) содержат четыре молекулы белка (A, B, C, D) в независимой части элементарной ячейки (рис. 2). В каждой молекуле фермента локализована связанная в активном центре молекула АДФ с заселенностью, близкой к единице.
Рис. 2.
Укладка мономеров в решетке кристаллов комплекса рибокиназы Th. sp. 2.9 с АДФ (а); независимая часть элементарной ячейки кристаллов комплекса рибокиназы Th. sp. 2.9 с АДФ – четыре мономера белка A, B, C, D (б).
При совмещении по Сα-атомам четырех молекул фермента, расположенных в независимой части ячейки, величина среднеквадратичного отклонения между парами молекул A–B, A–C, A–D равна соответственно 0.587, 0.760 и 0.570 Å. Значительные отклонения заметны на участках, соединяющих большой и малый домены, в сегментах, соединяющих стрэнды малого домена, и в петле, соединяющей спирали α5 и α6.
Укладка полипептидной цепи в молекуле рибокиназы Th. sp. 2.9. Пространственные структуры известны для ряда белков семейства рибокиназ, в том числе рибокиназы человека и ряда микробных рибокиназ: из E. coli, Vibrio cholera O395, Leishmania donovani, Thermotoga maritima [26, 30, 31]. Показано, что, хотя идентичность аминокислотных последовательностей между белками этого семейства менее 30%, рибокиназы сходны типом укладки полипептидной цепи, имеют два общих мотива в аминокислотной последовательности, а субъединица фермента состоит из двух доменов. Еще одной чертой, общей для большинства рибокиназ с известной структурой, является димерное строение биологически активной формы фермента.
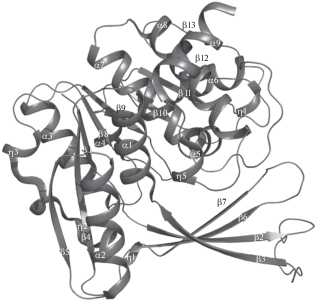
Типичная для рибокиназ укладка полипептидной цепи обнаружена и в молекуле термофильной рибокиназы Th. sp. 2.9. Полипептидная цепь молекулы рибокиназы Th. sp. 2.9, содержащая 301 аминокислотный остаток (а.о.), включает в себя 13 β-стрэндов, 10 α-спиралей и 3 η-спирали (η-спирали 310). Как и во всех белках семейства рибокиназ, молекула состоит из двух доменов: большого каталитического глобулярного α/β-домена и выступающего из него малого β-домена, который представляет собой изогнутый четырехстрэндовый β-слой (рис. 3). Малый β-домен образован двумя вставками в центральную α/β-укладку. Каждая из вставок состоит из пары антипараллельных β-стрэндов β2–β3 (а.о. 8–36) и стрэндов β6–β7 (а.о. 90–108). Большой домен состоит из центрального β-слоя, образованного девятью β-стрэндами, которые с обеих сторон окружены α-спиралями. Первые шесть параллельных β-стрэндов домена вместе с примыкающими α-спиралями имеют топологию классического NAD-связывающего домена Россмана [32], характерную для белков, связывающих нуклеотиды. Однако в рибокиназе к этому слою примыкают еще три стрэнда, соединенные друг с другом короткими поворотами. Три С-концевые β-cтрэнда, продолжающие центральный слой (β11–β13), антипараллельны и соединены короткими поворотами типа шпильки. Обе стороны C-концевого участка большого домена закрыты α-спиралями 6 и 7 с одной стороны и 8 и 9 с другой. Большой и малый домены соединены короткими неупорядоченными участками, которые дают возможность доменам двигаться друг относительно друга. На коротких переходных участках, соединяющих стрэнд β1 большого домена со стрэндом β2 малого домена или стрэнд β3 малого домена со спиралью α1 большого домена, повышенное содержание остатков глицина и пролина, они входят в один из двух общих для всего семейства мотивов аминокислотных последовательностей [11]. Остатки глицина востребованы по стерическим и конформационным причинам. Они увеличивают пространство для возможного изменения взаимного расположения доменов, а остатки пролина способствуют изменению направления полипептидной цепи. Повышенное содержание пролина и глицина наблюдается и в переходных между доменами участках β7–η2 и β5–β6. Температурные факторы а.о. фрагмента 85–89, соединяющего стрэнды β5 и β6, и фрагмента Pro108-Pro116 между стрэндом β7 и спиралью η2 выше среднего по структуре.
Рис. 3.
Укладка полипептидной цепи в мономере комплекса рибокиназы Th. sp. 2.9 с АДФ. Элементы вторичной структуры обозначены стрелками (β-стренды) и спиралями: α и 310 (η) – спирали.
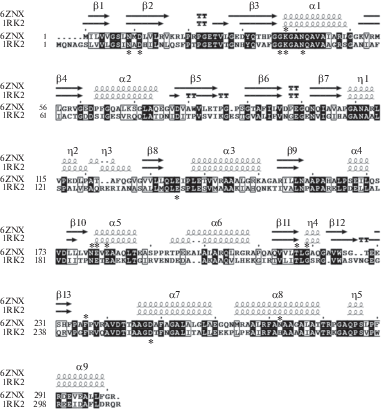
Для сравнения на рис. 4 представлены последовательности и распределение элементов вторичной структуры в мономерах рибокиназы Th. sp. 2.9 и E. coli. Cтепень идентичности аминокислотных последовательностей между этими ферментами превышает 40%. При совмещении по Cα-атомам субъединицы А рибокиназы Th. sp. 2.9 с субъединицей E. coli среднеквадратичное отклонение равно 1.445 Å. Распределение элементов вторичной структуры, а также количество а.о. в α-спиралях и β-стрэндах практически одинаковы в обоих белках.
Рис. 4.
Сравнение аминокислотных последовательностей и вторичных структур рибокиназ Th. sp. 2.9 и E. coli. Элементы вторичной структуры α и 310 (η) спирали и β-cтрэнды обозначены спиралями и стрелками соответственно. Черным цветом выделены идентичные в обоих белках аминокислотные остатки, звездочками – аминокислотные остатки, участвующие в связывании молекул рибозы и АДФ.
Активный центр фермента, включающий в себя центры связывания рибозы и АДФ, расположен на участке поверхности каталитического α/β-домена, находящегося между большим и малым доменами (рис. 5). Известно, что катализируемая ферментом реакция протекает посредством нуклеофильной атаки атомов кислорода О5' гидроксиметильных групп рибозы γ-фосфата АТФ [11]. Нуклеофильные свойства кислорода гидроксиметильной группы рибозы увеличиваются благодаря водородной связи между гидроксильной группой и остатком аспарагиновой кислоты, действующей как каталитическое основание. В молекуле рибокиназы E. coli – это Asp255, в рибокиназе Th. sp. 2.9 – Asp248. Атомы азота основной цепи остатков глицина, окружающие остаток аспарагиновой кислоты, участвуют в образовании анионной дыры, стабилизирующей переходное состояние реакции [11]. Сближение субстратов, необходимое для протекания реакции, – результат перехода фермента из открытого в закрытое состояние сразу после связывания рибозы [30]. Конформационные изменения, обеспечивающие этот переход, сопровождаются движением доменов. В закрытой конформации малый домен изолирует от растворителя область активного центра, в котором связана рибоза. Поэтому малому домену приписывают роль крышки, контролирующей доступ растворителя к активному центру.
О строении участка молекулы, в котором происходит связывание рибозы, можно судить, сравнивая структуру молекулы рибокиназы Th. sp. 2.9 и структуру комплекса рибокиназы E. coli с рибозой и АДФ [11]. Связывание рибозы имеет место на поверхности большого домена, расположенной наиболее близко к сегменту, связывающему большой и малый домены. В прямых контактах с рибозой в рибокиназе E. coli участвуют а.о. стрэнда β1 Asn14, Asp16 (Asn9, Asp11 в Th. sp. 2.9), Gly42 (Gly37 в Th. sp. 2.9), расположенные между β3 и α1, остатки спирали α1 Lys43 и Asn46 (Lys38 и Asn41 в Th. sp. 2.9), Glu143 (Glu135 в Th. sp. 2.9), расположенные между α8 и α3, и Asp255 (Asp248 в Th. sp. 2.9) спирали α7. Почти все перечисленные а.о. относятся к α/β-домену и взаимодействуют с полярной стороной рибозного кольца. Гидрофобная поверхность сахарного кольца взаимодействует в основном с алифатическими боковыми цепями внутренней стороны малого домена. Отметим, что в рибокиназе рибоза связана с областью большого домена, который имеет топологию Россмана, и в большинстве белков связываются нуклеотиды. Все взаимодействующие с рибозой а.о. инвариантны не только в структурах рибокиназ из E. coli и Th. sp. 2.9, но и входят в один из аминокислотных мотивов, характеризующих семейство рибокиназ [11, 30]. Хотя некоторые рибокиназы, в том числе рибокиназы Th. sp. 2.9 и E. coli, могут использовать дезоксирибозу как субстрат [33], в рибокиназе E. сoli это приводит к потере трех водородных связей с а.о. Asn14, Asp16, Gly42 (Asn9, Asp11, Gly37 в Th. sp. 2.9 соответственно), что объясняет предпочтительность рибозы как субстрата. В рибокиназе из E. coli связанная рибоза почти полностью закрыта малым доменом, который действует как крышка над центром связывания, а также второй субъединицей димера.
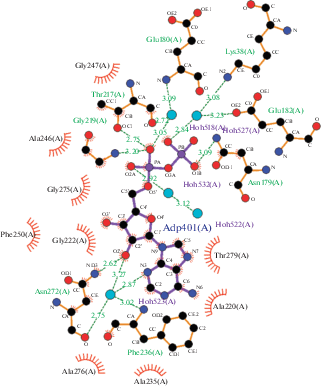
Молекула АДФ, локализованная в молекуле рибокиназы Th. sp. 2.9, образовалась в результате гидролиза АТФ, присутствующего в кристаллизационном растворе, и занимает в активном центре положение, схожее с положением молекулы АДФ в субъединице рибокиназы E. coli. Молекула АДФ локализована в полости между двумя доменами, но непосредственно связана только с а.о., находящимися на поверхности α/β-домена (рис. 5). По сравнению с рибозасвязывающим центром АДФ-связывающий участок находится на большем расстоянии от переходного участка между двумя доменами сегмента. Поэтому при переходе к закрытой конформации фермента молекула АДФ не накрывается полностью малым доменом и остается доступной для растворителя. В связывании молекулы АДФ участвуют а.о. нескольких фрагментов вторичной структуры: пяти спиралей (α1, α5, α7, α8, α9) и двух стрэндов (β12 и β13). Основание аденина локализовано в петле, соединяющей стрэнд β13 со спиралью α7 (а.о. 235–245) (рис. 5). Оксигруппы рибозного фрагмента расположены по одну сторону рибозного кольца. Оксигруппа при втором атоме углерода образует прямую водородную связь с атомом OD2_Asp272 спирали α8, а через молекулу воды – с азотом основной цепи Phe236 стрэнда β13. Вторая гидроксильная группа рибозы связана с атомом кислорода основной цепи Gly222 стрэнда β12. Фосфатные группы молекулы АДФ направлены в сторону центра связывания рибозы. Атомы кислорода α-фосфатной группы связаны непосредственно с атомом азота основной цепи Gly219 и OG1_Тhr217 (стрэнд β11), а через молекулу H2O – c атомом азота основной цепи Glu180 спирали α5. Один из атомов кислорода β-фосфата связан с атомом азота амидогруппы Asn79, который находится между стрэндом β10 и спиралью α5, а через молекулу воды – с OE2_Glu182 (α5) и Nε_Lys38 (α1) (рис. 6). Почти все а.о., непосредственно участвующие в связывании АДФ в рибокиназах Th. sp. 2.9 и E. coli, инвариантны в обоих белках. Имеется только одна замена. Со второй оксигруппой рибозного кольца в рибокиназе Th. sp. 2.9 связан остаток Asp272, а в рибокиназе E. coli соответствующую позицию занимает His279.
Особенности пространственной структуры рибокиназы Th. sp. 2.9. Несмотря на значительное сходство пространственных структур рибокиназы Th. sp. 2.9 и E. coli, оба фермента различаются четвертичной структурой. Фермент из E. coli, как и большинство рибокиназ с известной пространственной структурой, функционирует как димер, в котором субъединицы связаны посредством взаимодействия малых доменов – их β-стрэнды образуют связь, называемую β-claspstructure [34]. В кристаллах рибокиназы E. сoli (пр. гр. Р6122) в независимой части элементарной ячейки содержится один мономер, а димер, являющийся функционально активной формой фермента, образуется при действии оси второго порядка. Граница раздела между молекулами димера формируется вследствие взаимодействий между β-слоями малых доменов. Упакованные почти перпендикулярно друг другу, они образуют сплющенный баррель. Такая структура была названа β-claspstructure [34]. Непосредственно в катализе обе субъединицы димера функционируют независимо, однако при переходе фермента в закрытое состояние вторая субъединица димера закрывает часть активного центра, где связывается рибоза, что обеспечивает надежную изоляцию активного центра от растворителя во время реакции. Анализ расположения молекул в независимой части элементарной ячейки рибокиназы Th. sp. 2.9 показывает, что связь между протомерами через малые домены отсутствует. Однако анализ кристаллической упаковки, проведенный с использованием сервиса PISA [35], показывает, что димер может быть образован путем объединения субъединиц A и D после применения оператора –x, y – 1/2, –z + 1 или путем объединения субъединиц B и C после применения оператора x – 1, y, z. Данные димеры структурно аналогичны (рис. 7), площади контактирующих поверхностей, как и энергия формирования комплекса, согласно сервису PISA близки (3810 Å2 и –28.9 ккал/моль и 3410 Å2 и –29.9 ккал/моль соответственно), однако количество водородных связей различается (табл. 2). Четыре молекулы рибокиназы Th. sp. 2.9, расположенные в независимой части элементарной ячейки, несколько различаются взаимным расположением большого и малого доменов. В молекулах А, В, С, D расстояние между Сα-атомами а.о. Gly100 малого домена и Val241 большого домена равно 7.42, 12.33, 18.1 и 5.71 Å соответственно. Оба а.о. расположены на противоположных участках полипептидной цепи, ограничивающей активный центр, и могут характеризовать степень его открытости. Большая подвижность а.о. малого домена в рибокиназе Th. sp. 2.9 может вносить вклад в различия кинетических параметров между рибокиназами Th. sp. 2.9 и E. coli, как это показано в [22].
Рис. 7.
Молекулы рибокиназы, построенные из мономеров D, A (а) и B, C (б). В активных центрах мономеров показаны молекулы АДФ.
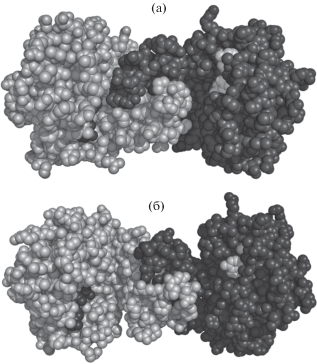
Таблица 2.
Контакты между мономерами D и А, связанными оператором симметрии –x, y – 1/2, –z + 1, и мономерами B и C, связанными оператором симметрии x – 1, y, z
| Мономер D (x, y, z) | Мономер A (–x, y – 1/2, –z + 1) | Расстояние, Å |
|---|---|---|
| N_GLU24 | OE1_GLN103 | 3.19 |
| OH_TYR31 | O_VAL106 | 2.72 |
| N_ILE104 | O_GLU24 | 3.24 |
| N_VAL106 | O_VAL26 | 3.04 |
| O_ARG18 | N_VAL106 | 3.44 |
| O_PRO20 | N_ILE104 | 3.28 |
| OH_TYR31 | N_ALA107 | 3.71 |
| OH_TYR31 | N_VAL106 | 3.70 |
| O_ASN102 | N_GLY23 | 2.57 |
| OE1_GLN103 | NE2_GLN103 | 2.71 |
| O_ILE104 | N_VAL26 | 3.11 |
| O_VAL106 | N_GLY28 | 2.68 |
| Мономер B (x, y, z) | Мономер C (x – 1, y, z) | Расстояние, Å |
| N_VAL26 | O_ILE104 | 3.36 |
| N_GLY28 | O_VAL106 | 3.05 |
| OE1_GLN103 | NE2_GLN103 | 2.83 |
| O_GLU24 | N_ILE104 | 3.68 |
Работа выполнена при поддержке ОКР “МКС (Наука)” в рамках Федеральной космической программы 2016–2025 в части решения и уточнения структуры белка, а также Министерства науки и высшего образования РФ в рамках Государственного задания ФНИЦ “Кристаллография и фотоника” РАН в части анализа пространственной структуры.
Список литературы
Mathews I., Erion M.D., Ealick S.E. // Biochemistry. 1998. V. 37. P. 15607. https://doi.org/10.1021/bi9815445
Schumacher M.A., Scott D.M., Mathews I.I. et al. // J. Mol. Biol. 2000. V. 296. P. 549. https://doi.org/10.1006/jmbi.1999.3474
Schimmel S.D., Hoffee P., Horecker B.L. // Arch. Biochem. Biophys. 1974. V. 164. P. 560. https://doi.org/10.1016/0003-9861(74)90067-8
Andersson C., Mowbray S. // J. Mol. Biol. 2002. V. 315. P. 409. https://doi.org/10.1006/jmbi.2001.5248
Furlong C.E. // Methods Enzymol. 1982. V. 90. P. 467. https://doi.org/10.1016/S0076-6879(82)90173-2
Park J., Gupta R.S. // Cell. Mol. Life Sci. 2008. № 65. P. 2875. https://doi.org/10.1007/s00018-008-8123-1
Anderson A., Cooper R.A. // Biochim. Biophys. Acta. 1969. V. 177. P. 163. https://doi.org/10.1016/0304-4165(69)90081-6
Lopilato J.E., Garwin J.L., Emr S.D. et al. // J. Bacteriol. 1984. V. 158 P. 665. https://doi.org/10.1128/JB.158.2.665-673.1984
Switzer R.L. // J. Biol. Chem. 1969. V. 244. P. 2854.
Becker M.A., Raivio K.O., Bakay B. et al. // J. Clin. Invest. 1980. V. 65. P. 109. https://doi.org/10.1172/JCI109640
Sigrell J.A., Cameron A.D., Jones T.A., Mowbray S.L. // Structure. 1998. V. 6. P. 183. https://doi.org/10.1016/s0969-2126(98)00020-3
Chuvikovsky D.V., Esipov R.S., Skoblov Y.S. et al. // Bioorg. Med. Chem. 2006. V. 14. P. 6327. https://doi.org/10.1016/j.bmc.2006.05.057
Miroshnikov A.I., Esipov R.S., Muravyova T.I. et al. // Open Conf. Proc. J. 2010. V. 1. P. 98.
Fateev I.V., Antonov K.V., Konstantinova I.D. et al. // Beilstein J. Org. Chem. 2014. V. 10. P. 1657. https://doi.org/10.3762/bjoc.10.173
Stepchenko V.A., Seela F., Esipov R.S. et al. // Synth. Lett. 2012. V. 23(10). P. 1541. https://doi.org/10.1055/s-0031-129067916
Konstantinova I.D., Antonov K.V., Fateev I.V. et al. // Synthesis. 2011. V. 10. P. 1555. https://doi.org/10.1055/s-0030-1260010
Mikhailopulo I.A., Miroshnikov A.I. // Biotechnol. Acta. 2013. V. 6. P. 3.
Mikhailopulo I.A., Miroshnikov A.I. // Acta Naturae. 2010. V. 2. P. 6. https://doi.org/10.32607/20758251-2010-2-2-36-58
Zhou X., Mikhailopulo I.A., Cruz-Bournazou N., Neubauer P. // J. Mol. Catalysis B: Enzymatic. 2015. V. 115. P. 119. https://doi.org/10.1016/j.molcatb.2015.02.009
Zhou X., Szeker K., Jiao L.-Y. et al. // Adv. Synthesis Catalysis. 2015. V. 357. № 6. P. 1237. https://doi.org/10.1002/adsc.201400966
Esipov R.S., Abramchik Yu.A., Fateev I.V. et al. // Acta Naturae. 2016. V. 8. № 3 (30). P. 160.
Esipov R.S., Abramchik Yu.A., Fateev I.V. et al. // Biofarm. Z. 2016. V. 8(2). P. 3.
Абрамчик Ю.А., Тимофеев В.И., Муравьева Т.И. и др. // Кристаллография. 2016. Т. 61. № 6. С. 940. https://doi.org/10.7868/S0023476116060023
Battye T.G., Kontogiannis L., Johnson O. et al. // Acta Cryst. D. 2011. V. 67. № 4. P. 271. ttps://doi.org/https://doi.org/10.1107/S0907444910048675
Long F., Vagin A.A., Young P., Murshudov G.N. // Acta Cryst. D. 2008. V. 64. № 1. P. 125. https://doi.org/10.1107/S0907444907050172
Paul R., Patra M.D., Sen U. // Adv. Exp. Med. Biol. 2015. V. 842. P. 293. https://doi.org/10.1107/S2053230X18000109
Murshudov G.N., Vagin A.A., Dodson E.J. // Acta Cryst. D. 1997. V. 53. P. 240. https://doi.org/10.1107/S0907444996012255
Emsley P., Cowtan K. // Acta Cryst. D. 2004. V. 60. P. 2126. https://doi.org/10.1107/S0907444904019158
Куранова И.П., Смирнова Е.А., Абрамчик и др. // Кристаллография. 2011. Т. 56. № 5. С. 944. https://doi.org/10.1134/S1063774511050154
Sigrell J.A., Cameron A.D., Mowbray S.L. // J. Mol. Biol. 1999. V. 290. P. 1009. https://doi.org/10.1006/jmbi.1999.2938
Gatreddi S., Vasudevan D., Qureshi I.A. // Int. J. Biol. Macromol. 2019. V. 136. P. 253. https://doi.org/10.1016/j.ijbiomac.2019.06.001
Rossmann M.G., Moras D., Olsen K.W. // Nature. 1974. V. 250. P. 194199.
Jargiello P. // Biochim. Biophys. Acta. 1982. V. 698. P. 78. https://doi.org/10.1016/0167-4781(82)90187-7
Chothia C., Janin J. // Biochemistry. 1982. V. 21. P. 3955. https://doi.org/10.1021/bi00260a009
Krissinel E., Henrick K. // J. Mol. Biol. 2007. V. 372. P. 774. https://doi.org/10.1016/j.jmb.2007.05.022
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография