Кристаллография, 2023, T. 68, № 6, стр. 959-970
Поиск новых потенциальных Т- и В-клеточных эпитопов в спайковом белке SARS-CoV-2
И. А. Колесников 1, В. И. Тимофеев 1, 2, М. В. Николенко 1, А. В. Ермаков 1, А. С. Ивановский 2, *, Ю. А. Дьякова 1, Ю. В. Писаревский 2, 1, М. В. Ковальчук 1, 2
1 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
2 Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника РАН”
Москва, Россия
* E-mail: a.1wanowskiy@gmail.com
Поступила в редакцию 19.04.2023
После доработки 15.06.2023
Принята к публикации 09.08.2023
- EDN: YEZWPQ
- DOI: 10.31857/S0023476123600106
Аннотация
Современная эпидемиологическая обстановка, в том числе новый вирус SARS-CoV-2 и его высокая мутагенность, требуют принципиально новых сроков создания вакцин, что может быть достигнуто только за счет применения современных вычислительных технологий и моделирования. С использованием методов иммуноинформатики найдены эпитопы в спайковом белке вируса SARS-CoV-2, для найденных эпитопов предсказаны их аллергенность и иммуногенность, показано, что на основе данных эпитопов возможно сконструировать вакцину против SARS-CoV-2.
ВВЕДЕНИЕ
Известно, что SARS-CoV-2 [1] – это вирус, вызывающий чрезвычайно опасное заболевание COVID-19 [2], которое может протекать как в легкой форме [3], так и бессимптомно, а также в крайне тяжелой форме, приводящей к смерти [4]. Существуют подходы к лечению этого заболевания, в том числе ряд вакцин различного типа действия. Однако высокая мутагенность вируса приводит к тому, что старые вакцины становятся менее эффективными или вообще перестают действовать. Таким образом, существует необходимость быстро реагировать на появление новых штаммов SARS-CoV-2.
Накопление большого объема генетической информации в единой базе и развитие биоинформатических инструментов позволяют рассчитывать целевую модель вакцины с учетом прогноза иммунного ответа хозяина и генетической информации патогена. Создание вакцины на основе технологии рекомбинантной ДНК позволяет сократить срок создания вакцины.
НИЦ “Курчатовский институт” создает национальную базу генетической информации, которая включает в себя средства депонирования, поиска по метаданным и гомологического поиска генетической информации, а также средства для управления биоинформатическими платформами Galaxy и NextFlow. Это позволяет работать с существующими и создавать собственные биоинформатические инструменты. В [5, 6] предложен иммуноинформатический подход, который позволяет конструировать основанные на эпитопах белковые вакцины.
В настоящей работе применили модификацию этого подхода для SARS-CoV-2. Одним из последних вариантов коронавируса, вызывающего COVID-19, является так называемый “Омикрон” [7, 8]. Выявлены пять его подвариантов [9]. Известно, что некоторые подварианты “Омикрона” могут заражать ранее вакцинированных людей [10]. Таким образом, создание эффективной вакцины против данного варианта коронавируса является актуальной задачей.
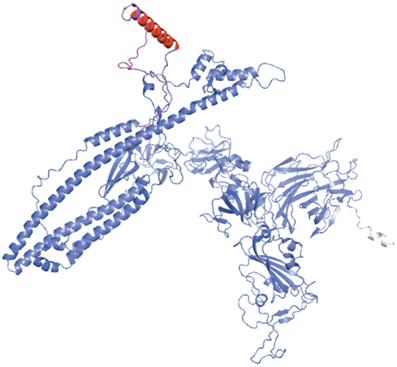
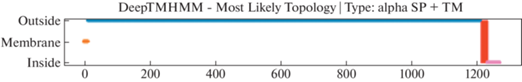
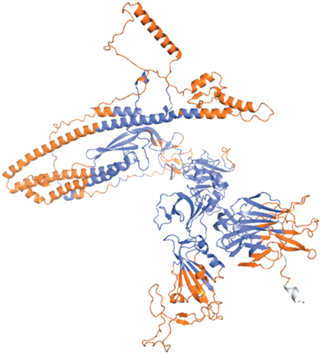
Выбранный для исследования так называемый спайк-протеин варианта “Омикрон” (PDB ID: 7WK3) служит мишенью для поиска вакцин [11]. Трехмерная структура белка приведена на рис. 1, его топология относительно мембраны представлена на рис. 2. На основе смоделированной трехмерной структуры и первичной последовательности исследуемого белка был проведен поиск В- и Т-клеточных эпитопов. Первичная последовательность взята из Национальной базы генетической информации (https://nbgi.ru/).
МАТЕРИАЛЫ И МЕТОДЫ
Т-клеточные эпитопы найдены с помощью программы NetCTL [12]. Сервер позволяет предсказывать эпитопы CTL, ограниченные 12 супертипами главного комплекса гистосовместимости (MHC) класса I. Связывание пептидов с MHC класса I и протеасомное расщепление были предсказаны с помощью искусственных нейронных сетей [12–14].
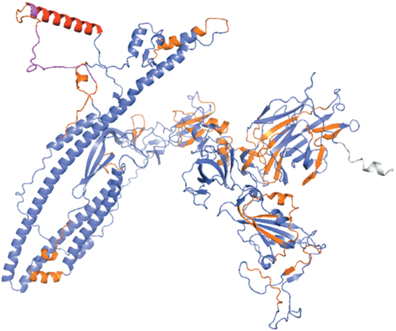
С использованием NetCTL получен прогноз Т-клеточных эпитопов (выделены оранжевым цветом на рис. 3). Настройки, с которыми проводился прогноз, следующие:
– weight of tap – 0.05;
– weight of MHC – 1;
– weight of Cleavage – 0.15;
– порог идентификации эпитопов – 0.75.
B-клеточные эпитопы предсказаны с помощью инструмента прогнозирования ElliPro (http://tools.iedb.org/ellipro/) сервера IEDB [15, 16]. При работе с этим методом используются доступность растворителя и гибкость для прогнозирования эпитопов. Инструмент прогнозирования ElliPro выбирает антигенные остатки из известной трехмерной структуры белка. Нижняя оценка и максимальное расстояние были откалиброваны с использованием значений по умолчанию с minimum score – 0.5 Å и maximum distance – 6 Å соответственно. 3D-модель белка S1 использовали для предсказания как конформационных (прерывистых), так и линейных В-клеточных эпитопов.
Для анализа аллергенности найденных эпитопов использовали программу AllerTOP [17], для анализа токсичности – ToxinPred [18]. Для оценки перспективности данного белка как антигена была применена программа VaxiJen [19].
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Спайковый белок вируса SARS-CoV-2 (СБ CoV) является главной антигенной детерминантой вируса и поэтому был выбран в качестве объекта исследования [11]. Данный белок подвержен мутациям в новых вариантах вируса SARS-CoV-2, что приводит к тому, что разработанные ранее вакцины оказываются неэффективными [20]. Для поиска антигенных детерминант спайкового белка варианта Омикрон вируса SARS-CoV-2 использовали подход, основанный на алгоритмах глубокого машинного обучения [12, 16–18]. Учитывая, что СБ CoV является трансмембранным белком, была определена его топология относительно цитоплазматической мембраны.
Для этого использовали основанный на алгоритмах глубокого машинного обучения веб-сервис DeepTMHMM [21] proteins using deep neural networks. Показано, что аминокислотные остатки 1214–1273 относятся к трансмембранному и внутриклеточному фрагменту белка, что затрудняет их использование для конструирования пептидной вакцины.
Далее провели поиск эпитопов. Выявлены как Т-, так и В-клеточные эпитопы СБ CoV (табл. 1–3). Всего выявлено 37 Т-клеточных эпитопов высокоаффинных лигандов главного комплекса гистосовместимости первого типа (MHC-1). Из них 35 находятся в надмембранной части. Каждый из найденных пептидов состоит из девяти аминокислот, что характерно для лигандов MHC-1. В табл. 1 Т-клеточные эпитопы перечислены в порядке убывания их аффинности к MHC-1. Наиболее аффинные и, следовательно, более многообещающие с точки зрения иммуногенности отличаются от менее аффинных в 4–7 раз. Отметим, что согласно [12] использованный метод позволяет предсказать, является ли пептид высокоаффинным лигандом MHC-1 с вероятностью 95%. В-клеточные эпитопы можно разделить на два типа в зависимости от пространственной структуры эпитопов: непрерывные (линейные) и прерывистые (конформационные) эпитопы [15]. Список линейных В-клеточных эпитопов приведен в табл. 2. Всего найдено 20 линейных В-клеточных эпитопов, все они относятся к надмембранной части СБ CoV. Наиболее иммуногенные из них представляют собой достаточно длинные пептиды (до 97 аминокислотных остатков). Конформационных эпитопов найдено 22 (табл. 3), из них 20 находятся в надмембранном фрагменте белка.
Таблица 1.
Предсказанные Т-клеточные эпитопы
| ID | Первичная последовательность | Афинность | Оценочная функция |
|---|---|---|---|
| 865 | LTDEMIAQY | 0.7953 | 3.6616 |
| 258 | WTAGAAAYY | 0.6735 | 3.1128 |
| 604 | TSNQVAVLY | 0.6559 | 3.0758 |
| 361 | CVADYSVLY | 0.5348 | 2.5759 |
| 746 | STECSNLLL | 0.5136 | 2.3492 |
| 733 | KTSVDCTMY | 0.4908 | 2.3795 |
| 652 | GAEHVNNSY | 0.4042 | 1.996 |
| 160 | YSSANNCTF | 0.3975 | 1.9531 |
| 196 | NIDGYFKIY | 0.3921 | 1.9606 |
| 152 | WMESEFRVY | 0.3902 | 1.9232 |
| 162 | SANNCTFEY | 0.3737 | 1.8739 |
| 687 | VASQSIIAY | 0.3529 | 1.7978 |
| 30 | NSFTRGVYY | 0.3389 | 1.6915 |
| 392 | FTNVYADSF | 0.2704 | 1.3208 |
| 136 | CNDPFLGVY | 0.2613 | 1.3355 |
| 285 | ITDAVDCAL | 0.235 | 1.168 |
| 1237 | MTSCCSCLK | 0.226 | 1.0963 |
| 261 | GAAAYYVGY | 0.2253 | 1.2194 |
| 357 | RISNCVADY | 0.2106 | 1.2032 |
| 465 | ERDISTEIY | 0.2097 | 1.1687 |
| 1039 | RVDFCGKGY | 0.2036 | 1.1403 |
| 1096 | VSNGTHWFV | 0.2012 | 0.9574 |
| 50 | STQDLFLPF | 0.1974 | 1.0468 |
| 343 | NATRFASVY | 0.1955 | 1.1138 |
| 370 | NSASFSTFK | 0.1671 | 0.8165 |
| 880 | GTITSGWTF | 0.1656 | 0.9433 |
| 612 | YQDVNCTEV | 0.1531 | 0.7502 |
| 296 | LSETKCTLK | 0.1515 | 0.7879 |
| 815 | RSFIEDLLF | 0.1421 | 0.8441 |
| 748 | ECSNLLLQY | 0.1413 | 0.8171 |
| 192 | FVFKNIDGY | 0.1358 | 0.7837 |
| 1264 | VLKGVKLHY | 0.1262 | 0.8253 |
| 1095 | FVSNGTHWF | 0.1232 | 0.7622 |
| 628 | QLTPTWRVY | 0.1189 | 0.7887 |
| 372 | ASFSTFKCY | 0.118 | 0.8085 |
| 445 | VGGNYNYLY | 0.1164 | 0.7698 |
| 83 | VLPFNDGVY | 0.113 | 0.7675 |
Таблица 2.
Предсказанные линейные В-клеточные эпитопы
| Эпитоп | Chain | Начало | Конец | Первичная последовательность | Количе-ство а.о. | Score |
|---|---|---|---|---|---|---|
| 1 | A | 1204 | 1273 | GKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCL KGCCSCGSCCKFDEDDSEPVLKGVKLHYT | 70 | 0.9 |
| 2 | A | 435 | 511 | AWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQ AGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVV | 77 | 0.827 |
| 3 | A | 946 | 1017 | GKLQDVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLD KVEAEVQIDRLITGRLQSLQTYVTQQLIRAAE | 72 | 0.821 |
| 4 | A | 730 | 775 | SMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRAL TGIAVEQD | 46 | 0.8 |
| 5 | A | 1 | 23 | MFVFLVLLPLVSSQCVNLTTRTQ | 23 | 0.731 |
| 6 | A | 1123 | 1184 | SGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTS PDVDLGDISGINASVVNIQKEID | 62 | 0.73 |
| 7 | A | 243 | 262 | ALHRSYLTPGDSSSGWTAGA | 20 | 0.725 |
| 8 | A | 809 | 905 | PSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARD LICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWT FGAGAALQIPFAMQMAYR | 97 | 0.706 |
| 9 | A | 67 | 80 | AIHVSGTNGTKRFD | 14 | 0.646 |
| 10 | A | 699 | 711 | LGAENSVAYSNNS | 13 | 0.644 |
| 11 | A | 134 | 166 | QFCNDPFLGVYYHKNNKSWMESEFRVYSSANNC | 33 | 0.636 |
| 12 | A | 336 | 356 | CPFGEVFNATRFASVYAWNRK | 21 | 0.632 |
| 13 | A | 397 | 425 | ADSFVIRGDEVRQIAPGQTGKIADYNYKL | 29 | 0.623 |
| 14 | A | 676 | 686 | TQTNSPRRARS | 11 | 0.618 |
| 15 | A | 1097 | 1102 | SNGTHW | 6 | 0.606 |
| 16 | A | 168 | 188 | FEYVSQPFLMDLEGKQGNFKN | 21 | 0.602 |
| 17 | A | 1073 | 1087 | KNFTTAPAICHDGKA | 15 | 0.58 |
| 18 | A | 121 | 127 | NNATNVV | 7 | 0.551 |
| 19 | A | 209 | 215 | PINLVRD | 7 | 0.538 |
| 20 | A | 97 | 102 | KSNIIR | 6 | 0.53 |
Таблица 3.
Предсказанные конформационные В-клеточные эпитопы
| Эпитоп | Аминокислотный остаток | Количе-ство а.о. | Score |
|---|---|---|---|
| 1 | A:S1239, A:C1240, A:C1241, A:S1242, A:C1243, A:L1244, A:K1245 | 7 | 0.993 |
| 2 | A:T1231, A:M1233, A:L1234, A:C1235, A:C1236, A:M1237, A:T1238 | 7 | 0.988 |
| 3 | A:L1218, A:G1219, A:F1220, A:I1221, A:A1222, A:G1223, A:L1224, A:I1225, A:A1226, A:I1227, A:V1228, A:M1229, A:V1230, A:I1232 | 14 | 0.955 |
| 4 | A:I818, A:G1246, A:C1247, A:C1248, A:S1249, A:C1250, A:G1251, A:S1252, A:C1253, A:C1254, A:F1256, A:D1257, A:E1258, A:D1259, A:D1260, A:S1261, A:E1262, A:P1263, A:V1264, A:L1265, A:K1266, A:G1267, A:V1268 | 23 | 0.906 |
| 5 | A:S1161, A:P1162, A:D1163 | 3 | 0.904 |
| 6 | A:W1212, A:P1213, A:W1214, A:Y1215, A:I1216 | 5 | 0.896 |
| 7 | A:K1269, A:L1270, A:H1271, A:Y1272 | 4 | 0.892 |
| 8 | A:S884, A:G885, A:W886, A:T887 | 4 | 0.885 |
| 9 | A:R457, A:K458, A:S459, A:N460, A:L461, A:K462, A:P463, A:F464, A:E465, A:R466, A:D467, A:I468, A:S469, A:T470, A:E471 | 15 | 0.819 |
| 10 | A:S730, A:M731, A:K733, A:S735, A:V736, A:D737, A:C738, A:T739, A:M740, A:Y741, A:I742, A:C743, A:G744, A:D745, A:S746, A:T747, A:E748, A:C749, A:S750, A:N751, A:L752, A:L753, A:L754, A:Q755, A:Y756, A:G757, A:S758, A:F759, A:C760, A:T761, A:Q762, A:L763, A:N764, A:R765, A:A766, A:L767, A:T768, A:G769, A:I770, A:A771, A:V772, A:E773, A:Q774, A:D775, A:S943, A:G946, A:K947, A:L948, A:Q949, A:D950, A:V951, A:V952, A:N953, A:Q954, A:N955, A:A956, A:Q957, A:A958, A:L959, A:N960, A:T961, A:L962, A:V963, A:K964, A:Q965, A:L966, A:S967, A:S968, A:N969, A:F970, A:G971, A:A972, A:I973, A:S974, A:S975, A:V976, A:L977, A:N978, A:D979, A:I980, A:L981, A:S982, A:R983, A:L984, A:D985, A:K986, A:E988, A:A989, A:E990, A:Q992, A:I993, A:D994, A:R995, A:L996, A:I997, A:T998, A:G999, A:R1000, A:L1001, A:Q1002, A:S1003, A:L1004, A:Q1005, A:Y1007, A:V1008, A:Q1011, A:L1012, A:R1014, A:A1015, A:A1016, A:E1017, A:D1165, A:L1166, A:G1167, A:D1168, A:I1169, A:S1170, A:G1171, A:I1172, A:N1173, A:A1174, A:S1175, A:V1176, A:V1177, A:N1178, A:I1179, A:K1181, A:E1182 | 128 | 0.804 |
| 11 | A:L7, A:L8, A:P9, A:L10 | 4 | 0.793 |
| 12 | A:L1145, A:D1146, A:S1147, A:F1148, A:K1149, A:E1150, A:E1151, A:L1152, A:D1153, A:F1156 | 10 | 0.787 |
| 13 | A:I1130, A:G1131, A:I1132, A:V1133 | 4 | 0.746 |
| 14 | A:C336, A:P337, A:G339, A:E340, A:V341, A:F342, A:N343, A:A344, A:T345, A:R346, A:F347, A:A348, A:S349, A:V350, A:Y351, A:A352, A:W353, A:N354, A:R355, A:K356, A:S371, A:A372, A:S373, A:F374, A:S375, A:A397, A:D398, A:S399, A:F400, A:V401, A:I402, A:R403, A:G404, A:D405, A:E406, A:V407, A:R408, A:Q409, A:I410, A:G416, A:K417, A:I418, A:A419, A:D420, A:Y421, A:Y423, A:A435, A:W436, A:N437, A:S438, A:N439, A:N440, A:L441, A:D442, A:S443, A:K444, A:V445, A:G446, A:G447, A:N448, A:Y449, A:N450, A:Y451, A:L452, A:Y453, A:R454, A:L455, A:F456, A:I472, A:Y473, A:Q474, A:A475, A:G476, A:S477, A:T478, A:P479, A:C480, A:N481, A:G482, A:V483, A:E484, A:G485, A:F486, A:N487, A:C488, A:Y489, A:F490, A:P491, A:L492, A:Q493, A:S494, A:Y495, A:G496, A:F497, A:Q498, A:P499, A:T500, A:N501, A:G502, A:V503, A:G504, A:Y505, A:Q506, A:P507, A:Y508, A:R509, A:V510, A:V511 | 108 | 0.744 |
| 15 | A:L821, A:L822, A:F823, A:K825, A:V826, A:T827, A:L828, A:A829, A:D830, A:A831, A:G832, A:F833, A:I834, A:K835, A:Q836, A:Y837, A:G838, A:D839, A:C840, A:L841, A:G842, A:D843, A:I844, A:A845, A:A846, A:R847, A:D848, A:L849, A:I850, A:C851, A:A852, A:Q853, A:K854, A:F855, A:N856, A:G857, A:L858, A:T859, A:V860, A:L861, A:P862, A:P863, A:L864, A:L865, A:T866, A:D867, A:E868, A:M869, A:I870, A:A871, A:Q872, A:Y873, A:T874, A:S875, A:A876, A:L877, A:L878, A:A879, A:G880, A:T881, A:I882, A:T883, A:G889, A:A890, A:G891, A:A892, A:A893, A:L894, A:I896, A:P897, A:F898, A:A899, A:M900, A:Q901, A:A903, A:Y904, A:R905, A:V911, A:Y1206, A:E1207, A:Q1208, A:Y1209 | 82 | 0.704 |
| 16 | A:N1134, A:N1135, A:T1136 | 3 | 0.674 |
| 17 | A:S12, A:S13, A:Q14, A:C15, A:N17, A:L18, A:T19, A:R21, A:T22, A:A67, A:I68, A:H69, A:V70, A:S71, A:G72, A:T73, A:N74, A:G75, A:T76, A:K77, A:R78, A:F79, A:D80, A:N81, A:K97, A:S98, A:N99, A:I100, A:R102, A:N121, A:N122, A:A123, A:T124, A:N125, A:V127, A:Q134, A:F135, A:C136, A:N137, A:D138, A:P139, A:F140, A:L141, A:G142, A:V143, A:Y144, A:Y145, A:H146, A:K147, A:N148, A:N149, A:S151, A:W152, A:M153, A:E154, A:S155, A:E156, A:F157, A:R158, A:V159, A:Y160, A:S161, A:S162, A:A163, A:N164, A:N165, A:C166, A:T167, A:V171, A:S172, A:Q173, A:P174, A:F175, A:L176, A:M177, A:D178, A:L179, A:E180, A:G181, A:K182, A:Q183, A:G184, A:N185, A:F186, A:K187, A:N188, A:N211, A:L212, A:V213, A:R214, A:D215, A:A243, A:L244, A:H245, A:R246, A:S247, A:Y248, A:L249, A:T250, A:P251, A:G252, A:D253, A:S254, A:S255, A:S256, A:G257, A:W258, A:T259, A:A260, A:G261, A:A262 | 111 | 0.635 |
| 18 | A:S698, A:L699, A:G700, A:A701, A:E702, A:N703, A:S704, A:V705, A:A706, A:Y707, A:S708, A:N709, A:N710, A:S711, A:T716, A:K1073, A:N1074, A:F1075, A:T1076, A:T1077, A:A1078, A:P1079, A:A1080, A:I1081, A:C1082, A:H1083, A:D1084, A:G1085, A:K1086, A:A1087, A:S1097, A:N1098, A:G1099, A:T1100, A:H1101, A:W1102, A:S1123, A:G1124, A:N1125, A:C1126, A:D1127, A:V1128 | 42 | 0.61 |
| 19 | A:N679, A:S680, A:P681, A:A684, A:S686, A:V687 | 6 | 0.56 |
| 20 | A:Q1180, A:I1183, A:D1184 | 3 | 0.552 |
| 21 | A:Q1142, A:P1143, A:E1144 | 3 | 0.551 |
| 22 | A:D808, A:P809, A:S810, A:K811, A:P812, A:S813, A:K814 | 7 | 0.524 |
Оценка аллергенности найденных пептидов с использованием AllerTOP [17] показала, что ни один из них не является аллергеном.
Токсичность найденных пептидов оценивали с использованием ToxinPred [18]. Выявлено, что два Т-эпитопа и два конформационных В-эпитопа, вероятно, являются токсинами (табл. 4–6).
Таблица 4.
Предсказание токсичности Т-клеточных эпитопов
| ID | Белок | SV Score |
|---|---|---|
| 865 | LTDEMIAQY | –0.52 |
| 258 | WTAGAAAYY | –1.07 |
| 604 | TSNQVAVLY | –1.17 |
| 361 | CVADYSVLY | –0.52 |
| 746 | STECSNLLL | –0.52 |
| 733 | KTSVDCTMY | 0.12 |
| 652 | GAEHVNNSY | –0.58 |
| 160 | YSSANNCTF | –0.48 |
| 196 | NIDGYFKIY | –1.13 |
| 152 | WMESEFRVY | –1.68 |
| 162 | SANNCTFEY | –0.49 |
| 687 | VASQSIIAY | –1.09 |
| 30 | NSFTRGVYY | –0.73 |
| 392 | FTNVYADSF | –1.1 |
| 136 | CNDPFLGVY | –0.82 |
| 285 | ITDAVDCAL | –0.67 |
| 1237 | MTSCCSCLK | 0.13 |
| 261 | GAAAYYVGY | –1.17 |
| 357 | RISNCVADY | –0.78 |
| 465 | ERDISTEIY | –1.79 |
| 1039 | RVDFCGKGY | –0.34 |
| 1096 | VSNGTHWFV | –0.92 |
| 50 | STQDLFLPF | –1.49 |
| 343 | NATRFASVY | –0.64 |
| 370 | NSASFSTFK | –1.04 |
| 880 | GTITSGWTF | –1.1 |
| 612 | YQDVNCTEV | –0.82 |
| 296 | LSETKCTLK | –1.22 |
| 815 | RSFIEDLLF | –0.82 |
| 748 | ECSNLLLQY | –0.39 |
| 192 | FVFKNIDGY | –0.75 |
| 1264 | VLKGVKLHY | –1.26 |
| 1095 | FVSNGTHWF | –0.92 |
| 628 | QLTPTWRVY | –1.5 |
| 372 | ASFSTFKCY | –0.66 |
| 445 | VGGNYNYLY | –0.82 |
| 83 | VLPFNDGVY | –1.29 |
Таблица 5.
Предсказание токсичности линейных В-эпитопов
| Эпитоп | Белок | SVM score |
|---|---|---|
| 1 | GKYEQYIKWPWYIWLGFIAGLIAIVMVTIMLCCMTSCCSCLKGCCSCGSCCKF DEDDSEPVLKGVKLHYT | –0.33 |
| 2 | AWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGF NCYFPLQSYGFQPTNGVGYQPYRVV | –0.98 |
| 3 | GKLQDVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDKVEAEVQIDRLIT GRLQSLQTYVTQQLIRAAE | –0.23 |
| 4 | SMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQD | –0.63 |
| 5 | MFVFLVLLPLVSSQCVNLTTRTQ | –1.07 |
| 6 | SGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINA SVVNIQKEID | |
| 7 | ALHRSYLTPGDSSSGWTAGA | –0.58 |
| 8 | PSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVL PPLLTDEM IAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYR | |
| 9 | AIHVSGTNGTKRFD | –1.25 |
| 10 | LGAENSVAYSNNS | –0.61 |
| 11 | QFCNDPFLGVYYHKNNKSWMESEFRVYSSANNC | –0.93 |
| 12 | CPFGEVFNATRFASVYAWNRK | –0.95 |
| 13 | ADSFVIRGDEVRQIAPGQTGKIADYNYKL | –0.48 |
| 14 | TQTNSPRRARS | –1.26 |
| 15 | SNGTHW | –0.71 |
| 16 | FEYVSQPFLMDLEGKQGNFKN | –1.55 |
| 17 | KNFTTAPAICHDGKA | –0.99 |
| 18 | NNATNVV | –0.68 |
| 19 | PINLVRD | –0.90 |
| 20 | KSNIIR | –0.83 |
Таблица 6.
Предсказание токсичности конформационных В-эпитопов
| Эпитоп | Аминокислотный остаток | SVM-Score |
|---|---|---|
| 1 | A:S1239, A:C1240, A:C1241, A:S1242, A:C1243, A:L1244, A:K1245 | –0.14 |
| 2 | A:T1231, A:M1233, A:L1234, A:C1235, A:C1236, A:M1237, A:T1238 | 0.56 |
| 3 | A:L1218, A:G1219, A:F1220, A:I1221, A:A1222, A:G1223, A:L1224, A:I1225, A:A1226, A:I1227, A:V1228, A:M1229, A:V1230, A:I1232 | –0.85 |
| 4 | A:I818, A:G1246, A:C1247, A:C1248, A:S1249, A:C1250, A:G1251, A:S1252, A:C1253, A:C1254, A:F1256, A:D1257, A:E1258, A:D1259, A:D1260, A:S1261, A:E1262, A:P1263, A:V1264, A:L1265, A:K1266, A:G1267, A:V1268 | 1.21 |
| 5 | A:S1161, A:P1162, A:D1163 | –0.80 |
| 6 | A:W1212, A:P1213, A:W1214, A:Y1215, A:I1216 | –0.53 |
| 7 | A:K1269, A:L1270, A:H1271, A:Y1272 | –0.79 |
| 8 | A:S884, A:G885, A:W886, A:T887 | –0.84 |
| 9 | A:R457, A:K458, A:S459, A:N460, A:L461, A:K462, A:P463, A:F464, A:E465, A:R466, A:D467, A:I468, A:S469, A:T470, A:E471 | –1.63 |
| 10 | A:S730, A:M731, A:K733, A:S735, A:V736, A:D737, A:C738, A:T739, A:M740, A:Y741, A:I742, A:C743, A:G744, A:D745, A:S746, A:T747, A:E748, A:C749, A:S750, A:N751, A:L752, A:L753, A:L754, A:Q755, A:Y756, A:G757, A:S758, A:F759, A:C760, A:T761, A:Q762, A:L763, A:N764, A:R765, A:A766, A:L767, A:T768, A:G769, A:I770, A:A771, A:V772, A:E773, A:Q774, A:D775, A:S943, A:G946, A:K947, A:L948, A:Q949, A:D950, A:V951, A:V952, A:N953, A:Q954, A:N955, A:A956, A:Q957, A:A958, A:L959, A:N960, A:T961, A:L962, A:V963, A:K964, A:Q965, A:L966, A:S967, A:S968, A:N969, A:F970, A:G971, A:A972, A:I973, A:S974, A:S975, A:V976, A:L977, A:N978, A:D979, A:I980, A:L981, A:S982, A:R983, A:L984, A:D985, A:K986, A:E988, A:A989, A:E990, A:Q992, A:I993, A:D994, A:R995, A:L996, A:I997, A:T998, A:G999, A:R1000, A:L1001, A:Q1002, A:S1003, A:L1004, A:Q1005, A:Y1007, A:V1008, A:Q1011, A:L1012, A:R1014, A:A1015, A:A1016, A:E1017, A:D1165, A:L1166, A:G1167, A:D1168, A:I1169, A:S1170, A:G1171, A:I1172, A:N1173, A:A1174, A:S1175, A:V1176, A:V1177, A:N1178, A:I1179, A:K1181, A:E1182 | |
| 11 | A:L7, A:L8, A:P9, A:L10 | –1.00 |
| 12 | A:L1145, A:D1146, A:S1147, A:F1148, A:K1149, A:E1150, A:E1151, A:L1152, A:D1153, A:F1156 | –1.34 |
| 13 | A:I1130, A:G1131, A:I1132, A:V1133 | –0.76 |
| 14 | A:C336, A:P337, A:G339, A:E340, A:V341, A:F342, A:N343, A:A344, A:T345, A:R346, A:F347, A:A348, A:S349, A:V350, A:Y351, A:A352, A:W353, A:N354, A:R355, A:K356, A:S371, A:A372, A:S373, A:F374, A:S375, A:A397, A:D398, A:S399, A:F400, A:V401, A:I402, A:R403, A:G404, A:D405, A:E406, A:V407, A:R408, A:Q409, A:I410, A:G416, A:K417, A:I418, A:A419, A:D420, A:Y421, A:Y423, A:A435, A:W436, A:N437, A:S438, A:N439, A:N440, A:L441, A:D442, A:S443, A:K444, A:V445, A:G446, A:G447, A:N448, A:Y449, A:N450, A:Y451, A:L452, A:Y453, A:R454, A:L455, A:F456, A:I472, A:Y473, A:Q474, A:A475, A:G476, A:S477, A:T478, A:P479, A:C480, A:N481, A:G482, A:V483, A:E484, A:G485, A:F486, A:N487, A:C488, A:Y489, A:F490, A:P491, A:L492, A:Q493, A:S494, A:Y495, A:G496, A:F497, A:Q498, A:P499, A:T500, A:N501, A:G502, A:V503, A:G504, A:Y505, A:Q506, A:P507, A:Y508, A:R509, A:V510, A:V511 | |
| 15 | A:L821, A:L822, A:F823, A:K825, A:V826, A:T827, A:L828, A:A829, A:D830, A:A831, A:G832, A:F833, A:I834, A:K835, A:Q836, A:Y837, A:G838, A:D839, A:C840, A:L841, A:G842, A:D843, A:I844, A:A845, A:A846, A:R847, A:D848, A:L849, A:I850, A:C851, A:A852, A:Q853, A:K854, A:F855, A:N856, A:G857, A:L858, A:T859, A:V860, A:L861, A:P862, A:P863, A:L864, A:L865, A:T866, A:D867, A:E868, A:M869, A:I870, A:A871, A:Q872, A:Y873, A:T874, A:S875, A:A876, A:L877, A:L878, A:A879, A:G880, A:T881, A:I882, A:T883, A:G889, A:A890, A:G891, A:A892, A:A893, A:L894, A:I896, A:P897, A:F898, A:A899, A:M900, A:Q901, A:A903, A:Y904, A:R905, A:V911, A:Y1206, A:E1207, A:Q1208, A:Y1209 | |
| 16 | A:N1134, A:N1135, A:T1136 | –0.80 |
| 17 | A:S12, A:S13, A:Q14, A:C15, A:N17, A:L18, A:T19, A:R21, A:T22, A:A67, A:I68, A:H69, A:V70, A:S71, A:G72, A:T73, A:N74, A:G75, A:T76, A:K77, A:R78, A:F79, A:D80, A:N81, A:K97, A:S98, A:N99, A:I100, A:R102, A:N121, A:N122, A:A123, A:T124, A:N125, A:V127, A:Q134, A:F135, A:C136, A:N137, A:D138, A:P139, A:F140, A:L141, A:G142, A:V143, A:Y144, A:Y145, A:H146, A:K147, A:N148, A:N149, A:S151, A:W152, A:M153, A:E154, A:S155, A:E156, A:F157, A:R158, A:V159, A:Y160, A:S161, A:S162, A:A163, A:N164, A:N165, A:C166, A:T167, A:V171, A:S172, A:Q173, A:P174, A:F175, A:L176, A:M177, A:D178, A:L179, A:E180, A:G181, A:K182, A:Q183, A:G184, A:N185, A:F186, A:K187, A:N188, A:N211, A:L212, A:V213, A:R214, A:D215, A:A243, A:L244, A:H245, A:R246, A:S247, A:Y248, A:L249, A:T250, A:P251, A:G252, A:D253, A:S254, A:S255, A:S256, A:G257, A:W258, A:T259, A:A260, A:G261, A:A262 | |
| 18 | A:S698, A:L699, A:G700, A:A701, A:E702, A:N703, A:S704, A:V705, A:A706, A:Y707, A:S708, A:N709, A:N710, A:S711, A:T716, A:K1073, A:N1074, A:F1075, A:T1076, A:T1077, A:A1078, A:P1079, A:A1080, A:I1081, A:C1082, A:H1083, A:D1084, A:G1085, A:K1086, A:A1087, A:S1097, A:N1098, A:G1099, A:T1100, A:H1101, A:W1102, A:S1123, A:G1124, A:N1125, A:C1126, A:D1127, A:V1128 | –0.72 |
| 19 | A:N679, A:S680, A:P681, A:A684, A:S686, A:V687 | –0.43 |
| 20 | A:Q1180, A:I1183, A:D1184 | –0.80 |
| 21 | A:Q1142, A:P1143, A:E1144 | –0.84 |
| 22 | A:D808, A:P809, A:S810, A:K811, A:P812, A:S813, A:K814 | –0.78 |
Отметим, что оба конформационных В-эпитопа и один из Т-эпитопов относятся к трансмембранному и внутриклеточному фрагментам белка. Алгоритмы, использованные для предсказания токсичности и аллергенности, разработаны с применением глубокого машинного обучения. Для алгоритма предсказания токсичности использовали три выборки: 8233 токсичных и 8233 нетоксичных пептида, 1924 токсичных и 1924 нетоксичных пептида, 1924 токсичных и 19240 нетоксичных пептидов. Выборки разделяли на тестовую и обучающую в соотношении 80:20 [22 ] . Такая модель позволила достичь высокой степени достоверности результата [22 ] . Модель для предсказания аллергенности имела обучающую выборку из 2427 аллергенных и столько же неаллергенных пептидов, что позволило получить достоверность предсказания выше 90% [17].
Полученные результаты позволят как сконструировать вакцину к данному варианту SARS-CoV-2, так и использовать предложенный подход в случае необходимости разработки вакцин к новым вариантам SARS-CoV-2.
ЗАКЛЮЧЕНИЕ
Предсказаны B- и Т-клеточные эпитопы спайкового белка SARS-CoV-2. Проведена оценка их иммуногенности, аллергенности и токсичности. Показано, что они не являются аллергенами, большинство из них не являются токсинами. Таким образом, большинство найденных эпитопов могут быть использованы при конструировании прототипа пептидной вакцины против SARS-CoV-2.
Работа выполнена при финансовой поддержке Министерства науки и высшего образования РФ в рамках Федеральной научно-технической программы развития синхротронных и нейтронных исследований и исследовательской инфраструктуры на 2019–2027 гг. (Соглашение № 075-15-2021-1355 (12 октября 2021 г.)
Список литературы
Coronaviridae Study Group of the International Committee on Taxonomy of Viruses // Nat Microbiol. 2020. V. 5 (4). P. 536. https://doi.org/10.1038/s41564-020-0695-z
Beeching N.J., Fletcher T.E., Flower R. // BMJ Best Practice Coronavirus Disease (COVID-19).
Heymann D.L., Shido N. // Lancet. 2020. V. 395 (10224). P. 542. https://doi.org/10.1016/S0140-6736(20)30374-3
Clinical management of severe acute respiratory infection when novel coronavirus (2019-nCoV) infection is suspected: interim guidance, WHO, 2020, 28 January.
Araf Y., Moin A.T., Timofeev V.I. et al. // Front. Immunol. 2022. V. 13. 863234. https://doi.org/10.3389/fimmu.2022.863234
Abass O.A., Timofeev V.I., Sarkar B. et al. // J. Biomolecular Structure and Dynamics. 2021. V. 40 (16). P. 7283. https://doi.org/10.1080/07391102.2021.1896387
Quarleri J., Galvan V., Delpino M.V. // GeroScience. 2022. V. 44 (1). P. 53. https://doi.org/10.1007/s11357-021-00500-4
Gowrisankar A., Priyanka T.M., Banerjee S. // Eur. Phys. J. Plus. 2022. V. 137. P. 100. https://doi.org/10.1140/epjp/s13360-021-02321-y
Yao L., Zhu K. L., Jiang X. L. et al. // Lancet. Infectious Diseases. 2022. V. 22. № 8. P. 1116. https://doi.org/10.1016/S1473-3099(22)00410-8
Cao Y., Yisimayi A., Jian F. et al. // Nature. 2022. V. 608 (7923). P. 593. https://doi.org/10.1038/s41586-022-04980-y
Gallagher T.M., Buchmeier M.J. // Virology. 2001. V. 279. № 2. P. 371.
Larsen M.V., Lundegaard C., Lamberth K. et al. // BMC Bioinformatics. 2007. V. 8. 424. https://doi.org/10.1186/1471-2105-8-424
Buus S., Lauemoller S.L., Worning P. et al. // Tissue Antigens. 2003. V. 62. № 5. P. 378.
Potocnakova L., Bhide M., Pulzova L.B. // J. Immunol. Res. 2016. 6760830. https://doi.org/10.1155/2016/6760830
Ponomarenko J., Bui H.H., Li W. et al. // BMC Bioinformatics. 2008. V. 9. 514. https://doi.org/10.1186/1471-2105-9-514
Dimitrov I., Bangov I., Flower D.R., Doytchinova I. // J. Mol. Model. 2014. V. 20. 2278. https://doi.org/10.1007/s00894-014-2278-5
Gupta S., Kapoor P., Chaudhary K. et al. // PLoS ONE. 2013 V. 8 (9). № 73957. https://doi.org/10.1371/journal.pone.0073957
Doytchinova I.A., Flower D.R. // BMC Bioinformatics. 2007. V. 8 (4). https://doi.org/10.1186/1471-2105-8-4
Kudriavtsev A.V., Vakhrusheva A.V., Novoseletsky V.N. et al. // Viruses. 2022. V. 14 (8). 1603. https://doi.org/10.3390/v14081603
Hallgren J., Konstantinos D.T., Pedersen M.D. et al. // bioRxiv. 2022. https://doi.org/10.1101/2022.04.08.487609
Sharma N., Naorem L.D., Jain S., Raghava G.P.S. // Briefings in bioinformatics. 2022. V. 23. № 5. 174. https://doi.org/10.1093/bib/bbac174
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография