Микробиология, 2019, T. 88, № 1, стр. 45-52
Филогенетическое происхождение α-глюкозидаз MAL и IMA международной генетической линии Saccharomyces cerevisiae S288C
Г. И. Наумов a, А. Н. Боровкова a, b, А. В. Шнырева b, Е. С. Наумова a, *
a Государственный научно-исследовательский институт генетики и селекции промышленных микроорганизмов Национального исследовательского центра “Курчатовский институт”
117545 Москва, Россия
b Московский государственный университет имени М.В. Ломоносова, кафедра микологии и альгологии
119991 Москва, Россия
* E-mail: lena_naumova@yahoo.com
Поступила в редакцию 18.07.2018
После доработки 27.08.2018
Принята к публикации 02.10.2018
Аннотация
Принимая во внимание общепринятую концепцию полной дупликации генома (ПДГ) у дрожжей рода Saccharomyces, проведен сравнительный анализ множественных α-глюкозидаз MAL и IMA генетической линии Saccharomyces cerevisiae S288C и α-глюкозидаз протоплоидных (геномы которых не прошли дупликации) дрожжей родов Kluyveromyces и Lachancea. Показано, что только некоторые изоформы MAL и IMA дрожжей Kluyveromyces и Lachancea находятся в близком филогенетическом родстве с α-глюкозидазами MAL12, MAL32 и IMA1–IMA4 дрожжей S. cerevisiae S288C, тогда как другие более близки дивергентной IMA5. Полученные результаты согласуются с концепцией ПДГ, согласно которой дрожжи Saccharomyces, Kluyveromyces и Lachancea возникли от общего протоплоидного предка и поэтому могут иметь общие близкородственные α-глюкозидазы MAL и IMA. Уровень сходства аминокислотных последовательностей изомальтаз IMA1–IMA4 дрожжей S. cerevisiae S288C и изомальтаз L. dasiensis, L. fantastica, L. fermentati, L. lanzarotensis, L. meyersii, L. quebecensis, L. thermotolerans составляет 75–100%, мальтазы MAL этих же видов идентичны на 75–99%. При этом α-глюкозидазы MAL и IMA независимо дивергировали в каждом роде, виде и даже штамме.
Генетическая линия Saccharomyes cerevisiae S288C была первым эукариотическим организмом, у которого определили полную нуклеотидную последовательность генома (Goffeau et al., 1996), который в настоящее время является международным ресурсом (Saccharomyces Genome Database, SGD htpp://www.yeast-genome.org). Гены, представленные в геноме этих дрожжей, приняты в качестве референсных для распознавания и изучения генов различных эукариотических организмов.
Генетика ферментации α-глюкозида мальтозы, важной для производства хлеба, кваса, пива, пищевого и технического спирта, имеет большое фундаментальное и прикладное значение. Так, у эукариотических микроорганизмов опероноподобная структура впервые была обнаружена на примере мальтозных полимерных (множественных) локусов MAL, состоящих из одного регуляторного и двух структурных генов: пермеазы мальтозы и α-глюкозидазы мальтазы (Needleman et al., 1984; Наумов, Юркевич, 1985).
Биохимический и генетический анализы показали, что существуют два типа родственных α‑глюкозидаз у дрожжей S. cerevisiae (семейство GH13, международная классификация CAZy http://www.cazy.org). Один тип (мальтаза, К.Ф. 3.2.1.20) отвечает за гидролиз и ферментацию α‑1,4-глюкозидов (мальтозы и туранозы), а второй (изомальтаза/α-метилглюкозидаза, К.Ф. 3.2.1.10) – за гидролиз и ферментацию α-1,6-глюкозидов (α‑метилглюкозида и изомальтозы) (Наумов, Наумов, 2012; Deng et al., 2014). Оба фермента имеют также общие субстраты: сахарозу и паранитрофенил-α-D-глюкопиранозид. Международный проект (Goffeau et al., 1996) по секвенированию и аннотированию генома генетической линии S. cerevisiae S288C позволил обнаружить, наряду с известными мальтазными генами MAL12 и MAL32, новое близкородственное им семейство изомальтазных генов IMA1–IMA5 (Наумов, Наумов, 2010; Brown et al., 2010; Teste et al., 2010). Уровень идентичности аминокислотных последовательностей известных мальтаз MAL12, MAL32, MAL62 и изомальтаз IMA1–IMA4 не превышает, соответственно, 99 и 92%, а между собой они имеют 71% идентичных аминокислотных остатков. Изомальтаза IMA5 имеет только 60–66% идентичных аминокислотных остатков с представителями обоих типов α-глюкозидаз.
В литературе имеются только неполные данные об эволюции α-глюкозидаз, депонированных в международных генетических базах (Наумов, Наумов, 2010; Naumoff, 2010; Brown et al., 2010; Teste et al., 2010). Цель нашего сообщения – установить происхождение α-глюкозидаз IMA и MAL дрожжей S. cerevisiae S288C.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Изученные штаммы дрожжей и их происхождение приведены в табл. 1. Поиск гомологичных α-глюкозидаз (мальтаз и изомальтаз) у изученных видов дрожжей проводили в базе данных GenBank (http://www.ncbi.nlm.nih.gov/genbank/) с помощью программы BLAST. В качестве запроса использовали полную аминокислотную последовательность мальтазы MAL12 (регистрационный номер в GenBank YGR292W) дрожжей S. cerevisiae S288C длиной 589 а.о. Частичные последовательности белков не использовали.
Таблица 1.
Происхождение изученных штаммов дрожжей
| Штамм | Вид | Источник выделения | α-Глюкозидазы (IMA, MAL) |
Название белка или регистрационный номер в GenBank |
|---|---|---|---|---|
| Saccharomyces | ||||
| S288C | S. cerevisiae | Генетическая линия | IMA1 | YGR287C |
| IMA2 | YOL157C | |||
| IMA3 | YIL172C | |||
| IMA4 | YJL221C | |||
| IMA5 | YJL216C | |||
| MAL32 | YBR299W | |||
| MAL12 | YGR292W | |||
| Kluyveromyces | ||||
| NRRL Y-1140 | K. lactis | Сливки, США | MAL | KLLA0D00231p |
| MAL | KLLA0_B00242g | |||
| CBS 2104 | K. dobzhanskii | Drosophila pseudoobscura, США | MAL | CDO93416.1 |
| Lachancea | ||||
| CBS 10888 | L. dassiensis | Листья папоротника Angiopterisly godiifolia, Тайвань | IMA | LADA_0G16754g1_1 |
| IMA | LADA_0F00320g1_1 | |||
| MAL | LADA_0G16688g1_1 | |||
| MAL | LADA_0H04346g1_1 | |||
| CBS 6924 | L. fantastica | Почва, Южная Африка (Претория) | IMA | LAFA_0F00782g1_1 |
| MAL | LAFA_0E00188g1_1 | |||
| MAL | LAFA_0D17568g1_1 | |||
| CBS 6772 | L. fermentati | Безалкогольные напитки, Южная Корея | IMA | LAFE_0G01134g1_1 |
| IMA | LAFE_0A00254g1_1 | |||
| MAL | LAFE_0G01090g1_1 | |||
| CBS 3082 | L. kluyveri | Drosophila pinicola, США | IMA | SAKL0A00154p |
| IMA | SAKL0C00176p | |||
| MAL | SAKL0A05698p | |||
| MAL | SAKL0A05654p | |||
| MAL | SAKL0C02112p | |||
| CBS 6340 | L. thermotolerans | Кондитерские изделия, консервы Мирабель, Россия | IMA | KLTH0B00308p |
| MAL | KLTH0E17006p | |||
| MAL | KLTH0G19470p | |||
| MAL | KLTH0H05324p | |||
| CBS 8951 | L. meyersii | Морская вода (мангровые заросли), Багамские острова | IMA | LAME_0A00232g1_1 |
| MAL | LAME_0D11342g1_1 | |||
| MAL | LAME_0C08394g1_1 | |||
| CBS 11611 | L. nothofagi | Сокотечение Nothofagus, Аргентина | IMA | LANO_0G18272g1_1 |
| MAL | LANO_0F01354g1_1 | |||
| CBS 11717 | L. mirantina | Кашаса (“Бразильский ром”), Бразилия | IMA | LAMI_0F07580g1_1 |
| MAL | LAMI_0B00122g1_1 | |||
| MAL | LAMI_0F07646g1_1 | |||
| CBS 12615 | L. lanzarotensis | Виноград, Канарские острова, Испания |
IMA | LALA0_S01e00672g |
| MAL | LALA0_S01e19174g | |||
| MAL | LALA0_S13e02894g | |||
| CBS 14138 | L. quebecensis | Кора клeна, Канада | IMA | LAQU0S17e02674g1_1 |
| MAL | LAQU0S06e05864g1_1 | |||
| MAL | LAQU0S02e00122g1_1 | |||
| MAL | LAQU0S02e11166g1_1 | |||
| Debaryomyces | ||||
| CBS 767 | D. hansenii | Неизвестно | MAL | DEHA2A13882p |
| MAL | DEHA2E00528p | |||
| Scheffersomyces | ||||
| CBS 6054 | Schef. stipitis | Фруктовые деревья, личинки насекомых, Франция | IMA | ABN65252.1 |
| MAL | ABN67312.1 | |||
| MAL | ABN67767.1 | |||
| MAL | ABN66628.1 | |||
| MAL | ABN64883.1 | |||
| Schizosaccharomyces | ||||
| CBS 7264 | Schi. pombe | Виноградный сок, Швеция | MAL | NP_595063.1 |
Для определения генетического родства изученных видов дрожжей был применен филогенетический анализ нуклеотидных последовательностей домена D1/D2 26S рДНК (600 п.н.). Множественные выравнивания изученных нуклеотидных и аминокислотных последовательностей проводили вручную, используя программу BioEdit (http:// www.mbio.ncsu.edu/BioEdit/bioedit.html). Филогенетические деревья строили методом ближайших соседей (Neighbor-Joining) в программе MEGA 6 (Tamura et al., 2013).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Обнаружение множественных родственных α‑глюкозидаз IMA и MAL позволило изучать их филогенетику. Была определена суммарная копийность этих генов в геномах различных дрожжей: S. cerevisiae – 8, Lachances (Saccharomyces) kluyveri – 5, Scheffersemyces (Pichia) stipitis – 5, Lachancea (Kluyveromyces) thermotolerans – 4, Kluyveromyces lactis – 3, Debaryomyces hansenii – 2, Candida albicans – 2 и Schizosaccharomyces pombe – 1 (Brown et al., 2010; Naumoff, 2010; Teste et al., 2010). У различных штаммов S. cerevisiae в разном количестве и комбинациях были идентифицированы пять мальтазных генов: MAL12, MAL22, MAL32, MAL42 и MAL62 (Needleman et al., 1984). Филогенетическое родство α-глюкозидаз дрожжей L. thermotolerans и L. kluyveri, наиболее важных и близких S. cerevisiae, не было изучено. Показано только, что одна и две α-глюкозидазы, соответственно, L. thermotolerans и L. kluyveri, содержат в субстрат-специфичном диагностическом сайте трипептид Val216-Gly-Ser (Teste et al., 2010). Последний характерен для изомальтаз, тогда как для мальтаз – Thr-Ala-Gly (Наумов, Наумов, 2010; Yamamoto et al., 2004). Сайт-направленный мутагенез подтвердил важность остатка Val216 для субстратной специфичности дрожжевой изомальтазы (Yamamoto et al., 2004).
На рис. 1 представлено построенное нами филогенетическое древо аминокислотных последовательностей α-глюкозидаз MAL и IMA. Для сравнительного анализа мы взяли известные мальтазы MAL12 (регистрационный номер в GenBank YGR292W), MAL32 (YBR299W) и изомальтазы IMA1–IMA5 (соответственно, YGR287C, YOL157C, YIL172C, YJL221C, YJL216C) дрожжей S. cerevisiae S288C, а также α-глюкозидазы их ближайших и дальних родственников. Диагностические сайты Val216-Gly-Ser и Thr-Ala-Gly позволили дифференцировать изомальтазы и мальтазы всех изученных дрожжей, кроме дивергентных мальтаз, которые представлены у Schi. pombe (NP_595063.1) и D. hansenii (DEHA2E00528p). Филогенетический анализ аминокислотных последовательностей α-глюкозидаз продемонстрировал существование нескольких изолированных кластеров (рис. 1). Отдельное положение на древе занимают мальтазы представителей неблизкородственных S. cerevisiae родов дрожжей: Schi. pombe, D. hansenii и Schef. stipitis. Обратим внимание, что D. hansenii обладает двумя дивергентными мальтазами: 46% идентичности. Уровень сходства аминокислотных последовательностей мальтазы DEHA2A13882p с мальтазами дрожжей Schef. stipitis составляет 70–72%, тогда как мальтаза DEHA2E00528p более сходна с мальтазой филогенетически неродственных дрожжей Schi. pombe.
Рис. 1.
Филогенетическое древо α-глюкозидаз дрожжей Saccharomyces cerevisiae S288C и родственных им видов родов Lachancea (L.) и Kluyveromyces (K.). Приводятся значения бутстрепа не менее 50%. Шкала соответствует 100 заменам на 1000 аминокислотных остатков. В скобках указан регистрационный номер α-глюкозидаз в GenBank. Изомальтазы, в отличие от мальтаз, помечены жирным шрифтом. В качестве внешней группы использованы мальтазы дрожжей Debaryomyces hansenii и Schizosaccharomyces pombe.
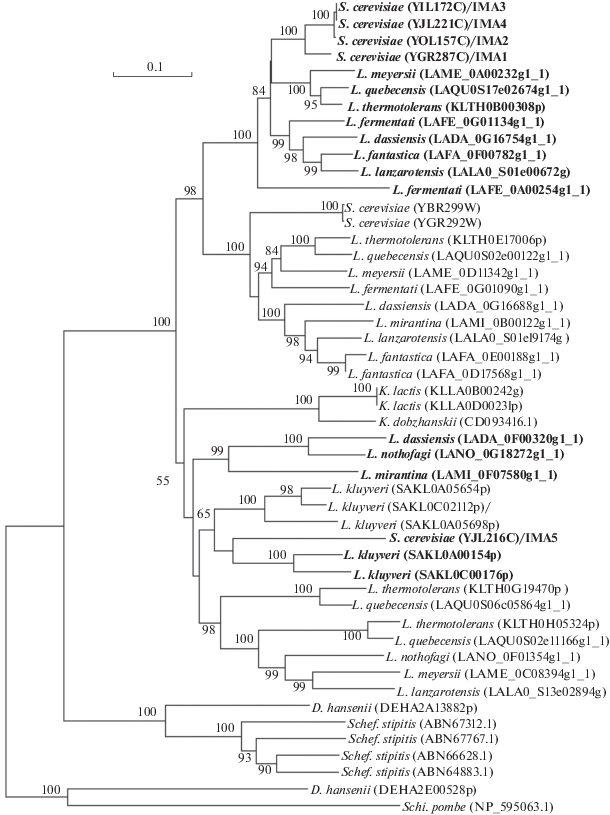
α-Глюкозидазы S. cerevisiae, K. lactis, K. dobzhanskii и десяти видов Lachancea образовали отдельный кластер со 100%-ной достоверностью. Среди α-глюкозидаз этих дрожжей выделяется хорошо обособленный кластер (бутстреп 98%), который включает два субкластера также с высокими поддержками (100%). Первый объединяет изомальтазы IMA1–IMA4 дрожжей S. cerevisiae S288C и изомальтазы L. dasiensis, L. fantastica, L. fermentati, L. lanzarotensis, L. meyersii, L. quebecensis, L. thermotolerans, уровень сходства аминокислотных последовательностей которых составляет 75–100%. Во втором субкластере объединились мальтазы MAL этих 7 видов и L. mirantina, идентичные на 75–99%. Сходство указанных изомальтаз и мальтаз ниже: 68–72%.
В гетерогенной группе (бутстреп 55%), наряду с мальтазами MAL и изомальтазами IMA дрожжей K. lactis, K. dobzhanskii, L. dasiensis, L. kluyveri, L. lanzarotensis, L. meyersii, L. mirantina, L. nothofagi, L. quebecensis, и L. thermotolerans, мы обнаруживаем изомальтазу IMA5 дрожжей S. cerevisiae S288C. В целом, уровень идентичности входящих в эту группу α‑глюкозидаз значительно ниже, чем в первом кластере (57–88%). Наибольшее сходство имеют изомальтазы (SAKL0A00154p, SAKL0C00176p) и мальтазы (SAKL0A05698p, SAKL0A05698p и SAKL0C02112p) дрожжей L. kluyveri, соответственно, 88 и 73–79%. Сопоставимый уровень сходства имеет также изомальтаза IMA5 S. cerevisiae и две изомальтазы L. kluyveri: 70–72%. Наиболее дивергентными оказались изомальтазы LADA_0F00320g1_1 дрожжей L. dasiensis, L. nothofagi (LANO_0G18272g1_1) и L. mirantina (LAMI_0F07580g1_1), аминокислотные последовательности которых идентичны на 73–83%, а уровень сходства с остальными α-глюкозидазами этого кластера составил 57–64%. Необходимо отметить, что у первых двух изомальтаз в диагностическом триплете Val216-Gly-Ser произошла замена Ser на Gly: Val216-Gly-Gly, тогда как у LAMI_0F07580g1_1 – произошла замена Gly на Ala: Val216-Ala-Gly. Следует отметить, что все обнаруженные α-глюкозидазы L. fantastica попали в первый кластер, тогда как α-глюкозидазы L. kluyveri, L. nothofagi, K. lactis и K. dobzhanskii обнаружены только во втором. α-Глюкозидазы остальных восьми изученных видов Lachancea распределились между двумя кластерами.
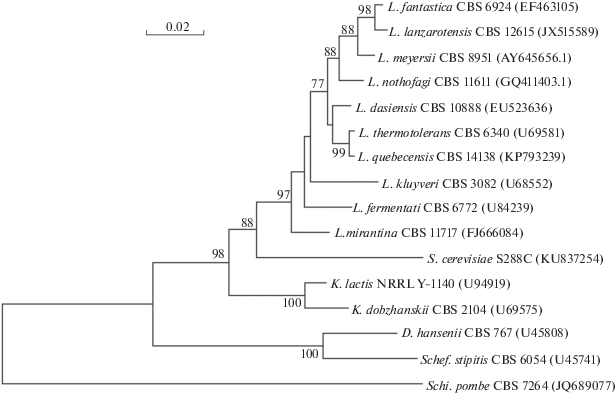
Современная классификация аскомицетовых дрожжей основана на филогенетическом анализе ряда молекулярных маркеров, прежде всего домена D1/D2 гена 26S рДНК (около 600 п.н.). Обширная компьютерная база данных GenBank по нуклеотидным последовательностям домена D1/D2 широко используется для определения таксономического положения новых штаммов. На рис. 2 представлено филогенетическое древо нуклеотидных последовательностей домена D1/D2 26S рДНК анализируемых дрожжей. Со 100%-ной достоверностью выделяется кластер, объединяющий дрожжи S. cerevisiae S288C и 10 видов рода Lachancea, K. lactis NRRL Y-1140 и K. dobzhanskii CBS 2104. Их дальние родственники, Schi. pombe, D. hansenii и Schef. stipitis, занимают отдельное положение на филогенетическом древе. Следует отметить, что топологии деревьев, построенных на основании аминокислотных последовательностей α-глюкозидаз и нуклеотидных последовательностей домена D1/D2 хорошо согласуются (рис. 1 и 2).
Рис. 2.
Филогенетическое древо нуклеотидных последовательностей домена D1/D2 26S рДНК дрожжей Saccharomyces cerevisiae S288C и родственных их родов Lachancea (L.) и Kluyveromyces (K.). Приводятся значения бутстрепа не менее 70%. Шкала соответствует 20 заменам на 1000 нуклеотидных остатков. В скобках указан регистрационный номер D1/D2-последовательностей в GenBank. В качестве внешней группы использованы дрожжи Schizosaccharomyces pombe.
Ранее был показан природный межвидовой перенос пектиназных генов PGU из S. cerevisiae в S. bayanus, из S. paradoxus в S. cerevisiae (Наумов и соавт., 2016), а также генов SUC, MAL, RTM и Y'-последовательности из S. cerevisiae в S. bayanus (Naumova et al., 2005, 2011). Кроме того, было обнаружено перемещение генов PGU у родственных дрожжей Galactomyces citri-aurantii, Geotrichum klebahii и Galactomyces candidus (Шаламитский, Наумов, 2017). Возможность межвидовых переносов генов, очевидно, обусловлена наличием общей системы типов спаривания у видов одного рода, позволяющей им скрещиваться в любой комбинации (Наумов и соавт., 2009; Наумов, 2015). В нашем случае такой механизм передачи генов MAL и IMA у представителей разных родов исключается. Виды разных родов не могут скрещиваться, а их геномы рекомбинировать. Известно только феромонное межродовое взаимодействие дрожжей разных родов, например, между S. cerevisiae и L. kluyveri (McCullough, Herskowitz, 1979; Наумов, Пишкур, 1999).
Таким образом, нахождение близкородственных α-глюкозидаз у представителей разных родов дрожжей, очевидно, можно объяснить только общим происхождением соответствующих родов дрожжей. В настоящее время хорошо обоснована концепция полной дупликации геномов в ходе эволюции некоторых родов дрожжей, в том числе видов рода Saccharomyces, тогда как у видов протоплоидных родов Lachancea (viz. L. kluyveri, L. dasiensis, L. fantastica, L. fermentati и L. thermotolerans) и Kluyveromyces (viz. K. lactis) она не проходила (Kellis et al., 2004; Scannell et al., 2007; Souciet et al., 2009; Dujon, Louis, 2017). Причем, полная дупликация восьми предковых хромосом у дрожжей S. cerevisiae произошла уже после их расхождения с дрожжами Lachancea (Souciet et al., 2009; Dujon, Louis, 2017). Гаплоидное число хромосом у дрожжей Saccharomyces равно 16, тогда как у видов Lachancea равно восьми. Полученные нами результаты указывают на то, что изомальтазы и мальтазы образовались у общего протоплоидного предка дрожжей родов Saccharomyces, Lachancea и Kluyveromyces, т.е. до их расхождения и до полной дупликации генома Saccharomyces. Затем в каждом роде, виде и даже штамме происходила дивергенция своих α-глюкозидаз, имеющих как IMA, так и MAL активности.
Созданные и идентифицированные изогенные генетические линии дрожжей S. cerevisiae (Mortimer, Johnston, 1986; Наумов и соавт., 1994) до сих пор обеспечивают проведение исследований по генетике, геномике и эволюции дрожжей рода Saccharomyces, а также являются основой изучения других аскомицетовых дрожжей.
Авторы признательны Д.Г. Наумову за помощь в работе. Исследование выполнено при финансовой поддержке Российского фонда фундаментальных исследований (проект № 17-04-00309).
Список литературы
Наумов Г.И. Дрожжи Komagataella – генетический род согласно межвидовой гибридизации // Микробиология. 2015. Т. 84. № 4. С. 449–455. Naumov G.I. The yeast Komagataella: a genetic genus in accordance with interspecies hybridization // Microbiology (Moscow). 2015. V. 84. P. 538–543.
Наумов Д.Г., Наумов Г.И. Обнаружение нового семейства α-глюкозидазных генов IMA у дрожжей Saccharomyces cerevisiae // ДАН. 2010. Т. 432. № 4. С. 549–551.
Naumoff D.G., Naumov G.I. Discovery of novel family of α-glucosidase genes IMA genes in yeast Saccharomyces cerevisiae // Dokl. Biochem. Biophys. 2010. V. 432. № 1. P. 114–116.
Наумов Г.И., Наумов Д.Г. Молекулярно-генетическая дифференциация α-глюкозидаз дрожжей: мальтазы и изомальтазы // Микробиология. 2012. Т. 81. № 3. С. 301–305.
Naumov G.I., Naumoff D.G. Molecular genetic differentiation of yeast α-glucosidases: maltases and isomaltases // Microbiology (Moscow). 2012. V. 81. P. 278–282.
Наумов Г.И., Кондратьева В.И., Наумова Е.С. Таксономическая генетика дрожжей рода Zygowilliopsis // Генетика. 2009. Т. 45. № 12. С. 1609–1615.
Naumov G.I., Kondratieva V.I., Naumova E.S. Taxonomic genetics of Zygowilliopsis yeasts // Russian J. Genet. 2009. V. 45. P. 1422–1427.
Наумов Г.И., Никоненко Т.А., Кондратьева В.И. Таксономическая идентификация сахаромицетов Дрожжевого генетического центра Калифорнийского университета // Генетика. 1994. Т. 30. № 1. С. 45–48.
Naumov G.I., Nikonenko T.A., Kondrat’eva V.I. Taxonomic identification of Saccharomyces from the yeast genetic stock center of the University of the California // Russian J. Genet. 1994. V. 30. P. 38–41.
Наумов Г.И., Пишкур Ю. О феромонной активности музейных штаммов дрожжей Saccharomyces sensu lato // Микробиология. 1999. Т. 68. № 6. С. 860–863.
Naumov G.I., Piskur J. Pheromone activity of collection strains of Saccharomyces sensu lato yeasts // Microbiology (Moscow). 1999. V. 68. P. 759–762.
Наумов Г.И., Шаламитский М.Ю., Мартыненко Н.Н., Наумова Е.С. Молекулярная филогения пектиназных генов PGU дрожжей рода Saccharomyces // Микробиология. 2016. Т. 85. № 6. С. 703–712.
Naumov G.I., Shalamitskiy M.Yu., Martynenko N.N., Naumova E.S. Molecular phylogeny of pectinase genes PGU in the yeast genus Saccharomyces // Microbiology (Moscow). 2016. V. 85. P. 734–743.
Наумов Г.И., Юркевич В.В. Опероноподобная система дрожжей // Вест. Моск. ун-та. Сер. 16. Биология. 1985. Т. 40. № 3. С. 40–42.
Шаламитский М.Ю., Наумов Г.И. Филогенетический анализ пектиназ аскомицетных дрожжей // Биотехнология. 2017. Т. 33. № 6. С. 28–36.
Shalamitskiy M.Yu., Naumov G.I. Phylogenetic analysis of pectinases of ascomycetous yeasts // Appl. Biochem. Microbiol. (Moscow). 2018. V. 54. № 7.
Brown C.A., Murray A.W., Verstepen K.J. Rapid expansion and functional divergence of subtelomeric gene families in yeast // Curr. Biol. 2010. V. 20. P. 895–903.
Deng X., Petitjean M., Teste M.-A., Kooli W., Tranier S., François J.M., Parrou J.L. Similarities and differences in the biochemical and enzymological properties of the four isomaltases from Saccharomyces cerevisiae // FEBS Open Bio. 2014. V. 4. P. 200–212.
Dujon B.A., Louis E.J. Genome diversity and evolution in the budding yeasts (Saccharomycotina) // Genetics. 2017. V. 206. № 2. P. 717–750.
Goffeau A., Barrell B.G., Bussey H., Davis R.W., Dujon B., Feldmann H., Galibert F., Hoheisel J.D., Jacq C., Johnston M., et al. Life with 6000 genes // Science. 1996. V. 274. № 5287. P. 546–567.
Kellis M., Birren B.W., Lander E.S. Proof and evolutionary analysis of ancient genome duplication in the yeast Saccharomyces cerevisiae // Nature. 2004. V. 428. P. 617–624.
McCullough J., Herskowitz I. Mating pheromones of Saccharomyces kluyveri: pheromone interactions between Saccharomyces kluyveri and Saccharomyces cerevisiae // J. Bacteriol. 1979. V. 138. P. 146–154.
Mortimer R.K., Johnston J.R. Genealogy of principal strains of the yeast genetic stock center // Genetics. 1986. V. 113. P. 35–43.
Naumoff D.G. Sequence-based classification of yeast glycoside hydrolases: CAZy vs. Génolevures // III Межд. Конф. “Математическая биология и биоинформатика”. Пущино (10–15 октября 2010 г.). М.: Макс-Пресс, 2010. С. 139–140.
Naumova E.S., Naumov G.I., Masneuf-Pomarède I., Aigle M., Dubourdieu D., Molecular genetic study of introgression between Saccharomyces bayanus and S. cerevisiae // Yeast. 2005. V. 22. P. 1099–1115.
Naumova E.S., Naumov G.I., Michailova Y.V., Martynenko N.N., Masneuf-Pomarède I. Genetic diversity study of the yeast Saccharomyces bayanus var. uvarum reveals introgressed subtelomeric Saccharomyces cerevisiae genes // Res. Microbiol. 2011. V. 162. P. 204–213.
Needleman R.B., Kaback D.B., Dubin R.A., Perkins E.L., Rosenberg N.G., Sutherland A., Forrest D.B., Michels C.A. MAL6 of Saccharomyces: a complex genetic locus containing three genes required for maltose fermentation // Proc. Natl. Acad. Sci. USA. 1984. V. 81. P. 2811–2815.
Scannell D.R., Butler G., Wolfe K.H. Yeast genome evolution – the origin of the species // Yeast. 2007. V. 24. P. 929–942.
Souciet J.-L., Dujon B., Gaillardin C. et al. Comparative genomics of protoploid Saccharomycetaceae // Genome Res. 2009. V. 19. P. 1696–1709. doi 10.1101/gr.091546.109
Tamura K., Stecher G., Peterson D., Filipski A., Kumar S. MEGA6: molecular evolutionary genetics analysis version 6.0. // Mol. Biol. Evol. 2013. V. 30. P. 2725–2729.
Teste M.-A., François J.M., Parrou J.-L. Characterization of a new multigene family encoding isomaltases in the yeast Saccharomyces cerevisiae, the IMA family // J. Biol. Chem. 2010. V. 285. P. 26815–26824.
Yamamoto K., Nakayama A., Yamamoto Y., Tabata S. Val 216 decides the substrate specificity of α-glucosidase in Saccharomyces cerevisiae // Eur. J. Biochem. 2004. V. 271. № 16. P. 3414–3420.
Дополнительные материалы отсутствуют.


