Микробиология, 2019, T. 88, № 3, стр. 328-335
Результаты генетического анализа пробиотического штамма Escheriсhia coli M-17 и потенциально пробиотического штамма Escheriсhia coli ВМ
И. В. Белова a, А. Г. Точилина a, *, И. В. Соловьева a, Е. И. Ефимов a, И. С. Горлова a, Т. П. Иванова a, В. А. Жирнов a
a Нижегородский научно-исследовательский институт эпидемиологии и микробиологии имени академика
И.Н. Блохиной Роспотребнадзора
603000 Нижний Новгород, Россия
* E-mail: lab-lb@yandex.ru
Поступила в редакцию 16.10.2018
После доработки 17.01.2019
Принята к публикации 01.02.2019
Аннотация
Изучены последовательности геномов двух представителей рода Escheriсhia: производственного штамма E. coli M-17 и вновь выделенного высокотехнологичного штамма E. coli BM. Подтверждено что штамм E. coli M-17 обладает характерным для вида биохимическим профилем, показано, что в его геноме не представлены трансмиссивные гены антибиотикорезистентности, гены патогенности, вирулентности и интегрированные плазмиды. Впервые был определен сиквенс-тип штамма ‒ ST141. Обнаружено, что штамм E. coli ВM обладает сниженной ферментативной активностью, в его геноме выявлены детерминанты антибиотикорезистентности (strA, strB, sul2, aadA1), ряд детерминант патогенности и вирулентности, транспозон Tn7, фрагменты встроенных плазмид – IncFIB и Col156, установлена принадлежность штамма к десятому сиквенс-типу – ST-10. Полученные данные свидетельствуют, что E. coli M-17 отвечает всем современным требованиям, предъявляемым к штаммам-продуцентам пробиотиков, тогда как выявленные особенности генома штамма E. coli ВM не позволяют рассматривать его как производственно-перспективный и использовать для создания пробиотических препаратов, несмотря на то, что штамм обладает хорошими технологическими характеристиками. Полногеномные последовательности штаммов E. coli M-17 и E. coli ВM депонированы в Международной базе данных GenBank под номерами NZ_LBDD00000000 и NZ_LBDC00000000 соответственно.
Для производства колисодержащих пробиотиков в России используется штамм Escherichia coli М‑17, автором которого является Л.Г. Перетц (Перетц, 1955). Данный штамм характеризуется широким спектром полезных свойств и используется в биотехнологии уже более 70 лет, однако обладает относительно невысокой технологичностью, что выражается в низкой скорости накопления биомассы. Это обусловливает необходимость поиска альтернативных производственно перспективных штаммов этого вида, пригодных для использования в технологических целях. В настоящей работе представлены результаты исследования геномов двух штаммов рода Escherichia – традиционно использующегося при производстве пробиотиков E. coli M-17 и нового штамма E. coli BM, выделенного из кишечника здорового человека и характеризующегося хорошей технологичностью.
Согласно современным нормативным документам безопасность штаммов-продуцентов пробиотиков должна быть подтверждена отсутствием у них трансмиссивных генов антибиотикорезистентности, генов вирулентности, островов патогенности, интегрированных плазмид и транспозонов (МУ № 2.3.2.2789-10, 2010; МУК № 4.2.2602-10, 2011). Биологические свойства и технологические характеристики обоих штаммов были изучены ранее, однако углубленное исследование их геномов не проводилось (Гайдеров, 2007; Ладыгина и соавт., 2009). В рамках настоящей работы было проведено изучение полногеномных последовательностей штаммов с применением следующих ресурсов, доступных в режиме он-лайн: Международной базы данных последовательностей геномов Escherichia и Shigella “EnteroBase” при университете Уорика (Warwick Medical School, UK, http://enterobase.warwick.ac.uk) и сервисов, доступных на сайте Центра геномной эпидемиологии (Center for Genomic Epidemiology, Denmark, http://www.genomicepidemiology.org/). Это позволило проанализировать геномы на наличие детерминант патогенности, антибиотикорезистентности, вирулентности, а также провести их молекулярное типирование с использованием метода мультилокусного сиквенс типирования (MLST).
Цель работы – изучение геномов штамма E. coli M-17 и штамма E. coli BM на наличие детерминант антибиотикорезистентности, патогенности, вирулентности, интегрированных плазмид и транспозонов.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объекты исследования. В работе использовали штаммы Escherichia coli M-17 и E. coli BM. Штамм E. coli M-17 получен из коллекции маточных культур ФГУП НПО “Микроген” Минздрава России, г. Нижний Новгород, Нижегородское предприятие по производству бактерийных препаратов “ИмБио”; штамм зарегистрирован в коллекции Научного центра экспертизы средств медицинского назначения (ГКПМ № 240418). Штамм E. coli BM, полученный из коллекции лаборатории микробиома человека и средств его коррекции ФБУН ННИИЭМ им. академика И.Н. Блохиной Роспотребнадзора, выделен из кишечника здорового человека и охарактеризован как высокотехнологичный. Штаммы получены в лиофильно высушенном состоянии.
Восстановление штаммов проводили с использованием мясо-пептонного бульона “HiMedia” (Индия). Подрощенную культуру титровали в диапазоне разведений 10–1–10–7 и проводили высевы по 0.05 мл на среду Эндо (питательная среда для выделения энтеробактерий – агар Эндо ГРМ, Оболенск, Россия), посевы инкубировали при 37 ± 1°С в течение 24 ч. Выросшие колонии микроорганизмов оценивали по морфологии и отбирали для расширенного изучения биохимических свойств с использованием пластин биохимических, дифференцирующих энтеробактерии (ПБДЭ) “Диагностические системы” (Россия), систем индикаторных бумажных для идентификации микроорганизмов (СИБ) – набор № 2 для межродовой и видовой дифференциации энтеробактерий (АО НПО “Микроген” МЗ РФ, Россия) а также сред “HiMedia” (Индия).
Геномную ДНК выделяли с использованием коммерческого набора QIAamp DNA Mini Kit (“Qiagen”, Германия), фрагментацию производили с использованием системы ультразвуковой фрагментации Covaris E210 (“Applied Biosystems”, США) согласно инструкции производителя. Очистку смеси и отбор фрагментов 300‒500 п.н. проводили при помощи магнитных частиц Agencourt AMP-ure beads (“Beckman Coulter”, США). Для подготовки библиотек использовали набор TrueSeq (“Illumina Inc.”, США), секвенирование выполняли на платформе MiSeq (“Illumina Inc.”, США). Короткие прочтения собирали в контиги с помощью программы CLC Bio v. 8.0 (“Aarhus”, Дания).
Аннотацию генома производили с помощью утилиты Prokka v. 1.11 (Seemann, 2014) и геномного сервера RAST (http://rast.nmpdr.org). Для подробного изучения генома использовали специализированные программные продукты, доступные на сайте Центра геномной эпидемиологии (https:// cge.cbs.dtu.dk/services/). Изучение CRISPR-региона проводили с использованием программы Cri-sprFinder (Grissa et al., 2017), поиск детерминант антибиотикорезистентности и патогенности с использованием ResFinder 2.1 и PathogenFinder (Zankari et al., 2012; Cosentino et al., 2013). Для поиска детерминант вирулентности применяли программу VirulenceFinder, для обнаружения интегрированных плазмид – PlasmidFinder 1.3, установление серотипа проводили с помощью SerotypeFinder 1.1, для изучения геноварианта адгезина FimH применяли сервис FimHTyper (Joensen et al., 2014; Carattoli et al., 2014; Joensen et al., 2015; Roer et al., 2017).
MLST типирование проводили с использованием программы MLST-1.8 Server (Larsen et al., 2012) и базы данных MLST университета Уорика (Mark Achtman Database, http://mlst.warwick.ac.uk/ mlst/dbs/Ecoli/) с помощью анализа последовательностей семи генов “домашнего хозяйства” (Alikhan et al., 2018).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
В результате исследования биохимических свойств штаммов E. coli с помощью стандартных тест-систем ПБДЭ и СИБ, а также дополнительных тестов (ксилоза, рамноза, дульцит и тест на желатиназную активность) с использованием сред HiMedia были выявлены отличия в биохимических профилях штаммов. E. coli M-17 обладает способностью ферментировать сахарозу, мальтозу, сорбит, глюкозу, лактозу, арабинозу, маннит, ксилозу, рамнозу, образует индол, обладает β-галактозидазной активностью, утилизирует цитрат натрия с глюкозой, обладает лизин- и орнитиндекарбоксилазами. Штамм не способен разжижать желатин, утилизировать цитрат и малонат натрия, не обладает аргининдегидролазой, фенилаланиндезаминазой, не образует ацетилметилкарбинол, не ферментирует инозит, дульцит, не гидролизует мочевину, не образует сероводород. Именно такой биохимический профиль характерен для штамма E. coli M-17, предложенного Л.Г. Перетцем в 1930 г. и заявленным в фармакопейной статье на сухой колибактерин (Перетц, 1955). Штамм E. coli BM отличается тем, что не способен ферментировать сахарозу, мальтозу, сорбит, не декарбоксилирует орнитин.
При проведении полногеномного секвенирования E. coli M-17 и E. coli BM было собрано 42 (N50 = 465.806, среднее покрытие 250) и 59 (N50 = 205.755, среднее покрытие 250) контигов соответственно. Дальнейший анализ позволил установить основные характеристики геномов штаммов (табл. 1).
Таблица 1.
Фенотипические и генетические характеристики штаммов E. coli M-17 и E. coli BM
| Штамм | E. coli M-17 | E. coli ВМ | |
|---|---|---|---|
| Биохимическая активность | САХ* | + | ‒ |
| МАЛ | + | ‒ | |
| СОР | + | ‒ | |
| ОРН | + | ‒ | |
| Размер генома, п.н. | 5 105 186 | 5 224 908 | |
| GC, % | 50.7 | 50.7 | |
| Количество генов | 5.107 | 5.559 | |
| CDS (последовательности, кодирующие белки) | 4.930 | 5.094 | |
| CRISPR-регион | 1 | 1 | |
| Опероны синтеза колицинов | Колицин V Contig_14 LBDD01000032 3025_2210 Колицин В Contig_72 NZ_LBDD01000012 40387_41076 |
Колицин V Contig_107 NZ_LBDC01000009 94132_94620 |
|
| Детерминанты антибиотикорезистентности | strB | ‒ | Contig_22 NZ_LBDC01000022.1 5610_6446 |
| aadA1 | ‒ | Contig_61 NZ_LBDC01000028.1 29677_30465 |
|
| strA | ‒ | Contig_22 NZ_LBDC01000022.1 4807_5610 |
|
| sul2 | ‒ | Contig_22 NZ_LBDC01000022.1 3931_4746 |
|
| DfrA1 | ‒ | Contig_61 NZ_LBDC01000028.1 31142_31615 |
|
| mdtABCD claster | Contig_55 LBDD01000001 1044601_1048971 |
Contig_78 LBDC01000013.1 73875_69381 |
|
| MAR locus | Contig_66 LBDD01000004 415943_417243 |
Contig_103 NZ_LBDC01000020 25244_23280 |
|
| Эффлюксные помпы | CmeA CmeB Contig_63 LBDD01000005 431924_436197 TolC Contig_63 LBDD01000005 202366_203847 AcrR Contig_63 LBDD01000005 431525_430863 MacA MacB Contig_57 LBDD01000002 117495_114479 MATE MDR Pump Contig_65 LBDD01000007 225090_223717 MFS Contig_76 LBDD01000003 414925_416151 |
CmeA Contig_32 LBDC01000025 10318_9161 CmeB Contig_56 LBDC01000005 102713_105862 TolC Contig_99 LBDC01000002 186357_184876 MacA Contig_60 NZ_LBDC01000016 47123_46008 MATE MDR pump Contig_100 LBDC01000003 292755_291382 MFS Contig_72 NZ_LBDC01000015 92555_93781 |
|
| Гены вирулентности и патогенности | TieB | ‒ | Contig_73 LBDC01000014 73595.74770 |
| iha | ‒ | Contig_51 LBDC01000012 2522.4612 |
|
| gad | ‒ | Contig_99 LBDC01000002 522096.523496 |
|
| exc1 | ‒ | Contig_22 NZ_LBDC01000022.1 1397.1837 |
|
| Интегрированные плазмиды и мобильные элементы | Транспозон Tn7 | ‒ | Contig_61 NZ_LBDC01000028 19928_27710 |
| IncFIB | ‒ | Contig_46 LBDC01000004 186596_217956 |
|
| Col156 | ‒ | Contig_73 LBDC01000014 68539_69105 |
|
| Серотип | О2:Н6 | О21:Н4 | |
| FimH тип | 14 | 215 | |
| ST | 141 | 10 | |
| Номер в БД GenBank | NZ_LBDD00000000.1 | NZ_LBDC00000000.1 | |
| Номер в БД EnteroBase | ESC_GA8607AA | ESC_IA6791AA | |
Очевидно, что оба штамма обладают сходными размерами геномов, в обоих геномах присутствуют опероны синтеза колицина V, а E. coli М-17 содержит еще дополнительно гены синтеза колицина В.
В геномах обоих штаммов присутствуют CRISPR-регионы, локус CRISPR E. coli M-17 находится в пределах 57 контига (LBDD01000002), а E. coli ВМ ‒ 68 (LBDC01000001). По структурной организации эти детерминанты обоих штаммов можно отнести к I-E типу, что типично для представителей рода Escherichia (Makarova et al., 2015). Отличительной чертой этого типа является наличие гена cas3, который кодирует одноцепочечную ДНК, стимулирующую хеликазу, раскручивающую двухцепочечную ДНК (рис. 1).
Рис. 1.
Организация CRISPR-кассет двух штаммов E. coli. Стрелками обозначены соотвествующие Cas-белки, ромбы обозначают повторы, прямоугольники – уникальные спейсеры.
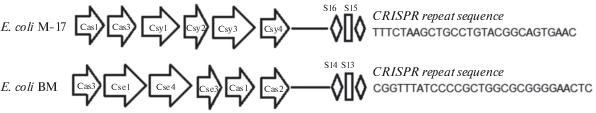
В ряде случаев CRISPR-кассеты микроорганизмов используют для штаммового типирования, так как данная область генома не коррелирует с филогенией, и ее строение может отличаться даже у разных штаммов одного вида (Makarova et al., 2015). Однако в ходе работы мы установили, что хотя CRISPR-локусы обоих штаммов отличаются компоновкой Cas-белков и имеют разные последовательности спейсеров и повторов (рис. 1), их спейсеры не штаммоспецифичны и имеют 100% идентичность с последовательностями ряда других E. coli, представленных в базе данных GenBank.
Геномный контекст метаболизма штаммов изучен с использованием RAST. Установлено, что в геномах детерминированы несколько путей центрального метаболизма углеводов: гликолиз, путь Энтнера‒Дудорова и пентозофосфатный путь. С использованием алгоритма KEGG map (http:// rast.nmpdr.org/seedviewer.cgi) показано, что метаболические пути штаммов не имеют особенностей, однако в геноме штамма E. coli M-17 присутствуют детерминанты, ответственные за утилизацию сахарозы: гены сахарозо-6-фосфат гидролазы (scrB), фосфофруктокиназы (fruK) и соответствующая транспортная система (PTS scrA). В то же время у штамма E. coli ВM данные детерминанты отсутствуют, что соотносится с особенностями его биохимического профиля – штамм не способен ферментировать сахарозу. Кроме того, E. coli ВM не декарбоксилирует орнитин, не ферментирует мальтозу и сорбит, что является его отличительной особенностью, так как более 90% штаммов E. coli утилизируют мальтозу и сорбит, а способность к утилизации сахарозы и орнитина у представителей вида вариабельна (De Vos et al., 2009). Отмечено, что в геноме штамма детерминанты метаболизма мальтозы, сорбита и орнитина имеются, но по какой-то причине не проявляются фенотипически. Подобная “потеря” отдельных детерминант или их “выключение” может быть связана с приспособлением микроорганизма к специализированной экологической нише – кишечнику человека (Равин, Шестаков, 2013). В целом, оба штамма обладают выраженным метаболическим потенциалом, способны синтезировать колицины и утилизировать широкий спектр субстратов. Полногеномные последовательности штаммов были проанализированы на наличие детерминант антбиотикорезистентности, патогенности, вирулентности, наличие интегрированных плазмид и транспозонов. При этом установлено, что в состав обоих геномов входит кластер транспортных генов (mdtABCD), наличие которого обусловливает устойчивость к новобиоцину и доксихолату, гены MAR локуса, отвечающего за систему эффлюкса лекарственных, в том числе и антибактериальных препаратов, а также другие виды молекулярных эффлюксных помп: cmeA, cmeB, tolC, acrR, macA, macB, семейств MATE и MFS. Все перечисленные детерминанты имеют хромосомную локализацию, типичны для представителей рода и не представляют угрозы в плане трансмиссивного распространения (Baranova, Nikaido, 2002; Alekshun, Levy, 2004).
При исследовании штамма E. coli M-17 не было обнаружено детерминант антибиотикорезистентности, способных к трансмиссивному переносу, в то время как в геноме штамма E. coli ВM обнаружен ген устойчивости к сульфаниламидам sul2, который кодирует сульфаниламид устойчивую дигидроптероат синтазу, гены устойчивости к аминогликозидам: ген aadA1, кодирующий аминогликозид-аденилилтрансферазу, гены strB и strA, кодирующие аминогликозидфосфотрансферазы, детерминанту устойчивости к триметоприму dfrA1. В изучаемом геноме детерминанты sul2, strB и strA, находящиеся в 22 контиге, фланкированы белками из семейства транспозаз (WP_001120891.1), а гены aadA1 и dfrA1, расположенные в пределах 61 контига, входят в состав транспозона Tn7, который играет немаловажную роль в переносе генетической информации и распространении генов антибиотикорезистентности (Петрова, 2008).
При изучении детерминант вирулентности и патогенности в геномах обоих штаммов не было обнаружено детерминант, кодирующих энтеротоксины Stx1 и Stx2, термостабильный токсин St, интимины Eae, энтерогемолизины Ehx, которые связаны с клинически значимыми диареегенными штаммами E. coli (Карцев и соавт., 2018). Однако в геноме E. coli BM обнаружены другие значимые детерминанты: ген плазмидного происхождения tieB, также кодирующий энтеротоксин, ген бифункционального адгезина iha, ген глутаматдекарбоксилазы gad и ген, кодирующий белок мобильного переноса Exc1 (табл. 1). По данным сервиса PathogenFinder штамм E. coli BM отнесен к патогенным для человека (score = 0.868). В геноме E. coli M-17 детерминант патогенности и вирулентности не выявлено, штамм охарактеризован как непатогенный (score = 2.0).
С использованием программы PlasmidFinder-1.3 геномы штаммов были проверены на наличие интегрированных плазмид и их фрагментов. Установлено, что геном E. coli M-17 не содержит интегрированных плазмид, а геном E. coli ВM содержит фрагменты двух плазмид – IncFIB, характерной для уропатогенных эшерихий, и Col156. Гены плазмидного происхождения расположены в пределах 46 и 73 контигов и кодируют комплекс белков конъюгативного переноса TraA, TraM, TraY и т.д. (табл. 1).
Далее было изучено наличие и геноварианты гена fimH, детерминирующего маннозочувствительные пили I типа, отвечающие за адгезию клеток E. coli (Маркова и соавт., 2018). Установлено, что оба штамма несут этот ген, причем ген fimH E. coli M-17 принадлежит к 14 типу, а fimH E. coli ВM – к 215. Отдельные исследователи рассматривают наличие fimH как признак, обусловливающий потенциальную вирулентность микроорганизма, связанную с высокими показателями адгезии и биопленкообразованием и, следовательно, непригодность для производственных целей (Нечаева и соавт., 2016; Слукин и соавт., 2017). Однако, согласно современным данным, наличие пилей первого типа и собственно адгезина fimH не всегда связано с проявлением вирулентности и встречается у штаммов E. coli, не обладающих адгезивно-инвазивным фенотипом, причем повышенную способность к адгезии и инвазии авторы связывают с точечными мутациями гена fimH в “горячих точках” (Dreux et al., 2013). В генах fimH исследованных штаммов не обнаружено мутаций, обусловливающих замены G73A/E/R/W и T158A/P и способствующих повышенной адгезии бактериальной клетки на эпителиоцитах кишечника, характерной для адгезивно-инвазивных E. coli (AIEC). Fim-типы 14 и 215, принадлежность к которым обнаружена в ходе исследования, не свойственны патогенным штаммам и достаточно редко встречаются среди популяции E. coli (Roer et al., 2017).
С использованием программного продукта SerotypeFinder-1.1 были проанализированы детерминанты антигенов штаммов E. coli: гены, кодирующие структурный белок флагеллин FliC (KLD44527.1; KLD52182.1) и белки O-антигена: Wzx (KLD49178.1; KLD50213.1) и Wzy (KLD49266.1; KLD50216.1). Установлено, что штамм E. coli M-17 принадлежит к серотипу О2:Н6, а штамм E. coli ВM имеет серотип О21:Н4.
В рамках настоящей работы было проведено MLST-типирование штаммов с использованием схемы, предложенной в университете Уорика. В данной схеме используются фрагменты семи генов “домашнего хозяйства” – гены аденилаткиназы (adk), фумаратгидратазы (fumC), ДНК-гиразы (gyrB), изоцитратдегидрогеназы (icd), малатдегидрогеназы (mdh), аденилосукцинатдегидрогеназы (purA), рекомбиназы А (recA). Установлено, что штамм E. coli M-17 принадлежит к 141 сиквенс-типу (ST-141), а E. coli ВM отнесен к 10 сиквенс-типу (ST-10) (табл. 2).
Таблица 2.
Аллельные профили исследуемых штаммов Eshcerihia
| Штамм | Аллели | ST | ||||||
|---|---|---|---|---|---|---|---|---|
| adk | fumC | gyrB | icd | mdh | purA | recA | ||
| E. coli M-17 | 10 | 11 | 4 | 8 | 8 | 8 | 2 | 141 |
| E. coli ВM | 13 | 52 | 10 | 14 | 17 | 25 | 17 | 10 |
Согласно информации международной базы данных EnteroBase (дата обращения 28.04.2018) 12 штаммов из 24, имеющих серотип О2:Н6, принадлежат к 141 сиквенс-типу; они характеризуются как непатогенные и не связаны с клиническими проявлениями заболеваний.
Штамм E. coli ВM имеет серотип О21:Н4 и отнесен к 10 сиквенс-типу (ST-10). На момент обращения (28.04.2018) в базе EnteroBase зарегистрировано четыре штамма с серотипом О21:Н4, два из которых представлены десятым сиквенс-типом, причем один из этих штаммов человеческого происхождения охарактеризован как энтероагрегативный (EAEC) и ассоциирован с диарейным синдромом. Кроме того, к этому же ST-комплексу (ST complex 10), принадлежат гетеропатогенные штаммы E. coli, проявляющие себя как энтеро- и уропатогены (Карцев и соавт., 2018).
Таким образом, в ходе проведенной работы показано, что штамм E. coli M-17 не содержит детерминант антибиотикорезистентности, вирулентности, интегрированных плазмид, по совокупности данных анализа полногеномной последовательности и молекулярного типирования штамм не является патогенным для человека и может использоваться для производства пробиотиков и БАД к пище.
В свою очередь штамм E. coli ВM обладает комплексом генов антибиотикорезистентности, вирулентности, несет гены мобильных элементов, интегрированные плазмиды и является патогенным для человека. Результаты молекулярного типирования позволили установить его сиквенс-тип – ST-10, и подтвердили принадлежность к эпидемически значимому клону. Полученные данные исключают использование данного штамма как пробиотического в соответствии с современными регламентирующими документами.
Полученные результаты доказывают, что использование полногеномного секвенирования, позволяющего выявить и проанализировать все интересующие исследователей детерминанты и провести молекулярное типирование штамма, является приоритетным при работе с производственно перспективными штаммами.
Список литературы
Гайдеров А.А. Изучение свойств штаммов Escherichia coli M-17 и Bacillus subtilis 1719 на модели экспериментального дисбиоза. Автореферат дис. … канд. мед. наук, 30.05.2007. Москва: Российский университет дружбы народов (РУДН), 2007. 18 с.
Карцев Н.Н., Светоч Э.А., Ершова М.Г., Абросимова Г.Н., Тазина О.И., Пинчук А.С., Фурсова Н.К., Шепелин А.П., Дятлов И.А. Характеристика диареегенных эшерихий, выделенных от детей в возрасте до 5 лет в Ярославле // Клиническая лабораторная диагностика. 2018. Т. 63. № 4. С. 249‒253. https://doi.org/10.18821/0869-2084-63-4-249-253
Ладыгина А.В., Терешкина Н.В., Григорьева Л.В., Чупринина Р.П. Взаимодействие культур Bifidobacterium bifidum штамм 1 и Escherihia coli штамм М-17 в бификоле в опытах in vivo на модели “острой” и “хронической” токсичности // Биопрепараты. Профилактика, диагностика, лечение. 2009. № 1‒2. С. 24‒28.
Маркова Ю.А., Анганова Е.В., Турская А.Л., Быбин В.А., Савилов Е.Д. Регуляция формирования биопленок Escherichia coli // Прикл. биохимия и микробиология. 2018. Т. 54. № 1. С. 3‒15. https://doi.org/10.7868/S0555109918010014
Markova J.A., Turskaya A.L., Bybin V.A., Anganova E.V., Savilov E.D. Regulation of Escherichia coli biofilm formation (review) // Appl. Biochem. Microbiol. 2018. V. 54. P. 3‒15.
Методические указания по контролю биологических и микробиологических факторов. Система предрегистрационного доклинического изучения безопасности препаратов. Отбор, проверка и хранение прозводственных штаммов, используемых при производстве пробиотиков: методические указания № 4.2.2602-10. М.: Роспотребнадзор, 2011. 61 с. [Электронный ресурс]. Режим доступа http://files.stroyinf.ru/Data2/1/ 4293816/4293816271.pdf (дата обращения 31.07.2018).
Методические указания по санитарно-эпидемиологической оценке безопасности и функционального потенциала пробиотических микроорганизмов, используемых для производства пищевых продуктов: методические указания № 2.3.2.2789-10. М.: Роспотребнадзор, 2010. 103 с. [Электронный ресурс]. Режим доступа http://files.stroyinf.ru/Data2/1/4293757/4293757373.pdf (дата обращения 31.07.2018).
Нечаева О.В., Аль-Баяти Б.М., Глинская Е.В., Ульянов В.Ю., Вакараева М.М., Заярский Д.А., Тихомирова Е.И., Беспалова Н.В. Особенности формирования микробных биопленок условно-патогенными штаммами Escherichia coli и разработка способов борьбы с ними // Изв. Самарского научного центра Российской академии наук. 2016. Т. 18. № 2 (3). С. 776‒782.
Перетц Л.Г. Значение нормальной микрофлоры для организма человека. М.: Медгиз, 1955. 410 с.
Петрова М.А., Горленко Ж.М., Соина В.С., Миндлин С.З. Изучение ассоциации генов strA-strB с плазмидами и транспозонами у современных и древних штаммов бактерий // Генетика. 2008. Т. 44. № 9. С. 1281‒1286.
Petrova M.A., Gorlenko Z.M., Mindlin S.Z., Soina V.S. Association of the strA-strB genes with plasmids and transposons in the present-day bacteria and in bacterial strains from permafrost // Russ. J. Genet. 2008. V. 44. P. 1116‒1120.
Равин Н.В., Шестаков С.В. Геном прокариот // Вавиловский журнал генетики и селекции. 2013. Т. 17. № 4/2. С. 972‒984.
Слукин П.В., Асташкин Е.И., Ермоленко З.М., Круглов А.Н. Ершова О.Н., Маликов В.Е., Светоч Э.А., Фурсова Н.К. Разнообразие клинических уропатогенных штаммов Escherichia coli по генотипам вирулентности // Сборник трудов IХ Всеросс. научно-практ. конф. с международным участием “Молекулярная диагностика 2017”. 2017. Т. 2. С. 345‒347.
Alekshun M.N., Levy S.B. The Escherichia coli mar locus ‒ Antibiotic resistance and more. The mar locus and related systems confer multiple antibiotic resistance and control expression of virulence factors and genes for metabolizing small molecules // ASM News. 2004. V. 70. P. 451‒456.
Alikhan N.F., Zhou Z., Sergeant M.J., Achtman M. A genomic overview of the population structure of Salmonella // PLoS Genet. 2018. V. 14. № 4. P. 1‒13 https://doi.org/10.1371/journal.pgen.1007261
Baranova N., Nikaido H. The baeSR two-component regulatory system activates transcription of the yegMNOB (mdtABCD) transporter gene cluster in Escherichia coli and increases its resistance to novobiocin and deoxycholate // J. Bacteriol. 2002. V. 184. P. 4168‒4176. PMID: https://doi.org/12107134
Bergey’s Manual of Systematic Bacteriology / Eds. De Vos P., Garrity G.M., Jones D., Krieg N.R., Ludwig W., Rainey F.A., Schleifer K.-H., Whitman W. Springer, 2009. V. 3. 1450 p.
Carattoli A., Zankari E., García-Fernández A., Voldby Larsen M., Lund O., Villa L., Møller Aarestrup F., Hasman H. In silico detection and typing of plasmids using PlasmidFinder and plasmid multilocus sequence typing // Antimicrob. Agents Chemother. 2014. V. 58. P. 3895‒3903. https://doi.org/10.1128/ AAC.02412-14
Cosentino S., Voldby L. M., Møller A.F., Lund O. PathogenFinder ‒ distinguishing friend from foe using bacterial whole genome sequence data // PLoS One. 2013. V. 8. № 10. e77302. https://doi.org/10.1371/journal.pone.0077302
Dreux N., Denizot J., Martinez-Medina M., Mellmann A., Billig M., Kisiela D., Chattopadhyay S., Sokurenko E., Neut C., Gower-Rousseau C., Colombel J.F, Bonnet R., Darfeuille-Michaud A., Barnich N. Point mutations in FimH adhesin of Crohn’s disease-associated adherent-invasive Escherichia coli enhance intestinal inflammatory response // PLoS Pathog. 2013. V. 9. № 1. https://doi.org/10.1371/journal.ppat.1003141
Grissa I., Vergnaud G., Pourcel C. CRISPRFinder: a web tool to identify clustered regularly interspaced short palindromic repeats // Nucl. Acids Res. 2017. V. 35. P. 52‒57. https://doi.org/10.1093/nar/gkm360
Joensen K., Scheutz F., Lund O. Real-time whole-genome sequencing for routine typing, surveillance, and outbreak detection of verotoxigenic Escherichia coli // J. Clin. Microbiol. 2014. V. 52. P. 1501‒1510. https://doi.org/10.1128/JCM.03617-13
Joensen K.G., Tetzschner A.M., Iguchi A., Aarestrup F.M., Scheutz F. Rapid and easy in silico serotyping of Escherichia coli isolates by use of whole-genome sequencing data // J. Clin. Microbiol. 2015. V. 53. P. 1501‒1510. https://doi.org/10.1128/ JCM.00008-15
Larsen M.V., Cosentino S., Rasmussen S., Friis C., Hasman H., Marvig R.L., Jelsbak L., Sicheritz-Pontén T., Ussery D.W., Aarestrup F.M., Lund O. Multilocus sequence typing of total-genome-sequenced bacteria // J. Clin. Microbiol. 2012. V. 50. P. 1355‒1361. https://doi.org/10.1128/JCM.06094-11
Makarova K.S., Wolf Y.I., Alkhnbashi O.S. An updated evolutionary classification of CRISPR-Cas systems // Nat. Rev. Microbiol. 2015. V. 13. P. 722‒736. https://doi.org/10.1038/nrmicro3569
Roer L., Tchesnokova V., Allesøe R., Muradova M., Chattopadhyay S., Ahrenfeldt J., Thomsen M., Lund O., Hansen F., Hammerum A.M., Sokurenko E., Hasman H. Development of a Web tool for Escherichia coli subtyping based on fimH alleles // J. Clin. Microbiol. 2017. V. 55. P. 2538–2543. https://doi.org/10.1128/JCM.00737-17
Seemann T. Prokka: rapid prokaryotic genome annotation // Bioinformatics. 2014. V. 30. P. 2068‒2069. https://doi.org/10.1093/ bioinformatics/btu153
Zankari E., Hasman H., Cosentino S., Vestergaard M., Rasmussen S., Lund O., Aarestrup F.M., Larsen M.V. Identification of acquired antimicrobial resistance genes // J. Antimicrob. Chemother. 2012. V. 67. P. 2640‒2644. https://doi.org/10.1093/ jac/dks261
Дополнительные материалы отсутствуют.


