Микробиология, 2019, T. 88, № 3, стр. 309-317
Селекция микробного сообщества при формировании кислого шахтного дренажа
В. В. Кадников a, Е. В. Груздев a, Д. А. Ивасенко b, А. В. Белецкий a, А. В. Марданов a, Э. В. Данилова c, О. В. Карначук b, Н. В. Равин a, *
a Институт биоинженерии, ФИЦ Биотехнологии РАН
119071 Москва, Россия
b Томский государственный университет
634050 Томск, Россия
c Институт общей и экспериментальной биологии СО РАН
670047 Улан-Удэ, Россия
* E-mail: nravin@biengi.ac.ru
Поступила в редакцию 28.11.2018
После доработки 09.12.2018
Принята к публикации 10.12.2018
Аннотация
Исследовано изменение состава микробного сообщества при формировании кислого шахтного дренажа на модели двух водоемов, расположенных в открытом карьере по добыче полиметаллических руд “Озерное” в Восточной Сибири. Первый водоем, Bu-18, наполнялся подземными водами, имел нейтральный рН и низкое содержание сульфата и растворенных ионов металлов. Второй водоем, Bu-16, наполнялся водой из Bu-18, проходящей через слой горных пород, содержащих сульфидные минералы, и представлял собой кислый шахтный дренаж с рН 2.85. В воде Bu-16 содержалось 1405 мг/л сульфата, 164 мг/л марганца, 78 мг/л магния и 26 мг/л железа. Молекулярный анализ состава микробных сообществ двух водоемов, проведенный с помощью высокопроизводительного секвенирования фрагментов гена 16S рРНК, показал, что формирование кислого шахтного дренажа сопровождается уменьшением общего разнообразия микроорганизмов и отбором нескольких доминирующих таксономических и функциональных групп. Большую часть сообщества прокариот в Bu-16 составляли хемолитоавтотрофные железо- и серо-окисляющие бактерии Leptospirillum, Acidithiobacillus, Gallionella, Sulfuriferula и Sulfobacillus. Гетеротрофные бактерии Ferrimicrobium и Metallibacterium, способные восстанавливать Fe(III) в анаэробных условиях, присутствовали в минорных количествах. Более 20% сообщества представляли бактерии группы Candidate Phyla Radiation и кандидатного филума Dependentiae (TM6), являющиеся паразитами или симбионтами других микроорганизмов. Эти группы ранее редко находили в кислых шахтных дренажах и лишь в минорных количествах. Среди эукариот в Bu-16 были обнаружены флагелляты рода Spumella, которые могут быть хозяевами Dependentiae.
Дренажные воды с высокой кислотностью, часто называемые кислыми шахтными дренажами (acid mine drainage), образуются в результате окисления пирита (FeS2) и других сульфидных минералов в присутствии кислорода и воды. Скорость окисления пирита увеличивается на порядки за счет активности аэробных микроорганизмов, окисляющих восстановленные соединения железа (Rohwerder et al., 2003; Johnson, Hallberg, 2003). В результате окисления пирита ионами Fe3+ (реакция FeS2 + 14Fe3+ + 8H2O > 15Fe2+ + + $2{\text{SO}}_{4}^{{2 - }}$ + 16H+) образуются протоны и сульфат, что приводит к повышению кислотности. В свою очередь, низкий рН среды способствует растворению содержащих металлы минералов и переходу металлов в раствор. Результатом этих процессов являются воды с высокой кислотностью (рН 0.5‒4), высоким содержанием сульфатов и ионов металлов (железа, цинка, меди, марганца, кадмия и др.), достигающим граммов на литр. Кислые шахтные дренажи характерны для мест складирования отходов добычи металлов, заброшенных шахт, хвостохранилищ обогатительных фабрик, открытых карьеров по добыче руд и т.п.
Высокая кислотность и содержание токсичных ионов металлов в кислых дренажных водах создают экстремальные условия для развития микроорганизмов, ограничивающие разнообразие микробных сообществ (Tyson et al., 2004; Baker, Banfield, 2003; Méndez-García et al., 2015). Однако в этих экосистемах присутствуют как неорганические доноры электронов (в основном Fe2+ и восстановленные соединения серы), так и кислород, что создает условия для роста хемолитоавтотрофных микроорганизмов. В анаэробных зонах, таких как осадки очистных прудов, в качестве акцепторов электронов может выступать Fe3+, а также сульфат и нитрат, а донорами электронов служить как аллохтонные органические соединения, доступные извне, так и первичная продукция, образуемая хемолитоавтотрофами.
Микробные сообщества кислых шахтных дренажей активно исследовали как классическими микробиологическими, так и молекулярными методами, основанными на анализе генов 16S рибосомной РНК (Baker, Banfield, 2003; Méndez-García et al., 2015), а также методами метагеномики, метатранскриптомики и метапротеомики (Tyson et al., 2004; Ram et al., 2005; Chen et al., 2015). Среди хемолитоавтотрофов, которые обычно доминируют в микробных сообществах кислых дренажных вод, чаще всего обнаруживают представителей родов Acidithiobacillus (филум Proteobacteria) и Leptospirillum (филум Nitrospirae), а в условиях умеренно-низких рН (2‒5) – бетапротеобактерии семейства Gallionellaceae (Méndez-García et al., 2015). Гетеротрофная часть микробных сообществ обычно более разнообразна и может включать протеобактерии классов альфа, бета и гамма, а также представителей филумов Firmicutes, Actinobacteria, Acidobacteria и TM7 (Méndez-García et al., 2015). Среди гетеротрофов обнаруживают как бактерии, участвующие в цикле железа и серы (представители родов Acidiphilum, Ferrimicrobium, Sulfobacillus и др.), так и относящиеся к группам, типичным для неэкстремальных экосистем (Xanthomonodales, Sphingomonodales, Burkholderiales и др.).
Большинство исследованных кислых шахтных дренажей являются “открытыми” экосистемами, в которые могут попадать и аллохтонные микроорганизмы, например, со стоком поверхностных вод с окружающих участков местности. При этом в кислом дренаже происходит селекция микробного сообщества, в соответствии с физико-химическими условиями.
Целью настоящей работы было провести сравнительное исследование составов микробных сообществ двух водоемов, расположенных в открытом карьере, подготовленном для добычи полиметаллических руд и выявить специфические для кислого дренажа группы микроорганизмов. Первый из водоемов (Bu-18) питается атмосферными осадками и грунтовыми водами и имеет близкие к нейтральным значения рН. Второй водоем (Bu-16) формируется в результате перетока воды из Bu-18 и, вследствие окисления подстилающих массивов полисульфидных руд, характеризуется низкими значениями рН.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Отбор проб и характеристика химического состава воды. Пробы воды из водоемов Bu-16 и Bu-18 отбирали 11 августа 2016 г. с помощью стерильных пластиковых бутылей. Температуру, рН и окислительно-восстановительный потенциал (Eh) определяли на месте отбора рН-метром HANNA HI 8314F. Воду для анализа содержания ионов (30 мл) и элементного анализа (15 мл) на месте отбора проб пропускали через фильтр с диаметром пор 0.45 мкм. Элементный анализ воды проводили в ООО “Химико-аналитический центр “Плазма” (г. Томск) методом масс-спектрометрии с индуктивно связанной плазмой (ICP-MS), а анализ ионов ‒ на ионном хроматографе.
Выделение метагеномной ДНК, ПЦР-амплификация и секвенирование фрагментов генов 16S рРНК. Для сбора микроорганизмов пробы воды объемом 5 л пропускали через фильтр с диаметром пор 0.22 мкм. Выделение суммарной ДНК из отфильтрованной биомассы осуществляли с использованием набора MO BIO Power Soil Kit (“MO BIO Laboratoties”, Carlsbad, США) в соответствии с инструкцией производителя.
Для ПЦР-амплификации фрагмента гена 16S рРНК, включающего вариабельные участки V3–V6, использовали “универсальные” праймеры U341F (5'-CCT ACG GGR SGC AGC AG) и PRK806R (5'-GGA CTA CYV GGG TAT CTA AT). Полученные ПЦР фрагменты секвенировали на геномном анализаторе GS FLX (“Roche”, Швейцария) по протоколу Titanium с использованием набора реактивов GS FLX Titanium Sequencing Kit XL+. Создание библиотеки, амплификацию и секвенирование на GS FLX проводили, следуя соответствующим протоколам производителя.
Нуклеотидные последовательности фрагментов генов 16S рРНК депонированы в базу данных Sequence Read Archive (SRA) NCBI под номерами SRX5073294 (Bu-16) и SRX5073295 (Bu-18), в рамках проекта BioProject PRJNA507138.
Анализ результатов секвенирования фрагментов генов 16S рРНК. Для проведения анализа из набора определенных при пиросеквенировании последовательностей генов 16S рРНК с помощью программы Mothur (Schloss et al., 2009) отбирали чтения, содержащие последовательность праймера U341F, и обрезали последовательности до одинаковой длины 250 нт для облегчения кластерного анализа. Затем с помощью Usearch (Edgar, 2010) удаляли чтения низкого качества и проводили кластеризацию всех последовательностей (для обоих образцов совокупно) в оперативные таксономические единицы (ОТЕ) на уровне 97% идентичности. На этом этапе алгоритм Usearch удалял химерные последовательности и синглтоны (последовательности, прочтенные только один раз; в основном они представляют ошибки пиросеквенирования (Behnke et al., 2011). Затем с помощью Usearch наборы исходных последовательностей, полученных для образцов Bu-16 и Bu-18, накладывали на репрезентативные последовательности полученных ОТЕ на уровне 97% идентичности. Таким образом, для каждой ОТЕ было определено число соответствующих ей последовательностей генов 16S рРНК в каждом из двух исследованных образцов. Окончательный набор данных содержал 8830 последовательностей фрагментов генов 16S рРНК для образца Bu-16 и 19 104 – для образца Bu-18.
Таксономическую идентификацию ОТЕ проводили с помощью SINA online alignment and classification platform и базы данных SILVA версия 1.2.11, с параметрами по умолчанию (Pruesse et al., 2012). Также идентификацию ОТЕ проводили путем сравнения с базой данных 16S рРНК в GenBank по протоколу BLASTN.
ПЦР-амплификация и секвенирование фрагментов генов 18S рРНК эукариот образца Bu-16. Для ПЦР-амплификации фрагмента гена 18S рРНК использовали праймеры V4_1F (5'-CCA GCA SCY GCG GTA ATW CC) и TAReukREV3 (5'-ACT TTC GTT CTT GAT YRA) и препарат метагеномной ДНК образца Bu-16 в качестве матрицы. ПЦР фрагменты секвенировали на Illumina MiS-eq, в результате чего было получено 52 516 пар чтений (2 × 300 нт). Пересекающиеся чтения с двух концов объединяли с помощью FLASH v. 1.2.11 (Magoc, Salzberg, 2011). Окончательный набор данных содержал 47 284 последовательностей генов 18S рРНК. Нуклеотидные последовательности фрагментов генов 18S рРНК образца Bu-16 депонированы в базу данных SRA NCBI под номером SRX5073293. Поиск последовательностей, гомологичных 18S рРНК Spumella elongata (GenBank AJ236859), проводили с помощью BLASTN, отбирая последовательности с уровнем идентичности свыше 94% на не менее чем 90% их длины (обнаружено 2512 шт.).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Физико-химические характеристики дренажных вод. Месторождение полиметаллических руд “Озерное” расположено в Еравнинском районе Республики Бурятия и входит в десятку крупнейших цинковых месторождений в мире по объемам запасов и качеству руды. К настоящему времени проведены вскрышные работы для разработки месторождения открытым способом, но промышленная добыча еще не начата. Объектами исследования являются два небольших водоема, расположенные в открытом карьере (рис. 1). Водоем Bu-18 имеет площадь около 2 кв. м, глубину не более 20 см и на момент отбора образцов наполнялся подземными водами из родника; поверхностный сток из него отсутствовал. Второй водоем, обозначенный Bu-16, располагался на расстоянии около 15 м от Bu-18 ниже по склону карьера, имел площадь около 15 кв. м и глубину не более 30 см. Перепад высот между двумя водоемами составлял около 2 м. По-видимому, Bu-16 питается грунтовой водой, перетекающей из Bu-18 и проходящей через слой подстилающих сульфидсодержащих пород.
Рис. 1.
Изменение состава микробных сообществ при образовании кислого шахтного дренажа. (а) Схема расположения водоемов Bu-18 и Bu-16. Направление потоков воды указано серыми стрелками. (б) Разнообразие микробных сообществ Bu-18 и Bu-16. Показаны доли (% от общего числа последовательностей генов 16S рРНК) ОТЕ, обнаруженных только в Bu-18 (Bu-18), в обоих водоемах (Bu-18 + Bu-16) или только в Bu-16 (Bu-16), в микробных сообществах Bu-18 (серые столбики) и Bu-16 (черные столбики). Подгруппы “общих” ОТЕ (Bu-18 + Bu-16): e < 0.5 ‒ ОТЕ, доли которых в Bu-16 снизились более, чем в 2 раза по сравнению с Bu-18; 0.5 < e < 2 ‒ ОТЕ, доли которых в Bu-18 и Bu-16 отличались не более, чем в 2 раза; e > 2 ‒ ОТЕ, доли которых в Bu-16 увеличились более чем в 2 раза по сравнению с Bu-18. Число ОТЕ в каждой категории указано цифрами над столбиками.
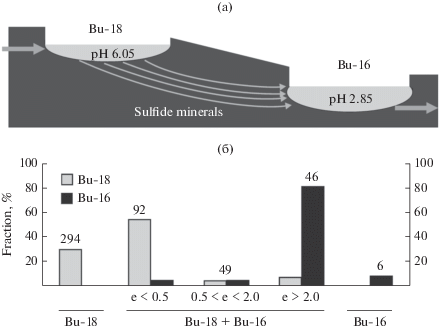
Такая схема питания водоемов согласуется с физико-химическими характеристиками воды. Температура воды в Bu-18 составляла 7.9°С, вода была слабо кислой (рН 6.05), имела невысокое содержание сульфатов и растворенных металлов (табл. 1). Водоем Bu-16, температура которого составляла 13.2°С, можно определить как кислый дренаж, он имеет низкий рН (2.85) и более высокое по сравнению с Bu-18 содержание сульфатов и ионов металлов, особенно железа и марганца, концентрация которых достигала 26 и 164 мг/л соответственно (табл. 1). По-видимому, различия в химическом составе воды двух водоемов обусловлены процессом окисления сульфидов при перетоке воды из Bu-18 в Bu-16, так как содержание анионов, хлорида и нитрата, примерно одинаковы. Повышенное содержание кремния и алюминия в Bu-16 также свидетельствует о частичном гидролизе алюмосиликатных минералов вследствие низкого рН, явления типичного для шахтных дренажей.
Таблица 1.
Физико-химические характеристики и элементный состав исследованных проб воды из водоемов Bu-16 и Bu-18
| Характеристика, единицы измерения | Bu-16 | Bu-18 |
|---|---|---|
| T, °C | 13.2°С | 7.9°С |
| рН | 2.85 | 6.05 |
| Eh, mV | +500 | +365 |
| Na (мг/л) | 11.5 | 4.7 |
| K (мг/л) | 0.89 | 0.81 |
| Ca (мг/л) | 279 | 120 |
| Si (мг/л) | 14.3 | 6.85 |
| Mg (мг/л) | 77.8 | 31.8 |
| Al (мг/л) | 23.7 | 0.033 |
| Fe (мг/л) | 26.4 | 0.43 |
| Mn (мг/л) | 164 | 28.4 |
| Cu (мг/л) | 0.56 | 0.001 |
| Zn (мг/л) | 10.5 | 1.95 |
| Sr (мг/л) | 0.74 | 0.14 |
| Cd (мг/л) | 0.24 | 0.011 |
| Pb (мг/л) | 1.24 | 0.003 |
| Ni (мг/л) | 0.023 | 0.0044 |
| As (мг/л) | <0.002 | <0.002 |
| ${\text{SO}}_{4}^{{2 - }}$ (мг/л) | 1405 | 452 |
| Cl– (мг/л) | 70 | 70 |
| ${\text{NO}}_{3}^{{2 - }}$ (мг/л) | 20 | 24 |
Микробное сообщество кислого дренажа Bu-16 является результатом селекции сообщества источника Bu-18. В результате анализа последовательностей фрагментов генов 16S рРНК всего было идентифицировано 487 ОТЕ (на уровне 97% идентичности последовательностей), из которых лишь 9 относились к археям. Индексы альфа-разнообразия указывают на более низкое разнообразие микробного сообщества кислого дренажа Bu-16, по сравнению с Bu-18, и доминирование в нем относительно небольшого числа ОТЕ (табл. 2).
Таблица 2.
Разнообразие прокариот в микробных сообществах водоемов Bu-16 и Bu-18
| Индексы разнообразия | Bu-16 | Bu-18 |
|---|---|---|
| Число ОТЕ | 193 | 481 |
| Chao1 | 199.8 | 481.0 |
| Shannon_e | 3.45 | 5.01 |
| Simpson | 0.0144 | 0.0567 |
| Berger-Parker | 0.131 | 0.0451 |
Данные по присутствию отдельных ОТЕ в двух сообществах и их относительной численности хорошо согласуются с предположением о том, что сообщество Bu-16 является результатом селекции сообщества Bu-18, сопровождающейся уменьшением общего разнообразия и отбором отдельных доминирующих групп. Из всех 487 обнаруженных ОТЕ в обоих сообществах присутствовали 187 ОТЕ, уникальными для Bu-18 были 294 ОТЕ, а для Bu-16 – всего 6 ОТЕ (рис. 1). Еще более четкая картина наблюдалась при анализе долей, которые эти ОТЕ составляли в двух сообществах. Так, 6 специфичных для Bu-16 ОТЕ представляли в нем 8.1% всех последовательностей генов 16S рРНК, и еще 82.5% приходилось на 46 “общих” ОТЕ, доля которых в Bu-16 выросла, по сравнению с Bu-18, более чем вдвое (рис. 1). “Общие” ОТЕ, доли которых менялись в пределах +/‒ 2 раза, представляли в каждом сообществе лишь около 4% всех последовательностей.
Микроорганизмы ‒ потенциальные участники циклов железа и серы в кислом дренаже Bu-16. Микробное сообщество источника Bu-18 насчитывало 481 ОТЕ, представляющих бактериальные филумы Acidobacteria, Actinobacteria, Bacteroidetes, Ignavibacteriae, Chlamydiae, Chloroflexi, Cyanobacteria, Deinococcus-Thermus, Dependentiae (TM6), Elusimicrobia, Fibrobacteres, Firmicutes, Gemmatimonadetes, Nitrospirae, Patescibacteria, Planctomycetes, Proteobacteria (классы альфа, бета, гамма и дельта), Spirochaetes, Verrucomicrobia и кандидатный филум WPS-2 (табл. 3). Археи составляли около 4.5% сообщества и относились к классу Thermoplasmata филума Euryarchaeota.
Таблица 3.
Состав микробных сообществ водоемов Bu-16 и Bu-18 по результатам анализа последовательностей генов 16S рРНК
| Таксономическая группа | Доля в сообществе (%) | |
|---|---|---|
| Bu-18 | Bu-16 | |
| Archaea (Thermoplasmatales) | 4.51 | 0.02 |
| Acidobacteria | 3.11 | 0.33 |
| Actinobacteria | 5.93 | 3.90 |
| Ferrimicrobium | 0.00 | 0.99 |
| Bacteroidetes | 18.32 | 1.65 |
| Cyanobacteria | 1.62 | 1.17 |
| Dependentiae (TM6) | 0.19 | 7.83 |
| Elusimicrobia | 1.80 | 0.29 |
| Firmicutes | 0.26 | 11.18 |
| Sulfobacillus | 0.17 | 6.22 |
| Nitrospirae | 1.30 | 13.26 |
| Leptospirillum | 1.30 | 13.26 |
| Candidate Phyla Radiation | 19.76 | 14.51 |
| Parcubacteria (OD1) | 15.52 | 11.94 |
| Saccharibacteria (TM7) | 4.20 | 2.57 |
| Alphaproteobacteria | 3.60 | 0.76 |
| Betaproteobacteria | 9.98 | 26.13 |
| Gallionellaceae | 3.48 | 17.50 |
| Sulfuriferula | 0.50 | 4.22 |
| Gammaproteobacteria | 10.93 | 15.97 |
| Acidithiobacillus | 1.67 | 13.06 |
| Metallibacterium | 0.00 | 0.96 |
| Deltaproteobacteria | 1.49 | 0.05 |
| Verrucomicrobia | 1.07 | 0.05 |
| Прочие и неклассифицированные | 16.13 | 2.90 |
В кислом дренаже Bu-16 снижались относительные доли таких характерных для пресных водоемов и почв групп бактерий, как Acidobacteria, Bacteroidetes, большинства видов альфа-, гамма- и дельтапротеобактерий, а также архей, вместе составлявших большинство сообщества в Bu-18. Более чем в 2 раза, по сравнению с Bu-18, возросли относительные доли лишь 52 ОТЕ, которые суммарно представляли более 90% микроорганизмов в Bu-16. Доминирующими микроорганизмами в Bu-16 являлись хемолитоавтотрофные бактерии, способные окислять железо и/или восстановленные соединения серы, – представители родов Leptospirillum (13.25%), Acidithiobacillus (13.06%) и Sulfobacillus (6.22%), а также бетапротеобактерии семейств Gallionellaceae (17.50%) и Sulfuricellaceae (4.22%). Leptospirillum являются облигатными хемолитоавтотрофами, использующими в качестве донора электронов только Fe2+, а в качестве акцептора – кислород. Хотя они обычно доминируют в сообществах, развивающихся при экстремальной кислотности (рН ниже 2) и повышенной температуре (25‒40°С), например, в шахтах Richmond Mine в Калифорнии (Tyson et al., 2004), они были обнаружены и в холодных кислых шахтных дренажах в Бурятии (Кадников и соавт., 2016а). Acidithiobacillus – более умеренные ацидофилы (оптимальный рН 2‒3), в отношении оптимальной температуры роста в основном являются мезофилами или психрофилами. Эти хемолиотоавтотрофные бактерии способны в аэробных условиях окислять не только Fe2+, но и восстановленные соединения серы или только их, фиксировать CO2 через цикл Кальвина и атмосферный азот с участием нитрогеназы (Liljeqvist et al., 2011). Многие штаммы Acidithiobacillus (например, A. ferrivorans) были выделены из кислых шахтных дренажей с рН 2‒3.5 в районах с холодным климатом (Kupka et al., 2007; Hallberg et al., 2010).
Наиболее многочисленной группой хемолитоавтотрофов были бетапротеобактерии семейств Gallionelaceae и Sulfuricellaceae. К Gallionelaceae относились 9 ОТЕ, большая часть которых представляла род Gallionella (6 ОТЕ, 15.64% сообщества). Эти автотрофные железоокисляющие бактерии обнаруживают в кислых шахтных дренажах с умеренными значениями рН (2‒5) и концентрациями растворенных металлов. Близкие к Gallionella бактерии входили в число доминирующих групп микроорганизмов в дренажных водах шахт, используемых для добычи свинца и цинка в районе Carnoules во Франции (Bruneel et al., 2006), шахт по добыче пирита Cae Coch в Уэльсе (Kimura et al., 2011) и Yunfu в Китае (He et al., 2007), урановой шахты Ronneburg в Германии (Fabisch et al., 2013) и др. Бактерии рода Gallionella доминировали в микробном сообществе воды скважины ShG14-8 месторождения полиметаллических руд Шерловая гора в Забайкалье, имеющей близкое к Bu-16 значение рН (2.65) (Кадников и соавт., 2016b). Наряду с Gallionella были обнаружены бактерии рода Nitrosospira (0.72%) и Nitrotoga (1.10%), культивируемые представители которых могут, соответственно, окислять аммоний и нитрит (Schramm et al., 1998; Ishii et al., 2017). Около 4.22% сообщества Bu-16 составляли сероокисляющие бактерии рода Sulfuriferula, использующие в качестве акцептора электронов кислород или нитрат в анаэробных условиях (Watanabe et al., 2015). Присутствие бактерий, энергетический метаболизм которых связан с окислением или восстановлением соединений азота, согласуется с достаточно высоким содержанием в воде нитрата, источником которого могут быть техногенные соединения азота, поступающие с используемой при добыче руды открытым способом взрывчаткой.
Среди бактерий, предположительно участвующих в цикле железа, также были обнаружены фирмикуты рода Sulfobacillus (6.22%) и близкая к ним ОТЕ, отнесенная к тому же семейству XVII Clostridiales (4.93%). Представители Sulfobacillus могут расти как автотрофно, окисляя железо и серу, так и гетеротрофно (Anderson et al., 2012). Около 1% последовательностей генов 16S рРНК в образце Bu-16 относилось к актинобактериям Ferrimicrobium acidiphilum. Это ацидофильные гетеротрофы, способные окислять Fe2+ в аэробных условиях, а при анаэробном росте восстанавливать Fe3+, используя в качестве донора электронов органические соединения (Johnson et al., 2009). Восстанавливать Fe3+ в анаэробных условиях также способны гаммапротеобактерии Metallibacterium scheffleri (Ziegler et al., 2013), доля которых в сообществе составляет 0.96%.
В целом, различные группы бактерий, культивируемые представители которых способны окислять железо или восстановленные соединения серы, составляют около половины всего микробного сообщества в Bu-16, а в источнике Bu-18 на эти ОТЕ приходилось лишь около 4%.
Микробное сообщество кислого дренажа Bu-16 содержит большую долю не свободноживущих микроорганизмов. Неожиданным результатом данной работы стало обнаружение в исследованных сообществах в значительных количествах представителей двух некультивируемых групп ‒ суперфилума Candidate Phyla Radiation (Patescibacteria) и кандидатного филума Dependentiae (TM6). Candidate Phyla Radiation являются монофилетичной группой, включающей несколько десятков линий уровня филума (Brown et al., 2015). Эти бактерии имеют небольшой размер клеток и геномов, у них отсутствуют многие ключевые биосинтетические пути, поэтому предполагается, что они могут расти только в ассоциации с другими микроорганизмами, как паразиты или симбионты (Castelle et al., 2018). В источнике Bu-18 было обнаружено 40 ОТЕ, представляющих кандидатные филумы Parcubacteria (OD1) и Saccharibacteria (TM7), из которых 21 присутствует и в Bu-16. Доли 5 ОТЕ в Bu-16 выросли по сравнению с Bu-18 более чем вдвое, и суммарно они представляют в нем около 13% микроорганизмов. По-видимому, увеличение численности этих ОТЕ отражает селекцию их хозяев в кислом дренаже Bu-16. Представителей Saccharibacteria ранее детектировали в различных кислых шахтных дренажах (Méndez-García et al., 2015), а обнаружение представителей Parcubacteria описано лишь в одной работе, посвященной анализу таких экосистем на острове Шпицберген (García-Moyano et al., 2015). Во всех описанных случаях эти линии были минорными компонентами микробных сообществ.
Кандидатный филум Dependentiae был представлен тремя ОТЕ, суммарная доля которых составляла 0.19% в Bu-18 и возрастала до 7.83% в Bu-16. Общими характеристиками бактерий этого филума являются: небольшой размер генома, редуцированная клеточная оболочка, отсутствие многих биосинтетических путей, наличие системы транспорта АТФ в клетку извне и наличие белков, обеспечивающих взаимодействие с эукариотическими клетками (Yeoh et al., 2016). Все эти характеристики указывают, что Dependentiae являются паразитами простейших эукариот (Yeoh et al., 2016). Обнаруженные нами Dependentiae были близки (95‒97% идентичность последовательностей генов 16S рРНК) к Candidatus Dependentiae bacterium SeV1 (GenBank CP025544), внутриклеточному паразиту простейшего Spumella elongata. Spumella (Stramenopiles; Chrysophyceae; Chromulinales; Chromulinaceae) – гетеротрофные флагелляты, широко распространенные в почвах и пресных водоемах (Boenigk et al., 2005). Анализ последовательностей фрагментов генов 18S рРНК эукариот из Bu-16 показал, что около 5% из них представляли флагелляты рода Spumella. Возможно, развитие микробных биопленок на поверхности минералов, характерное для кислых шахтных дренажей, создает благоприятные условия для питания Spumella. Несмотря на то, что различных простейших неоднократно обнаруживали в кислых шахтных дренажах (Méndez-García et al., 2015), о присутствии в них Spumella ранее не сообщалось.
Таким образом, полученные нами данные показывают, что при формировании кислого шахтного дренажа происходит снижение разнообразия микробного сообщества и его обогащение микроорганизмами – участниками циклов железа и серы, в первую очередь аэробными хемолитоавтотрофными железо- и сероокисляющими бактериями родов Acidithiobacillus, Gallionella, Sulfuriferula и Leptospirillum. Также увеличилось относительное содержание гетеротрофных бактерий, способных восстанавливать Fe3+ в анаэробных условиях. Особенностью исследованного кислого дренажа Bu-16 является высокое содержание несвободно живущих бактерий (представителей Candidate Phyla Radiation и Dependentiae), являющихся симбионтами или паразитами других микроорганизмов. Они редко встречаются в кислых шахтных дренажах и в ранее описанных случаях были минорными компонентами микробных сообществ. Присутствие в сообществе представителей Dependentiae и их вероятных хозяев, простейших рода Spumella, показывает важность эукариотических микроорганизмов как компонента экосистемы кислых дренажных вод.
ФИНАНСИРОВАНИЕ РАБОТЫ
Работа выполнена при поддержке РНФ (грант 14-14-01016, анализ прокариот) и РФФИ (грант 18-34-00356, анализ эукариот). Отбор и физико-химический анализ проб кислых дренажей были поддержаны грантом РФФИ № 16-54-150011.
Список литературы
Кадников В.В., Ивасенко Д.А., Белецкий А.В., Марданов А.В., Данилова Э.В., Пименов Н.В., Карначук О.В., Равин Н.В. Влияние содержания металлов на состав микробных сообществ кислых дренажных вод месторождения полиметалличеcких руд // Микробиология. 2016a. Т. 85. № 6. С. 732‒739.
Kadnikov V.V., Ivasenko D.A., Beletsky A.V., Mardanov A.V., Danilova E.V., Pimenov N.V., Karnachuk O.V., Ravin N.V. Effect of metal concentration on the microbial community in acid mine drainage of a polysulfide ore deposit // Microbiology (Moscow). 2016a. V. 85(6). P. 745–751.
Кадников В.В., Ивасенко Д.А., Белецкий А.В., Марданов А.В., Данилова Э.В., Пименов Н.В., Карначук О.В., Равин Н.В. Новая некультивируемая бактерия семейства Gallionellaceae, – описание и реконструкция генома на основе анализа метагенома микробного сообщества кислых шахтных вод // Микробиология. 2016b. Т. 85. № 4. С. 421–435.
Kadnikov V.V., Ivasenko D.A., Beletsky A.V., Mardanov A.V., Danilovac E.V., Pimenov N.V., Karnachuk O.V., Ravin N.V. A novel uncultured bacterium of the family Gallionellaceae: description and genome reconstruction based on metagenomic analysis of microbial community in acid mine drainage // Microbiology (Moscow). 2016b. V. 8(4). P. 449–461.
Anderson I., Chertkov O., Chen A., Saunders E., Lapidus A., Nolan M., Lucas S., Hammon N., Deshpande S., Cheng J.F., Han C., Tapia R., Goodwin L.A., Pitluck S., Liolios K., Pagani I., Ivanova N., Mikhailova N., Pati A., Palaniappan K., Land M., Pan C., Rohde M., Pukall R., Göker M., Detter J.C., Woyke T., Bristow J., Eisen J.A., Markowitz V., Hugenholtz P., Kyrpides N.C., Klenk H.P., Mavromatis K. Complete genome sequence of the moderately thermophilic mineral-sulfide-oxidizing firmicute Sulfobacillus acidophilus type strain (NAL(T)) // Stand. Genomic Sci. 2012. V. 6 (3). P. 1‒13.
Baker B.J., Banfield J.F. Microbial communities in acid mine drainage // FEMS Microbiol. Ecol. 2003. V. 44. P. 139–152.
Behnke A., Engel M., Christen R., Nebel M., Klein R.R., Stoeck T. Depicting more accurate pictures of protistan community complexity using pyrosequencing of hypervariable SSU rRNA gene regions // Environ. Microbiol. 2011. V. 13. P. 340–349.
Boenigk J., Pfandl K., Stadler P., Chatzinotas A. High diversity of the “Spumella-like” flagellates: an investigation based on the SSU rRNA gene sequences of isolates from habitats located in six different geographic regions // Environ. Microbiol. 2005. V. 7. P. 685‒697.
Brown C.T., Hug L.A., Thomas B.C., Sharon I., Castelle C.J., Singh A., Wilkins M.J., Wrighton K.C., Williams K.H., Banfield J.F. Unusual biology across a group comprising more than 15% of domain Bacteria // Nature. 2015. V. 523. P. 208‒211.
Bruneel O., Duran R., Casiot C., Elbaz-Poulichet F., Personne J.C. Diversity of microorganisms in Fe-As-rich acid mine drainage waters of Carnoules, France // Appl. Environ. Microbiol. 2006. V. 72. P. 551–556.
Castelle C.J., Brown C.T., Anantharaman K., Probst A.J., Huang R.H., Banfield J.F. Biosynthetic capacity, metabolic variety and unusual biology in the CPR and DPANN radiations // Nat. Rev. Microbiol. 2018. V. 16. P. 629‒645.
Chen L.X., Hu M., Huang L.N., Hua Z.S., Kuang J.L., Li S.J., Shu W.S. Comparative metagenomic and metatranscriptomic analyses of microbial communities in acid mine drainage // ISME J. 2015. V. 9. P. 1579‒1592.
Edgar R.C. Search and clustering orders of magnitude faster than BLAST // Bioinformatics. 2010. V. 26. P. 2460–2461.
Fabisch M., Beulig F., Akob D.M., Kusel K. Surprising abundance of Gallionella-related iron oxidizers in creek sediments at pH 4.4 or at high heavy metal concentrations // Front. Microbiol. 2013. V. 4. P. 390.
García-Moyano A., Austnes A.E., Lanzén A., González-Toril E., Aguilera Á., Øvreås L. Novel and unexpected microbial diversity in acid mine drainage in Svalbard (78° N), revealed by culture-independent approaches // Microorganisms. 2015. V. 3. P. 667‒694.
Hallberg K.B., González-Toril E., Johnson D.B. Acidithiobacillus ferrivorans, sp. nov.; facultatively anaerobic, psychrotolerant iron-, and sulfur-oxidizing acidophiles isolated from metal mine-impacted environments // Extremophiles. 2010. V. 14. P. 9‒19.
He Z., Xiao S., Xie X., Zhong H., Hu Y., Li Q., Gao F., Li G., Liu J., Qiu G. Molecular diversity of microbial community in acid mine drainages of Yunfu sulfide mine // Extremophiles. 2007. V. 11. P. 305–314.
Ishii K., Fujitani H., Soh K., Nakagawa T., Takahashi R., Tsuneda S. Enrichment and physiological characterization of a cold-adapted nitrite-oxidizing Nitrotoga sp. from an eelgrass sediment // Appl. Environ. Microbiol. 2017. V. 83. pii: e00549-17.
Johnson B.D., Hallberg K.B. The microbiology of acidic mine waters // Research in Microbiology. 2003. V. 154. P. 466‒473.
Johnson D.B., Bacelar-Nicolau P., Okibe N., Thomas A., Hallberg K.B. Ferrimicrobium acidiphilum gen. nov., sp. nov. and Ferrithrix thermotolerans gen. nov., sp. nov.: heterotrophic, iron-oxidizing, extremely acidophilic actinobacteria // Int. J. Syst. Evol. Microbiol. 2009. V. 59. P. 1082‒1089.
Kimura S., Bryan C.G., Hallberg K.B., Johnson D.B. Biodiversity and geochemistry of an extremely acidic, low-temperature subterranean environment sustained by chemolithotrophy // Environ. Microbiol. 2011. V. 13. P. 2092–2104.
Kupka D., Rzhepishevska O.I., Dopson M., Lindström E.B., Karnachuk O.V., Tuovinen O.H. Bacterial oxidation of ferrous sulfate at low temperatures // Biotechnol. Bioeng. 2007. V. 97. P. 1470‒1478.
Liljeqvist M., Valdes J., Holmes D.S., Dopson M. Draft genome of the psychrotolerant acidophile Acidithiobacillus ferrivorans SS3 // J. Bacteriol. 2011. V. 193. P. 4304–4305.
Magoč T., Salzberg S.L. FLASH: fast length adjustment of short reads to improve genome assemblies // Bioinformatics. 2011. V. 27. P. 2957‒2963.
Méndez-García C., Peláez A.I., Mesa V., Sánchez J., Golyshina O.V., Ferrer M. Microbial diversity and metabolic networks in acid mine drainage habitats // Front. Microbiol. 2015. V. 29 (6). P. 475.
Pruesse E., Peplies J., Glöckner FO. SINA: accurate high-throughput multiple sequence alignment of ribosomal RNA genes // Bioinformatics. 2012. V. 28. P. 1823–1829.
Ram R.J., Verberkmoes N.C., Thelen M.P., Tyson G.W., Baker B.J., Blake R.C. 2nd, Shah M., Hettich R.L., Banfield J.F. Community proteomics of a natural microbial biofilm // Science. 2005. V. 308. P. 1915‒1920.
Rohwerder T., Gehrke T., Kinzler K., Sand W. Bioleaching review part A: progress in bioleaching: fundamentals and mechanisms of bacterial metal sulfide oxidation // Appl. Microbiol. Biotechnol. 2003. V. 63. P. 239‒248.
Schramm A., De Beer D., Wagner M., Amann R. Identification and activities in situ of Nitrosospira and Nitrospira spp. as dominant populations in a nitrifying fluidized bed reactor // Appl. Environ. Microbiol. 1998. V. 64. P. 3480‒3485.
Schloss P.D., Westcott S.L., Ryabin T., Hall J.R., Hartmann M., Hollister E.B., Lesniewski R.A., Oakley B.B., Parks D.H., Robinson C.J., Sahl J.W., Stres B., Thallinger G.G., Van Horn D.J., Weber C.F. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities // Appl. Environ. Microbiol. 2009. V. 75. P. 7537–7541.
Tyson G.W., Chapman J., Hugenholtz P., Allen E.E., Ram R.J., Richardson P.M., Solovyev V.V., Rubin E.M., Rokhsar D.S., Banfield J.F. Community structure and metabolism through reconstruction of microbial genomes from the environment // Nature. 2004. V. 428. № 6978. P. 37–43.
Watanabe T., Kojima H., Fukui M. Sulfuriferula multivorans gen. nov., sp. nov., isolated from a freshwater lake, reclassification of “Thiobacillus plumbophilus” as Sulfuriferula plumbophilus sp. nov., and description of Sulfuricellaceae fam. nov. and Sulfuricellales ord. nov. // Int. J. Syst. Evol. Microbiol. 2015. V. 65. P. 1504‒1508.
Yeoh Y.K., Sekiguchi Y., Parks D.H., Hugenholtz P. Comparative genomics of candidate phylum TM6 suggests that parasitism is widespread and ancestral in this lineage // Mol. Biol. Evol. 2016. V. 33. P. 915‒927.
Ziegler S., Waidner B., Itoh T., Schumann P., Spring S., Gescher J. Metallibacterium scheffleri gen. nov., sp. nov., an alkalinizing gammaproteobacterium isolated from an acidic biofilm // Int. J. Syst. Evol. Microbiol. 2013. V. 63. P. 1499‒1504.
Дополнительные материалы отсутствуют.


