Микробиология, 2019, T. 88, № 5, стр. 541-552
Пути разложения 3-хлорбензоата штаммами Rhodococcus opacus 1CP и 6a
И. П. Соляникова a, *, Е. В. Емельянова a, Е. С. Шумкова b, В. М. Травкин c
a ФИЦ ПНЦ БИ РАН, Институт биохимии и физиологии микроорганизмов им. Г.К. Скрябина
142290 Пущино, Россия
b ФИЦ Биотехнологии РАН, Институт биохимии им. А.Н. Баха
119071 Москва, Россия
c ФГБОУ ВО Белгородский государственный аграрный университет имени В.Я. Горина
308503 п. Майский, Белгородской обл., Россия
* E-mail: innas@IBPM.Pushchino.ru
Поступила в редакцию 18.03.2019
После доработки 15.05.2019
Принята к публикации 29.05.2019
Аннотация
В промышленности и сельском хозяйстве в качестве прекурсора/консерванта широко используют 3-хлорбензойную кислоту (3-ХБК), вследствие чего она является одним из загрязнителей окружающей среды. Изучены ключевые этапы разложения 3-ХБК бактериями Rhodococcus opacus (штаммы 1CP и 6a). Дана характеристика субстратной специфичности 3-хлорбензоат-1,2-диоксигеназ (3‑ХБК-1,2-ДО), индуцирующихся при росте обоих штаммов в присутствии 3-ХБК. Показана более широкая специфичность 3-ХБК-1,2-ДО по сравнению со специфичностью бензоат-1,2-диоксигеназы (БК-1,2-ДО) штамма 1CP, индуцирующейся при росте культуры в присутствии бензоата. При разложении 3-ХБК клетками R. opacus 1CP были обнаружены как 3-хлор-, так и 4-хлорпирокатехин. Штамм R. opacus 6a эффективно разлагал 3-ХБК без накопления интермедиатов. Установлено различие в путях деструкции 3-ХБК данными штаммами: путь орто-расщепления 3-хлорпирокатехина у штамма 1CP и 4-хлорпирокатехина у штамма 6a. В геноме R. opacus 6a обнаружены гены, кодирующие хлорпирокатехин-1,2-диоксигеназу и хлормуконатциклоизомеразу, на 98–99% идентичные генам R. opacus 1CP, кодирующим 4-хлорпирокатехин-1,2-диоксигеназу (4-ХПК-1,2-ДО) и 3-хлормуконатциклоизомеразу (3-ХМЦИ) модифицированного пути орто-расщепления 4-хлорпирокатехина (интермедиата разложения 4-хлорфенола). На примере изучаемых штаммов показано, что реализация разных путей разложения 3-ХБК у исследованных культур определяется особенностями субстратной специфичности 3-ХБК-1,2-ДО – фермента, инициирующего разложение этого поллюта-нта.
По оценкам специалистов к концу прошлого века в окружающую среду в результате деятельности человека попало более 2000 различных галогенированных соединений (Gribble, 1994). Так, 3-хлорбензойную кислоту (3-ХБК) используют в качестве сырья при производстве красителей, фармацевтических препаратов, фунгицидов и в качестве консерванта для адгезивов и красок. Подобно другим хлорбензоатам, она может накапливаться как конечный продукт при кометаболизме полихлорбифенилов (ПХБ) и хлортолуолов и в процессе хлорирования воды (Baggi, Zangrossi, 2001; Zhang et al., 2009). Широкое использование 3-ХБК приводит к повышенному загрязнению промышленных стоков, рек и грунтовых вод. Практически все хлорароматические соединения как природного, так и антропогенного происхождения оказывают негативное влияние на живые организмы (Willes et al., 1993; Gribble, 1994). Введение хлор-заместителей в ароматическое кольцо увеличивает устойчивость молекулы к микробной атаке, и, в целом, чем больше заместителей содержится в кольце, тем она более устойчива к разложению (Boyle et al., 1992).
В процессе микробной деструкции можно выделить два ключевых момента – доступность субстрата для клеток (его растворимость, токсичнocть, наличие систем, способствующих поступлению субстратов в клетку) и присутствие в клетках специализированных ферментных систем, разлагающих поступающие соединения. Зачастую невозможность деградации какого-либо соединения обусловлена узкой субстратной специфичностью первого фермента, участвующего в деструкции. Наглядным примером этого является разложение полихлорбифенилов (Adebusoye, 2017). Большинство штаммов способно разлагать/трансформировать ПХБ только в условиях кометаболизма с бифенилом, т.к. бифенилдиоксигеназа является ферментом с широкой субстратной специфичностью (Aken et al., 2010). Хлорбензоаты (ХБК), образующиеся при разложении хлорбифенилов, несмотря на более простое по сравнению с ПХБ строение, слабо подвержены микробному разложению. Причина этого – относительно узкая субстратная специфичность бензоат-1,2-диоксигеназы (БК-1,2-ДО) (КФ 1.14.12.10), фермента, инициирующего деградацию (хлор)бензоатов. Такой фермент обнаружен у штаммов Acinetobacter calcoaceticus (Harayama et al., 1991), Rhodococcus sp. RHA1 (Kitagawa et al., 2001), Rhodococcus opacus 1CP (Solyanikova et al., 2016). Помимо бензоата, узкоспецифичная БК-1,2-ДО может атаковать 3-ХБК, но при этом не активна с 2- и 4-хлорбензоатами (Kitagawa et al., 2001). Однако известны БК-1,2-ДО и с более широкой субстратной специфичностью, например, у бактерий рода Pseudomonas (Hartmann et al., 1979; Yamaguchi, Fujisawa, 1981; Engesser et al., 1990).
Пути микробной деструкции хлорароматических соединений на примере разложения 3-ХБК были подробно изучены у бактерии Pseudomonas sp. B13 (Hartmann et al., 1979). Было обнаружено, что для полного превращения 3-ХБК в интермедиаты цикла трикарбоновых кислот штамму Pseudomonas sp. B13 требовалось присутствие фермента, способного осуществлять начальную атаку на хлорбензоат – хлорбензоат-1,2-диоксигеназы (3‑ХБК-1,2-ДО), а также ферментов так называемого модифицированного пути орто-расщепления 3-хлор- и 4-хлорпирокатехинов (3-ХПК и 4‑ХПК), которые образуются в результате последовательного действия 3-ХБК-1,2-ДО и дигидродигидроксибензоатдегидрогеназы (Hartmann et al., 1979) (рис. 1а).
Рис. 1.
Пути микробной деградации 3-ХБК бактериями: (a) – модифицированный путь орто-расщепления 3-хлорпирокатехина грамотрицательными бактериями; (б) – новый модифицированный путь орто-расщепления 3-хлорпирокатехина R. opacus 1CP; (в) –модифицированный путь орто-расщепления 4-хлорпирокатехина родококками, в том числе R. opacus 1CP.
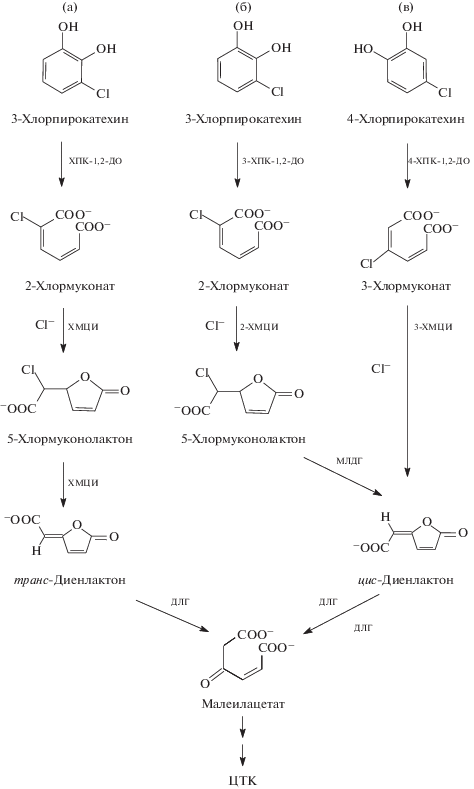
3-ХБК оказалась одним из соединений, которое многие исследователи использовали как “модельное” для изучения способности микроорганизмов адаптироваться к последствиям антропогенной нагрузки на окружающую среду. За относительно короткое время в этой области был достигнут значительный прогресс (Field, Sierra-Alvarez, 2008; Abbasian et al., 2016). Вместе с тем, наименее изученными до сих пор остаются первые реакции превращения не только 3-ХБК, но и ряда других монохлорароматических соединений (3-хлорфенола (3-ХФ), 3-хлоранилина (3-ХА)) бактериями разных групп. При микробном превращении мета-замещенных монохлорированных ароматических соединений возможно образование как 3-ХПК (рис. 1а), так и 4-ХПК (рис. 1в), и соотношение образующихся продуктов будет зависеть от свойств ферментов начальной атаки. Например, при окислении 3-ХБК клетками Pseudomonas sp. B13 образовывалась смесь 3-ХПК и 4‑ХПК в соотношении 67 : 33 (Reineke, Knackmuss, 1978). Аналогично установлено образование смеси 3‑ХПК и 4-ХПК (из 3-ХБК) рекомбинантным штаммом, несущим гены БК-1,2-ДО штамма Pseudomonas sp. 3CBA, деструктора 3‑хлорбензоата (Xu et al., 2017). Штамм Pseudomonas aeruginosa трансформировал 3-ХБК только в 3-ХПК (Ichihara et al., 1962; Ajithkumar, Kunhi, 2000). Как показали многочисленные исследования грамотрицательных бактерий, субстратная специфичность ферментов модифицированного орто-пути расщепления 3-ХПК позволяет им превращать 3‑ХПК в транс-диенлактон через 2‑хлормуконат и смесь 2- и 5-хлормуконолактонов (2-ХМЛ на рис. 1а не указан). Диенлактонгидролазы этих бактерий, описанные в литературе, были активны как с транс-диенлактоном, так и с его цис-изомером (Schlömann, 1994). Напротив, грамположительный штамм Arthrobacter sp. окислял 3‑ХБК только в 4-ХПК (Horvath, Alexander, 1970).
В наших исследованиях мы исходили из следующих предпосылок: 1) ферменты и метаболические пути микробной деградации хлорированных соединений впервые подробно исследовались на примере окисления 3-ХБК грамотрицательными бактериями; 2) успешное применение микробных препаратов для очистки окружающей среды от попадающих в нее токсикантов требует понимания процессов, происходящих не только на клеточном, но и на популяционном уровнях; 3) способность грамположительных бактерий разлагать 3-ХБК до сих пор изучена недостаточно, а информация о свойствах ферментов, катализирующих первые этапы разложения 3-ХБК у родококков, чрезвычайно ограничена. Учитывая вышесказанное, мы выбрали 3-ХБК для дальнейшего изучения процессов микробного превращения хлорбензоатов. Представляло интерес провести сравнение метаболизма 3-ХБК у двух представителей вида R. opacus: штаммов 1CP и 6a. Эти штаммы являются активными деструкторами токсичных соединений, например, фенола, 4‑хлорфенола, компонентов нефти (Горлатов и соавт., 1989; Моисеева и соавт., 1999; Коновалова и соавт., 2009). Кроме того, было показано, что ряд ферментных систем деструкции токсикантов этих штаммов обладаeт сходными физико-химическими свойствами.
Целью данной работы было провести сравнительный анализ основных этапов разложения 3‑ХБК штаммами Rhodococcus opacus 1CP и 6a.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объектами исследования были бактерии, выделенные из загрязненных почв: Rhodococcus opacus 1СР (Горлатов и соавт., 1989) (ВКМ Ac-2638 и DSM 46757) и R. opacus 6a (Коновалова и соавт., 2009). Изначально штамм 1CP был выделен на среде с 2,4-дихлорфенолом и активно разлагал 4‑ХФ. Получен вариант этого штамма, способный к росту в среде с 2-хлорфенолом (2-ХФ) и 3‑ХБК (Моисеева и соавт., 1999). Установлено, что введение в жизненный цикл этой бактерии стадии покоя приводит к значительному сокращению времени адаптации к новым субстратам. Поэтому для изучения разложения 3-ХБК культурой R. opacus 1CP использовали клетки, введенные в состояние покоя и поддерживаемые в неактивном состоянии в течение нескольких лет (Solyanikova et al., 2016).
Для выделения продуктов ПЦР использовали бактерии Escherichia coli DH5α, любезно предоставленную Я. Гренингом (Tech. Acc. J. Gröning).
Культивирование. Для культивирования штаммов 1СР и 6а R. opacus использовали минеральную среду следующего состава (г/л): Na2HPO4 0.7; KH2PO4 0.5; NH4NO3 0.75; MgSO4 ⋅ 7H2O 0.2; M-nSO4 0.001; FeSO4 0.02 (Горлатов и соавт., 1989). Активацию R. opacus 1CP проводили прямым высевом клеточной суспензии в минеральную среду с 3-ХБК (50 мг/л). После полного исчерпания субстрата его повторно вносили в концентрации 100 мг/л.
Получение биомассы. Для получения биомассы бактерии выращивали в минеральной среде с 3‑ХБК в качестве единственного источника углерода и энергии в колбах Эрленмейера объемом 750 мл, содержащих 200 мл среды, при 29°С на качалке при 220 об./мин. Для выращивания бактерий применяли метод периодического культивирования с дробным внесением по 100–200 мг/л субстрата. Клетки выращивали до плотности культуры 1.6–1.8 оптических единиц. Оптическую плотность (ОП) культур определяли при 545 нм. Остаточное количество субстрата определяли методом абсорбционной спектроскопии культуральной жидкости в диапазоне 220–340 нм после отделения клеток центрифугированием (7000 об./мин, 10 мин) на центрифуге Eppendorf 5414 (Германия). Для определения активности 3‑ХБК-1,2-ДО использовали свежесобранные клетки, осажденные центрифугированием и отмытые 50 мМ трис-HCl буфером (рН 8.0).
Геномную ДНК выделяли из 50 мл культуры R. opacus 6a, инкубируемой в среде с 4-хлорфенолом (100 мг/л) и 3-ХБК (200 мг/л). В среду вносили 20 г/л глицина за 24 ч до окончания культивирования. Экстракцию проводили смесью (фенол : : хлороформ : изоамилалкоголь) = (25 : 24 : 1) согласно протоколу [http://nbn-resolving.de/ urn:nbn:de:swb:105-1501974].
Для проведения ПЦР использовали следующие пары праймеров. Для ХПК-1,2-ДО (clcA): RhodF Fr (5'-CGACAAGTTCAAGGCCGAG-3'), RhodJ Rev (5'-CACGAAGTTGTAGGTGACGTAG-3'), RhodF Fr–Rhod Rev1 (5'-CTCGAAGTASAGCTGGGTSRTCA-3') (Thiel, 2004). Для ХМЦИ (clcB): ChpA Fr (5'-CAAGGTCATCATCGACTGCTAYYT- NGCNCC-3')–Rev1 (5'-CCAGGGCCRAASAGYTCRCAIC-3'); ChpА Fr (5'-CAAGGTCATCATCGACTGCTAYYTNGCNCC-3')–ChpD Rev (5'-TGGTGGAGTCCAGCATGGTNCCNCCRTA-3') (Thiel, 2004). Для хлормуконолактондегалогеназы (ХМЛД) (clcF): Fr (5'-GCTTCGACATATGTTGTACCTAGTTC-3')–Rev (5'-ATGGATCCTCAGTCTTTGCCGA-3') (Gröning et al., 2012).
Продукты ПЦР выделяли с помощью DNA Isolation Spin-Kit agarose A5193 (“AppliChem”). Лигировали их в вектор с использованием набора Clone JET PCR Cloning Kit K1231 Pure Extreme (“Thermo Scientific”). Далее вектором со вставкой трансформировали культуру E. coli DH5α. Трансформанты высевали на чашки с агаризованной LB средой, содержащей 100 мкг/мл ампициллина, и инкубировали при 37°C 24 ч для отбора колоний. Плазмидную ДНК после трансформации выделяли с помощью Gene JET Plasmid Miniprep Kit K0503 (“Fermentas”).
Амплификацию генов clcA и clcB осуществляли на приборе T-Gradient-Thermocycler (“Biometra”, Германия) при следующих условиях: 95°C – 5 мин, далее 8 циклов со снижением температуры отжига на 1°C в каждом цикле: 94°C – 30 с, (60–50°C) – 30 с, 72°C – 60 с; далее 20 циклов: 94°C – 30 с, 50°C – 30 с, 72°C – 60 с; конечная достройка: 72°C – 2 мин. Продукты реакции разделяли методом электрофореза в 1%-ном агарозном геле при напряжении 10 В/см, окрашивали раствором бромистого этидия (5 мкг/мл) и фотографировали в УФ-свете с использованием системы гель-документирования Gel DocTM XR (“Bio-Rad”, США).
Секвенирование ПЦР-продукта проводили в MWG Eurofins (Германия) (https://www.eurofinsgenomics.eu).
Анализ нуклеотидных последовательностей функциональных генов. Поиск гомологичных последовательностей проводили по базам данных GenBank/ DDBJ/EMBL (http://www.ncbi.nlm.nih.gov). Анализ нуклеотидных последовательностей осуществляли с помощью алгоритма BLAST (http:// www.ncbi.nlm.nih.gov).
Депонирование нуклеотидных последовательностей. Нуклеотидные последовательности фрагментов генов clcA и clcB, в двух вариантах каждый, депонированы в GenBank.
Полярографическое определение активности 3‑ХБК-1,2-ДО проводили, как описано ранее (Соляникова и соавт., 2016). Кислородный электрод Кларка использовали в комплекте с усилительной системой “Ingold 5313/10 O2 amplifier” (Швейцария–США). Регистрацию сигнала осуществляли с помощью двухкоординатного самописца “XY Recorder-4103” (Чехословакия). Реакцию выражали в pA/с, где 1 pA/с = 0.153 мкг O2/(л с). Оставшиеся после анализа клетки замораживали и хранили при –20°С до использования.
Приготовление бесклеточного экстракта проводили, как описано ранее (Solyanikova et al., 2016).
Определение активности ферментов – пирокатехин-1,2-диоксигеназы (ПК-1,2-ДО), хлормуконатциклоизомеразы (ХМЦИ), диенлактонгидролазы (ДЛГ) и хлормуконолактон дегалогеназы (ХМЛД) – проводили, как описано ранее (Моисеева и соавт., 2001; Gröning et al., 2012). За единицу каталитической активности принимали количество фермента, катализирующее превращение 1 мкмоль субстрата или образование 1 мкмоль продукта за 1 мин. Концентрацию белка определяли модифицированным методом Брэдфорд (Schlömann et al., 1990) с использованием бычьего сывороточного альбумина (V фракция) в качестве стандарта.
УФ-спектрометрия. Ферментативный анализ и мониторинг образования интермедиатов проводили с использованием спектрофотометра Varian Cary 50 UV-visible (“Agilent Technologies”, Германия). Спектры регистрировали в области поглощения 200–350 нм.
Идентификация продуктов, образующихся из 3‑ХБК. Для идентификации интермедиатов, образующихся при разложении 3-ХБК в ростовых условиях, клетки выращивали в минеральной среде с 200 мг/л 3-ХБК. ВЭЖХ проводили с использованием системы Dionex. Пробы отбирали каждый час, клетки отделяли центрифугированием и супернатант наносили на колонку Lichrospher 100 RP-18, элюцию вели 50% (v/v) метанолом с 1 г/л H3PO4 в качестве мобильной фазы. В качестве детектора использовали Dionex UVD 340 S Dioden array detektor (“Dionex Co.”, США). Сигналы соединений детектировали при длине волны 210 или 270 нм. Метаболиты сравнивали с референтными соединениями по времени удержания и УФ-спектрам.
Повторность измерений в экспериментах – трехкратная при постановке как минимум трех независимых серий опытов. Представленные результаты отражают усредненные величины.
Статистический анализ данных проводили с использованием теста Стьюдента, принимая достаточным критерий вероятности р < 0.05.
РЕЗУЛЬТАТЫ
Рост бактерий на 3-ХБК. Клетки штамма R. opacus 1CP после покоя быстро адаптировались к новым условиям роста в среде с 3-ХБК. При прямом высеве покоящихся клеток в жидкую минеральную среду с 3-хлорбензоатом видимый рост начинался после 48–60 ч инкубации. При последующих пересевах культуры в свежую минеральную среду с 3-ХБК рост начинался после лаг-периода, составляющего 6–8 ч. При дробных добавках 3-ХБК общее количество потребленного клетками субстрата достигало 1.0–1.5 г/л. Была определена субстратная специфичность диоксигеназы, ХБК-1,2-ДО, индуцирующейся при росте R. opacus 1CP в присутствии 3-ХБК. В табл. 1 приведены результаты оценки дыхательной активности клеток в ответ на внесение ряда замещенных бензоатов и фенолов. Установлено, что 3-ХБК-1,2-ДО, индуцирующаяся при культивировании R. opacus 1CP в присутствии 3-ХБК, отличалась от аналогичного фермента, индуцирующегося при росте клеток в присутствии бензоата. Эту разницу особенно наглядно показывало различие в активностях ферментов при использовании бензоата и 3‑ХБК в качестве субстрата. В целом фермент, индуцирующийся при росте клеток в среде с 3‑ХБК, характеризовался более широкой субстратной специфичностью по сравнению с аналогичным ферментом из клеток, выращенных в среде с бензоатом.
Таблица 1.
Относительная активность (%) фермента ХБК-1,2-ДО штаммов 1CP и 6a актинобактерии R. opacus
| Субстрат | Штаммы бактерии R. opacus | ||
|---|---|---|---|
| 6a | 1CP | ||
| Ростовой субстрат | |||
| 3-ХБК | 3-ХБК | бензоат (Solyanikova et al., 2015) | |
| Бензоат | 24 | 168 | 100 |
| 2-Хлорбензоат | 0 | 0 | 0 |
| 3-Хлорбензоат | 100 | 100 | 18 |
| 4-Хлорбензоат | 0 | 0 | 0 |
| 2-Гидроксибензоат | 48 | 56 | 0 |
| 3-Гидроксибензоат | 67 | 76 | 0 |
| 4-Гидроксибензоат | 0 | 48 | 38 |
| 2-Метилбензоат | Н.о. | 0 | 0 |
| 3-Метилбензоат | Н.о. | 56 | 9 |
| 4-Метилбензоат | Н.о. | 17 | 0 |
| 2,3-Дихлорбензоат | 18 | 34 | 0 |
| 2,4-Дихлорбензоат | 47 | 40 | 0 |
| 2,5-Дихлорбензоат | 0 | 0 | 0 |
| 2,6-Дихлорбензоат | Н.о. | 0 | 0 |
| 3,4-Дихлорбензоат | Н.о. | 0 | 0 |
| 3,5-Дихлорбензоат | Н.о. | 0 | 0 |
| Фенол | Н.о. | 0 | 4* |
| 2-Хлорфенол | Н.о. | 36 | 7* |
| 3-Хлорфенол | Н.о. | 23 | 11* |
| 4-Хлорфенол | Н.о. | 6 | 6* |
Штамм R. opacus 6a также был способен к росту в присутствии 3-ХБК. Диоксигеназа, 3-ХБК-1,2-ДО, этого штамма характеризовалась относительно широкой субстратной специфичностью. Однако обращала на себя внимание относительно низкая активность с бензоатом. В этом заключалось существенное различие 3‑ХБК-1,2-ДО сравниваемых штаммов.
Идентификация интермедиатов разложения 3‑ХБК. Были определены метаболиты, образующиеся при разложении 3-ХБК бактериями штаммов R. opacus 1CP и 6a. Анализ культуральной жидкости показал, что в случае культивирования штамма 1CP с субстратом 3-ХБК наблюдали присутствие небольшого количества 3-ХПК и 4-ХПК (максимальные значения достигали 50 и 3 мкМ соответственно). Таким образом, разложение 3‑ХБК данным штаммом сопровождалось образованием обоих монохлорпирокатехинов. В табл. 2 приведена динамика убыли 3-ХБК и образования интермедиатов в развивающейся культуре штамма 1CP.
Таблица 2.
Интермедиаты, обнаруженные нативном растворе культур R. opacus 1CP и R. opacus 6a, развивающихся в присутствии 3-ХБК. В скобках указано остаточное количество субстрата, %
| Время после добавления субстрата, ч | R. opacus 1СР | R. opacus 6a |
|---|---|---|
| 0 | 3-ХБК (100) | 3-ХБК (100) |
| 2.5 | 2-ХМ, следы 3-ХБК (99.8) |
3-ХБК (93.8) |
| 4 | 3-ХПК 4-ХПК, следы 2-ХМ 3-ХБК (98.7) |
3-ХБК (86.5) |
| 6 | 3-ХБК (88.2) | 3-ХБК (85.4) |
| 7 | 3-ХПК 4-ХПК, следы 3-ХБК (80.9) |
3-ХБК (69.1) |
| 8 | 4-ХПК, следы 3-ХБК (69.3) |
3-ХБК (64.5) |
| 21 | 3-ХБК (0) | 3-ХБК (<10) |
При развитии штамма R. opacus 6a, анализ культуральной жидкости показал только убыль 3‑ХБК, остаточное количество которой через 21 ч после ее внесения составило менее 10%, и отсутствие накопления каких-либо интермедиатов.
Клонирование генов разложения хлорпирокатехина. В работе было использовано более 10 вариантов пар праймеров, разработанных для клонирования генов, кодирующих ХПК-1,2-ДО и ХМЦИ грамположительных бактерий (Thiel, 2004). Продукты ПЦР были получены только с теми парами праймеров, последовательности которых приведены в “Материалах и методах исследования”. Использование в качестве матрицы ДНК из клеток R. opacus 6a и праймеров, специфичных к генам грамположительных бактерий, кодирующих ХПК-1,2-ДО и ХМЦИ, позволило получить продукты ожидаемого размера. Определение нуклеотидных последовательностей полученных продуктов ПЦР показало высокую степень их идентичности с соответствующими участками генов, кодирующих 4-хлорпирокатехин-1,2-диоксигеназу (4-ХПК-1,2-ДО) и 3-хлормуконатциклоизомеразу (3-ХМЦИ), участвующих в разложении 4‑ХПК штаммом 1CP (рис. 1в). Гены R. opacus 6a, кодирующие данные ферменты, были отправлены в банк генов под номерами KX137842 (4‑ХПК-1,2-ДО), KX137843 (4-ХПК-1,2-ДО), KX137844 (3-ХМЦИ), KX137845 (3-ХМЦИ).
Использование праймеров, специфичных к clcF гену, кодирующему хлормуконолактондегалогеназу штамма 1CP, и ДНК из штамма 6a в качестве матрицы не позволило получить специфический генный продукт. В положительном контроле, где реакционная смесь содержала в качестве матрицы ДНК из штамма 1CP, соответствующий продукт был получен.
Таким образом, проведенные исследования еще раз подтвердили, что у штамма 1CP стабильно присутствует локализованный на мегаплазмиде ген, кодирующий ХМЛД. А для штамма 6а было показано наличие генов, кодирующих ферменты превращения 4-ХПК (4-ХПК-1,2-ДО и 3‑ХМЦИ), и отсутствие генов, кодирующих ферменты превращения 3-ХПК по новому модифицированному орто-пути (как минимум, ХМЛД), которые у штамма 1CP участвуют в разложении 2-ХФ и 3‑ХБК.
Активность ферментов в бесклеточном экстракте. В бесклеточном экстракте культуры R. opacus 1CP, выращенной в присутствии 3-ХБК, были обнаружены активности 4-x ферментов, относящихся к модифицированному пути орто-расщепления хлорпирокатехина (табл. 3). В бесклеточном экстракте культуры R. opacus 6a, выращенной в среде с 3-ХБК, также детектировалась активность ферментов ХПК-1,2-ДО, ХМЦИ и ДЛГ. Однако не было обнаружено активности ХМЛД.
Таблица 3.
Ферменты бесклеточных экстрактов бактерий R. opacus 1CP и 6a, выращенных в присутствии 3-ХБК
| Фермент | Реакционный субстрат | Удельная активность | |
|---|---|---|---|
| ед./мг белка | % | ||
| R. opacus 1CP | |||
| ПК-1,2-ДО | Пирокатехин | 0.4283 ± 0.0126 | 100 |
| 3-Хлорпирокатехин | 0.0708 ± 0.0019 | 17 | |
| 4-Хлорпирокатехин | 0.2063 ± 0.0054 | 48 | |
| 3,5-Дихлорпирокатехин | 0.0397 ± 0.0014 | 9 | |
| 3-Метилпирокатехин | 0.4884 ± 0.0196 | 114 | |
| 4-Метилпирокатехин | 0.7755 ± 0.0401 | 181 | |
| ХМЦИ | цис,цис-Муконат | 0.0010 ± 0.0002 | 5 |
| 2-Хлормуконат | 0.0198 ± 0.0008 | 100 | |
| 2-Метилмуконат | 0.0044 ± 0.0002 | 22 | |
| ДЛГ | цис-Диенлактон | 0.1153 ± 0.0085 | |
| ХМЛИ | 5-Хлормуконолактон | 15.5100 ± 0.9716 | |
| R. opacus 6a | |||
| ПК-1,2-ДО | Пирокатехин | 0.2060 ± 0.0094 | 100 |
| 3-Хлорпирокатехин | 0.2041 ± 0.0067 | 100 | |
| 4-Хлорпирокатехин | 0.0513 ± 0.0019 | 25 | |
| 3,5-Дихлорпирокатехин | 0.0084 ± 0.0002 | 4 | |
| 3-Метилпирокатехин | 0.2072 ± 0.0101 | 100 | |
| 4-Метилпирокатехин | 0.3632 ± 0.0129 | 176 | |
| МЦИ | цис,цис-Муконат | 0 | 0 |
| 2-Метилмуконат | 0 | 0 | |
| 3-Метилмуконат | 0.0019 ± 0.0008 | 45 | |
| 2-Хлормуконат | 0.0042 ± 0.0003 | 100 | |
| ДЛГ | цис-Диенлактон | 0.1242 ± 0.0098 | |
| ХМЛИ | 5-Хлормуконолактон | 0 | |
УФ-спектроскопия. Проведение УФ-спектроскопии показало, что под действием ферментов бесклеточного экстракта штамма 1CP происходило снижение поглощения, характерное для раствора 2-хлормуконата (2-ХМ) (максимум поглощения соединения – 278 нм) (рис. 2а). Значительного увеличения пика (максимум поглощения при 207 нм), соответствующего накоплению 5-хлормуконолактона (5-ХМЛ), не было отмечено, по-видимому, потому, что в бесклеточном экстракте присутствовали и другие ферменты исследуемого пути, препятствующие накоплению метаболита. При использовании в качестве субстрата 5-ХМЛ под действием ферментов бесклеточного экстракта штамма 1CP наблюдали снижение поглощения при 207 нм, характерного для 5-ХМЛ, и образование пика c максимумом поглощения при 280 нм, который характерен для цис-диенлактона (рис. 2б).
Рис. 2.
Изменения в спектрах поглощения реакционной смеси, содержащей 2-ХМ (а) и 5-ХМЛ (б), под действием ферментов бесклеточного экстракта штамма R. opacus 1CP, развивающегося в среде с 3‑ХБК.
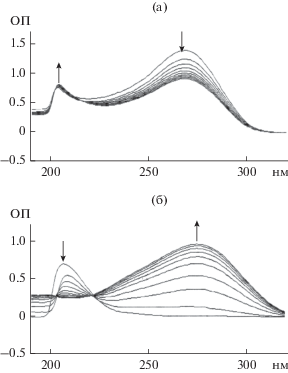
При добавлении бесклеточного экстракта штамма 6a к реакционной смеси, содержащей 2‑ХМ, также наблюдали изменения в спектре поглощения раствора: снижение пика при 270 нм, что соответствует убыли 2-ХМ, и незначительное увеличение пика при 207 нм, соответствующего образованию 5-ХМЛ. Для этого же штамма при добавлении бесклеточного экстракта к реакционной смеси, содержащей раствор 5-ХМЛ, изменений в спектре поглощения не было отмечено. Это указывало на отсутствие активности ХМЛД в бесклеточном экстракте штамма 6a, выращенного в минеральной среде с 3-ХБК.
ОБСУЖДЕНИЕ
Способность бактериальных штаммов разлагать устойчивые ксенобиотические соединения определяется генами биодеградации (Schlömann, 1994). Эти гены зачастую расположены на плазмидах и могут передаваться от бактерии к бактерии (Liang et al., 2012). Штаммы 1CP и 6a бактерии R. opacus являются эффективными деструкторами (хлор)ароматических соединений. Исследование ферментов деградации этих соединений у обоих штаммов показало, что они во многом идентичны. Однако вопрос о степени сходства деградативных генов до сих пор оставался открытым.
Первый этап деструкции (хлор)бензоатов происходит под действием (хлор)бензоат-1,2-диоксигеназы – двухкомпонентной системы, состоящей из редуктазного и оксидазного компонентов; последний представлен двумя типами субъединиц, α и β (Field, Sierra-Alvarez, 2008). Считается, что α-субъединица отвечает за субстратную специфичность БК-1,2-ДО (Parales, Resnick, 2006; Kweon et al., 2010). Как было показано ранее, R. opacus 1CP изначально был способен к росту на 3-ХБК после длительного, в несколько месяцев, периода адаптации (Моисеева и соавт., 2001). При прямом высеве покоящихся клеток в жидкую минеральную среду с 3-ХБК видимый рост культуры начинался в течение 48–60 ч, а последующие пересевы приводили к значительному сокращению лагфазы. Исследованные в данной работе 3-хлорбензоат-1,2-диоксигеназы были узкоспецифичны по отношению к монохлорированным бензоатам. 3-ХБК-1,2-ДО из клеток R. opacus 1CP и R. opacus 6a не были активны ни с 2-ХБК, ни с 4-ХБК. Аналогично, два штамма, идентифицированные как Rhodococcus ruber и R. pyridinivorans и способные разлагать 3-ХБК, проявили крайне низкую активность по отношению к 2- и 4-хлорбензоатам (Wang et al., 2018).
Не меньший интерес представлял ответ на вопрос, какой интермедиат образуется в качестве основного в результате воздействия хлорбензоат-1,2-диоксигеназы на 3-ХБК – 3-ХПК или 4-ХПК. Из литературы известно, что окисление 3-ХБК может проходить с образованием как 3-ХПК, так и 4-ХПК. Напомним, что псевдомонады способны деградировать 3-ХБК с образованием смеси 3‑ХПК и 4-ХПК (Reineke, Knackmuss, 1978; Xu et al., 2017). Процесс трансформации ряда моно-хлорароматических соединений, несущих заместитель в мета-положении (3-ХФ, 3-ХБК и 3-ХА) изучали у бактерий рода Rhodococcus (Janke et al., 1988a, 1988b, 1989; Ihn et al., 1989; Janke, Ihn, 1989). Полученные результаты показали, что в силу специфичности ферментов начальной атаки единственным продуктом, образующимся при трансформации родококками мета-хлорированных ароматических соединений, был 4-ХПК. Таким образом, на основании представленных ранее работ можно было бы предположить, что дальнейшее превращение мета-замещенных хлорированных соединений, таких как 3-ХБК, 3-ХФ и 3‑ХА, родококками, грамположительными актинобактериями, должно проходить с образованием только 4-ХПК, в то время как грамотрицательные бактерии способны к образованию как 3‑ХПК, так и 4-ХПК. Тем не менее, ранее мы показали, что разложение 3-ХБК культурой R. opacus 1CP проходило под действием ферментов нового модифицированного орто-пути с 3-ХПК в качестве ключевого интермедиата (рис. 1б). Этот путь катализируют 3-ХПК-1,2-ДО, 2-ХМЦИ, ХМЛД и ДЛГ (Моисеева и соавт., 2001; Moiseeva et al., 2002). Субстратная специфичность ферментов этого нового модифицированного орто-пути у R. opacus 1CP позволяла ему осуществлять превращение 3-хлорпирокатехина через 5-хлормуконолактон в диенлактон (Moiseeva et al., 2002). Диенлактон был представлен цис-изомером и под действием диенлактонгидролазы, активной только с этим изомером, превращался в β-кетоадипат. Субстратная специфичность ферментов модифицированного пути орто-расщепления 4-ХПК (рис. 1в) штамма 1CP культуры R. opacus такова, что при наличии только этих ферментов штамм не способен эффективно разлагать 3-ХПК: конечным продуктом превращения 3-ХПК ферментами 4-ХПК ветви был 5-ХМЛ (Solyanikova et al., 1995). Таким образом, у штамма 1CP для полной утилизации 3-ХБК (с образованием 3-ХПК в качестве интермедиата) имеется новый, модифицированный орто-путь, а 4-ХФ превращается по 4‑хлорпирокатехиновой ветви. Как видно из полученных данных, для штамма R. opacus 6а, как и для других актинобактерий, характерно окисление 3-ХБК до 4-ХПК. У штамма 6a 4-ХПК также является интермедиатом разложения 4-ХФ. Далее происходит превращение 4-ХПК по пути его орто-расщепления. Этот путь катализируют ферменты 4-ХПК-1,2-ДО, 3-ХМЦИ и ДЛГ. Более того, практически полная идентичность 4-ХПК-1,2-ДО и 3-ХМЦИ у штаммов R. opacus 1CP и 6a указывает на одинаковый источник ферментов биодеградации у этих микроорганизмов и на возможность превращения 3-ХБК обоими штаммами по пути образования 4-ХПК, что свойственно другим родококкам. Образование ключевого интермедиата разложения 3-ХБК, 3-ХПК или 4‑ХПК, и путь его дальнейшего превращения определяется особенностями первого фермента, 3-ХБК-1,2-ДО.
ХМЛД (=СlcF) – фермент, определяющий способность штамма 1CP использовать 2-ХФ в качестве единственного источника роста, разлагая образующийся из него 3-ХПК по новому модифицированному орто-пути (рис. 1б). Наличие гена, кодирующего СlcF, у R. opacus 1CP было показано ранее (Moiseeva et al., 2002). Ген clcF, идентичный гену штамма R. opacus 1CP, обнаружен у нескольких представителей родококков другого вида – R. wratislaviensis (Solyanikova et al., 2014). Однако клетки R. wratislaviensis G10 разлагали 3‑ХБК без индукции ключевого фермента, ClcF. Это позволило сделать вывод о том, что разложение 3-ХБК данной культурой также проходило с образованием не 3-, а 4-ХПК, как и у штамма 6а культуры R. opacus, не имеющего ХМЛД.
Известно, что под давлением внешних факторов, к которым относится и загрязнение окружающей среды ксенобиотиками разных классов и различного назначения, микроорганизмы получают способность разлагать большинство этих соединений в результате передачи и распространения генов биодеградации внутри микробного сообщества (Liang et al., 2012). К настоящему времени получены многочисленные свидетельства передачи генетического материала, что выражается в присутствии гомологичных или даже полностью идентичных генов у разных бактериальных штаммов. Вместе с тем, остается неясным, всегда ли необходима передача дополнительного генетического материала. Полученные результаты указывают на то, что разложение субстрата (в данном случае 3-ХБК) происходит с одинаковой эффективностью как у штамма, несущего дополнительные гены биодеградации (R. opacus 1CP, R. wratislaviensis G10), так и у штамма, их лишенного (R. opacus 6a). При этом анализ генного профиля (clcF+, clcF−), ферментного профиля ([4‑ХПК-1,2-ДО + 3-ХМЦИ] или [3-ХПК-1,2-ДО + + 2-ХМЦИ + ХМЛД]) и метаболитного профиля ([4-ХПК + 3-ХМК + цис-ДЛ] или [3-ХПК + 2-ХМК + + 5-ХМЛ + цис-ДЛ]) показывает, что даже при наличии соответствующих генов, как в случае штаммов R. opacus 1CP и R. wratislaviensis G10, разложение мета-замещенного бензоата определяется особенностями фермента первого этапа окисления, а не генным набором.
Список литературы
Горлатов С.Н., Мальцева О.В., Шевченко В.И., Головлева Л.А. Разложение хлорфенолов культурой Rhodococcus erythropolis // Микробиология. 1989. Т. 58. С. 802–806.
Gorlatov S.N., Maltseva O.V., Shevchenko V.I., Golovleva L.A. Degradation of chlorophenols by a culture of Rhodococcus erythropolis // Microbiology (Moscow). 1989. V. 58. P. 647–651.
Коновалова Е.И., Соляникова И.П., Головлева Л.А. Разложение 4-хлорфенола штаммом Rhodococcus opacus 6a // Микробиология. 2009. Т. 78. С. 847–849.
Konovalova E.I., Solyanikova I.P., Golovleva L.A. Degradation of 4-chlorophenol by bacterium Rhodococcus opacus 6a // Microbiology (Moscow). 2009. V. 78. P. 805–807.
Моисеева О.В., Белова О.В., Соляникова И.П., Шлеманн М., Головлева Л.А. Ферменты нового модифицированного орто-пути из Rhodococcus opacus 1CP, утилизирующего 2-хлорфенол // Биохимия. 2001. Т. 66. С. 678–687.
Moisseeva O.V., Belova O.V., Solyanikova I.P., Schlömann M., Golovleva L.A. Enzymes of novel modified ortho-pathway of Rhodococcus opacus 1CP, utilizing 2-chlorophenol // Biochemistry (Moscow). 2001. V. 66. P. 548–555.
Моисеева О.В., Линько Е.В., Баскунов Б.П., Головлева Л.А. Разложение 2-хлорфенола и 3-хлорбензоата культурой Rhodococcus opacus 1CP // Микробиология. 1999. Т. 68. С. 400–405.
Соляникова И.П., Борзова О.В., Емельянова Е.В., Шумкова Е.С., Присяжная Н.В., Плотникова Е.Г., Головлева Л.А. Диоксигеназы, индуцирующиеся при разложении бензоата деструкторами хлорбифенилов Rhodococcus wratislaviensis G10 и хлорфенолов Rhodococcus opacus 1CP, и гены, потенциально вовлеченные в этот процесс // Биохимия. 2016. Т. 81. С. 1239–1253.
Solyanikova I.P., Borzova O.V., Emelyanova E.V., Shumkova E.S., Prisyazhnaya N.V., Plotnikova E.G., Golovleva L.A. Dioxygenases of chlorobiphenyl-degrading species Rhodococcus wratislaviensis G10 and chlorophenol-degrading species Rhodococcus opacus 1CP induced in benzoate-grown cells and genes potentially involved in these processes // Biochemistry (Moscow). 2016. V. 81. P. 986–999.
Abbasian F., Lockington R., Megharaj M., Naidu R. A review on the genetics of aliphatic and aromatic hydrocarbon degradation // Appl. Biochem. Biotechnol. Part A: Enzyme Engineering and Biotechnology. 2016. V. 178. P. 224–250.
Adebusoye S.A. Biological degradation of 4-chlorobenzoic acid by a PCB-metabolizing bacterium through a pathway not involving (chloro)catechol // Biodegradation. 2017. V. 28. P. 37–51.
Ajithkumar P.V., Kunhi A.A.M. Pathways for 3-chloro- and 4-chlorobenzoate degradation in Pseudomonas aeruginosa 3mT // Biodegradation. 2000. V. 11. P. 247–261.
Aken B.V., Correa P.A., Schnoor J.L. Phytoremediation of polychlorinated biphenyls: new trends and promises // Environ. Sci. Technol. 2010. V. 44. P. 2767–2776.
Baggi G., Zangrossi M. Assessment of the biodegradative potential versus chlorobenzoates as single or mixed compounds in a stable microbial consortium // Annu. Rev. Microbiol. 2001. V. 51. P. 179–188.
Boyle A.W., Silvin C.J., Hassett J.P., Nakas J.P., Tanenbaum S.W. Bacterial PCB degradation // Biodegradation. 1992. V. 3. P. 285–298.
Engesser K.H., Auling G., Busse J., Knackmusss H.-J. 3-Fluorobenzoate enriched bacterial strain FLB300 degrades benzoate and all three isomeric monofluorobenzoates // Arch. Microbiol. 1990. V. 153. P. 193–199.
Field J.A., Sierra-Alvarez R. Microbial transformation and degradation of polychlorinated biphenyls // Environ. Pollut. 2008. V. 155. P. 1–12.
Field J.A., Sierra-Alvarez R. Microbial transformation of chlorinated benzoates // Reviews in Environmental Science and Bio/Technology. 2008. V. 7. P. 191–210.
Gribble G.W. The abundant natural sources and uses of chlorinated chemicals // Am. J. Public Health. 1994. V. 84. P. 1183.
Gröning J.A.D., Roth C., Kaschabek S.R., Sträter N., Schlömann M. Recombinant expression of a unique chloromuconolactone dehalogenase ClcF from Rhodococcus opacus 1CP and identification of catalytically relevant residues by mutational analysis // Arch. Biochem. Biophys. 2012. V. 526. P. 69–77.
Harayama S., Rekik M., Bairoch A., Neidle E.L., Ornston L.N. Potential DNA slippage structures acquired during evolutionary divergence of Acinetobacter calcoaceticus chromosomal benABC and Pseudomonas putida TOL pWW0 plasmid xylXYZ, genes encoding benzoate dioxygenases // J. Bacteriol. 1991. V. 173. P. 7540–7548.
Hartmann J., Reineke W., Knackmuss H.-J. Metabolism of 3-chloro-, 4-chloro-, and 3,5-dichlorobenzoate by a pseudomonad // Appl. Environ. Microbiol. 1979. V. 37. P. 421–428.
Horvath R.S., Alexander M. Cometabolism of meta-chlorobenzoate by an Arthrobacter // Appl. Microbiol. 1970. V. 20. P. 254–258.
Ichihara A., Adachi K., Hosokawa K., Takeda Y. The enzymatic hydroxylation of aromatic carboxylic acids; substrate specificities of anthranilate and benzoate oxidases // J. Biol. Chem. 1962. V. 237. P. 2296–2302.
Janke D., Al-Mofarji T., Schukat B. Critical steps in degradation of chloroaromatics by rhodococci. II. Whole-cell turnover of different monochloroaromatic non-growth substrates by Rhodococcus sp. An 117 and An 213 in the absence/presence of glucose // J. Basic Microbiol. 1988a. V. 28. P. 519–528.
Janke D. Al-Mofarji T., Straube G., Schumann P., Prauser H. Critical steps in degradation of chloroaromatics by rhodococci. I. Initial enzyme reactions involved in catabolism of aniline, phenol and benzoate by Rhodococcus sp. An 117 and An 213 // J. Basic Microbiol. 1988b. V. 28. P. 509–518.
Ihn W., Janke D., Tresselt D. Critical steps in degradation of chloroaromatics by rhodococci. III. Isolation and identification of accumulating intermediates and dead-end products // J. Basic Microbiol. 1989. V. 29. P. 291–297.
Janke D., Ihn W., Tresselt D. Critical steps in degradation of chloroaromatics by rhodococci IV. Detailed kinetics of substrate removal and product formation by resting pre-adapted cells // J. Basic Microbiol. 1989. V. 29. P. 305–314.
Janke D., Ihn W. Cometabolic turnover of aniline, phenol and some of their monochlorinated derivatives by the Rhodococcus mutant strain AM 144 // Arch. Microbiol. 1989. V. 152. P. 347–352.
Kitagawa W., Miyauchi K., Masai E., Fukuda M. Cloning and characterization of benzoate catabolic genes in the gram-positive polychlorinated biphenyl degrader Rhodococcus sp. strain RHA1 // J. Bacteriol. 2001. V. 183. P. 6598–6606.
Kweon O., Kim S.J., Freeman J.P., Song J., Baek S., Cerniglia C.E. Substrate specificity and structural characteristics of the novel Rieske nonheme iron aromatic ring-hydroxylating oxygenases NidAB and NidA3B3 from Mycobacterium vanbaalenii PYR-1 // mBio. 2010. V. 1. pii: e00135-10.
Liang B., Jiang J., Zhang J., Zhao Y., Li S. Horizontal transfer of dehalogenase genes involved in the catalysis of chlorinated compounds: evidence and ecological role // Crit. Rev. Microbiol. 2012. V. 38. P. 95–110.
Moiseeva O.V., Solyanikova I.P., Kaschabek S., Thiel M., Golovleva L.A., Schlömann M. A new modified ortho-cleavage pathway of 2-chlorophenol degradation by Rhodococcus opacus 1CP: genetic and biochemical evidences // J. Bacteriol. 2002. V. 184. P. 5282–5292.
Parales R.E., Resnick S.M. Aromatic ring hydroxylating dioxygenases // Pseudomonas. 2006. V. 4. P. 287–340.
Reineke W., Knackmuss H.-J. Chemical structure and biodegradability of halogenated aromatic compounds. Substituent effects on 1,2-dioxygenation of benzoic acid // Biochim. Biophys. Acta. 1978. V. 542. P. 412–423.
Schlömann M. Evolution of chlorocatechol catabolic pathways // Biodegradation. 1994. V. 5. P. 301–321.
Schlömann M., Schmidt E., Knackmuss H.-J. Different types of dienelactone hydrolase in 4-fluorobenzoate-utilizing bacteria // J. Bacteriol. 1990. V. 172. P. 5112–5118.
Solyanikova I.P., Emelyanova E.V., Borzova O.V., Golovleva L.A. Benzoate degradation by Rhodococcus opacus 1CP after a dormancy: characterization of dioxygenases involved in the process // J. Environ. Sci. Health. Part B. 2016. V. 51. P. 182–191.
Solyanikova I.P., Maltseva O.V., Vollmer M.D., Golovleva L.A., Schlömann M. Characterization of muconate and chloromuconate cycloisomerase from Rhodococcus erythropolis 1CP: indications for functionally convergent evolution among bacterial cycloisomerases // J. Bacteriol. 1995. V. 177. P. 2821–2826.
Solyanikova I.P., Plotnikova E.G., Shumkova E.S., Robota I.V., Prisyazhnaya N.V., Golovleva L.A. Chloromuconolactone dehalogenase ClcF of actinobacteria // J. Environ. Sci. Health. Part B. 2014. V. 49. P. 422–431.
Thiel M. Entwicklung von PCR-Primern zum Nachweis von Genen des Chloraromaten-Abbaus in mikrobiellen Lebensgemeinschaften. PhD Thesis. Technischen Universität Bergakademie, Freiberg, Germany. 2004. 183 p.
Wang H., Hu J., Xu K., Tang X., Xu X., Shen C. Biodegradation and chemotaxis of polychlorinated biphenyls, biphenyls, and their metabolites by Rhodococcus spp. // Biodegradation. 2018. V. 29. P. 1–10. https://doi.org/10.1007/s10532-017-9809-6
Willes R.F., Nestmann E.R., Miller P.A., Orr J.C., Munro I.C. Scientific principles for evaluating the potential for adverse effects from chlorinated organic chemicals in the environment // Regul. Toxicol. Pharmacol. 1993. V. 18. P. 313–356.
Xu C., Zang X., Hang X., Liu X., Yang H., Liu X., Jiang J. Degradation of three monochlorobenzoate isomers by different bacteria isolated from a contaminated soil // Int. Biodeter. Biodegr. 2017. V. 120. P. 192–202. https://doi.org/10.1016/j.ibiod.2017.02.020
Yamaguchi M., Fujisawa H. Reconstitution of iron-sulfur cluster of NADH-cytochrome c reductase, a component of benzoate 1,2-dioxygenase system from Pseudomonas arvilla C-1 // J. Biol. Chem. 1981. V. 256. P. 6783–6787.
Zhang G., Yang X., Xie F., Chao Y., Qian S. Cometabolic degradation of mono-chloro benzoic acids by Rhodococcus sp. R04 grown on organic carbon sources // World J. Microbiol. Biotechnol. 2009. V. 25. P. 1169–1174.
Дополнительные материалы отсутствуют.


