Микробиология, 2020, T. 89, № 2, стр. 235-246
Молекулярный анализ микробных сообществ ризосфер злаков, выращенных на контрастных почвах
А. О. Зверев a, *, Е. В. Першина a, В. М. Шапкин a, А. К. Кичко a, О. П. Митрофанова b, В. Д. Кобылянский b, О. С. Юзихин c, А. А. Белимов a, Е. Е. Андронов a, d
a Федеральное государственное бюджетное научное учреждение “Всероссийский научно-исследовательский институт сельскохозяйственной микробиологии”
196608 Санкт-Петербург, Россия
b Федеральный исследовательский центр “Всероссийский институт генетических ресурсов растений
им. Н.И. Вавилова”
190000 Санкт-Петербург, Россия
c Федеральное государственное бюджетное научное учреждение “Всероссийский научно-исследовательский институт защиты растений”
196608 Санкт-Петербург, Россия
d Санкт-Петербургский государственный университет
199034 Санкт-Петербург, Россия
* E-mail: azver.bio@gmail.com
Поступила в редакцию 19.03.2018
После доработки 25.07.2019
Принята к публикации 29.09.2019
Аннотация
В работе изучали изменения микробного состава ризосферных сообществ при выращивании различных видов и сортов сельскохозяйственных злаков (ржи и пшеницы) в контрастных по ряду агрохимических признаков почвах. Представлены результаты анализа микробного сообщества ризосфер (секвенирование V4-вариабельного участка гена 16S рДНК), а также данные ионного и масс-спектрометрического состава почвенной вытяжки. Индексы альфа-разнообразия ризосферных и контрольных почвенных сообществ практически не различались. Анализ бета-разнообразия показал большую изменчивость микробных сообществ ризосфер растений, выращенных на черноземе. Ризосферные сообщества растений, выращенных на дерново-подзолистой почве, различались меньше и обособлены в отдельные кластеры. Были выявлены таксоны, достоверно принимающие участие в формировании специфического ризосферного микробиома ржи и пшеницы. Найдены как общие ризосферные таксоны (Sphingobacteriia, Betaproteobacteria), так и видо- и сортоспецифичные (Oxalobacteraceae, Sphingobacteriaceae). Содержание ионов в составе почвенной вытяжки существенно зависело от типа почвы, а сходство масс-спектральных профилей в большей степени определялось видом растения, чем типом почвы.
Изучению воздействия растения на сообщество микроорганизмов почвы посвящено большое количество работ. Тем не менее, лишь с появлением высокопроизводительного секвенирования и развития метагеномики стало возможно наиболее полно описывать микробные сообщества, с учетом не только культивируемых, но и некультивируемых форм микроорганизмов (Handelsman, 2004).
В настоящее время исследованию структуры прикорневых микробных сообществ с использованием метагеномных методов посвящено множество статей и крупных научных проектов (Gottel et al., 2011; Knief et al., 2012; Mendes et al., 2014). Исследователями отмечены различия в таксономическом составе ризосферных микробных сообществ однодольных и двудольных растений. Так в сообществах двудольных, изучаемых, главным образом, на примере Arabidopsis thaliana, показано увеличение числа представителей Actinobacteria (Streptomicetaceae), протеобактерий (главным образом Pseudomonadaceae), однодольные же растения характеризовались сравнительно большей долей Bacteroidetes и Rhizobiales (Bulgarelli et al., 2015).
Основная масса работ посвящена исследованию двудольных, и, в частности, модельного объекта A. thaliana. Одним из самых значимых исследований является работа Лундберга и соавт. (Lundberg et al., 2012). В работе с использованием методов высокопроизводительного секвенирования было проведено исследование таксономической структуры микробиомов в исходных почвах, ризосфере и эндосфере различных сортов A. thaliana. Наиболее выраженные изменения в составе микробиомов были связаны с действием растительного фактора (с изменением состава сообщества микроорганизмов под действием развивающегося растения), следующим по силе воздействия были тип почвы, а затем стадия развития и его генотип (Lundberg et al., 2012). Было показано, что микробные сообщества эндосфер в значительной степени обособлены от сообществ почвы и ризосферы. По сравнению с эндосферой, почвенные и ризосферные микробные сообщества демонстрировали выраженное сходство таксономических составов. Таксономический анализ показал присутствие в сообществах исходных почв большого количества представителей Proteobacteria (35%), Bacteroidetes (20%), Acidobacteria (18%), также отмечено присутствие Actinobacteria, Firmicutes, Gemmatimonadates. В ризосферных сообществах достоверно увеличивалась доля протеобактерий за счет появления в сообществе бактерий из сем. Pseudomonadaceae (Lundberg et al., 2012).
Значительная часть сельскохозяйственных культур (пшеница, рожь, просо и т.д.), являются однодольными. Изучению микробных сообществ таких культур посвящено меньшее количество работ. Тем не менее, на сегодняшний день существуют исследования, описывающие микробиомы пшеницы (Donn et al., 2015), кукурузы (Peiffer et al., 2013), риса (Edwards et al., 2015), ячменя (Bulgarelli et al., 2015).
Результаты, полученные в целом ряде исследований однодольных растений, оказались схожи. Наиболее показательной можно назвать работу по изучению закономерностей формирования ризосферного сообщества ячменя обыкновенного (Hordeum vulgare) (Bulgarelli et al., 2015), в которой было показано снижение показателей альфа- и бета-разнообразия в ряду почва‒ризосфера‒эндосфера. Исходная почва была богата представителями фил Actinobacteria, Bacteroidetes, и Proteobacteria; они составляли до 92% сообщества. В ризосферном сообществе, по сравнению с сообществами исходной почвы, увеличивалась доля филы Bacteroidetes, в частности, Flavobacteriaceae, Rizobiaceae и Commamonodaceae. При этом численность бактерий из фил Firmicutes и Chloroflexi уменьшалась (Bulgarelli et al., 2015).
Как уже упоминалось выше, наиболее выраженные изменения в составе микробиомов были связаны с развитием растения и локализацией сообщества (почва, ризосфера, ризоплана). Вторым по силе воздействия на ризосферное сообщество являлся почвенный фактор (Berg et al., 2009; Girvan et al., 2013). Ряд исследований показывают, что в зависимости от того, насколько различаются почвы, воздействие этого фактора объясняет от 7.5 (Edwards et al., 2015) до 27% (Winston et al., 2014) различий в структуре микробиомов ризосферы. В контрастных почвах, микробный состав которых сильно различается, сильно различаются между собой и таксономические составы ризосферных сообществ на этих почвах (Peiffer et al., 2013).
Фактор вида и сорта растения – его генотипа – рассматривается в ряде экспериментов наряду с типом почвы и растительным фактором. Примером может служить изучение ризосферных сообществ трех видов агав ‒ Agave tequilana, A. salmiana и A. deserti (Coleman-Derr et al., 2016). В ризосферном сообществе A. tequilana, используемого в сельскохозяйственных посадках, было обнаружено существенное увеличение доли Pseudomonadales (около 25% общего состава сообщества), а также Enterobacteriales (около 40%). Дикие агавы показывали большее разнообразие. По сравнению с сообществом исходной почвы, доля представителей Bacilliales, Actinomycetales и Xanthomonadales увеличивалась (Coleman-Derr et al., 2016). Анализ ризосферных сообществ ячменя (Bulgarelli et al., 2015) и кукурузы (Peiffer et al., 2013) свидетельствует о том, что влияние генотипа растения сказывается не столько на присутствии тех или иных таксонов (как это показано для фактора растения и типа почвы) в ризосфере, сколько на изменении их частоты. При анализе микробиомов ячменя генотип растения оказался достоверно значимым фактором (5.7% различий в структуре микробиомов), определяющим состав ризосферного микробиома, в то время как для A. thaliana этот эффект выражен меньше (Lundberg et al., 2012). Эту же концепцию подтверждает исследование нескольких сортов Cannabis sativa и C. indica (Winston et al., 2014).
Влияние генотипа растения может сказываться не только на таксономической структуре сообщества, но и на показателях альфа- и бета-разнообразия сообщества. Так, анализ ризосферных сообществ пяти сортов риса, принадлежащих трем разным видам, продемонстрировал значительные (до полутора раз) различия в показателях альфа-разнообразия между микробными сообществами (Edwards et al., 2015).
Стоит подчеркнуть, что все представленные выше результаты должны восприниматься с осторожностью, поскольку методологическая база для подобного рода исследований пока разработана слабо. Особенно существенно сказывается отсутствие единой методики дифференциального фракционирования микробиомных сообществ в ряду почва‒ризосфера‒ризоплана‒эндосфера.
В настоящее время большинство исследований микробных сообществ прикорневой зоны растений проводится на модельных объектах, таких, как A. thaliana (Chaparro et al., 2013) или Zea mays (Aira et al., 2010). При этом сравнительно малый процент составляют работы, посвященные исследованию хозяйственно-ценных однодольных культур, таких как рожь и пшеница.
Целью настоящей работы было изучить изменения микробного сообщества ржи и пшеницы в двух контрастных, широко распространенных в сельском хозяйстве почвах, а также выяснить, существует ли видо- и сортоспецифичные изменения в ионном и молекулярном составе почвенных вытяжек.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объекты исследования. Для работы были отобраны почвы, принадлежащие к группам черноземных и дерново-подзолистых. Выбор объектов был обусловлен их контрастными свойствами и широким распространением на территории России. Данные почвы количественно различаются по общему содержанию органического вещества, гранулометрическому составу и другим физико-химическим показателям. Главной особенностью дерново-подзолистой почвы является кислая реакция среды. Чернозем, в свою очередь, содержит большое количество органических веществ, обладает высокой влагоемкостью и рН близким к нейтральному.
Для выявления влияния типа почвы на формирование ризосферного микробиома сравнивали образцы, принадлежащие разным типам почв (контролей), с образцами почв ризосфер всех растений; для выявления видовых и сортовых различий сравнивали образцы почвы ризосфер растений. Различия выявлялись как между отдельными образцами, так и между образцами, объединенными по одному из признаков (тип почвы, вид или сорт растения).
Дерново-подзолистая почва была предоставлена Псковским НИИСХ и совхозом “Родина” Псковской области (координаты точки сбора – 57°50′44.2′′ N, 28°12′03.7′′ E). Чернозем получен из заповедника “Каменная степь” Воронежской области (51°01′41.6′′ N, 40°43′39.3′′ E).
Отбор почв для проведения экспериментов производили на участках сельскохозяйственного назначения, на разделительных кромках полей (свободных от посевов в течение последних 50 лет), с глубины 3‒15 см.
С целью усреднения проб полученная почва была просеяна на грохоте с ячейкой 5 мм, подсушена, расфасована в пластиковые сосуды: 5 кг для чернозема и 5.5 кг для дерново-подзолистой почвы. Почва была увлажнена из расчета 75% максимальной влагоемкости.
Семена получены из коллекции ВИР. Сорта пшеницы: по каталогу ВИР К-54609 (далее обозначен как W5) и К-9084 (далее обозначен как W2). Сорта ржи: по каталогу ВИР К-6469 (далее обозначен как R1) и К-7856 (далее обозначен как R3).
Постановка вегетационного опыта. Закладка вегетационного опыта по культивированию двух контрастных сортов Triticum aestivum (Пшеница мягкая) и двух сортов Secale cereale (Рожь посевная) была произведена в дерново-подзолистой почве и черноземе. В сосуды с почвой вносили семена из расчета 25 шт. на сосуд на глубину 3‒5 см. На один сорт использовали по два сосуда с каждым типом почв.
Эксперимент проводили в течение 42 сут. Средняя дневная температура за время проведения эксперимента составляла 13°С, ночная – 4°С. Влажность почвы поддерживалась на уровне 75% от полной влагоемкости.
После завершения вегетационного опыта из каждого сосуда были отобраны по 2 образца корневой массы: отделенные от почвы корни были поделены на 2 части, после чего помещены в 50-мл флакон с водой для дальнейшего встряхивания в течение 1 мин. Таким образом, для каждого варианта опыта было отобрано 4 повторности. Из полученной однородной почвенной суспензии были отобраны по 2 мл для центрифугирования. Осадок был использован для выделения тотальной ДНК ризосферы. Поскольку во всех случаях навеска почвы была ориентировочная, дальнейший анализ включал только качественные характеристики микробных сообществ.
Выделение и очистку ДНК осуществляли в соответствии с методикой, разработанной во ВНИ-ИСХМ (Андронов и соавт., 2011). Разрушение образца производили на гомогенизаторе FastPrep с использованием высокоскоростного встряхивания по орбите, исключающей возникновение стационарно закрученных потоков в пробирке. Средняя концентрация ДНК составила 18 нг/мл. Очищенный препарат ДНК использовали в качестве матрицы в реакции ПЦР с универсальными праймерами к вариабельному участку V4 гена 16S рРНК F515 GTGCCAGCMGCCGCGGTAA и R806 GGACTACVSGGGTATCTAAT (Bates, 2011). Подготовку проб и секвенирование проводили на приборе GS Junior (“Roche”, США) согласно рекомендациям производителя.
Биоинформационный анализ данных. Анализ полученных по результатам секвенирования нуклеотидных последовательностей гена 16S рРНК производил с использованием программы QIIME (версия 1.8.0) (Caporaso et al., 2010). Были исключены последовательности длиной менее 200 нуклеотидов, имеющие параметр качества прочтения (qualityscore) менее 25, химерные и небактериальные/неархейные последовательности. Последовательности с процентом сходства, превышающим 97%, объединяли в операционные таксономические единицы (ОТЕ) с использованием алгоритма de novo (Caporaso et al., 2010). В качестве базы нуклеотидных последовательностей использовалacь база GreenGenes версии 12.8 (DeSantisetal., 2006).
Для оценки биоразнообразия и проведения сравнительного анализа сообществ при помощи того же пакета QIIME были рассчитаны параметры альфа- и бета-разнообразия. Альфа-разнообразие оценивали с использованием индексов видового богатства (число ОТЕ в образце) и индекса Шеннона, а достоверность различий по индексам альфа-разнообразия среди микробиомов ‒ с использованием t-теста. Для оценки бета-разнообразия использовали методы “weighted unifrac” и “unweighted unifrac”, позволяющие оценить процент сходств/различий между всеми парами сравниваемых микробиомов (Lozupone et al., 2007). Результаты были представлены с использованием методов многомерной статистики PCoA – principal components analysis (анализ главных компонент).
Для подсчета индексов разнообразия и проведения кластерного анализа использовали критерий Bray-Curtis, вычисления осуществляли в программе PAST (Hammer et al., 2001).
Различия в частотах таксонов между образцами опыта определяли посредством проведения точного теста Фишера с поправкой на множественные сравнения по процедуре Бенджамини‒Хохберга на 5% уровне значимости.
Для исследования таксономического состава сообщества использовали только те ОТЕ, суммарное количество сиквенсов которых (для всех исследуемых образцов) было больше 8. Это эмпирически найденное ограничение необходимо для исключения из анализа ОТЕ, присутствующих в образцах в незначительном количестве.
Масс-спектрометрия. Для получения данных о молекулярном составе почвы использовали водную экстракцию. Полученную почвенную суспензию центрифугировали, осаждая твердые частицы. Супернатант использовали для анализа масс-спектрального состава, проводившегося на установке WatersAcquityHPLC при помощи прямого ввода; ионизация в электроспрее (напряжение 3.8 кВ). Для регистрации использовался времяпролетный детектор. Обработку результатов производили в программе BioNumerics 7 (“BioNumerics”).
Анализ ионного состава. Содержание доступных растениям форм питательных (биогенных) и токсичных (абиогенных) элементов в ризосферной почве изучали в лабораторных условиях путем получения почвенных вытяжек 0.1 М раствором HNO3. Вытяжки центрифугировали 10 мин при 7000 g и в супернатантах определяли содержание питательных макро- и микроэлементов (P, K, Ca, Mg, S, Fe, B, Zn, Mn, Na, Co, Cr, Cu, Ni), а также токсичных элементов (Сd, Pb, Hg) с помощью оптического эмиссионного спектрометра параллельного действия с индуктивно-связанной плазмой ICPE-9000, по методикам производителя. Контролем служили образцы неризосферной почвы. Обработку результатов проводили в программе BioNumerics 7 (“Bionumerics”).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
В работе были проанализированы микробиомы образцов со следующими сочетаниями факторов:
ДП – дерново-подзолистая почва, контроль;
ЧЗ – чернозем, контроль;
ДП R1 – ризосфера Secale cereale (ВИР К-6469) на дерново-подзолистой почве;
ЧЗ R1 – ризосфера S. cereale (ВИР К-6469) на черноземе;
ДП R3 – ризосфера S. cereale (ВИР К-7856) на дерново-подзолистой почве;
ЧЗ R3 – ризосфера S. cereale (ВИР К-7856) на черноземе;
ДП W2 – ризосфера Triticum aestivum (ВИР К-9084) на дерново-подзолистой почве;
ЧЗ W2 – ризосфера T. aestivum (ВИР К-9084) на черноземе;
ДП W5 – ризосфера T. aestivum (ВИР К-54609) на дерново-подзолистой почве;
ЧЗ W5 – ризосфера T. aestivum (ВИР К-54609) на черноземе.
Каждый вариант рассмотрен в 4 повторностях.
Анализ нуклеотидного полиморфизма популяций
В результате секвенирования было получено 149132 нуклеотидных последовательности, из которых после проверки качества и фильтрации в дальнейшем анализе использовали 119306. Среднее количество нуклеотидных последовательностей в библиотеке на образец составляло 2508.
В результате секвенирования всех образцов была достигнута относительно небольшая глубина секвенирования (2500 последовательностей в образце). Анализ разнообразия показал, что ни одна из кривых насыщения не вышла на плато. Это ожидаемый результат, поскольку почва является крайне гетерогенным объектом (Berg et al., 2009). Микробные сообщества почвы различаются даже в пределах нескольких миллиметров, а также в различных участках почвенных агрегатов и между горизонтами (Ивлев, 2005).
Чтобы количественно оценить, какую часть сообщества удалось детектировать в эксперименте, наиболее часто используют отношение числа выявляемых ОТЕ к индексу ChaoI, который, в свою очередь, оценивает теоретически возможное количество видов в природном сообществе (генеральной совокупности) (табл. 1, 2). При анализе воспроизводимости таксономического состава микробиомов в повторностях опыта были получены сравнительно высокие показатели – больше половины таксонов в составе сообщества (69.3%) достоверно не изменяли свои частоты в повторностях. Данный показатель был в среднем ниже в повторностях черноземных почв, что свидетельствует о его зависимости от типа почвы. В дальнейшем для получения репрезентативных библиотек различных природных сообществ стоит использовать разную глубину секвенирования и количество проб. Параметры эти рекомендуется определять в ходе установочных экспериментов, однако можно рекомендовать увеличить количество повторностей в случае черноземной почвы до 7, а в случае дерново-подзолистой почвы можно ограничиться и 5‒6 повторностями.
Таблица 1.
Достоверные различия между повторностями опыта
| Вариант | Доля таксонов, обнаруженных во всех 4 повторностях, % в сообществе | Доля таксонов, достоверно не изменяющих численность в 4 повторностях, % в сообществе |
|---|---|---|
| ЧЗ | 76.3 | 69.3 |
| ЧЗ W2 | 82.7 | 46.3 |
| ЧЗ W5 | 81 | 59.3 |
| ЧЗ R1 | 81.6 | 61.4 |
| ЧЗ R3 | 82.3 | 73.7 |
| ДП | 78.0 | 87.6 |
| ДП W2 | 76.4 | 69.7 |
| ДП W5 | 78.1 | 68.0 |
| ДП R1 | 80.1 | 83.2 |
| ДП R3 | 84.1 | 78.1 |
Таблица 2.
Показатели альфа-разнообразия с учетом стандартного отклонения (между повторностями)
| Образец | ОТЕ | Индекс Шеннона | Chao-1 | Филогенетическое разнообразие Фейта (PDWholeTree) | Репрезентативность выборки |
|---|---|---|---|---|---|
| ДП | 1051.5 ± 325.6 | 5.924 ± 0.192 | 2198.5 ± 778.3 | 2.991 ± 0.406 | 47.83 |
| ЧЗ | 1519.8 ± 606.4 | 6.022 ± 0.440 | 3100.0 ± 1415.9 | 2.929 ± 0.2877 | 49.03 |
| ДП R1 | 789.5 ± 79.9 | 5.573 ± 0.172 | 1751.5 ± 290.0 | 2.938 ± 0.095 | 45.08 |
| ЧЗ R1 | 749.5 ± 197.2 | 5.336 ± 0.116 | 1854.8 ± 613.4 | 3.49 ± 0.208 | 40.41 |
| ДП R3 | 836.8 ± 145.0 | 5.145 ± 0.427 | 1919.5 ± 273.2 | 2.936 ± 0.22 | 43.59 |
| ЧЗ R3 | 734.5 ± 111.2 | 5.225 ± 0.344 | 1643.3 ± 397.0 | 3.163 ± 0.1703 | 44.70 |
| ДП W2 | 557.0 ± 95.7 | 4.613 ± 0.523 | 1327.8 ± 226.4 | 2.574 ± 0.3315 | 41.95 |
| ЧЗ W2 | 661.1 ± 56.8 | 5.381 ± 0.038 | 1313.0 ± 328.3 | 3.161 ± 0.44 | 50.35 |
| ДП W5 | 611.0 ± 151.8 | 4.707 ± 0.273 | 1425.3 ± 294.1 | 2.436 ± 0.136 | 42.87 |
| ЧЗ W5 | 501.5 ± 25.1 | 5.051 ± 0.200 | 1070.4 ± 93.9 | 3.021 ± 0.144 | 46.85 |
Также при сравнении повторностей опыта было отмечено снижение доли “воспроизводимых” таксонов в ризосфере пшеницы: доля “воспроизводимых” ОТЕ в различных вариантах опытах составила в среднем 69.3%, в то время как в сообществах ризосферы пшеницы на черноземе этот параметр составлял 52.6%. Этот феномен может быть связан с большим разнообразием микробиомов, населяющих ризосферу пшеницы, которое, в свою очередь, определяется особенностями корневой экссудации этого растения.
Важно подчеркнуть, что в данном исследовании мы приняли достаточно строгий критерий воспроизводимости данных секвенирования, при котором доля таксона не должна достоверно различаться между повторностями. Если принять за критерий воспроизводимости данных просто наличие таксона в повторности, воспроизводимость данных будет значительно выше, в среднем – 79.3% (табл. 1).
Анализ показателей альфа-разнообразия. Для оценки альфа-разнообразия сообществ были рассчитаны индекс видового богатства и индекс Шеннона (табл. 2). Ризосферные сообщества демонстрируют снижение уровня разнообразия в сравнении с контрольными почвами, хотя достоверные различия в показателях альфа-разнообразия были обнаружены только для сообществ ризосферы пшеницы на черноземной почве (ЧЗ W5; ЧЗ W2).
Такое снижение разнообразия хорошо согласуется с известными литературными данными. В исследованиях ризосферного микробиома нескольких сортов риса также были продемонстрированы различия в альфа-разнообразии (Edwards et al., 2015). Небольшое, не всегда достоверное, снижение числа ОТЕ и индекса разнообразия Шеннона в ризосфере, по сравнению с контрольными почвами, также отмечалось многими авторами (Winston et al., 2014, Edwards et al., 2015). Различия в численных показаниях снижения разнообразия, по-видимому, обусловлены влиянием различных спектров корневых экссудатов на микробное сообщество черноземной почвы.
Снижение микробного разнообразия в ризосферных сообществах, возможно, связано с селективным действием растительных факторов, т.е. растение “отбирает” определенные функциональные группы микроорганизмов, в том числе филогенетически не связанные друг с другом. К этим группам могут быть отнесены и микроорганизмы с высокой метаболической пластичностью, быстро адаптирующие клеточный метаболизм к изменившимся условиям.
Анализ показателей бета-разнообразия. Для интегральной оценки различий в составе сообществ обычно используют различные варианты многомерных статистических анализов, в частности, анализ главных компонент PCoA. На рис. 1 представлены данные PCoA почвенных контрольных и ризосферных образцов, для данных взвешенного (учитывающего численность ОТЕ) ‒ weighted (рис. 1а, 1б) и невзвешенного (учитывающего только представленность ОТЕ) ‒ unweighted (рис. 1в, 1г) Unifrac.
Рис. 1.
Анализ главных компонент (PCoA) для микробных сообществ с обозначением контрольных и ризосферных образцов различных видов культурных растений. На осях указан процент объясненной вариации. А, Б – weighted Unifrac (1 – ДП; 2 – ЧЗ; 3 – пДП, 4 – пЧЗ; 5 – рДП; 6 – рЧЗ), В, Г – unweighted Unifrac (7 – исходная почва; 8 – ризосфера пшеницы W2; 9 – ризосфера пшеницы W5; 10 – ризосфера ржи R1; 11 – ризосфера ржи R2).
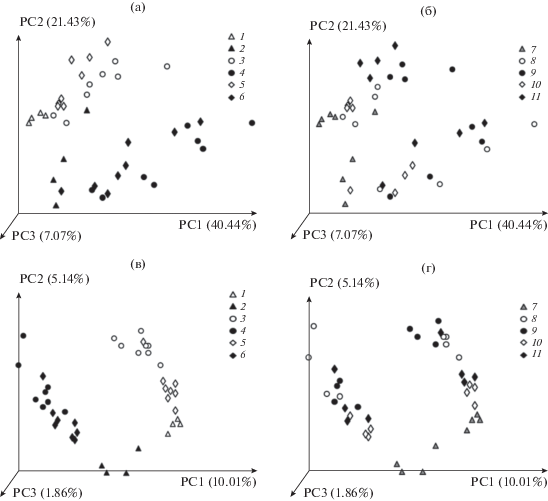
Существенные различия в составе ризосферных и почвенных сообществ подтверждаются высоким процентом объясненной вариации данных. Для PCoA анализа с использованием взвешенного Unifrac этот показатель составлял до 40% (рис. 1а, 1б).
Тип почвы оказался первым по значимости фактором, влияющим на состав микробного сообщества. С этим фактором ассоциировано более 20% различий в таксономическом составе микробиомов. Сообщества черноземной и дерново-подзолистой почвы различались по своей таксономической структуре, что выражается в присутствии кластеров на графиках PcoA (рис. 1а, 1в). Сообщества черноземной почвы проявляли некоторую гетерогенность по количественному составу микробиомов, что может быть связано с физико-химическими параметрами, создающими многообразие экологических ниш (рис. 1а, 1в). Микробные сообщества дерново-подзолистой почвы хорошо повторяют друг друга и образуют хорошо выраженный кластер (рис. 1а). Выраженное влияние типа почвы обусловлено физико-химическими различиями почвы – именно эти факторы в значительной мере определяют разнообразие прокариот.
Вторым по значимости фактором, определяющим состав ризосферного микробиома, является вид растения. Отчетливо этот фактор проявляется при сравнении сообществ дерново-подзолистой почвы и ризосфер, сформированных на ней, ‒ на графике РсоА отчетливо видны кластеры ризосфер ржи и пшеницы (рис. 1а, 1б). В случае чернозема этот эффект также присутствует, но является менее выраженным, что, возможно, связано с уже упомянутой ранее высокой гетерогенностью этой почвы.
Микробные сообщества ризосфер и пшеницы, и ржи на дерново-подзолистой почве отличаются от исходных почв, и отличия эти для обоих растений одинаково выражены. На дерново-подзолистых почвах также хорошо заметно влияние сорта растения. Так, ризосферные сообщества ржи сорта R1 оказываются ближе к исходным почвам, чем ризосферные сообщества ржи сорта R3. Аналогичный эффект можно наблюдать и для сортов пшеницы (рис. 1б). Такая разная дистанция между сообществами исходной почвы и ризосферными сообществами разных сортов в большей мере определяется количественным соотношением таксонов, поскольку по данным unweighted unifrac (учитывающим только наличие/отсутствие ОТЕ) дистанции между сообществами ДП‒ДП R1 и ДП‒ДП R2 существенно не отличаются.
Для микробиомных сообществ чернозема и ризосферных сообществ, сформированных на его основе, в целом характерны те же тенденции (рис. 1). Сообщества ризосфер ржи и пшеницы отличаются от сообществ исходных почв, дистанции между сообществами также зависят от сорта растения. Однако в рамках этого анализа однозначно разрешить эти изменения не удалось, вероятнее всего, из-за высокой агрохимической и биологической гетерогенности почвы.
Полученные результаты согласуются с опубликованными ранее работами. В ряде работ демонстрируется выраженное влияние растения на микробное сообщество уже в ризосфере (Bulgarelli et al., 2015; Edwards et al., 2015). В других работах, однако, указывается на отсутствие каких-либо изменений в составе сообществ ризосферы (по сравнению с сообществами контрольной почвы), и различия показаны только для сообществ ризопланы (Aira et al., 2010). Такое явное противоречие в оценках связано как с различной силой влияния растения на микробиом почвы, так и с уже упомянутой во введении проблемой фракционирования микробных сообществ (почва‒ризосфера‒ризоплана).
Анализ таксономической структуры сообществ. Как уже упоминалось в разделе Материалы и методы, для проведения таксономического анализа нами использовались только ОТЕ, достоверно не различающиеся между повторностями.
Преобладающим в сообществах являлся домен Bacteria, однако Archaea, представленный преимущественно филой Crenarchaeota, составлял значительную долю микробиома – 11% в случае черноземных и 5% в случае дерново-подзолистых образцов. Это соотношение практически не изменялось при рассмотрении ризосферных сообществ.
На уровне фил в почвенных и ризосферных образцах большую часть сообщества составляли представители Proteobacteria, Actinobacteria, Bacteroidetes, Verrucomicrobia, Acidobacteria, а также упомянутые выше Crenarchaeota. Такая картина в целом типична для почвенных сообществ. В литературе есть указания на присутствие большого количества Actinobacteria, Bacteroidetes, Proteobacteria даже в разных по своему происхождению и типу почвах (Lundberg et al., 2012).
При сравнении исходной почвы и ризосферных сообществ, сформированных на черноземе, наблюдалось увеличение доли филы Bacteroidetes и Proteobacteria. Этот же эффект, даже в большей степени, присутствовал и у ризосферных образцов на дерново-подзолистой почве. Доля представителей фил Planctomycetes, Acidobacteria, Chloroflexi и Actinobacteria, при сравнении обоих типов контрольных почв с соответствующими ризосферными сообществами, сокращалась. На уровне классов в переходах почва– ризосфера наблюдалось значительное увеличение числа Sphingobacteriia (Bacteroidetes) ‒ с 0.6 до 8% на черноземе и с 0.6 до 6.4% на дерново-подзолистой, а также Betaproteobacteria (Proteobacteria) ‒ с 6.8 до 19.4% на дерново-подзолистой почве и с 8.4 до 16.9% на черноземе (табл. 3).
Таблица 3.
Доли таксонов, достоверно изменяющих свою численность в попарных сравнениях ризосферных и почвенных сообществ (p < 0.05)
| Чернозем – ризосфера ржи на черноземе | ||
| Доля таксона в ЧЗ | Доля таксона в рЧЗ | |
| f__Oxalobacteraceae | 0.0058 | 0.0438 |
| f__Micrococcaceae | 0.0051 | 0.0298 |
| c__Acidobacteria-6 | 0.0194 | 0.0067 |
| Чернозем – ризосфера пшеницы на черноземе | ||
| Доля таксона в ЧЗ | Доля таксона в пЧЗ | |
| f__Oxalobacteraceae | 0.0058 | 0.0624 |
| c__Acidobacteria-6 | 0.0194 | 0.0058 |
| Дерново-подзолистая почва – ризосфера ржи на дерново-подзолистой почве | ||
| Доля таксона в ДП | Доля таксона в рДП | |
| f__Oxalobacteraceae | 0.0066 | 0.1116 |
| f__Gaiellaceae | 0.0518 | 0.0220 |
| o__Solirubrobacterales | 0.0338 | 0.0152 |
| f__Comamonadaceae | 0.0264 | 0.0113 |
| f__Micrococcaceae | 0.0086 | 0.0213 |
| Дерново-подзолистая почва – ризосфера пшеницы на дерново-подзолистой почве | ||
| Доля таксона в ДП | Доля таксона в пДП | |
| f__Oxalobacteraceae | 0.0066 | 0.0577 |
| f__Gaiellaceae | 0.0518 | 0.0303 |
| c__Acidobacteria-6 | 0.0172 | 0.0056 |
| Ризосфера ржи и ризосфера пшеницы на черноземе | ||
| Доля таксона в рЧЗ | Доля таксона в пЧЗ | |
| f__Micrococcaceae | 0.02983 | 0.01103 |
| Ризосфера ржи и ризосфера пшеницы на дерново-подзолистой почве | ||
| Доля таксона в рДП | Доля таксона в пДП | |
| f__Oxalobacteraceae | 0.1116 | 0.0577 |
| g__Rhodoplanes | 0.0224 | 0.0434 |
| f__Comamonadaceae | 0.0077 | 0.0204 |
| f__Sphingobacteriaceae | 0.0507 | 0.0287 |
| Ризосферы двух сортов ржи на черноземе | ||
| Доля таксона в рЧЗ R1 | Доля таксона в рЧЗ R3 | |
| f__Chthoniobacteraceae | 0.04756 | 0.0207 |
| Ризосферные сообщества двух сортов пшеницы на черноземе | ||
| Доля таксона в пЧЗ W2 | Доля таксона в пЧЗ W5 | |
| f__Oxalobacteraceae | 0.0741 | 0.0507 |
| Ризосферы двух сортов ржи на дерново-подзолистой почве | ||
| Доля таксона в рДП R1 | Доля таксона в рДП R3 | |
| f__Oxalobacteraceae | 0.0412 | 0.1821 |
| f__Chitinophagaceae | 0.0438 | 0.0227 |
| Ризосферы двух сортов пшеницы на дерново-подзолистой почве | ||
| Доля таксона в пДП W2 | Доля таксона в пДП W5 | |
| f__Sphingobacteriaceae | 0.0429 | 0.0144 |
Также в ризосфере ржи наблюдалось достоверное уменьшение доли бактерий семейств Ox-alobacteraceae на обоих типах почв (на дерново-подзолистой почве более чем в 12 раз и более чем в 5 раз на черноземе), Micrococcaceae (на дерново-подзолистой более чем в 2 раза, на черноземе – бо-лее чем в 4 раза), а в ризосфере пшеницы – уменьшение в сообществе доли семейства Oxalobacteraceae и достоверное увеличение доли Acidobacteria (табл. 3).
Сравнение различных сортов одного вида растения позволяют выделить несколько сортспецифических таксонов. Так между сортами ржи на черноземной почве достоверные различия долей в сообществе были выявлены в группе Chthoniobacteracea, а при аналогичном сравнении сортов пшеницы – в группе Oxalobacteraceae и Sphingobacteriaceae. Стоит отметить, что Sphingobacteriales также упоминается в литературе как сортспецифичный таксон для различных сортов конопли (Winston et al., 2014).
В ряде исследований отмечается специфичность ризосферных таксонов. Так, уже упоминавшиеся исследования ризосферных сообществ ячменя указывают на увеличение доли Commamonadaceaе (Bulgerelli et al., 2015), а ризосфер конопли ‒ на увеличение доли Sphingomonadales (Winston et al., 2014).
Видоспецифичность некоторых ризосферных таксонов также отражена в литературе (Edwards et al., 2015; Coleman-Derr et al., 2016), однако ни в одном из проанализированных литературных источников не было обнаружено тех таксонов, которые изменяли свою долю в проанализированных микробиомах ризосфер ржи и пшеницы.
Характеристика ризосферного эффекта с использованием данных масс-спектрометрии и ионного состава
Массив данных, полученных при анализе ионного и масс-спектрального состава, очень велик и требует детального рассмотрения. В рамках данной работы мы охарактеризуем сходство или различие ионных и масс-спектральных профилей.
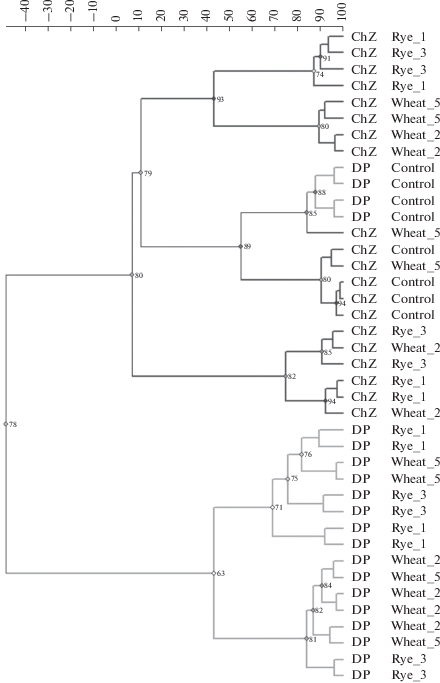
В графике, построенном по результатам кластерного анализа профилей масс-спектров (рис. 2), несмотря на отсутствие четкой кластеризации, можно выделить отдельную кладу, принадлежащую масс-спектрам исходных почвенных образцов. Также хорошо обособленной является клада, принадлежащая масс-спектрам ризосфер ржи. По-видимому, состав масс-спектров, в большей степени зависит от вида растения, а не от типа почвы. Это может быть связано с присутствием большого количества органических соединений ‒ продуктов корневой экссудации.
Рис. 2.
Дендрограмма, построенная по данным ICPE (Curtiss, алгоритм кластеризации Ward). Темными линиями отмечены образцы, полученные из чернозема, светлыми – из дерново-подзолистой почвы.
Кластерный анализ ионного состава (рис. 3) показывает несколько иную картину. Дендрограмма, построенная по данным ионного состава, характеризуется высокими значениями поддержки кластеров, а сам состав кластеров демонстрирует выраженную зависимость ионного состава почвенной вытяжки от типа почвы. Образцы ризосфер дерново-подзолистых почв группируются в отдельный кластер с высоким значением поддержки. Второй кластер включает в себя ризосферы, сформированные на черноземе, и обе исходных почвы (чернозем и дерново-подзолистая).
Рис. 3.
Дендрограмма, построенная по данным масс-спектрометрии (Jaccard, алгоритм кластеризации Ward). Темными линиями отмечены образцы, полученные из ризосфер ржи, светлыми – из ризосфер пшеницы. Пунктирные линии отмечают образцы, взятые из контрольных почв.
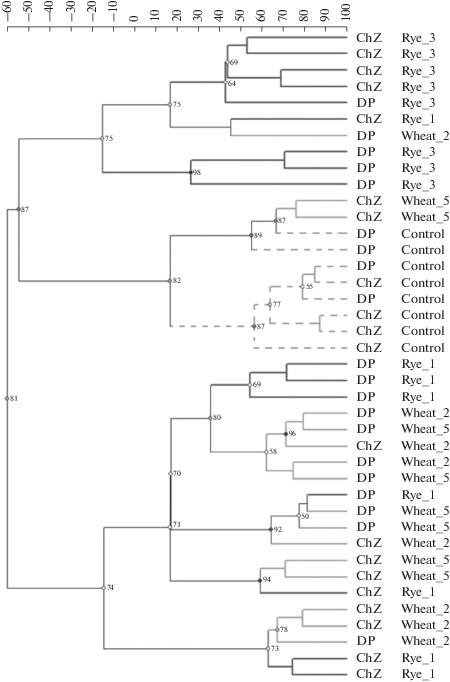
Таким образом, в качестве дальнейшей гипотезы можно считать, что именно тип почвы определяет ионный состав ризосферы. В целом химический состав (определяемый масс-спектрально) ризосферной почвы зависит от большего числа факторов, и одним из оказывающих существенное влияние является вид растения.
Исследование стало первой попыткой анализа ризосферных сообществ ржи и пшеницы на контрастных почвах с использованием системного подхода к анализу биоразнообразия. В ходе работы была применена комплексная методика отбора ризосферных образцов, а также дополнены методы статистической поддержки метагеномного анализа сообщества, позволяющие рассчитать требуемое количество повторностей эксперимента.
В ходе исследования было показано, что именно почвенное микробное сообщество является основой для формирования растением ризосферного микробиома. Также были выявлены таксоны, достоверно принимающие участие в формировании специфического ризосферного микробиома ржи и пшеницы. Были выявлены как общие ризосферные таксоны (Oxalobacteriaceae), так и видо- (Micrococcaceaе, Sphingobacteriaceae и др.) и сортоспецифичные. Изменение состава микробиома отчетливее проявляется на бедных почвах. Можно предположить, что растение “отбирает” из почвенного сообщества те или иные бактериальные таксоны, основываясь не на их таксономическом положении, но на функциональной роли в новой, формирующейся экологической системе.
Данное направление исследования, безусловно, является существенным как для разработки систем адаптивного земледелия, так и для расширения фундаментальных знаний о формировании и особенностях экологии ризосферых сообществ.
Список литературы
Андронов Е.Е., Пинаев А.Г., Першина Е.В., Чижевская Е.П. Научно-методические рекомендации по выделению высокоочищенных препаратов ДНК из объектов окружающей среды. Методические указания. С-Пб.: Российская академия сельскохозяйственных наук, Всероссийский научно-исследовательский институт сельскохозяйственной микробиологии, 2011. 23 с.
Ивлев А.М. Эволюция почв. Владивосток: Изд. Дальневосточного ун-та, 2005. 97 с.
Aira M., Gómez-Brandón M., Lazcano C., Baath E., Domínguez J. Plant genotype strongly modifies the structure and growth of maize rhizosphere microbial communities // Soil Biol. Biochem. 2010. V. 42. P. 2276–2281.
Bates S.T., Berg-Lyons D., Caporaso J.G., Walters W.A., Knight R., Fierer N. Examining the global distribution of dominant archaeal populations in soil // ISME J. 2011. V. 5. P. 908‒917.
Berg G., Smalla K. Plant species and soil type cooperatively shape the structure and function of microbial communities in the rhizosphere // FEMS Microbiol. Ecol. 2009. V. 68. P. 1–13.
Bulgarelli D., Garrido-Oter R., Munch P.C., Weiman A., Dröge J., Pan Y., McHardy A.C., Schulze-Lefert P. Structure and function of the bacterial root microbiota in wild and domesticated barley // Cell Host Microbe. 2015. V. 17. P. 392‒403. https://doi.org/10.1016/j.chom.2015.01.011
Caporaso J.G., Kuczynski J., Stombaugh J., Bittinger K., Bushman F.D., Costello E.K., Fierer N., Peña A.G., Goodrich J.K., Gordon J.I., Huttley G.A., Kelley S.T., Knights D., Koenig J.E., Ley R.E., Lozupone C.A., McDonald D., Muegge B.D., Pirrung M., Reeder J., Sevinsky J.R., Turnbaugh P.J., Walters W.A., Widmann J., Yatsunenko T., Zaneveld J., Knight R. QIIME allows analysis of high-throughput community sequencing data // Nat. Meth. 2010. V. 7. P. 335‒336. https://doi.org/10.1038/nmeth.f.303
Chaparro J.M., Badri D.V., Bakker M.G., Sugiyama A., Manter D.K., Vivanco J.M. Root exudation of phytochemicals in Arabidopsis follows specific patterns that are developmentally programmed and correlate with soil microbial functions // PLoS One. 2013. V. 8: e55731.
Coleman-Derr D., Desgarennes D., Fonseca-Garcia C., Gross S., Clingenpeel S., Woyke T., North G., Visel A., Partida-Martinez L.P., Tringe S.G. Plant compartment and biogeography affect microbiome composition in cultivated and native Agave species // New Phytol. 2016. V. 209. P. 798‒811. https://doi.org/10.1111/nph.13697
De Santis T.Z., Jr., Hugenholtz P., Keller K., Brodie E.L., Larsen N., Piceno Y.M., Phan R., Andersen G.L. NAST: a multiple sequence alignment server for comparative analysis of 16S rRNA genes // Nucleic Acids Res. 2006. V. 34. (Web Server issue) P. W394‒W399.
Donn S., Kirkegaard J.A., Perera G., Richardson A.E., Watt M. Evolution of bacterial communities in the wheat crop rhizosphere // Environ. Microbiol. 2015. V. 17. P. 610‒621.
Edwards J., Johnson C., Santos-Medellín C., Lurie E., Podishetty N.K., Bhatnagar S., Eisen J.A., Sundaresan V. Structure, variation, and assembly of the root-associated microbiomes of rice // Proc. Natl. Acad. Sci. USA. 2015. V. 112. P. E911‒E920. https://doi.org/10.1073/pnas.1414592112
Girvan M.S., Bullimore J, Pretty J.N., Osborn A.M., Ball A.S. Soil type is the primary determinant of the composition of the total and active bacterial communities in arable soils // Appl. Environ. Microbiol. 2003. V. 69. P. 1800–1809. https://doi.org/10.1111/1462-2920.12452
Gottel N.R., Castro H.F., Kerley M., Yang Z., Pelletier D.A., Podar M., Karpinets T., Uberbacher E., Tuskan G.A., Vilgalys R., Doktycz M.J., Schadt C.W. Distinct microbial communities within the endosphere and rhizosphere of Populus deltoides roots across contrasting soil types // Appl. Environ. Microbiol. 2011. V. 77. P. 5934‒5944. https://doi.org/10.1128/AEM.05255-11
Hammer O., Harper D., Ryan P. PAST: Paleontological Statistics software package for education and data analysis // Paleontologia Electronica. 2001. V. 4. Article 4. 9 p.
Handelsman J. Metagenomics: application of genomics to uncultured microorganisms // Microbiol. Mol. Biol. Rev. 2004.V. 68. P. 669–685.
Knief C., Delmotte N., Chaffron S., Stark M., Innerebner G., Wassmann R., von Mering C., Vorholt J.A. Metaproteogenomic analysis of microbial communities in the phyllosphere and rhizosphere of rice // ISME J. 2012. V. 6. P. 1378‒1390. https://doi.org/10.1038/ismej.2011.192
Lozupone C.A., Hamady M., Kelley S.T., Knight R. Quantitative and qualitative beta diversity measures lead to different insights into factors that structure microbial communities // Appl. Environ. Microbiol. Mar. 2007. V. 73. P. 1576‒1585.
Lundberg D.S., Lebeis S.L., Paredes S.H., Yourstone S., Gehring J., Malfatti S., Tremblay J., Engelbrektson A., Kunin V., del Rio T.G., Edgar R.C., Eickhorst T., Ley R.E., Hugenholtz P., Tringe S.G., Dangl J.L. Defining the core Arabidopsis thaliana root microbiome // Nature. 2012. V. 488. P. 86‒90.
Mendes L.W., Kuramae E.E., Navarrete A.A., van Veen J.A., Tsai S.M. Taxonomical and functional microbial community selection in soybean rhizosphere // ISME J. 2014. V. 8. P. 1577‒1587. https://doi.org/10.1038/ismej.2014.17
Peiffer J.A., Spor A., Koren O., Jin Z., Tringe S.G., Dangl J.L., Buckler E.S., Ley R.E. Diversity and heritability of the maize rhizosphere microbiome under field conditions // Proc. Natl. Acad. Sci. USA. 2013. V. 110. P. 6548‒6553.
Winston M.E., Hampton-Marcell J., Zarraonaindia I., Owens S.M., Moreau C.S., Gilbert J.A., Hartsel J.A., Kennedy S.J., Gibbons S.M. Understanding cultivar-specificity and soil determinants of the cannabis microbiome // PLoS One. 2014. V. 9. e99641.
Дополнительные материалы отсутствуют.


