Микробиология, 2020, T. 89, № 2, стр. 252-256
Простой метод выделения ДНК вирома фекалий лошади, пригодный для секвенирования по технологии Oxford Nanopore
Е. Е. Куликов a, b, *, А. К. Голомидова a, В. В. Бабенко c, А. В. Летаров a, d
a Институт микробиологии им. С.Н. Виноградского, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” РАН
119071 Москва, Россия
b Физтех-школа молекулярной и биологической физики,
НИУ Московский физико-технический институт
115184 Москва, Россия
c ФГБУ ФНКЦ физико-химической медицины ФБМА РФ
119435 Москва, Россия
d Биологический факультет, Московский государственный университет
им. М.В. Ломоносова
119192 Москва, Россия
* E-mail: eumenius@gmail.com
Поступила в редакцию 26.09.2019
После доработки 09.10.2019
Принята к публикации 29.11.2019
Аннотация
Исследования по метагеномике виромов, ассоциированных с симбиотическими микробными сообществами, представляют собой быстро развивающуюся область. Большая часть таких работ направлена на изучение вирусных сообществ фекалий человека или животных. Новая технология секвенирования компании Oxford Nanopore с получением длинных прочтений, которая стала доступна в последние годы, создает принципиально новые возможности при исследовании некультивируемых вирусных сообществ. Однако, по сравнению с традиционными методами NGS секвенирования, эта технология значительно более требовательна к количеству и качеству ДНК. Мы разработали простой, дешевый и эффективный протокол для выделения ДНК вирусного сообщества фекалий лошади, пригодной для секвенирования по данной технологии. Методика может быть адаптирована и для работы с другими объектами.
Микробные сообщества с высокой плотностью жизни весьма часто встречаются в природе. Такие сообщества могут быть как свободноживущими, так и симбиотическими, как, например, кишечные микробиомы животных. Ассоциированные с микробными экосистемами виромы (сообщества вирусов, преимущественно бактериофагов), оказывают многообразное влияние на структуру, адаптацию и другие аспекты активности бактерий в сообществе (Shkoporov, Hill, 2019). В настоящее время из всего многообразия природных высокоплотных экосистем обширные данные о составе, динамике и биогеографии виромов получены только для кишечного сообщества человека. Однако для расшифровки экологических “правил игры” вирусов и их хозяев в подобных сообществах и механизмов, контролирующих эти правила, необходимо подробное изучение некоторого числа альтернативных, существенно отличающихся симбиотических экосистем. Современные возможности обработки больших данных и глубокого обучения способны обеспечить выполнимость такого анализа, однако для его проведения необходима разработка комплексной методологии, включающей экспериментальные микробиологические и вирусологические методы, а также сравнительную геномику вирусных изолятов.
Длительная персистенция определенных групп колифагов, которая наблюдалась одновременно у нескольких животных, содержавшихся в одном и том же хозяйстве (Kulikov et al., 2012; Babenko et al., 2019), при отсутствии этих фагов в других популяциях косвенно свидетельствует о том, что состав вирома кишечника взрослых лошадей подвержен влиянию за счет продолжающегося переноса вирусов и бактерий между индивидуумами; последнее было предположено в результате исследований микробиоты кишечника лошадей (Costa et al., 2015; Julliand, Grimm, 2016). Это свойство отличает виром лошади от человеческого, ландшафт которого, видимо, отражает чрезвычайно индивидуальную историю заселения кишечника в раннем возрасте и в значительной мере сохраняется в течение последующей жизни (Shkoporov, Hill, 2019). Эти особенности экологии бактериофагов в кишечнике приводят к предположению, что локальные популяции лошадей могут содержать общие пулы ассоциированных фагов – локальные панвиромы, которые по своему составу могут значительно отличаться от панвиромов других популяций.
Для экспериментальной проверки данной гипотезы необходимо иметь возможность быстрого выделения вирусного компонента из фекалий лошади. Выделяемые препараты метавиромов должны отвечать ряду требований, а именно: быть свободными от геномной ДНК бактерий и лошади и позволять выделение высокоочищенных препаратов ДНК метавирома, которая может быть секвенирована, в том числе, наиболее чувствительными к качеству ДНК методами, такими как технология мономолекулярного секвенирования Oxford Nanopore (Jain et al., 2016). Предложенная нами методика выделения суммарных препаратов метавиромов фекалий лошади соответствует этим критериям.
Методика основана на свойстве полиэтиленгликоля осаждать фаговые частицы из растворов с высокой ионной силой (Vajda, 1978). Несмотря на ряд ограничений, указанных в исходной статье, метод после оптимизации и валидации активно применяется сегодня для выделения бактериофагов (Milani et al., 2018), а также метавиромов из фекалий человека для метагеномных исследований (Thurber et al., 2009; Castro-Mejia et al., 2015). Однако в указанных работах после осаждения вирусов ПЭГ использовали очистку центрифугированием в градиенте хлористого цезия. Такая методика хотя и приводит к получению препаратов очень высокой чистоты, сопряжена с большими потерями вирусов и искажением состава сообщества. Часть бактериофагов разрушается и агрегирует при высоких концентрациях CsCl. Кроме того, процедура диализа также сопряжена с существенными потерями материала.
Важной и затрудняющей работу особенностью фекалий лошади оказывается высокое содержание в них нерастворимой фракции (целлюлозные волокна, непереваренные остатки пищи и пр.), в том числе низкоплотной, легко флотирующей при центрифугировании. Эта особенность заставляет активно применять методы фильтрования вместо центрифугирования и вызывает большие трудности из-за ограниченной емкости существующих мелкопористых фильтрующих материалов (мембранных фильтров и пр.). Помимо этого, при работе с фекалиями лошади возникало дополнительное затруднение. После первичного осветления экстракта биоматериала низкоскоростным центрифугированием в супернатанте быстро формируется преципитат. По-видимому, этот преципитат образуется из-за окисления присутствующих в фекалиях растворимых органических соединений. Наличие преципитата делало практически невозможным использование мембранных фильтров для удаления бактериального и иного мелкодисперсного дебриса, так как эти фильтры быстро забиваются. Для преодоления этого затруднения мы использовали фильтрацию через многослойный фильтр.
После оптимизации методики мы остановились на следующем протоколе.
Навеску (около 20 г сырого веса) свежеотобранных фекалий лошади помещали в коническую колбу со 100 мл экстракционного раствора (0.2 M NaCl, 0.1 г/л NaN3, 1 г/л Tween 20; “Sigma-Aldrich”, США) и экстрагировали растворимую фракцию фекалий на орбитальном шейкере при комнатной температуре в течение 2 ч (100 об./мин). Полученный грубый экстракт фильтровали через воронку Бюхнера под вакуумом через 4 слоя вискозно-полиэстерного нетканого материала (“Чудо-тряпка”; “Комус”, Россия), к фильтрату добавляли панкреатическую ДНКазу до 10 мкг/мл и инкубировали 15 мин при комнатной температуре. Фильтрат центрифугировали в течение 15 мин при 10 000 g в роторе JA-14 (“Beckman”, СШA) и аккуратно отделяли супернатант от осадка. Далее в фильтровальной колбе с составным фильтром из пористого стекла (“Millipore”, СШA) готовили динамический многослойный одноразовый фильтрующий элемент, состоящий из слоя стеклянной фильтровальной бумаги (GF-C, “Whatman”, СШA), слоя толстой фильтровальной бумаги Whatman 3MM (“Whatman”, СШA) и слоя диатомита (Celite Hyflo Supercel, “Sigma-Aldrich”, СШA). Слой диатомита наносили путем фильтрации под вакуумом 10% суспензии диатомита в дистиллированной воде так, чтобы его слой был в итоге высотой 2–3 мм. На подготовленный фильтр наносили супернатант и фильтровали его под вакуумом. Полученный прозрачный фильтрат доводили до 0.6 М концентрации по NaCl добавлением сухой соли, к раствору добавляли сухой полиэтиленгликоль (Mr = 6000, “Panreac”, ЕС) и растворяли его аккуратным перемешиванием на орбитальном шейкере (45 об./мин). После полного растворения полиэтиленгликоля препараты выдерживали неделю при 4°С до образования коричнево-зеленоватого осадка. Осадок отделяли центрифугированием (15 мин при 10 000 g в роторе JA-14; “Beckman”, СШA) и ресуспендировали в 5 мл физиологического раствора. Полученный осадок содержал преимущественно вирусную фракцию фекалий.
Выделение ДНК из аликвоты осадка проводили методом лизиса катионными детергентами (цетилтриметиламмония бромид, CTAB), описанным в работе (Thomas et al., 1997) и хорошо зарекомендовавшим себя для выделения ДНК из сложных образцов, содержащих ингибирующие ДНК-полимеразы примеси (кровь, фекалии и пр.) (Tang et al., 2008). Полученная геномная ДНК характеризовалась высокой молекулярной массой (по данным гель-электрофореза в агарозных гелях) и отсутствием видимых примесей низкомолекулярных нуклеиновых кислот (рис. 1).
Рис. 1.
Гель-электрофорез образцов ДНК метавиромов кишечного сообщества лошади. Дорожки 1–4 – независимо выделенные препараты вирусных ДНК; дорожка 5 – маркер длин фрагментов ДНК (1 kb DNA Ladder, “Сибэнзим”, Россия).
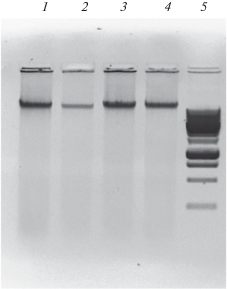
Выход ДНК был достаточно большим, что позволяло получить до 1.5 мкг ДНК из одного образца, чего достаточно для любых современных методов высокопроизводительного секвенирования (NGS).
Качество полученной ДНК оказалось достаточно высоким, чтобы ее можно было секвенировать не только методами NGS, включающими предварительную амплификацию (в нашей работе мы использовали платформу Ion Proton (“Thermo Fischer Scientific”, США)), но и прямым методом секвенирования длинных ридов без амплификации (секвенатор MinION производства “Oxford Nanopore”, США). Спектральные отношения оптической плотности полученных препаратов 260/280 нм и 260/230 нм составляли 1.85 и 1.1 соответственно. В обоих случаях секвенирование осуществляли по стандартным протоколам, рекомендованным производителями. При дополнительной процедуре очистки образца от короткой фракции фрагментов ДНК (100–3000 п.н.) с помощью протокола (SPRI size selection protocol) отношение поглощения на длинах волн 260 и 230 нм (A260/230) приближаются к рекомендованным значениям (1.6–1.8) пригодных для приготовления библиотек длинных ридов. Для проведения секвенирования длинных ридов использовался набор реактивов SQK-LSK109 и ячейка FLO-MIN106 (“Oxford Nanopore”, США).
Распределение длин и качества прочтений при секвенировании прибором MinION (рис. 2, 3) свидетельствуют о полной пригодности полученной ДНК для секвенирования данным методом. Предварительный анализ данных указал на низкий уровень контаминации ДНК вирома генетическим материалом бактерий и макроорганизма (не более 1%).
Рис. 2.
Распределение длин прочтений ДНК метавирома, выделенных из фекалий лошади по описанной методике и секвенированных без амплификации на приборе MinION Oxford Nanopore. На гистограмме указано значение N50 12.83 т.п.н., максимальная длина рида больше 180 т.п.н.
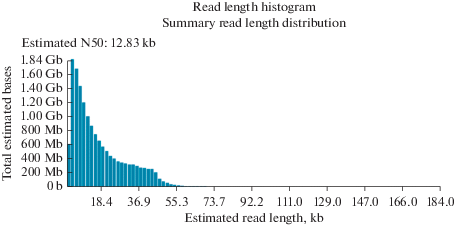
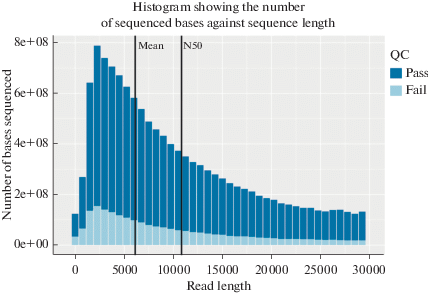
Рис. 3.
Гистограмма отображает распределение длин прочтений ДНК (бин-шаг 720 п.н.) и количество пар оснований, входящих в каждую бин-группу. QC – автоматический контроль качества прочтения, синим цветом указаны риды, прошедшие контроль качества (среднее значение 9.9), голубым цветом указаны риды низкого качества (среднее значение 5.2). На графике вертикальной чертой указаны (Mean) средняя длина прочтения (6117 п.н.) и N50 (10 841 п.н.). Данные параметры рассчитаны только для ридов, прошедших фильтр качественной оценки.
Минимальное количество шагов, низкая себестоимость, высокая воспроизводимость, возможность прямого секвенирования выделяемой ДНК и отсутствие потенциально травмирующих вирусы процедур (центрифугирование в градиенте плотности хлорида цезия, диализ) позволяет считать разработанный нами протокол выделения метавиромов адекватным для решения широкого круга задач, связанных с определением динамики вирусной компоненты симбиотической микрофлоры животных.
Список литературы
Babenko V.V., Golomidova A.K., Ivanov P.A., Letarova M.A., Kulikov E.E., Manolov A.I., Prokhorov N.S., Kostrukova E.S., Matyushkina D.M., Prilipov A.G., Maslov S., Belalov I.S., Clokie M.R.J.C., Letarov A.V. Phages associated with horses provide new insights into the dominance of lateral gene transfer in virulent bacteriophages evolution in natural systems // Biorxiv. 2019. https://doi.org/10.1101/542787
Castro-Mejia J.L., Muhammed M.K., Kot W., Neve H., Franz C.M., Hansen L.H., Vogensen F.K., Nielsen D.S. Optimizing protocols for extraction of bacteriophages prior to metagenomic analyses of phage communities in the human gut // Microbiome. 2015. V. 3. Article 64. https://doi.org/10.1186/s40168-015-0131-4
Costa M.C., Silva G., Ramos R.V., Staempfli H.R., Arroyo L.G., Kim P., Weese J.S. Characterization and comparison of the bacterial microbiota in different gastrointestinal tract compartments in horses // Vet. J. 2015. V. 205. P. 74–80.
Jain M., Olsen H.E., Paten B., Akeson M. The Oxford Nanopore MinION: delivery of nanopore sequencing to the genomics community // Genome Biol. 2016. V. 17. Article 239.
Julliand V., Grimm P. Horse Species Symposium: The microbiome of the horse hindgut: History and current knowledge1 // J. Animal Sci. 2016. V. 94. P. 2262–2274.
Kulikov E., Kropinski A.M., Golomidova A., Lingohr E., Govorun V., Serebryakova M., Prokhorov N., Letarova M., Manykin A., Strotskaya A., Letarov A. Isolation and characterization of a novel indigenous intestinal N4-related coliphage vB_EcoP_G7C // Virology. 2012. V. 426. P. 93–99.
Milani C., Casey E., Lugli G.A., Moore R., Kaczorowska J., Feehily C., Mangifesta M., Mancabelli L., Duranti S., Turroni F., Bottacini F., Mahony J., Cotter P.D., McAuliffe F.M., van Sinderen D., Ventura M. Tracing mother-infant transmission of bacteriophages by means of a novel analytical tool for shotgun metagenomic datasets: METAnnotatorX // Microbiome. 2018. V. 6. Article 145. https://doi.org/10.1186/s40168-018-0527-z
Shkoporov A.N., Hill C. Bacteriophages of the Human Gut: The “Known Unknown” of the Microbiome // Cell Host Microbe. 2019. V. 25. P. 195–209.
Tang J.N., Zeng Z.G., Wang H.N., Yang T., Zhang P.J., Li Y.L., Zhang A.Y., Fan W.Q., Zhang Y., Yang X., Zhao S.J., Tian G.B., Zou L.K. An effective method for isolation of DNA from pig faeces and comparison of five different methods // J. Microbiol. Methods. 2008. V. 75. P. 432–436.
Thomas J.C., Khoury R., Neeley C.K., Akroush A.M., Davies E.C. A fast CTAB method of human DNA isolation for polymerase chain reaction applications // Biochem. Educat. 1997. V. 25. P. 233–235.
Thurber R.V., Haynes M., Breitbart M., Wegley L., Rohwer F. Laboratory procedures to generate viral metagenomes // Nat. Protoc. 2009. V. 4. P. 470–483.
Vajda B.P. Concentration and purification of viruses and bacteriophages with polyethylene glycol // Folia Microbiol. 1978. V. 23. P. 88–96.
Дополнительные материалы отсутствуют.
Инструменты
Микробиология


