Микробиология, 2020, T. 89, № 3, стр. 324-333
Анализ полной последовательности генома нового представителя рода Methylococcus, штамма Concept-8
И. Ю. Ошкин a, В. Н. Хмеленина b, С. Ю. Бут b, К. К. Мирошников a, С. Э. Белова a, Н. С. Хохлачев c, Д. В. Чернушкин d, А. В. Белецкий e, А. В. Марданов e, Н. В. Равин e, В. О. Попов f, С. Н. Дедыш a, *, Н. В. Пименов a
a Институт микробиологии им. С.Н. Виноградского, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” РАН
119071 Москва, Россия
b Институт биохимии и физиологии микроорганизмов им. Г.К. Скрябина,
Пущинский научный центр биологических исследований РАН
142290 Пущино, Россия
c ООО “Газпром ВНИИГАЗ”
142717 Московская обл., п. Развилка, Россия
d ООО “НПО Биосинтез”
107045 Москва, Россия
e Институт биоинженерии, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” РАН
119071 Москва, Россия
f Институт биохимии им. А.Н. Баха, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” РАН
119071 Москва, Россия
* E-mail: dedysh@mail.ru
Поступила в редакцию 20.12.2019
После доработки 25.12.2019
Принята к публикации 26.12.2019
Аннотация
Определена и проанализирована полная последовательность генома облигатного термотолерантного метанотрофа Methylococcus sp. Concept-8, который является производной долгосрочного хранения, селекции и очистки промышленной культуры Methylococcus capsulatus БСВ-874, использованной в СССР в качестве штамма-продуцента для производства белка на основе метана. Размер генома штамма Concept-8 составил 3.46 млн пар нуклеотидов. В ходе его аннотации идентифицированы 3266 открытые рамки считывания, кодирующие белки. Ближайшим филогенетическим родственником штамма Concept-8 является Methylococcus capsulatus Bath (98.7% сходства последовательностей генов 16S рРНК). Сравнение геномов этих метанотрофов выявило 88.39% средней идентичности нуклеотидных последовательностей, что свидетельствует о принадлежности штамма Concept-8 к новому виду рода Methylococcus. Геномы штаммов Bath и Concept-8 содержат по две копии оперонов рРНК и мембранной метанмонооксигеназы pmoBAC и по одной копии оперона растворимой метанмонооксигеназы mmoXYBZDC. Рост на метаноле обеспечивается присутствием двух комплементарных метанолдегидрогеназ – MxaFJGIRACKLD и XoxF. Набор генов, кодирующих ферменты основных путей метаболизма С1-соединений у этих метанотрофов также высоко сходен. В геноме Methylococcus sp. Сoncept-8 обнаружено 2 региона, ассоциированных с профагами семейства Siphoviridae. Найденные в геноме Mc. capsulatus Bath профаговые регионы также гомологичны последовательностям вирусов семейства Siphoviridae, однако отличаются от выявленных в геноме штамма Сoncept-8.
Метанотрофные бактерии – это метаболически уникальная группа прокариотных микроорганизмов, способных использовать метан в качестве единственного источника углерода и энергии (Hanson, Hanson, 1996; Гальченко, 2001; Trotsenko, Murrell, 2008; Khmelenina et al., 2018). Эта метаболическаяособенность метанотрофов лежит в основе их применения в качестве продуцентов белка на основе метана (Григорян, Горская, 1970; Linton, Buckee, 1977; Hamer, Harrison, 1980; Лалов, 1991). Масштабное промышленное производство кормового белка (Гаприна) из природного газа с использованием термотолерантного метанотрофа Methylococcus capsulatus БСВ-874 было реализовано в СССР в середине 1980-х годов. Данные о последовательности генома этого метанотрофа получены не были, однако они доступны для другого штамма этого вида – Mc. capsulatus Bath, который с момента его описания в 1966 году (Foster, Davis, 1966) широко используется в качестве модельного микроорганизма для исследования метанотрофии. Геномная последовательность штамма Bath была опубликована в 2004 году и явилась первым геномом, определенным для метанотрофных бактерий (Ward et al., 2004). Многочисленные фундаментальные исследования метаболических процессов окисления метана Mc. capsulatus Bath, в том числе с использованием протеомики (Kao et al., 2004), транскриптомики (Larsen, Karlsen, 2016) и метаболического моделирования (Lieven et al., 2018), сделали его одним из наиболее детально изученных метанотрофов. Mc. capsulatus Bath используется в качестве штамма-продуцента в современных технологиях производства кормового белка из метана, реализованных в Дании и Норвегии.
Штамм Concept-8 является производной длительного хранения и селекции промышленной культуры Mc. capsulatus БСВ-874, поддерживаемой на протяжении последних десятилетий в ООО “Газпром ВНИИГАЗ”. Культура демонстрирует высокие скорости роста и высокий выход биомассы на метане, что позволяет рассматривать ее в качестве перспективного штамма-продуцента. В современной биотехнологии анализ последовательности генома является необходимым условием обоснованного выбора штамма-продуцента, так как позволяет получить информацию о метаболическом потенциале целевого организма и возможностях его оптимизации.
Настоящее исследование ставило целью определение полной последовательности генома штамма Concept-8 и сравнение ее с таковой у Mc. capsulatus Bath для выявления возможных отличий метаболической организации этих метанотрофов и установления точного филогенетического положения Concept-8.
ОБЪЕКТЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Источник культуры и ее очистка от ассоциированных микроорганизмов. Исходная культура штамма Concept-8, содержащая около 10% клеток сопутствующих гетеротрофных микроорганизмов, была предоставлена специалистами ООО “Газпром ВНИИГАЗ”, где она поддерживалась путем периодических пересевов на среде, использованной ранее для целей промышленного культивирования этого метанотрофа. Состав среды включал следующие компоненты (г л–1): NaNO3, 1; MgSO4 · · 7H2O, 0.2; CaCl2 · 2H2O, 0.02; Na2HPO4 · 5H2O, 1.5; KH2PO4, 0.7 и 1 мл раствора микроэлементов (Гальченко, 2001). Очистка метанотрофа от сопутствующих микроорганизмов была достигнута с помощью чередования серий предельных разведений культуры в жидкой среде NMS 1 : 5 и рассева суспензии клеток на агаризованную среду NMS 1 : 5 для получения колоний. Среда NMS 1 : 5 включала (г л−1): KNO3, 0.5; MgSO4 · 7H2O, 0.4; CaCl2 · 2H2O, 0.1; 20 мл 0.1 М фосфатного буфера (рН 7.0) и 1 мл раствора микроэлементов (Гальченко, 2001). Для приготовления предельных разведений использовали серии флаконов объемом 60 мл, содержащие 10 мл среды и 30% метана в газовой фазе. Флаконы инкубировали на качалке (100 об./мин) при 42°С. Суспензию культур из флаконов с наибольшими разведениями рассевали на агаризованную среду NMS 1 : 5, чашки помещали в эксикаторы с 30% метана в газовой фазе и инкубировали в течение 2-х недель при 37°С. Формирующиеся колонии отсевали во флаконы с жидкой средой, полученные культуры проверяли на чистоту с помощью фазово-контрастной микроскопии и вновь рассевали на агаризованную среду до получения чистой культуры.
Получение биомассы для геномного секвенирования. Полученную чистую культуру штамма Concept-8 выращивали во флаконах объемом 500 мл, содержащих 100 мл среды NMS 1 : 5 и 30% метана в газовой фазе. После 2-суточной инкубации на качалке (100 об./мин) при 42°С клетки осаждали центрифугированием и использовали для выделения ДНК.
Выделение ДНК и секвенирование генома. Препарат геномной ДНК выделяли с помощью набора DNeasy Power Soil Kit (“Qiagen”). Всего было выделено около 5 мкг ДНК. Последовательность генома штамма Concept-8 была получена с помощью комбинированной стратегии, включающей использование двух платформ высокопроизводительного секвенирования – Illumina (MiSeq) и мономолекулярного секвенирования на MinIon (“Oxford Nanopore”). Для приготовления геномной библиотеки, пригодной для секвенирования на приборе MiSeq (“Illumina”), использовали набор NEBNext ultra II DNA Library kit (“New England Biolabs”). Всего было получено 1.1 млн парных прочтений (2 × 300 нт), суммарная длина которых составила 662 млн нт. Для секвенирования на приборе MinIon (“Oxford Nanopore”) геномную библиотеку готовили с помощью набора 1D Ligation Sequencing Kit SQK-LSK108 и секвенировали с использованием ячейки FLO-MIN106 v R9.4.1. Было получено 39 915 чтений со средней длиной 14 686 нт. Сборку генома проводили с использованием всех полученных прочтений с помощью Unicycler (Wick et al., 2017).
Аннотация и анализ генома. Поиск и аннотацию генов проводили с использованием сервера RAST v. 2.0 (“Rapid Annotation using Subsystem Technology”) (Overbeek et al., 2014), Prokka (Seemann, 2014) и GhostKOALA (Kanehisa et al., 2016), с последующей проверкой и коррекцией аннотации вручную в результате сравнения предсказанных генов с базами данных NCBI (National Center for Biotechnology Information) (Altschul et al., 1997), Uniprot (https://www.uniprot.org/) и KEGG (Kyoto Encyclopedia of Genes and Genomes) (Kanehisa et al., 2014). Гены транспортных и рибосомальных РНК определяли с использованием tRNAScan-SE (Lowe, Eddy, 1996) и RNAmmer (Lagesen et al., 2007) соответственно. Поиск инсерционных последовательностей выполняли на ISsaga server (Varani et al., 2011).
Сравнительный геномный и филогенетические анализы. Последовательность генома Mc. capsulatus Bath была получена из базы данных GenBank (номер доступа AE017282.2). Показатели общего сходства геномов штамма Concept-8 и Mc. capsulatus Bath рассчитывали с использованием ANI calculator (Rodriguez-R, Konstantinidis, 2014) и Genome-to-Genome-Distance-Calculator (Meier-Kolthoff et al., 2013). Филогенетическую дендрограмму на основе сравнения геномов строили с использованием платформы Genome Taxonomy Database (GTDB) и программы GTDB-Tk, версия 04-RS89 (https:// github.com/Ecogenomics/GtdbTk) (Parks et al., 2018). Полногеномное выравнивание осуществляли с использованием алгоритма Nucmer, встроенного в программу Mummer (Marçais et al., 2018). Отклонения в составе пар Г + Ц рассчитывали по стандартному алгоритму в программе GenSkew (http:// genskew.csb.univie.ac.at/). Кластеры ортологичных генов определяли с помощью алгоритма Orthomcl программы Get_homologues (Contreras-Moreira, Vinuesa, 2013). Данные выравнивания и отклонения состава Г + Ц, а также координаты кластеров ортологичных генов использовали для построения круговой диаграммы сравнения двух геномов в программном пакете Circos (Krzywinski et al., 2009).
Выявление профагов. Для поиска потенциальных регионов профагов использовали сервер PHASTER (http://phaster.ca/) с использованием настроек, описанных в работе Arndt et al. (2016). Функции генов определяли по гомологии с известными вирусными белками в базе данных NCBI GenBank и пакетом VirFam (http://biodev.cea.fr/ virfam/), используя настройки, описанные в статье Lopes et al. (2014). Сравнение распределения генов в профаговых последовательностях проводили с помощью пакета tblastx и визуализировали программой Easyfig (Sullivan et al., 2011).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
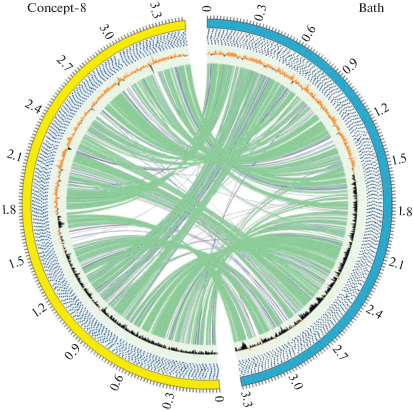
Общая характеристика генома и установление филогенетического положения штамма Concept-8. Геном штамма Concept-8 состоит из кольцевой хромосомы 3455527 п.н., кодирующей 3264 предполагаемых белков; среднее содержание пар Г + Ц составляет 62.3%. Плазмид выявлено не было. Размер генома Mc. capsulatus Bath несколько меньше (3 304 697 п.н.), а среднее содержание пар Г + Ц немного выше (63.6%), чем у штамма Concept-8. Общие характеристики геномов этих двух метанотрофов высоко сходны (табл. 1). В геномах штаммов Bath и Concept-8 имеется по две копии оперона рРНК. Сходство последовательностей генов 16S рРНК этих метанотрофов составляет 98.7%, что соответствует пределу различий видового уровня (Chun et al., 2018). Сравнение полных геномных последовательностей штаммов Bath и Concept-8 с помощью Genome-to-Genome-Distance-Calculator и ANI calculator, однако, выявило лишь 37.2 и 88.39% сходства соответственно, что свидетельствует о принадлежности штамма Concept-8 к новому виду рода Methylococcus (Chun et al., 2018). На рис. 1 представлено графическое изображение выравнивания геномов штамма Concept-8 и Mc. capsulatus Bath, демонстрирующее многочисленные регионы сходных участков последовательностей. В результате идентификации ортологов алгоритмом Orthomcl было обнаружено 2415 ортологичных кластера, что свидетельствует о единстве функций подавляющего большинства генов, кодируемых геномами Mc. capsulatus Bath и штамма Concept-8.
Таблица 1.
Характеристики геномов штамма Concept-8 и Mc. capsulatus Bath
| Характеристика генома | Concept-8 | Mc. capsulatus Bath |
|---|---|---|
| Размер генома, млн п.н. | 3.46 | 3.30 |
| Число контигов | 1 | 1 |
| Доля Г + Ц, мол. % | 62.3 | 63.6 |
| Открытые рамки считывания | 3266 | 3167 |
| рРНК (5S, 16S, 23S) | 2, 2, 2 | 2, 2, 2 |
| тРНК | 49 | 51 |
| CRISPR | 2 | 2 |
| мMMO оперон | 2 | 2 |
| рMMO оперон | 1 | 1 |
| Инсерционные последовательности | 64 | 61 |
| Профаги | 2 | 2 |
Рис. 1.
Графическое изображение выравнивания геномов штамма Concept-8 (желтый полукруг) и Mc. capsulatus Bath (голубой полукруг). Зеленые и сиреневые линии соединяют регионы сходных участков последовательностей длиной более 3000 и менее 3000 нуклеотидов соответственно. Концентрические окружности от центра к краю показывают: (1) график отклонений в составе пар Г + Ц, (2) кластеры ортологичных генов.
Филогеномный анализ с использованием базы данных GTDB показал, что штаммы Bath и Concept-8 формируют единый филогенетический кластер рода Methylococcus (рис. 2). Другими ближайшими филогенетическими родственниками являются представители родов Methylocaldum, Methylomagnum, Methyloterricola и Methylogaea.
Рис. 2.
Филогенетическое положение штамма Concept-8 по данным сравнительного анализа геномов представителей семейства Methylococcaceae, выполненного с помощью Genome Taxonomy Database toolkit, версия 04-RS89 (Parks et al., 2018). Бутстрeпы рассчитаны методом “maximum-likelihood” путем построения 100 альтернативных дендрограмм. Показаны значения бутстрепов более 70%. В качестве корня использованы 16 геномов метанотрофов семейства Methylocystaceae. Маркер – 0.1 замена на одну аминокислотную последовательность.

Ферменты цепи окисления метана. Штамм Concept-8 обладает двумя формами ключевого фермента окисления метана – мембранной и растворимой метанмонооксигеназами (мММО и рММО соответственно). В геноме этого метанотрофа обнаружены две копии полного кластера pmoBAC (peg. 123–125 и 1332–1334, см. файл с дополнительными материалами), кодирующего мMMO. Две аналогичные копии pmoBAC присутствуют в геноме Mc. capsulatus Bath, при этом последовательности pmoA у этих метанотрофов близки (99% идентичности транслированных аминокислотных последовательностей). Геном штамма Concept-8 имеет также дополнительную копию pmoC (pmoC3, peg. 1112), почти идентичную pmoC3 из Mc. capsulatus Bath. Гены pmoC3, как правило, составляют оперон с генами, кодирующими три белка неизвестной функции, предположительно имеющими отношение к окислению метана (Ward et al., 2004). Компоненты рMMO у штамма Concept-8 кодируются генным кластером mmoXYBZDC (peg. 2925–2930), аналогичным таковому у Mс. capsulatus Bath, и обнаруживают высокую гомологию последовательностей MmoX (98% идентичности). В том же локусе хромосомы расположены гипотетический белок (peg. 2931) и четыре открытые рамки считывания mmoGQSR (peg. 2932–2935), предположительно кодирующие большую субъединицу бактериального шаперонина GroEL (mmoG), двухкомпонентную сенсорно-регуляторную систему (mmoQ и mmoS) и активатор транскрипции (mmoR) (Csáki et al., 2003).
Геном штамма Concept-8 кодирует две метанолдегидрогеназы (МДГ), катализирующие вторую стадию окисления С1-субстрата. Структурные компоненты гетеродимерной Ca2+-зависимой метанолдегидрогеназы с пирролохинолинхиноном (PQQ) в активном центре, и белки, необходимые для ее каталитической функции, кодируются кластером MxaFJGIRACKLD (peg. 2400–2410), организация генов которого у Concept-8 и Mс. capsulatus Bath полностью идентична. Данный кластер включает гены mxaFI большой и малой субъединиц метанолдегидрогеназы и ген mxaG цитохрома cL – специфического акцептора электронов, а также гены белков, необходимых для встраивания Ca2+ в активный центр фермента (mxaACKL), и белков с неизвестными функциями (mxaRS) (Vuilleumier et al., 2009; Chistoserdova, 2011). Геномный анализ выявил наличие у штамма Concept-8 и Mс. capsulatus Bath альтернативной односубъединичной метанолдегидрогеназы XoxF-МДГ, которая содержит вместо кальция редкоземельный элемент в активном центре (peg. 1107) (Hibi et al., 2011).
Окислительные превращения формальдегида обеспечиваются ферментами тетрагидрофолатного (ТГФ) пути. В этом пути N5,N10-метилен-ТГФ, образующийся спонтанной конденсацией СН2О с ТГФ, с участием метилен-ТГФ-дегидрогеназы (peg. 810) окисляется в N5,N10-метенил-ТГФ, а последний метенил-ТГФ-циклогидролазой (peg. 810) модифицируется в N5-формил-ТГФ, и, наконец, формил-ТГФ-циклолигаза (peg. 1243) образует свободный формиат. На уровне метенил-ТГФ и/или формил-ТГФ С1-субстрат вступает в процесс синтеза серина и реакции серинового цикла ассимиляции углерода. Генетические детерминанты ТГФ-пути у штамма Concept-8 и Mс. capsulatus Bath практически идентичны.
Анализ генома штамма Concept-8 показал наличие полного набора ферментов тетрагидрометаноптерин (ТГМП)-зависимого пути окисления формальдегида в СО2, включая метилен-ТГМП дегидрогеназу (peg. 1610–1611) и метенил-ТГМП циклогидролазу (peg. 1341), а также формил-метанофуран-формилтрансферазу (peg. 1336) и ABC-субъединицы формил-метанофурандегидрогеназы (peg. 1337–1338, peg. 1335). Перечисленная последовательность ферментов, использующих кофакторы ТГМП и метанофуран, присутствует также у Mс. capsulatus Bath.
У штамма Concept-8 присутствуют гены трехсубъединичной формиатдегидрогеназы, катализирующей последнюю стадию окисления метана до CO2 (peg. 2339–2341). Штамм имеет также пятигенный оперон, кодирующий формиатдегидрогеназу (peg. 17–21), гомологичную цитоплазматической NAD+-зависимой формиатдегидрогеназе из Rhodobacter capsulatus, состоящей из (abc)2 субъединиц и катализирующей обратимую реакцию окисления формиата в СО2 (Hartmann, Leimkühler, 2013). Помимо структурных белков фермента, оперон кодирует полипептиды FdsC и FdsD, участвующие в модификации молибденового кофактора и его встраивание в фермент. Однако у штамма Concept-8 отсутствует гомолог двухсубъединичной формиатдегидрогеназы, которая имеется у Mс. capsulatus Bath (MCA2576-2577).
У штамма Concept-8 в окислении формальдегида до СО2 могут участвовать также компоненты РМФ-пути. В окислительном РМФ-цикле формальдегид реагирует с рибулозо-5-фосфатом с образованием гексулозо-6-фосфата, последний изомеризуется во фруктозо-6-фосфат и далее в глюкозо-6-фосфат. Штамм кодирует четыре изоформы глюкозо-6-фосфатдегидрогеназы (peg. 1745, peg. 1388, peg. 1965 и peg. 2738) и две 6-фосфоглюконатдегидрогеназы/декарбоксилазы (peg. 384, peg. 2737), которые производят молекулу CO2, восстанавливая две молекулы НАД(Ф) и регенерируя рибулозо-5-фосфат. В отличие от штамма Concept-8, Mс. capsulatus Bath имеет только две изоформы глюкозо-6-фосфатдегидрогеназы и одну 6-фосфоглюконатдегидрогеназу (Ward et al., 2004).
Пути ассимиляции углерода. Рибулозомонофосфатный (РМФ) цикл. Аналогично Mс. capsulatus Bath, штамм Concept-8 имеет по две идентичные копии генов гексулозофосфатсинтазы и гексулозофосфатизомеразы, а также ген, кодирующий слитый белок гексулозофосфатсинтаза/изомераза (peg. 1212). Компоненты РМФ-цикла линейно дуплицированы: нуклеотидная последовательность длиной 4946 п.н. имеет почти идентичный повтор с геном трансальдолазы в центральной части (peg. 1646), окруженным генами гексулозофосфатизомеразы (peg. 1645, peg. 1651), гексулозофосфатсинтазы (peg. 1650, peg.1644), фруктозобисфосфатальдолазы (peg. 1648, peg. 1641) и транскетолазы (peg. 1640, peg. 1647). Структура генного кластера РМФ-цикла у двух метилококков аналогична. Кроме данных штаммов, только у Methylomarinum vadi и Methylomicrobium buryatense 5G выявлена мультикопийность генов hps и phi. Дупликация гена hps (но не phi гена) характерна также для представителей рода Methylomonas. Как отражается мультикопийность ключевых генов РМФ-цикла на ростовые характеристики метанотрофов – важный предмет последующих исследований.
Сериновый путь. Штамм Сoncept-8 имеет генетические детерминанты, по крайней мере, части ферментов серинового пути С1 ассимиляции. Кластер sga-hpr-gck3 (peg. 3–5) кодирует серин-глиоксилат аминотрансферазу, гидроксипируватредуктазу и 3-глицераткиназу – индикаторные ферменты серинового пути, а также малил-КоА лиазу и малаттиокиназу (peg. 194–196) – ферменты, участвующие в синтезе глиоксилата – предшественника глицина, реакция которого с метенил-ТГФ и/или формил-ТГФ приводит к синтезу серина. Однако у штаммов Сoncept-8 и Bath отсутствует ФЕП-карбоксилаза – один из ключевых ферментов серинового пути. Восполнение С4-метаболитов цикла Кребса, по-видимому, осуществляет пируваткарбоксилаза, кодируемая последовательностью peg. 1178–1179. Однако ген пируваткарбоксилазы отсутствует у штамма Bath (Ward et al., 2004). Кроме реакции, катализируемой малил-КоА лиазой, регенерация акцептора С1-единиц, глицина, может происходить как результат превращений фосфогликолата, образуемого в оксигеназной реакции Рубиско через гликолат и глиоксилат с участием фосфогликолатфосфатазы (peg. 2032), гликолатоксидазы (peg. 3306–3308), и серингдиоксилат аминотрансферазы (peg. 3).
Цикл Кальвина. Геномный анализ выявил у Methylococcus Concept-8 гены, кодирующие большую (cbbL) и малую (cbbS) субъединицы рибулозо-1,5-бисфосфаткарбоксилазы/оксигеназы (Рубиско) (peg. 1217–1218), а также ген cbbQ (peg. 1220), кодирующий полипептид с предполагаемой функцией посттрансляционного активатора Рубиско (Baxter et al., 2002). Аналогично СbbL из других представителей Proteobacteria, включая Mс. capsulatus Bath и цианобактерий, у штамма Concept-8 данный фермент относится к “зеленому” типу Рубиско (clade A) (Baxter et al., 2002).
Пути распада фосфосахаров. Геном штамма Concept-8 кодирует несколько путей распада С6-фосфосахаров – первичных продуктов ассимиляции в РМФ цикле, в числе которых гликолиз, путь Энтнера–Дудорова (ЭД) и фосфокетолазный путь.
Штамм Concept-8 имеет полный набор ферментов модифицированного пути Эмбдена–Мейергофа–Парнаса, в котором реакция образования фруктозо-1,6-фосфата из фруктозо-6-фосфата обратима и катализируется ФФн-зависимой фосфофруктокиназой (peg. 3029). Этот фермент, а также ФФн-зависимая пируватфосфатдикиназа (peg. 1612), обеспечивают полную обратимость гликолитической последовательности, при этом ФФн-фосфофруктокиназа компенсирует отсутствие фруктозобисфосфатазы, как это показано ранее для Mс. capsulatus Bath (Reshetnikov et al., 2008).
В геноме штамма Concept-8 присутствуют также все ферменты ЭД пути распада фруктозо-6-фосфата в глицеральдегид-3-фосфат и пируват: глюкозо-6-фосфатизомераза (peg. 3024), четыре глюкозо-6-фосфатдегидрогеназы (peg. 1745, peg. 1388, peg. 1965 и peg. 2738), две 6-фосфоглюконолактоназы (peg. 1744, peg. 1387), 6-фосфоглюконатдегидратаза (peg. 1748) и 2-кето-3-дезокси-6-фосфоглюконатальдолаза (peg. 1749). Следует отметить, что у штамма Bath имеются только два изомера глюкозо-6-фосфатдегидрогеназы.
У штамма Сoncept-8 присутствуют генетические детерминанты альтернативного пути распада сахаров с участием фосфокетолазы (peg. 3117), катализирующей образование глицеральдегид-3-фосфата/эритрозо-4-фосфата и ацетилфосфата из первичных продуктов С1-ассимиляции – фруктозо-6-фосфата и/или ксилулозо-5-фосфата. В результате дальнейшего метаболизма ацетилфосфата с участием ацетаткиназы (peg. 3115) образуются ацетат и АТФ (Rozova et al., 2015; Henard et al., 2017), или напрямую ацетил-КоА с участием фосфоацетилтрансферазы (peg. 3288). Последняя имеется у штамма Сoncept-8, но отсутствует у штамма Bath. Оба метилококка кодируют ацетил-КоА синтетазу (EC 6.2.1.1, peg. 478), катализирующую синтез ацетил-КоА, сопровождающийся распадом АТФ до АМФ. Следует отметить, что фосфоацетилтрансфераза у Methylococcus Сoncept-8 имеет низкую гомологию с ферментами из других метанотрофных бактерий и, по-видимому, была приобретена в результате горизонтального переноса генов.
Синтез запасных соединений углерода. Штамм Сoncept-8 кодирует ферменты, необходимые для синтеза гликогена: 4-α-глюканотрансферазу (амиломальтазу) – глюкозо-1-фосфат аденилилтрансферазу – 1,4-α-глюкан разветвляющий фермент GlgB – гликоген синтазу 1 – АДФ-рибозопирофосфатазу, соответствующие гены организованы в кластере peg. 3328–3332, аналогичном таковому у Mс. capsulatus Bath (MCA1473–1477). Дополнительный генный кластер кодирует гликоген синтазу 2 и α-амилазу (peg. 2009–2010). Две гликогенсинтазы могут стать мишенью для модификации штамма-продуцента для предотвращения расхода углерода на синтез запасных углеводных соединений и повышения относительного содержания белка в биомассе.
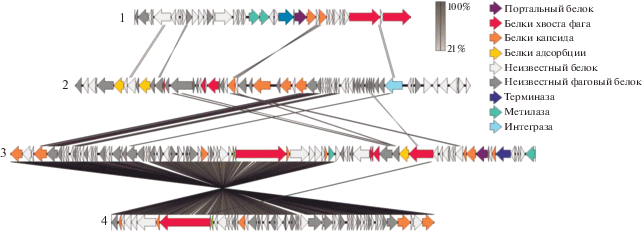
Выявление профагов. В геномах штаммов Mc. capsulatus Bath и штамм Concept-8 c помощью сервера PHASTER обнаружено по 2 потенциально полных профаговых региона (рис. 3). При этом последовательность генов одного из профаговых участков в геноме штамма Concept-8 имела высокое сходство с частью второго профага в этом же геноме (99.6% сходства кодируемых аминокислотных последовательностей). Вероятно, в данном случае имела место дупликация участка последовательности исходного профага. Обратная картина наблюдалась в геноме Mc. capsulatus Bath, где профаговые регионы выглядят как 2 разделенных участка одного исходного вирусного генома. Как для Mc. capsulatus Bath, так и для штамма Concept-8 в профаговых регионах были найдены гены, кодирующие вирусные структурные белки, характерные для представителей отряда Caudovirales (хвостатые бактериофаги). Однако генов, обеспечивающих репликацию ДНК и РНК профагов и лизис клетки-хозяина на поздних стадиях инфекции обнаружено не было. Как правило, это свидетельствует о потере данными профагами способности к индукции, но их влияние на регуляцию метаболизма бактерии хозяина и возможность репликации с помощью полимераз клетки-хозяина не могут быть исключены без проведения дополнительных исследований.
Рис. 3.
Структура генных кластеров, ассоциированных с профагами, в геномах Mc. capsulatus Bath (1, 2) и штамма Concept-8 (3, 4).
Таким образом, результаты анализа генома штамма Concept-8 подтвердили его принадлежность к роду Methylococcus, однако указали на его существенные, видового уровня отличия от Mс. capsulatus Bath. Сравнительный анализ метаболических путей, кодируемых в геномах этих двух метанотрофов, также выявил ряд отличий, уровень проявления которых нуждается в дальнейшей экспериментальной проверке.
Список литературы
Гальченко В.Ф. Метанотрофные бактерии. M.: ГЕОС, 2001. 500 с.
Григорян А.Н., Горская Л. Использование природного газа для микробиологического синтеза. М.: ОНТИ Микробиопром, 1970. 90 с.
Лалов В.В. Анализ и синтез энерготехнологических систем производства кормового белка из природного газа. Автореф. дис. … докт. биол. наук. Москва, 1991.
Altschul S.F., Madden T.L., Schäffer A.A., Zhang J., Zhang Z., Miller W., Lipman D.J. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs // Nucleic Acids Res. 1997. V. 25. P. 3389–3402.
Arndt D., Grant J.R., Marcu A., Sajed T., Pon A., Liang Y., Wishart D.S. PHASTER: a better, faster version of the PHAST phage search tool // Nucleic Acids Res. 2016. V. 44. P. 16–21.
Baxter N.J., Hirt R.P., Bodrossy L., Kovacs K.L., Embley M.T., Prosser J.I., Murrell C.J. The ribulose-1,5-bisphosphate carboxylase/oxygenase gene cluster of Methylococcus capsulatus (Bath) // Arch. Microbiol. 2002. V. 177. P. 279–289.
Chistoserdova L. Modularity of methylotrophy, revisited // Environ. Microbiol. 2011. V. 13. P. 2603–2622.
Chun J., Oren A., Ventosa A., Christensen H., Arahal D.R., da Costa M.S., Rooney A.P., Yi H., Xu X.W., De Meyer S., Trujillo M.E. Proposed minimal standards for the use of genome data for the taxonomy of prokaryotes // Int. J. Syst. Evol. Microbiol. 2018. V. 68. P. 461–466.
Contreras-Moreira B., Vinuesa P. GET_HOMOLOGUES, a versatile software package for scalable and robust microbial pangenome analysis // Appl. Environ. Microbiol. 2013. V. 79. P. 7696–7701.
Csáki R., Bodrossy L., Klem J., Murrell J.C., Kovács K.L. Genes involved in the copper-dependent regulation of soluble methane monooxygenase of Methylococcus capsulatus (Bath): Cloning, sequencing and mutational analysis // Microbiology (SGM). 2003. V. 149. P. 1785–1795.
Foster J.W., Davis R.H. A methane-dependent coccus, with notes on classification and nomenclature of obligate, methane-utilizing bacteria // J. Bacteriol. 1966. V. 91. P. 1924–1931.
Hamer G., Harrison D.E.F. Single cell protein: the technology, economics and future potential // Hydrocarbons in Biotechnology / Eds. Harrison D.E.F., Higgins I.J., London W. R. London: Heyden Institute of Petroleum, 1980. P. 59–73.
Hanson R., Hanson T. Methanotrophic bacteria // Microbiol. Rev. 1996. V. 60. P. 439–471.
Hartmann T., Leimkühler S. The oxygen-tolerant and NAD+-dependent formate dehydrogenase from Rhodobacter capsulatus is able to catalyze the reduction of CO2 to formate // FEBS J. 2013. V. 280. P. 6083–6096.
Henard C.A., Smith H.K., Guarnieri M.V. Phosphoketolase overexpression increases biomass and lipid yield from methane in an obligate methanotrophic biocatalyst // Metab. Eng. 2017. P. 152–158.
Hibi Y., Asai K., Arafuka H., Hamajima M., Iwama T., Kawai K. Molecular structure of La3+-induced methanol dehydrogenase-like protein in Methylobacterium radiotolerans // J. Biosci. Bioeng. 2011. V. 111. P. 547–549.
Kanehisa M., Goto S., Sato Y., Kawashima M., Furumichi M., Tanabe M. Data, information, knowledge and principle : back to metabolism in KEGG // Nucleic Acids Res. 2014. V. 42. P. D199–D205.
Kanehisa M., Sato Y., Morishima K. BlastKOALA and GhostKOALA: KEGG Tools for Functional Characterization of Genome and Metagenome Sequences // J. Mol. Bi-ol. 2016. V. 428. P. 726–731.
Kao W.C., Chen Y.R., Yi E.C., Lee H., Tian Q., Wu K.M., Tsai S.F., Yu S.S.F., Chen Y.J., Aebersold R., Chan S.I. Quantitative proteomic analysis of metabolic regulation by copper ions in Methylococcus capsulatus (Bath) // J. Biol. Chem. 2004. V. 279. P. 51554–51560.
Khmelenina V.N., Murrell J.C., Smith V.J., Trotsenko Y.A. Physiology and biochemistry of the aerobic methanotrophs // Aerobic Utilization of Hydrocarbons, Oils and Lipids. Handbook of Hydrocarbon and Lipid Microbiology / Ed. Rojo F. Cham: Springer, 2018. P. 1–25.
Krzywinski M., Schein J., Birol I., Connors J., Gascoyne R., Horsman D., Jones S.J., Marra M.A. Circos: An information aesthetic for comparative genomics // Genome Res. 2009. V. 19. P. 1639–1645.
Lagesen K., Hallin P., Rødland E.A., Stærfeldt H.H., Rognes T., Ussery D.W. RNAmmer: Consistent and rapid annotation of ribosomal RNA genes // Nucleic Acids Res. 2007. V. 35. P. 3100–3108.
Larsen Ø., Karlsen O.A. Transcriptomic profiling of Methylococcus capsulatus (Bath) during growth with two different methane monooxygenases // MicrobiologyOpen. 2016. V. 5. P. 254–267.
Lieven C., Petersen L.A.H., Jørgensen S.B., Gernaey K.V., Herrgard M.J., Sonnenschein N.A. A genome-scale metabolic model for Methylococcus capsulatus predicts reduced efficiency uphill electron transfer to pMMO // bioRxiv. 2018. P. 329714.
Linton J., Buckee J. Interactions in a methane-utilising mixed culture in a chemostat // J. Gen. Microbiol. 1977. V. 101. P. 219–225.
Lopes A., Tavares P., Petit M.A., Guérois R., Zinn-Justin S. Automated classification of tailed bacteriophages according to their neck organization // BMC Genomics. 2014. V. 15. P. 1–17.
Lowe V.M., Eddy S.R. TRNAscan-SE: A program for improved detection of transfer RNA genes in genomic sequence // Nucleic Acids Res. 1996. V. 25. P. 955–964.
Marçais G., Delcher A.L., Phillippy A.M., Coston R., Salzberg S.L., Zimin A. MUMmer4: A fast and versatile genome alignment system // PLoS Comput. Biol. 2018. V. 14. e1005944.
Meier-Kolthoff J.P., Auch A.F., Klenk H.P., Göker M. Genome sequence-based species delimitation with confidence intervals and improved distance functions // BMC Bioinform. 2013. V. 14. P. 60.
Overbeek R., Olson R., Pusch G.D., Olsen G.J., Davis J.J., Disz T., Edwards R.A., Gerdes S., Parrello B., Shukla M., Vonstein V., Wattam A.R., Xia F., Stevens R. The SEED and the Rapid Annotation of microbial genomes using Subsystems Technology (RAST) // Nucleic Acids Res. 2014. V. 42. P. 206–214.
Parks D.H., Chuvochina M., Waite D.W., Rinke C., Skarshewski A., Chaumeil P.A., Hugenholtz P. A standardized bacterial taxonomy based on genome phylogeny substantially revises the tree of life // Nat. Biotechnol. 2018. V. 36. P. 996–1004.
Reshetnikov A.S., Rozova O.N., Khmelenina V.N., Mustakhimov I.I., Beschastny A.P., Murrell J.C., Trotsenko Y.A. Characterization of the pyrophosphate-dependent 6-phosphofructokinase from Methylococcus capsulatus Bath // FEMS Microbiol. Lett. 2008. V. 288. P. 202–210.
Rodriguez-R L.M., Konstantinidis K.V. Bypassing cultivation to identify bacterial species // Microbe. 2014. V. 9. P. 111–118.
Rozova O.N., Khmelenina V.N., Gavletdinova J.Z., Mustakhimov I.I., Trotsenko Y.A. Acetate kinase – an enzyme of the postulated phosphoketolase pathway in Methylomicrobium alcaliphilum 20Z // Antonie van Leeuwenhoek. 2015. V. 108. P. 965–974.
Seemann T. Prokka: Rapid prokaryotic genome annotation // Bioinformatics. 2014. V. 30. P. 2068–2069.
Sullivan M.J., Petty N.K., Beatson S.A. Easyfig: A genome comparison visualizer // Bioinformatics. 2011. V. 27. P. 1009–1010.
Trotsenko Y.A., Murrell J.C. Metabolic aspects of aerobic obligate methanotrophy // Adv. Appl. Microbiol. 2008. V. 63. P. 183–229.
Varani A.M., Siguier P., Gourbeyre E., Charneau V., Chandler M. ISsaga is an ensemble of web-based methods for high throughput identification and semi-automatic annotation of insertion sequences in prokaryotic genomes // Genome Biol. 2011. V. 12. № 3. R30.
Vuilleumier S., Chistoserdova L., Lee M.C., Bringel F., Lajus A., Yang Z., Gourion B., Barbe V., Chang J., Cruveiller S., Dossat C., Gillett W., Gruffaz C., Haugen E., Hourcade E., Levy R., Mangenot S., Muller E., Nadalig T., Pagni M., Penny C., Peyraud R., Robinson D.G., Roche D., Rouy Z., Saenempechek C., Salvignol G., Vallenet D., Zaining W., Marx C.J., Vorholt J.A., Olson M. V., Kaul R., Weissenbach J., Médigue C., Lidstrom M.E. Methylobacterium genome sequences: A reference blueprint to investigate microbial metabolism of C1 compounds from natural and industrial sources // PLoS One. 2009. V. 4. № 5. e5584.
Ward N., Larsen Ø., Sakwa J., Bruseth L., Khouri H., Durkin A.S., Dimitrov G., Jiang L., Scanlan D., Kang K.H., Lewis M., Nelson K.E., Methé B., Wu M., Heidelberg J.F., Paulsen I.T., Fouts D., Ravel J., Tettelin H., Ren Q., Read T., DeBoy R.T., Seshadri R., Salzberg S.L., Jensen H.B., Birkeland N.K., Nelson W.C., Dodson R.J., Grindhaug S.H., Holt I., Eidhammer I., Jonasen I., Vanaken S., Utterback T., Feldblyum T.V., Fraser C.M., Lillehaug J.R., Eisen J.A. Genomic insights into methanotrophy: The complete genome sequence of Methylococcus capsulatus (Bath) // PLoS Biol. 2004. V. 2. P. 707–713.
Wick R.R., Judd L.M., Gorrie C.L., Holt K.E. Unicycler: Resolving bacterial genome assemblies from short and long sequencing reads // PLoS Comput. Biol. 2017. V. 13. № 6. e1005595.
Дополнительные материалы
- скачать ESM.docx
- Таблица. Гены основных метаболических путей Methylococcus sp. Concept-8 (представлены в том порядке, в котором они обсуждаются в статье).


