Микробиология, 2020, T. 89, № 5, стр. 503-510
Антимикробная активность нового пресноводного планктомицета Lacipirellula parvula PX69T
С. Э. Белова a, В. А. Салтыкова a, b, С. Н. Дедыш a, *
a Институт микробиологии им. С.Н. Виноградского, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” РАН
119071 Москва, Россия
b Московский государственный университет им. М.В. Ломоносова
119991 Москва, Россия
* E-mail: dedysh@mail.ru
Поступила в редакцию 28.03.2020
После доработки 29.03.2020
Принята к публикации 07.04.2020
Аннотация
Планктомицеты – это филогенетическая группа бактерий, отличающихся сложной организацией клеток, большими геномами и широким спектром кодируемых в них вторичных метаболитов. Анализ доступных геномов планктомицетов позволил предположить перспективность этих бактерий как источника потенциально новых биологически активных соединений, однако число полученных в культурах планктомицетов остается ограниченным. Настоящая работа была предпринята для оценки наличия и спектра антимикробной активности недавно описанного пресноводного планктомицета Lacipirellula parvula PX69T, представляющего новое семейство Lacipirellulaceae. В геноме штамма PX69T было выявлено 6 генных кластеров, кодирующих поликетидсинтазы III типа, синтетазы нерибосомных пептидов и другие вторичные метаболиты, обнаруживающие низкое сходство с таковыми у других бактерий. Экспериментальные исследования подтвердили наличие антимикробной активности штамма РХ69T в отношении ряда тест-культур микроорганизмов. Наибольшая антимикробная активность, до 83–87% подавления роста, была зарегистрирована в отношении Staphylococcus aureus и Candida albicans. В работе проведена оптимизация среды и длительности культивирования штамма РХ69T для достижения максимальной антимикробной активности.
Planctomycetes (планктомицеты) – это филогенетическая группа домена Bacteria, представители которой имеют необычную морфологию и организацию клеток, сложный жизненный цикл и отличаются уникальностью состава мембран, механизмов транспорта высокомолекулярных субстратов в клетку и других характеристик (Staley et al., 1992; Fuerst, 1995; Boedeker et al., 2017; Reintjes et al., 2017; Wiegand et al., 2018). Изначально планктомицеты были описаны как группа водных бактерий, однако с введением в практику методов молекулярной экологии они были обнаружены в широком спектре водных и наземных мест обитания с различными физико-химическими параметрами (Fuerst, 1995; Buckley et al., 2006; Lage, Bondoso, 2014; Dedysh, Ivanova, 2019; Wiegand et al., 2019). Из-за сложностей культивирования, перечень доступных для исследования изолятов планктомицетов остается ограниченным, причем подавляющее большинство ныне охарактеризованных представителей Planctomycetes принадлежат к классу Planctomycetia, который включает порядки Planctomycetales, Gemmatales, Isosphaerales и Pirellulales (Dedysh et al., 2020).
Представители Planctomycetia имеют большие геномы (Glöckner et al., 2003; Guo et al., 2012; Ivanova et al., 2017; Ravin et al., 2018; Wiegand et al., 2019), причем более половины закодированного в геномах потенциала пока остается нерасшифрованным. Средний размер генома у этих бактерий составляет 7.2 Mb (Wiegand et al., 2019), тогда как обладатель самого большого генома среди планктомицетов – Fimbriiglobus ruber SP5T – имеет геном размером 12.4 Mb (Ravin et al., 2018). Как известно, размер генома бактерий коррелирует со способностью кодировать синтез разнообразных вторичных метаболитов, многие из которых представлены биоактивными соединениями, имеющими медицинское значение (Donadio et al., 2007). Анализ первой геномной последовательности планктомицета, полученной для Rhodopirellula baltica (Glöckner et al., 2003), выявил наличие генов, кодирующих две синтетазы нерибосомных пептидов (NRPS), две поликетидсинтазы (PKS) и двухмодульный гибрид NRPS-PKS, расположенные на различных участках хромосомы (Donadio et al., 2007). Дальнейшее расширение спектра доступных геномов планктомицетов позволило установить, что генные кластеры, кодирующие синтез поликетидов, нерибосомных пептидов, а также терпенов, бактериоцинов, эктоина и других вторичных метаболитов присутствуют во всех из них в числе от 2 до 15 (Jeske et al., 2013; Graça et al., 2016; Wiegand et al., 2019). Наибольший интерес представляют PKS и NRPS, поскольку именно эти ферментные комплексы отвечают за синтез многих известных антибиотиков (Schwarzer et al., 2003; Fischbach, Walsh, 2006; Tracanna et al., 2017). Однако и синтез белков-бактериоцинов, ингибирующих развитие родственных видов бактерий, включая антибиотикорезистентные штаммы (Cotter et al., 2013), также представляет интерес. Таким образом, анализ геномов планктомицетов позволил предположить перспективность этих бактерий как источника новых биологически активных веществ и антибиотиков (Jeske et al., 2013; Graça et al., 2016; Tracanna et al., 2017; Wiegand et al., 2019). Дальнейшие экспериментальные исследования подтвердили наличие антимикробной активности у ряда планктомицетов, таких как Planctopirus limnophila DSM 377, Rhodopirellula baltica DSM 10527 (Jeske et al., 2016), Rhodopirellula lusitana (Graça et al., 2016) и Planctopirus hydrillae (Yadav et al., 2018), однако спектр исследованных культур планктомицетов пока остается узким.
В контексте проблемы возникновения множественной устойчивости патогенов к имеющимся антибиотикам, поиск продуцентов новых антимикробных веществ имеет высокую актуальность. Очевидно, что одним из объектов таких поисков должны быть новые культуры планктомицетов.
Настоящая работа была предпринята с целью оценки наличия и спектра антимикробной активности недавно описанного пресноводного планктомицета Lacipirellula parvula PX69T, представляющего новое семейство Lacipirellulaceae (Dedysh et al., 2020).
ОБЪЕКТЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Выявление генов, кодирующих синтез вторичных метаболитов. Анализ генома Lacipirellula parvula РХ69T (номер депонирования в ГенБанке AP021861) на наличие генных кластеров, кодирующих вторичные метаболиты, был проведен с использованием онлайн сервера antiSMASH (https:// antismash.secondarymetabolites.org) (Weber et al., 2015). Аналогичный анализ был также проведен для геномов других известных представителей семейства Lacipirellulaceae – “Bythopirellula goksoyri” Pr1d (GCF_008065115) и таксономически неохарактеризованного планктомицета штамма I41 (GCA_007746075), а также для ряда планктомицетов филогенетически родственного семейства Pirellulaceae – Blastopirellula marina (GCF_000153105), Mariniblastus fucicola (GCF_001642875), Rhodopirellula baltica (GCF_000196115), Roseimaritina ulvae (GCF_ 001642915), Rubripirellula reticaptiva (GCF_007860175) и Pirellula staleyi (GCF_000025185).
Среды и условия культивирования. Штамм РХ69T выращивали в периодической культуре на среде GPM1 с глюкозой и пептоном в качестве субстратов роста при температуре 28°С и скорости перемешивания 150 об./мин. Состав среды GPM1 (г/л): глюкоза – 1.0; пептон – 0.5; дрожжевой экстракт – 0.25; NH4NO3 – 0.1; раствор Хатнера (Staley et al., 1992) – 20 мл/л. При оптимизации роста штамма РХ69T использовали модифицированные версии среды GPM1 с увеличенным содержанием субстратов роста – среду GPM2 с концентрацией глюкозы и пептона, соответственно, 1.5 и 0.75 г/л, а также среду GPM3, содержащую 1.75 и 1.0 г/л глюкозы и пептона соответственно. Культивирование проводили во флаконах объемом 120 мл, содержащих 30 мл среды. Рост прослеживали путем измерения оптической плотности культуры (OD600) на спектрофотометре. Эксперименты проводили в четырехкратной повторности.
Получение экстрактов вторичных метаболитов. Для анализа антимикробной активности культуру штамма РХ69T наращивали во флаконах объемом 500 мл, содержащих 150 мл соответствующей среды. Экстракцию вторичных метаболитов из культуральной жидкости (КЖ) планктомицета проводили с использованием адсорбента “Amberlite®XAD®-2” (“Supelco”, “Sigma-Aldrich”, США) в соответствии с ранее описанным протоколом (Jeske et al., 2016). Для этого 600 мл культуры штамма РХ69T инкубировали с 2% (об./об.) адсорбента при 28°С и перемешивании со скоростью 80 об./мин в течение 3 сут. Затем адсорбент с комплексом связанных метаболитов собирали центрифугированием, переносили в колбу для экстракции, приливали 200 мл этилацетата и инкубировали 1 ч в темноте при комнатной температуре, после чего фильтровали. Этилацетат удаляли с помощью роторного вакуумного испарителя (Kika® WERKE HB4 basic) (36°С; 130 об./мин; 1 мбар). Полученные соединения растворяли в 1 мл метанола и хранили при –20°С.
Определение спектра и величины антимикробной активности. Проверку наличия и активности полученных экстрактов КЖ Lacipirellula parvula PX69T осуществляли в отношении ряда тест-культур микроорганизмов: бактерий Staphylococcus aureus ATCC 29213, Pseudomonas aeruginosa ATCC 27853, E. coli ATCC 25922 и K12 F+, Bacillus subtilis ATCC 6633, B. cereus var. mycoides HB и дрожжей Candida albicans 24433 и 10231. Тест-культуры бактерий выращивали на среде Nutrient Agar (“Difco”), дрожжи поддерживали на среде Сабуро следующего состава (г/л): глюкоза – 20; пептон – 5; дрожжевой экстракт – 5; агар – 15.
Наличие антимикробной активности выявляли по зоне подавления роста тест-культур микроорганизмов вокруг бумажных дисков (“Ahlstrom-Unksjoö Germany GmbH”, Grade BF 3, диаметр 9 мм), пропитанных экстрактами КЖ в количестве 60 мкл КЖ на диск и наложенных на газоны тест-культур. Последние получали рассевом тест-культур на соответствующие среды с “мягким” агаром (7 г/л).
Количественную оценку воздействия исследуемых экстрактов на рост тест-культур осуществляли с использованием микропланшетов и сканнера MR-96A (“Mindray”, КНР). В качестве положительного контроля роста использовали суспензии тест-культур на соответствующей питательной среде. В опытных вариантах к суспензии тест-культур на питательной среде добавляли экстракт КЖ штамма РХ69Т в концентрации 8% (об./об.). В качестве дополнительного контроля влияния растворителя (метанола) на рост тест-культур к их суспензиям в питательной среде добавляли такое же количество метанола. Эксперименты проводили в четырехкратной повторности. Микропланшеты с культурами инкубировали в течение 2 сут при 28°С, после чего проводили измерения оптической плотности культур при 450 нм и перемешивании средней интенсивности в течение 10 с. Результаты оценивали по разнице оптической плотности тест-культур в вариантах опыта с добавлением экстрактов КЖ штамма РХ69Т по сравнению с контролем.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Анализ генома Lacipirellula parvula PX69T на наличие генов синтеза вторичных метаболитов. Использование программного пакета antiSMASH позволило выявить в геноме штамма PX69T шесть генных кластеров, предположительно отвечающих за продукцию вторичных метаболитов (рис. 1). Три из них были классифицированы как кодирующие поликетидсинтазы III типа и два – как кодирующие синтетазы нерибосомных пептидов. Шестой из выявленных оперонов имел ряд признаков, позволяющих классифицировать его как связанный с синтезом вторичных метаболитов, но их оказалось недостаточно для предсказания природы этих метаболитов. Сходство выявленных в геноме штамма PX69T генов синтеза вторичных метаболитов по сравнению с таковыми у других микроорганизмов оказалось крайне низким. Ближайшие гомологи поликетидсинтаз штамма PX69T закодированы в геноме планктомицета семейства Gemmataceae, Gemmata obscuriglobus UQM 2246, причем лишь 17% кодирующих их генов обнаруживают сходство. Гены гомологов синтетаз нерибосомных пептидов штамма PX69T имеются в геноме планктомицета семейства Pirellulaceae, Rubripirellula obstinata CECT 8602 (14% генов обнаруживают сходство), а также миксобактерий Corallococcus coralloides DSM 2259 и Myxococcus xanthus DK 1622 (7–10% генов обнаруживают сходство).
Рис. 1.
Число генных кластеров, кодирующих вторичные метаболиты в геноме Lacipirellula parvula РХ69Т и двух других представителей семейств Lacipirellulaceae – “Bythopirellula goksoyri” Pr1d и таксономически неохарактеризованного штамма I41, а также некоторых представителей семейства Pirellulaceae. 1 – поликетидсинтазы I типа; 2 – поликетидсинтазы III типа; 3 – синтетазы нерибосомных пептидов; 4 – бактериоцины; 5 – терпены; 6 – эктоин; 7 – другие.
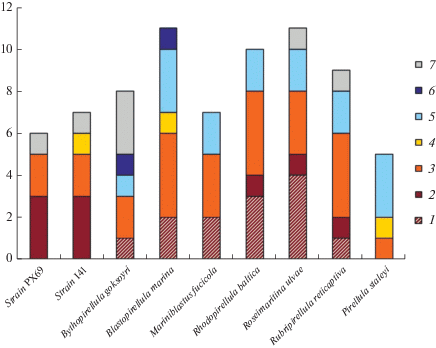
Спектр вторичных метаболитов, кодируемых в геноме другого, пока неохарактеризованного представителя рода Lacipirellula, штамма I41 (Wiegand et al., 2019), был высоко сходен с обнаруженным у штамма PX69T, но включал также и гены синтеза бактериоцина (рис. 1). В отличие от пресноводных планктомицетов рода Lacipirellula, геном морского представителя семейства Lacipirellulaceae, “Bythopirellula goksoyri” Pr1d, кодировал несколько иной, включающий эктоин и терпены, спектр вторичных метаболитов, что, возможно, объясняется спецификой местообитания этого планктомицета. Перечень метаболитов, кодируемых в геномах планктомицетов семейства Pirellulaceae, значительно варьировал у различных организмов, однако, в целом, он был шире у морских представителей этого семейства (рис. 1).
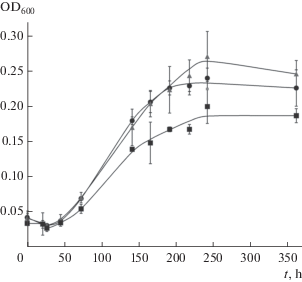
Влияние состава среды на динамику роста штамма РХ69Т. Ранее выполненные исследования антимикробной активности морских планктомицетов показали, что степень проявления их антимикробной активности зависит от состава среды культивирования (Graça et al., 2016). Наибольшая активность экстрактов КЖ планктомицетов была зарегистрирована при использовании среды с повышенным содержанием дрожжевого экстракта, пептона и глюкозы. В связи с этим была проведена оптимизация состава среды культивирования штамма РХ69Т путем прослеживания зависимости ростовых характеристик этого планктомицета от концентраций глюкозы и пептона в среде (рис. 2). Повышение содержания субстратов роста в среде приводило к увеличению скорости роста культуры и урожая клеток планктомицета. Ростовые характеристики штамма РХ69Т на средах GPM2 и GPM3 оказались близки, но максимальная оптическая плотность культуры достигалась при культивировании на среде GPM3. Дальнейшее увеличение концентраций глюкозы и пептона в среде не вызывало существенного увеличения оптической плотности культуры (данные не показаны).
Рис. 2.
Динамика роста Lacipirellula parvula РХ69Т на средах с различными концентрациями глюкозы и пептона. Квадратами показан рост на среде GPM1, кругами – на среде GPM2, треугольниками – на среде GPM3.
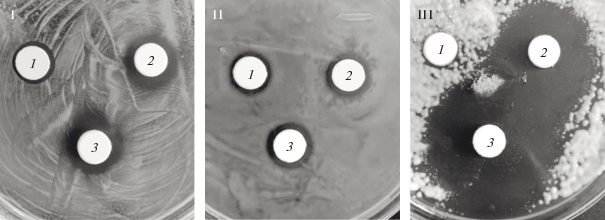
Спектр антимикробной активности нового планктомицета. Первоначальную проверку наличия и спектра антимикробной активности штамма РХ69Т проводили с помощью стандартного диско-диффузионного метода (рис. 3). Зоны подавления роста были зарегистрированы для всех использованных в работе тест-культур, но наиболее сильное ингибирование было отмечено в отношении грамположительных бактерий S. aureus (рис. 3, I) и дрожжей C. albicans (рис. 3, III). Экстракты КЖ, полученные на средах GPM2 и GPM3, обладали более сильным ингибирующим действием, что особенно ярко проявилось в отношении тест-культур дрожжей (рис. 3).
Рис. 3.
Зоны подавления роста тест-культур бактерий S. aureus (I), E. coli K12 F+ (II) и дрожжей C. albicans 24433 (III) экстрактами культуральной жидкости штамма РХ69Т, выращенного на средах GPM1 (1), GPM2 (2) и GPM3 (3). Фотографии сделаны после 2 сут инкубации.
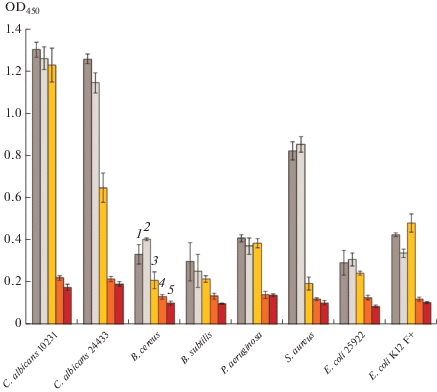
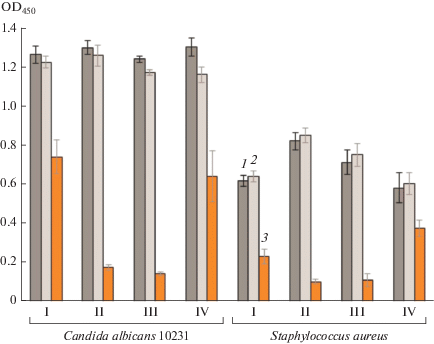
Количественная оценка антимикробной активности штамма РХ69Т. Культивирование в микропланшетах позволило количественно оценить степень ингибирования тест-культур экстрактами КЖ штамма РХ69Т. Наибольшее подавление роста тест-культур наблюдалось при использовании экстрактов, полученных на средах GPM2 и GPM3, тогда как активность полученных на среде GPM1 экстрактов в большинстве случаев была незначительной (рис. 4). Ингибирующая активность экстрактов КЖ, полученных на средах GPM2 и GPM3, была максимальной по отношению к S. aureus (85 и 87% подавления роста соответственно) и двум штаммам дрожжей C. albicans (83–86% соответственно). Менее выраженный ингибирующий эффект наблюдался по отношению к E. coli K12F+ (71 и 75% соответственно). Степень ингибирования роста остальных тест-культур – E. coli 25922, B. cereus, B. subtilis и P. aeruginosa – при использовании сред GPM2 и GPM3 составила 54–65 и 66–70% соответственно (рис. 4).
Рис. 4.
Антимикробная активность экстрактов культуральной жидкости Lacipirellula parvula РХ69Т в отношении тест-культур микроорганизмов: 1 – положительный контроль; 2 – контроль с добавлением метанола; 3, 4 и 5 – внесение экстрактов КЖ планктомицета, выращенного на средах GPM1, GPM2 и GPM3 соответственно. Данные приведены для экстракта 20-сут культуры планктомицета.
Зависимость антимикробной активности экстрактов КЖ штамма РХ69Т от длительности культивирования. Возраст культуры, использованной для получения экстракта КЖ, во многом определял его активность в отношении тест-культур (рис. 5). В случае всех исследованных культур, наибольшее ингибирующее воздействие оказывали экстракты КЖ 20- и 40-сут культур Lacipirellula parvula РХ69Т. Активность слишком “молодых” (11 сут инкубации) или “старых” (60 сут) экстрактов КЖ составляла менее половины таковой у 20- и 40-суточных культур. По-видимому, синтез соединений с антимикробной активностью имеет место только на определенной стадии развития культуры планктомицета, причем при слишком длительной инкубации может происходить их частичная деградация.
Рис. 5.
Зависимость антимикробной активности Lacipirellula parvula РХ69Т в отношении C. albicans 10231 и S. aureus от возраста культуры, использованной для экстракции вторичных метаболитов: 1 – положительный контроль; 2 – контроль с добавлением метанола; 3 – внесение экстракта КЖ планктомицета, выращенного на среде GPM3. Сроки инкубации культуры: I – 11 сут, II – 20 сут, II – 40 сут, IV – 60 сут.
Таким образом, проведенные нами исследования подтвердили наличие антимикробной активности Lacipirellula parvula РХ69T в отношении широкого ряда тест-культур микроорганизмов, причем наибольшая антимикробная активность наблюдалась в отношении штаммов S. aureus и Candida albicans. Степень проявления этой активности существенно зависела от состава среды и длительности культивирования планктомицета. Идентификация соединений, ответственных за наблюдаемую антимикробную активность Lacipirellula parvula PX69T, а также условий, инициирующих и стимулирующих их синтез, представляет предмет дальнейших исследований.
Список литературы
Boedeker C., Schüler M., Reintjes G., Jeske O., van Teeseling M.C., Jogler M., Rast P., Borchert D., Devos D.P., Kucklick M., Schaffer M., Kolter R., van Niftrik L., Engelmann S., Amann R., Rohde M., Engelhardt H., Jogler C. Determining the bacterial cell biology of Planctomycetes // Nat. Commun. 2017. V. 8. P. 14853. https://doi.org/10.1038/ncomms14853
Buckley D.H., Huangyutitham V., Nelson T.A., Rumberger A., Thies J.E. Diversity of Planctomycetes in soil in relation to soil history and environmental heterogeneity // Appl. Environ. Microbiol. 2006. V. 72. P. 4522–4531.
Cotter P.D., Ross R.P., Hill C. Bacteriocins – a viable alternative to antibiotics? // Nat. Rev. Microbiol. 2013. V. 11. P. 95–105.
Dedysh S.N., Ivanova A.A. Planctomycetes in boreal and subarctic wetlands: diversity patterns and potential ecological functions // FEMS Microbiol. Ecol. 2019. V. 95. fiy227. https://doi.org/10.1093/femsec/fiy227
Dedysh S.N., Kulichevskaya I.S., Beletsky A.V., Ivanova A.A., Rijpstra W.I.C., Sinninghe Damsté J.S., Mardanov A.V., Ravin N.V. Lacipirellula parvula gen. nov., sp. nov., representing a lineage of planctomycetes widespread in low-oxygen habitats, description of the family Lacipirellulaceae fam. nov. and proposal of the orders Pirellulales ord. nov., Gemmatales ord. nov. and Isosphaerales ord. nov. // Syst. Appl. Microbiol. 2020. V. 43. Art. 126050. https://doi.org/10.1016/j.syapm.2019.126050
Donadio S., Monciardini P., Sosio M. Polyketide synthases and nonribosomal peptide synthetases: the emerging view from bacterial genomics // Nat. Prod. Rep. 2007. V. 24. P. 1073–1109. https://doi.org/10.1039/b514050c
Fischbach M.A., Walsh C.T. Assembly-line enzymology for polyketide and nonribosomal peptide antibiotics: logic, machinery, and mechanisms // Chem. Reviews. 2006. V. 106. P. 3468–3496.
Fuerst J.A. The planctomycetes: emerging models for microbial ecology, evolution and cell biology // Microbiology (SGM). 1995. V. 141. P. 1493–1506.
Glöckner F.O., Kube M., Bauer M., Teeling H., Lombardot T., LudwigW., Gade D., Beck A., BorzymK., Heitmann K., Rabus R., Schlesner H., Amann R., Reinhardt R. Complete genome sequence of the marine planctomycete Pirellula sp. strain 1 // Proc. Natl. Acad. Sci. USA. 2003. V. 100. P. 8298–8303.
Graça A.P., Calisto R., Lage O.M. Planctomycetes as novel source of bioactive molecules // Front. Microbiol. 2016. V. 7. Art. 1241. https://doi.org/10.3389/fmicb.2016.01241
Guo M., Han X., Jin T., Zhou L., Yang J., Li Z., Chen J., Geng B., Zou Y., Wan D., Li D., Dai W., Wang H., Chen Y., Ni P., Fang C., Yang R. Genome sequences of three species in the family Planctomycetaceae // J. Bacteriol. 2012. V. 194. P. 3740–3741.
Jeske O., Jogler M., Petersen J., Sikorski J., Jogler C. From genome mining to phenotypic microarrays: Planctomycetes as source for novel bioactive molecules // Antonie van Leeuwenhoek. 2013. V. 104. P. 551–567. https://doi.org/10.1007/s10482-013-0007-1
Jeske O., Surup F., Ketteniß M., Rast P., Förster B., Jogler M., Wink J., Jogler C. Developing techniques for the utilization of Planctomycetes as producers of bioactive molecules // Front. Microbiol. 2016. V. 7. Art. 1242. https://doi.org/10.3389/fmicb.2016.01242
Ivanova A.A., Naumoff D.G., Miroshnikov K.K., Liesack W., Dedysh S.N. Comparative genomics of four Isosphaeraceae planctomycetes: a common pool of plasmids and glycoside hydrolase genes shared by Paludisphaera borealis PX4T, Isosphaera pallida IS1BT, Singulisphaera acidiphila DSM 18658T, and strain SH-PL62 // Front. Microbiol. 2017. V. 8. Art. 412. https://doi.org/10.3389/fmicb.2017.00412
Lage O.M., Bondoso J. Planctomycetes and macroalgae, a striking association // Front. Microbiol. 2014. V. 5. Art. 267. https://doi.org/10.3389/fmicb.2014.00267
Ravin N.V., Rakitin A.L., Ivanova A.A., Beletsky A.V., Kulichevskaya I.S., Mardanov A.V., Dedysh S.N. Genome analysis of Fimbriiglobus ruber SP5T, a planctomycete with confirmed chitinolytic capability // Appl. Environ. Microbiol. 2018. V. 84. Art. e02645-1.
Reintjes G., Arnosti C., Fuchs B.M., Amann R. An alternative polysaccharide uptake mechanism of marine bacteria// ISME J. 2017. V. 11. P. 1640–1650.
Schwarzer D., Finking R., Marahiel M.A. Nonribosomal peptides: from genes to products // Nat. Prod. Rep. 2003. V. 20. P. 275–287.
Staley J.T., Fuerst J.A., Giovannoni S., Schlesner H. The order Planctomycetales and the genera Planctomyces, Pirellula, Gemmata, and Isosphaera // The Prokaryotes: A Handbook on the Biology of Bacteria: Ecophysiology, Isolation, Identification, Applications / Eds. Balows A., Trüper H.G., Dworkin M., Harder W., Schleifer K.-H. New York, N.Y.: Springer New York, 1992. P. 3710–3731.
Tracanna V., de Jong A., Medema M.H., Kuipers O.P. Mining prokaryotes for antimicrobial compounds: from diversity to function // FEMS Microbiol. Rev. 2017. V. 41. P. 417–442.
Weber T., Blin K., Duddela S., Krug D., Kim H.U., Bruccoleri R., Lee S.Y., Fischbach M.A., Müler R., Wohlleben W., Breitling R., Takano E., Medema M.H. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters // Nucl. Acids Res. 2015. V. 43. W237–W243.
Wiegand S., Jogler M., Jogler C. On the maverick Planctomycetes // FEMS Microbiol. Rev. 2018. V. 42. P. 739–760.
Wiegand S., Jogler M., Boedeker C., Pinto D., Vollmers J., Rivas-Marín E., Kohn T., Peeters S.H., Heuer A., Rast P., Oberbeckmann S., Bunk B., Jeske O., Meyerdierks A., Storesund J.E., Kallscheuer N., Lücker S., Lage O.M., Pohl T., Merkel B.J., Hornburger P., Müller R.-W., Brümmer F., Labrenz M., Spormann A.M., Op den Camp H.J.M., Overmann J., Amann R., Jetten M.S.M., Mascher T., Medema M.H., Devos D.P., Kaster A.-K., Øvreås L., Rohde M., Galperin M.Y., Jogler C. Cultivation and functional characterization of 79 planctomycetes uncovers their unique biology // Nat. Microbiol. 2019. V. 5. P. 126–140. https://doi.org/10.1038/s41564-019-0588-1
Yadav S., Vaddavalli R., Siripuram S., Eedara R.V.V., Yadav S., Rabishankar O., Lodha T., Chintalapati S., Chintalapati V. Planctopirus hydrillae sp. nov., an antibiotic producing planctomycete isolated from the aquatic plant Hydrilla and its whole genome shotgun sequence analysis// J. Antibiotics. 2018. V. 71. P. 575–583.
Дополнительные материалы отсутствуют.
Инструменты
Микробиология


