Микробиология, 2021, T. 90, № 1, стр. 24-42
Бистабильность и формирование матрикса биопленки как механизмы адаптации Bacillus subtilis в стационарной фазе
М. Р. Шарипова a, *, А. М. Марданова a, Н. Л. Рудакова a, Д. С. Пудова a
a Казанский (Приволжский) федеральный университет
420008 Казань, Россия
* E-mail: marsharipova@gmail.com
Поступила в редакцию 25.02.2020
После доработки 01.07.2020
Принята к публикации 07.07.2020
Аннотация
Бациллы контролируют поведенческие реакции, такие как подвижность, формирование биопленок, биосинтез ферментов и метаболитов, дифференцировка и другие путем интеграции разнообразных экологических сигналов через сложную регуляторную сеть. Преобладающим образом жизни Bacillus subtilis в естественной среде является биопленка, будучи идеальной моделью для изучения молекулярной стратегии ее формирования. В обзоре систематизированы сведения об основных системах регуляции, ответственных за потерю подвижности и формирование биопленок B. subtilis, анализируется поведение бактерий внутри биопленочной популяции, приводящее к состоянию бистабильности и дифференцировке на разные типы субпопуляций, оценивается регуляторная взаимосвязь между системами контроля, ответственными за синтез структурных компонентов матрикса биопленки. Особый акцент сделан на данных о сигнальных механизмах, запускающих образование биопленки и ее дисперсию. Представлены обобщенные сведения о последних открытиях в этой области и их интеграции в общее представление об этих сложных микробных сообществах.
ВВЕДЕНИЕ
В стационарной фазе бациллы способны к формированию разных адаптационных ответов, сопровождающихся изменениями в регуляции экспрессии множества генов, направленных на выживание. В естественной среде биопленки являются преобладающим образом жизни бактерий, образование которых в лимитированных условиях представляет сложный и строго регулируемый процесс. Способность синтезировать биопленки является частью жизненного цикла большинства микроорганизмов и успешной стратегией защиты бактерий от неблагоприятных факторов окружающей среды, поэтому молекулярные основы формирования биопленок остаются темой возрастающего интереса в микробиологии. В настоящее время используют различные бактериальные модели для выяснения молекулярных механизмов образования и функционирования биопленок. Для этой цели идеальной моделью считают биопленки обитающих в почве бактерий Bacillus subtilis, которые характеризуются уникальными архитектурными особенностями как следствие сложных программ клеточной специализации и межклеточной коммуникации в сообществе (Ryan-Payseur, Freitag, 2018; Kovacs, Dragos, 2019). Эти исследования нацелены на выяснение эволюции, биологической роли, морфологических особенностей и структуры биопленок, а также молекулярно-генетических механизмов, лежащих в основе клеточной дифференцировки внутри микробного сообщества. Особый интерес представляет регуляторная и метаболическая взаимосвязь между процессом образования биопленки у бацилл и другими клеточными событиями стационарной фазы, такими как споруляция, подвижность, секреция вторичных метаболитов и белков, включая липопептиды и внеклеточные ферменты, в том числе протеиназы (Актуганов и соавт., 2019; Pisithkul et al., 2019). Установлено, что биопленка B. subtilis содержит протеиназо-продуцирующие клетки и их количество возрастает по мере развития биопленки (Kobayashi, Ikemoto, 2019). Кроме того, гены, продукты которых участвуют в формировании биопленок, регуляторно взаимосвязаны с синтезом и секрецией различных внеклеточных метаболитов и сигнальных молекул для кооперативного взаимодействия в микробном сообществе (Martin et al., 2020). На основе новых знаний разрабатывается платформа для создания искусственных биопленок методами 3D-печати для целевого практического использования (Huang et al., 2018; Balasubramanian et al., 2019). Мы делаем акцент на обобщении новых данных, полученных для формирования, развития и рассеивания биопленок B. subtilis.
Целью работы является систематизация данных о регуляции образования и дисперсии биопленки B. subtilis во взаимосвязи с другими физиологическими процессами стационарной фазы у этих бактерий.
СТРУКТУРА БИОПЛЕНКИ
B. subtilis являются подвижными грамположительными почвенными бактериями, способными формировать устойчивые биопленки, в том числе, в симбиозе с корнями растений. Образование биопленки является одним из ответов клеток B. subtilis на неблагоприятные факторы среды, в результате формируется структурированное многоклеточное сообщество бактерий, в котором клетки сцеплены друг с другом и заключены в полимерный матрикс собственного производства (Hobley et al., 2015). Матрикс включает экзополисахариды (EPS), белки, нуклеиновые кислоты и содержит каналы, через которые транспортируются питательные вещества и кислород и выводятся продукты бактериального метаболизма (Hobley et al., 2015). Матрикс обеспечивает стабильность биопленки, защиту содержащихся в нем бактерий от бактериофагов, неблагоприятных воздействий (осмотический шок, УФ-облучение, высыхание, изменение pH среды и др.) и антибактериальных препаратов (Стрелкова и соавт., 2013; Dragos et al., 2017; Vidakovic et al., 2018; Пономарева и соавт., 2018). Описан механизм “взаимопомощи” между клетками бактерий, способными и неспособными формировать матрикс, последние получают выгоду от кооперативного взаимодействия с клетками, синтезирующими экзополисахариды (Martin et al., 2020). Тесное связывание клеток бактерий через нанотрубки для совместного использования метаболитов и передачу сигналов на большие расстояния для соединения метаболических процессов направлено на создание единого микробного сообщества с рациональным распределением внеклеточных “общественных благ”. Отношения между бактериями основаны на активации сложных механизмов межклеточной коммуникации, в этих процессах решающую роль играют системы глобальной регуляции метаболизма: “кворум-сенсинг” и циклического ди-ГМФ, вследствие чего активируются процессы секреции ферментов и вторичных метаболитов, сидерофоров и противомикробных соединений (Bareia et al., 2018; Kalamara et al., 2018; Fu et al., 2019; Kovacs, Dragos, 2019). Биопленка является основой для метаболической кооперации клеток в сообществе, а также создает условия, благоприятные для симбиотических взаимоотношений между бактериями и макроорганизмами, например, между почвенными бациллами и корнями растений (Townsley et al., 2018).
Природные биопленки имеют сложную структуру и состоят из множества видов микроорганизмов, которые взаимодействуют между собой, либо конкурируют за питательные вещества и пространство для жизнедеятельности. Поскольку природные биопленки являются смесью видов, что затрудняет исследования, их часто проводят на биопленках одного вида в лабораторных условиях, которые можно контролировать. Наиболее распространенный способ получения биопленок в лабораториях – это формирование ассоциаций микроорганизмов на погруженных твердых поверхностях, которые визуализируют с помощью красителя кристаллического фиолетового, другой способ – формирование биопленки на границе раздела воздух–жидкость (Vlamakis et al., 2013; Hollenbeck et al., 2016). Плакунов и соавт. разработали альтернативный планшетному универсальный метод одновременного анализа планктонной и биопленочной культур для характеристики роста биопленки, синтеза внеклеточного матрикса, метаболической активности и минимального числа жизнеспособных клеток в биопленке на основе использования политетрафторэтилена, фильтров из стекловолокна в качестве поверхности для формирования биопленок (Плакунов и соавт., 2016). Метод пригоден для грамотрицательных и грамположительных бактерий. Кроме того, в настоящее время колонии, которые растут на поверхности чашек с агаром, также рассматривают как форму биопленки, поскольку они представляют собой устойчивые микробные сообщества клеток во внеклеточном матриксе (Branda et al., 2001; Gallegos-Monterrosa et al., 2016).
Отличительным признаком биопленок B. subtilis является множество архитектурных особенностей; их сложность обусловлена высокой степенью клеточной специализации и характером межклеточных коммуникаций субпопуляций, которые в совокупности образуют сообщество, по строению аналогичное многоклеточным организмам (Schafer, Turgay, 2019). Для формирования биопленки клетки B. subtilis переходят из планктонного в неподвижное состояние, при этом подавляется экспрессия жгутиковых генов и индуцируется экспрессия генов, необходимых для синтеза внеклеточного матрикса, обусловленных внешними сигналами (поверхностное сцепление, истощение питательных веществ, низкий уровень кислорода и др.) (Gingichashvili et al., 2019). Первоначально клетки бацилл представляют собой короткие подвижные палочки, которые по мере развития биопленки прилипают друг к другу и к поверхности и образуют длинные цепочки из неподвижных клеток, выделяющих внеклеточный матрикс. Матрикс биопленки B. subtilis характеризуется гидрофобностью и является источником механической жесткости (Dragos, Kovacs, 2017). Разработана инновационная технология оптической визуализации для пространственного и временнóго слежения за динамикой развития биопленки B. subtilis в различных условиях для определения неровностей и толщины биопленки, распределения в ней клеток с различным фенотипом (подвижность, продукция матрикса и споруляция) с применением метода меченой тройной флуоресценции (Wang et al., 2017).
Обширные складки или морщины на поверхности биопленки бацилл рассматривают как результат механической нестабильности в связи с передвижением клеток внутри биопленки или локализованной гибели клеток. Преимущество в образовании складок приводит к увеличению отношения поверхности биопленки к ее объему для обеспечения лучшего доступа клеток к кислороду (Asally et al., 2012; Trejo et al., 2013; Kolodkin-Gal et al., 2013). Морщины способствуют формированию сложной сети каналов внутри биопленки, облегчая циркуляцию жидкости и распределение питательных веществ в тех частях биопленки, которые недоступны диффузионным процессам; каналы внутри биопленки взаимосвязаны и их морфология и количество зависит от возраста биопленки и условий питания бактерий (Gingichashvili et al., 2019). Установлено, что каналы выполняют важную функциональную роль в транспорте молекул через структуры биопленки, что позволяет жидкости эффективно проходить в более глубокие слои периферии зрелой биопленки (Gingichashvili et al., 2019). На поверхности биопленки B. subtilis идентифицированы воздушные домены, представляющие участки для споруляции и распространения бактериальных спор (Branda et al., 2001). Такая трехмерная архитектура обеспечивает целостность биопленки B. subtilis и важна для адаптации и выживания микробных клеток.
РЕГУЛЯЦИЯ СИНТЕЗА МАТРИКСА БИОПЛЕНКИ
Гены, ответственные за образование экзополисахаридов матрикса, входят в состав eps-оперона (Nagorska et al., 2010; Marvasi et al., 2010; Roux et al., 2015). Дефектные по экзополисахаридам eps-мутанты бацилл образуют плоские и чрезвычайно хрупкие колонии, способные расти и формировать цепочки из клеток, частично содержащих внеклеточный материал благодаря синтезу дополнительных компонентов матрикса белковой природы (Branda et al., 2006). Мутации в генах ферментов, участвующих в биосинтезе матрикса, pgcA (ранее yhxB), кодирующий α-фосфоглюкомутазу, и gtaB, кодирующий УТФ-глюкозо-1-фосфат-уридилтрансферазу, приводят к нарушениям в образовании биопленки (Lazarevic et al., 2005). Мутанты, дефектные по синтезу уридин-дифосфат-галактозы (UDP-Gal), который в качестве предшественника необходим для биосинтеза экзополисахарида, не формируют биопленку (Chai et al., 2012). UDP-Gal является токсичным промежуточным продуктом в метаболизме галактозы, который преобразуется в нетоксичную UDP-глюкозу с помощью УДФ-глюкоза-4-эпимеразы (GalE); при мутации в гене galE, в присутствии галактозы в результате накопления UDP-Gal прекращается рост клеток. Наиболее изученным продуктом генов eps-оперона является белок EpsE. Это бифункциональный белок, который участвует в контроле биосинтеза компонентов EPS. Белок EpsE ингибирует вращение жгутиков, взаимодействуя с белком ротора жгутиков, FliG, и обладает активностью гликозилтрансферазы, которая необходима для биосинтеза EPS (Guttenplan, Kearns, 2013). При этом ингибирование движения происходит независимо от гликозилтрансферазной активности белка EpsE. Такой механизм регуляции способствует активному формированию матрикса биопленки при выключении подвижности клеток. У некоторых штаммов B. subtilis активно синтезируется и способствует усилению образования полимерного матрикса другой внеклеточный полимер, гамма-полиглутаминовая кислота (PGA) (Morikawa et al., 2006; Wu et al., 2008). Спектр экзополисахаридов В. subtilis, которые входят в матрикс биопленки, варьирует в зависимости от условий роста (Roux et al., 2015). Когда штамм B. subtilis NCIB3610 выращивали в среде, содержащей глутаминовую кислоту и глицерин, в углеводной биомассе присутствовали такие моносахариды как галактоза, глюкоза и N-ацетилгалактоза (Ga-lNAc); распространенность каждого сахара зависела от целостности генов eps-оперона (Chai et al., 2012). В соответствии с этими данными, гены, участвующие в метаболизме галактозы, важны для формирования биопленки.
Анализ полисахаридов штамма В. subtilis NCIB3610, выращенного в бульоне TY (LB с добавлением сульфата марганца и сульфата магния), выявил манноза-доминирующий профиль моносахаридов, зависимый от eps-оперона (Jones et al., 2014). По-видимому, природа полисахарида, продуцируемого компонентами eps-оперона, зависит от доступных субстратов. В дополнение к экзополисахаридам, синтезированным под контролем eps-оперона, штаммы B. subtilis для образования матрикса синтезируют экзогенный полисахарид леван (гомополимер фруктозы), биосинтез которого зависит от левансахаразы, кодируемой геном sacB (Benigar et al., 2014; Raga-Carbajal et al., 2016). При росте бактерий в присутствии в среде сахарозы леван включается в матрикс биопленки, что частично компенсирует отсутствие продуктов eps-оперона (Dogsa et al., 2013). Включение бактериями левана в матрикс биопленки, при условии, что сахароза продуцируется растениями, может быть актуальным для формирования биопленки B. subtilis в ризосфере в естественной среде.
Целостность структуры биопленки обуслoвливают 2 секретируемых белка B. subtilis, TasA и Ta-pA, оба кодируются генным кластером, состоящим из трех генов tapA–sipW–tasA (tapA-оперон) (Branda et al., 2006). Гомологи TasA широко распространены среди бацилл (Dragos, 2017). Белок TasA рассматривался как функциональный амилоидоподобный белок, который синтезируется как мономер, собирается в длинные волокна (фибриллы) и транслоцируется через мембрану с участием сигнальной пептидазы SipW; снаружи с помощью белка-помощника TapA волокна прикрепляются к клеточной стенке (Romero et al., 2014). Белок TasA является функциональным гомологом амилоидных белков курли E. coli, он полимеризуется in vitro и способен взаимодействовать с антителами, специфичными для амилоидных полимеров (Huang et al., 2018). Очищенный белок TasA находится в растворе в олигомерном состоянии, волокнообразование из олигомеров TasA стимулируется в кислой среде, либо в присутствии гидрофобной поверхности (Chai et al., 2013). Изучение in vitro различных олигомерных форм секретируемого белка TasA с помощью ЯМР, ЭМ, рентгеновской дифракции и аналитического ультрацентрифугирования показало конформационные изменения TasA от глобулярного состояния до протеиназо-устойчивой фибриллярной структуры, что не наблюдается для других биопленкообразующих белков (Diehl et al., 2018). На основе метода твердотельного ЯМР установлено, что основной амилоидный компонент TasA содержит β-листы и α-спирали во вторичной структуре, что свидетельствует о нетипичной амилоидной архитектуре у B. subtilis (Mammeri et al., 2019).
Мутанты по гену tasA биопленку не образуют, но этот дефект не такой существенный, как у мутантов по eps-генному кластеру (Branda et al., 2006). tasA-Мутанты продуцируют клеточные цепи, которые не способны удерживаться вместе, инактивация гена tasA также связана с отсутствием формирования морщинистой биопленки. Белок TasA участвует в упорядоченном созревании биопленки Bacillus. Его отсутствие приводит к ряду физиологических изменений, вызванных изменениями стабильности и динамики мембран, снижает жизнеспособность клеток при взаимодействии за счет отсутствия структурированного матрикса, что, в свою очередь, ограничивает возможности адаптации клеток к стрессам (Camara-Almiron et al., 2020). TasA проявляет антимикробные свойства; экспрессия tasA в биопленках B. subtilis усиливается в присутствии патогена Fusarium culmorum (Khezri et al., 2016). Интересно, что TasA также играет важную роль в распознавании родственных связей между близкородственными штаммами B. subtilis, что опосредуется различными ингибирующими молекулами (Lyons et al., 2016). Наконец, tаsА-мутантные штаммы дефектны в формировании связанных с корнем биопленок (Dragos et al., 2017). Поскольку ризосфера растений представляет естественную среду обитания B. subtilis, TasA может быть одним из ключевых секретируемых соединений, определяющих экологию и эволюцию этого вида.
Белок TapA образует минорный компонент волокон из TasA белка, связывается с TasA и участвует в прикреплении волокон TasA к клеточной стенке, играет важную роль в его локализации, а также в полимеризации и сборке волокон TasA из мономеров (Romero et al., 2014). В отсутствие гена tapA не образуются TasA-волокна дикого типа, и уменьшается общий уровень TasA-белка в экзогенном матриксе. Данные свидетельствуют, что белок TapA обеспечивает стабилизацию белка Ta-sA, в связи с чем фибриллярная форма белка TasA более устойчива к расщеплению протеиназами, по сравнению с олигомерной формой. Согласно этим выводам, делеция гена tapA также является критичной для образования биопленок B. subtilis.
Сигнальная пептидаза SipW расщепляет предшественник белка TapA, распознавая N-концевую сигнальную последовательность, чтобы белок высвобождался из мембраны и формировал фибриллярные волокна, связанные с клеточной стенкой (Stover, Driks, 1999). Мутации в гене sipW у штаммов B. subtilis приводили к нарушениям в прикреплении к поливинилхлоридным или стеклянным поверхностям в условиях лаборатории (Hamon et al., 2004). Это связано с тем, что пептидаза SipW является бифункциональным белком, которая работает как сигнальная пептидаза в процессинге белков TasA и TapA и функционирует для активации экспрессии eps-оперона при участии С-концевого домена белка. Такая активация необходима для инициации образования матрикса биопленки в результате прикрепления клеток к поверхности.
На заключительных этапах созревания биопленки синтезируется бактериальный гидрофобин BslA, который работает вместе с белком TasA (Hobley et al., 2015). Белок BslA (ранее YuaB) необходим для поверхностной гидрофобности биопленки B. subtilis и формирования ее сложной морфологии и архитектуры (Arnaouteli et al., 2017). Биопленка B. subtilis с дефектным фенотипом bslA– обладает амфифильными свойствами и напоминает мутанты с делецией генов eps и tasA (Ostrowski et al., 2011). Белок BslA является поверхностно-активным и состоит из двух доменов – иммуноглобулин-подобного домена и уникального высоко гидрофобного домена (Hobley et al., 2015). Белок BslA имеет две структурные конформации, которым соответствуют две функциональные формы: мономерный BslA необходим для создания сложной структуры биопленки, тогда как для гидрофобности поверхности биопленки требуется димерный белок (Arnaouteli et al., 2017). Димеризация BslA опосредуется образованием дисульфидной связи и зависит от двух консервативных остатков цистеина, расположенных в С-концевой области молекулы белка. Конформационный переход регулируется окислительно-восстановительным статусом среды: в нативной биопленке доступность акцептора электронов влияет на олигомеризацию BslA, и в обогащенной кислородом области с открытой поверхностью преобладающей формой является димер.
Таким образом, все компоненты матрикса биопленки, экзополисахариды (EPS, PGA) и белки (TasA, ТасА и BslA), необходимы для формирования внеклеточного матрикса B. subtilis и влияют на рост и конечные размеры биопленки. На основе методов интервальной микроскопии и световой профилометрии установлено, что два матричных компонента, экзополисахарид, продуцируемый геном epsA-O оперона, и белок поверхностного слоя BslA контролируют область покрытия биопленкой, причем высота зрелой биопленки зависит от белка BslA (Kesel et al., 2017). Взаимодействия между компонентами матрикса в биопленках ответственны за создание адаптируемой структуры в процессе развития биопленки. Показано, что на ранних этапах развития биопленки при формировании ее 3D-структуры с компонентами матрикса взаимодействует экзогенная ДНК, которая также вносит определенный вклад в созревание биопленки B. subtilis (Peng et al., 2020).
БИСТАБИЛЬНОСТЬ БИОПЛЕНКИ
Формирование и развитие биопленки, синтез компонентов внеклеточного матрикса строго регулируются на уровне транскрипции, посттранскрипционных и посттрансляционных модификаций (Reverdy et al., 2018). В планктонной популяции все клетки подвижны, экспрессия генов, необходимых для подвижности, обеспечивается, когда транскрипционные регуляторы Spo0A, DegU, ComA находятся в нефосфорилированном состоянии; их активация в ответ на присутствие разнообразных внеклеточных сигналов приводит к уменьшению популяции подвижных клеток (Vlamakis et al., 2013). В процессе созревания биопленки клетки бацилл дифференцируются на несколько типов, образуя субпопуляции генетически идентичных, но по фенотипу отличных клеток, которые реагируют на различные сигналы и выполняют разные функции для всего сообщества, чтобы минимизировать затраты энергии, например, предотвращая тотальный переход клеток в энергоемкий процесс споруляции (Lopez, Kolter, 2010). За активацию специфических генных каскадов, которые приводят к дифференцировке всех сосуществующих типов клеток в биопленках B. subtilis, ответственна сложная регуляторная сеть.
Впервые дифференциация клеток в биопленке установлена путем микроскопического исследования, в результате которого в воздушных доменах биопленки обнаружен специфичный для споруляции репортер (Branda et al., 2001). Впоследствии эти данные подтвердили эксперименты, полученные с помощью фьюжн-векторов с флуоресцентными репортерами (Veening et al., 2006). Внутри биопленки идентифицированы клетки, специализирующиеся на подвижности, формировании полимерного матрикса, споруляции, секреции экзопротеиназ. С помощью проточной цитометрии и флуоресцентной микроскопии установлено, что количество клеток, продуцирующих экзопротеиназы, увеличивается по мере созревания биопленки и обусловлено увеличением транскрипции генов экзопротеиназ, а также увеличением активности экзопротеиназ во времени (Marlow et al., 2014a). Протеиназы синтезируются клетками, продуцирующими матрикс, так и клетками, не продуцирующими матрикс, что указывает на значимость этих ферментов на разных стадиях развития биопленки. Данные этих экспериментов увеличили разнообразие типов специализированных клеток биопленки и свидетельствовали о регуляторной пластичности сообщества.
Активация Spo0A-транскрипционного фактора путем фосфорилирования запускает образование спор только в определенной субпопуляции клеток B. subtilis (Fujita et al., 2005). Существует два различных уровня активации транскрипционного фактора Spo0A-P: низкий уровень активации (клетки Spo0A-ON) приводит к образованию матрикса, а более высокий уровень – к споруляции (рис. 1б). Клетки Spo0A-ON становятся продуцентами матрикса, они теряют подвижность путем активации сцепления (белок EpsE) (Guttenplan, Kearns, 2013). Кроме того, клетки Spo0A-ON являются каннибалами, они невосприимчивы к действию двух самосекретируемых пептидных токсинов (пептиды Skf и Sdp), которые убивают их чувствительных к токсинам собратьев (Höfler et al., 2016). Этот процесс рассматривают как каннибализм: когда питательных веществ недостаточно, мертвые клетки служат пищей для оставшихся клеток популяции и задержки процесса спорообразования; этот процесс опосредуют секретируемые протеиназы (Kobayashi, 2019).
Рис. 1.
Регуляторные пути, контролирующие формирование биопленки у B. subtilis: а – схема действия SinR репрессора; б – механизм образования биопленки B. subtilis под контролем транскрипционного регулятора Spo0A-P; в – схема участия транскрипционных факторов DegU-P и ComA-P в контроле образования биопленки B. subtilis. Т-образная прямая обозначает ингибирование, а стрелками указаны пути активации. Описание схемы в тексте.
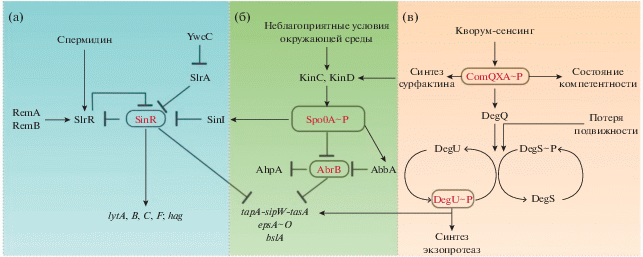
Активация фактора ComA (ComA-P) приводит к дифференцировке и образованию в сообществе субфракции компетентных клеток, которые способны поглощать экзогенную ДНК, чтобы увеличить генетическую изменчивость в популяции. Активация ComA-P регулируется механизмом кворум-сенсинга и запускается при достижении определенной плотности клеток (Zeriouh et al., 2014). Кроме того, клетки ComA-P экспрессируют паракринный сигнал сурфактин, который запускает экспрессию генов и оперонов для образования матрикса (рис. 1в) (Aleti et al., 2016). Активность пары регуляторов ответа ComA/ComP модулируется сигнальным пептидом ComX, который контролирует в качестве автоиндуцирующего сигнала процессы компетентности, продукции сурфактанта и, косвенно, синтез внеклеточных протеиназ в B. subtilis (Špacapan et al., 2018). Эти процессы регулируются в соответствии с размером бактериальной популяции, классифицируя ComX как сигнал кворум-сенсинга, контролирующий продукцию экзопротеиназ через промежуточный регулятор транскрипции DegQ.
Активация транскрипционного фактора DegU (DegU-P) приводит к образованию в сообществе субпопуляции клеток, называемых “майнеры”, которые специализируются на секреции внеклеточных протеиназ (рис. 1в) (Veening et al., 2008). Продукты этих клеток способствуют расщеплению крупных биополимеров на более мелкие олигопептиды. Показано, что пептид ComX индуцирует экспрессию гена субтилизина aprE и стимулирует секрецию экзопротеиназ в биопленках B. subtilis. В свою очередь, экзопротеиназы способны разрушать аутоиндуцирующий сигнальный пептид ComX. Снижение биологической активности ComX может быть восстановлено добавлением субтилизина, ингибирование металлопротеиназ ЭДТА снижает биологическую активность ComX, указывая на то, что сериновые и металлопротеиназы способны расщеплять пептид ComX (Špacapan et al., 2018). Эти данные свидетельствуют, что секретируемые протеолитические ферменты выполняют регуляторные функции и разрушают сигнальный аутоиндуцируемый пептид ComX, который является важным детерминантом их экспрессии в биопленках. Субпопуляция клеток с активными экзопротеиназами отличается от подвижных клеток или клеток, продуцирующих матрикс, и расположена ближе к поверхности биопленки (Marlow et al., 2014b). Это согласуется с фактом, что клетки DegU-ON активно экспрессируют водоотталкивающий белок BslA, который необходим для сохранения целостности биопленки B. subtilis (Hobley et al., 2015; Arnaouteli et al., 2017).
Анализ пространственной локализации отдельных субпопуляций в зрелых биопленках, проведенный с помощью флуоресцентных репортеров, показал, что подвижные клетки в нижнем слое биопленки способствуют ее пространственному расширению, а клетки, локализованные в ядре биопленки, специализируются на продукции внеклеточного матрикса и поддерживают его жесткость (Vlamakis et al., 2013). Разработаны новые методы визуализации внутренней структуры биопленки B. subtilis с сохранением архитектуры и ультраструктуры (Fuchs et al., 2018). Споры присутствуют в верхней части биопленки на воздушных структурах, которые облегчают их рассеивание. Пространственно-временное распределение компетентных клеток и клеток-продуцентов сурфактина как отдельные субпопуляции сложно выделить в общем микробном сообществе. Бистабильность внутри биопленки является способом адаптации всего сообщества к переменчивым условиям окружающей среды, когда генетически идентичная популяция способна образовывать отдельные субпопуляции с различными физиологическими свойствами, чтобы обеспечить необходимыми метаболитами все сообщество клеток. Эту стратегию назвали стратегией хеджирования роста (Veening et al., 2008).
РЕГУЛЯТОРНЫЙ ПУТЬ ФОРМИРОВАНИЯ БИОПЛЕНКИ
Генетическая схема регуляции образования биопленок в B. subtilis изучена лучше, чем передача сигналов, которые запускают этот процесс (Mielich-Süss, Lopez, 2015). Роль центрального регулятора транскрипции в клетках стационарной фазы у B. subtilis выполняет фактор Spo0A, который контролирует экспрессию более ста генов, необходимых для споруляции и формирования биопленки (рис. 1б) (Chastanet et al., 2011). Активность этого белка регулируется фосфорилированием: внеклеточные сигналы активируют сенсорные гистидинкиназы, которые запускают фосфореле, что приводит к фосфорилированию Spo0A. Содержание фосфорилированного Spo0A в клетках определяет профиль экспрессии генов (Schultz et al., 2016). Когда Spo0A фосфорилирируется, происходит индукция образования биопленки, по мере созревания которой регулятор Spo0A-P накапливается в определенных клетках и активирует в них споробразование. При пороговых уровнях фосфорилирования Spo0A запускаются два пути антирепрессии, чтобы активировать транскрипцию оперонов, критичных для синтеза матрикса биопленки.
Первый путь нацелен на подавление репрессора AbrB, который блокирует транскрипцию генов, участвующих во многих процессах, включая формирование биопленки (Chumsakul et al., 2011). Установлено, что от регулятора переходного состояния AbrB и фактора транскрипции Spo0A зависит экспрессия гена ahpA, кодирующего пероксидазу AhpA, которая специфически экспрессируется во время формировании биопленки, но не экспрессируется в стационарной фазе или при споруляции. Это позволило предположить, что для дерепрессии гена пероксидазы ahpA под действием регулятора AbrB требуется сигнал, отличающийся от сигналов при переходе клеток в стационарную фазу (рис. 1б) (Zwick et al., 2017). Регулятор AbrB контролируется посредством фактора Spo0A двумя различными путями: 1) Spo0A-P репрессирует транскрипцию гена abrB и 2) Spo0A-P способствует экспрессии фактора AbbA, AbrB-антирепрессора, который связывает AbrB и изолирует его от взаимодействия с целевой ДНК (Tucker et al., 2014).
Второй путь связан с небольшим ДНК-связывающим белком SinR, который ингибирует формирование биопленки и является центральным для определения судьбы клеток (рис. 1а) (Kampf et al., 2018). Белок SinR образует тетрамеры и связывается с сайтами в промоторных областях оперонов epsA-O и tapA–sipW–tasA, необходимых для синтеза внеклеточного полисахарида и амилоидных волокон во внеклеточном матриксе соответственно (Cairns et al., 2014; Stowe et al., 2014). Активность SinR контролируется антагонистическими белками, SinI и SlrR. Связывание SinI или SlrR с SinR ингибирует ДНК-связывающую способность последнего и освобождает опероны биопленки от репрессии (рис. 1а) (Newman et al., 2013). Более того, небольшие изменения в уровнях экспрессии белка SinR оказывают существенное влияние на экспрессию генов матрикса, главным образом, из-за сверхчувствительности регуляторных модулей SinI–SinR и SlrR–SinR, что указывает на строгую регуляцию их образования, в том числе метаболическую (Chai et al., 2011; Greenwich et al., 2019).
Один из них, антирепрессорный белок SinI, активируется после фосфорилирования Spo0A и связывается c SinR, образуя гетеродимерный комплекс, изолирует SinR от ДНК-мишеней, что приводит к экспрессии SinR-репрессированных генов (рис. 1а) (Newman et al., 2013; Stowe et al., 2014). Фенотипический переход из планктонного в биопленочное состояние регулируется активностью репрессора транскрипции SinR и его инактивацией первичным антагонистом, SinI. Установлен механизм разрушения тетрамера SinR с помощью белка SinI, который регулирует экспрессию SinR-репрессируемых генов (Milton et al., 2020). Если уровни SinR превышают уровни его антагонистов, то экспрессия генов матрикса подавлена, в то же время показано, что SinR экспрессируется в большинстве клеток, а SinI – только частью популяции. Функции белков SinI и SinR чувствительны к дозе гена: удвоение копий генов sinI и sinR приводило к подавлению синтеза полимеров матрикса (Chai et al., 2011; Ogura et al., 2016). В совокупности за счет сродства Spo0A-P к промотору гена sinI и числа копий генов sinI и sinR экспрессия генов матрикса является временной, и спорулирующие клетки не расходуют энергию на продукцию внеклеточного матрикса.
Другой антирепрессор SlrR белка SinR необходим для контроля образования биопленки двумя способами (Chai et al., 2010). Во-первых, он связывается с репрессором SinR с образованием комплекса SinR–SlrR, предотвращая связывание SinR с промоторами генов матрикса (рис. 1а). Так как регулятор SinR репрессирует ген антирепрессора slrR, который, в свою очередь, связывает регулятор SinR, это приводит к регуляции комплекса SinR–SlrR по механизму двойной петли отрицательной обратной связи. В результате ген slrR остается дерепрессированным при условии, что SlrR предотвращает репрессию со стороны регуляторного белка SinR. Когда уровень SlrR-белка в клетках высокий, происходит экспрессия генов оперонов матрикса, потому что в этих условиях уровень свободного репрессора SinR низкий. Когда уровень SlrR-белка в клетках низкий, регулятор SinR не ингибируется и блокирует опероны для синтеза матрикса биопленки. Во-вторых, комплекс SinR-SlrR участвует в контроле экспрессии гена hag, кодирующего флагеллин, необходимый для подвижности, а также генов аутолизинов, кодирующих белки для разделения клеток (гены lytABC и lytF) (рис. 1а). Идентифицирован новый регуляторный механизм взаимодействия SinR с промотором гена degU, после чего репрессор SlrR образует комплекс с SinR на промоторе degU. Таким образом, SlrR по-видимому, играет активную роль в экспрессии degU (Ogura et al., 2014).
Сцепление клеток имеет большое значение в начале формирования биопленки у B. subtilis. Регуляторный белок SlrR нестабилен и относится к суперсемейству LexA-пептидаз, в клетках он подвергается расщеплению, в том числе с помощью протеиназы ClpCP. Нестабильность белка SlrR приводит к дерепрессии генов, кодирующих аутолизины, активация которых приводит к разделению цепей на отдельные клетки (Chai et al., 2010). Таким образом, переключатель SlrR–SinR может существовать в двух состояниях: при низком уровне SlrR, что соответствует одиночным подвижным клеткам, и при высоком SlrR, что соответствует цепочкам матрикс-продуцирующих клеток. Переключение с низкого в высокое состояние достигается с помощью белка SinI, который синтезируется под контролем фактора Spo0A-P в ответ на активирующиеся гистидинкиназы. Накопление белка SinI ингибирует репрессор SinR, приводя к депрессии гена для продукции белка SlrR. Это приводит к накоплению SlrR, приводя переключатель SlrR–SinR в состояние высокого уровня SlrR, благоприятное для формирования биопленки.
Сложные взаимодействия между SinR и его родственными антагонистами приводят к бистабильности. На эти взаимодействия влияет фосфодиэстераза YmdB, которая участвует в контроле активности репрессора SinR: клетки, дефектные по гену ymdB, характеризуются гиперактивностью SinR и неспособны формировать биопленки (Kampf et al., 2018). У ymdB-мутанта большинство клеток экспрессируют гены, необходимые для подвижности и хемотаксиса. Супрессорный анализ показал, что мутанты ymdB легко приобретают мутации, влияющие на SinR, восстанавливая образование биопленки (Kampf et al., 2018). Эти данные свидетельствуют, что фосфодиэстераза YmdB необходима для гомеостаза SinR и/или его антагонистов. Показано, что на посттранскрипционном уровне транскрипты sinl–sinR и sinR контролируются путем их деградации с помощью РНКазы Y-содержащих белковых комплексов (DeLoughery et al., 2016). Важную роль в регуляции активности белков в клетках бацилл выполняют АТФ-зависимые протеиназы. Результаты глобального протеомного анализа и вестерн-иммуноблоттинга показали, что уровни SinR снижаются у мутанта с дефектом по высококонсервативной ClpYQ-протеиназе с изменениями в регуляции образования биопленки (Yu et al., 2018).
Для формирования биопленки также необходимы регуляторные белки RemA и RemB (рис. 1а) (Cairns et al., 2014). Регулятор RemA является ДНК-связывающим белком, который активирует транскрипцию eps- и tapA-оперонов, а также контролирует транскрипцию гена антирепрессора slrR. Установлено, что для регуляции экспрессии гена slrR требуются оба регуляторных белка, R-emA и RemB (Winkelman et al., 2013). Дополнительно, по результатам генетического анализа регуляторы RemA и RemB контролируют экспрессию оперонов матрикса способом, независимым от антирепрессора SlrR и действуют параллельно с другими регуляторами (DegU AbrB и SinR). Re-mA связывается с ДНК выше промотора eps-оперона и перекрывается с сайтом для взаимодействия с репрессором SinR, в этом случае белок SinR препятствует связыванию белка RemA с промотором (рис. 1а) (Winkelman et al., 2013). Аналогично, RemA связывается выше промотора tapA-оперона, но в этом случае SinR и RemA способны одновременно связываться с ДНК. Эти данные указывают на дополнительный путь для контроля экспрессии генов матрикса биопленки. На хромосомной ДНК B. subtilis ген remA расположен рядом с генами, продукты которых связаны со строгим ответом; этот факт может указывать на регуляторную взаимосвязь между экспрессией гена remA и метаболическим статусом клетки (Winkelman et al., 2013).
Установлено, что компоненты эпигенетического переключателя могут подвергаться дополнительной регуляции с участием регуляторных белков YwcC и SlrA, из которых белок SlrA является одним из антирепрессоров регулятора SinR, а белок YwcC является репрессором транскрипции TetR-типа и репрессирует ген slrA (рис. 1а) (Chai et al., 2009). Когда регулятор YwcC отвечает на неустановленный сигнал и его экспрессия подавляется, гены матрикса биопленки индуцируются SlrA-опосредованной инактивацией репрессора SinR. В отличие от SinI, белок SlrA продуцируется почти во всех клетках. В этом случае такой путь регуляции посредством YwcC–SlrA обеспечивает клеткам B. subtilis быструю реакцию для защиты бактериального сообщества на стрессовые изменения среды путем образования биопленки.
Регуляция экспрессии генов bslA и pgs у B. subtilis осуществляется системой трансдукции сигнала DegS–DegU, в которой DegU является транскрипционным регулятором. Он фосфорилируется родственной гистидинкиназой DegS и управляет многими клеточными процессами: плавательной и роящей моторикой, формированием биопленки, синтезом экзопротеиназ, производством гамма-полиглутаминовой кислоты (PGA) и споруляцией (Gabdrakhmanova et al., 2005; Каюмов и соавт., 2006; Sharipova et al., 2008; Cheremin et al., 2014). Установлено, что мутант по degU не способен к формированию погруженной биопленки, для которой требуется полимер PGA, продукт pgs-оперона (Stanley et al., 2005). Образование биопленки у мутанта degU нарушается из-за потери гидрофобности матрикса за счет нарушения в экспрессии гена гидрофобина BslA (Kobayashi, Iwano, 2012). Степень фосфорилирования фактора транскрипции DegU (DegU-P) определяет поведение клеток бактерий, при этом инициация образования биопленки нуждается в промежуточных уровнях активации фактора транскрипции DegU. При высоких уровнях фосфорилирования фактора DegU в клетках, наоборот, наблюдается опосредованное ингибирование биопленки (рис. 1в) (Marlow et al., 2014b). Активация образования биопленки начинается, когда фосфорилированный DegU-P запускает транскрипцию гена bslA, кодирующего гидрофобный белок (Hobley et al., 2015). Путь DegS–DegU активируется при ингибировании вращения жгутиков, что может произойти, когда клетка связывается с поверхностью и теряет подвижность (рис. 1в). Понижение вращения жгутика вызывало увеличение уровня фосфорилированного DegU-P, что запускало транскрипцию генов-мишеней, в том числе гена bslA (Chan et al., 2014; Cairns et al., 2014). Таким образом, остановка вращения жгутиков служит дополнительным сигналом для формирования полимерного матрикса.
Особенно важна в биопленках, где плотность клеток очень высока, автоиндуцирующая сигнальная система (Špacapan et al., 2018). Сигнальные молекулы связываются со специфическими рецепторами, вызывая транскрипцию и синтез необходимых секретируемых продуктов, называемых общественными благами (например, протеиназами). У B. subtilis автоиндуцирующая сигнальная система основана на пептидах как сигнальных молекулах, которые контролируют многие адаптационные процессы, включая синтез экзопротеиназ. Сигнальный аутоиндуцируемый пептид ComX участвует в регуляции бактериальной компетентности и продукции сурфактанта в B. subtilis (Aleti et al., 2016; Pollak et al., 2016). Оба признака регулируются в соответствии с размером бактериальной популяции, таким образом, классифицируя ComX как сигнал кворум-сенсинга. У B. subtilis основной системой на основе пептида автоиндукции является кластер генов comQXPA, известный как система определения кворума. ComX регулирует экспрессию srf-оперона, ответственного за синтез сурфактина, и участвует в развитии генетической компетентности (Aleti et al., 2016). В системе ComQXPA активность пары регуляторов ответа ComP–ComA модулируется сигнальным пептидом ComX. ComX модифицируется изопренилтрансферазой ComQ (Okada et al., 2005). Внеклеточное накопление модифицированного ComX приводит к фосфорилированию ComA и последующей индукции регулона ComA. Ген degQ также является частью этого регулона. Высокий уровень DegU-P положительно регулирует синтез внеклеточных ферментов, включая aprE, кодирующий сериновую экзопротеиназу B. subtilis (Veening et al., 2008). Таким образом, ComX и система ComQXPA положительно контролирует транскрипцию гена субтилизина aprE и продукцию экзопротеиназ во время роста биопленки. В свою очередь, экзопротеиназы способны деградировать сигнальный пептид ComX (Špacapan et al., 2018). Фактически описаны два типа регуляции с участием ComX: положительная генетическая регуляция и отрицательная биохимическая регуляция. Такая обратная связь балансирует спрос и предложение бактерий на полезные общественные блага через сигнальную молекулу ComX.
В дополнение к сложной регуляции транскрипционными факторам, экспрессия eps-оперона контролируется посредством цис-элемента РНК, который кодируется участком ДНК между вторым и третьим генами eps-оперона. Цис-элемент РНК обозначили как EAR-элемент (элемент, ассоциированный с РНК); он идентифицирован в подавляющем большинстве геномов у представителей семейства Bacillaceae. EAR-элемент действует как антитерминатор и увеличивает экспрессию eps-оперона путем взаимодействия с РНК (Irnov et al., 2010).
Если основная генетическая схема регуляции образования биопленки на уровне транскрипции охарактеризована, то о роли метаболизма (метаболизм углерода, пути биосинтеза, пути ферментации и вторичный метаболизм) в этом процессе сведений мало (Pisithkul et al., 2019). Вторичный мессенджер c-di-AMP играет важную роль в формировании биопленки B. subtilis и в прикреплении ее к корням растений (Townsley et al., 2018). В клетках B. subtilis, в отличие от большинства других генов биосинтеза аминокислот, экспрессия генов биосинтеза серина снижается при переходе в стационарную фазу, что является критическим внутриклеточным сигналом для активации биосинтеза матрикса биопленки (Greenwich et al., 2019). Была изучена связь между метаболизмом углерода и образованием биопленки (Chen et al., 2015). Анализ лизин-ацетилированных белков или ацетилома B. subtilis показал, что такая модификация белков играет важную регуляторную функцию в развитии биопленки (Reverdy et al., 2018). Установлена связь пути биосинтеза цистеина и образованием биопленки (Kobayashi, 2019). Формирование биопленки запускается, когда клетки испытывают недостаток глюкозы и зависят от контроля катаболита (Chen et al., 2015). Удаление гена ccpA стимулирует формирование биопленки и указывает, что белок CcpA негативно регулирует образование биопленки у B. subtilis. Другой глобальный регулятор метаболизма CodY также участвует в формировании биопленки у B. subtilis (Brinsmade et al., 2014). Бактерии способны синтезировать полиамины, и с помощью транскриптомного анализа спермидин-истощенных мутантов B. subtilis показано, что спермидин необходим для образования биопленки. Он активирует транскрипцию генов синтеза экзополисахаридов матрикса и tasA-оперона через регулятор SlrR (рис. 1а) (Hobley et al., 2017).
Рассмотренные пути регуляции указывают на сложную и многослойную регуляторную сеть, контролирующую дифференцировку на субпопуляции и образование биопленки у B. subtilis. Большинство регуляторов специфичны только для этого организма, их функциональные гомологи не обнаружены у других видов бактерий. Регуляторная сеть эволюционировала, чтобы бациллы адекватно реагировали на изменения метаболизма и окружающей среды путем формирования единого сообщества генетически идентичных, но отличающихся по физиологическим функциям микроорганизмов, заключенных в биопленку.
СИГНАЛЫ ДЛЯ ФОРМИРОВАНИЯ БИОПЛЕНКИ
Сведения о сигналах, ведущих к формированию биопленки и дифференцировке микроорганизмов в сообществе на субпопуляции, малочисленны. Описаны многочисленные рецепторы сигналов для регуляторных систем, которые свидетельствуют, что существует много условий, способных вызвать формирование биопленки. Концентрация Spo0A-P в клетке определяется активностью семейства гистидинкиназ (KinA, KinB, KinC, KinD), которые воспринимают сигналы и передают через фосфотрасферазы на белок Spo0A (Jiang et al., 2000; Grau et al., 2015). Фосфореле включает Spo0F-фосфотрансферазу, которая фосфорилируется киназой и передает фосфатную группу фосфотрансферазе Spo0B, в свою очередь фосфорилирующей фактор Spо0A. Пятая киназа, KinE может участвовать в этом пути и, по-видимому, влияет на экспрессию матричных генов (McLoon et al., 2011). Ни одна из идентифицированных киназ не отвечает полностью только за экспрессию генов полимерного матрикса; это скорее результат действия различных киназ в зависимости от разнообразия поступающих сигналов из среды.
Аутофосфорилирование гистидинкиназ происходит по цис-механизму, одна субъединица киназы фосфорилирует себя внутри мультимера, или транс-способом, одна субъединица мультимера фосфорилирует другую субъединицу. Установлено in vivo и in vitro, что аутофосфорилирование основной гистидинкиназы KinA при споруляции может происходить по транс-механизму и запускается в условиях истощения питательных веществ (Devi et al., 2015).
Первой сигнальной молекулой, идентифицированной в качестве индуктора генов биопленки, является секретируемый антибиотик сурфактин (Aleti et al., 2016). Сурфактин как сигнальная молекула через сенсорную киназу KinC инициирует фосфорилирование фактора Spo0A и способствует активации экспрессии генов полимерного матрикса биопленки (Mielich-Suss et al., 2015). Клетки, синтезирующие и секретирующие сурфактин, не являются клетками, которые реагируют на эту молекулу (Aleti et al., 2016). Эта концепция рассматривается как паракринная передача сигналов, когда продуцент сигнала не отвечает на свой сигнал. Это отличается от систем измерения кворума, в которой каждая клетка популяции продуцирует и реагирует на сигнальную молекулу (Aleti et al., 2016). Активация экспрессии генов в ответ на сигнальную молекулу сурфактин не происходит по традиционному механизму, включающему связывание сенсорного белка с лигандом. Сурфактин является липопептидом, молекула которого способна встраиваться в цитоплазматичекую мембрану, вследствие чего происходит утечка ионов калия, обуславливающая активацию гистидинкиназы KinC, передачу сигнала на Spo0A и индукцию экспрессии генов матрикса биопленки (Grau et al., 2015).
Бактерии рода Bacillus обладают большим сельскохозяйственным потенциалом, продуцируя липопептиды с высокой антимикробной активностью (Velmourougane et al., 2017; Актуганов и соавт., 2019). Рассматривают три семейства липопептидов Bacillus: сурфактин, итурин и фенгицин, молекулы которых имеют амфифильную природу, эффективно взаимодействуя с биологическими мембранными структурами. Их антимикробные свойства направлены против бактерий, грибков, оомицетов и вирусов. Установлено, что эти соединения стимулируют образование биопленок, что является ключевым фактором для успешной колонизации организмов биоконтроля (Penha et al., 2020). Другие соединения, которые вызывают утечку калия, фунгицид нистатин и антибиотик валиномицин, также вызывают экспрессию генов матрикса биопленки через гистидинкиназу KinC. Сурфактин, нистатин и валиномицин являются природными продуктами почвенных микроорганизмов, и бактерии B. subtilis встречаются с этими соединениями в природных условиях. Нистатин индуцирует передачу сигналов у B. subtilis путем связывания эргостерина мембраны; в совокупности полученные данные позволили предположить, что мембраны B. subtilis содержат микродомены по аналогии с липидными рафтами в плазматических мембранах эукариотических организмов (Lopez, 2015). Диоксид хлора также индуцирует экспрессию генов матрикса биопленки зависимым от гистидинкиназы KinC способом (Shemesh et al., 2010). Диоксид хлора, в отличие от сурфактина, запускает активацию гистидинкиназы KinC путем нарушения мембранного потенциала клетки. Увеличение синтеза компонентов матрикса в присутствии ингибиторов, нарушающих структуру мембраны, необходимо для выживания B. subtilis, когда экзополисахарид обеспечивает защиту от летальных последствий воздействия этих соединений.
Циклические липопептиды и особенно сурфактины, продуцируемые Bacillus sp., запускают формирование биопленки и колонизацию корней и имеют решающее значение для активности биоконтроля и системной устойчивости растений (Aleti et al., 2016). Важная функция сурфактина заключается в его способности разрушать мембранные везикулы у бацилл (Brown et al., 2014). Везикулы образуются клетками биопленки B. subtilis. Кодируемый профагом эндолизин может генерировать отверстия в клеточной стенке, через которые материал цитоплазматической мембраны выступает и выделяется в виде везикул (Brown et al., 2015; Toyofuku et al., 2017). Везикулы могут инкапсулировать важные для сообщества факторы и быть необходимыми для его выживания. Сурфактин лизирует внеклеточные везикулы с высвобождением содержимого у грамположительных бактерий.
У мутантов B. subtilis с нарушениями в синтезе матрикса с помощью транскрипционных репортеров наблюдали пролонгированную экспрессию eps- и tasA-оперонов и замедленную споруляцию в биопленке (Aguilar et al., 2010). Это связано с функционированием киназы KinD B. subtilis, обладающей киназной и фосфатазной активностями. В качестве фосфатазы KinD участвует в сохранении низкого уровня фосфорилированного Spo0A-P, что сопровождается формированием матрикса биопленки, и в качестве киназы KinD способствует развитию процесса спорообразования. Транскрипция основных оперонов, ответственных за образование матрикса, значительно усиливалась в ответ на присутствие в среде глицерина и марганца путем активации гистидинкиназы KinD, ответственной за восприятие присутствия этих соединений вне клетки (Shemesh et al., 2013). Есть данные, что KinD активируется в ответ на соединения, продуцируемые почвенными микроорганизмами и экссудатом корней растений (Chen et al., 2012).
Все генетические каскады контролируются семейством Rap фосфатаз, которые специфически взаимодействуют с экзогенными родственными пептидами (Phr-пептиды) (Veening et al., 2005). Известно, что фосфатаза RapGH дефосфорилирует DegU-P, в то время как фосфатаза RapABEJ действует на передачу фосфата в Spo0A-фосфореле, а фосфатаза RapCFGHK – на ComA-P. Вместе они могут представлять альтернативную систему передачи сигналов между клетками, нацеленную на реализацию программы бистабильности у B. subtilis, участвуют в регуляции процессов специализации и коллективного поведения, что обеспечивает взаимодействие и выживание вида (Verdugo-Fuentes et al., 2020).
Полная сеть регуляции Phr–Rap у B. subtilis далека от понимания из-за сложности регуляторных каскадов. Идентифицирован дополнительный уровень сложности в контроле сетей сигнализации у B. subtilis. Мембрана бацилл организована в микродомены, целостность которых важна для активации гистидинкиназы KinC и, следовательно, для дифференцировки клеток Spo0A-ON (Lopez, 2015). Показано, что для мембраны бацилл характерна компартментализация путей передачи сигнала через функциональные мембранные микродомены, которые играют важную роль в активации каскадов сигнальной трансдукции (Wagner et al., 2017). В составе микродоменов идентифицирована мембраносвязанная протеиназа FtsH, избирательно гидролизующая специфические Rap фосфатазы (Mielich-Suss et al., 2013). Поэтому целостность микродоменов цитоплазматической мембраны B. subtilis необходима для индукции формирования биопленки путем сохранения и стабилизации белков FtsH и KinC. Нарушения мембранных микродоменов (сарагозиновой кислотой) приводят к ингибированию сигнальных каскадов, ведущих к образованию биопленки у B. subtillis (Lopez, 2015; Wagner et al., 2017). Точно так же, протеиназная активность белка FtsH ингибируется пептидом SpoVM, который может использоваться в качестве ингибитора биопленки (Yepes et al., 2012). Причем микродомены ограничены набором белков, участвующих в специфических клеточных процессах, таких как секреция протеиназ и трансдукция сигнала (Lopez, Koch, 2017).
У B. subtilis для запуска генов, кодирующих компоненты матрикса, могут быть использованы не сигнальные механизмы. Так, в дополнение к механизмам активации транскрипции генов для синтеза матриксного полимера при низком уровне содержания в клетках фосфорилированного Spo0A-P у B. subtilis происходит экспрессия двух генных кластеров “каннибализма”, в результате часть клеток популяции синтезирует и секретирует токсичные пептиды SDP и SKF. Бактерии, которые синтезируют токсины, сами являются устойчивыми к этим токсичным пептидам (Höfler et al., 2016). Для транспорта пептида SKF во внеклеточное пространство необходим ABC-транспортер, который удаляет этот токсин из клетки (Gonzalez-Pastor, 2011). Устойчивость бактерий к другому пептиду SDP обусловлена наличием в мембране специфического белка SdpI, синтез которого индуцируется в присутствии токсичного SDP в клетках с высокой концентрацией фосфорилированного регуляторного белка Spo0A-P (Ellermeier et al., 2006; Kobayashi, Ikemoto, 2019). Клетки-каннибалы выделяют токсичные пептиды, которые целенаправленно убивают клетки B. subtilis, не экспрессирующие гены токсинов. Поскольку гены, кодирующие токсины, и гены, кодирующие полимер матрикса, активируются при низком уровнем фосфорилированного регуляторного белка Spo0A-P, популяции клеток, формирующих биопленку, и клеток, синтезирующих токсичные пептиды, перекрываются (Lopez et al., 2009). В результате клетки, которые инициировали синтез полимера матрикса, выделяют также токсичные пептиды, направленные на уменьшение популяции клеток, не продуцирующих матрикс биопленки. Это приводит к формированию популяции, состоящей из увеличенного количества клеток, продуцирующих матрикс. Токсины каннибализма не только убивают клетки B. subtilis, но и клетки других видов, когда B. subtilis выращивается в смешанных культурах (Liu et al., 2010). При совместном культивировании B. subtilis (опыты проводились с использованием флуоресцентных репортеров для матричных генов) с другими почвенными микроорганизмами в присутствии индукторов для образования биопленки было обнаружено, что подавляющее большинство составляли представители рода Bacillus, несмотря на разнообразие бактерий в образцах почвы.
Таким образом, разнообразные сигнальные молекулы, от сурфактина до токсинов каннибализма, могут способствовать росту числа клеток для стимуляции образования биопленки в популяции. Это происходит с помощью передачи сигналов, приводящих к дифференциальной экспрессии генов, или путем избирательного уничтожения клеток B. subtilis, не синтезирующих полимерный матрикс биопленки.
РАССЕИВАНИЕ И ДИССОЦИАЦИЯ БИОПЛЕНКИ
Бактериальные биопленки важны в естественных условиях, для биотехнологии и медицины. Однако понимание регуляции развития биопленки и ее сохранения в разных нишах является далеко не полным. Важной стадией в течение жизненного цикла биопленки является рассеивание, когда подвижные клетки покидают зрелую биопленку, чтобы распространиться и колонизировать новые ниши (Плакунов и соавт., 2017). Триггерами выступают различные экзо- и эндогенные факторы: изменение доступности питательных веществ, бактериальные аутоиндукторы, жирные кислоты, пептидные сигналы, оксид азота, стресс-сигналы (голод, избыток железа, фосфатов и др.). Такие сигналы приводят к изменению концентрации вторичных мессенджеров – циклического дигуанозинмонофосфата (c-di-GMP) и циклического диаденилатмонофосфата (c-di-AMP), гуанозинтетрафосфата (ppGpp), гуанозинпентафосфата (pppGpp) и малых регуляторных РНК, которые изменяют активность экспрессии бактериальных генов (Romling, Galperin, 2017; Townsley et al., 2018). Процесс рассеивания биопленки контролируется путем регуляции на уровне транскрипции оперонов, кодирующих матрикс биопленки. Установлено, что в определенных клетках бактерий B. subtilis запускается механизм ингибирования экспрессии генов матрикса, и они возвращаются в состояние планктонных клеток (Vlamakis et al., 2013; Norman et al., 2013). На уровне экспрессии генов это происходит в результате снижения уровня антирепрессора SlrR по мере созревания биопленки, что связано с его нестабильностью в результате расщепления протеиназой ClpCP и как результат ауторазрушения (Chai et al., 2010). Расщепление регулятора SlrR позволяет репрессору SinR взаимодействовать с областями промоторов оперонов матрикса, ингибируя биосинтез компонентов матрикса (Ogura, 2016; Milton et al., 2020).
Установлено, что у B. subtilis транскрипционный фактор SigB, активирующийся на общий стресс, необходим для остановки разрастания зрелой биопленки и запуска механизма рассеивания, когда питательные вещества становятся ограниченными (Bartolini et al., 2018). В частности, биопленки с дефектным геном sigB были больше, чем биопленки дикого типа, но демонстрировали ускоренную гибель клеток, большую чувствительность к стрессам и снижению дисперсии. Причем обнаружена регуляторная взаимосвязь между фактором SigB и экспрессией репрессора SinR. Эта новая регуляторная схема SigB–SinR важна для контроля пригодности биопленок в различных средах, в которой регуляторный статус факторов SigB и SinR определяет, остаются клетки в биопленке или покидают ее, если условия становятся неблагоприятными.
Диссоциация биопленки основана на контролируемом расщеплении макромолекул ее матрикса во внеклеточной среде (Плакунов и соавт., 2017). В этом процессе могут участвовать внеклеточные протеиназы (Marlow et al., 2014a). Имеются данные, что субтилизиноподобная протеиназа наттокиназа B. subtilis (Dabbagh et al., 2014) и субтилизиноподобная протеиназа B. pumilus способны разрушать амилоидный пептид (Danilova et al., 2014). Можно предположить, что внеклеточные протеиназы бацилл участвуют в разборке волокон TasA матрикса биопленки (Mitrofanova et al., 2017). Поиск путей для изучения диссоциации биопленок является важной социальной проблемой. Эти данные полезны при искоренении хронических инфекций, для предотвращения закупорки трубопроводов и др. (Hobley et al., 2015; Velmourougane et al., 2017; Vaccari et al., 2017). D-аминокислоты, добавленные к биопленочным культурам, вызывали диссоциацию биопленок B. subtilis путем включения в пептидогликан и блокирования встраивания TapA белка в клеточную стенку, что приводило к деградации волокон TasA (Romero et al., 2014; Yu et al., 2016). Инкорпорация D-аминокислот в белки может быть предотвращена в присутствии D-аминоацил-тРНК-деацилазы, которая удаляет D-аминокислоты из неправильно заряженных тРНК, штамм B. subtilis с мутацией в гене D‑аминоацил-тРНК-деацилазы (ген dtd) характеризовался подавлением биопленки, когда мутация была устранена, ингибирование биопленки не наблюдалось (Leiman et al., 2013). Молекулярная основа токсичности D-аминокислот зависит от способности замещать соответствующие L‑изомеры во время синтеза белка и вызывать нарушение в функционировании белков.
Другое соединение, способное участвовать в диссоциации биопленок, представляет собой полиамин норспермидин (Si et al., 2015; Wu et al., 2016). Норспермидин может специфически взаимодействовать с экзополисахаридом матрикса и вызывать нарушения в структуре биопленок, при этом мутанты B. subtilis, неспособные продуцировать норспермидин, формировали долгоживущие биопленки (Kolodkin-Gal et al., 2012). Биоинформатический поиск генов-гомологов пути биосинтеза норспермидина E. coli в геноме B. subtilis не выявил гомологичных генов (Hobley et al., 2014). Тем не менее, эти исследования показали, что такие специфические триггеры биопленки, как D-аминокислоты и норспермидин, способны дестабилизировать матриксный полимер зрелой биопленки B. subtilis.
ЗАКЛЮЧЕНИЕ
B. subtilis является исключительно удобным модельным организмом для исследований многообразия регуляторных путей стационарной фазы этих бактерий, поскольку они способны к выживанию в широком диапазоне физико-химических условий, включая экстремальные. В геноме этих бактерий наряду с генетической информацией о спорообразовании заложена информация о механизмах биосинтеза и дисперсии биопленок, основанная на сложных генетических программах, ответственных за создание единого сообщества субпопуляций генетически идентичных, но фенотипически различных клеток, которое является формальным аналогом сложных многоклеточных организмов (Kovacs, Dragos, 2019). Для биопленок B. subtilis выявлен эффект, сходный с так называемым “родственным отбором” (kin-selection), характерным для высших организмов и приводящим к обеспечению жизнеспособности дочерних клеточных поколений. Бистабильность, выраженная в фенотипическом изменении клеток, способствует приспособляемости вида, поскольку субпопуляции взаимовыгодно взаимодействуют внутри сообщества, а гетерогенная популяция способна быстро реагировать на любые изменение окружающей среды. Для B. subtilis идентифицирована развитая сеть регуляторных систем, участвующих в образовании гетерогенного микробного сообщества, установлены сигнальные и регуляторные пути, которые запускают формирование биопленки. Среди них одним из центральных событий является активация транскрипционного фактора Spo0A, запускающего экспрессию генных кластеров, кодирующих матрикс биопленки, в ответ на широкий спектр внеклеточных сигналов. Одной из особенностей клеток B. subtilis, формирующих биопленку, является синтез и секреция токсичных пептидов каннибализма для подавления клеток, не синтезирующих внеклеточные полимеры матрикса. Реальные механизмы регуляции могут быть более многообразными, поскольку бактерии B. subtilis в природе обитают в сообществе с корнями растений или в присутствии других организмов (Бузолева и соавт., 2016; Yannarell et al., 2019; Hashem et al., 2019; Bóka et al., 2019). В связи с этим, многие проблемы в развитии биопленок B. subtilis остаются нерешенными. Комплексный анализ регуляторных схем привел к открытию новых способов регуляции для бактерий, включая впервые описанное глобальное ацетилирование белков (Reverdy et al., 2018) или открытие особых участков, мембранных функциональных микродоменов, эквивалентных липидным рафтам в плазматических мембранах эукариотических клеток (Lopez, 2015; Bramkamp et al., 2015; Lopez, Koch, 2017; Wagner et al., 2017). Процесс передачи сигналов через регуляторные каскады сигнальной трансдукции у бацилл происходит при участии мембранных микродоменов в соответствии с набором окружающих факторов. Структурная организация микродоменов показала неожиданный уровень сложности, что стало беспрецедентным для бактерий, поскольку бактерии являются более сложными организмами, чем считалось ранее. Более того, данные свидетельствуют о том, что мембранные микродомены широко распространены среди бактерий. В отличие от информации о регуляции механизмов образования биопленки, изучение путей контроля за рассеиванием и диссоциацией биопленки у B. subtilis находится в начальном состоянии. Вопросы, которые предстоит решить, связаны с синтезом специфических пептидов и D-аминокислот, участвующих в разборке биопленки в разных условиях, кроме того, предстоит определить внешние и внутренние стимулы, запускающие эти процессы. Такие исследования имеют не только фундаментальную, но и практическую значимость и нацелены, в том числе, на борьбу с биопленками, формируемыми патогенными организмами, для разработки эффективных способов новой комбинированной терапии.
ФИНАНСИРОВАНИЕ РАБОТЫ
Работа выполнена в рамках государственной программы повышения конкурентоспособности Казанского (Приволжского) федерального университета среди ведущих мировых научно-образовательных центров и поддержана грантами РНФ 16-16-04062 и РФФИ 19-08-00853.
Список литературы
Актуганов Г.Э., Галимзянова Н.Ф., Гильванова Е.А., Рябова А.С., Сафина В.Р., Пудова Е.А., Кузьмина Л.Ю., Бойко Т.Ф., Мелентьев А.И. Влияние источников углерода и минеральных солей на формирование биопленок и синтез циклических липопептидов PGPB-штаммами Bacillus и Paenibacillus // Экобиотех. 2019. Т. 2. С. 468–481.
Бузолева Л.С., Цветкова Н.Б., Богатыренко Е.А. Формирование биопленок бактериями Listeria monocytogenes разных серовариантов в ассоциации с Bacillus pumilus // Микробиология. 2016. Т. 85. С. 293–299.
Buzoleva L.S., Bogatyrenko E.A., Tsvetkova N.B. Biofilm formation by different serological variants of Listeria monocytogenes in association with Bacillus pumilus // Microbiology (Moscow). 2016. V. 85. P. 311–316.
Каюмов А.Р., Балабан Н.П., Марданова А.М., Костров С.В., Шарипова М.Р. Биосинтез субтилизиноподобной сериновой протеиназы Bacillus intermedius в условиях солевого стресса // Микробиология. 2006. Т. 75. С. 557–563.
Kayumov A.R., Balaban N.P., Mardanova A.M., Sharipova M.R., Kostrov S.V. Biosynthesis of the subtilisin-like serine proteinase of Bacillus intermedius under salt stress conditions // Microbiology (Moscow). 2006. V. 75. P. 557–562.
Плакунов В.К., Мартьянов С.В., Тетенева Н.А., Журина М.В. Универсальный метод количественной характеристики роста и метаболической активности микробных биопленок в статических моделях // Микробиология. 2016. Т. 85. С. 484–489.
Plakunov V.K., Mart’yanov S.V., Teteneva N.A., Zhurina M.V. A universal method for quantitative characterization of growth and metabolic activity of microbial biofilms in static models // Microbiology (Moscow). 2016. V. 85. P. 509–513.
Плакунов В.К., Мартьянов С.В., Тетенева Н.А., Журина М.В. Управление формированием микробных биопленок: анти- и пробиотические агенты // Микробиология. 2017. Т. 86. С. 402–420.
Plakunov V.K., Mart’yanov S.V., Teteneva N.A., Zhurina M.V. Controlling of microbial biofilms formation: anti- and probiofilm agents // Microbiology (Moscow). 2017. V. 86. P. 423–438.
Пономарева А.Л., Бузолева Л.С., Богатыренко Е.А. Абиотические факторы внешней среды, влияющие на формирование биопленок микроорганизмов // Известия РАН. Сер. биол. 2018. Т. 45. С. 549–556.
Ponomareva A.L., Buzoleva L.S., Bogatyrenko E.A. Abiotic environmental factors affecting the formation of microbial biofilms // Biol. Bull. 2018. V. 45. P. 490–496.
Стрелкова Е.А., Позднякова Н.В., Журина М.В., Плакунов В.К., Беляев С.С. Роль внеклеточного полимерного матрикса в устойчивости бактериальных биопленок к экстремальным факторам среды // Микробиология. 2013. Т. 82. С. 131–138.
Strelkova E.A., Pozdnyakova N.V., Zhurina M.V., Plakunov V.K., Belyaev S.S. Role of the extracellular polymer matrix in resistance of bacterial biofilms to extreme environmental factors // Microbiology (Moscow). 2013. V. 82. P. 119–125.
Aguilar C., Vlamakis H., Guzman A., Losick R., Kolter R. KinD is a checkpoint protein linking spore formation to extracellular-matrix production in Bacillus subtilis biofilms // MBio. 2010. V. 1. P. e00035-10.
Aleti G., Lehner S., Bacher M., Compant S., Nikolic B., Plesko M., Schuhmacher R., Sessitsch A., Brader G. Surfactin variants mediate species-specific biofilm formation and root colonization in Bacillus // Environ. Microbiol. 2016. V. 18. P. 2634–2645.
Arnaouteli S., Ferreira A.S., Schor M., Morris R.J., Bromley K.M., Jo J. Bifunctionality of a biofilm matrix protein controlled by redox state // Proc. Natl. Acad. Sci. USA. 2017. V. 114. P. 6184–6191.
Asally M., Kittisopikul M., Rue P., Du Y., Hu Z., Cagatay T. Localized cell death focuses mechanical forces during 3D patterning in a biofilm // Proc. Natl. Acad. Sci. USA. 2012. V. 109. P. 18891–18896.
Balasubramanian S., Aubin-Tam M.E., Meyer A.S. 3D Printing for the fabrication of biofilm-based functional living materials // ACS Synth. Biol. 2019. V. 8. P. 1564–1567.
Bareia T., Pollak S., Eldar A. Self-sensing in Bacillus subtilis quorum-sensing systems // Nat. Microbiol. 2018. V. 3. P. 83–89.
Bartolini M., Cogliati S., Vileta D., Bauman C., Rateni L., Leñini C., Argañaraz F., Francisco M., Villalba J.M., Steil L., Völker U., Grau R. Regulation of biofilm aging and dispersal in Bacillus subtilis by the alternative sigma factor SigB // J. Bacteriol. 2018. V. 201. P. e00473-18.
Benigar E., Dogsa I., Stopar D., Jamnik A., Kralj Cigic I., Tomsic M. Structure and dynamics of a polysaccharide matrix: aqueous solutions of bacterial levan // Langmuir. 2014. V. 30. P. 4172–4182.
Bóka B., Manczinger L., Kocsubé S., Shine K., Alharbi N.S., Khaled J.M., Münsterkötter M., Vágvölgyi C., Kredics L. Genome analysis of a Bacillus subtilis strain reveals genetic mutations determining biocontrol properties // World J. Microbiol. Biotechnol. 2019. V. 35. P. 52.
Bramkamp M., Lopez D. Exploring the existence of lipid rafts in bacteria. Version 2 // Microbiol. Mol. Biol. Rev. 2015. V. 79. P. 81–100.
Branda S.S., Chu F., Kearns D.B., Losick R., Kolter R. A major protein component of the Bacillus subtilis biofilm matrix // Mol. Microbiol. 2006. V. 59. P. 1229–1238.
Branda S.S., Gonzalez-Pastor J.E., Ben-Yehuda S., Losick R., Kolter R. Fruiting body formation by Bacillus subtilis // Proc. Natl. Acad. Sci. USA. 2001. V. 98. P. 11621–11626.
Brinsmade S.R., Alexander E.L., Livny J., Stettner A.I., Segre D., Rhee K.Y., Sonenshein A.L. Hierarchical expression of genes controlled by the Bacillus subtilis global regulatory protein CodY // Proc. Natl. Acad. Sci. USA. 2014. V. 111. P. 8227–8232.
Brown L., Kessler A., Cabezas-Sanchez P., Luque-Garcia J.L., Casadevall A. Extracellular vesicles produced by the Gram-positive bacterium Bacillus subtilis are disrupted by the lipopeptide surfactin // Mol. Microbiol. 2014. V. 93. P. 183–198.
Brown L., Kessler A., Casadevall A. Vesicle isolation from Bacillus subtilis biofilm // Bio Protoc. 2015. V. 5. P. e1409.
Cairns L.S., Hobley L., Stanley-Wall N.R. Biofilm formation by Bacillus subtilis: new insights into regulatory strategies and assembly mechanisms // Mol. Microbiol. 2014. V. 93. P. 587–598.
Cámara-Almirón J., Navarro Y., Díaz-Martínez L., Magno-Pérez-Bryan M.C., Molina-Santiago C., Pearson J.R., Vicente A., Pérez-García A., Romero D. Dual functionality of the amyloid protein TasA in Bacillus physiology and fitness on the phylloplane // Nat. Commun. 2020. V. 11. P. 1859.
Chai L., Romero D., Kayatekin C., Akabayov B., Vlamakis H., Losick R., Kolter R. Isolation, characterization, and aggregation of a structured bacterial matrix precursor // J. Biol. Chem. 2013. V. 288. P. 17559–17568.
Chai Y., Beauregard P.B., Vlamakis H., Losick R., Kolter R. Galactose metabolism plays a crucial role in biofilm formation by Bacillus subtilis // MBio. 2012. V. 3. P. e00184-12.
Chai Y., Kolter R., Losick R. Paralogous antirepressors acting on the master regulator for biofilm formation in Bacillus subtilis // Mol. Microbiol. 2009. V. 74. P. 876–887.
Chai Y., Kolter R., Losick R. Reversal of an epigenetic switch governing cell chaining in Bacillus subtilis by protein instability // Mol. Microbiol. 2010. V. 78. P. 218–229.
Chai Y., Norman T., Kolter R., Losick R. Evidence that metabolism and chromosome copy number control mutually exclusive cell fates in Bacillus subtilis // EMBO J. 2011. V. 30. P. 1402–1413.
Chan J.M., Guttenplan S.B., Kearns D.B. Defects in the flagellar motor increase synthesis of polygamma-glutamate in Bacillus subtilis // J. Bacteriol. 2014. V. 196. P. 740–753.
Chastanet A., Losick R. Just-in-time control of Spo0A synthesis in Bacillus subtilis by multiple regulatory mechanisms // J. Bacteriol. 2011. V. 193. P. 6366–6374.
Chen Y., Cao S., Chai Y., Clardy J., Kolter R., Guo J.H., Losick R. A Bacillus subtilis sensor kinase involved in triggering biofilm formation on the roots of tomato plants // Mol. Microbiol. 2012. V. 85. P. 418–430.
Chen Y., Gozzi K., Yan F., Chai Y. Acetic acid acts as a volatile signal to stimulate bacterial biofilm formation // mBio. 2015. V. 6. P. e00392.
Cheremin M., Nyamsuren Ch., Toimentseva A.A., Sharipova M.R. Optimization of the expression of the subtilisin-like protease from Bacillus pumilus // Russ. J. Bioorg. Chem. 2014. V. 40. P. 694–698.
Chumsakul O., Takahashi H., Oshima T., Hishimoto T., Kanaya S., Ogasawara N., Ishikawa S. Genome-wide binding profiles of the Bacillus subtilis transition state regulator AbrB and its homolog Abh reveals their interactive role in transcriptional regulation // Nucl. Acids. Res. 2011. V. 39. P. 414–428.
Dabbagh F., Negahdaripour M., Berenjian A., Behfar A., Mohammadi F., Zamani M., Irajie C., Ghasemi Y. Nattokinase: production and application // Appl. Microbiol. Biotechnol. 2014. V. 98. P. 9199–9206.
Danilova Y.V., Shagimardanova E.I., Margulis A.B., Toymentseva A.A., Balaban N.P., Rudakova N.L., Rizvanov A.A., Sharipova M.R., Palotas A. Bacterial enzymes effectively digest Alzheimer’s P-amyloid peptide // Brain. Res. Bull. 2014. V. 108. P. 113–117.
DeLoughery A., Dengler V., Chai Y., Losick R. Biofilm formation by Bacillus subtilis requires an endoribonuclease-containing multisubunit complex that controls mRNA levels for the matrix gene repressor SinR // Mol. Microbiol. 2016. V. 99. P. 425–437.
Devi S.N., Kiehler B., Haggett L., Fujita M. Evidence that autophosphorylation of the major sporulation kinase in Bacillus subtilis is able to occur in trans // J. Bacteriol. 2015. V. 197. P. 2675–2684.
Diehl A., Roske Y., Ball L., Chowdhury A., Hiller M., Molière N., Kramer R., Stöppler D., Worth C.L., Schlegel B., Leidert M., Cremer N., Erdmann N., Lopez D., Stephanowitz H., Krause E., van Rossum B.-J., Schmieder P., Heinemann U., Turgay K., Akbey Ü., Oschkinat H. Structural changes of TasA in biofilm formation of Bacillus subtilis // PNAS. 2018. V. 115. P. 3237–3242.
Dogsa I., Brloznik M., Stopar D., Mandic-Mulec I. Exopolymer diversity and the role of levan in Bacillus subtilis biofilms // PLoS One. 2013. V. 8. P. e62044.
Dragos A., Kovacs A.T., Claessen D. The role of functional amyloids in multicellular growth and development of Gram-positive bacteria // Biomolecules. 2017. V. 7. P. 60.
Dragos A., Kovacs B.T. The peculiar functions of the bacterial extracellular matrix // Trends Microbiol. 2017. V. 25. P. 257–266.
Ellermeier C.D., Hobbs E.C., Gonzalez-Pastor J.E., Losick R. A three-protein signaling pathway governing immunity to a bacterial cannibalism toxin // Cell. 2006. V. 124. P. 549–559.
Fu H., Elena R.C., Marquez P.H. The roles of small RNAs: insights from bacterial quorum sensing // ExRNA. 2019. V. 1. P. 32.
Fuchs F.M., Holland G., Moeller R., Laue M. Directed freeze-fracturing of Bacillus subtilis biofilms for conventional scanning electron microscopy // J. Microbiol. Methods. 2018. V. 152. P. 165–172.
Fujita M., Gonzalez-Pastor J.E., Losick R. High- and low-threshold genes in the Spo0A regulon of Bacillus subtilis // J. Bacteriol. 2005. V. 187. P. 1357–1368.
Gabdrakhmanova L., Vishniakov I., Sharipova M., Balaban N., Kostrov S., Leshchinskaya I. Salt stress induction of glutamyl endopeptidase biosynthesis in Bacillus intermedius // Microbiol. Res. 2005. V. 160. P. 233–242.
Gallegos-Monterrosa R., Mhatre E., Kovács A.T. Specific Bacillus Subtilis 168 variants form biofilms on nutrient-rich medium // Microbiology (SGM). 2016. V. 162. P. 1922–1932.
Gingichashvili S., Duanis-Assaf D., Shemesh M., Featherstone J.D.B., Feuerstein O., Steinberg D. The adaptive morphology of Bacillus subtilis biofilms: a defense mechanism against bacterial starvation // Microorganisms. 2019. V. 8. P. 62. https://doi.org/10.3390/microorganisms8010062
Gonzalez-Pastor J.E. Cannibalism: a social behavior in sporulating Bacillus subtilis // FEMS Microbiol. Rev. 2011. V. 35. P. 415–424.
Grau R.R., de Oña P., Kunert M., Leñini C., Gallegos-Monterrosa R., Mhatre E., Vileta D., Donato V., Hölscher T., Boland W., Kuipers O.P., Kovács Á.T. A duo of potassium-responsive histidine kinases govern the multicellular destiny of Bacillus subtilis // mBio. 2015. V. 6. P. e00581.
Greenwich J., Reverdy A., Gozzi K., Di Cecco G., Tashjian T., Godoy-Carter V., Chai Y. A Decrease in serine levels during growth transition triggers biofilm formation in Bacillus subtilis // J. Bacteriol. 2019. V. 201. P. e00155-19.
Guttenplan S.B., Kearns D.B. Regulation of flagellar motility during biofilm formation // FEMS Microbiol. Rev. 2013. V. 37. P. 849–871.
Hamon M.A., Stanley N.R., Britton R.A., Grossman A.D., Lazazzera B.A. Identification of AbrB-regulated genes involved in biofilm formation by Bacillus subtilis // Mol. Microbiol. 2004. V. 52. P. 847–860.
Hashem A., Tabassum B., Fathi Abd Allah E. Bacillus subtilis: A plant-growth promoting rhizobacterium that also impacts biotic stress // Saudi J. Biol. Sci. 2019. V. 26. P. 1291–1297.
Hobley L., Harkins C., MacPhee C.E., Stanley-Wall N.R. Giving structure to the biofilm matrix: an overview of individual strategies and emerging common themes // FEMS Microbiol. Rev. 2015. V. 39. P. 649–669.
Hobley L., Kim S.H., Maezato Y., Wyllie S., Fairlamb A.H., Stanley-Wall N.R., Michael A.J. Norspermidine is not a self-produced trigger for biofilm disassembly // Cell. 2014. V. 156. P. 844–854.
Hobley L., Li B., Wood J.L., Kim S.H., Naidoo J., Ferreira A.S., Khomutov M., Khomutov A., Stanley-Wall N.R., Michael A.J. Spermidine promotes Bacillus subtilis biofilm formation by activating expression of the matrix regulator slrR // J. Biol. Chem. 2017. V. 292. P. 12041–12053.
Höfler C., Heckmann J., Fritsch A., Popp P., Gebhard S., Fritz G. Cannibalism stress response in Bacillus subtilis // Microbiology (SGM). 2016. V. 162. P. 164–176.
Hollenbeck E.C., Douarche C., Allain J.M., Roger P., Regeard C., Cegelski L., Fuller G.G., Raspaud E. Mechanical behavior of a Bacillus subtilis pellicle // J. Phys. Chem. B. 2016. V. 120. P. 6080–6088.
Huang J., Liu S., Zhang C., Wang X., Pu J., Ba F., Xue S., Ye H., Zhao T., Li K., Wang Y., Zhang J., Wang L., Fan C., Lu T.K., Zhong C. Programmable and printable Bacillus subtilis biofilms as engineered living materials // Nat. Chem. Biol. 2018. V. 15. P. 34–41.
Irnov I., Winkler W.C. A regulatory RNA required for antitermination of biofilm and capsular polysaccharide operons in Bacillales // Mol. Microbiol. 2010. V. 76. P. 559–575.
Jiang M., Shao W., Perego M., Hoch J.A. Multiple histidine kinases regulate entry into stationary phase and sporulation in Bacillus subtilis // Mol. Microbiol. 2000. V. 38. P. 535–542.
Jones S.E., Paynich M.L., Kearns D.B., Knight K.L. Protection from intestinal inflammation by bacterial exopolysaccharides // J. Immunol. 2014. V. 192. P. 4813–4820.
Kalamara M., Spacapan M., Mandic-Mulec I., Stanley-Wall N.R. Social behaviours by Bacillus subtilis: quorum sensing, kin discrimination and beyond // Mol. Microbiol. 2018. V. 110. P. 863–878.
Kampf J., Gerwig J., Kruse K., Cleverley R., Dormeyer M., Grunberger A., Kohlheyer D., Commichau F.M., Lewis R.J., Stulke J. Selective pressure for biofilm formation in Bacillus subtilis: differential effect of mutations in the master regulator SinR on bistability // mBio. 2018. V. 9. P. e01464-18.
Kesel S., Bronk B., García C.F., Götz A., Lieleg O., Opitz M. Matrix composition determines the dimensions of Bacillus subtilis NCIB 3610 biofilm colonies grown on LB agar // RSC Adv. 2017. V. 7. P. 31886–31898.
Khezri M., Jouzani G.S., Ahmadzadeh M. Fusarium culmorum affects expression of biofilm formation key genes in Bacillus subtilis // Braz. J. Microbiol. 2016. V. 47. P. 47–54.
Kobayashi K. Inactivation of cysL inhibits biofilm formation by activating the disulfide stress regulator Spx in Bacillus subtilis // J. Bacteriol. 2019. V. 201. https://doi.org/10.1128/JB.00712-18
Kobayashi K., Ikemoto Y. Biofilm-associated toxin and extracellular protease cooperatively suppress competitors in Bacillus subtilis biofilms // PLoS Genet. 2019. V. 15. P. e1008232.
Kobayashi K., Iwano M. BslA (YuaB) forms a hydrophobic layer on the surface of Bacillus subtilis biofilms // Mol. Microbiol. 2012. V. 85. P. 51–66.
Kolodkin-Gal I., Cao S., Chai L., Bottcher T., Kolter R., Clardy J., Losick R. A self- produced trigger for biofilm disassembly that targets exopolysaccharide // Cell. 2012. V. 149. P. 684–692.
Kolodkin-Gal I., Elsholz A.K., Muth C., Girguis P.R., Kolter R., Losick R. Respiration control of multicellularity in Bacillus subtilis by a complex of the cytochrome chain with a membrane-embedded histidine kinase // Genes Dev. 2013. V. 27. P. 887–899.
Kovacs A.T., Dragos A. Evolved biofilm: review on the experimental evolution studies of Bacillus subtilis pellicles // J. Mol. Biol. 2019. V. 431. P. 4749–4759.
Lazarevic V., Soldo B., Medico N., Pooley H., Bron S., Karamata D. Bacillus subtilis α-phosphoglucomutase is required for normal cell morphology and biofilm formation // Appl. Environ. Microbiol. 2005. V. 71. P. 39–45.
Leiman S.A., May J.M., Lebar M.D., Kahne D., Kolter R., Losick R. D-amino acids indirectly inhibit biofilm formation in Bacillus subtilis by interfering with protein synthesis // J. Bacteriol. 2013. V. 195. P. 5391–5395.
Liu W.T., Yang Y.L., Xu Y., Lamsa A., Haste N.M., Yang J.Y., Dorrestein P.C. Imaging mass spectrometry of intraspecies metabolic exchange revealed the cannibalistic factors of Bacillus subtilis // Proc. Natl. Acad. Sci. USA. 2010. V. 107. P. 16286–16290.
Lopez D. Molecular composition of functional microdomains in bacterial membranes // Chem. Phys. Lipids. 2015. V. 192. P. 3–11.
Lopez D., Koch G. Exploring functional membrane microdomains in bacteria: an overview // Curr. Opin. Microbiol. 2017. V. 36. P. 76–84.
Lopez D., Kolter R. Extracellular signals that define distinct and coexisting cell fates in Bacillus subtilis // FEMS Microbiol. Rev. 2010. V. 34. P. 134–149.
Lopez D., Vlamakis H., Losick R., Kolter R. Cannibalism enhances biofilm development in Bacillus subtilis // Mol. Microbiol. 2009. V. 74. P. 609–618.
Lyons N.A., Kraigher B., Stefanic P., Mandic-Mulec I., Kolter R. A Combinatorial kin discrimination system in Bacillus subtilis // Curr. Biol. 2016. V. 26. P. 733–742.
Mammeri N.E., Hierrezuelo J., Tolchard J., Cámara-Almirón J., Caro-Astorga J., Álvarez-Mena A., Dutour A., Berbon M., Shenoy J., Morvan E., Grélard A., Kauffmann B., Lecomte S., de Vicente A., Habenstein B., Romero D., Loquet A. Molecular architecture of bacterial amyloids in Bacillus biofilms // FASEB J. 2019. V. 33. P. 12146–12163.
Marlow V.L., Cianfanelli F.R., Porter M., Cairns L.S., Dale J.K., Stanley-Wall N.R. The prevalence and origin of exoprotease-producing cells in the Bacillus subtilis biofilm // Microbiology (SGM). 2014a. V. 160. P. 56–66.
Marlow V.L., Porter M., Hobley L., Kiley T.B., Swedlow J.R., Davidson F.A., Stanley-Wall N.R. Phosphorylated DegU manipulates cell fate differentiation in the Bacillus subtilis biofilm // J. Bacteriol. 2014b. V. 196. P. 16–27.
Martin M., Dragoš A., Otto S.B., Schäfer D., Brix S., Maróti G., Kovács Á.T. Cheaters shape the evolution of phenotypic heterogeneity in Bacillus subtilis biofilms // ISME J. 2020. https://doi.org/10.1038/s41396-020-0685-4
Marvasi M., Visscher P.T., Casillas Martinez L. Exopolymeric substances (EPS) from Bacillus subtilis: polymers and genes encoding their synthesis // FEMS Microbiol. Lett. 2010. V. 313. P. 1–9.
McLoon A.L., Kolodkin-Gal I., Rubinstein S.M., Kolter R., Losick R. Spatial regulation of histidine kinases governing biofilm formation in Bacillus subtilis // J. Bacteriol. 2011. V. 193. P. 679–685.
Mielich-Suss B., Lopez D. Molecular mechanisms involved in Bacillus subtilis biofilm formation // Environ. Microbiol. 2015. V. 17. P. 555–565.
Mielich-Suss B., Schneider J., Lopez D. Overproduction of flotillin influences cell differentiation and shape in Bacillus subtilis // MBio. 2013. V. 4. P. e00719-00713.
Milton M.E., Draughn G.L., Bobay B.G., Stowe S.D., Olson A.L., Feldmann E.A., Thompson R.J., Myers K.H., Santoro M.T., Kearns D.B., Cavanagh J. The solution structures and interaction of SinR and SinI: elucidating the mechanism of action of the master regulator switch for biofilm formation in Bacillus subtilis // J. Mol. Biol. 2020. V. 432. P. 343–357.
Mitrofanova O., Mardanova A., Evtugyn V., Bogomolnaya L., Sharipova M. Effects of Bacillus serine proteases on the bacterial biofilms // Biomed. Res. Int. 2017. V. 2017. P. 10.
Morikawa M., Kagihiro S., Haruki M., Takano K., Branda S., Kolter R., Kanaya S. Biofilm formation by a Bacillus subtilis strain that produces β-polyglutamate // Microbiology (SGM). 2006. V. 152. P. 2801–2807.
Nagorska K., Ostrowski A., Hinc K., Holland I.B., Obuchowski M. Importance of eps genes from Bacillus subtilis in biofilm formation and swarming // J. Appl. Genet. 2010. V. 51. P. 369–381.
Newman J.A., Lewis R.J. Exploring the role of SlrR and SlrA in the SinR epigenetic switch // Commun. Integr. Biol. 2013. V. 6. P. e25658.
Norman T.M., Lord N.D., Paulsson J., Losick R. Memory and modularity in cell-fate decision making // Nature. 2013. V. 503. P. 481–486.
Ogura M. Post-transcriptionally generated cell heterogeneity regulates biofilm formation in Bacillus subtilis // Genes Cells. 2016. V. 21. P. 335–349.
Ogura M., Yoshikawa H., Chibazakura T. Regulation of the response regulator gene degU through the binding of SinR/SlrR and exclusion of SinR/SlrR by DegU in Bacillus subtilis // J. Bacteriol. 2014. V. 196. P. 873–881.
Okada M., Sato I., Cho S.J., Iwata H., Nishio T., Dubnau D., Sakagami Y. Structure of the Bacillus subtilis quorum-sensing peptide pheromone ComX // Nat. Chem. Biol. 2005. V. 1. P. 23–24.
Ostrowski A., Mehert A., Prescott A., Kiley T.B., Stanley-Wall N.R. YuaB functions synergistically with the exopolysaccharide and TasA amyloid fibers to allow biofilm formation by Bacillus subtilis // J. Bacteriol. 2011. V. 193. P. 4821–4831.
Peng N., Cai P., Mortimer M., Wu Y., Gao C., Huang Q. The exopolysaccharide–eDNA interaction modulates 3D architecture of Bacillus subtilis biofilm // BMC Microbiol. 2020. V. 20. P. 115.
Penha R.O., Vandenberghe L., Faulds C., Soccol V.T., Soccol C.R. Bacillus lipopeptides as powerful pest control agents for a more sustainable and healthy agriculture: recent studies and innovations // Planta. 2020. V. 251. P. 70.
Pisithkul T., Schroeder J.W., Trujillo E.A., Yeesin P., Stevenson D.M., Chaiamarit T., Coon J.J., Wang J.D., Amador-Noguez D. Metabolic remodeling during biofilm development of Bacillus subtilis // mBio. 2019. V. 10. P. e00623-19.
Pollak S., Omer-Bendori S., Even-Tov E., Lipsman V., Bareia T., Ben-Zion I., Eldar A. Facultative cheating supports the coexistence of diverse quorum-sensing alleles // Proc. Natl. Acad. Sci. USA. 2016. V. 113. P. 2152–2157.
Raga-Carbajal E., Carrillo-Nava E., Costas M., Porras-Dominguez J., López-Munguía A., Olvera C. Size product modulation by enzyme concentration reveals two distinct levan elongation mechanisms in Bacillus subtilis levansucrase // Glycobiology. 2016. V. 26. P. 377–385.
Reverdy A., Chen Y., Hunter E., Gozzi K., Chai Y. Protein lysine acetylation plays a regulatory role in Bacillus subtilis multicellularity // PLoS One. 2018. V. 13. P. e0204687.
Romero D., Vlamakis H., Losick R., Kolter R. Functional analysis of the accessory protein TapA in Bacillus subtilis amyloid fiber assembly // J. Bacteriol. 2014. V. 196. P. 1505–1513.
Römling U., Galperin M.Y. Discovery of the second messenger cyclic di-GMP // Meth. Mol. Biol. 2017. V. 1657. P. 1–8.
Roux D., Cywes-Bentley C., Zhang Y.-F., Pons S., Konkol M., Kearns D.B. Identification of poly-N-acetylglucosamine as a major polysaccharide component of the Bacillus subtilis biofilm matrix // J. Biol. Chem. 2015. V. 290. P. 19261–19272.
Ryan-Payseur B.K., Freitag N.E. Bacillus subtilis biofilms: a matter of individual choice // mBio. 2018. V. 9. P. e02339-18.
Schafer H., Turgay K. Spx, a versatile regulator of the Bacillus subtilis stress response // Curr. Genet. 2019. V. 65. P. 871–876.
Schultz D. Coordination of cell decisions and promotion of phenotypic diversity in B. subtilis via pulsed behavior of the phosphorelay // Bioessays. 2016. V. 38. P. 440–445.
Sharipova M., Balaban N., Kayumov A., Kirillova Y., Mardanova A., Gabdrakhmanova, Leshchinskaya I., Rudenskaya G., Akimkina T., Safina D., Demidyuk I., Kostrov S. The expression of the serine proteinase gene of Bacillus intermedius in Bacillus subtilis // Microbiol. Res. 2008. V. 163. P. 39–50.
Shemesh M., Chai Y. A combination of glycerol and manganese promotes biofilm formation in Bacillus subtilis via histidine kinase KinD signaling // J. Bacteriol. 2013. V. 195. P. 2747–2754.
Shemesh M., Kolter R., Losick R. The biocide chlorine dioxide stimulates biofilm formation in Bacillus subtilis by activation of the histidine kinase KinC // J. Bacteriol. 2010. V. 192. P. 6352–6356.
Si X., Quan X., Wu Y. A Small-molecule norspermidine and norspermidine-hosting polyelectrolyte coatings inhibit biofilm formation by multi-species wastewater culture // Appl. Microbiol. Biotechnol. 2015. V. 99. P. 10861–10870.
Spacapan M., Danevčič T., Mandic-Mulec I. ComX-induced exoproteases degrade ComX in Bacillus subtilis PS-216 // Front. Microbiol. 2018. V. 9. P. 105.
Stanley N.R., Lazazzera B.A. Defining the genetic differences between wild and domestic strains of Bacillus subtilis that affect poly-γ-DL-glutamic acid production and biofilm formation // Mol. Microbiol. 2005. V. 57. P. 1143–1158.
Stover A.G., Driks A. Secretion, localization and antibacterial activity of tasA, a Bacillus subtilis spore-associated protein // J. Bacteriol. 1999. V. 181. P. 1664–1672.
Stowe S.D., Olson A.L., Losick R. Chemical shift assignments and secondary structure prediction of the master biofilm regulator, SinR, from Bacillus subtilis // J. Biomol. NMR Assign. 2014. V. 8. P. 155–158.
Townsley L., Yannarell S.M., Huynh T.N., Woodward J.J., Shank E.A. Cyclic di-AMP acts as an extracellular signal that impacts Bacillus subtilis biofilm formation and plant attachment // mBio. 2018. V. 9. P. e00341-18.
Toyofuku M., Carcamo-Oyarce G., Yamamoto T., Eisenstein F., Hsiao C.C., Kurosawa M., Gademann K., Pilhofer M., Nomura N., Eberl L. Prophage-triggered membrane vesicle formation through peptidoglycan damage in Bacillus subtilis // Nat. Commun. 2017. V. 8. P. 481.
Trejo M., Douarche C., Bailleux V., Poulard C., Mariot S., Regeard C., Raspaud E. Elasticity and wrinkled morphology of Bacillus subtilis pellicles // Proc. Natl. Acad. Sci. USA. 2013. V. 110. P. 2011–2016.
Tucker A.T., Bobay B.G., Banse A.V., Olson A.L., Soderblom E.J., Moseley M.A. A DNA mimic: the structure and mechanism of action for the antirepressor protein AbbA // J. Mol. Biol. 2014. V. 426. P. 1911–1924.
Vaccari L., Molaei M., Niepa T.H.R., Lee D., Leheny R.L., Stebe K.J. Films of bacteria at interfaces // Adv. Colloid. Interface Sci. 2017. V. 247. P. 561–572.
Veening J.W., Hamoen L.W., Kuipers O.P. Phosphatases modulate the bistable sporulation gene expression pattern in Bacillus subtilis // Mol. Microbiol. 2005. V. 56. P. 1481–1494.
Veening J.W., Igoshin O.A., Eijlander R.T., Nijland R., Hamoen L.W., Kuipers O.P. Transient heterogeneity in extracellular protease production by Bacillus subtilis // Mol. Syst. Biol. 2008. V. 4. P. 184.
Veening J.W., Kuipers O.P., Brul S., Hellingwerf K.J., Kort R. Effects of phosphorelay perturbations on architecture, sporulation, and spore resistance in biofilms of Bacillus subtilis // J. Bacteriol. 2006. V. 188. P. 3099–3109.
Velmourougane K., Prasanna R., Saxena A.K. Agriculturally important microbial biofilms: present status and future prospects // J. Basic Microbiol. 2017. V. 57. P. 548–573.
Verdugo-Fuentes A., Gastélum G., Rocha J., Torre M. Multiple and overlapping functions of quorum sensing proteins for cell specialization in Bacillus species // J. Bacteriol. 2020. V. 202. P. e00721-19.
Vidakovic L., Singh P.K., Hartmann R., Nadell C.D., Drescher K. Dynamic biofilm architecture confers individual and collective mechanisms of viral protection // Nat. Microbiol. 2018. V. 3. P. 26–31.
Vlamakis H., Chai Y., Beauregard P., Losick R., Kolter R. Sticking together: building a biofilm the Bacillus subtilis way // Nat. Rev. Microbiol. 2013. V. 11. P. 157–168.
Wagner R.M., Kricks L., Lopez D. Functional membrane microdomains organize signaling networks in bacteria // J. Membr. Biol. 2017. V. 250. P. 367–378.
Wang X., Meng S., Han J. Morphologies and phenotypes in Bacillus subtilis biofilms // J. Microbiol. 2017. V. 55. P. 619–627.
Winkelman J.T., Bree A.C., Bate A.R., Eichenberger P., Gourse R.L., Kearns D.B. RemA is a DNA-binding protein that activates biofilm matrix gene expression in Bacillus subtilis // Mol. Microbiol. 2013. V. 88. P. 984–997.
Wu Y., Quan X., Si X., Wang X. A small molecule norspermidine in combination with silver ion enhances dispersal and disinfection of multi-species wastewater biofilms // Appl. Microbiol. Biotechnol. 2016. V. 100. P. 5619–5629.
Wu Q., Xu H., Liang J., Yao J. Contribution of glycerol on production of poly(γ-glutamic acid) in Bacillus subtilis NX-2 // Appl. Biochem. Biotechnol. 2010. V. 160. P. 386–392.
Yannarell S.M., Grandchamp G.M., Chen S.Y., Daniels K.E., Shank E.A. A dual-species biofilm with emergent mechanical and protective properties // J. Bacteriol. 2019. V. 201. P. e00670-18.
Yepes A., Schneider J., Mielich B., Koch G., Garcia-Betancur J.C., Ramamurthi K.S. The biofilm formation defect of a Bacillus subtilis flotillin-defective mutant involves the protease FtsH // Mol. Microbiol. 2012. V. 86. P. 457–471.
Yu C., Li X., Zhang N., Wen D., Liu C., Li Q. Inhibition of biofilm formation by D-tyrosine: effect of bacterial type and D-tyrosine concentration // Water Res. 2016. V. 92. P. 173–179.
Yu Y., Yan F., He Y., Qin Y., Chen Y., Chai Y., Guo J.H. The ClpY-ClpQ protease regulates multicellular development in Bacillus subtilis // Microbiology (SGM). 2018. V. 164. P. 848–862.
Zeriouh H., de Vicente A., Perez-Garcia A., Romero D. Surfactin triggers biofilm formation of Bacillus subtilis in melon phylloplane and contributes to the biocontrol activity // Environ. Microbiol. 2014. V. 16. P. 2196–2211.
Zwick J.V., Noble S., Ellaicy Y.K., Coe G.D., Hakey D.J., King A.N., Sadauskas A.J., Faulkner M.J. AhpA is a peroxidase expressed during biofilm formation in Bacillus subtilis // MicrobiologyOpen. 2017. V. 6. P. e00403.
Дополнительные материалы отсутствуют.


