Микробиология, 2021, T. 90, № 2, стр. 179-190
Антибиотикорезистентные микроорганизмы и детерминанты множественной лекарственной устойчивости у бактерий рода Pseudomonas в очистных сооружениях г. Пущино
И. А. Кошелева a, Т. Ю. Измалкова a, *, О. И. Сазонова a, Т. В. Сиунова a, А. Б. Гафаров a, С. Л. Соколов a, А. М. Боронин a
a Федеральный исследовательский центр “Пущинский научный центр биологических исследований РАН”,
Институт биохимии и физиологии микроорганизмов им. Г.К. Скрябина Росийской академии наук
142290 Пущино, Московская область, Россия
* E-mail: tatiz@ibpm.pushchino.ru
Поступила в редакцию 30.06.2020
После доработки 20.10.2020
Принята к публикации 28.10.2020
Аннотация
В работе охарактеризованы антибиотикорезистентные штаммы, изолированные методом прямого высева из пяти проб, отобранных из очистных сооружений г. Пущино в апреле 2015 г. на различных стадиях очистки. Проведен первичный анализ коллекции (около 800 штаммов) на устойчивость к антибиотикам: карбенициллину, канамицину, стрептомицину, амикацину, тобрамицину, хлорамфениколу, рифампицину, гентамицину, тетрациклину, цефтазидиму, цефепиму и меропенему. Показано, что наиболее часто встречающимися антибиотикоустойчивыми микроорганизмами в очистных сооружениях г. Пущино являются бактерии родов Pseudomonas и Alcaligenes. Исследовано распространение генов резистентности к тетрациклину и показано, что в штаммах из очистных сооружений г. Пущино преобладают гены tetA/tetC, контролирующие активный вывод антибиотика из клетки. Штаммы, содержащие гены, ассоциированные с интегронами I-го типа (intI1, qacE/qacΔE1 и sul1) составили четвертую часть от исследованных образцов. У четырех штаммов псевдомонад обнаружены IncN плазмиды, семь штаммов Pseudomonas spp. содержат плазмиды группы несовместимости Р-9 (ε-подгруппы). Три IncP-9 плазмиды являются конъюгативными и несут детерминанты устойчивости к тетрациклину, стрептомицину и гентамицину одновременно, что показано впервые для ε-подгруппы IncP-9 плазмид.
Антибиотики являются широко распространенной группой лекарственных препаратов, наиболее успешно применяемой в медицине и ветеринарии. Потребление данных веществ по всему миру оценивается в сотни тысяч тонн в год. В медицине антибиотики являются третьей группой фармацевтических препаратов по частоте назначения (Puckowski et al., 2016). В ветеринарии эти соединения широко применяются не только для лечения, но и для стимуляции роста животных. Широкое применение антибиотиков в XXI в. привело к возникновению и быстрому распространению патогенов с множественной лекарственной устойчивостью, которое все чаще ставит под угрозу успешное лечение бактериальных инфекций и является глобальной проблемой в области здравоохранения, решение которой требует эффективных действий. Однако распространение резистентных патогенных микроорганизмов в клинике не может рассматриваться отдельно от процессов циркуляции детерминант устойчивости к антибиотикам в природных экосистемах. В настоящее время многие страны разрабатывают стратегию действий по предотвращению распространения резистентности к антибиотикам, которая основана на концепции неразрывной связи жизнедеятельности человека и животных с окружающей средой (Ashbolt et al., 2013). Загрязнение фармацевтическими отходами наземных и водных экосистем наблюдается повсеместно, причем порой концентрация антибиотиков в окружающей среде превышает их терапевтические уровни (Larsson et al., 2007; Li et al., 2016; Puckowski et al., 2016). Неизмененные остатки лекарственных средств попадают в больших количествах в окружающую среду вследствие физиологической экскреции людей, животных, в составе бытового фармацевтического мусора и при обработке антибиотиками сельхозугодий. Также оставшиеся без изменений исходные соединения и их биологически активные метаболиты сбрасываются в почву при использовании навоза в качестве удобрения. Распространение антибиотиков сточными водами приводит к загрязнению водной среды.
В последнее время все больше внимания уделяется очистным сооружениям, которые являются резервуарами устойчивых к антибиотикам бактерий, и, благодаря высокой плотности и разнообразию микробиоты активного ила, служат “горячими точками” горизонтального переноса, рекомбинации и распространения в окружающей среде генов антибиотикорезистентности (Dalkmann et al., 2012). Город Пущино Московской области (население около 20 тыс. чел.) не содержит на своей территории крупных производств, поэтому основным источником попадания в очистные сооружения антибиотиков и антибиотикорезистентных штаммов служат коммунально-бытовые сточные воды.
Целью данной работы было изучение культивируемых антибиотикорезистентных микроорганизмов и детерминант множественной лекарственной устойчивости в отсутствие влияния промышленных и сельхозпроизводств на примере очистных сооружений г. Пущино.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
В работе использовали бактериальные штаммы, устойчивые к антибиотикам, изолированные методом прямого высева из проб, отобранных из очистных сооружений г. Пущино на различных стадиях очистки (табл. 1).
Таблица 1.
Мультирезистентные штаммы, изолированные из очистных сооружений г. Пущино на разных этапах очистки
| Обозначение штамма* | Характеристика** | Плазмиды*** | tet-гены | intI1/sul1/qacΔE1 | Размер кассеты, п.н. |
|---|---|---|---|---|---|
| P. fragi 1C-5 | TcRKmRSmRGmR RifRCmRCazRFepR |
+ | tet A/C ++ | –/–/– | |
| Pseudomonas sp. 1Сb-36 | TcRCazRFepR | + | tet A/C ++ | +/–/– | |
| P. lundensis 1G-10 | TcRKmRSmRGmR CmRCazRFepR |
IncP-9 | tet A/C ++ | –/–/– | |
| Pseudomonas sp. 1G-5 | TcRKmRSmR GmR CmRMerR |
IncP-9 | tet A/C ++ | +/+/+ | ~700 |
| Pseudomonas sp. 1K-1 | TcRKmRSmRGmR RifRCbRCazRFepR |
Tra+ TcRSmRGmR |
tet A/C ++ | +/+/+ | |
| P. lundensis 1S-2 | TcRSmRGmRRifR | IncP-9 | tet A/C ++ | –/–/– | |
| Pseudomonas sp. 1Т-27 | TcRSmRGmRKmR | НО | – | +/–/– | |
| Pseudomonas sp. 1Т-31 | TcRSmRCazRFepR | IncN | tet A/C ++ | +/+/+ | ~1400 |
| Pseudomonas sp. 1Т-50 | TcRSmRAkRFepR | IncN | – | +/–/– | |
| P. fragi 2G-2 | TcRKmRSmRGmR CmRCazRMerRCbR |
IncP-9 | tet A/C ++ | –/–/– | |
| Pseudomonas sp. 2G-7 | TcRSmRGmR CbRMerR |
IncР-9 Tra+ TcRSmRGmR |
tet A/C ++ | –/–/– | |
| Pseudomonas sp. 2S-6 | TcRKmRSmRGmR CbRCmRCazRMerR |
Tra+ TcRSmR |
tet A/C ++ | +/+/+ | ~1500 |
| Pseudomonas sp. 2S-8 | TcRSmR GmRCmRFepR |
tet A/C ++ | –/–/– | ||
| Pseudomonas sp. 2S-9 | TcRSmRGmRCbR | IncР-9 Tra+ TcRSmRGmR |
tet A/C ++ | –/–/– | |
| Pseudomonas sp. 2Т-67 | TcRSmRGmR FepRMerR |
НО | tet A/C ++ | +/+/+ | ~1000 |
| Pseudomonas sp. 2Т-108 | TcRSmRCazR FepRMerR |
НО | tet A/C ++ | +/–/– | |
| Alcaligenes faecalis 3R-2 | ТcRGmRRif+CmR | tet A/C ++ tet Ribo ++ |
–/–/– | ||
| Pseudomonas sp. 3Сb-32 | TcRKmRSmRGmR AkRTobRFepR |
+ | – | +/+/+ | ~1000 |
| Pseudomonas sp. 3Сb-33 | KmRTobRCazRFepR | + | – | +/+/+ | ~2400 |
| Pseudomonas sp. 3Т-6 | TcRTobRCazR | ||||
| Pseudomonas sp. 3Т-35 | TcRKmRSmRGmR AkRTobRCazRFepR |
НО | – | +/+/+ | ~1000 |
| Alcaligenes faecalis 4C-8 | ТcRKmRSmRGmR Rif+CazRFepR |
tet Ribo ++ | –/–/– | ||
| Pseudomonas sp. 4Сb-25 | TcRKmRSmRGmR AkRTobRCazRFepR |
IncN | tet A/C +/– | +/+/+ | ~1200 |
| Pseudomonas sp. 4G-3 | TcRSmRGmR CbRCmR |
IncР-9 Tra+ TcRSmRGmR |
tet A/C +/– tet G ++ |
+/+/+ | ~1500 |
| Pseudomonas sp. 4S-2 | TcRKmRSmR GmRCazRFepR |
tet G ++ | +/+/+ | ~1500 | |
| Pseudomonas sp. 5Сb-1 | SmRGmRCazR FepRMerR |
IncN | – | +/+/+ | ~750 |
| Pseudomonas sp. 5Сb-24 | KmRSmRAkR FepRMerR |
+ | – | –/–/– | |
| Pseudomonas sp. 5Сb-35 | KmRSmRAkR TobR FepRMerR |
+ | – | –/–/– | |
| Pseudomonas sp. 5S-96 | TcRKmRSmRGmR CmRMerRCbR |
Tra+ SmRGmR |
tet G ++ | +/+/+ | ~1500 |
* В названии штамма первая цифра обозначает номер пробы, заглавная буква – антибиотик, на котором был изолирован штамм (С – хлорамфеникол, Cb – карбенициллин, T – тетрациклин, G – гентамицин, K – канамицин, S – стрептомицин, R – рифампицин). ** T cR, KmR – устойчивость к тетрациклину, канамицину, CbR – устойчивость к карбенициллину, SmR – устойчивость к стептомицину, CmR, GmR, RifR– устойчивость к хлорамфениколу, гентамицину, рифампицину, AkR – устойчивость к амикацину, TobR – устойчивость к тобрамицину, CazR – устойчивость к цефтазидиму, FepR – устойчивость к цефепиму, MerR – устойчивость к меропенему. *** Tra+ – способность плазмид к конъюгационному переносу; НО – плазмиды не обнаружены.
Штаммы выращивали при 28°С на богатой агаризованной среде LB (Sambrook et al., 1989) с добавлением одного из следующих антибиотиков (мкг/мл): карбенициллин – 500, цефтазидим – 40, цефепим – 40, меропенем – 40, тетрациклин – 30, канамицин – 100, стрептомицин – 100, амикацин – 30, тобрамицин – 50, гентамицин – 10, рифампицин – 100 и хлорамфеникол – 100. Концентрации антибиотиков подбирали на основании базы данных Европейского комитета по тестированию чувствительности к антибиотикам EUCAST (http:// www.eucast.org) с учетом того, что конечная концентрация антибиотика в среде должна быть выше минимальной ингибирующей концентрации и обеспечивать устойчивый рост резистентных штаммов. Общую численность культивируемых микроорганизмов (КОЕ/мл образца) для каждой пробы определяли путем прямого подсчета колоний, выросших на богатой агаризованной среде LB без добавления антибиотика; численность антибиотикоустойчивых микроорганизмов также определяли прямым подсчетом выросших колоний. Эксперименты проводили в трех повторностях. Для селекции штаммов, относящихся к роду Pseudomonas, использовали среду PIA – pseudomonas isolation agar (“Difco”, США); среду King B (“Pronadisa Conda”, Испания) использовали для выявления псевдомонад флуоресцирующей группы.
Идентификацию выделенных микроорганизмов проводили по протоколу фирмы-изготовителя, используя систему MALDI Biotyper (матрично-активированная лазерная десорбция/ионизация), основанную на сравнении масс-спектров рибосомальных белков исследуемого штамма с базой спектров референсных микроорганизмов известных видов (“Bruker Daltonics”, Германия). На основании полученных оценочных величин выше 2.000 (2.074–2.368) выделенные нами микроорганизмы были определены до вида, в диапазоне 1.845–1.921 – до рода. Пробы для анализа готовили согласно (De Bruyne et al., 2011).
Геномную ДНК бактерий выделяли с использованием AxyPrep Bacterial Genomic DNA MiniPrep Kit по протоколу фирмы-изготовителя (“Axygen Biosciences”, США). Плазмидную ДНК выделяли методом щелочного лизиса (Sambrook et al., 1989). Концентрацию ДНК определяли на флуориметре TKO-100 фирмы “Hoefer Scientific Instruments” (США) с красителем Hoechst 33258 (“Bio-Rad”, США) согласно протоколу фирмы-изготовителя.
Полимеразную цепную реакцию (ПЦР) осуществляли в циклере GeneAmp PCR System 9700 (“Applied Biosystems”, США). Реакцию проводили в стандартных условиях, при конечной концентрации дезоксирибонуклеотидтрифосфатов 200 мкМ, 1.5 мМ или 2 мМ MgCl2 и, в некоторых случаях, 5% диметилсульфоксида (ДМСО) (“Sigma”, США). Олигонуклеотидные праймеры, использованные в работе, представлены в табл. 2.
Таблица 2.
ПЦР праймеры, использованные в работе
| Ген | Праймеры | Нуклеотидная последовательность | Размер ПЦР продукта, п.н. |
Ссылка |
|---|---|---|---|---|
| intI1 | intI1F intI1R |
5'-CCTCCCGCACGATGATC-3' 5'-TCCACGCATCGTCAGGC-3' |
280 | Kraft et al., 1986 |
| sul1 | Sul1F Sul1B |
5'-CTTCGATGAGAGCCGGCGGC-3' 5'-GCAAGGCGGAAACCCGCGCC-3' |
437 | Sandvang et al., 1997 |
| qacΔE1 | qacΔE1F qacΔE1B |
5'-ATCGCAATAGTTGGCGAAGT-3' 5'-CAAGCTTTTGCCCATGAAGC-3' |
228 | |
| Генная кассета интегрона | 5_-CS: intIF 3_-CS: intIB |
5'-GGCATCCAAGCAGCAAGC-3' 5'-AAGCAGACTTGACCTGAT-3' |
Вариабельный | |
| sul2 | Sul2-F Sul2-B |
5'-GCGCTCAAGGCAGATGGCATT-3' 5'-GCGTTTGATACCGGCACCCGT-3' |
293 | Kerrn et al., 2002 |
| tet A,C | tetAC-150f tetAC-716r |
5'-GCTRTATGCGTTGRTGCAAT-3' 5'-TCCTCGCCGAAAATGACC-3' |
567 | Yu et al., 2005 |
| tet G | tetG-247f tetG-678r |
5'-GTCGATTACACGATTATGGC-3' 5'-CACTTGGCCGATCAGTTGA-3' |
432 | |
| tet M, O, P, Q, S, T, W |
Ribo2-FW Ribo2-RV |
5'-GGMCAYRTGGATTTYWTIGC-3' 5'-TCIGMIGGIGTRCTIRCIGGRC-3' |
1315 | |
| IncN (rep) | IncN-rep-1 IncN-rep-2 |
5'-AGTTCACCACCTACTCGCTCCG-3' 5'-CAAGTTCTTCTGTTGGGATTCCG-3' |
165 | Götz et al., 1996 |
| IncQ (oriV) | IncQ-oriV-1 IncQ-oriV-2 |
5'-CTCCCGTACTAACTGTCACG-3' 5'-ATCGACCGAGACAGGCCCTGC-3' |
436 | |
| IncW (oriW) |
IncW-oriV-1 IncW-oriV-2 |
5'-GACCCGGAAAACCAAAAATA-3' 5'-GTGAGGGTGAGGGTGCTATC-3' |
1140 | |
| IncP-1 trfA1 |
trfA1-1 trfA1-2 |
5'-ATGACGACCAAGAAGCG-3' 5'-AACCCCCAGCCGGAACTG-3' |
889 | |
| IncP-9 repAB | repF repR |
5'-CCAGCGCGGTACWTGGG-3' 5'-GTCGGCAICTGCTTGAGCTT-3' |
554 | Greated, Thomas, 1999 |
| IncP-7 rep-область |
Upper Lower |
5'-CCCTATCTCACGATGCTGTA-3' 5'-GCACAAACGGTCGTCAG-3' |
524 | Измалкова и соавт., 2005 |
Электрофорез ДНК проводили в 1–2.5% агарозе в 0.5× Трис-боратном буфере по стандартной методике (Sambrook et al., 1989). Визуализацию ДНК проводили путем окрашивания геля в растворе бромистого этидия. ДНК очищали из геля, используя AxyPrepTM DNA Gel Extraction Kit (“Axygen”, США), по протоколу фирмы-изготовителя.
В работе использовали ферменты и буферные растворы компании “Thermo Fisher Scientific” (США). Все процедуры проводили в соответствии с рекомендациями фирмы-производителя.
Конъюгационный перенос плазмид в реципиентный штамм P. putida КТ2442 (gfpKmR) осуществляли на мембранных фильтрах на агаризованной среде LB. Культуры донорных и реципиентных бактерий, выращенных на среде LB и находящихся в логарифимичекой фазе роста, смешивали в соотношении 1 : 1 и наносили на нитроцеллюлозные фильтры (Synpor 6, размер пор 0.45 мкм, Чехия), помещенные на поверхность полноценной агаризованной среды. Бактерии инкубировали 12–18 ч при 30°С. Выросшие клетки смывали физиологическим раствором и высевали на селективные среды. Трансконъюганты тестировали на присутствие плазмид и гена gfp.
Нуклеотидную последовательность ампликонов определяли c помощью генетического анализатора ABI 3130 xl Analysis System (“Applied Biosystems”, США) по протоколу фирмы-производителя. Анализ идентичности нуклеотидных последовательностей осуществляли при помощи программы BLASTN (http://blast.ncbi.nlm.nih.gov/).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Изоляция и идентификация микроорганизмов, устойчивых к антибиотикам. В апреле 2015 г. из Пущинских очистных сооружений было отобрано 5 проб для выделения и анализа микроорганизмов, устойчивых к антибиотикам. В состав очистных сооружений входят: решетки, песколовки, первичные отстойники, аэротенки, вторичные отстойники, система обеззараживания с помощью гипохлорита натрия, контактные резервуары, иловые площадки. Качество очистки сточных вод соответствует нормативу сброса сточных вод в водоемы рыбохозяйственного назначения (https://www.pushchino-tvk.ru/). Проба № 1 была отобрана из песколовки и представляла собой мутную, сильно загрязненную жидкость. Пробу № 2 отбирали из канала, по которому очищаемые стоки попадают в первичные отстойники. Проба № 3 была взята из аэротенка, проба № 4 – из вторичных отстойников. Проба № 5 отобрана после последнего этапа очистки и представляла собой прозрачную жидкость. Высевы из проб производили на богатую агаризованную среду (LA), содержащую один из следующих антибиотиков, относящихся к разным группам: карбенициллин (β-лактамные пенициллины); канамицин, стрептомицин и гентамицин (аминогликозиды); тетрациклин (тетрациклины); хлорамфеникол (левомицетин); рифампицин (ансамицины), в концентрациях, указанных в разделе “Материалы и методы исследования”.
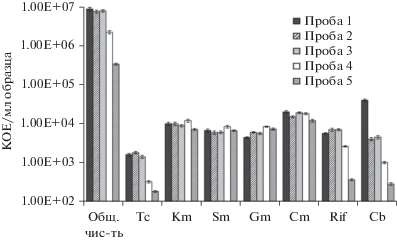
Общая численность культивируемых микроорганизмов (КОЕ/мл образца) для каждой пробы, а также численность микроорганизмов, полученных методом прямого высева на индивидуальных антибиотиках, представлена на рис. 1. Обнаружено, что если для одних антибиотикоустойчивых микроорганизмов обработка сточных вод приводила к значительному снижению численности, то для микроорганизмов, устойчивых к канамицину, стрептомицину и хлорамфениколу, общая численность была сходной во всех пяти пробах и составляла в среднем 103–104 клеток/мл образца (рис. 1). Небольшой рост, наблюдаемый в случае гентамицина, вероятно, связан с участием мобильных генетических элементов в распространении GmR-фенотипа (Schluter et al., 2007). Наиболее эффективно очистка сточных вод снижала количество микроорганизмов, устойчивых к карбенициллину, рифампицину и тетрациклину. Данный факт может быть связан с разложением молекулы антибиотика в процессе очистки сточных вод, как в случае β-лактамов (Chaturvedi et al., 2021), с деградацией микробным консорциумом (Chaturvedi et al., 2021), а также с элиминацией генов антибиотикорезистентности, что было показано для tet-генов (Wen et al., 2016).
Рис. 1.
Общая численность микроорганизмов (КОЕ/мл образца) и общая численность антибиотикоустойчивых микроорганизмов (КОЕ/мл образца), изолированных методом прямого высева из пяти проб очистных сооружений г. Пущино на разных стадиях очистки. Планки погрешностей соответствуют стандартному отклонению от средних значений измерений в трех повторностях (4–10% в зависимости от степени загрязненности пробы). Обозначения: Tc – тетрациклин, Km – канамицин, Sm – стрептомицин, Rif – рифампицин, Gm – гентамицин, Cm – хлорамфеникол, Cb – карбенициллин.
Выросшие на индивидуальных антибиотиках микроорганизмы (приблизительно по 100 резистентных штаммов c каждого антибиотика) методом перекрестных реплик проверялись на устойчивость к остальным вышеуказанным антибиотикам, а также к тобрамицину и амикацину (аминогликозиды II-го и III-го поколения соответственно); цефтазидиму и цефепиму (β-лактамные цефалоспорины III-го и IV-го поколения соответственно) и меропенему (β-лактамные карбапенемы). Всего было проверено 784 штамма. Среди общего числа перекрестно проверенных штаммов, 66% были устойчивы к карбенициллину, 45% – к канамицину, 48% – к стрептомицину, 41% – к амикацину, 29% – к тобрамицину, 35% – к хлорамфениколу, 33% – к рифампицину, 38% – к гентамицину, 15% – к тетрациклину. Наименьшее количество штаммов оказалось устойчиво к цефалоспоринам III и IV поколения (10% к цефтазидиму и 8% к цефепиму) и карбапенемам (2% к меропенему). Большинство исследованных штаммов (65%) оказались мультирезистентными и обладали устойчивостью к трем (21%) и более (до девяти – 1.6%) антибиотикам. В среднем 40% устойчивых изолятов росли на селективной среде для выделения псевдомонад, 18.5% флуоресцировали на среде King B. Всего было получено 167 фенотипов резистентности из 784 исследуемых штаммов.
Некоторые штаммы, устойчивые к трем и более антибиотикам (96 фенотипов резистентности), идентифицировали с использованием масс-спектрометрической системы MALDI Biotyper. Наиболее часто встречающимися культивируемыми антибиотикоустойчивыми микроорганизмами в очистных сооружениях г. Пущино были бактерии родов Pseudomonas (53% идентифицированных штаммов) и Alcaligenes (8%). Также были обнаружены представители родов Aeromonas и Serratia. Наиболее интересные штаммы представлены в табл. 1. Полученные данные не отражают реальный видовой состав популяции, тем не менее, они являются предварительной иллюстрацией встречаемости культивируемых форм резистентных микроорганизмов. В дополнение стоит отметить, что при анализе сезонных колебаний микробных сообществ очистных сооружений провинции Чжэцзян восточного Китая методом высокопроизводительного секвенирования V3–V4 гипервариабельных участков генов 16S рРНК показано высокое содержание псевдомонад (15.71%) именно в весенних пробах (Zhang et al., 2018).
Исследование разнообразия генов устойчивости к тетрациклину (tet) у резистентных микроорганизмов. Тетрациклины представляют собой группу соединений, к которой относятся как природные (тетрациклин, хлортетрациклин, окситетрациклин), так и полусинтетические (доксициклин, миноциклин) антибиотики. В настоящее время антибиотики тетрациклинового ряда, благодаря их высокой биологической активности, являются одними из наиболее широко применяемых препаратов в здравоохранении, сельском хозяйстве, животноводстве и рыбоводстве. Поскольку тетрациклины плохо усваиваются в организме человека и животных, их значительная часть попадает в сточные воды с отходами жизнедеятельности, провоцируя рост численности резистентных бактерий (Li et al., 2016). Штаммы, изолированные из очистных сооружений г. Пущино, методом ПЦР тестировали на наличие tet-генов, отвечающих за различные механизмы резистентности. По последним данным известно 62 гена, контролирующих резистентность микроорганизмов к тетрациклину (Roberts, 2020). Основными механизмами, обеспечивающими устойчивость к тетрациклину является активный вывод (эффлюкс) антибиотика из клеток и механизм защиты рибосомы, поскольку в основе антибактериального действия тетрациклинов лежит подавление синтеза белка за счет ингибирования связывания аминоацил-тРНК с А-участком 30S субъединицы бактериальной рибосомы (Nguyen et al., 2014). Среди исследованных к настоящему времени tet-генов, гены, контролирующие эффлюкс-систему, в частности, ген tetA, являются преобладающими и представлены 35 классами (Nguyen et al., 2014; Roberts, 2020). В качестве маркерных генов в данной работе мы использовали гены tetA, tetC и tetG, контролирующие активный вывод тетрациклина из клеток, а также группу генов tetRibo: tetM, tetO, tetB(P), tetQ, tetS, tetT и tetW, кодирующих белки, защищающие рибосому (Yu et al., 2005). Показано, что гены tetA/tetC, контролирующие активный вывод антибиотика из клетки, являются наиболее распространенными в штаммах из очистных сооружений г. Пущино, тогда как гены, обеспечивающие защиту рибосомы, встречаются значительно реже и обнаружены только у представителей Alcaligenes faecalis, которые составляют всего 8% от исследованных штаммов (табл. 1). Следует отметить, что в то время как одни авторы считают гены эффлюкс-систем устойчивости к тетрациклинам преобладающими в сточных водах или активном иле, отмечая их более быстрое распространение в микробной популяции (Zang et al., 2011; Yu et al., 2015), другие обнаруживают доминирование генов, кодирующие защиту рибосомы, в частности, tetW и tetM (Wen et al., 2016; Svobodova et al., 2018). Возможно, это связано как с видовым составом микробных сообществ (например, самая большая группа тетрациклиновых эффлюкс-помп TetA–TetL чаще встречается у грамотрицательных бактерий), локализацией tet-генов в составе мобильных элементов, например, плазмид, так и с присутствующими в среде антибиотиками. Гены активного вывода, как правило, обеспечивают резистентность к тетрациклину, но гораздо менее эффективны в отношении антибиотиков второго поколения, таких как доксициклин и миноциклин, и практически не обеспечивают устойчивость к третьему поколению глициклинов (Nguyen et al., 2014).
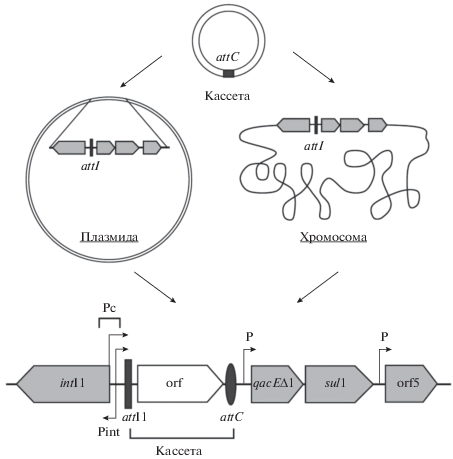
Анализ штаммов, устойчивых к антибиотикам на наличие интегронов класса 1. Ассоциация генов антибиотикорезистентности с мобильными генетическими элементами – плазмидами, транспозонами и интегронами, обеспечивает их быстрое распространение в микробной популяции. Ведущая роль в этом процессе принадлежит интегронам, которые представляют собой природные системы горизонтального переноса генов. Всего выделяют 5 классов интегронов на основании гомологии последовательностей кодируемых ими интеграз, три из которых могут нести гены устойчивости к антибиотикам (Rowe-Magnus, Mazel, 2002). Однако большинство интегронов антибиотикорезистентности относят к классу 1. Интегроны I-го типа включают два концевых региона, называемых константными последовательностями (constant sequences, CS), и высоковариабельный центральный участок. В 5'-CS, обычно находятся ген интегразы (intI), сайт рекомбинации кассеты (attI), и промотор, с которого экспрессируются гены кассеты. Кассета представляет собой сегмент ДНК, содержащий открытые рамки считывания (ОРС) без промотора и уникальный рекомбинационный сайт attC (57–141 п.н.). В 3'-CS интегрона I-го типа находится чаще делетированный или полноразмерный вариант гена устойчивости к четвертичным аммониевым соединениям (qacE/qacΔE1), ген резистентности к сульфониламидам (sul1), и ген с неустановленной функцией (orf5) (рис. 2). Дополнительные факторы, такие как внедрение в состав транспозонов (часто ассоциированы с Tn21-семейством) и использование плазмид с широким кругом хозяев, позволяют интегронам множественной лекарственной устойчивости эффективно распространяться среди микроорганизмов различных систематических групп (Partridge et al., 2018). Быстрое изменениe количества резистентных бактерий, содержащих интегроны I-го типа, в популяции в ответ на внешнее воздействие позволяет использовать ген интегразы, intI1, в качестве маркера антропогенного загрязнения антибиотиками (Gillings et al., 2014).
Рис. 2.
Роль мобильных генетических элементов (МГЭ) в распространении множественной лекарственной устойчивости. Схема интегрона резистентности класса 1. Кассеты встраиваются в вариабельную область интегрона путем сайт-специфической рекомбинации. Интегрированная кассета состоит из генов и сайта рекомбинации attC. intI1 – ген интегразы; qacEΔ1 – ген устойчивости к четвертичным аммонийным соединениям; sul1 – ген устойчивости к сульфонамидам; orf5 – ген с неизвестной функцией. Рисунок создан по материалам Partridge et al., 2018.
Коллекцию тестировали методом ПЦР в стандартных условиях на присутствие генов, ассоциированных с интегронами I-го типа: гена интегразы (intI1), генов устойчивости к четвертичным аммонийным соединениям (qacE/qacΔE1) и гена резистентности к сульфониламидам (sul1), который часто находится в 3'-консервативной области. Штаммы, содержащие гены intI1, qacE/qacΔE1 и sul1, составили четвертую часть от исследованных образцов. Двенадцать исследованных штаммов содержали кассеты генов резистентности к антибиотикам размером от 500 до 2400 п.н. (табл. 1).
Роль плазмид в распространении антибиотикорезистентности в очистных сооружениях г. Пущино. Плазмиды, генетические внехромосомные элементы, способные к автономной репликации, являются идеальным транспортным средством для горизонтального переноса генов антибиотикорезистентности, как обособленных, так и в составе транспозонов и интегронов. Часто генетические детерминанты резистентности к различным антибиотикам могут локализоваться на одной и той же плазмиде. Плазмиды с мультирезистентностью обычно имеют размер более 50 т.п.н. и являются конъюгативными. Для доказательства плазмидной локализации генов резистентности проводили конъюгационные скрещивания 43 штаммов из очистных сооружений г. Пущино, одновременно устойчивых к стрептомицину и тетрациклину, с использованием в качестве реципиента P. putida KT2442. В результате скрещиваний были получены 27 трансконъюгантов при отборе на селективной среде, содержащей стрептомицин и 16 – на среде с тетрациклином. “Перекрестная” проверка позволила отобрать 5 трансконъюгантов, одновременно устойчивых к стрептомицину и к тетрациклину. Таким образом, штаммы Pseudomonas sp. 1K-1, 2G-7, 2S-6, 2S-9, 4G-3 содержат плазмиды резистентности, по крайней мере, к этим двум антибиотикам. Проверка трансконъюгантов на устойчивость к остальным антибиотикам показала, что плазмиды в штаммах Pseudomonas sp. 1K-1, 2G-7, 2S-9, 4G-3 также несут гены устойчивости к гентамицину (табл. 1).
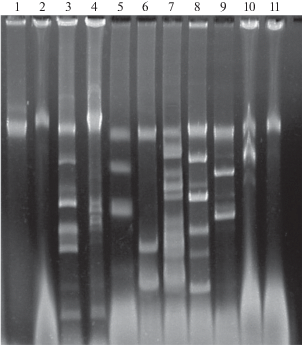
Поскольку большинство выделенных изолятов являются мультирезистентными, эксперименты по конъюгационному переносу для них связаны со сложностью подбора реципиентов, содержащих факторы для контрселекции. Около 40 штаммов, изолированных из очистных сооружений г. Пущино, тестировали на наличие плазмид методом щелочного лизиса. Оказалось, что ряд штаммов (в частности, 1С-36, 1Т-31, 1Т-50, и 5С-24) содержат плазмиды (50 т.п.н. и более), которые потенциально могут быть конъюгативными и нести детерминанты антибиотикорезистентности, а некоторые (например, 1Т-31, 1Т-50, 3С-32, 3С-33, 3Т-6, 4С-25, 5С-24 и 5С-35) являются мультиплазмидными и содержат от 2 до 5 плазмид (рис. 3). В некоторых штаммах плазмид не обнаружено, однако это может быть обусловлено ограничениями метода выделения плазмидной ДНК.
Рис. 3.
Плазмидный профиль штаммов, изолированных из очистных сооружений г. Пущино: 1 –1Cb-36, 2 – 1T-27, 3 – 1T-31, 4 – 1T-50, 5 – 4Cb-25, 6 – 5Cb-24, 7 – 5Cb-35, 8 – 3Cb-32, 9 – 3Cb-33, 10 – 3T-6, 11 – 3T-35.
Также исследуемые штаммы тестировали методом ПЦР с праймерами, специфичными для репликонов, характерных для плазмид антибиотикорезистентности с широким кругом хозяев (IncP-1, IncP-9, IncP-4 (IncQ), IncW и IncN) и In-cP-7 (табл. 1). Несмотря на то, что плазмиды группы несовместимости P-1 считаются одними из основных носителей генов резистентности и их часто обнаруживают не только в очистных сооружениях, но и в прошедшей очистку воде (Schluter A. et al., 2007), в штаммах из очистных сооружений города Пущино плазмид данной группы обнаружено не было. Также не было обнаружено плазмид IncP-4 (IncQ), IncP-7 и IncW. У четырех штаммов Pseudomonas sp. 1Т-31, 1Т-50, 4Сb-25, 5Сb-1 обнаружены IncN плазмиды. Плазмиды группы несовместимости N, несмотря на относительно небольшой размер (около 50 т.п.н.), являются конъюгативными и часто содержат транспозоны или интегроны класса 1 (Partridge et al., 2018). В составе ряда IncN плазмид были обнаружены гены резистентности к цефалоспоринам III и IV поколений, а также карбапенемам (blaKPC, blaCTX-M и blaIMP) (Caratolli et al., 2013). Штаммы Pseudomonas sp. 1Т-31, 1Т-50, 4Сb-25, 5Сb-1 устойчивы к цефтазидиму и/или к цефепиму, а 5Сb-1 также и к меропенему, однако связано ли это с наличием в штаммах IncN плазмид еще предстоит выяснить.
Семь штаммов Pseudomonas spp., изолированных на разных стадиях очистки (P. lundensis 1G-10, Pseudomonas sp. 1G-5, P. lundensis 1S-2, P. fragi 2G-2, Pseudomonas sp. 2G-7, Pseudomonas sp. 2S-9, Pseudomonas sp. 4G-3), содержат плазмиды группы несовместимости Р-9 (табл. 1). Эксперименты по конъюгационному переносу в рецепиентный штамм P. putida KT2442 и ПЦР-анализ полученных трансконъюгантов показали, что, по крайней мере, три IncP-9 плазмиды (p2G-7, p2S-9 и p4G-3) являются конъюгативными и несут детерминанты устойчивости к тетрациклину (tetA/tetC), стрептомицину и гентамицину. Плазмида p4G-3 также содержит в своем составе интегрон класса 1, размер кассеты составляет около 1500 п.н. (табл. 1). Вы-деление плазмидной ДНК показало, что трансконъюганты действительно содержат Р-9 плазмиды размером около 100 т.п.н. Плазмиды биодеградации IncP-9 группы широко распространены среди почвенных псевдомонад, однако плазмид резистентности этой группы описано крайне мало.
Анализ rep-генов плазмид антибиотикорезистентности IncP-9 группы. Невозможность сосуществования в одной клетке плазмид со сходной организацией областей, ответственных за репликацию и стабильное поддержание (так называемого базового репликона), послужило основой для традиционной системы классификации плазмид по группам несовместимости. К настоящему времени у псевдомонад описаны плазмиды, принадлежащие к 14 различным группам, 13 из которых связаны с распространением детерминант устойчивости к антибиотикам (Boronin, 1992). Представителей IncР-9 группы, помимо фенотипического разнообразия, характеризует вариабельность организации областей инициации репликации. В настоящее время известно по крайней мере 9 подгрупп IncP-9, которые выделены на основании анализа rep-генов и oriV-сайтов (α, β, γ, δ, ε, ζ, η, θ и ι) (Sevastsyanovich et al., 2008). Для определения места плазмид антибиотикорезистентности из очистных сооружений г. Пущино в системе классификации IncР-9, были определены нуклеотидные последовательности ампликонов rep-генов штаммов P. lundensis 1G-10 (MK820708), Pseudomonas sp. 1G-5 (MK820707), P. lundensis 1S-2 (MK820709), P. fragi 2G-2e (MK820704), Pseudomonas sp. 2G-7 (MK820706), Pseudomonas sp. 2S-9 (MK820710), Pseudomonas sp. 4G-3 (MK820705).
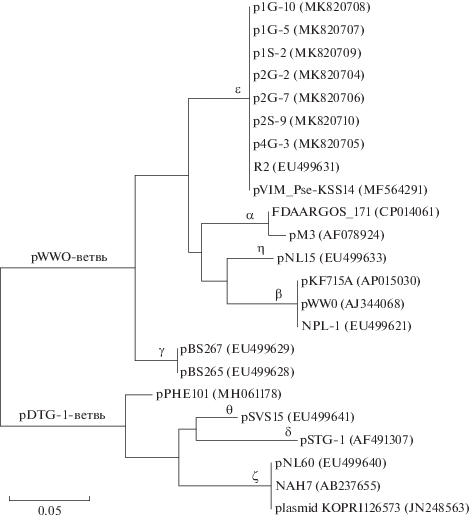
Результаты кластерного анализа нуклеотидных последовательностей rep-генов плазмид группы несовместимости Р-9, принадлежащим к различным подгруппам, который проводился при помощи программы MEGA (версия 7.0), показали, что IncP-9 плазмиды из очистных сооружений г. Пущино относятся к ε-подгруппе (рис. 4). Данная подгруппа включает плазмиды антибиотикорезистентности и принадлежит к ветви pWWO, куда относятся плазмиды устойчивости к антибиотикам подгрупп α, ε и η, а также плазмиды деградации ксенобиотиков (подгруппы β и γ). Нуклеотидные последовательности ПЦР фрагментов rep-генов IncP-9 плазмид из очистных сооружений г. Пущино полностью идентичны последовательности rep-гена мультирезистентной плазмиды pVIM_Pse-KSS14 штамма P. putida KSS14 (MF564291), несущей детерминанты устойчивости в аминогликозидам (aacA4 и aadA1), карбапенемам (blaVIM-2) и фосфомицину (fosE) (Hong et al., 2018).
Рис. 4.
Филогенетическое древо, иллюстрирующее эволюционные взаимоотношения между областями rep-генов плазмид группы несовместимости Р-9, принадлежащим к различным подгруппам (α, β, γ, δ, ε, ζ, η, θ). Относительные дистанции и конечный график построены при помощи программы MEGA (версия 7.0), метод максимального правдоподобия, алгоритм Neighbor-Joining (ближайших соседей). Шкала соответствует оценке эволюционного расстояния в пять замен на 100 нуклеотидов.
Принадлежность плазмиды к определенной группе несовместимости определяет не только круг ее бактериальных хозяев и потенциал распространения в определенных бактериальных популяциях, но и возможность совмещения различных признаков в одном штамме, а следовательно, косвенно обуслoвливает адаптивные возможности микроорганизмов к различным условиям окружающей среды. Нами впервые выделены IncP-9ε плазмиды резистентности к тетрациклину, стрептомицину и гентамицину, что говорит о широком распространении плазмид резистентности IncP-9ε и недооценке их вклада в распространение генов множественной лекарственной устойчивости. Полученные данные расширяют наши представления о роли IncP-9 плазмид в переносе и распространении генов устойчивости к антибиотикам. Обнаружение мультирезистентных штаммов, интегронов I-го типа и плазмид с широким кругом хозяев указывает на высокую скорость горизонтального переноса генов устойчивости к антибиотикам на всех стадиях очистки в очистных сооружениях г. Пущино и свидетельствует о потенциальной опасности увеличения детерминант антибиотикорезистентности в водной среде и необходимости мониторинга микробных сообществ водных экосистем.
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВНастоящая статья не содержит результатов исследований, в которых в качестве объектов использовались люди или животные.
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВНастоящая статья не содержит результатов исследований, в которых в качестве объектов использовались люди или животные.КОНФЛИКТ ИНТЕРЕСОВАвторы заявляют отсутствие конфликта интересов.
Список литературы
Измалкова Т.Ю., Сазонова О.И., Соколов С.Л., Кошелева И.А., Боронин А.М. Плазмиды биодеградации нафталина и салицилата Р-7 группы несовместимости в штаммах флуоресцирующих псевдомонад // Микробиология. 2005. Т. 74. С. 342–348.
Izmalkova T.Yu., Sazonova O.I., Sokolov S.L., Kosheleva I.A., Boronin A.M. The P-7 incompatibility group plasmids responsible for biodegradation of naphthalene and salicylate in fluorescent pseudomonads // Microbiology (Moscow). 2005. V. 74. P. 290–295.
Ashbolt N.J., Amézquita A., Backhaus T., Borriello P., Brandt K.K., Collignon P., Coors A., Finley R., Gaze W.H., Heberer T., Lawrence J.R., Larsson D.G., McEwen S.A., Ryan J.J., Schönfeld J., Silley P., Snape J.R., Van den Eede C., Top E. Human Health Risk Assessment (HHRA) for environmental development and transfer of antibiotic resistance // Environ. Health Perspect. 2013. V. 121. P. 993–1001.
Boronin A.M. Diversity of Pseudomonas plasmids: to what extent? // FEMS Microbiol Lett. 1992. V. 79. P. 461–467.
Carattoli A. Plasmids and the spread of resistance // Int. J. Med. Microbiol. 2013. V. 303. P. 298–304.
Chaturvedi P., Giri B.S., Shukla P., Gupta P. Recent advancement in remediation of synthetic organic antibiotics from environmental matrices: Challenges and Perspective // Bioresour. Technol. 2021. V. 319. 124161.
Dalkmann P., Broszat M., Siebe C., Willaschek E., Sakinc T., Huebner J., Amelung W., Grohmann E., Siemens J. Accumulation of pharmaceuticals, Enterococcus, and resistance genes in soils irrigated with wastewater for zero to 100 years in central Mexico // PLoS One. 2012. V. 7. e45397.
De Bruyne K., Slabbinck B., Waegeman W., Vauterin P., De Baets B., Vandamme P. Bacterial species identification from MALDI-TOF mass spectra through data analysis and machine learning // Syst. Appl. Microbiol. 2011. V. 34. P. 20–29.
Gillings M.R., Gaze W.H., Pruden A., Smalla K., Tiedje J.M., Zhu Y.G. Using the class 1 integron-integrase gene as a proxy for anthropogenic pollution // ISME J. 2015. V. 9. P. 1269–1279.
Götz A., Pukall R., Smit E., Tietze E., Prager R., Tschäpe H., van Elsas J.D., Smalla K. Detection and characterization of broad-host-range plasmids in environmental bacteria by PCR // Appl. Environ. Microbiol. 1996. V. 62. P. 2621–2628.
Greated A., Thomas C.M. A pair of PCR primers for IncP-9 plasmids // Microbiology (SGM). 1999. V. 145. P. 3003–3004.
Hong J.S., Yoon E.-J., Song W., Seo Y.B., Shin S., Park M.-J., Jeong S.H., Lee K. Molecular characterization of Pseudomonas putida group isolates carrying blaVIM-2 disseminated in a university hospital in Korea // Microb. Drug Resist. 2018. V. 24. P. 627–634.
Kraft C.A., Timbury M.C., Platt D.J. Distribution and genetic location of Tn7 in trimethoprim-resistant Escherichia coli // J. Med.Microbiol. 1986. V. 22. P. 125–131.
Kerrn M.B., Klemmensen T., Frimodt-Möller N., Espersen F. Susceptibility of Danish Escherichia coli strains isolated from urinary tract infections and bacteraemia, and distribution of sul genes conferring sulphonamide resistance // J. Antimicrob. Chemother. 2002. V. 50. P. 513–516.
Larsson D.G.J., de Pedro C., Paxeus N. Effluent from drug manufactures contains extremely high levels of pharmaceuticals // J. Hazard. Mater. 2007. V. 148. P. 751–755.
Li J., Zhang H., Chen Y., Luo Y., Zhang H. Sources identification of antibiotic pollution combining land use information and multivariate statistics // Environ. Monit. Assess. 2016. V. 188. P. 430.
Nguyen F., Starosta A.L., Arenz S., Sohmen D., Dönhöfer A., Wilson D.N. Tetracycline antibiotics and resistance mechanisms // Biol. Chem. 2014. V. 395. P. 559–575.
Partridge S.R., Kwong S.M., Firth N., Jensen S.O. Mobile genetic elements associated with antimicrobial resistance // Clin. Microbiol. Rev. 2018. V. 31. P. 1–61.
Puckowsk A., Mioduszewska K., Łukaszewicz P., Borecka M., Caban M., Maszkowska J., Stepnowsk P. Bioaccumulation and analytics of pharmaceutical residues in the environment: A review // J. Pharm. Biomed. Anal. 2016. V. 127. P. 232–255.
Roberts M.C. Mechanism of resistance for characterized tet and otr genes. http://faculty.washington.edu/marilynr/tetweb1.pdf (modified: Feb 2020).
Rowe-Magnus D.A., Mazel D. The role of integrons in antibiotic resistance gene capture // Int. J. Med. Microbiol. 2002. V. 292. P. 115–125.
Sambrook J., Fritsch E.F., Maniatis T. Molecular Cloning: A Laboratory Manual. 2nd ed. Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press, 1989.
Sandvang D., Aarestrup F.M., Jensen L.B. Characterisation of integrons and antibiotic resistance genes in Danish multiresistant Salmonella enterica Typhimurium DT104 // FEMS Microbiol. Lett. 1998. V. 160. P. 37–41.
Schluter A., Szczepanowski R., Puhler A., Top E.M. Genomics of IncP-1 antibiotic resistance plasmids isolated from wastewater treatment plants provides evidence for a widely accessible drug resistance gene pool // FEMS Microbiol. Rev. 2007. V. 31. P. 449–477.
Sevastsyanovich Y.R., Krasowiak R., Bingle L.E.H., Haines A.S., Sokolov S.L., Kosheleva I.A., Leuchuk A.A., Titok M.A., Smalla K., Thomas C.M. Diversity of IncP-9 plasmids of Pseudomonas // Microbiology (SGM). 2008. V. 154. P. 2929–2941.
Svobodová K., Semerád J., Petráčková D., Novotný Č. Antibiotic resistance in czech urban wastewater treatment plants: microbial and molecular genetic characterization // Microb. Drug Resist. 2018. V. 24. P. 830–838.
Wen Q., Yang L., Duan R., Chen Z. Monitoring and evaluation of antibiotic resistance genes in four municipal wastewater treatment plants in Harbin, Northeast China // Environ. Pollut. 2016. V. 212. P. 34–40.
Yu J., Liu D., Li K. Influence of tetracycline on tetracycline-resistant heterotrophs and tet genes in activated sludge process // Curr. Microbiol. 2015. V. 70. P. 415–422.
Yu Z., Michel F.C., Hansen G., Jr., Wittum Th., Morrison M. Development and application of real-time PCR assays for quantification of genes encoding tetracycline resistance // Appl. Environ. Microbiol. 2005. V. 71. P. 6926–6933.
Zhang B., Yu Q., Yan G., Zhu H., Xu X. Yang, Zhu L. Seasonal bacterial community succession in four typical wastewater treatment plants: correlations between core microbes and process performance // Sci. Rep. 2018. V. 8. 4566. https://doi.org/10.1038/s41598-018-22683-1
Zhang X.X., Zhang T. Occurrence, abundance, and diversity of tetracycline resistance genes in 15 sewage treatment plants across China and other global locations // Environ. Sci. Technol. 2011. V. 45. P. 2598–2604.
Дополнительные материалы отсутствуют.


