Микробиология, 2021, T. 90, № 3, стр. 357-361
Новые штаммы-деструкторы линдана Achromobacter sp. NE1 и Brevundimonas sp. 242
Д. О. Егорова a, *, Э. А. Назарова a, В. А. Демаков a
a Институт экологии и генетики микроорганизмов Уральского отделения Российской академии наук – филиал Федерального государственного бюджетного учреждения науки Пермского федерального исследовательского центра Уральского отделения Российской академии наук
Урал, Россия
* E-mail: daryao@rambler.ru
Поступила в редакцию 03.11.2020
После доработки 04.12.2020
Принята к публикации 05.12.2020
Аннотация
Впервые выделены штаммы Achromobacter sp. NE1 (GenBank MW132988) и Brevundimonas sp. 242 (GenBank MW132989), обладающие уникальной способностью использовать в качестве единственного источника углерода и энергии линдан и один из побочных продуктов, образующихся в процессе аэробной биотрансформации линдана, – 1,2,4-трихлорбензол (штамм NE1) и 2,5-дихлорфенол (штамм 242). Установлено, что штаммы содержат ген linA, кодирующий первый фермент деструкции линдана дегидрохлориназу, на 99.65–100% схожий с геном linA штаммов-деструкторов линдана рода Sphingobium.
Линдан (γ-гексахлорциклогексан, γ-ГХЦГ) – химически стабильное соединение, применявшееся в качестве пестицида в широких масштабах. В 2009 году линдан включен в перечень стойких органических загрязнителей в рамках Стокгольмской конвенции (Приложение А – уничтожение), ратифицированной Россией в 2011 г. (http://chm.pops.int). Исследования нескольких десятилетий показали, что одним из перспективных способов разложения линдана является бактериальная деструкция (Kumar, Pannu, 2018). Первые сообщения об анаэробном дехлорировании линдана появились в 1960-х годах, а об участии в процессе разложения линдана аэробных бактерий – в 1970-х годах (Lal et al., 2006, 2010; Camacho-Pérez et al., 2012). Из почв выделено и описано значительное количество бактериальных штаммов, осуществляющих полную или частичную трансформацию линдана, среди которых выявлены представители родов Actinobacteria, Alcaligenes, Arthrobacter, Azotobacter, Bacillus, Burkholderia, Chromohalobacter, Citribacter, Clostridium, Desulfovibrio, Escherichia, Flavobacterium, Kokuria, Microbacterium, Pandorea, Pseudomonas, Rhodanobacter, Sphingobium, Sphingomonas, Staphylococcus, Streptomyces, Trametes, Xanthomonas (Lal et al., 2010; Zheng et al., 2011; Camacho-Pérez et al., 2012; Pannu, Kumar, 2017; Saez et al., 2017; Kumar, Pannu, 2018). Большинство штаммов-деструкторов линдана являются представителями родов Sphingobium и Sphingomonas.
Биохимический путь трансформации линдана условно разделен на “верхний” и “нижний” и включает в себя стадии дегалогенирования и гидроксилирования с последующим расщеплением циклической структуры молекулы (рис. 1). Побочными продуктами данного процесса являются 1,2,4-трихлорбензол (1,2,4-ТХБ) и 2,5-дихлорфенол (2,5-ДХФ) – химически стабильные и токсичные соединения (Lal et al., 2010; Zheng et al., 2011; Camacho-Pérez et al., 2012) (рис. 1). Возможность бактериальной деструкции 2,5-ДХФ описана для штамма Cupriavidus nantongensis X1T (Fang et al., 2020). Несмотря на то, что разложение 1,2,4-ТХБ осуществляет широкий спектр бактерий, процесс биотрансформации является длительным (период полуразложения составляет 7.07–21.24 сут) (Song et al., 2011; Dong et al., 2015). Для штаммов-деструкторов линдана не описана способность осуществлять разложение 1,2,4-ТХБ и/или 2,5-ДХФ.
Рис. 1.
“Верхний” путь деструкции линдана и образующихся продуктов (Lal et al., 2006; Camacho-Pérez et al., 2012; Fang et al., 2020; www.kegg.jp): ГХЦГ – γ-гексахлорциклогексан (линдан); ПХЦГ – γ-пентахлорциклогексен; 2,5-ДХГХ – 2,5-дихлоргидрохинон; 1,2,4-ТХБ – 1,2,4-трихлорбензол; 3,4,6-ТХК – 3,4,6-трихлоркатехол; 2,5-ДХФ – 2,5-дихлорфенол; 2,5-ДХБХ – 2,5-дихлорбензохинон. Жирным шрифтом обозначены элементы схемы, определяемые в настоящем исследовании.
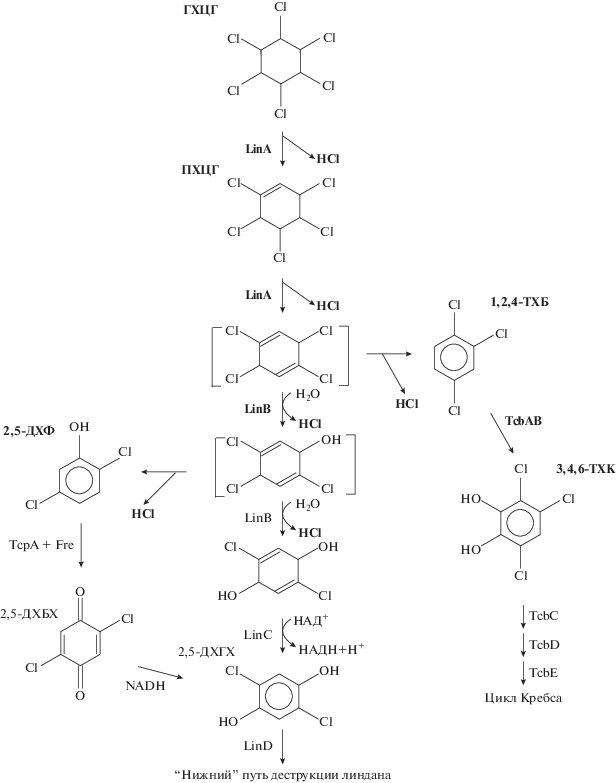
lin-Гены, кодирующие ферменты биодеструкции линдана, впервые были описаны для штамма Sphingobium japonicum UT26 (Lal et al., 2006). В настоящий момент lin-оперон выявлен у представителей родов Pseudomonas, Rhodanobacter, Shewanella, Sphingobium, Sphingomonas и Xanthomonas (http:// www.ncbi.nlm.nih.gov). Разложение 1,2,4-ТХБ обусловливается действием ферментов, кодируемых tcb-опероном (tcbABCDE), тогда как бактериальное окисление 2,5-ДХФ описано только для комплекса ферментов монооксигеназа TcpA и НАД(Ф)Н:ФАД редуктаза Fre, кодируемых генами tcpA и fre (Fang et al., 2020; www.kegg.jp; http:// www.ncbi.nlm.nih.gov).
Целью настоящей работы было выделение и изучение уникальных штаммов-деструкторов линдана.
Методом накопительного культивирования на минеральной среде К1 (Zaitsev et al., 1991) с линданом (0.3 г/л) в качестве селективного фактора, из почв, длительное время загрязненных хлорорганическими соединениями, было выделено 26 штаммов аэробных бактерий. В результате культивирования на среде К1, содержащей один из субстратов (0.3 г/л линдана, 0.1 г/л 1,2,4-ТХБ, 0.1 г/л 2,5-ДХФ, вносимых в виде ацетоновых растворов), для дальнейшего изучения были отобраны штаммы NE1 и 242, способные использовать в качестве источника углерода и энергии по два субстрата из указанных выше.
Морфо-физиологические признаки штаммов изучали по общепринятым методикам (Методы..., 1984). Тотальную ДНК штаммов получали методом щелочного лизиса. Амплификацию гена 16S рРНК проводили с использованием классических праймеров 27F и 1492R, как описано в работе (Weisburg et al., 1991). Секвенирование проводили на автоматическом секвенаторе Genetic Analyzer 3500xl (“Applied Biosystems”, США), с применением реактивов Big Dye Terminator Ready Reaction Kit v 3.1 (“Applied Biosystems”, США), согласно рекомендациям производителя. Анализ нуклеотидных последовательностей и перевод в аминокислотные последовательности осуществляли с применением программы MEGA10, поиск гомологичных последовательностей осуществляли по базам данных GenBank (http://www.ncbi.nlm.nih.gov) и EzBioCloud (https://www.ezbiocloud.net).
Анализ морфо-физиологических признаков штамма NE1 позволил предположить, что данный штамм относится к классу Betaproteobacteria, порядку Burkholderiales. Далее филогенетическую принадлежность штамма оценивали на основании анализа нуклеотидной последовательности фрагмента гена 16S рРНК, депонированной в GenBank под номером MW132988. Наибольший уровень сходства составил 99.79% с соответствующей последовательностью типового штамма Achromobacter marplatensis B2T (GenBank EU150134).
В результате изучения морфо-физиологических признаков штамма 242 было предположено, что он принадлежит классу Alphaproteobacteria, порядку Caulobacterales. Анализ фрагмента гена 16S рРНК выявил 99.85% сходства с соответствующей последовательностью типового штамма Brevundimonas diminuta ATCC15257T (GenBank GL883089). Нуклеотидная последовательность фрагмента гена 16S рРНК штамма Brevundimonas sp. 242 депонирована в GenBank под номером MW132989.
Для более подробного изучения разложения линдана, 1,2,4-ТХБ и 2,5-ДХФ штаммами Achromobacter sp. NE1 и Brevundimonas sp. 242 проводили опыты с отмытыми клетками (Egorova et al., 2020). Анализ концентрации субстрата и метаболитов проводили методами ГХ-МС, ВЭЖХ и спектрофотометрии (Egorova et al., 2020). Расчет скорости деструкции осуществляли по формуле: V = (Снач – Скон)/t, где V – скорость деструкции субстрата, Снач – начальная концентрация субстрата, Скон – концентрация субстрата в конечный момент времени, t – время деструкции.
Штаммы Achromobacter sp. NE1 и Brevundimonas sp. 242 осуществляли деструкцию линдана за 16 сут на 35.9 и 26.4% соответственно, при этом в среде были выявлены ионы хлора (соответственно, 23.9 и 17.6% от максимально возможного) и γ-пентахлорциклогексен (γ-ПХЦГ) (рис. 1). Скорость разложения линдана штаммом NE1 составила 6.73 ± 0.03 мг/л сут, а штаммом 242 – 4.95 ± ± 0.02 мг/л сут. По эффективности и скорости разложения линдана представленные штаммы превосходят большинство описанных деструкторов (Zheng et al., 2011; Camacho-Pérez et al., 2012; Pannu, Kumar, 2017; Saez et al., 2017).
Обнаружение γ-ПХЦГ в среде позволило предположить, что в процессе разложения линдана участвуют Lin-ферменты (Camacho-Pérez et al., 2012). Молекулярно-генетический анализ нуклеотидных последовательностей, амплифицированных с тотальной ДНК штаммов NE1 и 242, с использованием праймеров FGPlinA1-433-69 5'‑CGT AGA CAA GCG CCA AGA GG-3' и FGPli-nA2-434-411 5'-GGT GAA ATA GTT CGT GCA TC-3' и режима, описанного в работе (Thomas et al., 1996), показал высокий уровень сходства с последовательностями linA-генов (рис. 2). Уровень сходства фрагмента гена linA штамма Achromobacter sp. NE1 (GenBank MW150968) с наиболее близкородственными гомологичными последовательностями штаммов Sphingobium sp. S6 (GenBank MN649851.1) и Sphingobium japonicum UT26 (GenBank АР010803.1) составил 100 и 99.67% соответственно, а гена linA штамма Brevundimonas sp. 242 (GenBank MW150969) с гомологичной последовательностью штамма Sphingobium indicum B90A (GenBank CP013070.1) – 99.65%. Известно, что нуклеотидные последовательности гена linA штаммов-деструкторов линдана S. indicum B90A (Индия), S. japonicum UT26 (Япония) и S. framcense Sp+ (Франция) характеризуются 100%-ым сходством (Lal et al., 2006). Анализ дедуктивных аминокислотных последовательностей LinANE1, LinA242, LinAB90A, LinAUT26 и LinASp+ показал уровень сходства 100%. Таким образом, можно предположить, что штаммы Achromobacter sp. NE1 и Brevundimonas sp. 242 осуществляют начальное дегалогенирование линдана по “верхнему” пути, под действием дегидрохлориназы LinA, характерной для бактерий рода Sphingobium.
Рис. 2.
Положение linA-генов штаммов Achromobacter sp. NE1 и Brevundimonas sp. 242 на филогенетическом дереве, построенном с использованием метода UPGMA. Цифрами показана достоверность ветвления, вычисленная на основании “bootstrap”-анализа. В скобках указаны номера последовательностей linA-генов в GenBank.

Известно, что при разложении линдана по “верхнему” пути происходит образование опасных побочных продуктов – 1,2,4-ТХБ и 2,5-ДХФ (рис. 1) (Camacho-Pérez et al., 2012). При культивировании штамма Brevundimonas sp. 242 с линданом через 16 сут в среде зафиксировано 23.1 мг/л 1,2,4-ТХБ, а у штамма Achromobacter sp. NE1 – 32.8 мг/л 2,5-ДХФ. В условиях периодического культивирования на этапе отбора штаммов для исследования было установлено, что штамм NE1 разлагает 1,2,4-ТХБ со скоростью 2.71 ± 0.05 мг/л сут, а штамм 242 осуществляет деструкцию 2,5-ДХФ со скоростью 2.25 ± 0.02 мг/л сут. Анализ скорости деструкции субстратов, а также выявленных концентраций линдана, 1,2,4-ТХБ и 2,5-ДХФ через 16 сут культивирования позволяет предположить, что отсутствие в среде культивирования одного из побочных продуктов обусловлено деградативной активностью исследуемых штаммов.
При хроматографическом анализе культуральной среды в опытах с отмытыми клетками установлено, что разложение 1,2,4-ТХБ штаммом Achromobacter sp. NE1 происходит через стадию образования 3,4,6-трихлоркатехола (рис. 1). Известно, что данный путь биотрансформации обусловлен работой ферментов, кодируемых tcb-опероном (www.kegg.jp). Вероятно, штамм Achromobacter sp. NE1 обладает уникальным сочетанием Lin- и Tcb-ферментативных систем.
Проведенные исследования не позволили установить промежуточные продукты разложения 2,5-ДХФ у штамма Brevundimonas sp. 242. В настоящее время описан один путь аэробной бактериальной трансформации 2,5-ДХФ – через стадию образования 2,5-дихлорбензохинона до 2,5-дихлоргидрохинона (рис. 1) (Fang et al., 2020). На основании полученных результатов можно предположить, что штамм 242 обладает ферментативными системами, схожими с комплексом TcpA+Fre штамма Cupriavidus nantongensis X1T, что в сочетании с ферментами деструкции линдана позволяет эффективно разлагать 2,5-ДХФ без накопления промежуточных продуктов.
Таким образом, в результате проведенного исследования впервые выделены штаммы-деструкторы линдана, принадлежащие родам Achromobacter и Brevundimonas. Уникальность штаммов Achromobacter sp. NE1 и Brevundimonas sp. 242 состоит в том, что начальное дегалогенирование линдана обусловлено действием дегидрохлориназы, кодируемой геном linA, наиболее близким к linA-гену известных штаммов-деструкторов линдана из рода Sphingobium. Следует отметить, что штаммы Achromobacter sp. NE1 и Brevundimonas sp. 242 обладают уникальной способностью разлагать наряду с линданом образующиеся при его деструкции побочные продукты – 1,2,4-ТХБ и 2,5-ДХФ. Подобные свойства для аэробных бактерий-деструкторов линдана описаны впервые.
Список литературы
Методы общей бактериологии: Пер. с англ. / Под ред. Герхардта Ф. и соавт. М.: Мир, 1983. Т. 1–3.
Methods for General and Molecular Bacteriology / Philipp Gerhardt, editor-in-chief. Eds. Murray R.G.E., Wood W.A., Krieg N.R. Washington, D.C.: American Society for Microbiology, 1994. 791 p.
Camacho-Pérez B., Ríos-Leal E., Rinderknecht-Seijas N., Poggi-Varaldo M. Enzymes involved in the biodegradation of hexachlorocyclohexane: A mini review // J. Environ. Manag. 2012. V. 95. P. S306–S318.
Dong W.H., Zhang P., Lin X.Y., Zhang Y., Tabouré A. Natural attenuation of 1,2,4-trichlorobenzene in shallow aquifer at the Luhuagang’s landfill site, Kaifeng, China // Sci. Total Environ. 2015. V. 505. P. 216–222.
Egorova D.O., Gorbunova T.I., Pervova M.G., Kir’yanova T.D., Demakov V.A., Saloutin V.I., Chupakhin O.N. Biodegradability of hydroxylated derivatives of commercial polychlorobiphenyls mixtures by Rhodococcus-strains // J. Hazard. Mater. 2020. V. 400. 123328.
Fang L., Qin H., Shi T., Wu X., Li Q.X., Hua R. Ortho and para oxydehalogenation of dihalophenols catalyzed by the monooxygenase TcpA and NAD(P)H:FAD reductase Fre // J. Hazard. Mater. 2020. V. 388. 121787.
Kumar D., Pannu R. Perspectives of lindane (γ-hexachlorocyclohexane) biodegradation from the environment: a review // Bioresour. Bioprocs. 2018. V. 5. P. 29.
Lal R., Dogra C., Malhotra S., Sharma P., Pal R. Diversity, distribution and divergence of lin genes in hexachlorocyclohexane-degrading sphingomonads // Trends Biotech. 2006. V. 24. P. 121–130.
Lal R., Pandey G., Sharma P., Kumari K., Malhotra S., Pandey R., Raina V., Kohler H.P., Holliger C., Jackson C., Oakeshott J.G. Biochemistry of microbial degradation of hexachlorocyclohexane and prospects for bioremediation // Microbiol. Mol. Biol. Rev. 2010. V. 74. P. 58–80.
Pannu R., Kumar D. Process optimization of γ-hexachlorocyclohexane degradation using three novel Bacillus sp. strains // Biocat. Agric. Biothech. 2017. V. 11. P. 97–107.
Saez J.M., García V.C., Benimeli C.S. Improvenment of lindane removal by Streptomyces sp. M7 by using stable microemulsions // Ecotoxicol. Environ. Saf. 2017. V. 144. P. 351–359.
Song Y., Wang F., Kengara F.O., Bian Y.-R., Yang X.-L., Liu C.-Y., Jiang X. Improved biodegradation of 1,2,4-trichlorobenzene by adapted microorganisms in agricultural soil and in soil suspension cultures // Pedosphere. 2011. V. 21. P. 423–431.
Thomas J.-C., Berger F., Jacquier M., Bernillon D., Baud-Grasset F., Truffaut N., Norm P., Vogel T.M., Simonet P. Isolation and characterization of a novel γ-hexachlorocyclohexane-degrading bacterium // J. Bacteriol. 1996. V. 178. P. 6049–6055.
Weisburg W.G., Barns S.M., Pelletier D.A., Lane D.J. 16S Ribosomal DNA amplification for phylogenetic study // J. Bacteriol. 1991. V. 173. P. 697–703.
Zaitsev G.M., Tsoi T.V., Grishenkov V.G., Plotnikova E.G., Boronin A.M. Genetic control of degradation of chlorinated benzoic acids in Arthrobacter globiformis, Corynebacterium sepedonicum and Pseudomonas cepacia strains // FEMS Microbiol. Lett. 1991. V. 81. P. 171–176.
Zheng G., Selvam A., Wong J.W.C. Rapid degradation of lindane (γ-hexachlorocyclohexane) at low temperature by Sphingobium strains // Int. Biodeterior. Biodegrad. 2011. V. 65. P. 612–618.
Дополнительные материалы отсутствуют.


