Микробиология, 2021, T. 90, № 3, стр. 329-336
Таксономическое разнообразие бактериального сообщества плодовых тел миксомицета Lycogala epidendrum (L.) Fr.
Л. Р. Сизов a, *, Л. В. Лысак a, В. И. Гмошинский b
a Факультет почвоведения МГУ им. М.В. Ломоносова
119991 Москва, Россия
b Биологический факультет МГУ им. М.В. Ломоносова; ФГБУ
Государственный заповедник “Полистовский”
119991 Москва, Россия
* E-mail: Leo.Sizoff@yandex.ru
Поступила в редакцию 03.11.2020
После доработки 30.12.2020
Принята к публикации 04.01.2021
Аннотация
При помощи метода высокопроизводительного секвенирования гена 16S рРНК изучено таксономическое разнообразие бактериального сообщества на плодовых телах миксомицета Lycogala еpidendrum. Выявлено присутствие представителей 31 рода бактерий, относящихся к шести филумам. В микробиоме доминирует филум Proteobacteria, на родовом уровне преобладают представители родов Pseudomonas и Luteibacter. Анализ таксономического разнообразия бактериальных сообществ показал, что сообщество L. epidendrum значительно уступает по разнообразию сообществам подстилки и валежника – субстратам, находящимся в непосредственной близости с плодовыми телами. Это свидетельствует в пользу селективной роли плодового тела миксомицета в отношении филума Proteobacteria, в первую очередь родов Pseudomonas и Luteibacter.
Миксомицеты (Myxomycetes = Myxogastrea) – грибоподобные эукариотические организмы со сложным жизненным циклом, включающим вегетативные стадии в виде одноклеточных микроорганизмов (флагеллятные клетки, миксамебы) и надклеточных структур (плазмодий), генеративные стадии в виде плодовых тел (спорофоров), а также покоящиеся стадии (микроцисты, сферулы, склероции). В соответствии с современной классификацией миксомицеты, наряду с классами Dictyosteliomycetes и Ceratiomyxomycetes, образуют тип Eumycetozoa в пределах монофилетической группы Amoebozoa (Leontyev et al., 2019). Молекулярно-биологическими методами показано, что представители Eumycetozoa являются доминирующей группой почвенных простейших (Stephenson et al., 2011). На вегетативных стадиях миксомицеты потребляют путем фагоцитоза клетки бактерий, дрожжей, мицелий и споры грибов, простейших и водоросли. В составе фаготрофного комплекса миксомицеты способны влиять на численность бактерий в локусах их постоянного обитания в природных условиях (Feest, Madelin, 1988). Миксомицеты широко распространены в лесах умеренного пояса. На территории России было обнаружено более 440 видов (Bortnikov et al., 2020).
Исследование бактериальных сообществ, ассоциированных с миксомицетами на разных стадиях жизненного цикла, представляет значительный интерес как для раскрытия биоценотических связей между этими организмами, так и для изучения вклада миксомицетов в формирование микробных сообществ почвы и сопряженных субстратов в лесном биоценозе.
Все стадии жизненного цикла миксомицетов в природе проходят в локусах, богатых бактериями. На примере Dictyostelium discoideum Raper, принадлежащего к родственному миксомицетам классу Dictyosteliomycetes, продемонстрированы симбиотические взаимоотношения между бактериями и эукариотами. Бактерии рода Burkholderia обитают внутри амеб и способствуют сбору и запасанию съедобных бактерий внутри клетки-хозяина, что в итоге помогает D. discoideum выживать в бедной питательными ресурсами среде обитания (Haselkorn et al., 2018). В другом исследовании высказано предположение о возможной связи плазмодиев тропических миксомицетов и бактерий. Показано, что бактерии из семейства Enterobacteriaceae ассоциированы с плазмодиями определенных видов миксомицетов, и такие плазмодии приобретают новые свойства (Kalyanasundaram, 2004). Изучение миксомицетов Trichia decipiens и Metatrichia vesparia, отобранных на территории Государственного природного заповедника “Нургуш” (Кировская область, РФ), выявило доминирование на спорофорах грамотрицательных бактерий, среди которых преобладали представители родов Methylobacterium и Sphingobacterium, причем практически все они были способны к синтезу ауксинов при росте на среде с триптофаном (Широких, Широких, 2016). В недавнем исследовании при помощи метода секвенирования были изучены бактериальные сообщества плодовых тел 112 видов миксомицетов. Преобладающие порядки бактерий были Pseudomonadales, Rhizobiales, Sphingomonadales, Actinomycetales и Burkholderiales. Среди родов доминировали Acinetobacter и Pseudomonas. Доминантные бактерии преимущественно наследовались от субстрата, тогда как минорные компоненты бактериального сообщества различались для разных видов миксомицетов (Li et al., 2017). Кроме того, были исследованы бактериальные сообщества плазмодиев миксомицетов порядка Physarales. Как и на плодовых телах, в сообществах плазмодиев преобладали Proteobacteria, и доминанты в плазмодиях происходили в основном из субстрата, а большое количество бактерий более редких таксонов было специфично для плазмодиев разных видов миксомицетов (Wang et al., 2020).
Ранее нами при изучении сапротрофного бактериального комплекса 13 видов лигнофильных миксомицетов в лесном биоценозе было показано, что на поверхности большинства плодовых тел миксомицетов в значительных количествах присутствуют культивируемые грамотрицательные бактерии родов Cytophaga, Xanthomonas, Myxococcus, Aquaspirillum и Pseudomonas. Созревающие плодовые тела создают в подстилке и почве микролокусы с повышенной концентрацией культивируемых грамотрицательных бактерий (Сизов и соавт., 2020).
Миксомицет Lycogala epidendrum (L.) Fr., плодовые тела которого рассматриваются в данной работе, – космополит, широко распространен на территории лесопарков Москвы и лесов Московской области (Гмошинский, 2013). Он имеет достаточно крупные и заметные (на ранних стадиях развития – кораллово-розовые) спорофоры, формирующиеся на гниющей древесине (валежнике), что облегчает поиск и отбор образцов. Кроме того, спороношения L. epidendrum покрыты плотным перидием, в результате чего, в отличие от многих других миксомицетов, у L. epidendrum сравнительно медленно разрушаются плодовые тела, и на них можно ожидать формирование устойчивых бактериальных ассоциаций.
Целью нашей работы было изучение таксономического разнообразия бактериального сообщества спорофоров миксомицета Lycogala epidendrum при помощи метода высокопроизводительного секвенирования гена 16S рРНК тотальной ДНК и сравнение данного сообщества с сообществами лесной подстилки и валежника (гниющей древесины березы), на поверхности которого формируются плодовые тела.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объекты исследования. Основным объектом исследования являются плодовые тела Lycogala epidendrum. Зрелые спороношения мы определили по морфологическим признакам (бурые крупные шаровидные спорофоры 4–7 мм в диаметре и сравнительно мелкие споры 6–7 мкм в диаметре). Сбор плодовых тел произвели с одного локуса в октябре 2018 года в Москве, в Битцевском лесопарке, в овраге к северу от Деревлевского ручья (N 55.645485, E 37.571095). Также отобрали образцы валежника (гниющей древесины березы), на котором развивались спорофоры L. epidendrum, и верхнего слоя подстилки рядом с валежником (листья березы и липы) для сравнения бактериальных сообществ этих субстратов с сообществом спорофоров L. epidendrum. До дальнейшей обработки образцы хранили в морозильной камере бытового холодильника (–18°С).
Выделение и анализ ДНК. Для экстракции ДНК использовали 3 спорофора L. epidendrum и по 0.25 г древесины и подстилки. Выделение проводили с помощью набора реактивов PowerSoil DNA Isolation Kit (“MO BOI”) по инструкции производителя, для спорофоров и подстилки проводили смыв микроорганизмов с их поверхности в суспензию.
Высопроизводительное секвенирование участка гена 16S рРНК. Высокопроизводительное секвенирование проводили при помощи системы Illumina MiSeq (ООО “БИОСПАРК”, Троицк, Россия). Подготовку ДНК-библиотек для секвенирования осуществили с использованием технологии двустадийной полимеразной цепной реакции. На первом этапе производили амплификацию гипервариабельного V3–V4 участка гена 16S рРНК с использованием праймеров: Pro341F и Pro806R (Takahashi et al., 2014). На втором этапе амплифицировали ПЦР продукт, полученный на первом этапе, с целью баркодирования библиотеки. Впоследствии ДНК-библиотеки анализировали методом парно-концевого чтения (2 × 300 п.о.) генерацией не менее 10 000 парных прочтений на образец.
Обработка данных секвенирования. Обработка данных была проведена с использованием автоматизированного алгоритма QIIME (Caporaso et al., 2010) и включала объединение прямых и обратных прочтений, удаление технических последовательностей, фильтрацию химерных последовательностей. Для разбиения последовательностей на операционные таксономические единицы (ОТЕ) использовали алгоритм с открытым референсным порогом классификации 97%. Выравнивание прочтений на последовательность гена 16S рРНК и распределение последовательностей по таксономическим единицам провели с использованием базы данных Silva версии 132 (Pruesse et al., 2007).
С помощью программных пакетов QIIME мы определили индексы разнообразия, рассчитанные при объединении нуклеотидных последовательностей в ОТЕ с уровнем схожести в 97%. Для оценки альфа-разнообразия использовали индекс разнообразия Шеннона (H = Σ pi ln pi, где pi – доля i-го вида в сообществе) и индекс выровненности Пиелу, который является нормировкой индекса Шеннона между 0 и 1 (Мэгарран, 1992). Различия между бактериальными сообществами исследуемых локусов (бета-разнообразие) анализировали вычислением матрицы расстояний с использованием коэффициента Брея–Кертиса (Bray, 1956) и метрики weighted UniFrac, при которой учитывается филогенетическая близость таксонов в сравниваемых сообществах (Lozupone et al., 2011). Полученные меры несходства бактериальных сообществ равны 0 при полной идентичности двух сообществ и 1 при полном отсутствии сходств.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Филогенетическое разнообразие бактериальных сообществ. В результате проведенного высокопроизводительного секвенирования вариабельного участка гена 16S рРНК была получена информация о филогенетическом разнообразии бактериальных сообществ спорофоров (плодовых тел) L. epidendrum, лесной подстилки и валежника.
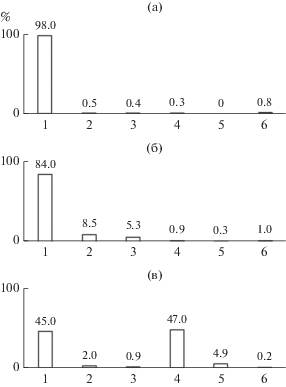
Бактериальные сообщества плодовых тел L. epidendrum и подстилки состояли в основном из представителей филума Proteobacteria (соответственно, 98 и 84% от общего числа последовательностей). В подстилке, кроме того, значительную долю составили филумы Bacteroidetes (8.5%) и Actinobacteria (5.3%). В сообществе древесины валежника преобладали представители филумов Acidobacteria (47%) и Proteobacteria (45%) (рис. 1).
Рис. 1.
Таксономическая структура бактериальных сообществ плодовых тел L. epidendrum (а); подстилки (б); валежника (в). Филумы (% от общего числа последовательностей в образце): 1 – Proteobacteria; 2 – Bacteroidetes; 3 – Actinobacteria; 4 – Acidobacteria; 5 – Verrucomicrobia; 6 – прочие.
Анализ таксономического состава бактериального сообщества плодовых тел L. epidendrum выявил представителей 31 рода, принадлежащих к 6 филумам. Преобладающие в микробиоме протеобактерии принадлежали к 3 классам: Gammaproteobacteria (90% от числа всех последовательностей), Alphaproteobacteria (5%) и Betaproteobacteria (3%). Содержание представителей других филумов (Firmicutes, Bacteroidetes, Actinobacteria, Acidobacteria и Chlorobi) в сумме составило около 2%. Класс Gammaproteobacteria был представлен четырьмя порядками: Pseudomonadales, Legionellales, Enterobacteriales и Xanthomonadales. Порядок Pseudomonadales был представлен доминирующим в сообществе родом Pseudomonas (79% от общего числа последовательностей), Legionellales включал один род Aquicella, среди Enterobacteriales было два рода – Pantoea и Enterobacter. Среди Xanthomonadales преобладал род Luteibacter (7%), а также присутствовали Rhodanobacter, Stenotrophomonas и Dokdonella. Класс Alphaproteobacteria был представлен родами Sphingomonas, Rhizobium, Bradirhizobium, Acidisoma, Acidiphilium и Roseomonas. Betaproteobacteria были представлены, главным образом, некультивируемыми представителями семейства Nitrosomonadaceae. В следовых количествах присутствовали представители родов Paraburkholderia, Massilia, Variovorax, Acidovorax и некоторые другие. Из других филумов в качестве минорного компонента присутствовали роды Lactobacillus и “Tyzzerella” (филум Firmicutes), Mucilaginibacter (филум Bacteroidetes), Microbacterium, Jatrophihabitans и Streptomyces (филум Actinobacteria) и Terriglobus (филум Acidobacteria). Филум Chlorobi был представлен лишь некультивируемыми представителями.
Из 31-го рода бактерий девять встретились только на плодовых телах (Dokdonella, Rhodanobacter, Pantoea, Aquicella, Noviherbaspirillum, Undibacterium, “Tyzzerella”, Microbacterium и Streptomyces), суммарно составляя долю в 1.74%. Девять родов из сообщества спорофоров были обнаружены также в древесине и подстилке – Acidovorax, Paraburkholderia, Bradirhizobium, Acidisoma, Acidiphilium, Sphingomonas, Mucilaginibacter, Jatrophihabitans и Terriglobus (суммарно 2.64%).
Таким образом, для бактериального сообщества спороношений L. epidendrum, характерно доминирование рода Pseudomonas (79%) и значительное содержание рода Luteibacter (7%) (табл. 1). В меньшей степени представлены роды грамотрицательных бактерий из филумов Bacteroidetes, Acidobacteria, Chlorobi. На последнем месте по обилию находятся грамположительные бактерии (Firmicutes и Actinobacteria). Доминирование рода Pseudomonas вполне закономерно. Бактерии из этого рода – обычные обитатели почв (Смирнов, Киприанова, 1990). Они обладают гибким метаболизмом, демонстрируют значительное физиологическое разнообразие и способны к колонизации различных экологических ниш. Многие представители рода Pseudomonas являются типичными копиотрофами. Однако среди Pseudomonas обнаружены и гидролитики, так, например, целлюлолитическая и хитинолитическая активность была обнаружена у бактерий Pseudomonas fluorescens, выделенных с плодовых тел трюфелей. Численность этих бактерий увеличивалась по мере созревания трюфелей, что позволило предположить их участие в разрушении стенок плодовых тел (Citterio et al., 2001). Схожую экологическую функцию род Pseudomonas может выполнять и на плодовых телах L. epidendrum, выполняя гидролиз внешних оболочек спорофоров (перидия), которые состоят из одного–двух слоев мукополисахаридов (Clark, Haskins, 2014). Бактерии вида Luteibacter rhizovicinus были впервые выделены из ризосферы ячменя, и они, судя по описанию, являются копиотрофами, которые активно используют мономеры, но неспособны к разложению трудногидролизуемых субстратов (Johansen et al., 2005). Наличие большого количества копиотрофов среди бактерий, ассоциированных с плодовыми телами миксомицета, позволяет предположить, что многие бактерии являются трофическим окружением активных гидролитиков (мицелиальных грибов и актиномицетов), значительное присутствие которых было выявлено нами на поверхности плодовых тел (Сизов и соавт., 2020).
Таблица 1.
Доминирующие роды в бактериальных сообществах плодовых тел L. epidendrum и сопряженных субстратов (% от общего числа последовательностей в образце)
| Основные филогенетические группы | Доминирующие роды | ||
|---|---|---|---|
| плодовые тела L. epidendrum | подстилка | валежник | |
| Alphaproteobacteria | – | Sphingomonas (3%), Methylobacterium (2%), Zymomonas (1%) | Afipia (3%), Acidisoma (2%), Rhizomicrobium (2%), Phenylobacterium (2%), Acidocella (1%), Bradyrhizobium (1%) |
| Betaproteobacteria | – | Massilia (11%), Variovorax (3%) | – |
| Gammaproteobacteria | Pseudomonas (79%), Luteibacter (7%) | Pseudomonas (43%), Luteibacter (2%) | – |
| Bacteroidetes | – | Spirosoma (4%), Hymenobacter (3%) |
Mucilaginibacter (2%) |
| Actinobacteria | – | Actinoplanes (2%) | – |
| Acidobacteria | – | – | Granulicella (35%) |
В микробиоме подстилки выявлено 80 родов бактерий из 11 филумов. Так же как на плодовых телах, в сообществе подстилки доминировали представители Proteobacteria (84%), меньшую долю составили филумы Bacteroidetes (8.5%) и Actinobacteria (5.3%), еще ниже (менее 1%) было содержание филумов Acidobacteria, Verrucomicrobia, Firmicutes, Chlorobi, Fusobacteria, Chloroflexi, Deinococcus–Thermus (рис. 1).
Среди Proteobacteria, как и на плодовых телах миксомицетов, преобладали представители Gammaproteobacteria (52%), ниже было содержание Betaproteobacteria (17%) и Alphaproteobacteria (14%). В минорных количествах присутствовали Deltaproteobacteria и Epsilonproteobacteria. Среди Gammaproteobacteria в подстилке доминировали, как и в случае со спорофорами L. epidendrum, род Pseudomonas (43%), предположительно, со схожей экологической нишей, и род Luteibacter (2%), представители которого являются копиотрофами. Betaproteobacteria были представлены, главным образом, родами Massilia, Variovorax, Acidovorax. Бактерии Massilia sp. выделяются из разных природных местообитаний и также предпочитают потреблять легкодоступные соединения (Altankhuu, Kim, 2017). Бактерии рода Variovorax являются частью сапротрофного бактериального комплекса в экосистемах, разлагая различные органические соединения. Некоторые представители способны восстанавливать нитраты (Jin et al., 2012). Класс Alphaproteobacteria был представлен теми же родами, что и в сообществе плодовых тел – Rhizobium, Bradyrhizobium, Sphingomonas, Caulobacter. Кроме того, был выявлен род Methylobacterium, представители которого являются метилотрофами (Троценко и соавт., 2010). Среди Deltaproteobacteria были определены представители родов Myxococcus, Sorangium, Polyangium, Bdellovibrio и Bacteriovorax. Epsilonproteobacteria были представлены одним родом Sulfuricurvum. В отличие от сообщества плодовых тел в подстилке ощутимый вклад вносили также представители филумов Bacteroidetes (роды Spirosoma, Hymenobacter, Flavobacterium, Mucilaginibacter) и Actinobacteria (род Actinoplanes) (табл. 1). Таким образом, в этом локусе наблюдалось высокое разнообразие таксонов, представители которых входят в различные функциональные группы, что свидетельствует в пользу активно протекающих процессов деструкции органического вещества, поступающего на поверхность почвы.
Иная таксономическая структура была определена для бактериального сообщества валежника. В прокариотном сообществе древесины были выявлены представители 58 родов из 7 филумов. Здесь доминировали представители двух филумов: Proteobacteria (45%) и Acidobacteria (47%), меньшее содержание было у филумов Verrucomicrobia (4.9%), Bacteroidetes (2%) и Аctinobacteria (0.9%). В минорных компонентах были определены филум Saccharibacteria и филум-кандидат TM6 (Yeoh et al., 2016) (рис. 1).
В микробиоме древесины в составе филума Acidobacteria доминировал род Granulicella (35%). Бактерии этого рода были впервые изолированы из верховых торфяников Сибири и Ярославской области (Pankratov, Dedysh, 2010). Было показано, что представители этого рода проявляют высокую целлюлолитическую активность (Yamada et al., 2014). Таким образом, бактерии Granulicella способны участвовать в разложении древесины наряду с грибами, обитающими в этом локусе. Бактериальное сообщество древесины характеризовалось иным спектром родов филума Proteobacteria по сравнению с подстилкой и плодовыми телами. Среди Alphaproteobacteria преобладали роды Afipia, Acidisoma, Rhizomicrobium, Phenylobacterium и Acidocella (табл. 1). Бактерии родов Acidisoma и Acidocella были выделены из кислых почв и сфагнума, и также как и доминантный род Granulicella, предпочитают кислую реакцию среды (Kishimoto et al., 1995; Belova et al., 2009). Содержание представителей классов Betaproteobacteria и Gammaproteobacteria было минорное, при этом доминанты родов Pseudomonas и Luteibacter, присущие сообществам плодовых тел и подстилки, определены не были. Из представителей других филумов вклад более 1% был лишь у рода Mucilaginibacter из филума Bacteroidetes. Представители этого рода выделяются из различных местообитаний (например, Mucilaginibacter pineti из древесины сосны) и активны в основном по отношению к легкодоступным субстратам (Paiva et al., 2014).
По числу обнаруженных филумов и родов сообщество плодовых тел L. epidendrum (31 род из 6 филумов) значительно уступало сообществам подстилки (80 родов из 12 филумов) и валежника (58 родов из 7 филумов). В подстилке зафиксировано наибольшее количество ОТЕ, определенных до рода, однако общее количество ОТЕ со сходством 97% было максимальным на древесине (186 ОТЕ) и минимальным на плодовых телах (55 ОТЕ). Наибольшее альфа-разнообразие сообщества, развивающегося на древесине, подтверждается величиной индекса Шеннона (5.5), ниже показатель для подстилки (4.7), минимальное значение зарегистрировано для сообщества плодовых тел L. epidendrum (0.8). Микробиомы лесной подстилки и валежника характеризуются большей выровненностью по сравнению с сообществом плодовых тел, что подтверждается значениями индекса выровненности Пиелу для подстилки и валежника (0.69 и 0.73 соответственно) и низким значением этого показателя для плодовых тел (0.14) (табл. 2). Различия между сообществами подстилки и спорофоров по метрикам weighted UniFrac и Брея–Кертиса принимали значения 0.91 и 0.97 соответственно, что меньше, чем для сообществ спорофоров и валежника (0.94 и 0.99). Это соотносится с тем, что по таксономической структуре бактериальное сообщество миксомицета ближе к сообществу подстилки, а не валежника. По-видимому, состав бактериального сообщества спороношений L. epidendrum не наследуется полностью от субстрата, а зависит в большей мере от специфики перидия как среды обитания.
Таблица 2.
Характеристика бактериальных сообществ спорофоров миксомицета, подстилки и валежника при помощи показателей разнообразия
| Показатели | Плодовые тела L. epidendrum | Подстилка | Валежник |
|---|---|---|---|
| Количество филумов | 6 | 11 | 7 |
| Количество определенных родов | 31 | 80 | 58 |
| Количество ОТЕ (97% сходства) | 55 | 105 | 186 |
| Индекс Шеннона | 0.8 | 4.7 | 5.5 |
| Индекс Пиелу | 0.14 | 0.69 | 0.73 |
По результатам, которые мы получили ранее методом посева на поверхность плотной среды, сапротрофный бактериальный комплекс на плодовых телах L. epidendrum был представлен в основном родами Cytophaga, Pseudomonas и Polyangium, а также в некоторых посевах преобладал род Xanthomonas (последний, также как и Luteibacter, из семейства Xanthomonadaceae) (Сизов и соавт., 2020). Это свидетельствует в пользу того, что представители доминантных родов Pseudomonas и Luteibacter, которые были определены при анализе гена 16S рРНК тотальной ДНК, активно участвуют в процессах, происходящих на поверхности плодовых тел. Однако роды Cytophaga и Polyangium не были обнаружены высокопроизводительным секвенированием, что, возможно, связано с тем, что вне условий искусственной питательной среды представители этих родов составляют незначительную долю в сообществе.
Таким образом, на плодовых телах миксомицета L. еpidendrum выявлено присутствие представителей шести филумов бактерий (31 род). Доминировал в микробиоме филум Proteobacteria (98%). На родовом уровне преобладали представители родов Pseudomonas (79%) и Luteibacter (7%), что связано с их гибким метаболизмом, способностью использовать широкий спектр мономеров, образующихся в результате распада внешней оболочки плодовых тел под действием гидролитиков (микромицетов, актиномицетов). Бактерии, попадая внутрь спорофоров при их разрушении, могут служить потенциальным питательным субстратом для будущих миксамеб. Анализ таксономического разнообразия бактериальных сообществ показал, что сообщество L. epidendrum значительно уступало по разнообразию сообществам подстилки и валежника – субстратам, находящимся в контактной близости с плодовыми телами. Это свидетельствует в пользу селективной роли плодового тела миксомицета в отношении филума Proteobacteria, в первую очередь родов Pseudomonas и Luteibacter.
Список литературы
Гмошинский В.И. Миксомицеты Москвы и Московской области. Автореферат дис. … канд. биол. наук, 03.02.12. М.: Моск. гос. ун-т им. М.В. Ломоносова, 2013. 23 с.
Мэгарран Э. Экологическое разнообразие и его измерение. М.: Мир, 1992. 184 с.
Сизов Л.Р., Захарова Н.Б., Лысак Л.В., Гмошинский В.И. Микробные сообщества на плодовых телах миксомицетов в лесном фитоценозе // Бюл. МОИП. Отд. биол. 2020. Т. 125. № 5. С. 29–35.
Смирнов В.В., Киприанова Е.А. Бактерии рода Pseudomonas. Киев: Наукова думка, 1990. 262 с.
Троценко Ю.А., Доронина Н.В., Торгонская М.Л. Аэробные метилобактерии. Пущино: ОНТИ ПНЦ РАН, 2010. 325 с.
Широких А.А., Широких И.Г. Фиторегуляторная активность бактерий, ассоциированных со спорофорами миксомицетов // Материалы II Международной научной конференции. “Биология, систематика и экология грибов и лишайников в природных экосистемах и агрофитоценозах”. Минск, 2016. С. 287–292.
Altankhuu K., Kim J. Massilia solisilvae sp. nov., Massilia terrae sp. nov. and Massilia agilis sp. nov., isolated from forest soil in South Korea by using a newly developed culture method // Int. J. Syst. Evol. Microbiol. 2017. V. 67. P. 3026–3032.
Belova S.E., Pankratov T.A., Detkova E.N., Kaparullina E.N., Dedysh S.N. Acidisoma tundrae gen. nov., sp. nov. and Acidisoma sibiricum sp. nov., two acidophilic, psychrotolerant members of the Alphaproteobacteria from acidic northern wetlands // Int. J. Syst. Evol. Microbiol. 2009. V. 59. P. 2283–2290.
Bortnikov F.M., Matveev A.V., Gmoshinskiy V.I., Novozhilov Yu.K., Zemlyanskaya I.V., Vlasenko A.V., Schnittler M., Shchepin O.N. Myxomycetes of Russia: a history of research and a checklist of species // Karstenia. 2020. V. 58. P. 316–373.
Bray J.R. A study of mutual occurrence of plant species // Ecology. 1956. V. 37. P. 21–28.
Caporaso J.G., Kuczynski J., Stombaugh J., Bittinger K., Bushman F.D., Costello E.K., Fierer N., Peña A.G., Goodrich J.K., Gordon J.I., Huttley G.A., Kelley S.T., Knights D., Koenig J.E., Ley R.E., Lozupone C.A., McDonald D., Muegge B.D., Pirrung M., Reeder J., Sevinsky J.R., Turnbaugh P.J., Walters W.A., Widmann J., Yatsunenko T., Zaneveld J., Knight R. QIIME allows analysis of high throughput community sequencing data // Nature Methods. 2010. V. 7. P. 335–336.
Citterio B., Malatesta M., Battistelli S., Marcheggiani F., Baffone W., Saltarelli R., Stocchi V., Gazzanelli G. Possible involvement of Pseudomonas fluorescens and Bacillaceae in structural modifications of Tuber borchii fruit bodies // Can. J. Microbiol . 2001. V. 47. P. 264–268.
Clark J., Haskins E. Sporophore morphology and development in the myxomycetes: A review // Mycosphere. 2014. V. 5. P. 153–170.
Haselkorn T.S., DiSalvo S., Miller J.W., Bashir U., Brock D.A., Queller D.C., Joan E. Strassmann J.E. The specificity of Burkholderia symbionts in the social amoeba farming symbiosis: Prevalence, species, genetic and phenotypic diversity // Mol. Ecol. 2018. V. 28. P. 847–862.
Feest A., Madelin M.F. Seasonal population changes of myxomycetes and associated organisms in five non-woodland soils, and correlations between their numbers and soil characteristics // FEMS Microbiol. Ecol. 1988. V. 4. P. 141–152.
Jin L., Kim K.K., Ahn C.Y., Oh H.M. Variovorax de-fluvii sp. nov., isolated from sewage // Int. J. Syst. Evol. Microbiol. 2012. V. 62. P. 1779–1783.
Johansen J.E., Binnerup S.J., Kroer N. Mølbak L. Luteibacter rhizovicinus gen. nov., sp. nov., a yellow-pigmented gammaproteobacterium isolated from the rhizosphere of barley (Hordeum vulgare L.) // Int. J. Syst. Evol. Microbiol. 2005. V. 55. P. 2285–2289.
Kalyanasundaram I. A positive ecological role for tropical myxomycetes in association with bacteria // Syst. Geogr. Plants. 2004. V. 74. P. 239–242.
Kishimoto N., Kosako N., Wakao N., Tatsuo T., Hiraishi A. Transfer of Acidiphilium facilis and Acidiphilium aminolytica to the genus Acidocella gen. nov., and emendation of the genus Acidiphilium // Syst. Appl. Microbiol. 1995. V. 18. P. 85–91.
Leontyev D.V., Schnittler M., Stephenson S.L., Novozhilov Y.K., Shchepin O.N. Towards a phylogenetic classification of the Myxomycetes // Phytotaxa. 2019. V. 399. P. 209–238.
Li S., Qi B., Peng X., Wang W., Wang W., Wang Q., Li Y. Bacteria diversity within myxomycetes based on 16S amplicon sequencing // The Ninth Int. Congr. on the Systematics and Ecology of Myxomycetes. Japan. Tanabe. Program and Abstracts. 2017. P. 43.
Lozupone C., Lladser M.E., Knights D., Stombaugh J., Knight R. UniFrac: an effective distance metric for microbial community comparison // ISME J. 2011. V. 5. P. 169–172.
Paiva G., Abreu P., Proença D.N., Santos S., Nobre M.F., Morais P.V. Mucilaginibacter pineti sp. nov., isolated from Pinus pinaster wood from a mixed grove of pines trees // Int. J. Syst. Evol. Microbiol. 2014. V. 64. P. 2223–2228.
Pankratov T.A., Dedysh S.N. Granulicella paludicola gen. nov., sp. nov., Granulicella pectinivorans sp. nov., Granulicella aggregans sp. nov. and Granulicella rosea sp. nov., acidophilic, polymer-degrading acidobacteria from Sphagnum peat bogs // Int. J. Syst. Evol. Microbiol. 2010. V. 60. P. 2951–2959.
Pruesse E., Quast C., Knittel K., Fuchs B.M., Ludwig W., Peplies J., Glöckner F.O. SILVA: a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB // Nucl. Acids Res. 2007. V. 35. P. 7188–7196.
Stephenson S.L., Fiore-Donno A.M., Schnittler M. Myxomycetes in soil // Soil Biol. Biochem. 2011. V. 43. P. 2237–2242.
Takahashi S., Tomita J., Nishioka K., Hisada T., Nishijima M. Development of a prokaryotic universal primer for simultaneous analysis of bacteria and archaea using next-generation sequencing // PLoS One. 2014. V. 9. e105592.
Wang Q., Li S., Qi B., Peng X., Dou W., Li Y. Study on the selection of environmental bacteria by plasmodia // Proc. of Tenth Int. Congr. on the Systematics and Ecology of Myxomycetes. Costa Rica, Turrialba. 2020. P. 23.
Yamada K., Okuno Y., Meng X.Y., Tamaki H., Kamagata Y., Hanada S. Granulicella cerasi sp. nov., an acidophilic bacterium isolated from cherry bark // Int. J. Syst. Evol. Microbiol. 2014. V. 64. P. 2781–2785.
Yeoh Y.K., Sekiguchi Y., Parks D.H., Hugenholtz P. Comparative genomics of candidate phylum TM6 suggests that parasitism is widespread and ancestral in this lineage // Mol. Biol. Evol. 2016. V. 33. P. 915–927.
Дополнительные материалы отсутствуют.


