Микробиология, 2021, T. 90, № 4, стр. 421-429
Fervidicoccus fontis 3639Fd – первая кренархеота, способная расти на липидах
А. И. Карасева a, b, А. Г. Ельченинов b, А. А. Перевалова b, К. С. Заюлина b, Т. В. Кочеткова b, *, И. В. Кубланов b
a Московский физико-технический институт, Физтех-школа биологической и медицинской физики
141700 Долгопрудный, Россия
b Институт микробиологии им. С.Н. Виноградского, ФИЦ Биотехнологии РАН
119071 Москва, Россия
* E-mail: kochetkova.tatiana.v@gmail.com
Поступила в редакцию 11.12.2020
После доработки 13.03.2021
Принята к публикации 15.03.2021
Аннотация
К настоящему времени способность прокариот расти на липидах показана только для представителей бактерий. Единственным исключением является Thermococcus sibiricus, представитель филума Euryarchaeota, выделенный из высокотемпературной нефтедобывающей скважины и способный расти на оливковом масле. В данной работе из горячего источника Камчатки нами была выделена чистая культура строгой анаэробной археи, штамм 3639Fd (=VKM B-3509, =KCTC 25228), растущей на различных липидах (трибутирине, триолеине, кунжутном, хлопковом и подсолнечном маслах) при 70°С и рН 5.5–6.0. При росте на трибутирине образуется бутират, углекислый газ и водород. Согласно анализу последовательности гена 16S рРНК, а также ДНК–ДНК гибридизации in silico, выделенный микроорганизм является штаммом вида Fervidicoccus fontis, представителя филума Crenarchaeota. В геномах F. fontis 3639Fd и типового штамма этого вида, Kam940T, были обнаружены гены α/β-гидролаз, ближайшими охарактеризованными гомологами которых являются различные карбоксилэстеразы (КФ 3.1.1) – ферменты, ответственные за гидролиз липидов. Таким образом, F. fontis является первой кренархеотой, способной получать энергию за счет гидролиза липидных субстратов.
Липиды – сложные органические соединения, являющиеся, наряду с белками, углеводами и нуклеиновыми кислотами, основными компонентами живых клеток. Ферменты, осуществляющие гидролиз липидов, относятся к группе карбоксилэстераз (КФ 3.1.1). Карбоксилэстеразы, расщепляющие сложные эфиры с короткой цепью жирных кислот (С2–С10) часто называются эстеразами, а расщепляющие сложные эфиры с длинной цепью жирных кислот (более 10 атомов углерода) – липазами. При этом механизм действия обеих групп карбоксилэстераз одинаков: в активном центре располагается аминокислотная триада Ser–Asp(Glu)–His, которая участвует в расщеплении сложноэфирных связей (Bornscheuer, 2002). Более того, зачастую один и тот же фермент обладает обоими типами активностей (Yang et al., 2019). Помимо широкой субстратной специфичности, липазы и эстеразы не нуждаются в кофакторах и проявляют высокую активность в органических растворителях (López-Iglesias, Gotor–Fernández, 2015).
Липазы и эстеразы имеют широкий потенциал применения: их используют в очистке сточных вод; синтезе оптически чистых соединений (фармацевтических производных, сложных эфиров и аминокислот), биодизеля и биополимеров; в производстве моющих средств, косметики, бумаги, кожи, пищевых продуктов (Bornscheuer, 2002; Anobom et al., 2014; Salihu, Alam, 2015; Cabrera, Blamey, 2018). Продажи липаз на мировом рынке достигли 590.5 млн долларов США к 2020 году при среднегодовом темпе роста 6.5% (Chandra et al., 2020).
Поиск новых липаз и/или микроорганизмов, растущих на липидах, является одним из актуальных вопросов современной биотехнологии и микробиологии. В существующих технологиях источниками этих ферментов чаще всего являются бактерии (представители родов Bacillus, Achromobacter, Alcaligenes, Arthrobacter, Pseudomonas и др.) и грибы (например, представители родов Candida, Rhizomucor, Aspergillus, Thermomucor). По сравнению с растениями и животными, прокариотические и грибные продуценты предпочтительнее из-за простоты культивирования и проведения генетических модификаций, большого выхода требуемого фермента и его повышенной активности (Chandra et al., 2020). Липолитические археи также являются перспективными для применения, поскольку среди архей встречается большое количество экстремофильных организмов, включая “рекордсменов”, живущих у известных нам границ параметров жизни (температуры, рН, солености, давления и т.д.). Это делает их самих и их ферменты устойчивыми к экстремальным воздействиям (Littlechild, 2015), необходимым во многих производственных процессах. Наиболее изученной группой архей в отношении липолитических свойств являются галофилы (Delgado-García et al., 2018). Что касается термофильных архей, то гены карбоксилэстераз были детектированы в геномах некоторых гипертермофилов: Pyrococcus furiosus, Metallosphaera sedula, Pyrobaculum caldifontis, Aeropyrum pernix, Thermococcus kodakarensis, Archaeglobus fulgidus, Picrophilus torridus (Soni et al., 2019), большинства видов рода Sulfolobus – S. solfataricus (Mandrich et al., 2007), S. acidophilus (Soni et al., 2019), S. shibatae (Huddleston et al., 1995), S. acidocaldarius (Sobek, Gorisch, 1988), S. tokodaii (Suzuki et al., 2004) и S. islandicus (Stiefler-Jensen et al., 2017). Эстеразные и липазные активности перечисленных выше архей показаны на экстрактах их клеток, выросших на комплексных пептидных субстратах (триптоне/пептоне/дрожжевом экстракте) или сахарозе, или после экспрессии генов их карбоксилэстераз в Escherichia coli (Soni et al., 2019). В качестве субстрата для определения эстеразной и липазной активностей чаще всего используют трибутирин (сложный эфир глицерина и трех остатков масляной кислоты) и триолеин (сложный эфир глицерина и трех остатков олеиновой кислоты) (Hotta et al., 2002). В то же время до сих пор нет работ по культивированию архей на липидных субстратах, за исключением краткого упоминания способности гипертермофильной археи T. sibiricus расти на оливковом масле (Mardanov et al., 2009), не включающего четких микробиологических и генетических доказательств этого свойства.
В данной работе описывается экстремально термофильный анаэробный штамм 3639Fd, первый представитель филума Crenarchaeota, выделенный на трибутирине и способный расти на различных липидах.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Выделение и характеристика объекта исследования. Штамм 3639Fd был выделен из накопительной культуры, содержащей осадки и воду из горячего источника Вертолетный (N 54°30.005, E 160°00.732; 59°C, pH 6.5), расположенного в кальдере Узон (Камчатка, Россия), и инкубированной в течение недели in situ с добавлением трибутирина (1/100 об./об.) (“Fluka”, США) и дрожжевого экстракта (ДЭ) (0.1 г/л) (“Хеликон”, Россия). Для последующей работы, выделения и характеристики чистой культуры использовали строго анаэробную модифицированную среду Пфеннига (Podosokorskaya et al., 2011). Пробирки со средой автоклавировали при 121°C, 101 кПа в течение 1 ч или, если в среду была добавлена сера, при 50 кПа в течение 40 мин. Растворимые субстраты: глицерин (“Химреактив”, Россия), бутират (“Sigma Aldrich”, США), ДЭ, пептон (“Хеликон”, Россия), триптон (“Диаэм”, Россия), казеин (“Реахим”, Россия), пируват (“AppliChem”, Германия), лактат (“Sigma”, США), фумарат (“Merck”, Германия), тартрат (“Fluka”, США), сукцинат (“Реахим”, Россия), малат (“Sigma Aldrich”, США), глюкозу, ксилозу, рамнозу, мальтозу, сахарозу (все “Реахим”, Россия), целлобиозу (‘Sigma Aldrich”, США), галактозу (“Serva”, Германия), арабинозу (“Fluka”, США) – вносили после стерилизации в виде стерильных растворов до конечной концентрации 1 г/л. Жирорастворимые субстраты: триглицериды (трибутирин, триолеин – “Sigma”, США), пищевые масла (кунжутное, хлопковое, оливковое, подсолнечное), эфиры длинноцепочечных жирных кислот (пальмитат и стеарат – “Sigma”, США) – вносили после стерилизации по каплям до конечной концентрации 5–10 мл/л. Нерастворимые субстраты: гуаровую камедь (“CPKelco”, США), ксилан (“Megazyme”, Ирландия), аморфную целлюлозу – вносили до стерилизации до конечной концентрации 1 г/л. Чистая культура штамма 3639Fd была получена методом предельных разведений.
Все эксперименты по описанию чистой культуры проводили в 18-мл пробирках Хангейта (объем среды 10 мл) с плотными резиновыми пробками и завинчивающимися крышками в атмосфере N2. Для определения оптимальной температуры роста штамм 3639Fd инкубировали в течении трех–шести дней с трибутирином в качестве субстрата при температурах 50, 55, 60, 65, 70, 78 и 83°С и рН 6.0. Для определения оптимального рН для роста штамм инкубировали при 70°С с использованием следующих буферов: ацетатного (диапазон значений рН 3.0–5.5) (“Sigma Aldrich”, США), бикарбонатного (5.5–6.5), MOPS (7.0–7.5) (“Диаэм”, Россия) и HEPBS (8.0–8.5) (“Sigma”, США) в конечной концентрации 10 мМ. Способность штамма восстанавливать различные акцепторы электронов при росте на трибутирине проверяли с ${\text{SO}}_{3}^{{2 - }},$ ${\text{SO}}_{4}^{{2 - }},$ ${{{\text{S}}}_{2}}{\text{O}}_{3}^{{2 - }}$ (в виде солей натрия в конечной концентрации 10 мМ) или S0 (1 г/л). Параметры роста и субстратные предпочтения определяли в оптимальных для штамма условиях (70°С, рН 5.5–5.6). Для выявления роста на определенном субстрате, штамм 3639Fd последовательно пересевали три раза в одних и тех же условиях. Все эксперименты проводили в трех повторностях.
Рост штамма оценивали прямым подсчетом клеток с использованием фазово-контрастного микроскопа Olympus CX-41RF (“Olympus”, Япония). Образование сероводорода определяли колориметрически с N,N-диметилпарафенилендиамином в модификации Трюпера и Шлегеля (Trüper, Schlegel, 1964) с использованием спектрофотометра Beckman (модель 35) при λ = 670 нм. Летучие жирные кислоты определяли методом ГЖХ на хроматографе Кристалл 5000.2 (“Хроматэк”, Россия) с пламенно-ионизационным детектором. Разделение проводили на капиллярной колонке Zebron ZB-WAXplus (“Phenomenex”, США) в режиме программирования температуры. Определение сахаров и спиртов проводили при помощи ВЭЖХ на хроматографе Стайер (“Аквилон”, Россия). Разделение проводили на колонке Aminex HPX-87H (“BioRad”, США) в изократическом режиме с элюентом 25 мМ H2SO4 и расходом 0.6 мл/мин. Регистрацию сигналов проводили двумя детекторами: рефрактометрическим Smartline 2300 (“Knauer”, Германия) и ультрафиолетовым UVV 104 при 210 нм (“Аквилон”, Россия) по последовательной схеме. Газообразные продукты измеряли с помощью “3700” модифицированного газового хроматографа (“ZIOC RAS”, Россия) с поддержкой аналитического ПО Phoenix v. 3.6.0 (“BSoft”, Россия).
Штамм 3639Fd был депонирован во Всероссийской коллекции микроорганизмов (ВКМ) под номером VKM B-3509 и в Корейской коллекции типовых культур (KCTC) под номером KCTC 25228.
Выделение геномной ДНК. Для выделения геномной ДНК штамма 3639Fd клетки, находящиеся на ранней стационарной фазе, собирали центрифугированием при 9000 об./мин в течение 15 мин. Осадок ресуспендировали в буфере TNE (10 мМ Трис-HCl, 10 мМ NaCl, 5 мМ ЭДТА; pH 8.0), добавляли протеиназу К (100 мкг/мл), SDS (0.5% по объему), 0.015 М ЭДТА и инкубировали смесь при 50°С в течение 1 ч. К инкубационной смеси добавляли смесь фенол–хлороформ (1 : 1), перемешивали и центрифугировали при 9000 об./мин в течение 15 мин. Супернатант дважды экстрагировали равным объемом хлороформа, затем к водной фазе добавляли 1/10 часть 3 М ацетата Na (pH 5.2) и 2 части 96% этанола. Полученную слизь (или “медузу”) собирали стерильным шпателем, два раза промывали в 70% этаноле, подсушивали и растворяли в стерильном TE буфере (10 мМ Трис-HCl, 1 мМ ЭДТА; pH 8.0). Полученную ДНК обрабатывали раствором РНК-азы А (10 мкг/мл), для этого смесь инкубировали в течение 30 мин при 37°С, затем ДНК дважды экcтрагировали равным объемом хлороформа и “высаживали” 96% этанолом, дополнительно промывали 70% этанолом и растворяли в стерильной воде. Концентрацию ДНК измеряли на флуориметре Qubit 2.0 (“Invitrogen/Life Technologies”, США).
Секвенирование и сборка генома были выполнены с помощью высокопроизводительной технологии Illumina. Геномная ДНК штамма 3639Fd была фрагментирована на участки размером ~500 п.н. с использованием ультразвукового гомогенизатора BioRuptor® UCD-200 (“Diagenode”, Бельгия). Библиотеки были подготовлены с использованием NEBNext® UltraTM DNA Library Prep kit for Illumina® (“New England Biolabs”, США) в соответствии с инструкциями производителя. Библиотеки клонов секвенировали на MiSeq Sequencing System (“Illumina”, США) с помощью 2 × 150 bp reagent kit. После фильтрации по качеству с использованием CLC Genomic Workbench 10.0 (“Qiagen”, Германия) было получено 1 307 626 прочтений.
Сборку генома проводили с использованием программы SPAdes 3.13.0 (Antipov et al., 2016). На конечном этапе было получено 10 контигов, общий размер которых составлял 1 248 168 п.н. Последовательность генома была депонирована в GenBank под номером JADEZV000000000.1.
Аннотацию генома проводили с помощью пайплайна NCBI PGAP (Arefiev et al., 2020). Сравнительные анализы, основанные на полной последовательности генома, выполняли следующим образом: среднее сходство по нуклеотидам (ANI) вычисляли с использованием модуля pyani 0.2.8 с выбором метода ANIb (Pritchard et al., 2016); среднее сходство по аминокислотам (AAI) – с помощью скрипта aai.rb (Rodriguez-R, Konstantinidis, 2016); in silico ДНК–ДНК гибридизацию – с использованием сервера GGDC 2.1 (Meier-Kolthoff et al., 2013) с выбором BLAST+ в качестве метода выравнивания.
Для определения пан-геномов штаммов F. fontis Kam940T и 3639Fd использовали пакет Proteinortho 5.16 (Lechner et al., 2011) со следующими параметрами: 95% сходство последовательностей (нижняя граница для одного вида по AAI), 50% взаимное покрытие. Визуализацию анализа проводили в пакете Venn (https://cran.r-project.org/ web/packages/venn/index.html). Для уникальных белков определяли распределение по функциональным категориям COG (clusters of orthologous groups) с использованием webMGA (Wu et al., 2011). Выравнивание последовательностей генов проводили с помощью алгоритма Mafft v. 7 (Katoh, Standley, 2002).
Проверка способности представителей рода Fervidicoccus к росту на липидах. Типовой штамм Fervidicoccus fontis Kam940Т (=DSM 19380T, =VKM B-2539T), а также имеющиеся в коллекции лаборатории метаболизма экстремофильных прокариот (ФИЦ Биотехнологии РАН) штаммы этого рода (FoA-16 и 1507) проверяли на способность расти на липидных субстратах на той же минеральной среде (Podosokorskaya et al., 2011) и при оптимальных для каждого отдельного штамма условиях: Kam940Т – при pH 5.6 c добавлением 0.2 г/л ДЭ при 70°С; FoA-16 – при pH 4.8, с добавлением 0.1 г/л ДЭ при 78°С; штамм 1507– при pH 6.0 c добавлением 0.1 г/л ДЭ при 70°С. Для выявления роста на определенном субстрате, штаммы последовательно пересевали три раза в одних и тех же условиях. Все эксперименты проводили в двух повторностях.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Выделение и характеристика штамма 3639Fd. Штамм 3639Fd был выделен на трибутирине с добавлением 0.05 г/л ДЭ при 70°С и рН 5.5 методом предельных разведений и представлял собой небольшие кокки правильной формы. Выделенный штамм оказался экстремально термофильным, умеренно ацидофильным микроорганизмом, растущим в диапазоне температур от 55 до 83°C и рН от 4.5 до 6.5 с оптимумом при 70°С и рН 5.5–5.6. Архея облигатно зависела от присутствия в среде дрожжевого экстракта (ДЭ) в оптимальной концентрации 0.1 г/л.
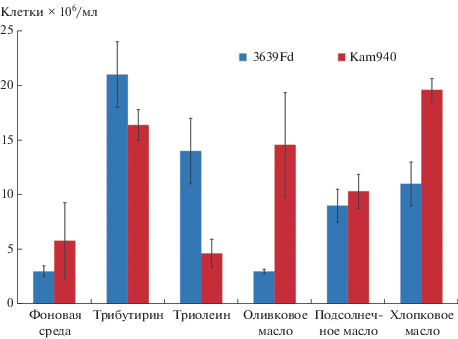
Штамм 3639Fd был способен расти на трибутирине с образованием бутирата (рис. 1), достигая максимального урожая клеток 2.1 ± 0.3 × 107 кл./мл за 67 ч при оптимальных условиях (70°С, рН 5.6, 0.1 г/л ДЭ). Также во время роста на трибутирине наблюдали образование СО2 и H2 (данные не показаны). Минимальное время удвоения в данных условиях составило 1.2 ч. Урожай клеток на фоновой среде с 0.1 г/л ДЭ без добавления трибутирина составлял 0.3 ± 0.05 × 107 кл./мл (рис. 2). При росте на трибутирине с серой плотность клеток увеличивалась вдвое (5.4 ± 0.5 × 107 кл./мл) и отмечалось образование сероводорода. Тиосульфат незначительно стимулировал рост штамма (3.2 ± ± 0.2 × 107 кл./мл), присутствие сульфата не оказывало влияния, а сульфит ингибировал рост штамма на трибутирине. 3639Fd также рос на триолеине (1.4 ± 0.03 × 107 кл./мл) и некоторых маслах: кунжутном (1.1 ± 0.1 × 107 кл./мл), хлопковом (1.1 ± ± 0.2 × 107 кл./мл) и подсолнечном (0.9 ± 0.15 × × 107 кл./мл) (рис. 2). Помимо липидов, штамм 3639Fd сбраживал триптон (1.2 ± 0.3 × 107 кл./мл), казеин, ДЭ (2 ± 0.5 × 107 кл./мл); органические кислоты, в том числе бутират (1 ± 0.05 × 107 кл./мл); моно- и дисахара. Штамм не рос на глицерине, оливковом масле, пептоне, полисахаридах. Неспособность к росту на оливковом масле, при том, что микроорганизм рос на чистом триолеате, возможно, обусловлена присутствием в его составе неизвестных примесей, которые могли ингибировать рост данной археи.
Рис. 1.
Рост Fervidicoccus fontis 3639Fd (1) и образование бутирата (2) на трибутирине в оптимальных условиях (рН 5.6, 70°С, 0.1 г/л ДЭ). Концентрация бутирата в контрольных образцах: на незасеянной среде с трибутирином (3) и на среде, выросшей на 1.0 г/л ДЭ (4).
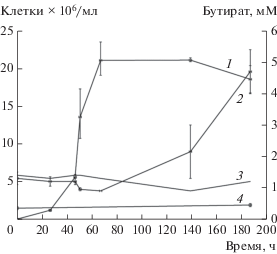
Рис. 2.
Рост Fervidicoccus fontis Kam940T (красные/черные столбики) и 3639Fd (синие/серые столбики) на липидных субстратах.
Филогенетическое и таксономическое положение штамма 3639Fd. Последовательность гена 16S рРНК штамма 3639Fd оказалась идентичной на 99.93% последовательности гена Fervidicoccus fontis Kam940T (Perevalova et al., 2010). Значения AAI, ANI и in silico ДНК–ДНК гибридизации для штамма 3639Fd и F. fontis Kam940T, составили 99.38, 99.63 и 97.1% соответственно. Такие значения превышают пороговые для отдельных видов – 95% по AAI (Konstantinidis, Tiedje, 2005), 95% по ANI (Kim et al., 2014), 70% по ДДГ (Wayne et al., 1987). Таким образом, основываясь на сходстве последовательностей генов 16S рРНК и полногеномных сравнениях, штамм 3639Fd был отнесен к виду F. fontis.
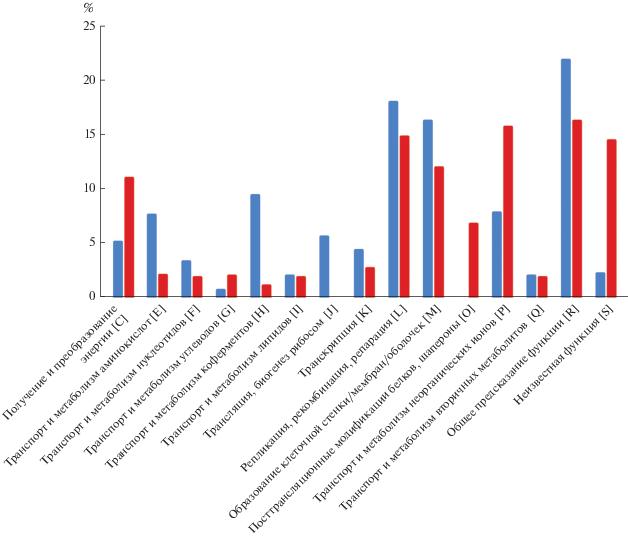
Анализ генома Fervidicoccus fontis 3639Fd и генетические доказательства способности гидролизовать липиды. Согласно аннотации PGAP геном F. fontis 3639Fd состоит из 1385 генов, включая 1322 белок-кодирующих генов, 48 РНК (3 рРНК, 43 тРНК, 2 нкРНК) и 15 псевдогенов. Поскольку фенотипически штамм 3639Fd отличается от типового штамма Kam940T, а среднее сходство их геномов и протеомов довольно высоко (ANI и AAI = 99.63, 99.38% соответственно), было решено определить общую (core genome) и уни-кальную части генома вида F. fontis. Оказалось, что 1125 белков являются общими; в то же время 159 найдены только у штамма Kam940T, 111 – у штамма 3639Fd (рис. 3). Распределение уникальных для каждого из штаммов белков по функциональным категориям COG (рис. 4) показало, что количество белков большинства групп (категории F, I, K, L, M и R) примерно равное. В то же время белки, входящие в категории E (метаболизм и транспорт аминокислот) и H (метаболизм и транспорт кофакторов), более многочисленны у штамма 3639Fd. И, наоборот, белки категории С (образование и запасание энергии), P (метаболизм и транспорт неорганических ионов) и S (неизвестная функция) сильнее представлены в штамме Kam940T.
Рис. 3.
Количество общих (кор-геном) и уникальных белков между штаммами Fervidicoccus fontis Kam940T и 3639Fd.
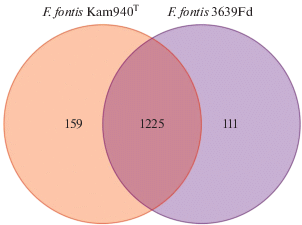
Рис. 4.
Распределение уникальных белков штаммов Fervidicoccus fontis Kam940T (красные столбики) и 3639Fd (синие столбики) по выбранным функциональным категориям COG.
Поскольку в этой работе центральным свойством штамма 3639Fd была способность расти на триглицеридах, нами были предприняты попытки обнаружения в геноме генов, кодирующих липазы/эстеразы. В геноме штамма 3639Fd обнаружен ген α/β-гидролазы 6 семейства, ближайшими охарактеризованными гомологами которой являются различные карбоксилэстеразы (КФ 3.1.1), включая моноацилглицерол-липазы из эукариот (процент сходства аминокислотной последовательности ~30–33% при покрытии ~60%). При выравнивании последовательности данной карбоксилэстеразы с представителями 15 семейств эстераз (Zarafeta et al., 2016) был определен ее каталитический мотив – GASMGG, который соответствует семействам III и V эстераз (Zarafeta et al., 2016). Ближайшими неохарактеризованными белками являются α/β-гидролазы из архей, включая гомолога типового штамма F. fontis Kam940Т. В геноме 3639Fd не было обнаружено генов, кодирующих ферменты известных путей окисления глицерина и жирных кислот. Первое подтверждается, а второе не подтверждается ростовыми экспериментами, так как нами было показано, что штамм способен расти на органических кислотах. Таким образом, в окислении этих субстратов, по-видимому, участвуют другие, пока еще неизвестные ферменты.
Проверка способности представителей рода Fervidicoccus к росту на липидах. После 4 сут инкубации при 70°C урожай клеток типового штамма F. fontis Kam940Т на трибутирине составил 1.6 ± ± 0.1 × 107 кл./мл, на триолеине 0.4 ± 0.1 × 107 кл./мл, на оливковом масле 1.4 ± 0.4 × 107 кл./мл, на хлопковом масле 2.0 ± 0.1 × 107 кл./мл, на подсолнечном масле 1.0 ± 0.1 × 107 кл./мл, на контрольной фоновой среде (без субстрата) 0.8 ± 0.3 × 107 кл./мл (рис. 2). Роста штаммов F. fontis 1507 и FoA-16 после 7 сут культивирования на трибутирине не наблюдали. Таким образом, было показано, что кроме штамма 3639Fd, только Kam940T способен расти на некоторых липидных субстратах, таких как трибутирин и хлопковое масло.
Fervidicoccus fontis является единственным видом внутри порядка Fervidicoccales филума Crenarchaeota. Типовой штамм этого вида, Kam940T, был выделен из горячего источника Камчатки и рос при 70°C на белках и пептидах (Perevalova et al., 2010). Молекулярно-экологические исследования показали, что представители этого рода повсеместно встречаются в горячих источниках вулканических регионов Йеллоустонского национального парка, Камчатки, Исландии, Новой Зеландии (Lebedinsky et al., 2013), Китая (Jiang et al., 2016), а также являются преобладающим архейным представителем и в некоторых мезофильных местах обитания, например, в болотных почвах (Lv et al., 2014). Это говорит о значительной экологической роли представителей данного рода в анаэробных экотопах, где он занимает место деструктора органики, успешно конкурируя с другими хемоорганотрофными прокариотами.
В данной работе мы показали новое свойство представителей рода Fervidicoccus, заключающееся в способности расти на разнообразных триглицеридах. Также в геноме 3639Fd был выявлен ген, кодирующий карбоксилэстеразу, относящуюся к III, V или новому семейству эстераз. Подобный ген был найден и в геноме F. fontis Kam940T, и, впоследствии, была продемонстрирована способность этого штамма расти на липидных субстратах.
Способность двух штаммов F. fontis расти на липидах не только расширяет представления об экологических возможностях рода Fervidicoccus, но и выделяет его среди всех известных культивируемых представителей Crenarchaeota, для которых роста на липидах не было показано. По имеющимся данным, среди других архей известен лишь один штамм, растущий на липидах (на оливковом масле) – это представитель филума Euryarchaeota – Thermococcus sibiricus. В геноме этой археи были найдены гены эстераз и липаз (Mardanov et al., 2009). Однако наш анализ четырех предполагаемых эстераз и липаз данного микроорганизма указывает на то, что ни одна из них не относится к карбоксилэстерзам и, таким образом, не может участвовать в гидролизе глицеролипидов. Одной из основных причин подобного недостатка информации о росте архей на липидных субстратах является трудоемкость выделения архей в чистые культуры и их культивирования (Makarova et al., 2019).
Дальнейший детальный анализ карбоксилэстеразы, обнаруженной в данной работе, поможет выявить механизмы липолитической активности архей и способствовать выделению новых штаммов архей-липолитиков с последующей характеристикой новых липаз, обладающих высоким биотехнологическим потенциалом.
Список литературы
Anobom C.D., Pinheiro A.S., De-Andrade R.A., Aguieiras E.C., Andrade G.C., Moura M.V., Almeida R.V., Freire D.M. From structure to catalysis: recent developments in the biotechnological applications of lipases // BioMed Res. Int. 2014. V. 2014. P. 684506.
Antipov D., Korobeynikov A., McLean J.S., Pevzner P.A. HybridSPAdes: an algorithm for hybrid assembly of short and long reads // Bioinformatics. 2016. V. 32. P. 1009–1015.
Arefiev V., Kovalenko G., Frant M., Chumachenko T., Polyvianna Y., Pivnenko S., Bolotin V., Mayboroda O., Solodiankin O., Tarasov O., Bezyemenni M., Lyon C., Redlinger M., Sapachova M., Mezhenskyi A.A., Ducluzeau A., Bortz E., Gerilovych A., Drown D.M., Bezyemenni M. Complete genome sequence of Salmonella enterica subsp. enterica serovar Kottbus Strain Kharkiv, isolated from a commercial pork production facility in Ukraine // Microbiol. Resour. Announc. 2020. V. 9. e01171-20.
Bornscheuer U.T. Microbial carboxyl esterases: classification, properties and application in biocatalysis // FEMS Microbiol. Rev. 2002. V. 26. P. 73–81.
Cabrera M.Á., Blamey J.M. Biotechnological applications of archaeal enzymes from extreme environments // Biol. Resh. 2018. V. 51. https://doi.org/10.1186/s40659-018-0186-3
Chandra P., Singh E.R., Arora P.K. Microbial lipases and their industrial applications: A comprehensive review // Microbial Cell Factories. 2020. V. 19. P. 169. https://doi.org/10.1186/s12934-020-01428-8
Delgado-García M., Rodríguez J.A., Mateos-Díaz J.C., Aguilar C.N., Rodríguez-Herrera R., Camacho-Ruíz R.M. Halophilic archaeal lipases and esterases: activity, stability, and food applications // Enzymes in Food Technology. Singapore: Springer, 2018. P. 243–262.
Hotta Y., Ezaki S., Atomi H., Imanaka T. Extremely stable and versatile carboxylesterase from a hyperthermophilic archaeon // Appl. Environ. Microbiol. 2002. V. 68. P. 3925–3931.
Huddleston S., Yallop C.A., Charalambous B.M. The identification and partial characterization of a novel inducible extracellular thermostable esterase from the archaeon Sulfolobus shibatae // Biochem. Biophys. Res. Commun. 1995. V. 216. P. 495–500.
Jiang Z., Li P., Jiang D., Dai X., Zhang R., Wang Y., Wang Y. Microbial community structure and arsenic biogeochemistry in an acid vapor-formed spring in Tengchong geothermal area, China // Publ. Library Sci. 2016. V. 11. P. e0146331.
Katoh K., Standley D.M. MAFFT multiple sequence alignment software version 7: improvements in performance and usability // Mol. Biol. Evol. 2013. V. 30. P. 772–780.
Kim M., Oh H.S., Park S.C., Chun J. Towards a taxonomic coherence between average nucleotide identity and 16S r-RNA gene sequence similarity for species demarcation of prokaryotes // Int. J. Syst. Evol. Microbiol. 2014. V. 64. P. 346–351.
Konstantinidis K.T., Tiedje J.M. Towards a genome-based taxonomy for prokaryotes // J. Bacteriol. ASM. 2005. V. 187. P. 6258–6264.
Lebedinsky A.V., Mardanov A.V., Kublanov I.V., Gumerov V.M., Beletsky A.V., Perevalova A.A., Bidzhieva S.Kh., Bonch-Osmolovskaya E.A., Skryabin K.G., Ravin N.V. Analysis of the complete genome of Fervidococcus fontis confirms the distinct phylogenetic position of the order Fervidicoccales and suggests its environmental function // Extremophiles. 2014. V. 18. P. 295–309.
Lechner M., Findeiß S., Steiner L., Marz M., Stadler P.F., Prohaska S.J. Proteinortho: Detection of (Co-)orthologs in large-scale analysis // BMC Bioinform. 2011. V. 12. P. 124.
Littlechild J.A. Archaeal enzymes and applications in industrial biocatalysts // Archaea. 2015. V. 2015. P. 10.
Lõpez-Iglesias M., Gotor-Fernández V. Recent advances in biocatalytic promiscuity: Hydrolase-catalyzed reactions for nonconventional transformations // Chem. Rec. 2015. V. 15. P. 743–759.
Lv X., Yu J., Fu Y., Ma B., Qu F., Ning K., Wu H. A meta-analysis of the bacterial and archaeal diversity observed in wetland soils // Sci. World J. 2014. Art. 437684.
Makarova K.S., Wolf Y.I., Koonin E.V. Towards functional characterization of archaeal genome dark matter // Biochem. Soc. Trans. 2019. V. 19. P. 389–398.
Mandrich L., Pezzullo M., Rossi M., Manco G. SSoNΔ and SsoNΔlong: two thermostable esterases from the same ORF in the archaeon Sulfolobus solfataricus? // Archaea. 2007. V. 2. P. 109–115.
Mardanov A.V., Ravin N.V., Svetlitchnyi V.A., Beletsky A.V., Miroshnichenko M.L., Bonch-Osmolovskaya E.A., Skryabin K.G. Metabolic versatility and indigenous origin of the archaeon Thermococcus sibiricus, isolated from a siberian oil reservoir, as revealed by genome analysis // Appl. Environ. Microbiol. 2009. V. 75. P. 4580–4588.
Meier-Kolthoff J.P., Auch A.F., Klenk H.P., Göker M. Genome sequence-based species delimitation with confidence intervals and improved distance functions // BMC Bioinform. 2013. V. 14. P. 60.
Perevalova A.A., Bidzhieva S.K., Kublanov I.V., Hinrichs K.-U., Liu X.L., Mardanov A.V., Lebedinsky A.V., Bonch-Osmolovskaya E.A. Fervidicoccus fontis gen. nov., sp. nov., an anaerobic, thermophilic crenarchaeote from terrestrial hot springs, and proposal of Fervidicoccaceae fam. nov. and Fervidicoccales ord. nov. // Int. J. Syst. Evol. Microbiol. 2010. V. 60. P. 2082–2088.
Pinheiro A.S., De-Andrade R.A., Aguieiras E.C., Andrade G.C., Moura M.V., Almeida R.V., Freire D.M. From structure to catalysis: Recent developments in the biotechnological applications of lipases // BioMed Res. Int. 2014. V. 2014. P. 684506.
Podosokorskaya O.A., Merkel A.Y., Kolganova T.V., Chernyh N.A., Miroshnichenko M.L., Bonch-Osmolovskaya E.A., Kublanov I.V. Fervidobacterium riparium sp. nov., a thermophilic anaerobic cellulolytic bacterium isolated from a hot spring // Int. J. Syst. Evol. Microbiol. 2011. V. 61. P. 2697–2701.
Pritchard L., Glover R.H., Humphris S., Elphinstone J.G., Toth I.K. Genomics and taxonomy in diagnostics for food security: Soft-rotting enterobacterial plant pathogens // Anal. Methods. 2016. V. 8. P. 12–24.
Rodriguez-R L.M., Konstantinidis K.T. The enveomics collection: a toolbox for specialized analyses of microbial genomes and metagenomes // PeerJ Preprints. 2016. e1900v1.
Salihu A., Alam M.Z. Solvent tolerant lipases: A review // Proc. Biochem. 2015. V. 50. P. 86–96.
Sobek H., Görisch H. Purification and characterization of a heat-stable esterase from the thermoacidophilic archaebacterium Sulfolobus acidocaldarius. // Biochem. J. 1988. V. 250. P. 453–458.
Soni S., Sathe S.S., Sheth R.R., Tiwari P., Vadgama R.K.N., Odaneth A.A., Lali A.M., Chandrayan S.K. N-terminal domain replacement changes an archaeal monoacylglycerol lipase into a triacylglycerol lipase // Biotechnol. Biofuels. 2019. V. 12. P. 110.
Stiefler-Jensen D., Schwarz-Linnet T., de Lichtenberg C., Nguyen T.T., Rand K.D., Huang L., She Q., Teilum K. The extraordinary thermal stability of EstA from S. islandicus is independent of post translational modifications // Protein Sci. 2017. V. 26. P. 1819–1827.
Suzuki Y., Miyamoto K., Ohta H. A novel thermostable esterase from the thermoacidophilic archaeon Sulfolobustokodaii strain 7 // FEMS Microbiol. Lett. 2004. V. 236. P. 97–102.
Trüper H.G., Schlegel H.G. Sulphur metabolism in Thiorhodaceae I. Quantitative measurements on growing cells of Chromatium okenii // Antonie Van Leeuwenhoek. 1964. V. 30. P. 225–238.
Wayne L.G., Moore W.E.C., Brenner D.J., Colwell R.R., Grimont P.A.D., Kandler O., Krichevsky M.I., Murray G.E., Stackebrandt E., Starr M.P., Truper H.G. Report of the ad hoc committee on reconciliation of approaches to bacterial systematics // Int. J. Syst. Evol. Microbiol. 1987. V. 37. P. 463–464.
Wu S., Zhu Z., Fu L., Niu B., Li W. WebMGA: A customizable web server for fast metagenomic sequence analysis // BMC Genomics. 2011. V. 12. P. 444.
Yang D., Li S., Stabenow J., Zalduondo L., Kong Y. Mycobacterium tuberculosis LipE has a lipase/esterase activity and is important for intracellular growth and in vivo infection // Infect. Immun. 2019. V. 88. e00750–19.
Zarafeta D., Moschidi D., Ladoukakis E., Gavrilov S., Chrysina E.D., Chatziioannou A., Kublanov I., Skretas G., Kolisis F.N. Metagenomic mining for thermostable esterolytic enzymes uncovers a new family of bacterial esterases // Sci. Rep. Nature. 2016. V. 6. P. 1–16.
Дополнительные материалы отсутствуют.


