Микробиология, 2021, T. 90, № 4, стр. 471-479
Функциональная специфичность продукта гена nifA в пределах группы клубеньковых бактерий
А. А. Владимирова a, *, Р. С. Гуменко a, Е. С. Акимова a, Ал. Х. Баймиев a, Ан. Х. Баймиев a
a Институт биохимии и генетики Российской академии наук
450054 Уфа, Россия
* E-mail: vladimirovaw@bk.ru
Поступила в редакцию 22.01.2021
После доработки 04.03.2021
Принята к публикации 10.03.2021
Аннотация
Проведен анализ функциональной активности разных филогенетических вариантов белка-регулятора транскрипции генов нитрогеназного комплекса NifA в клетках клубеньковых бактерий (ризобий) разных таксонов. Для этого были созданы рекомбинантные штаммы ризобий родов Mesorhizobium, Ensifer (Sinorhizobium) и Rhizobium с генно-инженерными конструкциями на основе плазмиды pJN105, содержащие разные филогенетические варианты гена nifA, характерные для бактерий родов Mesorhizobium, Ensifer и Rhizobium под управлением бактериального промотора ParaBAD. Показано, что у свободноживущих рекомбинантных клеток всех трех родов бактерий привнесение в их геном дополнительной экспрессирующейся копии гена nifA приводит к появлению детектируемой нитрогеназной активности. При этом уровень нитрогеназной активности не имеет прямой связи с филогенией привнесенного гена nifA, что, скорее всего, объясняется универсальностью продукта исследуемого гена внутри проанализированной группы бактерий. Полученные данные указывают на возможность комбинаторных процессов в эволюции генов азотфиксации.
Симбиотическая азотфиксация опосредуется тремя основными группами генов: nod-гены (участвуют в формировании клубенька), nif-гены (от англ. nitrogen fixation, кодируют ферментный комплекс – нитрогеназу) и fix-гены (участвуют в работе нитрогеназного комплекса) (Laranjo et al., 2014). У всех азотфиксирующих микроорганизмов nif-гены организованы в один (характерно чаще для свободноживущих азотфиксаторов) или несколько кластеров (свойственно клубеньковым бактериям) (Dai et al., 2014). При этом данные гены у ризобий могут локализоваться как на симбиотической плазмиде (Rhizobium, Ensifer), так и на хромосомных островках (Mesorhizobium, Bradyrhizobium), но во всех случаях отмечается активное участие этих генов в процессе горизонтального переноса. Биологическая фиксация азота на транскрипционном уровне находится под управлением одноименного продукта гена nifA, который участвует в запуске синтеза белковых компонентов ферментного комплекса – нитрогеназы. Белок NifA является транскрипционным активатором и относится к группе бактериальных энхансер-связывающих белков (bEBPs). Он связывается совместно с сигма-фактором РНК-полимеразы RpoN (σ54) с активирующей последовательностью (UAS; 5'-TGT-N10-ACA-3'), тем самым запуская экспрессию nif-генов (Dixon, Kahn, 2004; Wongdee et al., 2018). В структуре белка NifA выделяют три домена. N‑концевой домен является сигнальным мотивом, центральный – взаимодействует с σ54 и обладает АТФ-азной активностью, необходимой для активации транскрипции, С-концевой домен участвует непосредственно в связывании с ДНК за счет мотива спираль–поворот–спираль (HTH), распознающий специфические сайты посадки белка (Xu et al., 2004; Bush, Dixon, 2012).
Многочисленные молекулярно-генетические исследования природы азотфиксации у различных диазотрофов привели к возможности искусственного создания азотфиксирующих микроорганизмов. Такие попытки были предприняты посредством переноса кластеров nif-генов от различных азотфиксаторов к бактериям, не обладающим данной способностью (Dixon, Postgate, 1972; Setten et al., 2013; Wang et al., 2013; Yang et al., 2014; Han et al., 2015; Li et al., 2016). Однако вопрос о том, как интегрировать гены азотфиксации в клетку-реципиент так, чтобы экспрессия nif-генов не ингибировалась различными факторами, а процесс фиксации азота был эффективным по сравнению с диазотрофами, остается открытым. Некоторые исследователи имели иной подход к созданию эффективных азотфиксирующих штаммов бактерий, основанный на изменении регуляции экспрессии гена nifA. Впервые конститутивная экспрессия nifA в присутствии кислорода и азота была продемонстрирована Buchanan-Wollaston и соавт. в начале 80-х годов (Buchanan-Wollaston et al., 1981). Позднее Kennedy и соавт. отметили активацию экспрессии nif-генов у Azotobacter vinelandii и Azotobacter chroococcum при привнесении экспрессирующегося гена nifA Klebsiella pneumonia (Kennedy, Robson, 1983). Прикладной аспект использования азотфиксатора с измененной регуляцией генов нитрогеназного комплекса описан в работе An и соавт., где было показано, что свободноживущий ассоциативный азотфиксирующий микроорганизм Enterobacter gergoviae с конститутивной экспрессией транскрипционного активатора nifA гена стимулировал (улучшал) рост кукурузы (An et al., 2007). Таким образом, искусственное изменение экспрессии nif-генов оказывает влияние на азотфиксирующий статус микроорганизмов.
Ризобии в свободноживущем состоянии не способны к преобразованию атмосферного азота, поскольку индукция генов азотфиксации (в том числе и nifA гена) происходит в условиях низкого содержания кислорода: в тканях центральной части клубенька (Fischer, 1998). Однако ранее нами была описана возможность активации экспрессии nif-генов ex planta посредством введения в бактериальную клетку дополнительной копии nifA под искусственной регуляцией. Таким образом, в ризобиальные клетки была введена собственная активная копия nifA гена (Баймиев и соавт., 2019).
Ген nifA проявляет невысокую сцепленность с генами, кодирующими белки коровой части нитрогеназного комплекса, и может вовлекаться в горизонтальный перенос отдельно от остальных nif-генов. Это может привести к различным сочетаниям регуляторной и коровой части генов нитрогеназы. На сегодняшний день известно достаточно большое количество случаев, когда бактерии одного штамма в составе своего генома содержат разные филогенетические варианты одноименных nif-генов, что, скорее всего, является результатом комбинаторной эволюции вследствие рекомбинационных процессов между разными таксонами ризобий (Norel, Elmerich 1987; Sullivan et al., 2013; Wongdee et al., 2018). Исходя из этого, исследование функциональной специфичности белка NifA среди клубеньковых бактерий позволит приблизиться к пониманию эволюции генетики азотфиксации и даст возможность целенаправленного создания эффективных сельскохозяйственных штаммов ризобий.
Целью данной работы являлось выявление функциональной специфичности разных филогенетических вариантов регуляторного белка NifA в гетерологичных системах внутри группы клубеньковых бактерий.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объектами исследования были штаммы бактерий из коллекции “Симбионт” ИБГ УФИЦ РАН, выделенные из клубеньков дикорастущих бобовых растений Южного Урала: Vicia sylvatica L. (горошек лесной), Lathyrus pisiformis L. (чина гороховидная) Lotus zhegulensis Klokov (лядвенец жигулевский) и Medicago lupulina L. (люцерна хмелевидная). Для генно-инженерных манипуляций использовали штамм XL-1 Blue E. coli.
Культивирование бактериальных клеток. Выращивание клубеньковых бактерий дикого типа осуществляли на питательной среде YM (1% маннитол, 0.01% NaCl, 0.05% K2HPO4, 0.02% MgSO4, 0.1% дрожжевой экстракт) при 28°С. Отбор трансформированных штаммов проводили на среде YM с селективным антибиотиком – гентамицином (15 мкг/мл). Для анализа азотфиксирующей активности модифицированные штаммы ризобий выращивали на жидкой безазотной среде С40 (0.02% KH2PO4, 0.08% K2HPO4, 0.02% MgSO4 ⋅ 7H2O, FeCl3 (следы), Na2MoO4 (следы), 0.04% дрожжевой экстракт, 2% сахароза) с добавлением антибиотика. Индукцию экспрессии гена nifA, находящегося под управлением промотора ParaBAD, активировали арабинозой в диапазоне концентраций от 0.1 до 2 мM. Клетки E. coli культивировали на LB среде (1% бактотриптон, 0.5% дрожжевой экстракт, 0.5% NaCl) при 37°С.
Работа с ДНК. Выделение тотальной ДНК из клубеньковых бактерий проводили лизированием клеток в 1% Triton X100 и 1% суспензии Chelex100 (Баймиев и соавт., 2011).
Создание генно-инженерной конструкции. В качестве целевого гена использовалась последовательность гена nifA, принадлежащая к трем основным родам: Rhizobium (Rhizobium leguminosarum), Ensifer (Sinorhizobium meliloti) и Mesorhizobium (Mesorhizobium loti). Данные последовательности были амплифицированы с помощью ПЦР с использованием следующих праймеров: nifAvicF 5'-cagtgactgcgaggagttttgtaggcatgatta-3'; nifAvicR 5'-tggcgatcgcggtcactccttcttcaca-3'; nifAsinoF 5'-catgcgcaaacaggacaa-3'; nifAsinoR 5'-gctcgcgacgcctttatc-3'; nifAmesoF 5'-catgcacgatactcgcaac-3'; nifAmesoR 5'-cgcgagccggttcagag-3'. Данные варианты гена nifA клонировали в вектор pJN105 по сайту рестрикции SmaI. Вектор с целевым геном проклонировали в штамме XL-1 Blue E. coli. Затем методом щелочного лизиса по стандартной методике выделили плазмиду в достаточном количестве для трансформации компетентных клеток ризобий (Sambrook, Russell, 2001). Трансформацию штаммов клубеньковых бактерий проводили методом электропорации по протоколу для агробактерий в кювете 0.1 см в приборе MicroPulser (“BioRad”, США).
Анализ азотфиксирующей активности клубеньковых бактерий, как дикого типа, так и модифицированных штаммов проводили методом редукции ацетилена, предложенным Умаровым (Умаров, 1989). Для этого штаммы ризобий выращивали на безазотной среде С40 в течение 1–2 сут (для бактерий рода Rhizobium и Ensifer) и 3–5 сут (род Mezorhizobium) до концентрации клеток 1–2 × 108 КОЕ/мл на термостатируемом орбитальном шейкере ES20 при 180 об./мин и температуре 28°С. Рекомбинантные штаммы выращивались при тех же условиях, но с добавлением селективного антибиотика. Суспензию бактериальных клеток объемом 1 мл переносили в стерильные стеклянные флаконы на 15 мл, содержащие 5 г стерильного песка. Затем флаконы закрывали ватными пробками и помещали в термостат на 28°С на сутки. После инкубации заменяли ватные пробки на стерильные резиновые и во флаконы вводили ацетилен в объеме 1 мл. Через 1 ч проводили анализ содержания ацетилена и этилена по времени выхода каждого газа в исследуемых образцах на газовом хроматографе с пламенным ионизационным детектором Shimadzu GC-2014 (“Shimadzu”, Япония).
Оценку экспрессии nifA-гена у рекомбинантных штаммов проводили методом ОТ-ПЦР. РНК выделяли с применением набора FastRNA Pro Blue Kit (США). Для удаления остатков ДНК препарат РНК обрабатывали ДНКазой свободной от РНКаз (“Thermo Scientific”, США). Используя фермент MMLV-ревертазу (обратная транскриптаза вируса лейкемии мышей) (“Евроген”, Россия) получали первую цепь ДНК, комплементарную мРНК. Реакционная смесь содержала следующие компоненты: 1–5 мкг РНК, 20 мкМ случайного гексамерного праймера (“Fermentas”, США), однократный буфер для ревертазы (50 мМ трис-HCl рН 8.3, 10 мМ MnCl2, 10 мМ DTT, 0.5 мМ спермидина), по 500 мкМ каждого из дезоксинуклеотид-трифосфатов и 5 единиц MMLV-ревертазы. Инкубировали в течение 2 ч при 42°С, затем реакционную смесь инактивировали нагреванием до 70°С 10 мин. Полученные образцы использовали для постановки ОТ-ПЦР.
Выделение тотального белка из культуры бактериальных клеток проводили по описанной ранее методике (Towbin et al., 1979). Вестерн-блот-анализ осуществляли по методике, описанной в работе Mongiardini (Mongiardini et al., 2008). В качестве мембраны для переноса белков методами Вестерн-блот и дот-блот использовали PVDF-мембрану (“Bio-Rad”, США). Дот-блоттинг (метод вакуумной фильтрации) осуществляли по следующей схеме: PVDF-мембрану помещали в прибор Bio-Dot Microfiltration System (“Bio-Rad”, США), затем наносили по 500 мкл суспензии в лунки предварительно разрушенных бактериальных клеток после ультразвуковой дезинтеграции в ресуспендирующем буфере (50 мM Tris-HCl, 1 мM ЭДТА, 100 мM NaCl, 1 мM PMSF).
Обнаружение корового белка нитрогеназы – NifH проводили с использованием коммерческих поликлональных куриных антител (первичные антитела) (AS01021A, “Agrisera”, Швеция). После блокировки мембраны в 0.5% сухом обезжиренном молоке иммобилизированные белки обрабатывали первичными антителами, которые предварительно разводили в 2000 раз в ТBS-буфере c добавлением 0.5% сухого обезжиренного молока (10 мM трис-основание, 0.9% NaCl, 0.1% Tween-20, 0.5% сухое молоко). По окончании инкубации в течение 10–16 ч при 4°С мембраны трижды промывали в TBS-буфере от остатков первичных антител. Для обнаружения связавшихся первичных антител использовали моноклональные кроличьи антитела в разведении 1 : 2000, конъюгированные с пероксидазой хрена HPR (“Agrisera”, Швеция). Инкубацию проводили в течение 1.5–2 ч при комнатной температуре. После трех отмывок в TBS-буфере связывание вторичных антител детектировали с помощью субстрата DAB (“Cell Marque Corporation”, США).
Филогенетический анализ последовательностей генов nifA проводили в программе Megalign (Lasergene, DNASTAR) на основе множественного выравнивания ClustalW. Нуклеотидные последовательности гена nifA штаммов клубеньковых бактерий были взяты из GenBank.
Статистическую обработку полученных данных осуществляли с использованием программы Microsoft Office Excel 2010. Эксперименты проводили в трехкратной повторности. При оценке полученных результатов использовался критерий Краскела–Уоллиса. Представленные данные имеют соответствующие доверительные интервалы при уровне доверительной вероятности равной 0.95.
Обработку результатов дот-блот-анализа проводили с использованием компьютерной программы TatalLab v2.01.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
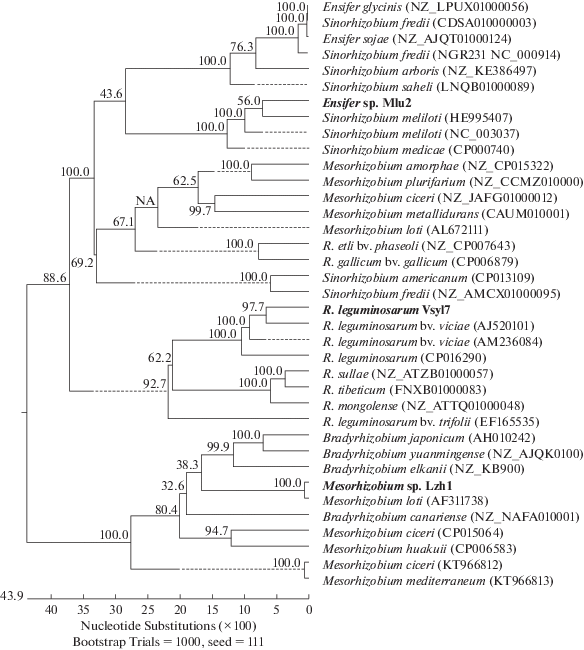
Для выявления степени функциональной специфичности гена nifA внутри группы клубеньковых бактерий были созданы генно-инженерные плазмидные конструкции с его различными филогенетическими вариантами под управлением бактериального промотора. В качестве основы была выбрана плазмида широкого круга хозяев pJN105, содержащая репликон pBBR1 (Newman, Fuqua, 1999). При сравнительном анализе последовательностей гена nifA клубеньковых бактерий, несмотря на высокую гетерогенность, тем не менее, наблюдалось определенное филогенетическое разделение по группам в зависимости от принадлежности к основным родам ризобий (рис. 1). В этой связи в качестве целевых генов при получении генно-инженерных конструкций были использованы три гена nifA, принадлежащие преимущественно к бактериям родов Rhizobium, Mesorhizobium, Ensifer, которые в плазмидe pJN105 были встроены под управление промотора ParaBAD, индуцируемого сахаром арабинозой. Известно, что бактерии рода Mesorhizobium содержат две копии гена регуляторного белка NifA: nifA1 и nifA2 (Sullivan et al., 2013). Нами для работы была клонирована последовательность гена, которая соответствует варианту гена nifA2.
Рис. 1.
Филогенетическое древо, построенное на основании сравнительного анализа последовательностей гена nifA клубеньковых бактерий.
Для того чтобы удостовериться, что полученные векторы функциональны в штаммах ризобий, нами параллельно была создана подобная же конструкция, где вместо целевого гена был вставлен репортерный ген зеленого флуоресцентного белка. При трансформации данной плазмидой ризобий образовывались зелено-окрашенные клоны микроорганизмов, что показывает функциональность полученных конструкций в клубеньковых бактериях.
Наиболее распространенными родами ризобий, вступающими в симбиоз с бобовыми растениями умеренного климата, являются бактерии рода Rhizobium, Mesorhizobium, Bradyrhizobium и Ensifer. Выбор штаммов реципиентов для получения рекомбинантных по созданным конструкциям микроорганизмов осуществлялся из числа представителей перечисленных родов ризобий по следующим критериям: во-первых, штаммы должны были быть эффективными, т.е. иметь хорошую клубенек-образующую и азотфиксирующую активности, что свидетельствовало бы о присутствии полного набора необходимых для азотфиксирующего симбиоза генов; во-вторых, неустойчивыми к действию антибиотика гентамицина для возможности селекции рекомбинантных бактерий после их трансформации. Штаммы, удовлетворяющие нас по условиям эффективности, были взяты из коллекции симбиотических микроорганизмов ИБГ УФИЦ РАН “Симбионт”. Далее, более 130 штаммов клубеньковых бактерий были проверены на устойчивость к действию антибиотика гентамицина (Gm), используемого в качестве маркера при скрининге рекомбинантных клонов. Таким образом, для данного исследования были отобраны штаммы VSyl1, VSyl3, VSyl7, VSyl8, LPis4 (род Rhizobium); MLu2 (род Ensifer); LZh1 и LZh3 (род Mesorhizobium), не проявляющие резистентность к гентаминицину и имеющие азотфиксирующую активность в симбиозе с растением-хозяином.
В дальнейшем на основе выбранных штаммов были получены их рекомбинантные варианты со всеми созданными векторными конструкциями, несущими гены nifA, принадлежащие родам Rhizobium, Ensifer, Mesorhizobium.
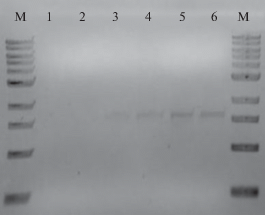
Поскольку белок NifA является основным активатором генов нитрогеназного комплекса, функциональную активность привнесенных копий его гена проверяли определением индукции наработки корового белка нитрогеназы NifH методом вестерн-блот анализа с использованием поликлональных куриных антител, полученных к данному белку (рис. 2).
Рис. 2.
Вестерн-блот анализ белков NifН, выделенных из штаммов клубеньковых бактерий дикого типа и их рекомбинантных вариантов. 1 – белок NifH, полученный из клубеньков гороха посевного; 2–4 – белок NifH, выделенный у штаммов дикого типа VSyl7, MLu2 и LZh1 соответственно; 5–7 – белок NifH, полученный из рекомбинантных штаммов VSyl7ParaBADVic, MLu2ParaBADSino и LZh1ParaBADMeso соответственно. Концентрация индуктора арабинозы 1 мM.

Было обнаружено, что при индукции привнесенного гена nifA арабинозой в концентрации 1 мM белок NifH нарабатывается во всех рекомбинантных штаммах, тогда как у диких вариантов в свободноживущем состоянии он отсутствует.
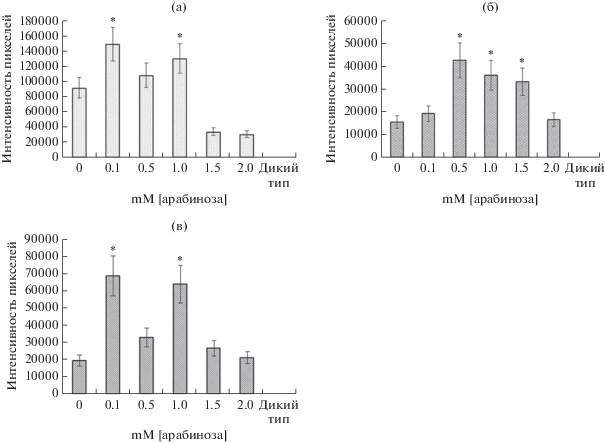
Анализ влияния различных концентраций индуктора бактериального промотора ParaBAD, под регуляцией которого находятся привнесенные копии генов nifA, на уровень наработки белка NifH показал неоднозначные результаты. Так, при исследовании концентраций индуктора арабинозы в диапазоне от 0.1 до 2 мM было показано, что наибольший уровень наработки целевого белка у разных рекомбинантных штаммов происходит при различных его концентрациях, хотя транскрипционная активность самого гена увеличивалась с повышением концентрации индуктора (рис. 3).
Рис. 3.
Анализ транскрипционной активности гена nifA методом ОТ-ПЦР у рекомбинантного штамма бактерий Rhizobium sp. VSyl7 c дополнительной ко-пией данного гена рода Rhizobium при воздействии различных концентраций индуктора арабинозы: 1 – без индуктора; 2 – 0.1 мM; 3 – 0.5 мM;4 – 1 мM; 5 – 1.5 мM; 6 – 2 мM.
Так, например, у рекомбинантного штамма R. leguminosarum VSyl7 c конструкцией pJN105Rh-izonifA и Mesorhizobium sp. с конструкцией pJN105MesonifA было обнаружено повышение индукции гена nifH уже при концентрации 0.1 мM, которая при 0.5 мM падала и опять повышалась при 1 мM. Выявить какую-то закономерность влияния определенных концентраций индуктора гена nifA на активацию генов нитрогеназного комплекса не получилось, поскольку индуктор на конечный белок влияет опосредовано через продукт гена nifA, который, в свою очередь может подвергаться различным воздействиям, например, действию ингибиторов. Поэтому каждый вариант штаммов имеет свою особенность при ответе на воздействие на них индуктором. Но, тем не менее, было показано, что у всех штаммов при воздействии арабинозой в концентрации 1 мM происходит наработка белка NifH на близких к максимальному уровню (рис. 4). Поэтому при выполнении следующего этапа работы была использована именно эта концентрация индуктора. Наибольший ответ происходил при концентрациях близких к 1 мM. Хотя есть и исключения.
Рис. 4.
Гистограммы анализа наработки белка NifH рекомбинантными бактериями и штаммами дикого типа дот-блот-методом, выращенными на питательной среде в присутствии/отсутствии индуктора – арабинозы в диапазоне концентраций 0.1–2 мM. (а) – Рекомбинантный штамм бактерий Rhizobium sp. VSyl7 c дополнительной активной копией nifA гена рода Rhizobium, штамм дикого типа – VSyl7. (б) – Рекомбинантный штамм бактерий Ensifer sp. MLu2 c дополнительной активной копией nifA гена рода Ensifer, штамм дикого типа – MLu2. (в) – Рекомбинантный штамм бактерий Mesorhizobium sp. LZh1 c дополнительной активной копией nifA гена рода Mesorhizobium, штамм дикого типа – LZh1. Средняя интенсивность пикселей по точкам представлена в графическом режиме. Знаком “*” помечены столбцы, где данные представляют статистическую значимость при p < 0.05.
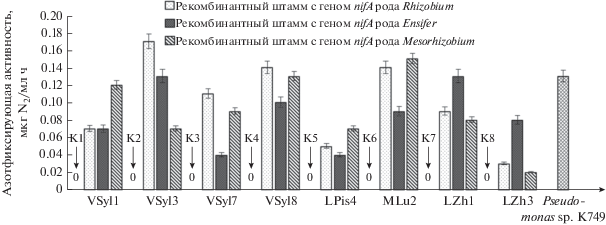
Далее, все полученные рекомбинантные штаммы были проанализированы на наличие азотфиксирующей активности методом редукции ацетилена. В результате этого было выявлено, что привнесение активной дополнительной копии nifA гена у ризобий приводит к появлению процесса фиксации молекулярного азота ex planta. Так же как и на предыдущем этапе, азотфиксирующая активность рекомбинантных штаммов в свободноживущем состоянии наблюдалась как при привнесении конструкций с геном nifA, характерным бактериям своего рода, так и аналогичными генами, клонированными из микроорганизмов других родов ризобий (рис. 5 ). Это говорит о том, что продукты генов nifA имеют универсальность среди исследованных родов клубеньковых бактерий. Тем не менее, нами были обнаружены и некоторые различия в активности при разных сочетаниях nifA и штаммов ризобий. При этом большее значение имел именно штамм, а не родовая принадлежность микроорганизмов. Так, наибольшая азотфиксирующая активность нами была обнаружена у штамма R. leguminosarum VSyl3 при привнесении гена nifA, характерного для бактерий рода Rhizobium. В то же время другой штамм R. leguminosarum – VSyl1, трансформированный той же конструкцией, показал значительно меньшие значения. При этом у него наибольшую азотфиксирующую активность вызывала трансформация конструкцией, содержащей ген nifA бактерий рода Mesorhizobium. Интересные данные были получены по штамму Ensifer sp. Штамм MLu2 показал наименьшую азотфиксирующую активность при привнесении в его клетки дополнительной активной копии гена nifA характерной для своего рода, тогда как рекомбинантные штаммы, трансформированные конструкциями, содержащими ген nifA бактерий рода Rhizobium и Mesorhizobium, отличались более высокой нитрогеназной активностью. Рекомбинантные штаммы рода Mesorhizobium также характеризовались тем, что привнесение в их клетки гена nifA бактерии своего же рода приводила к меньшей степени активации азотфиксации по сравнению с привнесением гена nifA других родов ризобий. Наибольшие показатели нитрогеназной активности у штаммов рода Mesorhizobium наблюдали при трансформации их конструкциями с геном nifA бактерий рода Ensifer.
Риc. 5. Анализ азотфиксирующей активности рекомбинантных штаммов клубеньковых бактерий. В качестве образца для сравнения взяты дикие варианты штаммов клубеньковых бактерий К1−К8.
Индукция азотфиксирующей активности исследованных родов клубеньковых бактерий при искусственной активации экспрессии гена регуляторного белка NifA, как было показано в работе, возможна. При этом наблюдается функциональная универсальность NifA внутри бактерий трех взятых в анализ родов ризобий. Уровни активации азотфиксации у штаммов одного вида разными филогенетическими вариантами NifA имеют различные значения, что говорит о том, что кроме эволюционной составляющей большое значение имеет генетическое сочетание набора генов, участвующих в процессе азотфиксации. Работы по искусственной комбинаторной эволюции nif-генов проводятся регулярно. Но до сих пор не известен механизм совместной работы всех продуктов данных генов, значение разных их сочетаний в эффективности азотфиксации (Smanski et al., 2014). И поскольку в природе встречаются штаммы бактерий даже одного вида с отличающейся эффективностью азотфиксации и, соответственно, разным набором и локализацией nif-генов, то значит процесс образования разных их комбинаций постоянный и имеет огромное значение в биологической минерализации атмосферного азота. Скорее всего, именно универсальность отдельных компонентов группы nif-генов делает возможным участие комбинаторных механизмов в эволюции генов азотфиксации, а также дает объяснение возможности образования химерных штаммов ризобий, когда в клетках клубеньковых бактерий обнаруживаются симбиотические гены другой филогенетической принадлежности. Таким образом, еще раз подтверждается ключевая роль горизонтального переноса генов между клубеньковыми бактериями в формировании большого разнообразия потенциально способных к азотфиксирующему симбиозу генетических вариантов ризобий и главенствующая роль бобового растения в отборе наиболее оптимальных для него штаммов клубеньковых бактерий.
Работа выполнена с использованием оборудования РЦКП “Агидель” и УНУ “КОДИНК”.
Список литературы
Баймиев Ан.Х., Гуменко Р.С., Владимирова А.А., Акимова Е.С., Вершинина З.Р., Баймиев Ал.Х. Искусственная активация экспрессии nif-генов у клубеньковых бактерий ex planta // Экологическая генетика. 2019. Т. 17. № 2. С. 35–42. https://doi.org/10.17816/ecogen17235-42
Baymiev An.K., Gumenko R.S., Vladimirova A.A., Akimova E.S., Vershinina Z.R., Baymiev Al.K. Artificial activation of nif gene expression in nodule bacteria ex planta // Ecological Genetics. 2019. V. 17. № 2. P. 35–42.
Баймиев Ан.Х., Птицын К.Г., Мулдашев А.А., Баймиев Ал.Х. Генетическая характеристика клубеньковых бактерий бобовых рода Lathyrus, произрастающих на территории Республики Башкортостан // Экологическая генетика. 2011. Т. 9. № 2. С. 3–8.
Умаров М.М. Ассоциативная азотфиксация. М: Изд-во МГУ, 1989. 136 с.
An Q., Dong Y., Wang W., Li Y., Li J. Constitutive expression of the nifA gene activates associative nitrogen fixation of Enterobacter gergoviae 57-7, an opportunistic endophytic diazotroph // J. Appl. Microbiol. 2007. V. 103. P. 613–620. https://doi.org/10.1111/j.1365-2672.2007.03289.x
Buchanan-Wollaston V., Cannon M.C., Beynon J.L., Cannon F.C. Role of the nifA gene product in the regulation of nif expression in Klebsiella pneumonia // Nature. 1981. V. 294. P. 776–778. https://doi.org/10.1038/294776a0
Bush M., Dixon R. The role of bacterial enhancer binding proteins as specialized activators of σ54-dependent transcription // Microbiol. Mol. Biol. Rev. 2012. V. 76. P. 497–529. https://doi.org/10.1128/MMBR.00006-12
Dai Z., Guo X., Yin H., Liang Y., Cong J., Liu X. Identification of nitrogen-fixing genes and gene clusters from metagenomic library of acid mine drainage // PLoS One. 2014. V. 9. P. e87976. https://doi.org/10.1371/journal.pone.0087976
Dixon R., Kahn D. Genetic regulation of biological nitrogen fixation // Nat. Rev. Microbiol. 2004. V. 2. P. 621–631. https://doi.org/10.1038/nrmicro954
Dixon R.A., Postgate J.R. Genetic transfer of nitrogen fixation from Klebsiella pneumoniae to Escherichia coli // Nature. 1972. V. 237. P. 102–103.
Fischer H.M. Environmental regulation of rhizobial symbiotic nitrogen fixation genes // Trends Microbiol. 1996. V. 4. P. 317–320. https://doi.org/10.1016/0966-842x(96)10049-4
Han Y., Lu N., Chen Q., Zhan Y., Liu W., Lu W., Yan Y. Interspecies transfer and regulation of Pseudomonas stutzeri A1501 nitrogen fixation island in Escherichia coli // J. Microbiol. Biotechnol. 2015. V. 25. P. 1339–1348. https://doi.org/10.4014/jmb.1502.02027
Kennedy C., Robson R.L. Activation of nif gene expression in Azotobacter by the nifA gene product of Klebsiella pneumonia // Nature. 1983. V. 301. P. 626–628. https://doi.org/10.1038/301626a0
Laranjo M., Alexandre A., Oliveira S. Legume growth-promoting rhizobia: an overview on the Mesorhizobium genus // Microbiol. Res. 2014. V. 169. P. 2–17. https://doi.org/10.1016/j.micres.2013.09.012
Li X.X., Liu Q., Liu X.M., Shi H.W., Chen S.F. Using synthetic biology to increase nitrogenase activity // Microbial Cell Factor. 2016. V. 15. № 43. P. 1–11. https://doi.org/10.1186/s12934-016-0442-6
Mongiardini E.J., Ausmees N., Pérez-Giménez J., Julia Althabegoiti M., Ignacio Quelas J., López-García S.L., Lodeiro A.R. The rhizobial adhesion protein RapA1 is involved in adsorption of rhizobia to plant roots but not in nodulation // FEMS Microbiol. Ecol. 2008. V. 65. P. 279–288. https://doi.org/10.1111/j.1574-6941.2008.00467.x
Newman J.R., FuquaC. Broad-host-range expression vectors that carry the L-arabinose-inducible Escherichia coli araBAD promoter and the araC regulator // Gene. 1999. V. 227. P. 197–203. https://doi.org/10.1016/s0378-1119(98)00601-5
Norel F., Elmerich C. Nucleotide sequence and functional analysis of the two nifH copies of Rhizobium ORS571 // Microbiology (SGM). 1987. V. 133. P. 1563–1576. https://doi.org/10.1099/00221287-133-6-1563
Sambrook J., Russell D.W. Molecular Cloning: A Laboratory Manual. 3rd ed. Cold Spring Harbor Laboratory Press, 2001. 2100 p.
Setten L., Soto G., Mozzicafreddo M., Fox A.R., Lisi C., Cuccioloni M., Ayub N.D. Engineering Pseudomonas protegens Pf-5 for nitrogen fixation and its application to improve plant growth under nitrogen-deficient conditions // PLoS One. 2013. V. 8. P. e63666. https://doi.org/10.1371/journal.pone.0063666
Smanski M.J., Bhatia S., Zhao D., Park Y., Woodruff L.B., Giannoukos G., Ciulla D., Busby M., Calderon J., Nicol R., Gordon D.B., Densmore D., Voigt C.A. Functional optimization of gene clusters by combinatorial design and assembly // Nature Biotechnol. 2014. V. 32. P. 1241–1249. https://doi.org/10.1038/nbt.3063
Sullivan J.T., Brown S.D., Ronson C.W. The NifA-RpoN regulon of Mesorhizobium loti strain R7A and its symbiotic activation by a novel LacI/GalR-family regulator // PLoS One. 2013. V. 8. P. e53762. https://doi.org/10.1371/journal.pone.0053762
Towbin H., Staehelin T., Gordon J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications // Proc. Natl. Acad. Sci. USA. 1979. V. 76. P. 4350–4354. https://doi.org/10.1073/pnas.76.9.4350
Wang L., Zhang L., Liu Z., Zhao D., Liu X., Zhang B., Dixon R. A minimal nitrogen fixation gene cluster from Paenibacillus sp. WLY78 enables expression of active nitrogenase in Escherichia coli // PLoS Genet. 2013. V. 9. P. e1003865. https://doi.org/10.1371/journal.pgen.1003865
Wongdee J., Boonkerd N., Teaumroong N., Tittabutr P., Giraud E. Regulation of nitrogen fixation in Bradyrhizobium sp. strain DOA9 involves two distinct NifA regulatory proteins that are functionally redundant during symbiosis but not during free-living growth // Front. Microbiol. 2018. V. 9. P. 1644.https://doi.org/10.3389/fmicb.2018.01644
Xu H., Gu B., Nixon B.T., Hoover T.R. Purification and characterization of the AAA+ domain of Sinorhizobium meliloti DctD, a σ54-dependent transcriptional activator // J. Bacteriol. 2004. V. 186. P. 3499–3507. https://doi.org/10.1128/JB.186.11.3499-3507.2004
Yang J., Xie X., Wang X., Dixon R., Wang Y.P. Reconstruction and minimal gene requirements for the alternative iron-only nitrogenase in Escherichia coli // Proc. Natl. Acad. Sci. USA. 2014. V. 111. P. 3718–3725. https://doi.org/10.1073/pnas.1411185111
Дополнительные материалы отсутствуют.


