Микробиология, 2021, T. 90, № 5, стр. 574-588
Филогенетическое разнообразие и потенциальная активность бактерий и грибов в подземных горизонтах месторождения урана
Т. Л. Бабич a, Е. М. Семенова a, Д. Ш. Соколова a, Т. П. Турова a, С. Х. Биджиева a, Н. Г. Лойко a, Г. И. Авдонин b, Н. И. Луценко c, Т. Н. Назина a, *
a Институт микробиологии им. С.Н. Виноградского, ФИЦ Биотехнологии РАН
119071 Москва, Россия
b Всероссийский научно-исследовательский институт минерального сырья
им. Н.М. Федоровского
119017 Москва, Россия
c АО “Далур”
Курганская обл., Россия
* E-mail: nazina@inmi.ru
Поступила в редакцию 05.03.2021
После доработки 08.03.2021
Принята к публикации 12.03.2021
Аннотация
В настоящей работе определен состав микроорганизмов в образцах пород, пластовой воды и накопительных культурах из подземных горизонтов месторождения урана (Россия). Исследованные горизонты располагаются в зоне затрудненного водообмена на глубине 513–544 м, характеризуются умеренной соленостью (до 15 г/л) пластовой воды и наличием обугленного растительного органического вещества. Методом высокопроизводительного секвенирования V4 региона гена 16S рРНК было определено биоразнообразие автохтонных подземных прокариот. В образцах пород обнаружено преобладание бактерий филумов Firmicutes (24.2%), Fusobacteriota (23.0%), Proteobacteria (18.7%), Actinobacteriota (15.5%) и Bacteroidota (9.0%). В пластовой воде доминировали бактерии филума Proteobacteria (90.7%), включающие представителей родов Methylophaga, Porphyrobacter, Roseovarius, Pseudomonas и Methylococcus. Секвенирование внутреннего транскрибируемого спейсера (ITS) позволило выявить в образцах пород низшие грибы родов Mucor и Thamnidium (филума Mucoromycota) и рода Penicillium (филума Ascomycota), известные устойчивостью к тяжелым металлам и способностью использования сложных органических субстратов. Функциональные характеристики бактериальных сообществ породы и пластовой воды, предсказанные с помощью программы iV-icodak и базы данных KEGG, показали бóльшую потенциальную способность бактерий пластовой воды по сравнению с микробиотой породы осуществлять пути метаболизма углеводов, азота и серы, деградации ксенобиотиков, бензоата, полициклических ароматических углеводородов и хлорсодержащих органических соединений. Получены накопительные культуры железоредуцирующих и сульфатредуцирующих бактерий и чистые культуры денитрифицирующих бактерий родов Bacillus, Paenibacillus и Acinetobacter. Приведенные результаты свидетельствуют о наличии малочисленного, но жизнеспособного микробного сообщества, способного при активизации водообмена участвовать в процессах трансформации углерода, азота, серы и металлов в подземном горизонте.
Способность микроорганизмов участвовать в процессах преобразования металлов и радионуклидов детально исследована на чистых культурах и микробных сообществах из поверхностных и подземных местообитаний, загрязненных техногенными радионуклидами (Newsome et al., 2014). Взаимодействие микроорганизмов с радионуклидами происходит по разным механизмам и включает биосорбцию (адсорбция на клеточной поверхности), биоаккумуляцию (проникновение и накопление внутри клетки), биотрансформацию (изменение степени окисления радионуклида за счет биохимических процессов) и др. Микробные популяции могут изменять геохимические параметры окружающих пород, воздействовать на состав и миграцию металлов и радионуклидов, осуществлять их диссимиляционное восстановление (U6+, Tc7+, V5+ и др.), сорбцию и концентрирование в биопленках, продуцировать комплексообразующие агенты, изменять величины рН и Еh.
Наиболее исследованы микроорганизмы подземных водоносных горизонтов, используемых для захоронения жидких радиоактивных отходов (РАО) или загрязненных радионуклидами в научных целях (North et al., 2004; Akob et al., 2007; Green et al., 2012). В глубинных хранилищах жидких РАО субстратами для роста микроорганизмов являются поступающие с отходами органическое вещество, нитраты и сульфаты, окисленные формы радионуклидов и других металлов (Fe3+), бикарбонат, присутствующий в подземных водах, а также молекулярный водород, который может образовываться в радиоактивных растворах за счет радиолиза воды. Помимо превращений радионуклидов в глубинных хранилищах наблюдается биогенное газообразование (N2, CH4, H2S, CO2), обусловленное жизнедеятельностью денитрифицирующих, бродильных, сульфатредуцирующих и метаногенных прокариот (Nazina et al., 2010).
Многочисленными исследованиями подтверждено участие денитрифицирующих бактерий в удалении нитратов из подземных горизонтов, загрязненных радионуклидами и нитрат-ионами (North et al., 2004; Green et al., 2012; Safonov et al., 2018). Продукты восстановления нитрата способствуют окислению восстановленных металлов и радионуклидов, усиливая их растворимость и миграцию. Восстановление радионуклидов и сульфатредукция в загрязненных ураном и нитратами осадках протекают только после исчерпания нитрата в подземных водах (Senko et al., 2005; Wu et al., 2010).
Микроорганизмы природных подземных залежей радионуклидов исследованы в меньшей степени, чем микробиота техногенных местообитаний, загрязненных радионуклидами (Mondani et al., 2011; Islam et al., 2011; Bhakat et al., 2019). В породах месторождения урана на северо-востоке Индии (Domiasiat, North East India) обнаружены бактерии филумов Firmicutes (51%), Actinobacteria (11%) и Bacteroidetes (10%) и классов Gammaproteobacteria (26%) и Betaproteobacteria (2%) (Kumar et al., 2013). Бактерии, выделенные из этого месторождения, принадлежали к родам Bacillus, Paenibacillus, Arthrobacter, Microbacterium, Pseudomonas, Acinetobacter и др. Показана адсорбция урана этими штаммами и высокая фосфатазная активность, также способствующая осаждению урана на микробной биомассе. Присутствие бактерий этих таксонов обнаружено также в ряде загрязненных радионуклидами и тяжелыми металлами почв (Radeva, Selenska-Pobell, 2005; Martinez et al., 2006).
Настоящая работа направлена на изучение разнообразия микроорганизмов подземных горизонтов месторождения урана Добровольное (Курганская обл., Россия), которое находится на стадии исследований и пока не эксплуатируется. Месторождение локализовано в аллювиальных отложениях средне-верхнеюрского возраста, залегающих на глубинах 480–600 м от земной поверхности (Долбилин и соавт., 2009). Породы, обогащенные углефицированным растительным детритом, содержат дисульфиды железа. На месторождении Далматово, аналогичном месторождению Добровольное, ведется добыча урана методом скважинного подземного выщелачивания (СПВ) с использованием серной кислоты и нитрита натрия в качестве окислителя. Контроль баланса подаваемых в пласт нитрит-ионов и содержания нитритов в продуктивных урансодержащих растворах показывает уменьшение общего содержания азотистых соединений, которое невозможно объяснить дисперсией состава подземных вод или депонированием в глины. Было высказано предположение о восстановлении нитрита до молекулярного азота денитрифицирующими бактериями подземных горизонтов и его удалении из системы.
В связи с вышесказанным, было необходимо определить состав микробного сообщества и наличие жизнеспособных денитрифицирующих и других популяций в подземных горизонтах месторождения Добровольное. Изучение фоновых значений разнообразия микробного сообщества необходимо для прогноза экологических последствий процесса выщелачивания урана и его воздействия на биогеохимические процессы в подземных горизонтах.
Целью настоящей работы было определение филогенетического разнообразия бактерий и грибов в образцах пород, пластовой воды и накопительных культурах из подземных горизонтов месторождения урана Добровольное (Россия), потенциальной функциональной активности бактерий, а также выделение и идентификация денитрифицирующих бактерий, способных восстанавливать нитрат/нитрит в подземных водах.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объекты исследования. Объектом микробиологических исследований были образцы пород и пластовая вода, отобранные в 2020 г. из керна разведочных скважин на месторождении урана Добровольное (Россия). Образцы пород отбирали на разной глубине из керна геологоразведочных скважин Р-123 (513.1 и 523.7 м), Р-78 (544.5 м), Р-76 (541.8 и 519.7 м) и Р-224 (521.4 м), и проба пластовой воды была отобрана из скважины Г-15 (табл. S1 , доп. материалы). Скважина 224 пробурена на месте проведения опыта по добыче урана. В 1989–1990 гг. на месторождении был проведен 2-х скважинный опыт по добыче урана методом скважинного подземного выщелачивания с использованием серной кислоты (Долбилин и соавт., 2009).
Получение накопительных и чистых культур. Для учета и выделения микроорганизмов из образцов пород получали суспензию, для чего усредненную навеску породы массой 10 г вносили в 90 мл стерильной водопроводной воды и тщательно перемешивали на качалке в течение 10 мин. Полученную суспензию выдерживали 5 мин для осаждения твердой фазы и использовали для засева жидких питательных сред методом предельных десятикратных разведений. Результаты оценивали методом наиболее вероятного числа по таблице Мак-Креди. Численность аэробных органотрофных микроорганизмов оценивали микроскопированием посевов серии разведений полученной суспензии в среде следующего состава (г/л водопроводной воды): глюкоза – 1.0; бактотриптон – 5.0; дрожжевой экстракт – 2.5; NaCl – 5; pH 7.0–7.2. Анаэробную технику Хангейта использовали для приготовления сред для анаэробных бактерий. В качестве газовой фазы использовали очищенный от кислорода аргон. Численность бродильных микроорганизмов оценивали микроскопированием посевов и обнаружением образования молекулярного водорода в газовой фазе в посевах серии разведений суспензии керна в среде (г/л воды): пептон – 4.0; глюкоза – 10.0; Na2SO4 – 2.0; MgSO4 – 1.0; NaCl – 5; соль Мора (FeSO4·(NH4)2SO4 · 6H2O) – 0.5; Na2S · 9H2O – 0.1; рН 7.2 (Postgate, 1984). Численность сульфатвосстанавливающих бактерий определяли по образованию сероводорода в посевах суспензии в среде (г/л воды): MgCl2 · 6H2O – 3.0; CaCl2 · 2H2O – 0.15; Na2SO4 – 4.0; NH4Cl – 0.25; KH2PO4 – 0.2; KCl – 0.5; NaCl – 5; лактат натрия – 4.0; дрожжевой экстракт – 0.5; NaHCO3 вносили, доводя рН среды до 7.0; Na2S · 9H2O – 0.2 (Widdel, Bak, 1992). Численность денитрифицирующих бактерий оценивали по появлению молекулярного азота в посевах серии разведений суспензии в среде следующего состава (г/л дистиллированной воды): KCl – 0.1; MgSO4 · 7H2O – 0.2; CaCl2 · 2H2O – 0.2; KH2PO4 – 0.75; K2HPO – 1.5; NH4Cl – 0.9; NaCl – 5; NaNO3 – 0.85; ацетат натрия – 2.0. Посевы инкубировали в стационарных условиях при температуре 20–23°C в течение 20 сут.
Чистые культуры аэробных органотрофных и денитрифицирующих бактерий были выделены методом последовательных пересевов с жидких сред на соответствующие плотные среды, содержащие 15 г агар-агара на 1 л. Процедуру выделения из единичных колоний повторяли трижды. Чистоту культур контролировали микроскопическим методом и посредством анализа гена 16S рРНК культуры из жидкой среды. В качестве субстратов для денитрифицирующих бактерий помимо ацетата использовали также пептон (2 г/л), глюкозу (2 г/л) и рибозу (2 мл/л). Среды готовили аэробно и анаэробно. Аэробную среду разливали в пенициллиновые пробирки с воздушной фазой, анаэробную среду разливали в пробирки Хангейта в атмосфере Ar.
Микроскопические методы. Морфологию клеток бактерий изучали на живых препаратах, используя эпифлуоресцентный микроскоп Axio Imager.D1 (“Carl Zeiss”, Германия) с цифровой камерой Axio Cam HRc и компьютерным программным обеспечением Axio Vision. Для получения фотографий в сканирующем микроскопе накопительные культуры наращивали в соответствующих жидких средах с тефлоновыми кубиками для получения биопленочного роста, среду декантировали и биомассу на тефлоновых кубиках дважды промывали фосфатным буфером (рН 7.0), поэтапно обезвоживали в растворах этилового спирта возрастающей концентрации (от 30 до 100%). Затем дважды отмывали образцы в 100% ацетоне и высушивали в критической точке с помощью специальной камеры. Высушенные препараты монтировали на специальные столики, а затем напыляли на них тонкий слой металла для создания токопроводящего покрытия. Полученные образцы просматривали в сканирующем электронном микроскопе JSM-6380LA (JEOL, Япония) при ускоряющем напряжении 20 кВ. Работа была выполнена с использованием оборудования, приобретенного в рамках Программы развития Московского государственного университета до 2020 г.
Аналитические методы. Молекулярный водород, азот и метан в газовой фазе определяли газохроматографическим методом; сероводород определяли колориметрически по методу Пахмайра с N,N-диметил-р-фенилендиамином, как описано ранее (Nazina et al., 2010). Образование нитритов в среде определяли по цветной реакции с реактивом Грисса. Прирост биомассы в жидкой среде оценивали по величине оптической плотности при длине волны 600 или 540 нм (ОД600 или ОД540) на приборе Ultrospec 2100 pro (“Amersham Biosciences”). В качестве контроля использовали незараженную и зараженную среду, инкубируемую при 6°C.
Молекулярно-биологические методы идентификации чистых культур и определения состава микробного сообщества пород и пластовой воды. Чистые культуры бактерий были идентифицированы методом анализа гена 16S рРНК с праймерами 8‑27f/1492r (Lane, 1991). Филогенетический анализ последовательностей генов 16S рРНК выделенных штаммов выполняли с использованием программы BLAST сервера NCBI (www.ncbi.nlm.nih.gov/blast/). Филогенетическое разнообразие прокариот (бактерий и архей) и эукариот (грибов) в исследованных пробах пород и пластовой воды определяли посредством секвенирования ампликонов гена 16S рРНК прокариотических организмов и ITS региона грибов и последующей биоинформатической обработки полученных данных. Ампликоны гена 16S рРНК и ITS региона получали путем полимеразной цепной реакции с использованием препаратов тотальной ДНК микробных сообществ анализируемых образцов.
Выделение ДНК, амплификация и секвенирование генов 16S рРНК прокариот и ITS региона грибов. ДНК из образцов пород и пластовой воды выделяли с использованием коммерческих наборов для выделения ДНК FastDNA Spin Kit (“MP Bio”, США) в соответствии с инструкцией производителя. Полученные препараты ДНК использовали для амплификации V4 участка гена 16S рРНК прокариот или ITS региона грибов путем полимеразной цепной реакции (ПЦР). Для получения библиотек генов 16S рРНК прокариот был амплифицирован V4 гипервариабельный регион этого гена и на основе двойного баркодирования были приготовлены библиотеки, как описано ранее (Fadrosh et al., 2014). Смысловые участки праймеров были взяты в соответствии с парой праймеров Pro341F–Pro805R (Takahashi et al., 2014). Библиотеки ITS региона получали по аналогичной схеме с использованием праймерных систем, описанных в статье (Bokulich, Mills, 2013). Для получения ампликонов ITS региона использовали прямой праймер ITS86F 5'-GTGAATCATCGAATCTTTGAA-3' и обратный праймер ITS4R 5'-TCCTCCGCTTATTGATATGC-3'. Получаемые при этом ампликоны за счет наличия на 5'-концах праймеров технических последовательностей были после очистки секвенированы на платформе Illumina MiSeq (США). Технические последовательности включали в себя адаптеры Illumina, сайты посадки секвенирующих праймеров, баркоды и спейсеры гетерогенности. Очистку ампликонов осуществляли путем препаративного агарозного гель-электрофореза с последующим использованием стандартных наборов реактивов для очистки ампликонов из агарозного геля и реакционных смесей со стандартными спин-колонками в соответствии с рекомендациями производителя наборов. Высокопроизводительное секвенирование полученных на предыдущей стадии ампликонов производили с использованием набора реагентов MiSeq Reagent Micro Kit v2 (300-cycles) MS-103-1002 (“Illumina”, США) на секвенаторе MiSeq (“Illumina”, США) в соответствии с инструкцией производителя. Обозначения полученных библиотек гена 16S рРНК приведены в табл. S1 (доп. материалы).
Биоинформатический анализ. После проведения секвенирования получали fastq файлы, содержащие информацию о последовательностях фрагментов гена 16S рРНК или ITS региона, а также информацию о достоверности прочтения каждого отдельного нуклеотида. Полученные фрагменты генов 16S рРНК были обрезаны с использованием программ trimmomatic-0.36 по алгоритму SLIDINGWINDOW:4:15 (Bolger et al., 2014). Прямые и обратные прочтения объединяли программой SeqPrep (https://github.com/jstjohn/SeqPrep). Для обнаружения химер и демультиплексирования использовали фильтры QIIME (Caporaso et al., 2010). Полученные последовательности распределяли по операционным таксономическим единицам (ОТЕ) с уровнем сходства более 97%. Полученные ОТЕ идентифицировали с использованием баз данных QIIME, онлайн-ресурса SILVA (Quast et al., 2014; https://www.arb-silva.de/ngs/) и Knomics Biota. Написание таксонов прокариот в настоящей публикации соответствует таковому, приведенному в онлайн-ресурсе SILVA. Относительное количество прочтений, входящих в ту или иную ОТЕ, соответствует относительной представленности данного таксона микроорганизмов в исследуемом микробном сообществе. Статистические подсчеты осуществляли с помощью Microsoft Excel. Онлайн-ресурс ClustVis (Metsalu, Vilo, 2015; https:// biit.cs.ut.ee/clustvis/) был использован для создания тепловых карт (heatmaps) членов сообщества на родовом уровне. Индексы разнообразия были рассчитаны с использованием программы EstimateS (http://purl.oclc.org/estimates). OTE, идентифицированные с использованием онлайн-ресурса SILVA, использовали для предсказания функциональных характеристик бактериальных сообществ с использованием пакета программ iVikodak (Nagpal et al., 2019). Модуль Global Mapper этой программы на основании базы данных KEGG использовали для получения функциональных профилей, а Local Mapper – для прогнозирования индивидуальных профилей ферментов метаболизма азота, серы, бензоата и метана, представленных у бактерий исследуемых микробных сообществ. Тепловые карты функциональных профилей и ферментов, предсказанных для сообществ, были построены с использованием интернет-ресурса ClustVis (http:// biit.cs.ut.ee/clustvis/, ноябрь 2020 г.).
Последовательности гена 16S рРНК чистых культур депонированы в базе данных NCBI под номерами MW541935, MW542185, MW542204, MW542594, MW543416, MW543704, MW543921, MW543938, MW544018, MW544028, MW544052, MW542313, MW542572, MW542576. Библиотеки фрагментов гена 16S рРНК породы, пластовой воды и накопительных культур депонированы в NCBI, BioProject PRJNA706554.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Физико-химические условия и численность культивируемых микроорганизмов в подземных горизонтах. Образцы пород и пластовой воды были отобраны с глубины 513–544 м месторождения Добровольное. Урановое оруденение относится к инфильтрационно-эпигенетическому, генетически связанному с развитием зон окисления. Оруденение на месторождении связано с сероцветными породами, насыщенными обугленным растительным органическим веществом и располагается на границе выклинивания зон обеленных пород, представляющих зону пластового окисления, восстановленную последующими процессами. Пирит в породах содержится в концентрации от 1 до 16%, причем его наибольшее количество приурочено к обогащенным ураном разностям. Отмечаются урансодержащие поликомпонентные гели в ассоциации с гипсом и пиритом. По данным Долбилина и соавт. (2009), химический состав вод верхнеюрского водоносного комплекса в пределах месторождения соответствует зоне затрудненного водообмена. Воды преимущественно соленые, с минерализацией 11.1–13.0 г/л. В составе компонентов преобладают гидрокарбонаты, сульфаты и хлориды натрия. Воды слабощелочные – pH колеблется от 7.2 до 7.5. Содержание сероводорода до 1.5 мг/л. Значения окислительно-восстановительного потенциала от +60 до +120 мВ, что свидетельствует о слабовосстановительной обстановке. Состав пластовой воды из скважины 224 приведен в табл. S2 (доп. материалы). Содержание урана в воде достигает 7.8 × 10–6 г/л.
Поскольку для выщелачивания металлов и радионуклидов обычно используется серная кислота и иногда азотная/азотистая кислоты, то представлялось необходимым определить наличие сульфатредуцирующих, железо-(металл)-редуцирующих и денитрифицирующих бактерий в пласте. Микробиологические исследования образцов пород из рудоносных горизонтов были направлены на обнаружение микроорганизмов, способных участвовать в преобразованиях поливалентных элементов и восстановлении нитратов и сульфата. В результате посевов на селективные питательные среды во всех образцах пород были обнаружены аэробные органотрофные бактерии, однако их численность была низка и не превышала 103 клеток в пересчете на 1 г породы (рис. S1 , дополнительные материалы). При микроскопировании посевов на средах для аэробных органотрофных и денитрифицирующих бактерий часто встречались биопленки и нити микроскопических грибов (рис. 1а, 1б). Бродильные бактерии, образующие молекулярный водород, были обнаружены лишь в посеве пород из скважины Р-123 (523.7 м). Микроскопическим методом эти бактерии обнаружены в посевах из большинства образцов пород, где их численность не превышала сотен клеток/г; в образце из скважины 224, используемой ранее в эксперименте по добыче урана методом скважинного подземного выщелачивания, численность бродильных бактерий достигала 106 кл./мл. Денитрифицирующие/нитратредуцирующие бактерии, восстанавливающие нитрат до стадии нитрита, были обнаружены в посевах всех образцов пород, максимальная численность составляла 103–104 кл./г породы из скв. Р-123 (513.1 и 523.7 м). Численность денитрифицирующих бактерий, восстанавливающих нитрат до молекулярного азота, была в среднем невелика – 102 кл./г породы, составляя 103 кл./г породы из скв. Р-78 (544.5 м). Бактерии, восстанавливающие Fe(III) до Fe(II) в среде с ацетатом, обнаружены в посевах породы из скв. Р-123 (513.1 и 523.7 м) и Р-76 (519.7 м) (рис. 1в). Популяция сульфатредуцирующих бактерий была малочисленной (рис. 1г), вероятно, из-за слабовосстановленных условий местообитания. Жизнеспособные сульфатредуцирующие бактерии не были обнаружены в посевах образцов из скв. Р-123 (523.7 м) и Р-224 (521.4 м) и достигали численности 104 кл./г породы в образце из скв. Р-76 (519.7 м). В пластовой воде из скв. Г-15 культивируемые сульфатредуцирующие бактерии и метаногены не были обнаружены. Первичные накопительные культуры, полученные при посеве пород, были использованы для определения состава микробного сообщества.
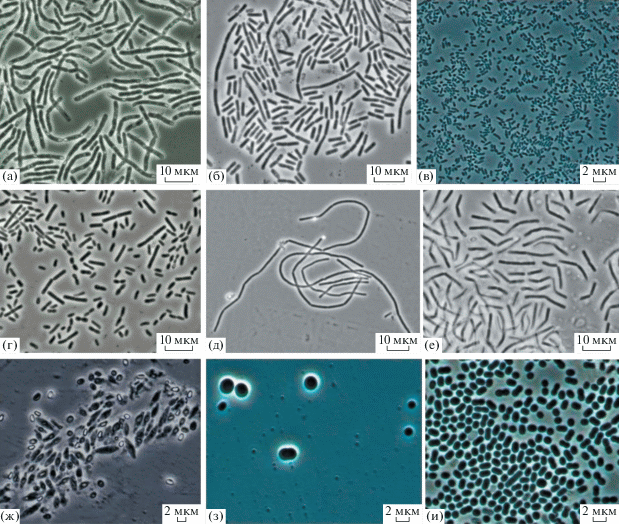
Рис. 1.
Морфология клеток и биопленки в накопительных культурах денитрифицирующих DNB-R123a (а, б), железоредуцирующих FeRB-R123a (в) и сульфатредуцирующих SRB-R76b (г) бактерий после 14 сут инкубации на соответствующих средах.

Филогенетическое разнообразие бактерий и архей в породе, пластовой воде и накопительных культурах. Для определения разнообразия микробного сообщества подземных горизонтов было проведено выделение ДНК из пробы пластовой воды скважины Г-15 и из шести образцов пород. Однако ДНК была выделена только из пробы пластовой воды Г-15 и из образца Р-224. Из пяти образцов пород ДНК выделить не удалось, вероятно, из-за низкой численности микроорганизмов в породах. Для получения более полного представления о составе подземного сообщества была выделена ДНК из 14 первичных накопительных культур, полученных при посеве пород на питательные среды. ДНК была использована для секвенирования V4 региона гена 16S рРНК. Всего было получено 267 131 фрагментов гена (ридов). Полученные последовательности принадлежали в основном прокариотам домена Bacteria, гены архей обнаружены при анализе пластовой воды (<0.1% от суммы последовательностей в библиотеке) и накопительных культур (≤0.2%). Покрытие разнообразия филотипов Bacteria, рассчитанное с помощью Good’s индекса, варьировало от 91 до 99% (табл. S3 , доп. материалы). Количественное распределение фрагментов гена 16S рРНК прокариот домена Bacteria в библиотеках на уровне высших таксонов – филумов или классов у Proteobacteria, приведено на рис. 2.
Рис. 2.
Относительная доля последовательностей фрагментов гена 16S рРНК Bacteria представленных на уровне филумов/классов в библиотеках из образцов породы из скв. P-224 (=R-224), пластовой воды из скв. Г-15 (=G-15) и из накопительных культур. Обозначения библиотек на рис. 2 и 3: AOB – аэробные органотрофные бактерии, DNB – денитрифицирующие бактерии, SRB – сульфатредуцирующие бактерии, FB – бродильные бактерии; номера соответствуют номерам скважин, из которых получали образцы породы или пластовую воду. Перечислены таксоны, составляющие >1% в каждой библиотеке.
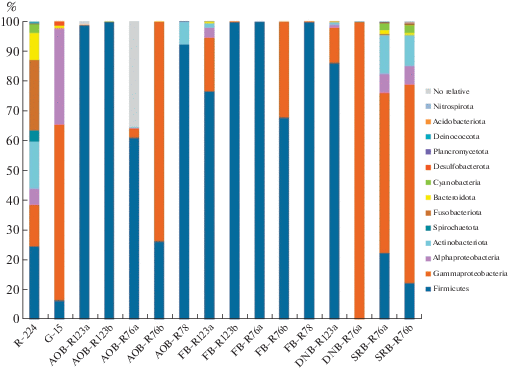
В составе микробного сообщества образца породы Р-224 присутствовали Firmicutes (24.2%), Fusobacteriota (23.0%), Proteobacteria (18.7%), Actinobacteriota (15.5%) и Bacteroidota (9.0%). В пластовой воде доминировали бактерии филума Proteobacteria (90.7%); в меньшей степени были представлены Firmicutes (6.4%) и Desulfobacterota (1.4%). Последовательности архей, выявленные в библиотеках из накопительных культур SRB-R76a и SRB-R76b, принадлежали некультивируемым Bathyarchaeia филума Crenarchaeota, археям рода Cuniculiplasma и некультивируемым A-plasma сем. Ferroplasmaceae (филум Thermoplasmatota). В пластовой воде G-15 и в накопительных культурах денитрифицирующих бактерий присутствовали археи филумов Thermoplasmatota (Marine Group II), Nanoarchaeota (Woesearchaeales, некультивируемые SCGC AAA011-D5), а также Crenarchaeota (род Candidatus Nitrosopumilus и некультивируемые Nitrososphaeraceae), способные участвовать в цикле азота.
Перечень 25 родов, наиболее представленных в исследованных микробных сообществах, приведен на рис. 3. В образце породы отмечено наибольшее разнообразие гетеротрофных бактерий родов Leptotrichia (13.9%), Fusobacterium (9.1%), Streptococcus (7.8%) и Actinomyces (7.6%). В пластовой воде преобладали протеобактерии родов Methylophaga (41.1%), Porphyrobacter (19.9%), Roseovarius (7.9%), Pseudomonas (6.9%) и Methylococcus (6.0%). В культурах аэробных органотрофных бактерий доминировали спорообразующие бактерии родов Bacillus и Paenibacillus и не образующие спор бактерии рода Anaerosinus, а также протеобактерии родов Acinetobacter и Arthrobacter. Бактерии родов Bacillus и/или Paenibacillus доминировали также в культурах бродильных бактерий и в культуре денитрифицирующих бактерий (DNB-R123a). Второе денитрифицирующее сообщество (DNB-R76a) почти полностью состояло из бактерий рода Acinetobacter (99.3%). Железо-(металл)-редуцирующие бактерии рода Shewanella были обнаружены среди минорных компонентов накопительной культуры бродильных бактерий FB-P123a. В среде для сульфатвосстанавливающих бактерий обнаружены Gammaproteobacteria родов Pseudomonas, Acinetobacter и Halomonas, являющиеся обычными компонентами подземных микробных сообществ, связанных с залежами углеводородов; минорные компоненты сообщества SRB-R76b (менее 0.04% последовательностей в библиотеке) включали собственно сульфидогенные Deltaproteobacteria родов Desulfotalea, Desulfocapsa, Desulfobulbus, Desulfomicrobium и Desulfovibrio.
Рис. 3.
Тепловая карта (heatmap) распределения 25 доминантных родов в библиотеках фрагментов гена 16S рРНК бактериальных сообществ из образцов породы из скв. P-224 (R-224), пластовой воды из скв. Г-15 (G-15) и накопительных культур, полученных из пород месторождения Добровольное. Двойная иерархическая дендрограмма показывает распределение бактерий в этих библиотеках. Переход цвета от синего до красного обозначает относительные величины содержания отдельных родов (от менее представленного к более представленному роду в библиотеке). Цифры на диаграмме обозначают % от общего количества последовательностей в библиотеке из каждой пробы.
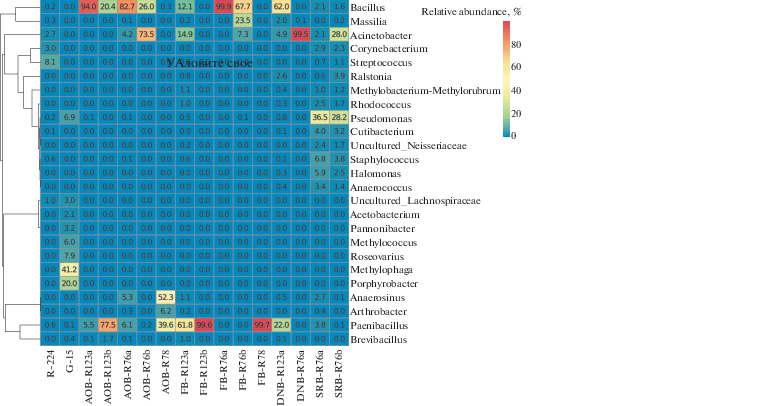
Gammaproteobacteria рода Methylophaga (41.1%) были наиболее представленной группой протеобактерий в пластовой воде. Спектр субстратов, используемых метилофагами в качестве единственных источников углерода и энергии, ограничен С1-соединениями (метанол, метиламин, диметилсульфид), за исключением ряда видов, которые используют также фруктозу (Janvier, Grimont, 1995). Однако недавно была доказана способность бактерий рода Methylophaga деградировать высокомолекулярные полициклические ароматические углеводороды (ПАУ) (Vila et al., 2010). C использованием [13C]n-гексадекана с равномерно мечеными атомами углерода, впервые показано, что ряд представителей этого рода, обладает способностью использовать n-гексадекан (Mishamandani et al., 2014; Gutierrez, Aitken, 2014). Эти сведения расширяют роль метилотрофов рода Methylophaga в превращении органического вещества нефтяного генезиса. Бактерии рода Methylophaga были неоднократно обнаружены при исследовании микробных сообществ зоны нефтяного загрязнения морской воды в Мексиканском заливе (Kessler et al., 2011; Yang et al., 2014). Вероятно участие этих бактерий в деградации захороненного органического вещества в исследованных глубинных горизонтах. Сульфатредуцирующая популяция пластовой воды была представлена бактериями родов Desulfobacterium (1.2%), Desulfomicrobium, Desulfocapsa и другими.
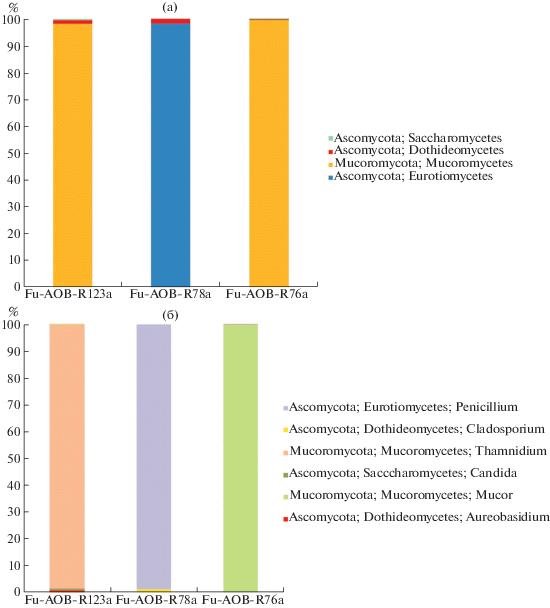
Филогенетическое разнообразие низших грибов. Секвенирование межгенного транскрибируемого спейсера (ITS) грибов, выросших в первичных культурах аэробных бактерий, позволило обнаружить низшие плесневые грибы в подземных пробах. В посевах образцов породы из скважин Р-76 (519.7 м), P-123 (513.1 м) и Р-78 (544.5 м) обнаружены, соответственно, низшие грибы родов Mucor (99.4%) и Thamnidium (97.6%) филума Mucoromycota и рода Penicillium (97.5%) филума Ascomycota (рис. 4а, 4б). Присутствие этих грибов в подземном местообитании, содержащем радионуклиды, представляется неслучайным. Известно, что грибы рода Mucor растут в анаэробных условиях, физиологически устойчивы к повышенному содержанию разных металлов (Al, Cd, Co, Cr, Cu, Hg, Ni, Pb, U и Zn) и накапливают их в виде наносфер в спорангиоспорах (Hoque, Fritscher, 2019). Эти свойства позволили рекомендовать грибы рода Mucor к применению в биотехнологиях биоремедиации местообитаний от металлов. Неоднократно отмечалась способность грибов рода Mucor использовать нефтяные углеводороды – алифатические и ароматические (BTEX = бензол, толуол, этилбензол и ксилол) соединения и полициклические ароматические углеводороды (PAH). Аскомицеты рода Penicillium были обнаружены при метагеномном анализе загрязненных ураном почв (Savannah River Site, США) (Jaswal et al., 2019). Выделенные из почв штаммы рода Penicillium демонстрировали устойчивость к урану в концентрации 10 мM и представляют интерес для биотехнологий биоремедиации экосистем от урана. Обнаруженные нами грибы родов Mucor, Thamnidium и Penicillium, вероятно, приспособлены к специфическим условиям подземного местообитания, характеризующегося повышенной концентрацией урана и присутствием захороненного растительного органического вещества.
Рис. 4.
Филогенетическое разнообразие грибов на уровне высших таксонов (а) и родов (б), обнаруженных в первичных культурах аэробных бактерий из образцов пород из скважин Р-76 (519.7 м), Р-78 и Р-123 (513.1 м).
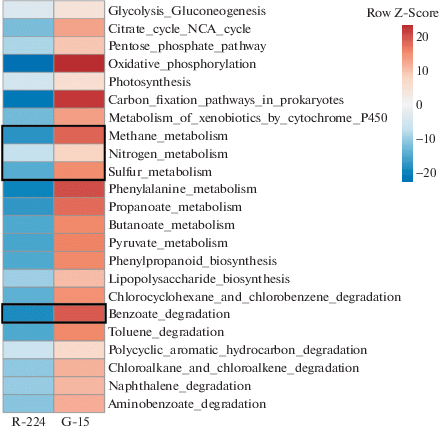
Потенциальные функциональные характеристики микробных сообществ образцов пород и пластовой воды из месторождения Добровольное. Результаты определения филогенетического разнообразия бактерий в образце породы из скв. P-224 (R-224) и пластовой воде из скв. Г-15 (G-15) были дополнительно проанализированы с помощью программного обеспечения iVikodak. На основании взаимосвязи между филогенией и функцией платформа iVikodak позволяет прогнозировать потенциальную функциональную активность сообщества с использованием данных 16S рРНК (Nagpal et al., 2019).
С помощью модуля “Global Mapper” программы iVicodak на основании использования базы данных KEGG были предсказаны функциональные характеристики исследуемых бактериальных сообществ образцов породы R-224 и пластовой воды G-15 (рис. 5). Бактерии, входящие в оба сообщества, осуществляли основные пути метаболизма углеводов и энергетического метаболизма. Однако сообщество пластовой воды G-15 обладало большей потенциальной способностью к осуществлению этих путей, а также путей метаболизма азота и серы, деградации ксенобиотиков, бензоата и связанных с ним путей деградации полициклических ароматических углеводородов, аминобензоата, толуола и хлорсодержащих органических соединений. Особое внимание было уделено превращениям соединений серы и азота, поскольку серную и азотистую кислоты используют для выщелачивания урана, а также превращениям бензоата, как центраболита деградации многих ароматических углеводородов, и превращениям метана и одноуглеродных соединений.
Рис. 5.
Тепловая карта, показывающая сравнение функциональных профилей бактериальных сообществ образцов породы R-224 и пластовой воды G-15 (в относительных процентах) в программе iVikodak. Анализируемые ниже метаболические пути отмечены черной рамкой.
У бактерий, входящих в исследуемые сообщества, было предсказано наличие ферментов комплекса “Метаболизм азота”, участвующих в диссимиляционном восстановлении нитратов до молекулярного азота (собственно денитрификация) и нитратредукции с образованием аммония, а также в ассимиляционном восстановлении нитрата до аммония (рис. S2 , доп. материалы). Микроорганизмы обладали наибольшей потенциальной активностью фермента нитратредуктазы (EC 1.7.5.1), который катализирует восстановление нитрата до нитрита в процессах денитрификации и диссимиляционной нитратредукции. Кроме него присутствовали остальные ферменты этих путей, катализирующие восстановление нитрита до молекулярного азота и аммония соответственно. В меньшей степени у микроорганизмов этих сообществ был представлен фермент ферредоксин-нитратредуктаза (EC 1.7.7.2), катализирующий восстановление нитрата до нитрита в процессе ассимиляционной нитратредукции, и ферредоксин-нитритредуктаза (EC 1.7.7.1), завершающий этот процесс восстановлением нитрита до аммония. Все ферменты пути метаболизма азота были наиболее представлены у микроорганизмов пластовой воды G-15. Наибольший вклад в преобразование азота в сообществе породы R-224 вносили Gammaproteobacteria рода Stenotrophomonas и Alphaproteobacteria рода Kordiimonas, а в сообществе пластовой воды G‑15 – Gammaproteobacteria родов Methylophaga и Pseudomonas, среди которых известны штаммы, способные к денитрификации (рис. S3 , доп. материалы). Тепловые карты относительного содержания ферментов, участвующих в метаболизме азота (а), серы (б), бензоата (в) и метана (г) в бактериальных сообществах породы R‑224 и пластовой воды G-15 приведены на рис. S4 (доп. материалы).
Бактерии родов Methylophaga, Porphyrobacter, Pseudomonas и Ruegeria входили в число ключевых микроорганизмов в метаболизме метана, азота, серы и бензоата в пробе пластовой воды G-15, хотя их относительный вклад в эти процессы немного различался. В образце породы в число ключевых микроорганизмов метаболизма метана, азота и серы входили Kordiimonas, Stenothrophomonas и Filifactor; в превращениях бензоата участвовали преимущественно Kordiimonas, Stenotrophomonas, Acinetobacter и Porphyrobacter (рис. S5–S8 , доп. материалы).
У бактерий, входящих в исследуемые сообщества, среди предсказанных ферментов комплекса “Метаболизм серы” выявлен фермент 3'-фосфоаденозин 5'-фосфосульфат синтаза (EC 2.7.7.4), катализирующий восстановление сульфата до аденилил сульфата (рис. S5 а, S5б). Микроорганизмы обладали также ферментами, катализирующими дальнейшее восстановление аденилил сульфата до сульфита, а затем до сульфида через пути ассимиляционной и диссимиляционной сульфатредукции. В сообществе G-15 диссимиляционная сульфатредукция обусловлена присутствием сульфатредуцирующих бактерий родов Desulfotalea, Desulfobulbus, Desulfovibrio и Desulfomicrobium (рис. S6 а, S6б).
Среди ферментов комплекса “Метаболизм метана” у микроорганизмов образцов породы и пластовой воды было предсказано наличие ферментов метилотрофии, т.е. использования в качестве источников углерода и энергии С1-соединений, вклю-чающих окисленные или замещенные производные метана, не имеющие С–С связи (около 50 соединений), но неспособные расти на самом метане. У бактерий исследуемых сообществ был представлен фермент метанол:5-гидроксибензимидазолилкобамид ко-метилтрансфераза (EC 2.1.1.90), ключевой фермент катаболизма метанола, а также ферменты, катаболизирующие одноуглеродные ксенобиотики – триметиламин, диметиламин и метиламин до формальдегида. При этом представленность этих ферментов была значительно выше у микроорганизмов пластовой воды G-15, что, по-видимому, связано с преобладанием в этом сообществе метилотрофных бактерий рода Methylophaga.
В обоих сообществах присутствовали бактерии, обладающие двумя ключевыми ферментами пути аэробной деградации бензоата (“Benzoate degradation I”), бензоат-1,2-диоксигеназой (EC 1.14.12.10) и дигидрооксициклогексадиен дегидрогеназой (EC 1.3.1.25), которые катаболизируют бензоат до катехола (рис. S7 а, S7б, доп. материалы). У бактериальных сообществ предсказано наличие всех ферментов катаболизма катехола, как до сукцинил-КоА, так и до пирувата/ацетил-КоА. Кроме того, у бактерий обеих сообществ присутствовал фермент бензоат-КoA-лигаза (EC 6.2.1.25), катализирующий превращение бензоата до бензоил-КоА – интермедиата биодеградации многих ароматических соединений. Однако ферменты осуществляемого через бензоил-КоА другого пути деградации бензоата (”Benzoate degradation II”), преимущественно используемого анаэробными бактериями, при анализе исследуемых сообществ не были обнаружены. Этот же модуль программы iVicodak позволил оценить вклад бактерий различных таксонов в осуществление пути “Деградация бензоата”. В сообществе G-15 за деградацию бензоата отвечали Alphaproteobacteria родов Porphyrobacter и Ruegeria и Gammaproteobacteria родов Pseudomonas, Methylophaga и Marinospirillum, а в сообществе R-224 наибольший вклад в деградацию бензоата вносили бактерии родов Kordiimonas (Alphaproteobacteria) и Stenotrophomonas и Acinetobacter (Gammaproteobacteria) (рис. S8 а, S8б, доп. материалы). Известно, что бактерии рода Pseudomonas являются модельным объектом для изучения биодеградации широкого ряда соединений, в том числе, бензоата, катехола и других ароматических и полициклических соединений (Nogales et al., 2017). Имеются также сведения о присутствии генов деградации бензоата и активности детерминируемых ими ферментов и у представителей других выявленных программой родов.
Выделение чистых культур денитрифицирующих бактерий и их идентификация. Из накопительных культур аэробных органотрофных и денитрифицирующих бактерий были выделены чистые культуры (рис. 6). Поскольку большинство денитрифицирующих бактерий способно расти аэробно, чистые культуры получали путем высева нако-пительных культур на указанную выше агаризованную среду для аэробных бактерий и последовательных пересевов выросших единичных колоний в анаэробные условия на среду с нитратом. Выделенные штаммы идентифицировали методом анализа гена 16S рРНК. Уровень сходства полученных последовательностей с соответствующими генами из генетических банков составлял 99–100%, что позволяет отнести штаммы к известным видам родов Bacillus, Peribacillus, Neobacillus, Acinetobacter и Variovorax (табл. 1). Последовательности гена 16S рРНК, близкие генам выделенных штаммов, были выявлены также в составе библиотек образцов породы, пластовой воды и накопительных культур (табл. S4 , рис. S9 , доп. материалы). Из 14 штаммов, проверенных на способность восстанавливать нитрат до нитрита и/или молекулярного азота, 8 штаммов восстанавливали нитрат с образованием нитрита и N2, 3 штамма – до стадии нитрита и 3 штамма не восстанавливали нитрат. С помощью реактива Грисса отмечено накопление нитрита штаммами V3, V4, V3P, V5a, V5b, выросшими в среде с сахарозой, тогда как штамм V2RSG не образовывал ${\text{NO}}_{2}^{ - }$ в этой среде. Штамм Bacillus megaterium V1 накапливал ${\text{NO}}_{2}^{ - }$ в средах с молочной сывороткой и глюкозой, а штамм Bacillus thuringiensis V3 – в средах с глицерином, глюкозой, сахарозой, молочной сывороткой и ацетатом. Наибольшее образование молекулярного азота в газовой фазе отмечено при росте штаммов Acinetobacter lwoffii V7T и V8T в среде с ацетатом и Peribacillus simplex V5b в среде с сахарозой.
Таблица 1.
Таксономическая принадлежность чистых культур бактерий, выделенных из образцов пород месторождения Добровольное, и продукты восстановления нитрата
| Штамм | Номер последовательности гена 16S рРНК в Генбанке | Ближайший типовой штамм вида, номер последовательности гена 16S рРНК в Генбанке | Сходство генов 16S рРНК, % | Образуют ${\text{NO}}_{2}^{ - }$ или N2 | Номер скважины, глубина отбора керна |
|---|---|---|---|---|---|
| V1 | MW541935 | Bacillus megaterium NBRC 15308, CP035094.1 | 100 | ${\text{NO}}_{2}^{ - }$, N2 | Р-123 (513.1 м) |
| V3 | MW542185 | Bacillus thuringiensis ATCC 10792, NR_114581 | 99.9 | ${\text{NO}}_{2}^{ - }$, N2 | Р-78 (544.5 м) |
| V4 | MW542204 | Acinetobacter lwoffii NCTC 5866, NZ_KB851227 | 100 | ${\text{NO}}_{2}^{ - }$, N2 | Р-76 (541.8 м) |
| V5a | MW542594 | Peribacillus simplex NBRC 15720, NR_118967 | 99 | ${\text{NO}}_{2}^{ - }$, N2 | Р-76 (519.7 м) |
| V5b | MW543416 | Peribacillus simplex NBRC 15720, NR_118967 | 99.9 | ${\text{NO}}_{2}^{ - }$, N2 | Р-76 (519.7 м) |
| V1P | MW543704 | Bacillus cereus ATCC 14579, NR_074540 | 99.9 | ${\text{NO}}_{2}^{ - }$ | Р-123 (513.1 м) |
| V2RSG | MW543921 | Neobacillus drentensis NBRC 102427, AB681783 | 99.9 | Р-123 (523.7 м) | |
| V2RS | MW543938 | Neobacillus drentensis NBRC 102427, AB681783 | 99.9 | ${\text{NO}}_{2}^{ - }$ | Р-123 (523.7 м) |
| V2PS | MW544018 | Neobacillus drentensis NBRC 102427, AB681783 | 99.9 | Р-123 (523.7 м) | |
| V3P | MW544028 | Variovorax boronicumulans NBRC 103145, AB681954 | 99.2 | ${\text{NO}}_{2}^{ - }$ | Р-78 (544.5 м) |
| V3RG | MW544052 | Variovorax boronicumulans NBRC 103145, AB681954 | 99.2 | Р-78 (544.5 м) | |
| V6T | MW542313 | Paenibacillus peoriae KCTC 3763, NR_117743.1 | 99.3 | ${\text{NO}}_{2}^{ - }$, N2 | Р-123 (513.1 м) |
| V7T | MW542572 | Acinetobacter lwoffii CIP 64.10, JQ638586 | 99.9 | ${\text{NO}}_{2}^{ - }$, N2 | Р-76 (541.8 м) |
| V8T | MW542576 | Acinetobacter lwoffii CIP 64.10, JQ638586 | 99.9 | ${\text{NO}}_{2}^{ - }$, N2 | Р-76 (541.8 м) |
Рис. 6.
Морфология клеток выделенных штаммов бактерий Bacillus megaterium V1 (а), Bacillus thuringiensis V3 (б), Variovorax boronicumulans V3P (в), Acinetobacter lwoffii V4 (г), Bacillus simplex V5a (д), Peribacillus simplex V5b (е), Paenibacillus peoriae V6T (ж), Acinetobacter lwoffii V7T (з), Acinetobacter lwoffii V8T (и).
Приведенные материалы свидетельствуют о присутствии в подземных горизонтах в зоне месторождения урана жизнеспособного микробного сообщества, включающего бактерии и низшие грибы. Вследствие затрудненного водообмена и массообмена численность культивируемых микроорганизмов в исследованных образцах пород была низка и достигала 106 кл./г породы в зоне ранее проводившегося эксперимента по выщелачиванию урана. Близкие значения численности культивируемых бактерий (104–105 кл./г) были обнаружены также в пробах из месторождения урана (Banduhurang) в Индии (Islam et al., 2011). Бактерии, выделенные из подземных горизонтов месторождения урана Добровольное, относились к родам Bacillus, Peribacillus, Paenibacillus, Neobacillus, Variovorax и Acinetobacter. Этот состав бактерий существенно совпадал с таковым бактерий, выделенных из месторождения урана (Domiasiat) в Индии (Kumar et al., 2013). Проведенный KEGG анализ позволил выявить потенциальную функциональную активность исследованного подземного бактериального сообщества в превращениях компонентов захороненного органического вещества, соединений серы и азота. Вероятно, потери азотистых соединений, выявленные на Далматовском месторождении, аналогичном по геологическим характеристикам месторождению Добровольное, могут быть обусловлены жизнедеятельностью денитрифицирующих бактерий, обитающих в подземных горизонтах.
Список литературы
Долбилин С.И., Мезенов И.А., Галашов С.Г., Ладейщиков А.В., Попонина Г.Ю., Самсонов Б.Г. Месторождение Добровольное // Месторождения урана в речных палеодолинах Уральского региона / Сост. Халезов А.Б. Ред. Машковцев Г.А. . М.: ВИМС, 2009. С. 68–77.
Akob D.M., Mills H.J., Kostka J.E. Metabolically active microbial communities in uranium-contaminated subsurface sediments // FEMS Microbiol. Ecol. 2007. V. 59. P. 95–107. https://doi.org/10.1111/j.1574-6941.2006. 00203.x
Bhakat K., Chakraborty A., Islam E. Characterization of arsenic oxidation and uranium bioremediation potential of arsenic resistant bacteria isolated from uranium ore // Environ. Sci. Pollut. Res. Int. 2019. V. 26. P. 12907–12919. https://doi.org/10.1007/s11356-019-04827-6
Bokulich N.A., Mills D.A. Improved selection of internal transcribed spacer-specific primers enables quantitative, ultra-high-throughput profiling of fungal communities // Appl. Environ. Microbiol. 2013. V. 79. P. 2519–2526. https://doi.org/10.1128/AEM.03870-12
Bolger A.M., Lohse M., Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data // Bioinformatics. 2014. V. 30. P. 2114–2120.
Caporaso J.G., Kuczynski J., Stombaugh J., Bittinger K., Bushman F.D., Costello E.K., Fierer N., Pena A.G., Goodrich J.K., Gordon J.I., Huttley G.A., Kelley S.T., Knights D., Koenig J.E., Ley R.E., Lozupone C.A., McDonald D., Muegge B.D., Pirrung M., Reeder J., Sevinsky J.R., Turnbaugh P.J., Walters W.A., Widmann J., Yatsunenko T., Zaneveld J., Knight R. QIIME allows analysis of high-throughput community sequencing data // Nat. Methods. 2010. V. 7. P. 335–336.
Fadrosh D.W., Ma B., Gajer P., Sengamalay N., Ott S., Brotman R.M., Ravel J. An improved dual-indexing approach for multiplexed 16S rRNA gene sequencing on the Illumina MiSeq platform // Microbiome. 2014. V. 2. P. 6. https://doi.org/10.1186/2049-2618-2-6
Green S.J., Prakash O., Jasrotia P., Overholt W.A., Cardenas E., Hubbard D., Tiedje J.M., Watson D.B., Schadt C.W., Brooks S.C., Kostka J.E. Denitrifying bacteria from the genus Rhodanobacter dominate bacterial communities in the highly contaminated subsurface of a Nuclear Legacy Waste Site // Appl. Environ. Microbiol. 2012. V. 78. P. 1039–1047. https://doi.org/10.1128/AEM.06435-11
Gutierrez T., Aitken M.D. Role of methylotrophs in the degradation of hydrocarbons during the Deepwater Horizon oil spill // ISME J. 2014. V. 8. P. 2543–2545. https://doi.org/10.1038/ismej.2014.88
Hoque E., Fritscher J. Multimetal bioremediation and biomining by a combination of new aquatic strains of Mucor hiemalis // Sci. Rep. 2019. V. 9. Art. 10318. https://doi.org/10.1038/s41598-019-46560-7
Islam E., Dhal P.K., Kazy S.K., Sar P. Molecular analysis of bacterial communities in uranium ores and surrounding soils from Banduhurang open cast uranium mine, India: A comparative study // J. Environ. Sci. Health. Part A: Tox. Hazard. Subst. Environ. Eng. 2011. V. 46. P. 271–280. https://doi.org/10.1080/10934529.2011.535433
Janvier M., Grimont P.A.D. The genus Methylophaga, a new line of descent within phylogenetic branch γ of Proteobacteria // Res. Microbiol. 1995. V. 146. P. 543–550.
Jaswal R., Pathak A., Edwards B., III, Lewis R., III, Seaman J.C., Stothard P., Krivushin K., Blom J., Rupp O., Chauhan A. Metagenomics-guided survey, isolation, and characterization of uranium resistant microbiota from the Savannah River Site, USA // Genes. 2019. V. 10. Art. 325. https://doi.org/10.3390/genes10050325
Kessler J.D., Valentine D.L., Redmond M.C., Du M., Chan E.W., Mendes S.D., Quiroz E.W., Villanueva C.J., Shusta S.S., Werra L.M., Yvon-Lewis S.A., Weber T.C. A persistent oxygen anomaly reveals the fate of spilled methane in the deep Gulf of Mexico // Science. 2011. V. 331. P. 312–315. https://doi.org/10.1126/science.1199697
Kumar R., Nongkhlaw M., Acharya C., Joshi S.R. Uranium (U)-tolerant bacterial diversity from U ore deposit of Domiasiat in North-East India and its prospective utilisation in bioremediation // Microbes Environ. 2013. V. 28. P. 33–41. https://doi.org/10.1264/jsme2.me12074
Lane D.J. 16S/23S rRNA sequencing // Nucleic Acid Techniques in Bacterial Systematics / Ed. Stackebrandt E., Goodfellow M. N.Y.: John Wiley & Sons, 1991. P. 115–175.
Martinez R.J., Wang Y., Raimondo M.A., Coombs J.M., Barkay T., Sobecky P.A. Horizontal gene transfer of PIB-type ATPases among bacteria isolated from radionuclide and metal-contaminated subsurface soils // Appl. Environ. Microbiol. 2006. V. 72. P. 3111–3118.
Metsalu T., Vilo J. ClustVis: A web tool for visualizing clustering of multivariate data using Principal Component Analysis and heatmap // Nucleic Acids Res. 2015. V. 43. P. W566–W570.
Mishamandani S., Gutierrez T., Aitken M.D. DNA-based stable isotope probing coupled with cultivation methods implicates Methylophaga in hydrocarbon degradation // Front. Microbiol. 2014. V. 5. Art. 76. https://doi.org/10.3389/fmicb.2014.00076
Mondani L., Benzerara K., Carrière M, Christen R., Mamindy-Pajany Y., Laureline Février L., Marmier N., Achouak W., Nardoux P., Berthomieu C., Chapon V. Influence of uranium on bacterial communities: a comparison of natural uranium-rich soils with controls // PLoS One. 2011. V. 6. Art. e25771. https://doi.org/10.1371/journal.pone.0025771
Nagpal S., Haque M.M., Singh R., Mande S.S. iVikodak – a platform and standard workflow for inferring, analyzing, comparing, and visualizing the functional potential of microbial communities // Front. Microbiol. 2019. V. 9. Art. 3336. https://doi.org/10.3389/fmicb.2018.03336
Nazina T.N., Luk’yanova E.A., Zakharova E.V., Konstantinova L.I., Kalmykov S.N., Poltaraus A.B., Zubkov A.A. Microorganisms in a disposal site for liquid radioactive wastes and their influence on radionuclides // Geomicrobiol. J. 2010. V. 27. P. 473–486. https://doi.org/10.1080/01490451003719044
Newsome L., Morris K., Lloyd J.R. The biogeochemistry and bioremediation of uranium and other priority radionuclides // Chem. Geol. 2014. V. 363. P. 164–184. https://doi.org/10.1016/j.chemgeo.2013.10.034
Nogales J., García J.L., Díaz E. Degradation of aromatic compounds in Pseudomonas: A systems biology view // Aerobic Utilization of Hydrocarbons, Oils and Lipids. Handbook of Hydrocarbon and Lipid Microbiology / Ed. Rojo F. Springer, Cham., 2017. P. 1–49. https://doi.org/10.1007/978-3-319-39782-5_32-1
North N., Dollhopf S.L., Petrie L., Istok J.D., Balkwill D.L., Kostka J.E. Change in bacterial community structure during in situ biostimulation of subsurface sediment cocontaminated with uranium and nitrate // Appl. Environ. Microbiol. 2004. V. 70. P. 4911–4920. https://doi.org/10.1128/AEM.70.8.4911-4920.2004
Postgate J.R. The Sulfate-Reducing Bacteria. 2nd ed. Cambridge: Cambridge Univ. Press, 1984.
Quast C., Pruesse E., Yilmaz P., Gerken J., Schweer T., Yarza P., Peplies J., Glöckner F.O. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools // Nucl. Acids Res. 2013. V. 41 (Database issue). Art. D590-596. https://doi.org/10.1093/nar/gks1219
Radeva G., Selenska-Pobell S. Bacterial diversity in water samples from uranium wastes as demonstrated by 16S rDN-A and RISA retrievals // Can. J. Microbiol. 2005. V. 1. P. 910–923.
Safonov A.V., Babich T.L., Sokolova D.S, Grouzdev D.S., Tourova T.P., Poltaraus A.B., Zakharova E.V., Merkel A.Y., Novikov A.P., Nazina T.N. Microbial community and in situ bioremediation of groundwater by nitrate removal in the zone of a radioactive waste surface repository // Front. Microbiol. 2018. V. 9. Art. 1985. https://doi.org/10.3389/fmicb.2018.01985
Senko J.M., Dewers T.A., Krumholz L.R. Effect of oxidation rate and Fe(II) state on microbial nitrate-dependent Fe(III) mineral formation // Appl. Environ. Microbiol. 2005. V. 71. P. 7172–7177.
Takahashi S., Tomita J., Nishioka K., Hisada T., Nishijima M. Development of a prokaryotic universal primer for simultaneous analysis of Bacteria and Archaea using next-generation sequencing // PLoS One. 2014. V. 9. Art. e105592. https://doi.org/10.1371/journal.pone.0105592
Vila J., Nieto J.M., Mertens J., Springael D., Grifoll M. Microbial community structure of a heavy fuel oil-degrading marine consortium: linking microbial dynamics with polycyclic aromatic hydrocarbon utilization // FEMS Microbiol. Ecol. 2010. V. 73. P. 349–362.
Widdel F., Bak F. Gram-negative mesophilic sulfate-reducing bacteria // The Prokaryotes / Ed. Balows A., Truper H.G., Dworkin M., Harder W., Schleifer, K.-Z. N.Y.: Springer-Verlag, 1992. Ch. 183. P. 3352–3337.
Wu W.-M., Carley J., Green S.J., Luo J., Kelly S.D., Van Nostrand J., Lowe K., Mehlhorn T., Carroll S., Boonchayanant B., Löfller F.E., Watson D., Kemner K.M., Zhou J., Kitanidis P.K., Kostka J.E., Jardine P.M., Criddle C.S. Effects of nitrate on the stability of uranium in a bioreduced region of the subsurface // Environ. Sci. Technol. 2010. V. 44. P. 5104–5111. https://doi.org/10.1021/es1000837
Yang T., Nigro L.M., Gutierrez T., D’Ambrosio L., Joye S.B., Highsmith R., Teske A.P. Pulsed blooms and persistent oil-degrading bacterial populations in the water column during and after the Deepwater Horizon blowout // Deep-Sea Research Part II: Topical Studies in Oceanography. 2016. V. 129. P. 282–291. https://doi.org/10.1016/j.dsr2.2014.01.014
Дополнительные материалы отсутствуют.


