Микробиология, 2021, T. 90, № 5, стр. 553-563
Термотолерантные метанотрофные бактерии из осадков реки Черная, Крым, и оценка их ростовых характеристик
И. Ю. Ошкин a, О. В. Данилова a, Р. З. Сулейманов a, Е. Н. Тихонова a, Т. В. Малахова b, А. И. Мурашова b, Н. В. Пименов a, С. Н. Дедыш a, *
a Институт микробиологии им. С.Н. Виноградского, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” РАН
119071 Москва, Россия
b Федеральный исследовательский центр “Институт биологии южных морей им. А.О. Ковалевского РАН”
299011 Севастополь, Россия
* E-mail: dedysh@mail.ru
Поступила в редакцию 13.04.2021
После доработки 23.04.2021
Принята к публикации 28.04.2021
Аннотация
Технология производства микробного белка из природного газа опирается на использование термотолерантных метанотрофных бактерий, характеризующихся высокой скоростью роста на метане. Спектр ранее задействованных в производстве штаммов-продуцентов ограничен представителями рода Methylococcus, что накладывает ограничения на развитие этой технологии и делает актуальным поиск новых культур быстрорастущих метанотрофов. Настоящая работа посвящена решению этой задачи с использованием образцов донного осадка реки Черная, Крым. Молекулярный анализ состава микробного сообщества осадка выявил доминирование представителей Gammaproteobacteria (33–42% полученных фрагментов генов 16S рРНК), а также Chloroflexi, Actinobacteria, Alphaproteobacteria, Bacteroidota и Acidobacteria в качестве численно значимых компонентов сообщества. В полученной из осадка первичной накопительной культуре метанотрофных бактерий около 60% фрагментов генов 16S рРНК принадлежали представителям рода Methylomonas. Применение различных стратегий выделения чистых культур метанотрофов позволило получить три изолята целевых бактерий родов Methylomonas, Methylomagnum и Methylocystis, температурные оптимумы роста которых составили 25, 35 и 40°С соответственно. Наиболее высокая скорость роста в периодической культуре, 0.21 ч–1, была зарегистрирована для Methylocystis sp. Kr9, обнаруживающего 99.22 и 99.13% сходства генов 16S рРНК с типовыми штаммами Methylocystis echinoides IMET 10491T и Methylocystis parvus OBBPT.
Аэробные метанотрофные бактерии – это уникальная группа прокариот, структурно и функционально специализированных на использовании метана (СН4) в качестве источника углерода и энергии (Hanson, Hanson, 1996; Гальченко, 2001; Trotsenko, Murrell, 2008; Khmelenina et al., 2018). В настоящее время известно около трех десятков родов метанотрофных бактерий, которые принадлежат к классам Gammaproteobacteria (метанотрофы I типа) и Alphaproteobacteria (метанотрофы II типа), а также несколько родов метанотрофов, относящихся к филе Verrucomicrobia (Dedysh, Knief, 2018). Метанотрофные бактерии широко распространены в местообитаниях с высокой доступностью метана, таких как донные осадки пресноводных экосистем (Beck et al., 2013; Deutzmann et al., 2014), болота (Dedysh, 2009; Danilova et al., 2016), рисовые чеки (Ma et al., 2013; Vaksmaa et al., 2017), покрывающие почвы полигонов твердых бытовых отходов (Cébron et al., 2007; Kumaresan et al., 2009) и др. Биологическое окисление метана является важным звеном глобального цикла СН4, а также и глобального круговорота углерода в природе (Conrad, 2009). Метанотрофы, однако, имеют также и значительный биотехнологический потенциал (Троценко, Хмеленина, 2009; Conrado, Gonzalez, 2014; Kalyuzhnaya et al., 2020). Микробная конверсия метана, являющегося доступным и сравнительно дешевым сырьем, открывает перспективы производства продуктов с добавленной стоимостью (Strong et al., 2015).
Устойчивый интерес к развитию биотехнологий на основе использования метанотрофных бактерий появился в 70-е годы XX в. Было показано, что метанотрофные бактерии могут быть источником биопротеина, синтезируемого на основе метана (Григорян, Горская, 1970). Масштабное промышленное производство кормового белка (Гаприна) из природного газа с использованием термотолерантного метанотрофа Methylococcus capsulatus ВСБ-874 было реализовано в СССР в середине 1980-х годов. По составу и количеству незаменимых аминокислот биомасса метанотрофов сопоставима с рыбной и соевой мукой и, следовательно, имеет определенные преимущества относительно ряда продуктов растительного происхождения (Coty, 1969). Особенно выделяются метанотрофы класса Gammaproteobacteria, имеющие наиболее высокое содержание белка и незаменимых аминокислот (Плясов, 1988).
Экономическая целесообразность производства микробного белка из природного газа напрямую зависит от продуктивности метанотрофных микроорганизмов (Ritala et al., 2017). Производственные штаммы должны обладать высокой скоростью роста, что характерно в первую очередь для термотолерантных или умеренно термофильных метанотрофных бактерий. Исходная технология получения бактериального белка из метана была реализована с использованием термотолерантного метанотрофа Methylococcus capsulatus. Аналогичные производства, налаженные в других странах, опираются на использование штаммов того же вида, однако подобная узость спектра продуцентов существенно тормозит развитие этой технологии. Число таких штаммов ограничено, так как процедура выделения метанотрофов в чистых культурах сложна методически и может занимать до нескольких лет. Долгое время Methylococcus capsulatus Bath оставался самым быстрорастущим метанотрофным микроорганизмом с максимально зарегистрированной удельной скоростью роста 0.37 ч–1 (Joergensen, Degn, 1987). За последние годы, однако, опубликованы несколько работ по получению штаммов рода Methylomonas, для которых аналогичный показатель составил 0.39 и 0.40 ч–1 (Guo et al., 2017; Kim et al., 2018). Это наиболее высокие значения удельной скорости роста, зарегистрированные для метанотрофных бактерий. Таким образом, вопрос о возможностях и пределах роста метанотрофных бактерий остается открытым, и поиск альтернативных продуцентов с улучшенными свойствами является перспективной задачей биотехнологии получения кормового белка на основе метанотрофных микроорганизмов.
Охарактеризованные представители термотолерантных и умеренно термофильных метанотрофных бактерий принадлежат к родам Methylococcus, Methylocaldum, Methylothermus и Methylomarinovum (Троценко и соавт., 2009; Hirayama et al., 2014; Dedysh, Knief, 2018). Метанотрофы рода Methylothermus населяют термальные пресноводные источники; эти бактерии крайне нестабильны в культивировании и быстро теряют жизнеспособность. Метанотрофы рода Methylomarinovum населяют морские гидротермы и требуют морскую воду для культивирования. Представители Methylothermus и Methylomarinovum, таким образом, малопригодны для производственных целей. Спектр местообитаний Methylococcus и Methylocaldum включает воду и осадки пресноводных экосистем, рисовые чеки, полигоны твердых бытовых отходов и компосты (Knief, 2015).
Целью настоящей работы являлось выделение новых штаммов термотолерантных метанотрофных бактерий, отличающихся высокими скоростями роста, из образцов донного осадка реки Черная, Крым.
ОБЪЕКТЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Отбор образцов и аналитические методы. В работе был использован образец верхнего слоя (0–2 см) донного осадка реки Черная, Крым (44.59° с.ш., 33.61° в.д.). Отбор проб донных осадков и придонной воды проводили акриловой грунтовой трубкой с вакуумным затвором. При помощи экструдера в полевых условиях с керна срезали верхний 2-см слой донных осадков. Пористость и влажность осадка определяли, соответственно, как процентную долю объема и массы поровой воды в осадке. Гидрохимические параметры проб воды определяли стандартными аналитическими методиками. Температуру воды и общую минерализацию измеряли в полевых условиях портативным анализатором качества воды TDS-3 М. Определение содержания углерода органического (Сорг) в колонках донных отложений выполняли, согласно спектрофотометрической методике, после сульфохромового сжигания органического вещества. Окисление органических соединений проводили в воздушно-сухих пробах донных отложений. Органические вещества донных осадков окисляли при температуре 125°С 0.4 N раствором бихромата калия в концентрированной серной кислоте в соотношении 1 : 1. Измерение оптических плотностей выполняли после осаждения минеральных частиц на спектрофотометре при длине волны 590 нм на приборе Helios Alfa (“Thermo Fisher Scientific”, США). Для извлечения метана из проб воды и донных отложений использовали способ фазово-равновесной дегазации (Большаков, Егоров, 1987). Определение концентраций метана в воде и донных отложениях проводили на хроматографе HP 5890 (“Hewlett Packard”, США) со стальной набивной колонкой (длина колонки – 1 м, внутренний диаметр – 2 мм, сорбент “Porapack Q” 80/100 меш., “Serva”) и пламенно-ионизационнным детектором при следующих условиях: газ-носитель – азот, объемная скорость 30 мл мин–1, температура детектора – 225°С, инжектора – 120°С. Часть отобранного ила использовали в качестве инокулята для получения накопительной культуры метанотрофных бактерий, тогда как другую его часть хранили при ‒20°С для последующей экстракции тотальной ДНК.
Получение накопительных культур метанотрофных бактерий. Для получения накопительной культуры метанотрофов использовали минеральную среду NMS (Nitrate Mineral Salts), разбавленную в соотношении 2 : 5 и содержащую (г/л): KNO3 – 0.2; MgSO4 · 7H2O – 0.2; CaCl2 · 2H2O – 0.04; 1% (об./об.) 200 мМ фосфатного буфера (рН 7.0) и 0.1% (об./об.) раствора микроэлементов для метанотрофов (Гальченко, 2001) следующего состава (г/л): ЭДТА – 5; FeSO4 · 7H2O – 2; ZnSO4 · 7H2O – 0.1; MnCl2 · 4H2O – 0.03; Co-Cl2 · H2O – 0.2; CuCl2 · 5H2O – 0.1; NiCl2 · · 6H2O – 0.02; Na2MoO4 – 0.03. Один грамм ила вносили во флаконы общим объемом 500 мл со 100 мл жидкой среды. В газовую фазу флаконов вводили метан до 30 об. % и инкубировали на шейкере (120 об./мин) при температуре 35°С в течение 10 дней.
Молекулярное профилирование состава микробного сообщества образца ила и полученной из него накопительной культуры метанотрофов. Для выделения тотальной ДНК из исследуемого речного осадка использовали три навески ила по 0.5 г каждая. Выделение ДНК из полученной накопительной культуры метанотрофных бактерий проводили в двукратной повторности с использованием образцов клеток, осажденных из 5 мл исследуемой культуры. ДНК выделяли с использованием набора FastDNA SPIN kit for soil (“MP Biomedicals”, США) в соответствии с рекомендациями фирмы-изготовителя. Полученные образцы ДНК хранили до анализа при –20°С. Состав сообщества прокариот определяли на основании анализа последовательностей вариабельного региона V3–V4 гена 16S рРНК, амплифицированного с помощью ПЦР с использованием праймеров PRK341F (5'‑CCTACGGGRBGCASCAG-3') и PRK806R (5'-GGACTACYVGGGTATCTAAT-3'). Полученные ПЦР фрагменты использовали для приготовления библиотеки для секвенирования с помощью наборов Nextera XT DNA Library Prep Kit (“Illumina”, США) по протоколам производителя. Полученные библиотеки секвенировали на MiSeq (“Illumina”, США) с использованием наборов MiSeq Reagent Kit V3 (в формате парных чтений 2 × 300 нт). Полученный набор фрагментов последовательностей 16S рРНК анализировали с использованием программных модулей пакета QIIME 2 v.2018.8 (https://qiime2.org) (Bolyen et al., 2019). Объединение нуклеотидных последовательностей, контроль качества и выбраковку химерных последовательностей проводили с помощью плагина DADA2 (Callahan et al., 2016). Далее последовательности кластеризовали в операционные таксономические единицы (ОТЕ) на видовом уровне идентичности (97% сходства) с помощью плагина VSEARCH (Rognes et al., 2016) и базы данных Silva v. 138, содержащей последовательности гена 16S рРНК (Quast et al., 2013; Yilmaz et al., 2014). Таксономическую идентификацию ОТЕ проводили методом BLASTN по базе Silva v. 138 (Altschul et al., 1997). Полученный из образцов ила и накопительной культуры пул фрагментов нуклеотидных последовательностей гена 16S рРНК депонирован в ГенБанке под номером PRJNA722466.
Получение и идентификация изолятов метанотрофных бактерий. Получение чистых культур метанотрофных бактерий проводили с использованием минеральной среды NMS 2 : 5 и следующих альтернативных подходов: 1) многократных предельных разведений культуры в жидкой среде; 2) рассева разбавленной суспензии клеток на агаризованный вариант среды для получения колоний; 3) рассева суспензии клеток на жидкую среду с использованием культуральных планшетов. Для приготовления предельных разведений использовали серии флаконов объемом 60 мл, содержащие 5 мл среды. Флаконы герметично закрывали, вводили метан шприцем с фильтром до 30% СН4 в газовой фазе и инкубировали на качалке (120 об./мин). Культуру, выросшую во флаконе с наибольшим разведением, микроскопировали и подвергали последующему рассеву до получения культуры, состоящей из морфологически одинаковых клеток. В случае рассевов суспензии культур на агаризованную среду, чашки помещали в эксикаторы с 30% метана в газовой фазе и инкубировали в течение 10 сут при 30, 35 и 42°С. Формирующиеся колонии отсевали во флаконы с жидкой средой, полученные культуры проверяли на чистоту с помощью фазово-контрастной микроскопии и вновь рассевали на агаризованную среду до получения чистой культуры. Процедура получения изолятов с использованием культуральных планшетов была предложена ранее для селекции быстрорастущих штаммов метанотрофов (Hoefman et al., 2012). Аликвоты накопительной культуры рассевали методом предельных разведений на 48-луночные культуральные планшеты (“Eppendorf”). Использовали серии из 15 последовательных предельных разведений; объем среды в каждой из лунок составлял 100 мкл. Планшеты помещали в герметичные пакеты “Genbag anaerobic” (“BioMérieux”, Франция) с заданным составом газовой фазы (20–40% метана и 60–80% воздуха) и инкубировали на шейкере (120 об./мин) при температурах инкубации 35 и 42°С. Замену газовой фазы в пакетах проводили каждые 2 дня. Рост культур определяли микроскопически и с помощью регистрации оптической плотности при длине волны 600 нм, которую измеряли на спектрофотометре Biophotometer AG 22331 (“Eppendorf”).
Идентификацию полученных изолятов осу-ществляли путем анализа последовательностей генов 16S рРНК, ПЦР-амплификацию которых проводили с использованием универсальных для бактерий праймеров (Weisburg et al., 1991). Очистку ПЦР-амплифицированных фрагментов осуществляли с использованием набора Wizard® SV Gel and PCR Clean-Up System (“Promega”, США) в соответствии с рекомендациями фирмы производителя. Секвенирование амплифицированных фрагментов генов 16S рРНК проводили на базе ЦКП “Биоинженерия” ФИЦ Биотехнологии РАН. Определенные в работе последовательности генов 16S рРНК изолятов метанотрофных бактерий депонированы в ГенБанке под номерами MW830155–MW830156 и MW884260.
Анализ морфологии и ультратонкого строения клеток изолятов. Фотографии клеток изолятов получали с помощью микроскопа Zeiss Axioplan 2 и цифровой фотокамеры Zeiss с матрицей 6 М-пкс, с использованием программного пакета AxioVision v. 4.2.
Исследование ультратонкого строения клеток изолятов метанотрофов проводили с помощью электронной микроскопии. Для этого образцы клеток фиксировали раствором 2.5% глутарового альдегида в какодилатном буфере (0.05 М раствор какодилата натрия, рН 7.0–7.5) и выдерживали в течение суток при 4°С; затем трижды промывали тем же буферным раствором в течение 5 мин и фиксировали в растворе OsO4 (1% OsO4 + 0.7% раствор рутениевого красного в какодилатном буфере) 1.5 ч при 4°С. После фиксации образцы заключали в 2% агар-агар и последовательно выдерживали в 3% растворе уранилацетата в 30% этиловом спирте в течение 4 ч, далее в 70% этаноле в течение 12 ч при 4°С. Материал обезвоживали в 96% этиловом спирте (2 раза по 15 мин), затем в абсолютном ацетоне (3 раза по 10 мин). Образцы пропитывали смолой ЭПОН-812 (Ероху Embedding Medium Эпон® 812, “Sigma-Aldrich”, США) выдерживая в смеси смола : ацетон в соотношении 1 : 1 (1 ч), затем в смеси смола : ацетон в соотношении 2 : 1 (1 ч). Полученный материал заливали в капсулы со смолой и проводили полимеризацию при температуре 37°С в течение суток, затем при 60°С в течение суток. Ультратонкие срезы получали на микротоме LKB-III (“LKB”, Швеция) и контрастировали в водном растворе 3% уранилацетата (30 мин), затем в водном растворе 4% цитрата свинца (30 мин). Полученные препараты анализировали с помощью электронного микроскопа JEM 100CXII (“JEOL”, Япония) при ускоряющем напряжении 80 кВ и рабочем увеличении 5000–50 000. Фотодокументирование материалов проводили с помощью цифровой системы вывода оптических изображений Morada G2.
Оценка ростовых характеристик культур метанотрофов. Определение температурного оптимума и ростовых характеристик изолятов проводили при культивировании в жидкой среде NMS 2 : 5 во флаконах с 30 об. % метана в газовой фазе и инкубации на шейкере (150 об./мин) в диапазоне температур от 20 до 50°С, с шагом в 5°С. Рост оценивали путем регулярных измерений оптической плотности культуры на спектрофотометре Eppendorf Biophotometer AG 22331 (Германия) при длине волны 600 нм. Расчет удельной скорости роста (µ, ч–1) проводили в экспоненциальной фазе роста культур по формуле: µ = (ln(OD2) – ln(OD1)/(t2 – – t1), где OD1 и OD2 – значения оптической плотности культуры в начале и конце экспоненциальной фазы роста, t1 и t2 – соответствующие им временные точки.
РЕЗУЛЬТАТЫ
Характеристика образцов донного осадка реки Черная. Осадок был представлен тонкодисперсным илом светло-серого цвета с рыжим наилком, без запаха сероводорода, без вкраплений гравия и без остатков водорослей-гидрофитов. Пористость осадка составляла 85%, содержание органического углерода – 7% от массы пробы, влажность – 75%, соленость – менее 0.3‰. Концентрация растворенного метана была равна 20.5 мкмоль/л. Величина общей минерализации воды составила 2.9 г л–1; в составе растворенных соединений были идентифицированы (мкг/л): ${\text{NO}}_{2}^{ - }$ – 27.6; ${\text{NO}}_{3}^{ - }$ – 869; ${\text{NH}}_{4}^{ + }$ – 57.9; ${\text{PO}}_{4}^{{3 - }}$ – 48.3. Температура воды и осадка в момент отбора образцов составила 22°C.
Молекулярный анализ состава микробного сообщества в донном осадке. В общей сложности, из образца осадка реки Черная было получено 88 655 частичных последовательностей генов 16S рРНК со средней длиной ~300 нт. В результате процедуры контроля качества, объединения парных последовательностей и выбраковки химер было получено 17 437 последовательностей, задействованных для анализа. Индекс Good’s coverage, рассчитываемый для оценки глубины секвенирования, составил 0.99 для всех повторностей. Индексы разнообразия Chao1 и Shannon варьировали, соответственно, в диапазонах 102–179 и 6.29–7.06. Число операционных таксономических единиц (ОТЕ), соответствующих видовому уровню идентичности последовательностей (97%), составляло от 100 до 172.
Наиболее многочисленная группа полученных из осадка реки Черная фрагментов гена 16S рРНК принадлежала представителям Gammaproteobacteria (32.1% всех прочтений). Другими численно значимыми компонентами микробного сообщества ила являлись Chloroflexi (20.2%), Alphaproteobacteria (9.1%), Bacteroidota (8.2%), Acidobacteria (7.2%), Desulfobacterota (6.2%), Actinobacteria (5.8%) и Firmicutes (3.0%) (рис. 1а). На уровне семейств доминирующими компонентами микробного сообщества ила являлись представители Anaerolineaceae (15.2–16.7%), Thermoanaerobaculaceae (4.1–5.3%), Halieaceae (3.4–5.9%) и неописанного пока семейства порядка Rhizobiales (1.5–8.4%). Микроорганизмы, принадлежащие к вышеперечисленным семействам, однако, не могли быть классифицированы до рода и представляли новые, неописанные пока таксоны. Ни одна из идентифицированных в речном иле ОТЕ не могла быть классифицирована как принадлежащая метанотрофным бактериям, что свидетельствовало о низкой численности метанотрофов в исследуемом образце ила. Этап получения накопительных культур, таким образом, был необходимым условием получения изолятов целевых микроорганизмов.
Рис. 1.
Состав бактериального сообщества: a – в природном образце ила; б – в накопительной культуре, сформированной после 1 мес. культивирования в условиях повышенных температур и в присутствии метана в качестве источника углерода.
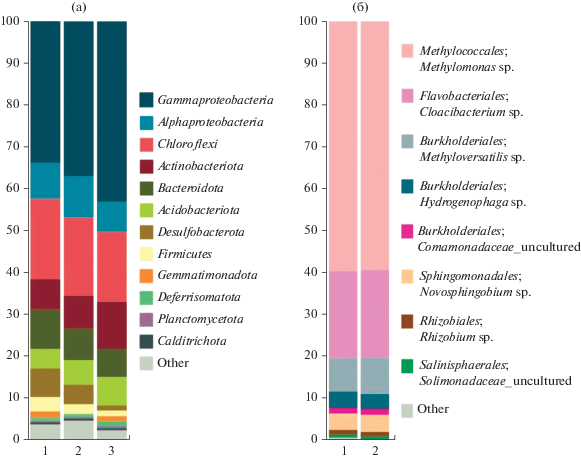
Получение и молекулярный анализ накопительных культур. Во флаконах со средой NMS 2 : 5, инокулированной образцами осадка, после 10 сут культивирования при 35°С наблюдалось активное развитие метанотрофного микробного сообщества. Его анализ с помощью фазово-контрастной микроскопии выявил доминирование подвижных клеток палочковидной морфологии, характерной для представителей рода Methylomonas. В результате молекулярного анализа из этой накопительной культуры было получено 26 986 частичных последовательностей генов 16S рРНК со средней длиной ~250 нт. Из них после процедуры контроля качества и выбраковки химер было задействовано для анализа 23 970 последовательностей, что составило не менее 87% от общего пула прочтений гена 16S рРНК. Общее видовое разнообразие было представлено 18 ОТЕ. Около 60% фрагментов всех генов 16S рРНК принадлежали представителям рода Methylomonas (рис. 1б). Последние были представлены одной ОТЕ видового уровня, обнаруживающей 98% сходства последовательностей гена 16S рРНК с Methylomonas koyamae Fw12E-YT. Другие основные компоненты полученной накопительной культуры были представлены бактериями родов Cloacibacterium (20.8%), Methyloversatilis (8.0%), Novosphingobium (3.9%) и Hydrogenophaga (3.9%).
Выделение изолятов метанотрофных бактерий. Полученная на раннем этапе культивирования накопительная культура содержала в себе лишь один доминирующий вид метанотрофов. Для получения этих бактерий в чистой культуре был проведен прямой высев клеточной суспензии на агаризованную среду NMS 2 : 5, что позволило получить изолят М9, клетки которого были представлены короткими подвижными палочками длиной 2–4 мкм (рис. 2а). Анализ ультратонких срезов клеток выявил наличие стопок внутрицитоплазматических мембран (ВЦМ), характерных для метанотрофов I типа (рис. 2б). В жидкой среде штамм М9 показывал тенденцию к хлопьевидному росту, благодаря наличию ярко выраженной слизистой капсулы, типичной для метанотрофов рода Methylomonas. Анализ гена 16S рРНК подтвердил принадлежность штамма М9 к роду Met-hylomonas (табл. 1), а также показал идентичность последовательности гена 16S рРНК штамма М9 таковой у соответствующей ОТЕ, идентифицированной в составе накопительной культуры. Уровень сходства с ближайшим типовым штаммом M. koyamae Fw12E-YT составил 97.2%, что может означать принадлежность штамма М9 к новому виду рода Methylomonas.
Рис. 2.
Морфология (а, в, д) и ультратонкие срезы клеток (б, г, е) изолятов метанотрофных бактерий: Methylomonas sp. М9 (a, б), Methylomagnum sp. Kr7 (в, г), Methylocystis sp. Kr9 (д, е). Стрелками указаны внутрицитоплазматические мембраны. Маркер – 5 мкм (а, в, д) и 1 мкм (б, г, е).
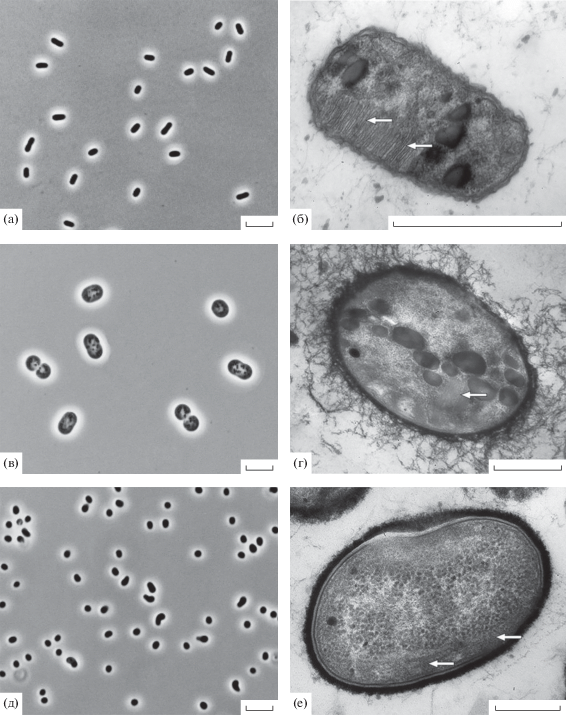
Таблица 1.
Результаты идентификации изолятов метанотрофных бактерий, полученных из осадков реки Черная
| Штамм | Номер последовательности гена 16S рРНК в ГенБанке | Ближайший филогенетически родственный типовой штамм метанотрофов | % сходства генов 16S рРНК |
|---|---|---|---|
| М9 | MW884260 | Methylomonas koyamae Fw12E-YT | 97.2 |
| Kr7 | MW830155 | Methylomagnum ishizawai RS11D-PrT | 99.53 |
| Kr9 | MW830156 | Methylocystis echinoides IMET 10491T Methylocystis parvus OBBPT |
99.22 99.13 |
Для более полного выявления разнообразия метанотрофов, присутствующих в исходной накопительной культуре в низкой численности, в виде единичных клеток, диапазон условий культивирования был расширен за счет варьирования температур инкубации от 30 до 42°С и рН среды от 5 до 7 с одновременным увеличением временного диапазона между пересевами до десяти дней. Эта стратегия позволила получить ряд смешанных культур с несколькими новыми морфотипами метанотрофных бактерий, отличными от морфотипа штамма М9. Так, культивирование на среде с рН 7.0 при температуре 30°С позволило получить смешанную культуру, в которой, наряду с Methylomonas-подобными метанотрофами, присутствовал также метанотроф с крупными клетками овоидной морфологии. Изолят этих метанотрофов, штамм Kr7, был получен с применением метода выделения в планшетах. Анализ последовательности гена 16S рРНК штамма Kr7 показал его принадлежность к виду Methylomagnum ishizawai (табл. 1). Штамм Kr7 был представлен одиночными подвижными клетками овальной формы, размером до 5 мкм (рис. 2в), на ультратонких срезах которых выявлялись ВЦМ I типа (рис. 2г).
Изменение условий культивирования первичной накопительной культуры в сторону понижения рН среды до 5.8 с повышением температуры до 40°С привело к появлению в составе сообщества единичных клеток метанотрофов грушевидной формы. Последующее использование метода выделения в планшетах позволило получить изолят этих метанотрофов, штамм Kr9. Клетки штамма Kr9 представляли собой подвижные в экспоненциальной фазе, изогнутые палочки или вибриоиды размером 1–4 мкм (рис. 2д). Анализ ультратонких срезов клеток показал наличие ламеллярных ВЦМ II типа, расположенных на периферии клетки, параллельно цитоплазматической мембране (рис. 2е). Анализ гена 16S рРНК показал принадлежность штамма Kr9 к роду Methylocystis, со сходством 99.22 и 99.13% сходства генов 16S рРНК с типовыми штаммами Methylocystis echinoides IMET 10491T и Methylocystis parvus OBBPT. Точное установление видовой принадлежности штамма Kr9, таким образом, требует применения сравнительного геномного анализа.
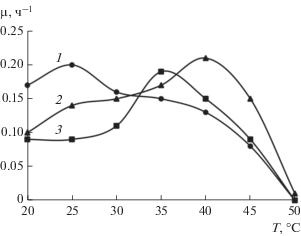
Ростовые характеристики изолятов метанотрофов. Зависимость удельной скорости роста изолятов от температуры инкубации представлена на рис. 3. Все культуры были способны к росту до 48°С, однако имели различные температурные предпочтения. Наиболее активный рост Methylomonas sp. М9 имел место в диапазоне 20–30°С, с оптимумом при 25°С (µ = 0.20 ч–1). Methylomagnum ishizawai Kr7 демонстрировал активный рост в диапазоне температур 30–40°С с оптимумом при 35°С (µ = 0.19 ч–1). Наиболее высокий температурный оптимум, 40°С, и наиболее широкий температурный диапазон активного роста, 25–45°С, был зарегистрирован для Methylocystis sp. Kr9. Удельная скорость роста штамма Kr9 при 40°С составила 0.21 ч–1 (рис. 3). Таким образом, все полученные в работе штаммы метанотрофов могли быть охарактеризованы как термотолерантные мезофилы. Наиболее активный рост при температурах 40–45°С демонстрировал Methylocystis sp. Kr9.
ОБСУЖДЕНИЕ
В настоящей работе нами был применен молекулярный метод оценки состава микробного сообщества на основе секвенирования гена 16S рРНК в сочетании с классическими подходами культивирования микроорганизмов, направленными на поиск новых быстрорастущих изолятов метанотрофных бактерий. Сайт отбора образцов располагался в субтропическом поясе, для которого характерны повышенные среднемесячные температуры. Концентрация растворенного метана в образцах (20 мкмоль/л) была сопоставима с обнаруженными в пресноводных осадках озера Вашингтон в США (2–170 мкмоль/л) (Auman et al., 2000) и Боденского озера в Германии (до 140 мкмоль/л) (Rahalkar et al., 2009). Оценка обилия метанотрофных бактерий в этих местообитаниях дала величины численности ≈109 клеток г–1 сухой биомассы для верхнего слоя осадка озера Вашингтон и 106–109 клеток г–1 влажного осадка в различных локациях Боденского озера (Costello et al., 2002; Deutzmann et al., 2011). Исследование состава микробного сообщества осадка озера Вашингтон с помощью высокопроизводительного секвенирования генов 16S рРНК показало, что доля фрагментов метанотрофных бактерий составила около 10% (Beck et al., 2013). В настоящей работе молекулярный анализ не выявил наличия метанотрофов в составе исследуемого микробного сообщества. Более того, для образца в целом было характерно низкое микробное разнообразие. Специфика места отбора образцов заключается в том, что для полузакрытой Севастопольской бухты характерны сейши, которые могут значительно влиять на термохалинные параметры воды кутовой части бухты, захватывая также исследованный район. В результате волновых процессов может меняться направление течения, и, как следствие, гидрохимические показатели воды. В точке отбора образцов соленость может меняться от 0 до 4‰ (неопубликованные данные). В связи с этим результаты одномоментного отбора проб показывают некоторое состояние в диапазоне возможных состояний гидрологических характеристик воды реки. Вполне вероятно, что изменчивые условия не способствуют формированию устойчивого сообщества, что потенциально может проявляться в его низком разнообразии и, как следствие, в отсутствии достаточного для детекции молекулярными методами количества метанотрофных бактерий. Тем не менее, отрицательный результат по наличию последовательностей гена 16S рРНК метанотрофных бактерий не исключал их присутствия в образцах осадка реки Черная. Ранее было показано, что применение культуральных подходов позволяет выявлять микроорганизмы, детекция которых молекулярными методами затруднена из-за их низкой представленности в сообществе (Houghton, Stewart, 2020). В настоящей работе использование селективных температур инкубации в диапазоне от 30 до 42°С позволило получить ряд чистых культур метанотрофных бактерий с различающимися температурными оптимумами.
Изолят Kr7 обнаруживал 99.5% сходства последовательности гена 16S рРНК с отмеченной у Methylomagnum ishizawai RS11D-PrT, мезофила, растущего при 20–37°C c оптимумом 31–33°C (Khalifa et al., 2015). Температурный оптимум штамма Kr7 (35°C) был несколько выше описанного ранее для этого вида метанотрофов. Для изолята рода Me-thylomonas, штамма М9, максимальная скорость роста (0.2 ч–1) была зарегистрирована при 25°C, что выше опубликованных величин для ряда других охарактеризованных представителей этого рода (Morinaga et al., 1976; Danilova et al., 2013). По опубликованным данным наиболее высокий показатель скорости роста (0.386 ч–1) среди всех известных на данный момент метанотрофных бактерий был получен как раз для одного из штаммов Methylomonas (Guo et al., 2017), однако он был зарегистрирован при росте в ферментере. Скорость роста этого же штамма при периодическом культивировании во флаконах была существенно ниже и составила 0.2 ч–1. Третий изолят, штамм Kr9, отличался стабильным ростом, как в жидкой культуре, так и на агаризованной среде, с оптимумом при 42°С. Для близкородственного штамма Methylocystis parvus OBBPT была зарегистрирована скорость роста 0.1 ч–1 (Pieja et al., 2011), в то время как штамм Kr9 рос в два раза быстрее (0.2 ч–1). В ряде исследований было показано, что Methylocystis parvus является перспективным продуцентом полигидроксибутирата (Rostkowski et al., 2013; Sundstrom, Criddle, 2015; Rumah et al., 2021). Штаммы этого вида метанотрофов использовали для укрепления бетона биогенным способом за счет окисления формиата (Ganendra et al., 2015), а также для получения продуктов с добавленной стоимостью при совместном культивировании в аквакультуре с водорослью Scenedesmus sp. (van der Ha et al., 2012). Methylocystis parvus относится к метанотрофам II типа, которые, в отличие от метанотрофов I типа, не склонны к спонтанному лизису клеток, хорошо сохраняются при длительном хранении и могут достигать более высоких значений оптической плотности (Murrell, 2010). В сочетании с высокой скоростью роста эти качества штамма Kr9 могут быть востребованы в промышленном производстве микробного белка.
Таким образом, в результате данной работы было получено 3 новых изолята термотолерантных метанотрофных бактерий, один из которых – Methylocystis sp. Kr9 – демонстрировал наиболее высокую удельную скорость роста в периодической культуре и наиболее широкий температурный диапазон роста. Подбор оптимальных параметров и условий культивирования в модельных ферментерах представляет следующий шаг на пути установления ростового потенциала этих бактерий и определения их применимости для производства белка из природного газа.
Список литературы
Большаков А.М., Егоров А.В. Об использовании методики фазоворавновесной дегазации при газометрических исследованиях // Океанология. 1987. Т. 27. № 5. С. 861–862.
Гальченко В.Ф. Метанотрофные бактерии. М.: ГЕОС, 2001. 500 с.
Григорян А.Н., Горская Л. Использование природного газа для микробиологического синтеза. М.: ОНТИ Микробиопром, 1970. 90 с.
Плясов Ю.М. Комплексная оценка питательной ценности кормового микробного белка // Биотехнология. 1988. Т. 4. № 3. С. 402–408.
Троценко Ю.А., Медведкова К.А., Хмеленина В.Н., Ешинимаев Б.Ц. Термофильные и термотолерантные аэробные метанотрофы// Микробиология. 2009. Т. 78. № 4. С. 435–450.
Trotsenko Y.A., Medvedkova K.A., Khmelenina V.N., Eshinimayev B.Ts. Thermophilic and thermotolerant aerobic methanotrophs // Microbiology (Moscow). 2009. V. 78. P. 387–401.
Троценко Ю.А., Хмеленина В.Н. Экстремофильные метанотрофы. М.: ОНТИ ПНЦ РАН, 2009. 204 с.
Altschul S.F., Madden T.L., Schäffer A.A., Zhang J., Zhang Z., Miller W., Lipman D.J. Gapped BLAST and PSI-BLAST: a new generation of protein // Nucleic Acids Res. 1997. V. 25. P. 3389–3402.
Auman A.J., Stolyar S., Costello A.M., Lidstrom M.E. Molecular characterization of methanotrophic isolates from freshwater lake sediment // Appl. Environ. Microbiol. 2000. V. 66. P. 5259–5266.
Beck D.A.C., Kalyuzhnaya M.G., Malfatti S., Tringe S.G., del Rio T.G., Ivanova N., Lidstrom M.E., Chistoserdova L. A metagenomic insight into freshwater methane-utilizing communities and evidence for cooperation between the Methylococcaceae and the Methylophilaceae // PeerJ. 2013. V. 1:e23.
Bolyen E., Rideout J.R., Dillon M.R., Bokulich N.A., Abnet C.C., Al-Ghalith G.A., Alexander H., Alm E.J., Arumugam M., Asnicar F. et al. Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2 // Nat. Biotechnol. 2019. V. 37. P. 852–857.
Callahan B.J., McMurdie P.J., Rosen M.J., Han A.W., Johnson A.J.A., Holmes S.P. DADA2: High-resolution sample inference from Illumina amplicon data // Nat. Methods. 2016. V. 13. P. 581–583.
Cébron A., Bodrossy L., Chen Y., Singer A.C., Thompson I.P., Prosser J.I., Murrell J.C. Identity of active methanotrophs in landfill cover soil as revealed by DNA-stable isotope probing // FEMS Microbiol. Ecol. 2007. V. 62. P. 12–23.
Conrad R. The global methane cycle: Recent advances in understanding the microbial processes involved // Environ. Microbiol. Rep. 2009. V. 1. P. 285–292.
Conrado R.J., Gonzalez R. Envisioning the bioconversion of methane to liquid fuels // Science. 2014. V. 343. P. 621–623.
Costello A.M., Auman A.J., Macalady J.L., Scow K.M., Lidstrom M.E. Estimation of methanotroph abundance in a freshwater lake sediment // Environ. Microbiol. 2002. V. 4. P. 443–450.
Coty V. A critical review of the utilization of methane // Biotech. Bioeng. Symp. 1969. V. 1. P. 105–117.
Danilova O.V., Kulichevskaya I.S., Rozova O.N., Detkova E.N., Bodelier P.L.E., Trotsenko Y.A., Dedysh S.N. Methylomonas paludis sp. nov., the first acidtolerant member of the genus Methylomonas, from an acidic wetland // Int. J. Syst. Evol. Microbiol. 2013. V. 63. № 6. P. 2282–2289.
Danilova O.V., Belova S.E., Gagarinova I.V., Dedysh S.N. Microbial community composition and methanotroph diversity of a subarctic wetland in Russia // Microbiology (Moscow). 2016. V. 85. P. 583–591.
Dedysh S.N. Exploring methanotroph diversity in acidic northern wetlands: Molecular and cultivation-based studies // Microbiology (Moscow). 2009. V. 78. P. 655–669.
Dedysh S.N., Knief C. Diversity and phylogeny of described aerobic methanotrophs // Methane Biocatalysis: Paving the Way to Sustainability / Ed. Kalyuzhnaya M., Xing X. Springer, Cham, 2018. P. 17–42.
Deutzmann J.S., Hoppert M., Schink B. Characterization and phylogeny of a novel methanotroph, Methyloglobulus morosus gen. nov., spec. nov // Syst. Appl. Microbiol. 2014. V. 37. P. 165–169.
Deutzmann J.S., Wörner S., Schink B. Activity and diversity of methanotrophic bacteria at methane seeps in eastern Lake Constance sediments // Appl. Environ. Microbiol. 2011. V. 77. P. 2573–2581.
Ganendra G., Wang J., Ramos J.A., Derluyn H., Rahier H., Cnudde V., Ho A., Boon N. Biogenic concrete protection driven by the formate oxidation by Methylocystis parvus OBBP. // Front. Microbiol. 2015. V. 6. P. 786.
Guo W., Li D., He R., Wu M., Chen W., Gao F., Zhang Z., Yao Y., Yu L., Chen S. Synthesizing value-added products from methane by a new Methylomonas // J. Appl. Microbiol. 2017. V. 123. P. 1214–1227.
van der Ha D., Nachtergaele L., Kerckhof F.M., Rameiyanti D., Bossier P., Verstraete W., Boon N. Conversion of biogas to bioproducts by algae and methane oxidizing bacteria // Environ. Sci. Technol. 2012. V. 46. P. 13425–13431.
Hanson R., Hanson T. Methanotrophic bacteria // Microbiol. Rev. 1996. V. 60. P. 439–471.
Hirayama H., Abe M., Miyazaki M., Nunoura T., Furushima Y., Yamamoto H., Takai K. Methylomarinovum caldicuralii gen. nov., sp. nov., a moderately thermophilic methanotroph isolated from a shallow submarine hydrothermal system, and proposal of the family Methylothermaceae fam. nov. // Int. J. Syst. Evol. Microbiol. 2014. V. 64. P. 989–999.
Hoefman S., van der Ha D., De Vos P., Boon N., Heylen K. Miniaturized extinction culturing is the preferred strategy for rapid isolation of fast-growing methane-oxidizing bacteria // Microb. Biotechnol. 2012. V. 5. P. 368–378.
Houghton K.M., Stewart L.C. Temperature-gradient incubation isolates multiple competitive species from a single environmental sample // Access Microbiol. 2020. V. 2. P. 1–9.
Joergensen L., Degn H. Growth rate and methane affinity of a turbidostatic and oxystatic continuous culture of Methylococcuscapsulatus (Bath) // Biotechnol. Lett. 1987. V. 9. P. 71–76.
Kalyuzhnaya M.G., Kumaresan D., Heimann K., Caetano N.S., Visvanathan C., Parthiba Karthikeyan O. Editorial: Methane: A bioresource for fuel and biomolecules // Front. E-nviron. Sci. 2020. V. 8. P. 8–10.
Khalifa A., Gyu Lee C., Ogiso T., Ueno C., Dianou D., Demachi T., Katayama A., Asakawa S. Methylomagnum ishizawai gen. nov., sp. nov., a mesophilic type I methanotroph isolated from rice rhizosphere // Int. J. Syst. Evol. Microbiol. 2015. V. 65. P. 3527–3534.
Khmelenina V.N., Murrell J.C., Smith T.J., Trotsenko Y.A. Physiology and biochemistry of the aerobic methanotrophs // Aerobic Utilization of Hydrocarbons, Oils and Lipids. Handbook of Hydrocarbon and Lipid Microbiology / Ed. F. Rojo. Springer, Cham, 2018. P. 1–25.
Kim J., Kim D.H.D., Yoon S. Rapid isolation of fast-growing methanotrophs from environmental samples using continuous cultivation with gradually increased dilution rates // Appl. Microbiol. Biotechnol. 2018. V. 102. P. 5707–5715.
Knief C. Diversity and habitat preferences of cultivated and uncultivated methanotrophic bacteria evaluated based on pmoA as molecular marker // Front. Microbiol. 2015. V. 6. Art. 1346.
Kumaresan D., Abell G.C.J., Bodrossy L., Stralis-Pavese N., Murrell J.C. Spatial and temporal diversity of methanotrophs in a landfill cover soil are differentially related to soil abiotic factors // Environ. Microbiol. Rep. 2009. V. 1. P. 398–407.
Ma K., Conrad R., Lu Y. Dry/wet cycles change the activity and population dynamics of methanotrophs in rice field soil // Appl. Environ. Microbiol. 2013. V. 79. P. 4932–4939.
Morinaga Y., Yamanaka S., Otsuka S.I., Hirose Y. Characteristics of a newly isolated methane oxidizing bacterium, Methylomonas flagellata nov. sp. // Agric. Biol. Chem. 1976. V. 40. P. 1539–1545.
Murrell J.C. The aerobic methane oxidizing bacteria (Methanotrophs) BT // Handbook of Hydrocarbon and Lipid Microbiology / Ed. Timmis K.N. Springer, Heidelberg, 2010. P. 1953–1966.
Pieja A.J., Sundstrom E.R., Criddle C.S. Poly-3-hydroxybutyrate metabolism in the type II methanotroph Methylocystis parvus OBBP // Appl. Environ. Microbiol. 2011. V. 77. P. 6012–6019.
Quast C., Pruesse E., Yilmaz P., Gerken J., Schweer T., Yarza P., Peplies J., Glöckner F.O. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools // Nucleic Acids Res. 2013. V. 41. P. 590–596.
Rahalkar M., Deutzmann J., Schink B., Bussmann I. Abundance and activity of methanotrophic bacteria in littoral and profundal sediments of lake constance (Germany) // Appl. Environ. Microbiol. 2009. V. 75. P. 119–126.
Ritala A., Häkkinen S.T., Toivari M., Wiebe M.G. Single cell protein-state-of-the-art, industrial landscape and patents 2001–2016 // Front. Microbiol. 2017. V. 8. P. 2009.
Rognes T., Flouri T., Nichols B., Quince C., Mahé F. VSEARCH: A versatile open source tool for metagenomics // PeerJ. 2016. V. 4. e2584.
Rostkowski K.H., Pfluger A.R., Criddle C.S. Stoichiometry and kinetics of the PHB-producing type II methanotrophs Methylosinus trichosporium OB3b and Methylocystis parvus OBBP // Bioresour. Technol. 2013. V. 132. P. 71–77.
Rumah B.L., Stead C.E., Claxton Stevens B.H., Minton N.P., Grosse-Honebrink A., Zhang Y. Isolation and characterisation of Methylocystis spp. for poly-3-hydroxybutyrate production using waste methane feedstocks // AMB Express. 2021. V. 11. Art. 6.
Strong P.J., Kalyuzhnaya M., Silverman J., Clarke W.P. A methanotroph-based biorefinery: Potential scenarios for generating multiple products from a single fermentation // Bioresour. Technol. 2016. V. 215. P. 314–323.
Strong P.J., Xie S., Clarke W.P. Methane as a resource: Can the methanotrophs add value? // Environ. Sci. Technol. 2015. V. 49. P. 4001–4018.
Sundstrom E.R., Criddle C.S. Optimization of methanotrophic growth and production of poly(3-hydroxybutyrate) in a high-throughput microbioreactor system // Appl. E-nviron. Microbiol. 2015. V. 81. P. 4767–4773.
Trotsenko Y.A., Murrell J.C. Metabolic aspects of aerobic obligate methanotrophy // Adv. Appl. Microbiol. 2008. V. 63. P. 183–229.
Vaksmaa A., van Alen T.A., Ettwig K.F., Lupotto E., Valè G., Jetten M.S.M., Lüke C. Stratification of diversity and activity of methanogenic and methanotrophic microorganisms in a nitrogen-fertilized Italian paddy soil // Front. Micr-obiol. 2017. V. 8. P. 1–15.
Weisburg W.G., Barns S.M., Pelletier D.A., Lane D.J. 16S ribosomal DNA amplification for phylogenetic study // J. Bacteriol. 1991. V. 173. P. 697–703.
Yilmaz P., Parfrey L.W., Yarza P., Gerken J., Pruesse E., Quast C., Schweer T., Peplies J., Ludwig W., Glöckner F.O. The SILVA and “all-species Living Tree Project (LTP)” taxonomic frameworks // Nucleic Acids Res. 2014. V. 42. P. 643–648.
Дополнительные материалы отсутствуют.