Микробиология, 2022, T. 91, № 6, стр. 685-694
Ацидобактерии в низинных болотах: филогенетическое разнообразие и анализ геномов ключевых представителей
С. Н. Дедыш a, А. А. Иванова a, Ш. А. Бегматов b, А. В. Белецкий b, А. Л. Ракитин b, А. В. Марданов b, Д. А. Филиппов c, Н. В. Равин b, *
a Институт микробиологии им. С.Н. Виноградского, ФИЦ Биотехнологии РАН
199071 Москва, Россия
b Институт биоинженерии им. К.Г. Скрябина, ФИЦ Биотехнологии РАН
199071 Москва, Россия
c Институт биологии внутренних вод им. И.Д. Папанина РАН
152742 Борок, Россия
* E-mail: nravin@mail.ru
Поступила в редакцию 31.05.2022
После доработки 07.06.2022
Принята к публикации 09.06.2022
- EDN: OHPZJB
- DOI: 10.31857/S0026365622600511
Аннотация
Бактерии филума Acidobacteriota являются одной из основных групп прокариот в почвах и торфяниках. Особенно многочисленны ацидобактерии в кислых верховых болотах, где доминируют представители хорошо изученного класса Acidobacteriia. Разнообразие и метаболический потенциал ацидобактерий, населяющих низинные болота с нейтральным рН, исследованы меньше. Мы провели сравнительный анализ состава сообществ ацидобактерий в четырех верховых и шести низинных болотах Вологодской области. Фрагменты последовательностей генов 16S рРНК ацидобактерий составляли 30–42 и 7–22% всех прочтений, полученных из образцов торфа верховых и низинных болот, соответственно. Ацидобактерии верховых болот были представлены порядками Acidobacteriales и Bryobacterales класса Acidobacteriia, тогда как в торфе низинных болот преобладали классы Vicinamibacteria и Blastocatellia. Из метагеномов торфа низинных болот были собраны геномы четырех представителей Blastocatellia (семейство Pyrinomonadaceae, 2 генома), Vicinamibacteria (семейство UBA2999) и Thermoanaerobaculia (порядок UBA5066). Анализ геномов выявил у всех четырех микроорганизмов ключевые пути аэробного гетеротрофного метаболизма, в том числе Эмбдена‒Мейергофа и глюконеогенеза, неокислительный этап пентозофосфатного пути, цикл трикарбоновых кислот, а также электрон-транспортную цепь аэробного дыхания, включающую терминальные цитохром с-оксидазы. Все геномы кодируют молибдоптериновые оксидоредуктазы CISM семейства и мультигемовые цитохромы с, что указывает на возможность диссимиляционного восстановления соединений серы и Fe(III) в анаэробных условиях. Вероятными субстратами для роста могут являться аминокислоты, пептиды и жирные кислоты. Представители Pyrinomonadaceae также могут использовать углеводы, включая полисахариды и, вероятно, участвуют в деструкции различных биополимеров в торфяных болотах. Представители Thermoanaerobaculia и Vicinamibacteria не имеют генов секретируемых гликозил-гидролаз и могут использовать ограниченный спектр простых сахаров. Геном представителя Vicinamibacteria содержал набор генов, кодирующих ранее не описанные у ацидобактерий бактериальные микрокомпартменты (метаболосомы), вероятно, участвующие в метаболизме L-рамнозы.
Представители филума Acidobacteria, недавно переименованного в филум Acidobacteriota (Oren, Garrity, 2021) составляют одну из численно доминирующих групп бактерий в почвах и торфяниках (Ludwig et al., 1997; Janssen, 2006; Jones et al., 2009; Lauber et al., 2009; Kielak et al., 2016; Dedysh, Oren, 2020; Ivanova et al., 2020a, 2020b). По данным молекулярного профилирования состава микробных сообществ почв по генам 16S рРНК, доля ацидобактерий обычно составляет от 5 до 50% всего пула прочтений ПЦР-фрагментов этих генов (Janssen, 2006; Lee et al., 2008; Lauber et al., 2009; Foesel et al., 2014). Знания о функциональной роли ацидобактерий в почвенных экосистемах включают сведения о деструкции различных биополимеров и участии в глобальных циклах углерода, железа и водорода (Kielak et al., 2016; Eichorst et al., 2018), однако этот перечень далеко не полон, так как спектр культивируемых ацидобактерий остается ограниченным. Известное разнообразие ацидобактерий составляет 26 филогенетических подгрупп (Barns et al., 2007), которые относятся к 15 классам (Dedysh, Yilmaz, 2018). Лишь пять из этих классов ‒ Acidobacteriia, Blastocatellia, Holophagae, Vicinamibacteria и Thermoanae-robaculia ‒ содержат таксономически охарактеризованных представителей (Dedysh, Yilmaz, 2018). Подавляющее большинство полученных в культурах и охарактеризованных ацидобактерий принадлежат к классу Acidobacteriia, который объединяет ацидофильных и ацидотолерантных, аэробных и анаэробных хемоорганотрофов, использующих широкий спектр моно-, ди- и полисахаридов, обладающих гидролитическим потенциалом и населяющих преимущественно кислые наземные местообитания (Dedysh, Oren, 2020).
Болота являются одной из экосистем, обильно колонизованных ацидобактериями. Их доля в составе микробных сообществ особенно высока в кислых верховых болотах (Dedysh et al., 2006; Dedysh, 2011; Ivanova et al., 2020a), а доминирующие популяции представлены ацидобактериями порядков Acidobacteriales и Bryobacterales (Ivanova et al., 2020a). Пул охарактеризованных представителей этих порядков довольно велик. Он включает бактерии родов Granulicella (Pankratov, Dedysh, 2010), способных к деструкции ряда полисахаридов растительного происхождения, представителей рода Bryobacter, утилизирующих специфические продукты разложения фитомассы сфагновых мхов (Kulichevskaya et al., 2010), целлюлолитических и хитинолитических ацидобактерий родов Telmatobacter (Pankratov et al., 2012) и Acidisarcina (Belova et al., 2018) соответственно, а также ряда других родов этих порядков. В противоположность верховым болотам, состав и функциональный потенциал ацидобактерий, населяющих низинные болота с нейтральной реакцией среды, изучены слабо. Как было отмечено в ранее проведенном анализе микробных сообществ двух низинных болот, в составе ацидобактерий преобладают представители класса Blastocatellia и ряда некультивируемых филогенетических подгрупп этого филума (Ivanova et al., 2020a).
Настоящее исследование было предпринято с целью более детального анализа состава популяций ацидобактерий в низинных болотах. Мы расширили спектр болотных экосистем и провели сравнение сообществ ацидобактерий на примере четырех верховых и шести низинных болот Вологодской области. Мы также проанализировали метагеномы двух низинных болот и собрали геномы представителей ацидобактерий, типичных для этих экосистем, что позволило получить данные о метаболическом потенциале этих микроорганизмов.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объекты исследования и отбор образцов. Образцы торфа для исследования были отобраны на территории Вологодской области из четырех верховых болот (Барское и Алексеевское – южная подзона тайги, Шиченгское и Пиявочное – средняя подзона тайги) и шести низинных болот (Родионское – южная подзона тайги, Шиченгское, Пиявочное, Илекса, Поврека и Чарозерское – средняя подзона тайги). Координаты точек отбора образцов и основные характеристики исследованных торфяных болот приведены в табл. 1.
Таблица 1.
Расположение сайтов отбора образцов торфа и основные характеристики болот, исследованных в настоящей работе
| Характеристики | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| координаты | pH | TOC, % | Nобщ, % | сульфат, мг/л | Fe, ppm | Ca, ppm | Mg, ppm | P, ppm | ||
| Верховые болота* | 1 | 59°56′56′′ N 41°16′59′′ E | 4.3 | 88.5 | 0.605 | 172 | 343 | 3522 | 634 | 614 |
| 2 | 60°46′29′′ N 36°49′35′′ E |
3.7 | 85.1 | 0.923 | 220 | 1347 | 4190 | 682 | 791 | |
| 3 | 59°27′10′′ N 40°30′45′′ E | 4.3 | 88 | 0.685 | 211 | 662 | 4191 | 905 | 721 | |
| 4 | 59°22′33′′ N 39°59′26′′ E | 4.1 | 81.5 | 1.16 | 200 | 5306 | 3765 | 816 | 1020 | |
| Низинные болота** | 1 | 59°56′31′′ N 41°15′53′′ E | 7.4 | 73.6 | 2.31 | 202 | 9387 | 29 834 | 2575 | 1179 |
| 2 | 60°46′08′′ N 36°49′30′′ E | 6.9 | 71.6 | 1.65 | 222 | 16 344 | 27 373 | 1078 | 1305 | |
| 3 | 59°47′08′′ N 37°52′08′′ E | 7.6 | 41.8 | 1.06 | 186 | 106 966 | 32 196 | 1599 | 8920 | |
| 4 | 61°08′18′′ N 36°33′27′′ E | 6.9 | 83.2 | 2.55 | 230 | 3455 | 15 968 | 2583 | 1049 | |
| 5 | 61°07′16′′ N 36°33′21′′ E | 6.5 | 48.6 | 1.51 | 607 | 19 264 | 8494 | 2665 | 1192 | |
| 6 | 60°30′42′′ N 38°38′59′′ E | 7.1 | 66.2 | 2.4 | 188 | 5333 | 31 193 | 2695 | 985 | |
Молекулярный анализ филогенетического разнообразия ацидобактерий. Анализ состава сообществ прокариот в образцах торфа путем экстракции ДНК и секвенирования пула ПЦР-амплифицированных фрагментов гена 16S рРНК был проведен в ранее выполненном исследовании (Ivanova et al., 2020a). Пул полученных в этом исследовании последовательностей фрагментов гена 16S рРНК, депонированный в GenBank под номером PRJNA610704, был использован в настоящей работе для анализа филогенетического разнообразия болотных ацидобактерий. Биоинформатический анализ полученных последовательностей проводили с помощью программного пакета QIIME v. 2019.10 (Caporaso et al., 2010). Таксономическую классификацию последовательностей осуществляли согласно базе данных Silva v.138 (Pruesse et al., 2007; Quast et al., 2013) с уровнем сходства 80%. Денойзинг и выбраковку химерных последовательностей проводили с помощью программы DADA2 (Callahan et al., 2016).
Секвенирование метагеномов, сборка и анализ геномов ацидобактерий. Препараты суммарной ДНК из образцов торфа низинных болот Шиченгское и Чарозерское выделяли с помощью набора DNeasy PowerSoil Kit (“Qiagen”). Полученные препараты секвенировали с использованием платформы Illumina HiSeq2500 в соответствии с инструкциями производителя (“Illumina”). В результате секвенирования TruSeq библиотек ДНК (парные чтения 2 × 150 нт.) было получено 92686766 (27.8 млрд нт.) и 125441212 (37.6 млрд нт.) пар чтений для образцов из болот Шиченгское и Чарозерское соответственно. Удаление адаптеров и низкокачественных последовательностей (Q < 30) проводили с помощью Cutadapt v. 1.17 (Martin, 2011) и Sickle v.1.33 (https://github.com/najoshi/sickle) соответственно. Чтения были собраны de novo в контиги с использованием программы MEGAHIT v.1.2.9 (Li et al., 2015).
Полученные контиги длиной более 1500 нт. были кластеризованы в MAG (metagenome-assembled genomes) с использованием MetaBAT v. 2.15 (Kang et al., 2015). Собранные MAG были таксономически идентифицированы с использованием Genome Taxonomy Database Toolkit (GTDB-Tk) v.1.3.0 (Chaumeil et al., 2020) и базы данных Genome Taxonomy database (GTDB) (Parks et al., 2022). Параметры полноты и загрязнения (избыточности) MAG оценивали по наличию набора однокопийных маркерных генов с помощью программы CheckM 1.1.3 (Parks et al., 2015).
Поиск генов и аннотацию геномов проводили с помощью сервера RAST 2.0 (Brettin et al., 2015) с последующей коррекцией аннотации путем сравнения предсказанных последовательностей белков с базами данных NCBI. N-концевые сигнальные пептиды были предсказаны с помощью Signal P v.5.0 (Almagro Armenteros et al., 2019); для классификации и анализа гидрогеназ использовали HydDB сервер (Søndergaard et al., 2016).
Последовательности MAG ацидобактерий депонированы в NCBI GenBank под номерами JAMQPH000000000 (геном SH2), JAMQPI000000000 (геном CH3), JAMQPJ000000000 (геном CH5) и JAMQPK000000000 (геном CH22).
РЕЗУЛЬТАТЫ
Филогенетическое разнообразие ацидобактерий в верховых и низинных болотах. В общей сложности, из исследованных образцов торфа верховых и низинных болот было получено 2 174 164 фрагмента (средняя длина ~440 п.н.) последовательностей гена 16S рРНК. После процедуры фильтрации по качеству и длине прочтений и выбраковке химер были отсортированы 1 024 783 последовательности. Пул фрагментов представителей филы Acidobacteriota включал 233 178 последовательностей, что составляло 30–42% всех прочтений, полученных из торфа верховых болот, и 7–22% прочтений, полученных из торфа низинных болот.
Сообщество ацидобактерий верховых болот почти целиком (>99%) состояло из представителей класса Acidobacteriia (рис. 1). В пределах этого класса преобладали бактерии порядков Acidobacteriales (от 55.4% общего числа всех последовательностей ацидобактерий в болоте Барское до 64.9% в болоте Алексеевское) и Bryobacterales (от 26.3% общего числа всех прочтений в болоте Алексеевское до 32.9% в болоте Барское). Третьей численно значимой группой ацидобактерий являлась филогенетическая подгруппа 2 (Subdivision 2), также относящаяся к классу Acidobacteriia, но пока не имеющая культивируемых представителей. Доля фрагментов гена 16S рРНК ацидобактерий подгруппы 2 составляла от 2.5% всех прочтений в болоте Пиявочное до 10.6% всех прочтений в болоте Шиченгское. Среди малочисленных групп ацидобактерий в верховых болотах были выявлены представители подгрупп 12 и 13 (рис. 1). Наиболее многочисленные группы последовательностей гена 16S рРНК, которые могли быть классифицированы на уровне рода, принадлежали представителям Occallatibacter (3.3‒9.7%), Bryobacter (8.5‒17.3%) и Candidatus Solibacter (13.4‒22.9%).
Рис. 1.
Состав микробного сообщества ацидобактерий в торфе верховых (I) и низинных (II) болот согласно результатам анализа генов 16S рРНК. Цифрами обозначены верховые болота: 1 ‒ Шиченгское, 2 ‒ Алексеевское, 3 ‒ Барское, 4 ‒ Пиявочное; низинные болота: 5 ‒ Шиченгское, 6 ‒ Родионское, 7 ‒ Пиявочное, 8 ‒ Илекса, 9 ‒ Поврека, 10 ‒ Чарозерское. Результаты анализа разнообразия представлены на уровне порядков и классов. Представители каждого класса ацидобактерий обозначены в одной цветовой гамме. Данные представляют собой среднее значение, полученное из 5 повторностей.
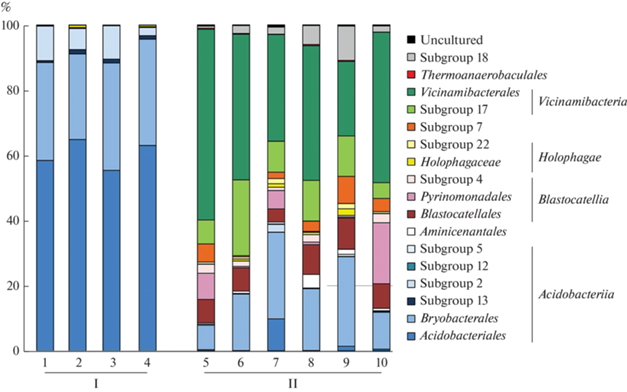
Ацидобактериальные сообщества низинных и верховых болот коренным образом различались (рис. 1 и 2). В низинных болотах численно доминировали представители класса Vicinamibacteria, относящиеся к порядку Vicinamibacteriales (от 23.1% всех последовательностей ацидобактерий в болоте Поврека до 58.9% в болоте Шиченгское) и некультивируемой филогенетической подгруппе 17 (Subdivision 17) (от 4.9% всех последовательностей ацидобактерий в болоте Чарозерское до 23.4% в болоте Родионское). Вторым по численности был класс Blastocatellia, представители которого составляли от 9.2 до 29.0% всех ацидобактерий. В пределах этого класса доминировали бактерии семейства Blastocatellaceae (от 4.1% полученных фрагментов ацидобактерий в болоте Пиявочное до 9.4% в болоте Поврека) и Pyrinomonadaceae (от 0.3% в болоте Поврека до 18.8% в болоте Чарозерское) (рис. 2). Представители класса Acidobacteriia также были выявлены в составе сообществ ацидобактерий низинных болот, однако их доля была значительно ниже, чем в верховых болотах и составляла от 8.6% в болоте Шиченгское до 39.5% в болоте Пиявочное. В пределах этого класса наиболее представленными группами были ацидобактерии рода Bryobacter (2.4‒12.2% всех последовательностей ацидобактерий) и Candidatus Solibacter (2.9‒12.4%). Несколько минорных групп ацидобактерий, которые были обнаружены в низинных болотах, но отсутствовали в верховых, составляли представители класса Thermoanaerobaculia, некультивируемых филогенетических подгрупп 7, 18, 22 и порядка “Aminicenantales”.
Рис. 2.
Состав микробного сообщества ацидобактерий в торфе низинных болот согласно результатам анализа генов 16S рРНК. Цифрами обозначены следующие болота: 5 ‒Шиченгское, 6 ‒ Родионское, 7 ‒ Пиявочное, 8 ‒ Илекса, 9 ‒ Поврека, 10 ‒ Чарозерское. Результаты анализа разнообразия представлены на уровне родов и таксонов высокого порядка. Представители каждого класса ацидобактерий обозначены в одной цветовой гамме. Данные представляют собой среднее значение, полученное из 5 повторностей.
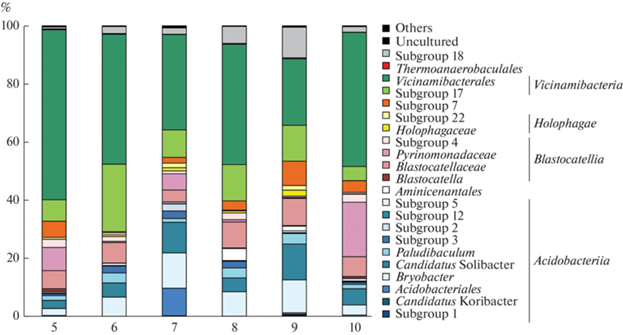
В целом, доля фрагментов гена 16S рРНК ацидобактерий, которые могли быть классифицированы на уровне рода, составила 38‒48% в верховых болотах и лишь 8‒28% в низинных болотах. Таким образом, подавляющее большинство ацидобактерий в низинных болотах были представлены неизвестными таксонами.
Определение последовательностей геномов некультивируемых ацидобактерий. Для получения MAG членов микробного сообщества мы просеквенировали метагеном образцов торфа из низинных болот Шиченгское и Чарозерское. Анализ таксономической принадлежности полученных MAG показал, что четыре из них, один из Шиченгского и три из Чарозерского болота, относились к ацидобактериям (табл. 2).
Таблица 2.
Основные характеристики геномов ацидобактерий, полученных из метагеномов торфа низинных болот
| Геном | Полнота, % | Загрязнение, % | Размер MAG, н.т. | Число контигов | Таксономическая классификация по GTDB |
|---|---|---|---|---|---|
| Болото Шиченгское | |||||
| SH2 | 91.1 | 5.3 | 7 378 535 | 909 | p__Acidobacteriota; c__Blastocatellia; o__Pyrinomonadales; f__Pyrinomonadaceae; g__PSRF01 |
| Болото Чарозерское | |||||
| CH3 | 91.0 | 2.9 | 6 905 000 | 735 | p__Acidobacteriota; c__Blastocatellia; o__Pyrinomonadales; f__Pyrinomonadaceae |
| CH5 | 88.7 | 4.7 | 3 735 478 | 687 | p__Acidobacteriota; c__Thermoanaerobaculia; o__UBA5066; f__UBA5066; g__Fen-183 |
| CH22 | 89.7 | 4.4 | 3 781 114 | 242 | p__Acidobacteriota; c__Vicinamibacteria; o__Vicinamibacterales; f__UBA2999 |
Два полученных генома, SH2 и СН3, относились к семейству Pyrinomonadaceae класса Blastocatellia, в котором представляли некультивируемый род-кандидат PSRF01 и новую линию уровня рода соответственно. Два других генома были отнесены к некультивируемому порядку-кандидату UBA5066 класса Thermoanaerobaculia (CH5) и семейству UBA2999 класса Vicinamibacteria (CH22).
Присутствие в геномах генов, кодирующих белки MreBCD и RodA, указывает на то, что клетки всех четырех ацидобактерий имеют палочковидную форму. Во всех геномах отсутствовали гены флагеллярного аппарата, но содержался набор генов пилей IV типа, наличие которых указывает на “ползающую” подвижность бактерий и их способность к адгезии к твердым субстратам (Mandlik et al., 2008).
Анализ центральных путей метаболизма новых ацидобактерий. Анализ геномов всех четырех бактерий выявил ключевые пути аэробного гетеротрофного метаболизма, в том числе Эмбдена‒Мейергофа, глюконеогенеза, неокислительный этап пентозофосфатного пути и цикл трикарбоновых кислот. Во всех геномах кодируется электрон-транспортная цепь для аэробного дыхания, включающая терминальные цитохром с-оксидазы. Все четыре генома кодируют оксидазы aa3 типа, а геномы SH2, СН3 и СН22 дополнительно содержат гены оксидазы cbb3 типа. Эти оксидазы различаются по своему сродству к кислороду: в то время как ферменты типа аа3 имеют низкое сродство и обычно используются аэробными микроорганизмами, оксидазы типа cbb3 обычно имеют очень высокое сродство к кислороду, обеспечивая дыхание в микроаэробных условиях (Pitcher et al., 2002). Предсказанная способность ацидобактерий расти в аэробных условиях согласуется с наличием в их геномах супероксиддисмутазы и каталазы, участвующих в защите от активных форм кислорода у аэробов.
Все четыре ацидобактерии обладают генетическими детерминантами, необходимыми и для анаэробного дыхания. Все геномы кодируют молибдоптериновые оксидоредуктазы CISM (complex iron–sulfur molybdoenzyme) семейства (Rothery et al., 2008), гомологичные полисульфид-, тиосульфат- и тетратионатредуктазам. Бактерии SH2 и CH5 также содержат гены цитохром с-зависимой нитритредуктазы, восстанавливающей нитрит до аммония; у SH2 имеется редуктаза закиси азота (N2О), а у СН3 – редуктаза оксида азота (NО). Отметим, что все геномы содержали гены мультигемовых (до 10 гем-связывающих сайтов) цитохромов с, число которых у SH2, CH3, CH5 и CH22 составляло 36, 29, 49 и 16 соответственно. Такие цитохромы c-типа играют ключевую роль в переносе электронов из клетки к внеклеточному акцептору электронов у хорошо изученных грамотрицательных бактерий Shewanella и Geobacter, осуществляющих диссимиляционное восстановление железа Fe(III) (Shi et al., 2007; Richter et al., 2012). Взаимодействию с нерастворимыми минералами Fe(III) также могут способствовать пили типа IV, кодируемые во всех четырех геномах.
Помимо органических субстратов (см. ниже), все анализируемые ацидобактерии могут использовать неорганические доноры электронов для дыхания. Все геномы кодируют СО-дегидрогеназу аэробного типа, а геномы представителей Pyrinomonadaceae (SH2 и CH3) также [NiFe]-гидрогеназы группы 1h, обладающие очень высоким сродством к водороду и способные окислять следовые количества атмосферного Н2 (Greening et al., 2015).
Потенциальные субстраты для роста и экологическая роль. Анализ геномов показал наличие генов транспортеров аминокислот и коротких пептидов, что указывает на способность этих бактерий использовать соответствующие субстраты. Все геномы также кодируют путь β-окисления жирных кислот, которые, вероятно, также могут использоваться в качестве субстратов. Бактерия SH2 кодирует набор ферментов пути утилизации бензоата (бензоил-CoA оксигеназная система), а бактерия CH22 – транспортеры ди- и трикарбоновых кислот (цитрат, фумарат, малат, сукцинат), которые могут использоваться в цикле трикарбоновых кислот.
Представители Pyrinomonadaceae обладают разнообразными возможностями использования углеводных субстратов, о чем свидетельствует наличие в их геномах большого числа гликозил-гидролаз и транспортеров сахаров. Бактерии SH2 и CH5 обладают генетическим потенциалом для утилизации ксилана, в том числе содержат гены эндо-1,4-β-ксиланаз, содержащих N-концевые сигналы секреции и карбогидрат-связывающие домены, гены секретируемых ксилан-1,4-β-ксилозидаз, α- и β-ксилозидаз, а также транспортеров и ферментов пути утилизации ксилозы (ксилозоизомераза и ксилулозокиназа). Наличие транспортеров АВС типа для мальтозы/мальтодекстрина и амилолитических ферментов указывает на возможность использования крахмала и его производных. Также оба генома кодируют пектатлиазы с N-концевыми сигналами секреции, ключевые ферменты, обеспечивающие утилизацию пектина. Особенностью бактерии СН3 является наличие кластера генов, кодирующих эндоглюканазы семейств GH9 и GH48. Некоторые из них помимо сигналов секреции содержат связывающие целлюлозу домены (CBM2 или CBM3). По-видимому, бактерия СН3 также способна использовать целлюлозу. Обе бактерии обладают генетическим потенциалом для утилизации простых сахаров, включая маннозу, галактозу и N-ацетилглюкозамин.
Анализ геномов бактерий СН5 и СН22 не выявил у них генов, кодирующих секретируемые гликозил-гидролазы, которые могли бы обеспечивать гидролиз сложных полисахаридов. Особенностью бактерии СН22 является наличие у нее секретируемой α-L-рамнозидазы, которая может отщеплять терминальные остатки α-L-рамнозы от α-L-рамнозидов, и полного пути метаболизма L‑рамнозы. Импортированная в клетку L-рамноза преобразуется в L-рамнулозу изомеразой, далее происходит ее фосфорилирование рамнулокиназой и расщепление альдолазой на дигидроксиацетонфосфат и лактальдегид. Последующий метаболизм лактальдегида, в результате которого образуется пируват, осуществляется в бактериальных микрокомпартментах-метаболосомах ‒ органеллах, ограниченных белковой оболочкой и предназначенных для метаболизма токсичных для клетки соединений (Chowdhury et al., 2014). Метаболосомы, участвующие в деградации 1,2-пропандиола и этаноламина, были впервые описаны у Salmonella (Bobik et al., 1999; Kofoid et al., 1999). Гены, кодирующие структурные белки метаболосомы, обнаружены в геноме бактерии СН22. Подобные механизмы утилизации рамнозы были ранее обнаружены у планктомицетов Planctomyces limnophilus (Erbilgin et al., 2014) и Humisphaera borealis (Dedysh et al., 2021), но не были описаны для ацидобактерий. Интересно, что геном СН5 также содержит гены структурных компонентов микрокомпартмента, но ферменты утилизации рамнозы у него отсутствуют. Возможно, это связано с неполнотой собранного MAG, или микрокомпартмент у этой бактерии используется для метаболизма других токсичных соединений.
ОБСУЖДЕНИЕ
Сравнительный анализ разнообразия представителей филума Acidobacteriota в четырех верховых и шести низинных болотах Вологодской области подтвердил ранее выявленные отличия состава сообществ ацидобактерий в этих экосистемах (Ivanova et al., 2020a). Ацидобактерии кислых верховых болот были представлены порядками Acidobacteriales и Bryobacterales класса Acidobacteriia, тогда как в торфе нейтральных низинных болот преобладали ацидобактерии классов Vicinamibacteria и Blastocatellia. Составляющие минорную группу ацидобактерий представители класса Thermoanaerobaculia были выявлены только в низинных болотах. Так как охарактеризованное разнообразие и пул доступных последовательностей геномов представителей классов Blastocatellia, Vicinamibacteria и Thermoanaerobaculia крайне ограничены, нами были собраны и проанализированы геномы четырех типичных представителей этих бактерий из низинных болот.
В настоящее время в семействе Pyrinomonadaceae класса Blastocatellia имеется четыре культивируемых представителя ‒ Pyrinomonas methylaliphatogenes, Brevitalea aridisoli, Brevitalea deliciosa и Arenimicrobium luteum (Crowe et al., 2014; Wüst et al., 2016), однако последовательность генома определена только для P. methylaliphatogenes (Lee et al., 2015). Эта бактерия является облигатным аэробным гетеротрофом, способным использовать преимущественно белковые субстраты, но также ряд простых сахаров и ксилан (Crowe et al., 2014). Геном P. methylaliphatogenes имеет почти вдвое меньший размер (3.8 млн нт.), чем геномы СН5 и СН22, но кодирует большой набор генов гликозил-гидролаз, в том числе эндо-1,4-β-ксиланазу и целлюлазу семейства GH5 (Lee et al., 2015). B. aridisoli, B. deliciosa и A. luteum также являются аэробными гетеротрофами, растут преимущественно на белковых субстратах, но могут использовать некоторые простые сахара. Штамм B. aridisoli Ac_11_E3T также может расти на ксилане и целлюлозе (Wüst et al., 2016). Таким образом, как культивируемые представители Pyrinomonadaceae, так и бактерии СН5 и СН22, по-видимому, используют широкий спектр субстратов, включающий белки, простые сахара и некоторые полисахариды.
В классе Thermoanaerobaculia имеется лишь один культивируемый представитель, Thermoanaerobaculum aquaticum, ‒ выделенная из горячего источника строго анаэробная бактерия, осуществляющая бродильный тип метаболизма на пирувате и белковых субстратах, также способная к диссимиляционному восстановлению Fe(III) и Mn(IV) (Losey et al., 2013). Другой представитель этого класса, “Candidatus Sulfomarinibacter”, описан на основе MAG, полученного из метагенома морских осадков (Flieder et al., 2021). Анализ этого генома показал возможность как аэробного, так и анаэробного (с оксидом азота, оксидами металлов и окисленными соединениями серы) дыхания, и использование в качестве субстратов белков и целлюлозы (Flieder et al., 2021). По наличию респираторных путей бактерия СН5 ближе к “Ca. Sulfomarinibacter”, а общим свойством всех вышеуказанных Thermoanaerobaculia, по-видимому, является способность к диссимиляционному восстановлению железа и узкий спектр используемых субстратов.
В классе Vicinamibacteria имеется два культивируемых вида, Vicinamibacter silvestris (Huber et al., 2016) и Luteitalea pratensis (Vieira et al., 2017), геном второго был просеквенирован (Huang et al., 2016). Обе ацидобактерии были выделены из почв и являются аэробными хемоорганотрофами, использующими различные белковые субстраты и простые сахара, причем геном L. pratensis содержит более ста генов гликозил-гидролаз и пептидаз. Следует отметить потенциальную способность V. silvestris расти на рамнозе, что было предсказано и для бактерии СН22. Возможно, способность к утилизации рамнозы и образованию необходимых для этого внутриклеточных микрокомпартментов-метаболосом распространена у представителей Vicinami-bacteria и некоторых других ацидобактерий.
Выполненный в настоящей работе анализ геномов четырех представителей ацидобактерий из низинных болот позволил получить данные о метаболическом потенциале этих микроорганизмов, которые проясняют их функциональную роль в этих экосистемах и могут быть использованы для оптимизации сред и подходов для культивирования этих бактерий.
Список литературы
Almagro Armenteros J.J., Tsirigos K.D., Sønderby C.K., Petersen T.N., Winther O., Brunak S., von Heijne G., Nielsen H. SignalP 5.0 improves signal peptide predictions using deep neural networks // Nat. Biotechnol. 2019. V. 37. P. 420–423.
Barns S.M., Cain E.C., Sommerville L., Kuske C.R. Acidobacteria phylum sequences in uranium-contaminated subsurface sediments greatly expand the known diversity within the phylum // Appl. Environ. Microbiol. 2007. V. 7. P. 3113–3116.
Belova S.E., Ravin N.V., Pankratov T.A., Rakitin A.L., Ivanova A.A., Beletsky A.V., Mardanov A.V., Sinninghe Damsté J.S., Dedysh S.N. Hydrolytic capabilities as a key to environmental success: chitinolytic and cellulolytic Acidobacteria from acidic sub-arctic soils and boreal peatlands // Front. Microbiol. 2018. V. 9. P. 2775.
Bobik T.A., Havemann G.D., Busch R.J., Williams D.S., Aldrich H.C. The propanediol utilization (pdu) operon of Salmonella enterica serovar Typhimurium LT2 includes genes necessary for formation of polyhedral organelles involved in coenzyme B12-dependent 1,2-propanediol degradation // J. Bacteriol. 1999. V. 181. P. 5967–5975.
Brettin T., Davis J.J., Disz T., Edwards R.A., Gerdes S., Olsen G.J., Olson R., Overbeek R., Parrello B., Pusch G.D., Shukla M., Thomason J.A. 3rd, Stevens R., Vonstein V., Wattam A.R., Xia Fangfang. RASTtk: a modular and extensible implementation of the RAST algorithm for building custom annotation pipelines and annotating batches of genomes // Sci. Rep. 2015. V. 5. P. 8365.
Callahan B.J., McMurdie P.J., Rosen M.J., Han A.W., Johnson A.J.A., Holmes S.P. DADA2: High-resolution sample inference from Illumina amplicon data // Nat. Methods. 2016. V. 13. P. 581–583.
Caporaso J., Kuczynski J., Stombaugh J. QIIME allows analysis of high-throughput community sequencing data // Nat. Methods. 2010. V. 7. P. 335–336.
Chaumeil P.-A., Mussig A.J., Hugenholtz P., Parks D.H. GTDB-Tk: a toolkit to classify genomes with the Genome Taxonomy Database // Bioinform. 2020. V. 36. P. 1925–1927.
Chowdhury C., Sinha S., Chun S., Yeates T.O., Bobik T.A. Diverse bacterial microcompartment organelles // Microbiol. Mol. Biol. Rev. 2014. V. 78. P. 438–468.
Crowe M.A., Power J.F., Morgan X.C., Dunfield P.F., Lagutin K., Rijpstra W.I.C., Vyssotski M., Sinninghe Damste J.S., Houghton K.M., Ryan J.L.J., Stott M.B. Pyrinomonas methylaliphatogenes gen. nov. sp. nov. a novel group 4 thermophilic member of the phylum Acidobacteria from geothermal soils // Int. J. Syst. Evol. Microbiol. 2014. V. 64. P. 220–227.
Dedysh S.N. Cultivating uncultured bacteria from northern wetlands: knowledge gained and remaining gaps // Front. Microbiol. 2011. V. 2. P. 184.
Dedysh S.N., Beletsky A.V., Ivanova A.A., Kulichevskaya I.S., Suzina N.E., Philippov D.A., Rakitin A.L., Mardanov A.V., Ravin N.V. Wide distribution of Phycisphaera-like planctomycetes from WD2101 soil group in peatlands and genome analysis of the first cultivated representative // Environ. Microbiol. 2021. V. 23. P. 1510–1526.
Dedysh S.N., Oren A. “Acidobacteriia” // Bergey’s Manual of Systematics of Archaea and Bacteria / Eds Trujillo M.E., Dedysh S., DeVos P., Hedlund B., Kämpfer P., Rainey F.A. and Whitman W.B.). https://doi.org/10.1002/9781118960608.cbm00001.pub2.
Dedysh S.N., Pankratov T.A., Belova S.E., Kulichevskaya I.S., Liesack W. Phylogenetic analysis and in situ identification of bacteria community composition in an acidic Sphagnum peat bog // Appl. Environ. Microbiol. 2006. V. 72. P. 2110–2117.
Dedysh S.N., Yilmaz P. Refining the taxonomic structure of the phylum Acidobacteria // Int. J. Syst. Evol. Microbiol. 2018. V. 68. P. 3796–3806.
Eichorst S.A., Trojan D., Roux S., Herbold C., Rattei T., Woebken D. Genomic insights into the Acidobacteria reveal strategies for their success in terrestrial environments // Environ. Microbiol. 2018. V. 20. P. 1041–1063.
Erbilgin O., McDonald K.L., Kerfeld C.A. Characterization of a planctomycetal organelle: a novel bacterial microcompartment for the aerobic degradation of plant saccharides // Appl. Environ. Microbiol. 2014. V. 80. P. 2193–2205.
Flieder M., Buongiorno J., Herbold C.W., Hausmann B., Rattei T., Lloyd K.G., Loy A., Wasmund K. Novel taxa of Acidobacteriota implicated in seafloor sulfur cycling // ISME J. 2021. V. 15. P. 3159–3180.
Foesel B.U., Nägele V., Naether A., Wüst P.K., Weinert J., Bonkowski M., Lohaus G., Polle A., Alt F., Oelmann Y., Fischer M., Friedrich M.W., Overmann J. Determinants of Acidobacteria activity inferred from the relative abundances of 16S rRNA transcripts in German grassland and forest soils // Environ. Microbiol. 2014. V. 16. P. 658–675.
Greening C., Carere C.R., Rushton-Green R., Harold L.K., Hards K., Taylor M.C., Morales S.E., Stott M.B., Cook G.M. Persistence of the dominant soil phylum Acidobacteria by trace gas scavenging // Proc. Natl. Acad. Sci. USA. 2015. V. 11. P. 10497–10502.
Huang S., Vieira S., Bunk B., Riedel T., Spröer C., Overmann J. First complete genome sequence of a Subdivision 6 Acidobacterium strain // Genome Announc. 2016. V. 4. P. e00469-16.
Huber K.J., Geppert A.M., Wanner G., Fösel B.U., Wüst P.K., Overmann J. The first representative of the globally widespread subdivision 6 Acidobacteria Vicinamibacter silvestris gen. nov. sp. nov. isolated from subtropical savannah soil // Int. J. Syst. Evol. Microbiol. 2016. V. 66. P. 2971–2979.
Ivanova A.A., Beletsky A.V., Rakitin A.L., Kadnikov V.V., Philippov D.A., Mardanov A.V., Ravin N.V., Dedysh S.N. Closely located but totally distinct: highly contrasting prokaryotic diversity patterns in raised bogs and eutrophic fens // Microorganisms. 2020a. V. 8. P. 484.
Ivanova A.A., Zhelezova A.D., Chernov T.I., Dedysh S.N. Linking ecology and systematics of acidobacteria: Distinct habitat preferences of the Acidobacteriia and Blastocatellia in tundra soils // PLoS One. 2020b. V. 15. P. e0230157.
Janssen P.H. Identifying the dominant soil bacterial taxa in libraries of 16S rRNA and 16S rRNA genes // Appl. Environ. Microbiol. 2006. V. 72. P. 1719–1728.
Jones R.T., Robeson M.S., Lauber C.L., Hamady M., Knight R., Fierer N. A comprehensive survey of soil acidobacterial diversity using pyrosequencing and clone library analyses // ISME J. 2009. V. 3. P. 442–453.
Kang D.D., Froula J., Egan R., Wang Z. MetaBAT an efficient tool for accurately reconstructing single genomes from complex microbial communities // PeerJ. 2015. V. 3. P. e1165.
Kielak A.M., Barreto C.C., Kowalchuk G.A., van Veen J.A., Kuramae E.E. The ecology of Acidobacteria: moving beyond genes and genomes // Front. Microbiol. 2016. V. 7. P. 744.
Kofoid E., Rappleye C., Stojiljkovic I., Roth J. The 17-gene ethanolamine (eut) operon of Salmonella typhimurium encodes five homologues of carboxysome shell proteins // J. Bacteriol. 1999. V. 181. P. 5317–5329.
Kulichevskaya I.S., Suzina N.E., Liesack W., Dedysh S.N. Bryobacter aggregatus gen. nov. sp. nov. a peat-inhabiting aerobic chemo-organotroph from subdivision 3 of the Acidobacteria // Int. J. Syst. Evol. Microbiol. 2010. V. 60. P. 301–306.
Lauber C.L., Hamady M., Knight R., Fierer N. Pyrosequencing-based assessment of soil pH as a predictor of soil bacterial community structure at the continental scale // Appl. Environ. Microbiol. 2009. V. 75. P. 5111–5120.
Lee K.C.Y., Morgan X.C., Power J.F., Dunfield P.F., Huttenhower C., Stott M.B. Complete genome sequence of the thermophilic Acidobacteria Pyrinomonas methylaliphatogenes type strain K22T // Stand. Genom. Sci. 2015. V. 14. P. 101.
Lee S.H., Ka J.O., Cho J.C. Members of the phylum Acidobacteria are dominant and metabolically active in rhizosphere soil // FEMS Microbiol. Lett. 2008. V. 285. P. 263–269.
Li D., Liu C.M., Luo R., Sadakane K., Lam T.W. MEG-AHIT: An ultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph // Bioinformatics. 2015. V. 31. P. 1674–1676.
Losey N.A., Stevenson B.S., Busse H.J., Damsté J.S.S., Rijpstra W.I.C., Rudd S., Lawson P.A. Thermoanaerobaculum aquaticum gen. nov. sp. nov. the first cultivated member of Acidobacteria subdivision 23 isolated from a hot spring // Int. J. Syst. Evol. Microbiol. 2013. V. 63. P. 4149–4157.
Ludwig W., Bauer S.H., Bauer M., Held I., Kirchhof G., Schulze R., Huber I., Spring S., Hartmann A., Schleifer K.H. Detection and in situ identification of representatives of a widely distributed new bacterial phylum // FEMS Microbiol. Lett. 1997. V. 153. P. 181–190.
Mandlik A., Swierczynski A., Das A., Ton-That H. Pili in Gram-positive bacteria: assembly involvement in colonization and biofilm development // Trends Microbiol. 2008. V. 16. P. 33–40.
Martin M. Cutadapt removes adapter sequences from high-throughput sequencing reads // EMBnet. J. 2011. V. 17.1. P. 10–12.
Oren A., Garrity G.M. Valid publication of the names of forty-two phyla of prokaryotes // Int. J. Syst. Evol. Microbiol. 2021. V. 71. P. 005056.
Pankratov T.A., Dedysh S.N. Granulicella paludicola gen. nov. sp. nov. Granulicella pectinivorans sp. nov. Granulicella aggregans sp. nov. and Granulicella rosea sp. nov. acidophilic polymer-degrading acidobacteria from Sphagnum peat bogs // Int. J. Syst. Evol. Microbiol. 2010. V. 60. P. 2951–2959.
Pankratov T.A., Kirsanova L.A., Kaparullina E.N., Kevbrin V.V., Dedysh S.N. Telmatobacter bradus gen. nov. sp. nov. a cellulolytic facultative anaerobe from subdivision 1 of the Acidobacteria and emended description of Acidobacterium capsulatum Kishimoto et al. 1991 // Int. J. Syst. Evol. Microbiol. 2012. V. 62. P. 430–437.
Parks D.H., Chuvochina M., Rinke C., Mussig A.J., Chaumeil P.-A., Hugenholtz P. GTDB: an ongoing census of bacterial and archaeal diversity through a phylogenetically consistent rank normalized and complete genome-based taxonomy // Nucl. Acids Res. 2022. V. 50. P. D785–D794.
Parks D.H., Imelfort M., Skennerton C.T., Hugenholtz P., Tyson G.W. CheckM: assessing the quality of microbial genomes recovered from isolates single cells and metagenomes // Genome Res. 2015. V. 25. P. 1043–1055.
Pitcher R.S., Brittain T., Watmough N.J. Cytochrome cbb3 oxidase and bacterial microaerobic metabolism // Proc. Biochem. Soc. Trans. 2002. V. 30. P. 653–658.
Pruesse E., Quast C., Knittel K., Fuchs B.M., Ludwig W., Peplies J., Glöckner F.O. SILVA: a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB // Nucl. Acids Res. 2007. V. 35. P. 7188–7196.
Quast C., Pruesse E., Yilmaz P., Gerken J., Schweer T., Yarza P., Peplies J., Glöckner F.O. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools // Nucl. Acids Res. 2013. V. 41. P. D590–D596.
Richter K., Schicklberger M., Gescher J. Dissimilatory reduction of extracellular electron acceptors in anaerobic respiration // Appl. Environ. Microbiol. 2012. V. 78. P. 913–921.
Rothery R.A., Workun G.J., Weiner J.H. The prokaryotic complex iron–sulfur molybdoenzyme family // Biochim. Biophys. Acta ‒ Biomembr. 2008. V. 1778. P. 1897–1929.
Shi L., Squier T.C., Zachara J.M., Fredrickson J.K. Respiration of metal (hydr)oxides by Shewanella and Geobacter: a key role for multihaem c-type cytochromes. MicroReview // Mol. Microbiol. 2007. V. 65. P. 12–20.
Søndergaard D., Pedersen C.N.S., Greening C. HydDB: A web tool for hydrogenase classification and analysis // Sci. Rep. 2016. V. 6. P. 34212.
Vieira S., Luckner M., Wanner G., Overmann J. Luteitalea pratensis gen. nov. sp. nov. a new member of subdivision 6 Acidobacteria isolated from temperate grassland soil // Int. J. Syst. Evol. Microbiol. 2017. V. 67. P. 1408–1414.
Wüst P.K., Foesel B.U., Geppert A., Huber K.J., Luckner M., Wanner G., Overmann J. Brevitalea aridisoli, B. deliciosa and Arenimicrobium luteum three novel species of Acidobacteria subdivision 4 (class Blastocatellia) isolated from savanna soil and description of the novel family Pyrinomonadaceae // Int. J. Syst. Evol. Microbiol. 2016. V. 66. P. 3355–3366.
Дополнительные материалы отсутствуют.


