Молекулярная биология, 2019, T. 53, № 1, стр. 142-153
Идентификация и характеристика вырожденных и сложных микросателлитов в геномах штаммов Burkholderia pseudomallei
М. Л. Леденева a, *, Г. А. Ткаченко a, И. М. Шпак a
a Волгоградский научно-исследовательский противочумный институт Роспотребнадзора
400131 Волгоград, Россия
* E-mail: volresin@yandex.ru
Поступила в редакцию 02.02.2018
После доработки 17.07.2018
Принята к публикации 17.07.2018
Аннотация
Эволюция микросателлитов (или простых коротких тандемных повторов, SSRs) – комплексный процесс, в результате которого совершенные повторы превращаются в новые структурные элементы генома с еще мало изученной функциональной ролью, такие как вырожденные и сложные (или компаунды) микросателлиты. В результате анализа in silico геномов 10 штаммов Burkholderia pseudomallei обнаружено 215 683 микросателлита, более 98% из которых оказались вырожденными. Плотность микросателлитов в геноме изменялась в диапазоне от 2922.7/млн.п.н. до 3022.6/млн.п.н. В зависимости от штамма 10.20–10.67% обнаруженных тандемных повторов в каждом геноме входили в состав компаундов. Плотность сложных микросателлитов в геноме варьировала в пределах от 144.7/млн.п.н. до 150.6/млн.п.н. Выявленные межштаммовые различия в количественной представленности микросателлитов были обусловлены сопряженным увеличением плотности SSRs с GC-содержанием и обратной взаимосвязью плотности SSRs с размером генома. Аналогичный характер корреляции наблюдали между плотностью SSRs и структурными особенностями (размер и GC-содержание) каждой из хромосом B. pseudomallei; при этом достоверная корреляционная связь плотности SSRs и компаундов выявлена только для хромосомы 2 (r = 0.93, p < 10–3). Характер ассоциации плотности вырожденных и сложных микросателлитов со структурными особенностями каждой из хромосом, наряду с вырожденностью и малой копийностью мотивов в составе большинства выявленных в геномах B. pseudomallei микросателлитов, согласуются с ранее выдвинутой гипотезой о подверженности отрицательному отбору протяженных локусов SSRs. Механизмом такого отбора может быть накопление точечных мутаций, приводящее к прерыванию блока повторов в процессе репликации в результате формирования легко преодолимых вторичных структур, стабилизирующих длину микросателлитного локуса.
ВВЕДЕНИЕ
Микросателлиты, называемые также простыми повторяющимися последовательностями (Simple Sequence Repeats, SSRs), представляют собой тандемно организованные фрагменты ДНК (мотивы, “коровые” единицы), содержащие от 1 до 6 пар нуклеотидов (п.н.) [1]. Доступность полногеномных последовательностей в сочетании с разработанными и программноимплементированными алгоритмами поиска периодических структур в ДНК позволили выявить микросателлиты в геномах всех известных организмов – от вирусов до эукариот [2–6]. Показано, что SSRs распределены в геномах неслучайно [7, 8] и могут влиять не только на транскрипционную активность генов [9], но и выполнять функциональную роль в эволюции их регуляции [10–12]. Однако в большинстве работ изучались главным образом совершенные (невырожденные) повторы. Изменение копий мотива при замене исходных нуклеотидов, а также при вставках и делециях, как единичных, так и нескольких нуклеотидов, ведет к образованию вырожденных и сложных микросателлитов. Вероятно, именно такие, деградировавшие, повторы рекрутируются для различных биологических функций, включая регуляцию транскрипции и репликации, структурные модификации белков и т.д. [13].
Сложные микросателлиты, или компаунды, представляют собой блоки из мотивов одного или разных типов, разделенные не более чем 100 п.н. Компаунды выявлены в геномах человека [14, 15], ряда эукариотических организмов [16, 17], а также некоторых видах бактерий, [18, 19] и вирусов [20–22]. Сложные микросателлиты предположительно участвуют в регуляции экспрессии генов и функциональной активности белков у ряда видов [12, 18]. Компаунды отражают динамичное изменение и свидетельствуют о более сложной эволюции SSRs, чем предложенная некоторыми авторами концепция жизненного цикла микросателлита от его “рождения” до полной деградации (“смерти”) [16].
Изучение вырожденных микросателлитов и компаундов в геномах патогенных микроорганизмов c высоким уровнем SSRs, таких как Burkholderia pseudomallei, возбудителя мелиоидоза (опасного и потенциально смертельного заболевания людей и животных), необходимо для понимания механизмов адаптационной пластичности их геномов. Структурно-функциональная неоднородность генома B. pseudomallei, включающего две двухцепочечные кольцевые хромосомы, определяет необходимость выявления взаимосвязей между количественной представленностью вырожденных и сложных микросателлитов и особенностями структурной организации каждой из хромосом. Цель представленной работы заключалась в идентификации с последующим изучением строения и локализации вырожденных и сложных микросателлитных повторов в геномах B. pseudomallei.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Объектами исследования служили полногеномные последовательности 10 штаммов B. pseudomallei из базы данных GenBank Национального центра биотехнологической информации США (http://www.ncbi.nlm.nih.gov/genome). Выбор штаммов определяли с учетом популяционной структуры вида B. pseudomallei, состоящей из двух субпопуляций разных географических регионов: Австралии и Юго-Восточной Азии. Различия между субпопуляциями определялись не только географическим распределением, но и частотой рекомбинационных событий. Ранее выявлено, что уровень рекомбинации выше в таиландских штаммах по сравнению с австралийскими изолятами [23]. Результатом высокой скорости рекомбинации может быть гомоплазия, приводящая к единообразию сиквенс-типов изолятов B. pseudomallei, несмотря на их генетическую и географическую гетерогенность. В связи с этим в работу были включены австралийские и таиландские штаммы B. pseudomallei разных сиквенс-типов, принадлежащие к разным клональным комплексам, а также штамм из Китая с сиквенс-типом, идентичным одному из таинландских штаммов (табл. 1).
Таблица 1.
Характеристика исследованных штаммов B. pseudomallei
| Штамм* | Источник выделения | Страна выделения | Сиквенс-тип (аллельный профиль) |
Ссылки на опублико-ванные работы |
|---|---|---|---|---|
| K96243 (id11) |
Человек | Таиланд | 10 (1, 1, 13, 1, 1, 1, 1) | [24, 25] |
| 1710b (id3829) |
Кровь человека | Таиланд | 177 (1, 1, 4, 3, 1, 3, 1) | [26] |
| 1026b (id208) |
Кровь человека | Таиланд | 102 (3, 4, 12, 1, 1,4, 1) | [27] |
| 1106a (id3832) |
Человек (внутренний абсцесс) | Таиланд | 70 (3, 4, 11, 3, 5, 4, 6) | [26] |
| BPC006 (id3947) |
Кровь человека | Китай | 70 (3, 4, 11, 3, 5, 4, 6) | [28] |
| NAU20B-16 (id3823) |
Почва | Австралия | 617 (1, 16, 3, 4, 2, 8, 1) | [29] |
| 668 (id244) |
Кровь человека | Австралия | 129 (1, 15, 13, 2, 1, 22, 1) | [26] |
| NCTC 13178 (id3825) |
Человек | Австралия | 286 (15, 2, 13, 2, 20, 19, 1) | [30] |
| NCTC 13179 (id4002) |
Человек (язва кожи) | Австралия | 613 (1, 4, 43, 4, 1, 48, 11) | [31] |
| MSHR305 (id414) |
Человек | Австралия | 36 (1, 7, 14, 7, 1, 12, 11) | [32] |
* В скобках указан номер штамма в базе данных https://pubmlst.org/bpseudomallei.
Определение аллельного профиля и соответствующего ему сиквенс-типа штамма проводили с использованием базы данных https://pubmlst.org/ bpseudomallei [33]. Информация о структурных особенностях хромосом каждого из штаммов приведена в табл. 2.
Таблица 2.
Количественная представленность вырожденных и сложных микросателлитов в геномах B. pseudomallei
| № п/п | Номер хромосомы | Номер доступа GenBank | Размер, п.н. | GC (%) | SSRs | Компаунды | |||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| CM | RACM | CСM | RACCM | CcSSR | cSSR% | Z-критерий | |||||
| 1 | Burkholderia pseudomallei K96243 | ||||||||||
| Chr-1 | NC_006350.1 | 4 074 542 | 67.7 | 11 692 | 2869.5 | 551 | 135.2 | 1146 | 9.8 | ||
| Chr-2 | NC_006351.1 | 3 173 005 | 68.5 | 9926 | 3128.3 | 522 | 164.5 | 1085 | 10.93 | ||
| Геном | 7 247 547 | 68.05 | 21 618 | 2982.8 | 1073 | 148.1 | 2231 | 10.32 | 0.07 | ||
| 2 | Burkholderia pseudomallei 1710b | ||||||||||
| Chr-1 | NC_007434.1 | 4 126 292 | 67.6 | 11 738 | 2844.7 | 539 | 130.6 | 1124 | 9.58 | ||
| Chr-2 | NC_007435.1 | 3 181 762 | 68.5 | 9880 | 3105.2 | 519 | 163.1 | 1086 | 10.99 | ||
| Геном | 7 308 054 | 67.99 | 21 618 | 2958.1 | 1058 | 144.8 | 2210 | 10.22 | –0.08 | ||
| 3 | Burkholderia pseudomallei 1026b | ||||||||||
| Chr-1 | NC_017831.1 | 4 092 668 | 67.9 | 11 823 | 2888.8 | 578 | 141.2 | 1189 | 10.06 | ||
| Chr-2 | NC_017832.1 | 3 138 747 | 68.5 | 9781 | 3116.2 | 511 | 162.8 | 1062 | 10.86 | ||
| Геном | 7 231 415 | 68.16 | 21 604 | 2987.5 | 1089 | 150.6 | 2251 | 10.42 | 0.26 | ||
| 4 | Burkholderia pseudomallei 1106a | ||||||||||
| Chr-1 | NC_009076.1 | 3 988 455 | 68.0 | 11 625 | 2914.7 | 542 | 135.9 | 1132 | 9.74 | ||
| Chr-2 | NC_009078.1 | 3 100 794 | 68.6 | 9790 | 3157.3 | 512 | 165.1 | 1078 | 11.01 | ||
| Геном | 7 089 249 | 68.26 | 21 415 | 3020.8 | 1054 | 148.7 | 2210 | 10.32 | –0.21 | ||
| 5 | Burkholderia pseudomallei NAU20B-16 | ||||||||||
| Chr-1 | CP004003.1 | 4 067 895 | 67.8 | 11 723 | 2881.8 | 564 | 138.6 | 1161 | 9.9 | ||
| Chr-2 | CP004004.1 | 3 245 956 | 68.3 | 9893 | 3047.8 | 494 | 152.2 | 1044 | 10.55 | ||
| Геном | 7 313 851 | 68.02 | 21 616 | 2955.5 | 1058 | 144.7 | 2205 | 10.20 | –0.01 | ||
| 6 | Burkholderia pseudomallei BPC006 | ||||||||||
| Chr-1 | NC_018527.1 | 4 001 777 | 68.0 | 11 673 | 2917.0 | 538 | 134.4 | 1124 | 9.63 | ||
| Chr-2 | NC_018529.1 | 3 153 284 | 68.5 | 9829 | 3117.1 | 514 | 163.0 | 1077 | 10.96 | ||
| Геном | 7 155 061 | 68.22 | 21 502 | 3005.1 | 1052 | 147.0 | 2201 | 10.24 | –0.13 | ||
| 7 | Burkholderia pseudomallei 668 | ||||||||||
| Chr-1 | NC_009074.1 | 3 912 947 | 68.0 | 11 457 | 2928.0 | 537 | 137.2 | 1120 | 9.78 | ||
| Chr-2 | NC_009075.1 | 3 127 456 | 68.6 | 9823 | 3140.9 | 517 | 165.3 | 1088 | 11.08 | ||
| Геном | 7 040 403 | 68.27 | 21 280 | 3022.6 | 1054 | 149.7 | 2208 | 10.38 | –0.18 | ||
| 8 | Burkholderia pseudomallei NCTC 13 178 | ||||||||||
| Chr-1 | CP004001.1 | 4 029 450 | 67.8 | 11 555 | 2867.6 | 586 | 145.4 | 1208 | 10.45 | ||
| Chr-2 | CP004002.1 | 3 361 911 | 68.1 | 10 130 | 3013.2 | 527 | 156.8 | 1106 | 10.92 | ||
| Геном | 7 391 361 | 67.94 | 21 685 | 2933.8 | 1113 | 150.6 | 2314 | 10.67 | 0.07 | ||
| 9 | Burkholderia pseudomallei NCTC 13 179 | ||||||||||
| Chr-1 | NC_022659.1 | 3 997 084 | 67.9 | 11 593 | 2900.4 | 581 | 145.4 | 1194 | 10.3 | ||
| Chr-2 | NC_022658.1 | 3 340 066 | 68.1 | 10 042 | 3006.5 | 500 | 149.7 | 1050 | 10.46 | ||
| Геном | 7 337 150 | 67.99 | 21 635 | 2948.7 | 1081 | 147.3 | 2244 | 10.37 | 0.12 | ||
| 10 | Burkholderia pseudomallei MSHR305 | ||||||||||
| Chr-1 | CP006470.1 | 4 054 155 | 67.8 | 11 690 | 2883.5 | 576 | 142.1 | 1192 | 10.2 | ||
| Chr-2 | CP006469.1 | 3 373 916 | 68.0 | 10 020 | 2969.8 | 512 | 151.8 | 1068 | 10.66 | ||
| Геном | 7 428 071 | 67.89 | 21 710 | 2922.7 | 1088 | 146.5 | 2260 | 10.41 | 0.10 | ||
Примечание. CM – число микросателлитов; RACM – плотность микросателлитов; CСM – число компаундов; RACCM – плотность компаундов; CcSSR – число микросателлитов, входящих в состав компаундов; cSSR% – процентная доля CcSSR от общего числа обнаруженных микросателлитов; Z-критерий – показатель статистической значимости различий между наблюдаемым (CCMobs) и ожидаемым (CCMexp) числом компаундов в каждом геноме.
Поиск вырожденных и сложных микросателлитов. Для идентификации SSRs и компаундов использовали модуль Advanced Mode web-версии программы Imperfect Microsatellite Extractor (IMEx) [34]. Входными данными служили нуклеотидные последовательности в формате FASTA и соответствующие им файлы формата .ptt, содержащие информацию о белоккодирующих регионах. Поиск проводили при следующих параметрах: минимальное число повторов для мононуклеотидных микросателлитов – 12, для динуклеотидных – 6, для тринуклеотидных – 4, для тетра-/пента-/гексануклеотидных – 3; макcимальное расстояние между любыми двумя блоками микросателлитов (dMAX) при поиске компаундов – 10 п.н. Остальные параметры оставляли заданными по умолчанию.
Cтатистический анализ полученных данных. Для описания и статистического исследования выявленных SSRs и компаундов использовали следующие термины: cSSRs – микросателлиты, входящие в состав компаундов; С – сложность компаунда, определяемая числом cSSRs в его составе; CM – число микросателлитов в каждом геноме/хромосоме; CCM – число компаундов в каждом геноме/хромосоме; CcSSR – число cSSRs в каждом геноме/хромосоме; cSSR% – доля микросателлитов, входящих в состав компаундов, от общего числа всех обнаруженных микросателлитов в каждом геноме/хромосоме (cSSR% = CcSSR/CM).
Взаимосвязь таких параметров как размер и GC-содержание (%) генома/хромосомы с количественной представленностью в их составе вырожденных и сложных микросателлитов оценивали с помощью коэффициента корреляции Пирсона (r). С целью обеспечения возможности межштаммового сравнения результатов поиска повторов найденные SSRs и компаунды были стандартизированы относительно их содержания (relative abundance, RA) в каждом геноме/хромосоме: RACM – число микросателлитов на 1 млн.п.н., или плотность микросателлитов; RACCM – число компаундов на 1 млн.п.н., или плотность компаундов.
В качестве показателя статистической значимости различий между наблюдаемым (CCMobs) и ожидаемым (CCMexp) числом компаундов в каждом геноме использовали значение Z-критерия, определяемого по формуле: $\frac{{{\text{O}} - {\text{E}}}}{{\sqrt {\text{E}} }}$, где O – наблюдаемое число компаундов в каждом геноме (CCMobs), а E – ожидаемое число компаундов в каждом геноме (CCMexp). Значение Z-критерия рассчитывали на основании следующих уравнений:
(1)
${\bar {C} = }\frac{1}{n}\sum\limits_{i = 1}^n {\left( {\frac{{{\text{CcSS}}{{{\text{R}}}_{i}}}}{{{\text{CC}}{{{\text{M}}}_{i}}}}} \right)} ,$РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Влияние расстояния между блоками микросателлитов на обнаружение компаундов
Микросателлиты входят в состав компаунда, если расстояние между любыми двумя соседними SSRs не превышает величины dMAX. В связи с этим значение dMAX считается ключевым фактором при поиске сложных микросателлитов [16]. В программе IMEx величина dMAX может принимать значения от 0 до 50 [34]. Оценку влияния этого параметра на выявление компаундов проводили путем измерения среднего значения плотности компаундов (RACCM) для 10 штаммов B. pseudomallei на хромосомном и геномном уровнях с заданным значением dMAX (10, 20, 30, 40 и 50 п.н.). Как и ожидалось, средние значения RACCM постепенно возрастали с увеличением dMAX, но зависимость не удовлетворяла требованиям линейности (рис. 1).
Рис. 1.
Влияние различных значений dMAX на величину средней плотности компаундов (RACCM) в геноме и в каждой из хромосом для 10 штаммов B. pseudomallei.
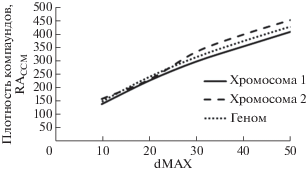
Таким образом, выбор оптимальной величины расстояния между блоками микросателлитов в составе компаундов далеко не тривиальная задача. Для идентификации компаундов мы использовали значение dMAX, равное 10 п.н., что позволяло провести сравнение полученных нами результатов с ранее опубликованными данными, так как пороговое значение 10 п.н. было использовано при изучении сложных микросателлитов в геноме эукариот и ряда прокариот [16, 18, 19].
Содержание, плотность и расположение вырожденных и сложных микросателлитов в геномах B. pseudomallei
В ходе проведенного компьютерного анализа нуклеотидных последовательностей 10 штаммов B. pseudomallei из базы данных GenBank выявлено в общей сложности 215 683 микросателлита. Число повторов на геном варьировало от 21 280 для B. pseudomallei 668 до 21 710 для B. pseudomallei MSHR305 (табл. 2); при этом плотность микросателлитов изменялась в обратном направлении: от 2922.7/млн.п.н. (B. pseudomallei MSHR305) до 3022.6/млн.п.н. (B. pseudomallei 668).
В результате анализа in silico выявлено 10 720 компаундов (табл. 2). Их содержание варьировало от 1052 в геноме штамма B. pseudomallei BPC006 до 1113 в геноме B. pseudomallei NCTC 13178. Величина СcSSR, отражающая число SSRs в составе компаундов, изменялась в диапазоне от 2201 (B. pseudomallei BPC006) до 2314 (B. pseudomallei NCTC 13178). Плотность компаундов изменялась в пределах от 144.7/млн.п.н. (B. pseudomallei NAU20B-16) до 150.6/млн.п.н. (B. pseudomallei 1026b и B. pseudomallei NCTC 13178). Значения процентной доли CcSSR от общего числа обнаруженных микросателлитов (cSSR%) варьировали в зависимости от штамма в диапазоне от 10.2% (B. pseudomallei NAU20B-16) до 10.67% (B. pseudomallei NCTC 13 178).
Полученные значения Z-критерия не позволили выявить статистически значимых различий между наблюдаемым (CCMobs) и ожидаемым (CCMexp) числом компаундов в каждом геноме. Для штаммов B. pseudomallei 1710b, 1106a, NAU20B-16, BPC006 и 668 величина CCMobs оказалась меньше, чем CCMexp. Напротив, значение CCMobs превышало CCMexp в геномах штаммов B. pseudomallei K96243, 1026b, NCTC 13178, NCTC 13179 и MSHR305 (табл. 2). Межштаммовые различия в значениях Z-критерия обусловлены использованием усредненной величины сложности компаундов (${\bar {C}}$) в составе проанализированных геномов при расчете ожидаемого числа сложных микросателлитов. Установлена статистически значимая корреляция Z-критерия со сложностью компаундов (r = –0.99, p < 10–4). Для оценки величины среднего значения сложных микросателлитов в геноме рассчитывали 95%-ный доверительный интервал на основе выборочных данных, пределы которого составили 1057–1086 компаундов.
Необходимо учитывать, что установленные параметры поиска микросателлитов приводят к обнаружению как вырожденных SSRs, так и совершенных повторов. На долю вырожденных микросателлитов приходилось около 98% от всех выявленных в общей сложности 215 683 микросателлитов в геномах 10 штаммов B. pseudomallei. Компаунды, в свою очередь, также премущественно были составлены из вырожденных микросателлитов и лишь около 1% SSRs в их составе представлены совершенными повторами.
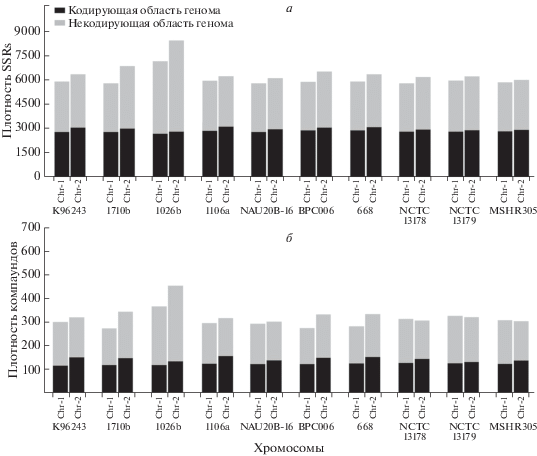
С целью выявления распределения вырожденных и сложных микросателлитов в геномах исследуемых штаммов B. pseudomallei проанализирована их представленность в кодирующих и некодирующих областях генома. В среднем число SSRs и компаундов в кодирующей области генома соответствовало 84.49 и 81.56% от общего числа найденных повторов. Однако при нормировании числа SSRs и сложных микросателлитов на общий размер генома бóльшая обогащенность повторами выявлена для некодирующей области. Средняя плотность SSRs и компаундов составляла соответственно 5859.1/млн.п.н. и 282.8/млн.п.н. в кодирующих регионах и 6759.9/млн.п.н. и 396.3/млн.п.н. в некодирующих областях (рис. 2).
Линейный корреляционный анализ связей количественной представленности микросателлитов и компаундов со структурными особенностями генома B. pseudomallei
Выявление достоверных корреляционных взаимосвязей количества и плотности вырожденных и сложных микросателлитов с такими параметрами генома B. pseudomallei, как его размер и GC-содержание, необходимо для понимания факторов, связаных с возникновением и распространением микросателлитов по геному. Результаты проведенного корреляционного анализа систематизированы в табл. 3.
Таблица 3.
Результаты корреляционного анализа связей количественной представленности микросателлитов и компаундов со структурными особенностями генома B. pseudomallei
| Число и плотность SSRs и компаундов | Размер генома/хромосомы | GC-содержание генома/хромосомы | |
|---|---|---|---|
| Геном | CM | r = 0.95, p < 10–4 | r = –0.89, p < 10–3 |
| RACM | r = –0.99, p < 10–7 | r = 0.98, p < 10–6 | |
| CCM | r = 0.68, p = 0.03 | r = –0.62, p = 0.06 | |
| RACCM | r = –0.32, p = 0.37 | r = 0.37, p = 0.29 | |
| Chr-1 | CM | r = 0.86, p < 10–2 | r = –0.42, p = 0.23 |
| RACM | r = –0.86, p < 10–2 | r = 0.96, p < 10–5 | |
| CCM | r = 0.24, p = 0.51 | r = –0.13, p = 0.72 | |
| RACCM | r = –0.19, p = 0.61 | r = 0.21, p = 0.72 | |
| Chr-2 | CM | r = 0.93, p < 10–4 | r = –0.88, p < 10–3 |
| RACM | r = –0.98, p < 10–6 | r = 0.99, p < 10–7 | |
| CCM | r = –0.11, p = 0.76 | r = 0.2, p = 0.58 | |
| RACCM | r = –0.87, p < 10–3 | r = 0.91, p = 10–3 | |
При анализе возможных корреляций между размером генома и GC-содержанием исследованных штаммов B. pseudomallei с числом и плотностью SSRs выявлен разнонаправленный характер их взаимовлияния: увеличение размера генома коррелировало с увеличением числа (r = = 0.95, p < 10–4) и уменьшением плотности (r = = –0.99, p < 10–7) SSRs, а увеличение GC-содержания, напротив, сопровождалось уменьшением содержания SSRs (r = –0.89, p < 10–3) и увеличением их плотности в геноме (r = 0.98, p < 10–6). В то же время не выявлено достоверной корреляционной связи размера генома и GC-содержания с плотностью компаундов.
Аналогичный характер корреляционных взаимосвязей наблюдался между количественной представленностью SSRs и размером каждой из хромосом B. pseudomallei. Однако для хромосомы 2 выявлена более значимая корреляция ее размера с числом и плотностью SSRs (r = 0.93 при p < 10–4 и r = –0.98 при p < 10–6 соответственно), чем для хромосомы 1 (r = 0.86 при p < 10–2 и r = –0.86 при p < 10–2 соответственно). Увеличение GC-содержания было сопряжено с увеличением плотности SSRs, как в составе хромосомы 1 (r = 0.96 при p < < 10–5), так и в составе хромосомы 2 (r = 0.99 при p < 10–7).
При анализе взаимосвязи плотности компаундов с размером и GC-содержанием каждой хромосомы выявлены статистически значимые корреляции только для хромосомы 2. Важно, что плотность компаундов коррелировала с размером (r = –0.87 при p < 10–3) и GC-содержанием (r = 0.91 при p < 10–3) хромосомы 2 – аналогично плотности SSRs, что нашло отражение в прямой очень сильной корреляции между плотностями SSRs и компаундов в составе хромосомы 2 (r = = 0.93, p < 10–3).
Сложность компаундов. Структура мотивов вырожденных и сложных микросателлитов
Сложность компаунда определяется числом индивидуальных микросателлитов, cSSRs, входящих в его состав. Например, компаунд (GCG)4-x3-(CG)6 содержит 2 cSSRs, а компаунд (CGC)4-x5-(GC)6-x6-(GCG)5 включает 3 cSSRs, поэтому их сложности выражаются как C = 2 и C = 3 соответственно. Первый тип сложных микросателлитов обозначают как 2-SSR-, а второй как 3-SSR-компаунды [22]. Для всех исследованных штаммов B. pseudomallei выявлена обогащенность 2-SSR-компаундами как хромосомы 1, так и хромосомы 2, что в среднем составило 93.13% и 91.99% от общего числа найденных в составе каждой хромосомы компаундов. Следующими по частоте встречаемости оказались 3-SSR-компаунды (6.47% для хромосомы 1 и 7.08% для хромосомы 2) и 4-SSR-компаунды (0.38% для хромосомы 1 и 0.70% для хромосомы 2). Cложные микросателлиты со значением C ≥ 5 представлены единичными вариантами. Компаунды наибольшей сложности обнаружены в составе хромосомы 2 штаммов B. pseudomallei NAU20B-16 и B. pseudomallei 668 и составили C = 8 и C = 9 соответсвенно. В целом, хромосома 2 проанализированных штаммов возбудителя мелиоидоза содержала более сложно организованные компаунды, чем хромосома 1. Показано, что доля компаундов со значением C ≥ 4 от общего числа сложных микросателлитов составила в среднем 0.4% для хромосомы 1, в то время как для хромосомы 2 этот показатель был равен 0.94%.
Нуклеотидный состав и размер мотива определяют частоту его встречаемости в составе микросателлитного локуса. Полученные результаты анализа вырожденных и сложных микросателлитов в геномах штаммов B. pseudomallei продемонстрировали более высокую распространенность ди- и тринуклеотидных последовательностей по сравнению с тетра-, пента- и гексаповторами при общей обогащенности всех мотивов GC-нуклеотидами. Интересно также отметить, что в геноме B. pseudomallei мотивы, наиболее часто образующие тандемные повторы, составляли довольно ограниченное множество и имели узкий диапазон копийности в составе микросателлитного локуса (табл. 4). Как видно из табл. 4, самые распространенные мотивы в составе вырожденных и сложных микросателлитов были идентичны для обеих хромосом B. pseudomallei. Установлено, что около 94% динуклеотидных CG/GC-микросателлитов представлены шести- или семикратной копийностью мотива, в то время как более 85% тринуклеотидных микросателлитов представлены четырехкратным вхождением “коровой” единицы.
Таблица 4.
Наиболее часто встречающиеся мотивы и их средняя копийность в составе SSRs-локусов и компаундов для каждой из хромосом B. pseudomallei
| SSRs | Компаунды | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| Хромосома 1 | Хромосома 2 | Хромосома 1 | Хромосома 2 | ||||||||
| мотив | число повторов | копийность мотива и число SSRs | мотив | число повторов | копийность мотива и число SSRs | мотив | число повторов | мотив | число повторов | ||
| ×na | Nb | ×na | Nb | ||||||||
| CG | 2453 | ×6 ×7 |
1724 574 |
CG | 2165 | ×6 ×7 |
1502 546 |
CG | 239 | CG | 221 |
| GC | 1838 | ×6 ×7 |
1329 382 |
GC | 1588 | ×6 ×7 |
1134 355 |
GC | 184 | GC | 155 |
| CGC | 897 | ×4 | 763 | CGC | 765 | ×4 | 657 | GCG | 95 | CGC | 87 |
| GCG | 852 | ×4 | 725 | GCG | 711 | ×4 | 603 | CGC | 95 | GCG | 85 |
| TCG | 422 | ×4 | 392 | CGG | 390 | ×4 | 349 | CGG | 51 | GCC | 50 |
| CGG | 415 | ×4 | 367 | GCC | 358 | ×4 | 320 | GCC | 48 | CGG | 39 |
| GCC | 410 | ×4 | 354 | TCG | 310 | ×4 | 282 | TCG | 26 | TCG | 27 |
| CGA | 379 | ×4 | 347 | CGA | 266 | ×4 | 244 | CCG | 26 | CGA | 26 |
| CCG | 210 | ×4 | 191 | CCG | 187 | ×4 | 162 | CGA | 24 | CCG | 26 |
| GGC | 208 | ×4 | 191 | GGC | 167 | ×4 | 147 | GGC | 24 | GGC | 25 |
| CGT | 182 | ×4 | 162 | CGT | 138 | ×4 | 127 | ACG | 16 | CGT | 15 |
| ACG | 171 | ×4 | 159 | ACG | 114 | ×4 | 110 | CGT | 12 | GCCG | 11 |
| TGC | 119 | ×4 | 114 | GCA | 98 | ×4 | 93 | CGGC | 11 | ACG | 11 |
| AGC | 119 | ×4 | 105 | AGC | 95 | ×4 | 87 | GCCG | 11 | CGGC | 11 |
| GCT | 105 | ×4 | 93 | TGC | 90 | ×4 | 83 | GAC | 9 | AGC | 8 |
| GCA | 104 | ×4 | 100 | GCT | 80 | ×4 | 67 | TGC | 9 | CTG | 8 |
| GTC | 97 | ×4 | 89 | GCCG | 74 | ×4 | 67 | GTC | 9 | GCA | 8 |
| GAC | 88 | ×4 | 81 | CGGC | 72 | ×4 | 64 | AGC | 8 | TCGC | 8 |
| CGGC | 82 | ×3 | 76 | GAC | 69 | ×3 | 64 | CCGG | 8 | GCT | 7 |
| GCCG | 79 | ×3 | 67 | GTC | 65 | ×3 | 55 | GCCC | 7 | CCGG | 7 |
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Простые повторяющиеся последовательности ДНК, состоящие из тандемных итераций коротких олигонуклеотидов, представляют собой гипермутируемые локусы и способны к обратимому изменению длины [12]. В ряде работ показано [36–38], что в геноме патогенных микроорганизмов сильному положительному отбору подвергаются высоко изменчивые микросателлитные локусы в составе генов, как правило, кодирующих поверхностные антигены возбудителей. Динамичное изменение числа повторов в структуре таких генов вносит существенный вклад в антигенные вариации бактерий и обеспечивает им ускользание от иммунного надзора организма-хозяина; при этом полиморфизм генов обусловлен протяженными участками SSRs с числом вхождения “коровой” единицы не менее 8 раз [39, 40]. Согласно существующей гипотезе, изменение числа повторов в составе микросателлитного локуса направляется проскальзыванием полимеразы при репликационных, репарационных и других процессах [41]. Однако мало что известно о механизмах проскальзывания полимеразы при чрезвычайно коротких локусах SSRs, включающих от двух до пяти тандемно повторяющихся мотивов и широко представленных в бактериальных геномах. В подобных коротких локусах SSRs некоторых микроорганизмов выявлен высокий уровень мутаций [42, 43]. Накопление точечных мутаций в составе микросателлитного кластера приводит к образованию несовершенных повторов и компаундов, которые могут прерывать рост тандемов и, тем самым, стабилизировать их в результате нарушения процесса проскальзывания полимеразы.
На широком круге разнообразных видов прокариотических организмов было показано, что протяженные SSR-локусы характерны для бактерий, адаптированных к экологической нише своего хозяина; причем для большинства таких бактерий размер генома и GC-содержание не превышали соответственно 2 млн.п.н. и 40%. По-видимому, подобная взаимосвязь между уникальными условиями средовой ниши и структурными особенностями генома поддерживается отбором и обеспечивает антигенную вариативность как часть защитных реакций бактерий, узко адаптированых к биотопам организма хозяина [3]. Однако для бактериальных видов c крупными геномами, в качестве среды обитания которых характерны разнообразные и сложные по структуре экосистемы с широким диапазоном условий, выявлена низкая представленность протяженных SSR-локусов. К числу таких видов относится возбудитель мелиоидоза, адаптировавшийся к возможностям как сапрофитного, так и паразитического существования. Геном B. pseudomallei представлен двумя хромосомами с размерами около 4 млн.п.н. и 3 млн.п.н., каждая из которых несет гены рибосомальных и транспортных РНК. Нуклеотидный состав генома обогащен GC-парами, доля которых в среднем составляет около 68%, а плотность белоккодирующих генов превышает 90%. С целью прояснить возможную функциональную роль SSRs в механизмах адаптационной пластичности возбудителя мелиоидоза мы проанализировали вырожденные и сложные микросателлиты в геномах 10 штаммов B. pseudomallei.
Проведен сравнительный анализ полученных результатов с опубликованными данными исследований SSRs и компаундов в геномах видов Lactobacillus spp. [19] и Escherichia coli [18]. В качестве общей характеристики, присущей геномам B. pseudomallei, Lactobacillus spp. и E. coli, нужно отметить экологическую пластичность, что позволяет этим патогенам существовать в различных экологических нишах, включая объекты окружающей среды и различные биотопы организма человека и животных. Вместе с тем, структурная организация геномов Lactobacillus spp. и E. coli заметно отличается от генома B. pseudomallei – прежде всего, это меньший размер их геномов и более низкое содержание GC-пар (2 млн.п.н и 42% для Lactobacillus spp. и 5 млн.п.н и 50% для E. coli).
При количественном сопоставлении результатов оказалось, что диапазон плотности микросателлитов (2922.7–3022.6/млн.п.н.) и компаундов (144.7–150.6/млн.п.н.) в геномах проанализированных штаммов B. pseudomallei значительно превосходил соответствущие показатели для Lactobacillus spp. (506.7–1040.9 микросателлитов/млн.п.н.; 3.3–16.4 компаундов/млн.п.н.) и E. coli (644.6–675.7 микросателлитов/млн.п.н.; 5.5–9.4 компаундов/млн.п.н.). Различную плотность SSRs и компаундов в геномах B. pseudomallei, Lactobacillus spp. и E. coli нельзя объяснить исключительно вариативностью размеров их геномов. При анализе представленности вырожденных и сложных микросателлитов в каждой из хромосом B. pseudomallei обнаружен сходный с полногеномным диапазон плотности: 2844.7–2928 микросателлитов/млн.п.н. и 130.6–145.4 компаундов/млн.п.н. для хромосомы 1; 2969.8–3157.3 микросателлитов/млн.п.н. и 149.7–165.3 компаундов/млн.п.н. для хромосомы 2. Размер каждой из хромосом B. pseudomallei не превосходит размера генома сравниваемых штаммов E. coli, что может свидетельствовать о вероятной видоспецифичности плотности микросателлитов, определяемой структурными особенностями генома.
Для установления характера структуры взаимосвязей между показателями количественного содержания SSRs и компаундов с такими параметрами генома B. pseudomallei, как его размер и GC-содержание, был проведен корреляционный анализ. Нами выявлено сопряженное увеличение плотности SSRs с GC-содержанием, а также обратная взаимосвязь плотности SSRs с размером, как для генома в целом, так и для каждой из хромосом. Однако в случае сложных микросателлитов достоверную корреляционную связь между плотностью компаундов и структурными сособенностями генома удалось установить только для хромосомы 2.
Определение закономерностей взаимосвязи плотности вырожденных и сложных микросателлитов позволяет предполагать возможный механизм образования компаундов. Так, Kofler с соавт. [16], при исследовании кластеризации микросателлитов в геномах 8 разных эукариотических видов, в качестве возможного механизма образования компаундов предложили процесс замещения нуклеотидов в концевых мотивах микросателлитного тракта с последующей экспансией таких несовершенных повторов. Подобный механизм формирования сложных микросателлитов должен приводить к тесной прямой корреляционной связи между плотностями SSRs и компаундов, что и было показано авторами [16]. Однако Chen и др. [18] при исследовании содержания сложных микросателлитов в геномах штаммов E. coli выявили очень слабую корреляцию между плотностями SSRs и компаундов, а разный характер ассоциации плотностей микросателлитов и компаундов в эукариотических и прокариотических геномах объяснили функционированием различных ДНК-полимераз и механизмов репликации. В дальнейшем Basharat & Yasmin [19] при анализе представленности компаундов в геномах Lactobacillus spp. пришли к заключению о зависимости ассоциации плотностей SSRs и компаундов у прокариот от видовой принадлежности и рекомбинационного потенциала. При анализе штаммов B. pseudomallei мы выявили статистически значимую взаимосвязь между плотностью вырожденных и сложных микросателлитов только для хромосомы 2. Эти результаты вполне согласуются с выводами Basharat & Yasmin [19] о видоспецифичности и структурных особенностях генома как факторов, определяющих плотности SSRs и компаундов. Более того, нами показано, что даже в рамках одного вида структурная организация каждой из хромосом может быть ассоциирована с разной плотностью SSRs и компаундов.
В результате анализа размера и нуклеотидного состава мотивов в структуре выявленных SSRs обнаружено преобладание GC-богатых ди- и тринуклеотидных повторов в геноме B. pseudomallei. Показано почти полное отсутствие мононуклеотидных треков и AT/TA-микросателлитов. Особенно интересным оказалось обнаружение перепредставленности определенной копийности мотива для ди- и тринуклеотидных микросателлитных последовательностей. Так, выявлено преимущественное шести- и семикратное вхождение “коровой” единицы в состав динуклеотидных микросателлитных локусов, в то время как размер тринуклеотидных повторов соответствовал в основном четырехкратной копийности мотива. Именно возможность формирования стабильных шпилечных структур некоторыми тринуклеотидными повторами: (CGG)n, (CTG)n, (CAG)n, (CСG)n и др., – которые способствуют проскальзыванию ДНК-полимеразы в ходе репликации, считается основой для исключительной нестабильности микросателлитов, что проявляется в изменении числа “коровых” единиц в их составе [44]. Однако при анализе геномов штаммов B. pseudomallei нами выявлена широкая представленность тринуклеотидных микросателлитных повторов и малая копийность мотивов в их составе, что свидетельствут скорее о низком уровне проскальзывания полимеразой этих участков генома в процессе репликации. Согласно существующей теории [41], при возникновении микросателлиты не содержат ошибок (замен, делеций и вставок), но появляющиеся в процессе биологической эволюции точечные мутации приводят к их накоплению, что становится препятствием для проскальзывания полимеразы и для дальнейшей экспансии повторов в составе микросателлитного локуса. Возможно, именно вырожденность большинства SSRs, составившая около 98% от всех обнаруженных микросателлитов в геномах B. pseudomallei, способствовала дестабилизации образуемых ими шпилечных структур и ограничивала увеличение длины блока микросателлитов.
При исследовании сложности компаундов, выявленных в геномах B. pseudomallei, продемонстрирована преимущественная представленность 2-SSR- и 3-SSR-компаундов в обеих хромосомах; лишь незначительная часть сложных микросателлитов оказалась представленной 4-SSR-компаундами. За исключением штамма B. pseudomallei MSHR305, компаунды со сложностью C ≥ 5 в исследованных штаммах возбудителя мелиоидоза представлены исключительно в составе хромосомы 2. Такой характер распределения сложноструктурированных компаундов в геноме B. pseudomallei может быть обусловлен более высокой плотностью SSRs в составе хромосомы 2, что повышает вероятность их вовлечения в формирование сложных микросателлитов. Оказалось, что большинство сложных микросателлитов составлено из разных мотивов, отличающихся как по размеру, так и по нуклеотидному составу, и это вполне согласуется с данными по структуре компаундов в геноме E. coli [18] и у видов рода Lactobacillus [19]. В то же время у эукариот, по меньшей мере в 90% всех обнаруженных 2-SSR-компаундов, выявленные мотивы имели очень схожую структуру [16]. Подобные различия в структурной организации сложных микросателлитов прокариот и эукариот, возможно, определяются разными механизмах их возникновения: фиксация полиморфизмов в составе разных SSR-локусов с последующим их вовлечением в формирование сложных микросателлитов у прокариот и последовательная экспансия вырожденных повторов в составе одного SSR-локуса у эукариот.
Таким образом, нами обнаружена вырожденность и малая копийность мотивов в составе большинства выявленных в геномах B. pseudomallei микросателлитов. Эти результаты можно рассматривать как подтверждение гипотезы, согласно которой протяженные локусы SSRs подвержены отрицательному отбору в геномах большинства прокариот, способных выживать как в организме хозяина, так и во внешней среде, наряду с увеличением размера и GC-обогащенностью их геномов [3]. Несмотря на значительный диапазон вариативности размера и GC-содержания геномов B. pseudomallei, Lactobacillus spp. и E. coli, единый характер направленности изменений в содержании микросателлитов может быть отражением экологических особенностей этих бактерий. Возможно, в основе выявленных тенденций, вырожденности и низкой копийности мотивов в составе микросателлитов, лежит механизм накопления точечных мутаций в локусах SSRs, что приводит к прерыванию блока повторов в результате формирования легко преодолимых в процессе репликации вторичных структур, стабилизирующих длину микросателлитного локуса.
Список литературы
Tautz D. (1993) Notes on the definition and nomenclature of tandemly repetitive DNA sequences. EXS. 67, 21–28.
Zhao X., Tan Z., Feng H., Yang R., Li M., Jiang J., Shen G., Yu R. (2011) Microsatellites in different Potyvirus genomes: survey and analysis. Gene. 488(1–2), 52–56.
Mrazek J., Guo X., Shah A. (2007) Simple sequence repeats in prokaryotic genomes. Proc. Natl. Acad. Sci. USA. 104(20), 8472–8477.
Saeed A.F., Wang R., Wang S. (2016) Microsatellites in pursuit of microbial genome evolution. Front. Microbiol. 6, 1462.
Toth G., Gaspari Z., Jurka J. (2000) Microsatellites in different eukaryotic genomes: survey and analysis. Genome Res. 10(7), 967–981.
Dieringer D., Schlotterer C. (2003) Two distinct modes of microsatellite mutation processes: evidence from the complete genomic sequences of nine species. Genome Res. 13(10), 2242–2251.
Hong C.P., Piao Z.Y., Kang T.W. (2007) Genomic distribution of simple sequence repeats in Brassica rapa. Mol. Cells. 23(3), 349–356.
Rajendrakumar P., Biswal A.K., Balachandran S.M., Srinivasarao K., Sundaram R.M. (2007) Simple sequence repeats in organellar genomes of rice: frequency and distribution in genic and intergenic regions. Bioinformatics. 23(1), 1–4.
Kashi Y., King D., Soller M. (1997) Simple sequence repeats as a source of quantitative genetic variation. Trends Genet. 13(2), 74–78.
Gur-Arie R., Cohen C.J., Eitan Y., Shelef L., Hallerman E.M., Kashi Y. (2000) Simple sequence repeats in Escherichia coli: abundance, distribution, composition, and polymorphism. Genome Res. 10(1), 62–71.
Huang T.S., Lee C.C., Chang A.C., Lin S., Chao C.C., Jou Y.S., Chu Y.W.,Wu C.W., Whang-Peng J. (2003) Shortening of microsatellite deoxy (CA) repeats involved in GL331-induced down-regulation of matrix metalloproteinase-9 gene expression. Biochem. Biophys. Res. Commun. 300(4), 901–907.
Kashi Y., King D.G. (2006) Simple sequence repeats as advantageous mutators in evolution. Trends Genet. 22(5), 253–259.
Boeva V., Regnier. M., Papatsenko D., Makeev V. (2006) Short fuzzy tandem repeats in genomic sequences, identification, and possible role in regulation of gene expression. Bioinformatics. 22(6), 676–684.
Bull L.N., Pabon-Pena C.R., Freimer N.B. (1999) Compound microsatellite repeats: practical and theoretical features. Genome Res. 9(9), 830–838.
Weber. J.L. (1990) Informativeness of human (dC‑dA)n.(dG-dT)n polymorphisms. Genomics. 7(4), 524–530.
Kofler R., Schlotterer C., Luschutzky E., Lelley T. (2008) Survey of microsatellite clustering in eight fully sequenced species sheds light on the origin of compound microsatellites. BMC Genomics. 9, 612.
Jia D. (2016) Survey and analysis of simple sequence repeats (SSRs) in three genomes of Candida species. Gene. 584(2), 129–135.
Chen M., Zeng G., Tan Z., Jiang M, Zhang J., Zhang C., Lu L., Lin Y., Peng J. (2011) Compound microsatellites in complete Escherichia coli genomes. FEBS Lett. 585(7), 1072–1076.
Basharat Z., Yasmin A. (2015) Survey of compound microsatellites in multiple Lactobacillus genomes. Can. J. Microbiol. 61(12), 898–902.
Alam C.M., Singh A.K., Sharfuddin C., Ali S. (2014) Genome-wide scan for analysis of simple and imperfect microsatellites in diverse carlaviruses. Infect. Genet. Evol. 21, 287–294.
Alam C.M., Singh A.K., Sharfuddin C., Ali S. (2014) Incidence, complexity and diversity of simple sequence repeats across potexvirus genomes. Gene. 537(2), 189–196.
Wu X., Zhou L., Zhao X., Tan Z. (2014) The analysis of microsatellites and compound microsatellites in 56 complete genomes of Herpesvirales. Gene. 551(1), 103–109.
Liguori A.P., Warrington S.D., Ginther J.L., Pearson T., Bowers J., Glass M.B., Mayo M., Wuthiekanun V., Engelthaler D., Peacock S.J., Currie B.J., Wagner D.M., Keim P., Tuanyok A. (2011) Diversity of 16S–23S rDNA Internal Transcribed Spacer (ITS) reveals phylogenetic relationships in Burkholderia pseudomallei and its near-neighbors. PLoS One. 6(12), e29323.
Godoy D., Randle G., Simpson A.J., Aanensen D.M., Pitt T.L., Kinoshita R., Spratt B.G. (2003) Multilocus sequence typing and evolutionary relationships among the causative agents of melioidosis and glanders, Burkholderia pseudomallei and Burkholderia mallei. J. Clin. Microbiol. 41(5), 2068–2079.
Holden M.T., Titball R.W., Peacock S.J., Cerdeño-Tárraga A.M., Atkins T., Crossman L.C., Pitt T., Churcher C., Mungall K., Bentley S.D., Sebaihia M., Thomson N.R., Bason N., Beacham I.R., Brooks K., Brown K.A., Brown N.F., Challis G.L., Cherevach I., Chillingworth T., Cronin A., Crossett B., Davis P., DeShazer D., Feltwell T., Fraser A., Hance Z., Hauser H., Holroyd S., Jagels K., Keith K.E., Maddison M., Moule S., Price C., Quail M.A., Rabbinowitsch E., Rutherford K., Sanders M., Simmonds M., Songsi-vilai S., Stevens K., Tumapa S., Vesaratchavest M., Whitehead S., Yeats C., Barrell B.G., Oyston P.C., Parkhill J. (2004) Genomic plasticity of the causative agent of melioidosis, Burkholderia pseudomallei. Proc. Natl. Acad. Sci. USA. 101(39), 14240–14245.
Nandi T., Ong C., Singh A.P., Boddey J., Atkins T., Sarkar-Tyson M., Essex-Lopresti A.E., Chua H.H., Pearson T., Kreisberg J.F., Nilsson C., Ariyaratne P.,Ronning C., Losada L., Ruan Y., Sung W.K., Woods D., Titball R.W., Beacham I., Peak I., Keim P., Nierman W.C., Tan P. (2010) A genomic survey of positive selection in Burkholderia pseudomallei provides insights into the evolution of accidental virulence. PLoS Pathog. 6(4), e1000845.
DeShazer D. (2004) Genomic diversity of Burkholderia pseudomallei clinical isolates: subtractive hybridization reveals a Burkholderia mallei-specific prophage in B. pseudomallei 1026b. J. Bacteriol. 186(12), 3938–3950.
Fang Y., Huang Y., Li Q., Chen H.,Yao Z., Pan J., Gu J., Tang B., Wang H.G., Yu B., Tong Y.G., Zou Q.M., Mao X.H. (2012) First genome sequence of a Burkholderia pseudomallei isolate in China, strain BPC006, obtained from a melioidosis patient in Hainan. J. Bacteriol. 194(23), 6604–6605.
Challacombe J.F., Stubben C.J., Klimko C.P., Wel-kos S.L., Kern S.J., Bozue J.A., Worsham P.L., Cote C.K., Wolfe D.N. (2014) Interrogation of the Burkholderia pseudomallei genome to address differential virulence among isolates. PLoS One. 9(12), e115951.
Spring-Pearson S.M., Stone J.K., Doyle A., Allender C.J., Okinaka R.T., Mayo M., Broomall S.M., Hill J.M., Karavis M.A., Hubbard K.S., Insalaco J.M., McNew L.A., Rosenzweig C.N., Gibbons H.S., Currie B.J., Wagner D.M., Keim P., Tuanyok A. (2015) Pangenome analysis of Burkholderia pseudomallei: genome evolution preserves gene order despite high recombination rates. PLoS One. 10(10), e0140274.
Johnson S.L., Bishop-Lilly K.A., Ladner J.T., Daligault H.E., Davenport K.W., Jaissle J., Frey K.G., Koroleva G.I., Bruce D.C., Coyne S.R., Broomall S.M., Li P.E., Teshima H., Gibbons H.S., Palacios G.F., Rosenzweig C.N., Redden C.L., Xu Y., Minogue T.D., Chain P.S. (2015) Complete genome sequences for 59 Burkholderia isolates, both pathogenic and near neighbor. Genome Announc. 3(2), e00159–15.
Stone J.K., Johnson S.L., Bruce D.C., Detter J.C., Mayo M., Currie B.J., Gelhaus H.C., Keim P., Tuanyok A. (2013) Complete genome sequence of the encephalomyelitic Burkholderia pseudomallei strain MSHR305. Genome Announc. 1(4), e00656-13.
Jolley K.A, Maiden M.C. (2010) BIGSdb: scalable analysis of bacterial genome variation at the population level. BMC Bioinformatics. 11, 595.
Mudunuri S.B., Nagarajaram H.A. (2007) IMEx: Imperfect Microsatellite Extractor. Bioinformatics. 23(10), 1181–1187.
Mrazek J. (2006) Analysis of distribution indicates diverse functions of simple sequence repeats in Mycoplasma genomes. Mol. Biol. Evol. 23(7), 1370–1385.
Moxon E.R. (1994) Adaptive evolution of highly mutable loci in pathogenic bacteria. Curr. Biol. 4(1), 24–33.
Deitsch K.W. (2009) Common strategies for antigenic variation by bacterial, fungal and protozoan pathogens. Nat. Rev. Microbiol. 7(7), 493–503.
Jordan P.W. (2005) Strain-specific differences in Neisseria gonorrhoeae associated with the phase variable gene repertoire. BMC Microbiol. 5, 21.
Потатуркина-Нестерова Н.И., Немова И.С., Магомедова А.М., Нестеров А.С. (2012) Патогенный потенциал микоплазм, эпидемиологически ассоциированных с воспалительными заболеваниями урогенитального тракта. Фундаментальные исследования. 1, 89–92.
Groisman E.A., Casadesus J. (2005) The origin and evolution of human pathogens. Mol. Microbiol. 56(1), 1–7.
Schlotterer C. (2000) Evolutionary dynamics of microsatellite DNA. Chromosoma. 109(6), 365–371.
Field D., Eggert L., Metzgar D., Rose R., Wills C. (1996) Use of polymorphic short and clustered coding- region microsatellites to distinguish strains of Candida albicans. FEMS Immunol. Med. Microbiol. 15(2–3), 73–79.
Field D., Wills C. (1998) Abundant microsatellite polymorphism in Saccharomyces cerevisiae, and the different distributions of microsatellites in eight prokaryotes and S. cerevisiae, result from strong mutation pressures and a variety of selective forces. Proc. Natl. Acad. Sci. USA. 95(4), 1647–1652.
Paiva A.M., Sheardy R.D. (2004) Influence of sequence context and length on the structure and stability of triplet repeat DNA oligomers. Biochemistry. 43(44), 14218–14227.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология