Молекулярная биология, 2019, T. 53, № 2, стр. 282-289
Вариабельность гена MIR196A2 как фактор риска развития первично-прогрессирующего рассеянного склероза
И. С. Киселев a, *, О. Г. Кулакова a, Н. М. Баулина a, В. В. Башинская a, Е. В. Попова a, А. Н. Бойко a, О. О. Фаворова a
a Российский национальный исследовательский медицинский университет имени Н.И. Пирогова Министерства здравоохранения Российской Федерации
117997 Москва, Россия
* E-mail: kiselev.ivan.1991@gmail.com
Поступила в редакцию 20.11.2018
После доработки 20.11.2018
Принята к публикации 20.11.2018
Аннотация
Рассеянный склероз – хроническое заболевание центральной нервной системы, сочетающее в своем патогенезе аутоиммунную и нейродегенеративную компоненты и характеризующееся выраженной гетерогенностью клинического фенотипа. Вклад наследственной предрасположенности в развитие наиболее частой ремиттирующей формы заболевания активно исследуется, в то время как генетическая архитектура первично-прогрессирующего рассеянного склероза остается малоизученной. Мы провели анализ ассоциации полиморфных вариантов генов микроРНК MIR146A, MIR196A2 и MIR499A с риском развития первично-прогрессирующего рассеянного склероза при сравнении больных этой формой заболевания со здоровыми индивидами и с больными ремиттирующей формой рассеянного склероза как по отдельности, так и в составе биаллельных сочетаний с вариантами генов, вовлеченных в развитие иммунного ответа. Аллель MIR196A2*С оказался дискриминирующим в отношении двух основных форм рассеянного склероза как по отдельности, так и в составе сочетаний с аллелями генов IFNAR2, IL7RA, IL6, PVT1, CD86, CCL5 и PSMB9. Представленные в работе данные могут быть использованы при составлении панели биомаркеров, которая позволит дифференцировать первично-прогрессирующую и ремиттирующую формы рассеянного склероза на начальных стадиях заболевания.
ВВЕДЕНИЕ
Рассеянный склероз (РС) – хроническое заболевание центральной нервной системы (ЦНС), сочетающее в своем патогенезе аутоиммунную и нейродегенеративную компоненты и сопровождающееся прогрессирующей неврологической дисфункцией (при отсутствии эффективной патогенетической терапии) [1]. РС обладает выраженной гетерогенностью клинического фенотипа. В большинстве случаев (примерно у 85% больных) наблюдается ремиттирующая форма РС (РРС), которая характеризуется чередованием клинических обострений и стадий частичной или полной ремиссии. Известно, что обострения при РРС связаны с активацией в различных отделах мозга очагов воспалительного процесса, который опосредуется мигрирующими в ЦНС иммунными клетками [2]. Однако у 10–15% больных наблюдают первично-прогрессирующую форму заболевания (ППРС), характеризующуюся устойчивым необратимым нарастанием неврологического дефицита [3] и отличающуюся бóльшим вкладом нейродегенеративного компонента в развитие болезни. Для ППРС характерны диффузные изменения в ткани мозга при умеренно выраженном очаговом поражении, что во многом опосредовано активацией микроглии провоспалительными цитокинами [4].
Хотя дебют заболевания при ППРС происходит в среднем на 10 лет позже, чем при РРС, быстро развивающаяся инвалидизация больных в молодом трудоспособном возрасте, характерная для этой агрессивной формы РС, обусловливает ее высокую социальную и экономическую значимость [3]. Подавляющее большинство иммуномодулирующих препаратов, эффективных при лечении РРС, не оказывает желаемого эффекта в случае ППРС [5], что подчеркивает необходимость использования при ППРС иной терапевтической стратегии. Поскольку для предотвращения необратимых неврологических нарушений необходимы быстрый выбор препарата и немедленное начало лечения РС, поиск биомаркеров, позволяющих дифференцировать ППРС от РРС на начальных стадиях заболевания, остается актуальной исследовательской задачей.
РС ‒ комплексное заболевание: клинические и патогенетические особенности основных его форм могут определяться через взаимодействие факторов внешней среды и наследственной предрасположенности полигенной природы. Связующим звеном в этом взаимодействии выступают эпигенетические механизмы, позволяющие в ответ на воздействие окружающей среды изменять экспрессию генов, в том числе и тех, для которых ранее была показана ассоциация с риском РС [6]. Один из таких механизмов – посттранскрипционное подавление генной экспрессии, опосредованное микроРНК [7].
МикроРНК представляют собой небольшие (18‒25 нуклеотидов) одноцепочечные некодирующие молекулы РНК, которые могут комплементарно или частично комплементарно связываться с мРНК-мишенью, преимущественно в 3'-нетранслируемой области, и тем самым опосредовать ее деградацию или подавление синтеза белкового продукта [8]. Благодаря плейотропности действия, микроРНК модулируют экспрессию до 60% белоккодирующих генов организма, а также опосредованно – генов других микроРНК [9]. Таким образом, они образуют сложную регуляторную систему, подобную цитокиновой сети, которая играет решающую роль в контроле функционирования многих биологических процессов, включая аутоиммунные и воспалительные реакции [7]; при этом незначительные изменения в уровне микроРНК могут оказывать влияние на экспрессию их генов-мишеней, что приводит к патологическим изменениям в работе подконтрольных им процессов.
При развитии РС наблюдается изменение уровня ряда микроРНК в различных фракциях и форменных элементах крови, а также в спинномозговой жидкости и тканях мозга больных [10]. При этом спектр микроРНК, дифференциально экспрессирующихся у больных РРС и ППРС, существенно различается [11‒13], что подтверждает представления о вовлечении этих молекул в формирование клинического фенотипа заболевания.
На уровень продукции и функциональную активность микроРНК могут влиять однонуклеотидные полиморфизмы (SNP), расположенные в их генах или в непосредственной близости от них. Для SNP rs2910164, rs11614913 и rs3746444 в генах MIR146A, MIR196A2 и MIR499A, соответственно, ранее уже была показана ассоциация с риском развития РРС и ряда других аутоиммунных заболеваний [14‒16]. Чтобы оценить возможное участие этих полиморфных вариантов в формировании клинического фенотипа РС, мы провели анализ ассоциации SNP MIR146A rs2910164, MIR196A2 rs11614913 и MIR499A rs3746444 с риском развития ППРС в сравнениях “ППРС vs Контроль” и “ППРС vs РРС”. Проведен также поиск ассоциированных с ППРС аллельных сочетаний, включающих SNP микроРНК и варианты ряда генов, участвующих в развитии иммунного ответа. Для выявленных аллельных сочетаний проведена оценка природы ген-генных взаимодействий.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
В исследование было включено 110 больных ППРС (50 женщин, 60 мужчин; средний возраст дебюта заболевания 47.5 ± 10.7 лет; средняя продолжительность заболевания 9.7 ± 7.2 лет; средний уровень инвалидизации по шкале EDSS (Expanded Disability Status Scale) 5.20 ± 1.49) и 564 больных РРС (393 женщины, 171 мужчина; возраст дебюта 27.4 ± 9.2 лет; продолжительность заболевания 11.4 ± 7.4 лет; EDSS 2.76 ± 1.17). Образцы крови больных ППРС и РРС собраны в центрах и отделениях рассеянного склероза в шести городах России (Москве, Ярославле, Екатеринбурге, Тюмени, Новосибирске и Казани). Диагноз “рассеянный склероз” (РС) устанавливали согласно критериям МакДональда в редакции 2010 года [17].
В контрольную группу вошли 424 здоровых индивида (285 женщин, 139 мужчин; средний возраст 44.2 ± 16.0 лет) без признаков неврологических заболеваний. Все включенные в исследование индивиды (этнические русские, по данным анкетирования) подписывали информированное согласие на участие в эксперименте. Исследование одобрил этический комитетет ФГБОУ ВО РНИМУ им. Н.И. Пирогова МЗ РФ.
Выделение геномной ДНК из цельной крови проводили с помощью наборов “QIAamp DNA Blood Midi Kit” (“QIAGEN”, Германия). Для генотипирования использовали полимеразную цепную реакцию в реальном времени на детектирующих амплификаторах “StepOnePlus” (“Thermo Fisher Scientific”, США) с применением наборов “TaqMan Pre-designed SNP Genotyping Assay Kit” (“Thermo Fisher Scientific”, США).
Отклонение частот носительства генотипов в исследуемых группах от равновесия Харди–Вайнберга оценивали с помощью критерия χ2 с использованием веб-инструмента SNPStats [18]. Анализ ассоциации носительства отдельных аллелей/генотипов SNP MIR146A rs2910164, MIR196A2 rs11614913 и MIR499A rs3746444 с риском развития ППРС проводили с применением программного обеспечения (ПО) APSampler [19]. Ассоциацию считали достоверной, если уровень значимости, определенный в соответствии с точным критерием Фишера (pf), для частот носительства аллеля/генотипа был менее 0.05, а 95%-ный доверительный интервал (ДИ) для отношения шансов (ОШ) не пересекал 1.
Для поиска ассоциированных с риском развития ППРС биаллельных сочетаний, в которые помимо SNP в гене микроРНК входят SNP в генах иммунного ответа, проводили мультилокусный анализ с помощью ПО APSampler. В исследование включены полиморфные варианты панели белоккодирующих генов, вовлеченных в развитие иммунного ответа: CCR5 rs333, IL7RA rs6897932, TNF rs1800629, IFNB1 rs1051922, TGFB1 rs1800471, IFNAR1 rs1012335, CTLA4 rs231775, TNFRSF1A rs1800693, IL4 rs2243250, IL6 rs1800795, IL2RA rs2104286, CLEC16A rs6498169, CCL5 rs2107538, CD58 rs2300747, EVI5 rs11804321, EOMES rs2371108, CD86 rs2255214, TCF7 rs756699, IL17A rs2275913, PSMB9 rs17587, IL22RA2 rs202573, IRF5 rs4728142, CXCR5 rs523604, IRF8 rs17445836, STAT3 rs744166, TYK2 rs2304256, TNFSF14 rs1077667, CD40 rs6074022, и IFNAR2 rs2248202, а также SNP rs2114358 в гене длинной некодирующей РНК PVT1, для которой показана ассоциация с РС по данным полногеномного поиска ассоциации (genome wide association study, GWAS). Ассоциацию считали значимой, если значение pf для носительства сочетания было менее 0.01, в то время как 95%-ный ДИ для ОШ не пересекал 1. Природу взаимодействия между компонентами сочетаний оценивали с помощью точного трехфакторного теста, подобного критерию Фишера (the exact three-way Fisher-like interaction numeric test, FLINT), и фактора синергии (synergy factor, SF), входящих в состав инструментов ПО APSampler [20]. Взаимодействие считали эпистатическим, если величина pFLINT была менее 0.05, а значение 95%-ного ДИ для SF не пересекало 1.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Определены частоты носительства аллелей и генотипов исследуемых SNP в генах микроРНК в группах больных ППРС, РРС и здоровых индивидов контрольной группы (табл. 1). Распределение генотипов в каждой из групп соответствовало равновесию Харди–Вайнберга для всех SNP.
Таблица 1.
Частоты носительства аллелей и генотипов генов микроРНК в сравнениях “ППРС vs Контроль” и “ППРС vs РРС”
| Поли- морфный вариант | Аллель/ генотип | Число (процент) носителей | “ППРС vs Контроль” | “ППРС vs РРС” | ||||
|---|---|---|---|---|---|---|---|---|
| ППРС, n = 110 |
контроль, n = 424 |
РРС, n = 564 |
значение pf | ОШ (95% ДИ)* | значение pf | ОШ (95% ДИ)* | ||
| MIR146A rs2910164 |
C | 43 (39.4) | 165 (38.9) | 202 (35.8) | Н.з. | – | Н.з. | – |
| G | 103 (94.5) | 406 (95.8) | 534 (94.7) | Н.з. | – | Н.з. | – | |
| C/C | 6 (5.5) | 18 (4.2) | 30 (5.3) | Н.з. | – | Н.з. | – | |
| C/G | 37 (33.9) | 147 (34.7) | 172 (30.5) | Н.з. | – | Н.з. | – | |
| G/G | 66 (60.6) | 259 (61.1) | 362 (64.2) | Н.з. | – | Н.з. | – | |
| MIR196A2 rs11614913 |
C | 103 (93.6) | 371 (87.5) | 484 (85.8) | 0.044 | 2.10 (0.93–4.76) | 0.014 | 2.43 (1.09−5.42) |
| T | 65 (59.1) | 261 (61.6) | 344 (61.0) | Н.з. | – | Н.з. | – | |
| C/C | 45 (40.9) | 163 (38.4) | 220 (39.0) | Н.з. | – | Н.з. | – | |
| C/T | 58 (52.7) | 208 (49.1) | 264 (46.8) | Н.з. | – | Н.з. | – | |
| T/T | 7 (6.4) | 53 (12.5) | 80 (14.2) | 0.044 | 0.48 (0.21–1.08) | 0.014 | 0.41 (0.18−0.92) | |
| MIR499A rs3746444 |
C | 42 (38.2) | 145 (35.0) | 196 (34.8) | Н.з. | – | Н.з. | – |
| T | 107 (97.3) | 414 (97.6) | 543 (96.3) | Н.з. | – | Н.з. | – | |
| C/C | 3 (2.7) | 10 (2.4) | 21 (3.7) | Н.з. | – | Н.з. | – | |
| C/T | 39 (35.5) | 135 (31.8) | 175 (31.0) | Н.з. | – | Н.з. | – | |
| T/T | 68 (61.8) | 279 (65.8) | 368 (65.2) | Н.з. | – | Н.з. | – | |
У больных ППРС в сравнении со здоровыми индивидами контрольной группы частота носительства аллеля MIR196A2*С значимо выше в соответствии с точным критерием Фишера (pf = 0.044), ОШ = 2.10, однако 95%-ный ДИ пересекает единицу, в связи с чем эта ассоциация не классифицирована как значимая. Для других полиморфных вариантов в сравнении “ППРС vs Контроль” незначимыми оказались и значения pf, и величины 95%-ного ДИ для ОШ.
При сравнении частот носительства аллелей и генотипов исследуемых SNP в группах больных ППРС и РРС аллель MIR196A2*С оказался значимо ассоциирован с высоким риском развития ППРС (pf = 0.014; ОШ = 2.43, ДИ: 1.09 − 5.42), в то время как генотип MIR196A2*Т/Т чаще встречался у больных РРС (pf = 0.014; ОШ = 0.41, ДИ: 0.18 − 0.92). Для других SNP, как и в случае сравнения “ППРС vs Контроль”, значимых ассоциаций не выявлено.
Для выявления возможных составных маркеров клинического фенотипа РС нами проведен мультилокусный анализ, направленный на поиск ассоциированных с ППРС биаллельных сочетаний, одним компонентом которых был SNP в гене микроРНК, а вторым – полиморфный вариант из панели других включенных в исследование генов. Генотипирование последних проведено нами ранее (направлено в печать). Биаллельные сочетания, включающие аллель MIR196A*C и ассоциированные с высоким риском, обнаружены при сравнении больных ППРС как со здоровыми индивидами, так и с больными РРС (табл. 2); сочетаний, содержащих гены других микроРНК, не наблюдали. В сравнении “ППРС vs Контроль” значимо ассоциированными с высоким риском ППРС оказались сочетания MIR196A*C + CCL5*G и MIR196A*C + PSMB9*G (pf = 0.0058 и 0.0040, ОШ = 2.61 и 2.59 соответственно). Эти же два сочетания выявлены при сравнении групп больных ППРС и РРС; они характеризовались более высоким уровнем значимости ассоциации (pf = 0.0018 и 0.0026 соответственно), а также бóльшим значением ОШ (2.87 и 2.65 соответственно). Кроме того, при сравнении больных ППРС с больными РРС обнаружены биаллельные сочетания высокого риска ППРС, включающие помимо MIR196A*C также аллель PVT1*A, CD86*T, IFNAR2*C, IL7RA*C или IL6*G (pf = 0.0013−0.0056, ОШ = 1.82−2.40).
Таблица 2.
Аллельные сочетания, включающие SNP в генах микроРНК, значимо ассоциированные с риском развития ППРС хотя бы в одном из двух сравнений: “ППРС vs Контроль” и/или “ППРС vs РРС”
| Аллельное сочетание | Число (процент) носителей | “ППРС vs Контроль” | “ППРС vs РРС” | ||||
|---|---|---|---|---|---|---|---|
| ППРС, n = 110 | контроль, n = 424 | РРС, n = 564 | значение pf | ОШ (95% ДИ)* | значение pf | ОШ (95% ДИ) | |
| MIR196A2*C +PVT1*A | 94 (85.5) | 323 (76.5) | 402 (71.7) | Н.з. | – | 0.0013 | 2.32 (1.33−4.07) |
| MIR196A2*C + CCL5*G | 96 (92.3) | 335 (82.1) | 455 (80.7) | 0.0058 | 2.61 (1.22−5.62) | 0.0018 | 2.87 (1.36−6.09) |
| MIR196A2*C + CD86*T | 78 (70.9) | 261 (62.9) | 315 (56.0) | Н.з. | – | 0.0022 | 1.92 (1.23−2.99) |
| MIR196A2*C + PSMB9*G | 101 (91.8) | 342 (81.2) | 453 (80.9) | 0.0040 | 2.59 (1.26−5.35) | 0.0026 | 2.65 (1.30−5.41) |
| MIR196A2*C + IFNAR2*C | 64 (58.7) | 193 (45.8) | 247 (43.9) | Н.з. | – | 0.0031 | 1.82 (1.20−2.76) |
| MIR196A2*C + IL7RA*C | 100 (90.9) | 326 (83.2) | 454 (80.6) | Н.з. | – | 0.0050 | 2.40 (1.21−4.75) |
| MIR196A2*C + IL6*G | 85 (77.3) | 287 (68.2) | 364 (64.5) | Н.з. | – | 0.0056 | 1.87 (1.16−3.01) |
Для выявленных сочетаний в сравнениях “ППРС vs Контроль” и “ППРС vs РРС” проведен анализ природы кумулятивного эффекта между компонентами, входящими в их состав. Значения pFLINT и SF для всех биаллельных сочетаний оказались незначимыми (данные не представлены), что говорит о возможном отсутствии эпистатических ген-генных взаимодействий между компонентами сочетаний. Вероятно, вклад этих вариантов аддитивен.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В результате проведенного исследования впервые показано, что носительство аллеля MIR196A2*C (rs11614913) ассоциировано с высоким риском развития ППРС. Эти данные получены при сравнении группы больных этой формой РС с больными РРС. При сравнении со здоровыми индивидами контрольной группы ассоциация с носительством аллеля MIR196A2*C оказалась значимой только по одному из двух используемых критериев оценки; можно предположить, что при увеличении размера выборки и в этом сравнении ассоциация с высоким риском ППРС станет значимой. Показательно, что ранее нами были получены данные об ассоциации этого аллеля в составе биаллельного сочетания с более тяжелым течением РРС, оцениваемым по шкале инвалидизации (Multiple Sclerosis Severity Score, MSSS) [14]. В совокупности эти данные позволяют предполагать, что аллель MIR196A2*C ассоциирован с более тяжелыми клиническими проявлениями РС, будь то развитие первично-прогрессирующей формы заболевания или же высокая скорость прогрессирования инвалидизации при РРС. Для аллелей/генотипов других микроРНК значимых ассоциаций с предрасположенностью к ППРС в проведенном исследовании не выявлено.
Ген MIR196A2, который расположен в хромосоме 12 (12q13.13) в кластере генов HOXC, участвующих в регуляции развития позвоночных организмов, как и его паралог MIR196A1 (хромосома 17, 17q21.32, кластер генов HOXB), кодирует miR-196a [21, 22]. Эта микроРНК участвует в регуляции развития и функционирования ряда органов и систем [23‒26], включая нейрональную ткань, где она вовлечена в поддержание межклеточного транспорта, синаптической пластичности и активности нейронов [23]. Важность miR-196a для функционирования нервной системы подтверждается тем фактом, что с использованием различных модельных организмов и клеточных линий, а также при моделировании in silico показано ее участие в патогенезе болезни Гентингтона – тяжелого нейродегенеративного заболевания [23, 27‒29]. Кроме того, miR-196a играет существенную роль в регуляции NF-kB-зависимого сигнального пути, необходимого для нормального развития и функционирования систем врождённого и адаптивного иммунитета [30, 31].
Полиморфный вариант rs11614913 расположен в предшественнике pre-miR-196a2 и поэтому может влиять на эффективность процессинга, а значит, и на количество зрелой микроРНК. В ряде работ показана связь носительства аллеля MIR196A2*С с повышенным уровнем продукции miR-196a [31‒33]. Однако известны и другие данные, согласно которым этот аллель не влияет на уровень продукции микроРНК или даже снижает ее [34, 35]. Ранее была показана ассоциация rs11614913 с рядом аутоиммунных и воспалительных заболеваний. Так, аллель MIR196A2*С оказался значимо ассоциированным с высоким риском развития язвенного колита в индийской популяции и с его клиническими характеристиками у больных из Италии и Японии [36, 37], а также с тяжелыми формами астмы у больных из Южной Кореи и Египта [38, 39]. Аллель MIR196A2*Т ассоциирован с предрасположенностью к болезни Бехчета у пациентов из Китая [32].
Использование мультилокусного анализа позволило обнаружить ряд ассоциированных с ППРС биаллельных сочетаний, которые включают помимо SNP в гене MIR196A2 полиморфные варианты генов, вовлеченных в развитие иммунного ответа (см. табл. 2). Их можно разделить на три группы (рис. 1). В первую группу вошли сочетания, которые включают аллели генов IFNAR2, IL7RA и IL6, поодиночке ассоциированные с ППРС (pf = = 0.011–0.030) при сравнении со здоровыми индивидами и/или больными РРС (по данным нашей статьи, направленной в печать). Для этих аллелей значимость ассоциации существенно повышается в составе биаллельных сочетаний, выявляемых в сравнении “ППРС vs РРС” (pf = 0.0031–0.0056). Еще одну группу составили сочетания, включающие аллели генов PVT1 и CD86, для которых ранее была показана связь с предрасположенностью к РС в GWAS [40], однако связь с риском развития ППРС в сравнении с РРС для этих генов впервые выявлена в представленной работе. В третью группу вошли сочетания с аллелями генов CCL5 и PSMB9, представляющие наибольший интерес в рамках этого исследования. Для генов CCL5 и PSMB9 поодиночке ранее не наблюдали ассоциации с РС, в том числе с привлечением GWAS. Мы также ранее не выявляли ассоциации этих генов с ППРС. Примечательно, что эти сочетания обнаружены нами как при сравнении больных ППРС со здоровыми индивидами контрольной группы, так и с больными РРС, что говорит об участии продуктов генов CCL5 и PSMB9 преимущественно в патогенезе первично-прогрессирующей формы заболевания.
Рис. 1.
Схематическое представление выявленных в работе сочетаний, ассоциированных с ППРС и содержащих наряду с аллелем MIR196A2*C (слева) аллели генов, вовлеченных в развитие иммунного ответа (справа). Не выделены цветом белоккодирующие гены, для которых нами ранее уже была показана ассоциация с ППРС поодиночке (направлено в печать). Светло-серым цветом обозначены гены, ассоциированные, по данным полногеномного поиска ассоциации, с РРС [40], темно-серым – гены, связь которых с предрасположенностью к ППРС в составе биаллельных сочетаний впервые показана в этой работе.
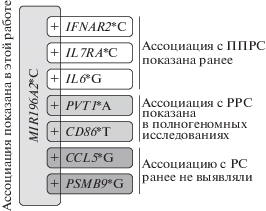
Ген CCL5 кодирует хемокин ССL5, который является хемоаттрактантом для моноцитов крови, Т-хелперов и эозинофилов и участвует в миграции иммунных клеток через гематоэнцефалический барьер [41]. Хотя связи SNP rs2107538 гена CCL5 с риском развития РС ранее не выявляли, показана его ассоциация с тяжестью течения болезни по шкале MSSS [42, 43]. Продукт гена PSMB9 – одна из субъединиц иммунопротеасомного комплекса, участвующего в процессировании антигенов для последующей их презентации на поверхности клеток в составе MHC класса I [44]. Поиск ассоциации полиморфного варианта rs17587 с риском развития РС ранее не проводили, однако была показана его ассоциация с ревматоидным артритом и с тяжелым нейродегенеративным заболеванием – болезнью Паркинсона [45, 46].
Анализ ген-генных взаимодействий во всех обнаруженных биаллельных сочетаниях показал, что вклад составляющих их вариантов, вероятно, аддитивен. Однако нельзя исключить, что эпистатические взаимодействия между компонентами тех или иных сочетаний не удалось обнаружить из-за недостаточной статистической мощности. В таком случае расширение группы больных ППРС может привести к их выявлению. О вероятном существовании подобных взаимодействий, по крайней мере в некоторых сочетаниях, может свидетельствовать тот факт, что мРНК генов PSMB9 и IL7RA представляют собой прямые мишени miR-196a-3p по данным базы предсказанных мишеней микроРНК TargetScanHuman [47].
В заключение следует отметить, что сравнение “ППРС vs РРС” позволяет получить результаты, согласующиеся с таковыми для сравнения “ППРС vs Контроль”, причем при первом сравнении число выявляемых ассоциаций и уровень их значимости возрастает. Возможно, это связано с тем, что размер группы больных РРС превышает размер контрольной группы. Аллель MIR196A2*C можно считать дискриминирующим ППРС от РРС как по отдельности, так и в составе ряда сочетаний с аллелями генов IFNAR2, IL7RA, IL6, PVT1, CD86, CCL5 и PSMB9. Показано, что в составе сочетаний с аллелями CCL5*G и PSMB9*G белоккодирующих генов аллель MIR196A2*C ассоциирован с риском развития ППРС. Скорее всего, увеличение размера выборки больных ППРС приведет к включению аллеля MIR196A2*C в категорию предрасполагающих к ППРС в сравнении со здоровыми добровольцами и поодиночке. В целом, полученные в работе данные могут быть использованы при составлении панели биомаркеров, которая позволит дифференцировать ППРС от РРС на начальных стадиях заболевания.
Авторы выражают благодарность врачам-неврологам Бабичевой Н.Н., Волковой Л.И., Караевой А.В., Касаткину Д.С., Коробко Д.С., Малковой Н.А., Сиверцевой С.А., Спириной Н.Н., Спирину Н.Н., Туровой Е.Л. и Хабирову Ф.А. за участие в формировании группы больных ППРС.
Работа выполнена при финансовой поддержке Госзадания ФГБОУ ВО РНИМУ им. Н.И. Пирогова Минздрава России и РФФИ (грант № 17-00-00206).
Авторы заявляют об отсутствии потенциального конфликта интересов.
Список литературы
Didonna A., Oksenberg J.R. (2017) The genetics of multiple sclerosis. In: Multiple Sclerosis: Perspectives in Treatment and Pathogenesis. Eds Zagon I.S., McLaughlin P.J. Brisbane: Codon Publications, Chapter 1, 3–16.
Podbielska M., O’Keeffe J., Hogan E.L. (2018) Autoimmunity in multiple sclerosis: role of sphingolipids, invariant NKT cells and other immune elements in control of inflammation and neurodegeneration. J. Neurol. Sci. 385, 198–214.
Koch M., Kingwell E., Rieckmann P., Tremlett H. (2009) The natural history of primary progressive multiple sclerosis. Neurology. 73, 1996–2002.
Correale J. (2014) The role of microglial activation in disease progression. Mult. Scler. 20, 1288–1295.
Thompson A.J. (2017) Challenge of progressive multiple sclerosis therapy. Curr. Opin. Neurol. 30, 237–240.
Oksenberg J.R. (2013) Decoding multiple sclerosis: an update on genomics and future directions. Expert. Rev. Neurother. 13, 11–19.
Баулина Н.М., Кулакова О.Г., Фаворова О.О. (2016) МикроРНК: роль в развитии аутоиммунного воспаления. Acta Naturae. 8, 23–36
Mohr A.M., Mott J.L. (2015) Overview of microRNA biology. Semin. Liver. Dis. 35, 3–11.
Diao L., Marcais A., Norton S., Chen K.C. (2014) MixMir: microRNA motif discovery from gene expression data using mixed linear models. Nucleic Acids. Res. 42, e135.
Dolati S., Marofi F., Babaloo Z., Aghebati-Maleki L., Roshangar L., Ahmadi M., Rikhtegar R., Yousefi M. (2018) Dysregulated network of miRNAs involved in the pathogenesis of multiple sclerosis. Biomed. Pharmacother. 104, 280–290.
Vistbakka J., Elovaara I., Lehtimaki T., Hagman S. (2017) Circulating microRNAs as biomarkers in progressive multiple sclerosis. Mult. Scler. 23, 403–412.
Ebrahimkhani S., Vafaee F., Young P.E., Hur S.S.J., Hawke S., Devenney E., Beadnall H., Barnett M.H., Suter C.M., Buckland M.E. (2017) Exosomal microRNA signatures in multiple sclerosis reflect disease status. Sci. Rep. 7, 14293.
Mancuso R., Hernis A., Agostini S., Rovaris M., Caputo D., Clerici M. (2015) MicroRNA-572 expression in multiple sclerosis patients with different patterns of clinical progression. J. Transl. Med. 13, 148.
Kiselev I., Bashinskaya V., Kulakova O., Baulina N., Popova E., Boyko A., Favorova O. (2015) Variants of microRNA genes: gender–specific associations with multiple sclerosis risk and severity. Int. J. Mol. Sci. 16, 20067–20081.
Li Y., Du C., Wang W., Ma G., Cui L., Zhou H., Tao H., Yao L., Zhao B., Li K. (2015) Genetic association of MiR-146a with multiple sclerosis susceptibility in the Chinese population. Cell. Physiol. Biochem. 35, 281–291.
Lu L., Tu Y., Liu L., Qi J., He L. (2014) MicroRNA-499 rs3746444 polymorphism and autoimmune diseases risk: a meta-analysis. Mol. Diagn. Ther. 18, 237–242.
Polman C.H., Reingold S.C., Banwell B., Clanet M., Cohen J.A., Filippi M., Fujihara K., Havrdova E., Hutchinson M., Kappos L., Lublin F.D., Montalban X., O’Connor P., Sandberg-Wollheim M., Thompson A.J., Waubant E., Weinshenker B., Wolinsky J.S. (2011) Diagnostic criteria for multiple sclerosis: 2010 revisions to the McDonald criteria. Ann. Neurol. 69, 292–302.
SNPStats: https://www.snpstats.net/start.htm.
Favorov A.V. (2005) APSampler: http://apsampler.sourceforge.net/.
Barsova R.M., Lvovs D., Titov B.V., Matveeva N.A., Shakhnovich R.M., Sukhinina T.S., Kukava N.G., Ruda M.Y., Karamova I.M., Nasibullin T.R., Mustafi-na O.E., Osmak G.J., Tsareva E.Y., Kulakova O.G., Favorov A.V., Favorova O.O. (2015) Variants of the coagulation and inflammation genes are replicably associated with myocardial infarction and epistatically interact in Russians. PLoS One. 10, e0144190.
Qiu R., Liu Y., Wu J.Y., Liu K., Mo W., He R. (2009) Misexpression of miR-196a induces eye anomaly in Xenopus laevis. Brain. Res. Bull. 79, 26–31.
Kim Y.J., Bae S.W., Yu S.S., Bae Y.C., Jung J.S. (2009) miR-196a regulates proliferation and osteogenic differentiation in mesenchymal stem cells derived from human adipose tissue. J. Bone Miner. Res. 24, 816–825.
Her L.S., Mao S.H., Chang C.Y., Cheng P.H., Chang Y.F., Yang H.I., Chen C.M., Yang S.H. (2017) miR-196a Enhances neuronal morphology through suppressing RANBP10 to provide neuroprotection in Huntington’s disease. Theranostics. 7, 2452–2462.
Mori M., Nakagami H., Rodriguez-Araujo G., Nimura K., Kaneda Y. (2012) Essential role for miR-196a in brown adipogenesis of white fat progenitor cells. PLoS Biol. 10, e1001314.
Go H., La P., Namba F., Ito M., Yang G., Brydun A., Igarashi K., Dennery P.A. (2016) MiR-196a regulates heme oxygenase-1 by silencing Bach1 in the neonatal mouse lung. Am. J. Physiol. Lung. Cell. Mol. Physiol. 311, L400–L411.
Tripurani S.K., Lee K.B., Wee G., Smith G.W., Yao J. (2011) MicroRNA-196a regulates bovine newborn ovary homeobox gene (NOBOX) expression during early embryogenesis. BMC Dev. Biol. 11, 25.
Fu M.H., Li C.L., Lin H.L., Tsai S.J., Lai Y.Y., Chang Y.F., Cheng P.H., Chen C.M., Yang S.H. (2015) The potential regulatory mechanisms of miR-196a in Huntington’s disease through bioinformatic analyses. PLoS One. 10, e0137637.
Kunkanjanawan T., Carter R.L., Prucha M.S., Yang J., Parnpai R., Chan A.W. (2016) miR-196a ameliorates cytotoxicity and cellular phenotype in transgenic Huntington’s disease monkey neural cells. PLoS One. 11, e0162788.
Cheng P.H., Li C.L., Chang Y.F., Tsai S.J., Lai Y.Y., Chan A.W., Chen C.M., Yang S.H. (2013) miR-196a ameliorates phenotypes of Huntington disease in cell, transgenic mouse, and induced pluripotent stem cell models. Am. J. Hum. Genet. 93, 306–312.
Yuan Y., Anbalagan D., Lee L.H., Samy R.P., Shanmugam M.K., Kumar A.P., Sethi G., Lobie P.E., Lim L.H. (2016) ANXA1 inhibits miRNA-196a in a negative feedback loop through NF-kB and c-Myc to reduce breast cancer proliferation. Oncotarget. 7, 27007–27020.
Yang G., Han D., Chen X., Zhang D., Wang L., Shi C., Zhang W., Li C., Chen X., Liu H., Zhang D., Kang J., Peng F., Liu Z., Qi J., Gao X., Ai J., Shi C., Zhao S. (2014) MiR-196a exerts its oncogenic effect in glioblastoma multiforme by inhibition of IκBα both in vitro and in vivo. Neuro Oncol. 16, 652–661.
Qi J., Hou S., Zhang Q., Liao D., Wei L., Fang J., Kijlstra A., Yang P. (2013) A functional variant of pre-miRNA-196a2 confers risk for Behcet’s disease but not for Vogt-Koyanagi-Harada syndrome or AAU in ankylosing spondylitis. Hum. Genet. 132, 1395–1404.
Hu Z., Chen J., Tian T., Zhou X., Gu H., Xu L., Zeng Y., Miao R., Jin G., Ma H., Chen Y., Shen H. (2008) Genetic variants of miRNA sequences and non-small cell lung cancer survival. J. Clin. Invest. 118, 2600–2608.
Qi P., Wang L., Zhou B., Yao W.J., Xu S., Zhou Y., Xie Z.B. (2015) Associations of miRNA polymorphisms and expression levels with breast cancer risk in the Chinese population. Genet. Mol. Res. 14, 6289–6296.
Sibin M.K., Harshitha S.M., Narasingarao K.V., Dhananjaya I.B., Dhaval P.S., Chetan G.K. (2017) Effect of rs11614913 polymorphism on mature miR196a2 expression and its target gene HOXC8 expression in human glioma. J. Mol. Neurosci. 61, 144–151.
Ciccacci C., Politi C., Biancone L., Latini A., Novelli G., Calabrese E., Borgiani P. (2017) Polymorphisms in MIR122, MIR196A2, and MIR124A genes are associated with clinical phenotypes in inflammatory bowel diseases. Mol. Diagn. Ther. 21, 107–114.
Okubo M., Tahara T., Shibata T., Yamashita H., Nakamura M., Yoshioka D., Yonemura J., Kamiya Y., Ishizuka T., Nakagawa Y., Nagasaka M., Iwata M., Yamada H., Hirata I., Arisawa T. (2011) Association study of common genetic variants in pre-microRNAs in patients with ulcerative colitis. J. Clin. Immunol. 31, 69–73.
Trinh H.K.T., Pham D.L., Kim S.C., Kim R.Y., Park H.S., Kim S.H. (2017) Association of the miR-196a2, miR-146a, and miR-499 polymorphisms with asthma phenotypes in a Korean population. Mol. Diagn. Ther. 21, 547–554.
Hussein M.H., Toraih E.A., Aly N.M., Riad E., Fawzy M.S. (2016) A passenger strand variant in miR-196a2 contributes to asthma severity in children and adolescents: a preliminary study. Biochem. Cell. Biol. 94, 347–357.
Bashinskaya V.V., Kulakova O.G., Boyko A.N., Favo-rov A.V., Favorova O.O. (2015) A review of genome-wide association studies for multiple sclerosis: classical and hypothesis-driven approaches. Hum. Genet. 134, 1143–1162.
Ubogu E.E., Callahan M.K., Tucky B.H., Ranso-hoff R.M. (2006) Determinants of CCL5-driven mononuclear cell migration across the blood-brain barrier. Implications for therapeutically modulating neuroinflammation. J. Neuroimmunol. 179, 132–144.
Макарычева О.Ю., Царева Е.Ю., Судомоина М.А., Кулакова О.Г., Титов Б.В., Быкова О.В., Гольцова Н.В., Кузенкова Л.М., Бойко А.Н., Фаворова О.О. (2011) Семейный анализ сцепления и ассоциации полиморфизма генов DRB1, CTLA4, TGFB1, IL4, CCR5, RANTES, MMP9 и TIMP1 с рассеянным склерозом. Acta Naturae. 3, 91–98.
van Veen T., Nielsen J., Berkhof J., Barkhof F., Kamphorst W., Bo L., Ravid R., Verweij C.L., Huitinga I., Polman C.H., Uitdehaag B.M. (2007) CCL5 and CCR5 genotypes modify clinical, radiological and pathological features of multiple sclerosis. J. Neuroimmunol. 190, 157–164.
Liu Y., Li H.J., Qiu X.T., Guo H.W., Li Y.H., Zhang Q. (2007) Molecular characterization, expression and mapping of porcine LMP2 and MECL-1 genes. DNA Seq. 18, 257–264.
Yu L., Li Q., Lin J., Yu J., Li Q., Yi W., Sun H., Chu J.Y., Yang Z.Q. (2013) Association between polymorphisms of PSMB8, PSMB9 and TAP2 genes with rheumatoid arthritis in ethnic Han Chinese from Yunnan. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 30, 222–226.
Mo M.S., Huang W., Sun C.C., Zhang L.M., Cen L., Xiao Y.S., Li G.F., Yang X.L., Qu S.G., Xu P.Y. (2016) Association analysis of proteasome subunits and transporter associated with antigen processing on chinese patients with Parkinson’s disease. Chin. Med. J. (Engl.). 129, 1053–1058.
Agarwal V., Bell G.W., Nam J.W. Bartel D.P. (2015) TargetScanHuman: http://www.targetscan.org.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


