Молекулярная биология, 2019, T. 53, № 2, стр. 225-239
Эволюционный консерватизм генных регуляторных сетей временно́й спецификации нейробластов
А. В. Спиров a, Е. М. Мясникова b, *
a Институт эволюционной физиологии и биохимии им. И.М. Сеченова Российской академии наук
194223 Санкт-Петербург, Россия
b Санкт-Петербургский политехнический университет им. Петра Великого
195251 Санкт-Петербург, Россия
* E-mail: ekmyasnikova@yandex.ru
Поступила в редакцию 16.08.2018
После доработки 25.10.2018
Принята к публикации 30.10.2018
Аннотация
Ансамбль генов семейства gap плодовой мушки Drosophila melanogaster относится к наиболее исследованным и наиболее консервативным генным регуляторным сетям (ГРС). Гены семейства gap, а именно hunchback (hb), Krüppel (Kr), pou-domain (pdm; pdm-1 и pdm-2) и castor (cas) входят в хорошо известные семейства Ikaros (IKZF1/hb), круппельподобных (Krüppel-like factor, KLF/Kr), POU domain (BRN1/pdm-1, BRN2/pdm-2) и гомологов Castor (CASZ1/cas), описанные у позвоночных и кодирующие сайтспецифические факторы транскрипции. Гены семейства gap составляют ядро одной из подсетей контроля эмбриональной сегментации дрозофилы, а также участвуют в процессе временно́й спецификации нейробластов у эмбрионов. Известно, что ансамбль gap регулирует сегментацию и нейрогенез у дрозофилы сходным образом. Более того, обсуждаемые гены эволюционно консервативны в своей роли функционального ядра ГРС временно́й спецификации в нейрогенезе позвоночных, в том числе человека. Особый интерес представляет вопрос, насколько эволюционно консервативны детали молекулярных механизмов регуляторики ансамбля генов gap. Ответ на него чрезвычайно важен, так как упоминаемые семейства гомологов вовлечены во многие процессы и у человека, включая функции супрессоров опухолевого роста.
ВВЕДЕНИЕ
Современные исследования в области эволюционной биологии развития, evo-devo, нацелены на выявление закономерностей в механизмах и процессах индивидуального развития путем сравнительного анализа разных модельных объектов на разных ступенях эволюционной лестницы. Оказалось, что многие процессы и механизмы или эволюционно консервативны, или заново открываются эволюцией. Современная системная биология охарактеризовала немало случаев, когда сходные процессы и механизмы, включая генные сети и сигнальные пути, неоднократно используются в ходе индивидуального развития на уровне одного организма. Возможность сопоставить сходные процессы и механизмы, как в онтогенезе одного экспериментального объекта, так и у разных объектов (на разных ступенях эволюции), позволяет не только глубже их изучить, но и лучше понять саму эволюцию.
Известно, что некоторые гены на разных ступенях эволюции контролируют разные процессы в онтогенезе; при этом механизмы контроля часто эволюционно консервативны. К таким генам относится IKZF1 – ген млекопитающих и человека, ортологичный гену дрозофилы hunchback (hb) [1, 2].
ДНК-связывающий белок Ikaros кодируется у человека геном IKZF1 [3, 4] и относится к сайтспецифическим транскрипционным факторам (ТФ) с доменом типа “цинкового пальца”. У человека (как и у модельных млекопитающих) в семейство Ikaros входят близкородственные гены: IKZF2 (ТФ Helios), IKZF3 (ТФ Aiolos) и IKZF4 (ТФ Eos). Известно, что белок Ikaros функционирует как регулятор транскрипции в процессах дифференцировки гемопоэтических клеток, то есть играет ключевую роль в системе кроветворения. Кроме того, Ikaros – один из основных супрессоров лимфоидного лейкоза у человека [5].
Помимо гематопоэза Ikaros играет важную роль в процессах нейрогенеза у млекопитающих и человека. Это как раз тот случай, когда один и тот же белок критичен для различных процессов в онтогенезе. Более того, Ikaros непосредственно репрессирует ген CASZ1 в нейрогенезе человека и модельных млекопитающих (CASZ1 кодирует ТФ с доменом “цинкового пальца”) [6]. Речь идет об эволюционно консервативном генном регуляторном мотиве IKZF1 → CASZ1, прослеживаемом ниже по эволюционной лестнице (наиболее детально исследована пара гомологов этих генов hb → cas у D. melanogaster [6].) Показано, что белок Castor супрессирует рост нейробластомы у млекопитающих и человека [7].
Семейство факторов, называемых круппельподобными (Krüppel-like), как и их гены (KLF), найдено у млекопитающих и человека и привлекает внимание исследователей в области биомедицины [8]. До сих пор остается открытым вопрос об истинности гомологии KLF с генами Krüppel (Kr) дрозофилы. У человека насчитывается 17 генов KLF и они вовлечены во многие процессы клеточного роста и дифференцировки [2]. Кодируемые ими сайтспецифические ТФ характеризуются доменами типа “цинкового пальца”. Известно, что KLF6 супрессирует рост опухоли простаты [8], а KLF4 входит в состав “магической четверки” факторов (KLF4, OCT4, SOX2 и MYC), которые могут перепрограммировать обычные дифференцированные клетки человека в стволовые [9]. У дрозофилы ген Kr, наряду с hb, формирует детально исследованное функциональное ядро сети генов gap в эмбриональной сегментации и сети временно́й спецификации эмбриональных нейробластов [10].
Еще одно семейство, функции которого в нейрогенезе консервативны, включает гены pdm-1 и pdm-2. Эти гены кодируют транскрипционные POU-факторы у дрозофилы (известны как Brn1 и Brn2 у млекопитающих), которые получили название по характерному для них гомеодомену типа POU. Они вовлечены в нейрогенез и у дрозофилы, и у позвоночных (в том числе у млекопитающих) [11, 12].
Рассмотренные выше гены (hb, Kr, pdm, cas) вовлечены в две сходные по функциональной организации генные сети в эмбриогенезе дрозофилы (рис. 1). Первая сеть играет ключевую роль в механизмах ранней эмбриональной сегментации и содержит гены дрозофилы класса gap и gap-like [13, 14]. Вторая сеть относится к временнóй спецификации нейробластов эмбриона дрозофилы [15].
Рис. 1.
Соответствия между пространственной последовательностью экспрессии генов дрозофилы hunchback (hb), Krüppel (Kr), POU-domain (pdm) и castor (cas) вдоль передне-задней (A→P) оси раннего эмбриона (а) и временнóй последовательностью их экспрессии в дочерних клетках нейробластов (б) при эмбриональном нейрогенезе (НБ – нейробласт, ГМК – ганглиолярная материнская клетка, t1–t3 – временные интервалы экспрессии; указана апикально-базальная ось). На рисунках (а) представлены воспроизведенные программой PointCloudXplore [16] изображения паттернов экспрессии генов у раннего эмбриона. Верхний – схема эмбриона с клеточным разрешением; нижний – 3D-реконструкция. Механизмы временнóй спецификации производят цепочки дочерних дифференцирующихся клеток с пространственной последовательностью экспрессии hb, Kr, pdm, cas в том же порядке, что и вдоль оси раннего эмбриона.
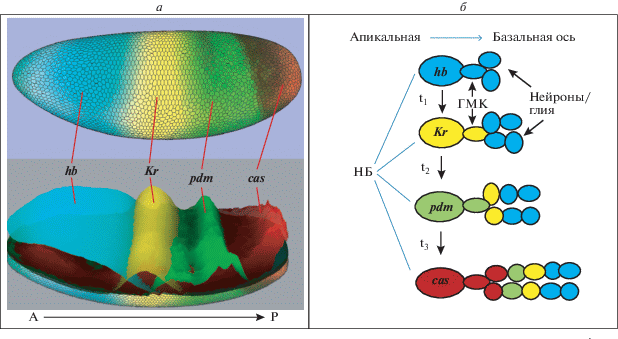
Isshiki и др. [10], первые исследователи, охарактеризовавшие роль ансамбля hb–Kr–pdm–cas в процессах временнóй спецификации (см. также [15]), сразу обратили внимание на соответствие пространственного и временнóго паттернов экспрессии генов этого ансамбля в эмбриональной сегментации и эмбриональном нейрогенезе (см. рис. 1). Некоторые авторы предполагают [15], что это соответствие обусловлено эволюционной консервативностью функций рассматриваемого генного ансамбля, хотя четких доказательств этой гипотезе пока нет.
В этой обзорно-теоретической статье мы обсуждаем и обосновываем возможные пути и молекулярные механизмы эволюционного перехода ансамбля hb–Kr–pdm–cas в его функции как сети временнóй спецификации эмбриональных нейробластов к его функции пространственного паттернинга (spatial patterning)11 раннего эмбриона (подсеть генной сети эмбриональной сегментации) (рис. 1). В процессе сегментации ансамбль формирует и поддерживает пространственную последовательность четырех доменов экспрессии генов hb, Kr, pdm и cas вдоль передне-задней оси раннего эмбриона (рис. 1а). В функции сети временнóй спецификации этот ансамбль в той же последовательности (hb → Kr → pdm → cas) экспрессируется во времени в эмбриональных нейробластах при серии их асимметричных делений в процессах эмбрионального нейрогенеза (рис. 1б).
Эволюционный анализ функций рассматриваемой генной регуляторной сети (ГРС) имеет особое значение, поскольку ее роль в процессах временнóй спецификации нейробластов высоко консервативна – вплоть до млекопитающих и человека. Медико-биологическое значение анализа этого консервативного механизма в том, что мутации генов ансамбля hb–Kr–pdm–cas приводят к нарушениям развития нервной системы. Большое значение имеет и то, что у млекопитающих и человека гены IKZF1 (гомолог гена hb дрозофилы), CASZ1 (гомолог cas) и, возможно, Brn1 и Brn2 (гомологи pdm-1 и pdm-2) относятся к генам-супрессорам опухолевого роста [5, 7].
АНСАМБЛЬ hb–Kr–pdm–cas В НЕЙРОГЕНЕЗЕ И СЕГМЕНТАЦИИ ДРОЗОФИЛЫ
Эмбриональный нейрогенез
Пространственный и временно́й паттернинг в нейрогенезе. Один из фундаментальных вопросов в нейробиологии развития – каким образом возникает поразительное разнообразие нейронов и глиальных клеток во взрослом мозге из небольшого числа нейронных стволовых клеток в эмбрионе [15]. Пространственный паттернинг нервных стволовых клеток22, достигаемый при помощи различных морфогенов и сигнальных каскадов, способствует появлению нейронного разнообразия [17, 18]. Более того, одиночные нейральные стволовые клетки могут генерировать различные типы нейронов в стандартном (стереотипном) порядке посредством временно́го паттернинга33 экспрессии регуляторных факторов в нейральных стволовых клетках.
Только относительно недавно стали понятны молекулярные механизмы, посредством которых отдельные клетки-предшественницы (прогениторные клетки) генерируют последовательность разных типов дочерних клеток. Этот процесс получил название временнóго паттерна, или временнóй спецификации (temporal-identity specification).
Паттернинг нейроэпителия дрозофилы. Нейронные стволовые клетки называются нейробластами (НБ). НБ дрозофилы обычно подвергаются нескольким циклам асимметричных делений, каждый раз продуцируя самообновляющийся НБ и небольшую дочернюю клетку, называемую ганглиолярной материнской клеткой (ГМК) (рис. 2). ГМК обычно делятся асимметрично один раз, в результате чего образуются две постмитотические дочерние клетки с разными судьбами [19–21].
Рис. 2.
Схема нейрогенеза в вентральном нервном стволе (ВНС) у эмбрионов дрозофилы, асимметричное деление нейробластов и временнáя спецификация их потомства. Три стадии нейрогенеза при формировании ВНС: ранний нейроэпителий (1), отслойка нейробластов (2) и генерация нейробластами различных видов нейронов на примере нейробласта 7-1 (NB7-1) (3). ГМК – ганглиолярная материнская клетка. (См. детали в тексте.)
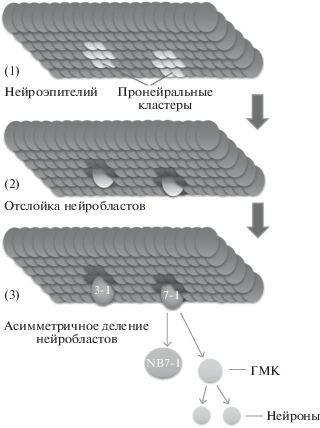
Не так давно дрозофила стала действенной модельной системой для изучения механизмов временнóй спецификации нейронов, что обсуждается в нескольких обширных и исчерпывающих обзорах по этой теме [22–25]. Несколько линий НБ в центральной нервной системе (ЦНС) дрозофилы хорошо охарактеризованы. Они генерируют несколько различных клеточных потомств в зависимости от порядка появления во времени [10, 26–34].
Паттернинг на основе генов gap в ранней сегментации запускает процессы все большей детализации эмбриональных пластов [35], что в результате приводит к детерминации клеток, дающих начало эмбриональной нервной системе [17, 33, 35–40]. А те же гены семейства gap заново активируются в этих клетках, что обеспечивает непрерывность процессов развития.
Генетический контроль временнóй спецификации. В результате исследований эмбрионального вентрального нервного ствола (cord) дрозофилы (ВНС) выявлены молекулярные механизмы временнóй спецификации, где ряд факторов последовательно экспрессируется в нейробластах по мере их созревания [41].
В раннем развитии ВНС зародыша дрозофилы 30 зародышевых НБ обособляются в каждом полусегменте в определенном порядке и отвечают за появление всех нейронов и глии ВНС [42–44] (рис. 2). Эти НБ претерпевают множественные циклы асимметричного деления, производя дочерние НБ и ГМК, которые в итоге делятся на два нейрона или глиальные клетки.
НБ образуют слой на вентральной поверхности ЦНС и их клетки-потомки раннего происхождения вытесняются клетками-потомками более позднего происхождения, что приводит к формированию “ламинарной” (“многослойной”) ЦНС, отражающей порядок появления нейронов – так что каждому типу нейронов соответствует свой слой [10] (рис. 1 и 2).
Каждый НБ уникально идентифицируется при помощи специфических молекулярных маркеров, а также своей конкретной позицией – положением в ламинарной структуре [27, 31, 32]. Более того, каждый конкретный НБ всегда порождает воспроизводимый набор нейронных клеток-потомков в порядке их появления [22, 27, 31, 32, 45]. Эти характеристики позволили детально изучить отдельные линии НБ и разработать уникальную платформу для идентификации и характеристики кандидатов в факторы временнóй спецификации.
Ансамбль hb–Kr–pdm–cas – одна из трех наиболее изученных ГРС, контролирующих у дрозофилы временну́ю спецификацию нейронов (или предетерминацию нейронов их очередностью как дочерних клеток, Birth-Order-Dependent Neuron Fates). Два других каскада временнóй спецификации в нейрогенезе дрозофилы менее изучены.
Факторы временнóй спецификации впервые идентифицировали при наблюдении за ламинарной экспрессией фактора Hb с одновременной экспрессией факторов Nubbin (Pdm-1), Pdm-2 и Cas в зрелой ЦНС дрозофилы [46]. Впоследствии показали, что большинство НБ в эмбриогенезе также последовательно экспрессируют Hb, Kr, Pdm и Cas [10, 41].
Постмитотическое клеточное потомство, возникающее в течение каждого временнóго окна44, поддерживает экспрессию специфически “своего” ТФ (рис. 1б) [10, 46–50]. Хотя различные НБ в ЦНС формируют слои и начинают дифференцироваться в разное время, в большинстве из них ТФ экспрессируются в одной и той же последовательности. Показано, что последовательная экспрессия ТФ необходима и достаточна для предетерминации нейронов в нескольких различных линиях НБ [10, 22, 45–47, 51–54].
ТФ Grainyhead (Grh) (характеризуется мотивом basic helix-loop-helix) считается кандидатом в факторы временнóй спецификации – он экспрессируется после Cas во многих линиях НБ [48, 55, 56]. В эмбриональной линии NB5-6 этот фактор необходим для предетерминации более поздно появляющихся нейронов [47].
Таким образом, серия ТФ, последовательно экспрессирующихся в НБ, контролирует последовательную генерацию различных типов нейронов в нескольких линиях НБ. Разные НБ генерируют разные линии, поэтому эти ТФ не идентифицируют определенный тип нейрона, но контролируют зависящую от порядка появления нейронную идентичность. Временнáя спецификация интегрируется с пространственной идентичностью НБ во всех сегментах эмбриона и, в итоге, предопределяет конкретные типы будущих нервных клеток. Например, Hb контролирует судьбы появившихся первыми клеток в нескольких линиях, которые могут быть моторными нейронами, интернейронами или глиальными клетками – в зависимости от линии НБ [10].
Кроме того, появляется все больше данных, из которых можно сделать вывод, что гены рассматриваемого ансамбля участвуют в роли контролеров временнóй спецификации и в других системах нейрогенеза дрозофилы [56–59].
По имеющимся на сегодня данным, белки Hb, Kr и Cas можно рассматривать в качестве истинных факторов временнóй спецификации, так как они определяют идентичность потомства НБ на основании сроков их появления в нескольких линиях НБ. Для выяснения вопроса о возможности использования Grh в качестве фактора временнóй спецификации для НБ необходимо проанализировать много линий.
Эмбриональная сегментация
Гены hb, Kr, pdm и cas относятся к классу туловищных (trunk) генов семейства gap (hb и Kr) или к группе gap-подобных генов (pdm и cas). Гены hb, Kr и pdm участвуют в процессах сегментации ранних эмбрионов (рис. 1а); причем, пара hb и Kr функционально охарактеризована в этой роли для многих модельных видов насекомых (см. обзор [13]). Пара функционально и структурно близких генов pdm-1 (nubbin) и pdm-2 регулируется генами hb и Kr и участвует в контроле других генов сегментации [14]. Роль cas в сегментации пока не исследована. Еще один ген, grh, замыкающий функциональный каскад hb–Kr–pdm–cas в некоторых НБ, тоже вовлечен в процессы раннего паттернинга эмбриона [60, 61].
Семейства генов объединены под названием gap (пробел, брешь) из-за того, что их нокаут приводит к выпадению в эмбриональной сегментации сразу нескольких соседних сегментов – пробел в их последовательности [13]. К настоящему времени роль генов gap и механизмы действия кодируемых ими сайтспецифических ТФ хорошо исследованы. Это одна из самых изученных ГРС в современной системной биологии. Гены этих семейств начинают экспрессироваться в раннем синцитиальном эмбрионе, формируя к началу стадии целлюляризации достаточно протяженные домены экспрессии вдоль передне-задней оси эмбриона (рис. 1а), в результате чего вдоль этой оси регулярно размещается с десяток таких доменов экспрессии ТФ.
В свою очередь эти домены экспрессии (вместе с первичными морфогенетическими градиентами) обусловливают становление паттернов экспрессии генов семейства pair-rule. Гены pair-rule “считывают” и детализируют начальную разметку доменами ТФ так, что формируют в итоге регулярные семиполосные паттерны экспрессии. Эти наборы семиполосных (периодических) паттернов ТФ семейства pair-rule – первые манифестации закладываемой сегментации эмбриона. Именно в этом роль gap и gap-подобных генов в процессах разметки и закладки эмбриональных сегментов дрозофилы.
Далее остановимся на рассмотрении функции этих генов в процессах эмбрионального паттернинга.
Гены hb и Kr. Это канонические и наиболее исследованные представители семейства туловищных генов gap (туда входят еще гены giant и knirps). Их домены экспрессии (антериорный hb и центральный Kr) соседствуют (рис. 1а), и оба гена, как полагают, взаимно репрессируются кодируемыми ими белками (Hb и Kr соответственно) [13].
Гены pdm-1 и pdm-2. Гены pdm относят к группе gap-подобных [14, 62, 63]. По функциям они сходны с генами семейства gap, но менее изучены. Функционально и структурно тесно связанные гены pdm-1 и pdm-2 сначала экспрессируются на ранней стадии во время целлюляризации в презумптивном брюшном отделе широким доменом, который вскоре подразделяется на две полосы. Эта динамика экспрессии регулируется теми же механизмами (репрессивным действием генов семейства gap), которые определяют домены экспрессии типичного гена семейства gap.
Ген cas. Насколько обширна литература по регуляторным связям гена cas в нейрогенезе (см. следующий раздел), настолько ее мало в области регуляции и функций cas в сегментации. Пожалуй, это только работа Isshiki и др. [10], где показано, что этот ген экспрессируется на стадии клеточной бластодермы (ядерный цикл 14А) широким доменом в задней части эмбриона – последним из генов анализируемого здесь ансамбля (рис. 1а).
Ген grh. Белок Grh синтезируется во время оогенеза и накапливается в развивающемся ооците. Материнская мРНК гена grh транскрибируется в раннем, синцитиальном зародыше. Насколько известно, самое раннее участие Grh в процессах эмбриогенеза – репрессия гена tailless (tll) [60, 61], который тоже относят к классу gap.
Таким образом, только функциональное ядро ансамбля gap + gap-подобных генов изучено в деталях. Это пара взаимно регулирующих друг друга генов hb и Kr и их мишень – гены pdm. Как это ядро связано с генами cas и grh в процессах сегментации, пока непонятно. На уровне наблюдений динамики паттернов экспрессии для нас существенно, что ансамбль генов hb–Kr–pdm–cas формирует домены экспрессии вдоль главной оси раннего эмбриона в определенном порядке.
РЕГУЛЯТОРНАЯ ОРГАНИЗАЦИЯ АНСАМБЛЯ hb–Kr–pdm–cas У ДРОЗОФИЛЫ
Рассмотрение регуляторной организации ансамбля hb–Kr–pdm–cas логично начать с его участия в нейрогенезе, где в отличие от сегментации роль ансамбля изначально исследовалась в целом.
Регуляторные связи ансамбля в нейрогенезе
Как осуществляется в НБ контроль переключений от экспрессии одного ТФ временнóй спецификации к следующему? Взаимная регуляция (кросс-регуляция) между ТФ играет важную роль в этих переключениях [45–48, 64] (рис. 3). Так, показано, что каждый фактор может активировать следующий ген в ансамбле, подавляя при этом ген, следующий за ним через один. Однако потеря Hb или Kr не влияет на продукцию клеток позднего созревания, что может означать наличие еще одного независимого механизма, вносящего свой вклад в последовательную экспрессию факторов временнóй спецификации [53] (рис. 3б).
Рис. 3.
Генные регуляторные сети временнóй спецификации нейронов у дрозофилы. а – Перекрестные регуляторные связи между генами посредством кодируемых ими ТФ для трех каскадов генов, контролирующих события временнóй спецификации у нейробластов или промежуточных нейронов-предшественников дрозофилы. Гены: hth – homothorax, klu – klumpfuss, ey – eyeless, slp – sloppy paired, D – Dichaete, tll – tailless, grh – grainyhead (все кодируют ТФ дрозофилы). б – Детализация регуляторных связей генов hb, Kr, pdm, и cas при их последовательной экспрессии в нейробластах дрозофилы, выявленных в мутантах с полной потерей функции гена (loss-of-function). Стрелки – активация, тупые концы – репрессия, пунктирные стрелки – недоказанные, но предполагаемые регуляторные связи. Svp –фактор Seven up. (См. детали в тексте.)
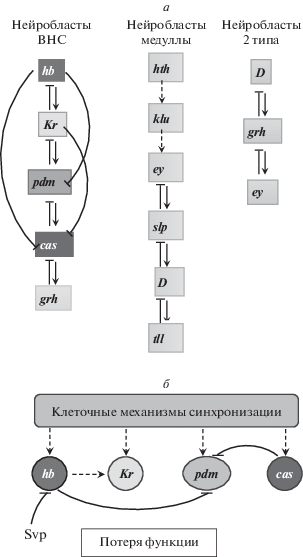
Предполагали, что во временну́ю регуляцию вовлечен механизм клеточных часов [10]. Однако в случае эмбриональной ВНС последовательность экспрессии факторов Hb–Kr–Pdm–Cas–Grh может быть экспериментально воспроизведена в изолированных НБ, культивируемых in vitro, что указывает на внутренний автономный механизм для переключений ТФ [48, 53]. Интересно, что переходы Kr → Pdm → Cas могут происходить в НБ при аресте клеточного цикла [53]. Следовательно, в НБ есть внутренние механизмы синхронизации, не зависящие от прохождения клеточного цикла [48, 53]. Вместе с тем переход от экспрессии Hb к экспрессии Kr требует как цитокинеза НБ, так и экспрессии фактора Seven-up (Svp) (рис. 3) [41, 65, 66].
Существенно, что в большинстве линий потеря Hb, Kr или Pdm не приводит к блокировке временнóй прогрессии, а значит их кросс-регуляция совсем не критичный процесс и в механизм переключения вовлечены другие факторы (рис. 3в). Однако потеря Cas приводит к блокировке временнóй прогрессии факторов (и постоянной во времени экспрессии Pdm) [10, 45, 46, 48, 53, 56].
Таким образом, на сегодня главная задача – идентифицировать конкретные гены/ТФ и регуляторные механизмы, которые вовлечены во временнýю регуляцию.
Регуляторные связи ансамбля в сегментации
Как отмечалось выше, функциональное ядро ансамбля, пара генов hb и Kr, изучены наиболее детально, как и связи этого ядра с генами pdm.
Гены hb и Kr. Подробно описан сложный механизм взаимодействия этой ключевой пары генов, особенность которого состоит в том, что низкие концентрации ТФ активируют ген-мишень, а высокие его репрессируют [67–69]. Такой механизм регуляторного “действия, зависимого от концентрации” (concentration-dependent action), давно обсуждался для фактора Hb на ген Kr и затем подтвержден экспериментально [67]. Показано, что и фактор Hb дрозофилы, и его гомолог Ikaros млекопитающих (вместе с другими представителями семейства) способны образовывать специфические гомо- и гетеродимеры за счет домена димеризации [68]. Предполагают, что мономер Hb/Ikaros действует на ген-мишень как активатор. При повышении внутриклеточных/внутриядерных концентраций Hb/Ikaros формирует димеры, которые и функционируют как репрессоры (см. ниже). Кроме того, активно обсуждали и зависимое от концентрации действие фактора Kr на ген hb [69] и не так давно представлены экспериментальные доказательства реализации такого регуляторного механизма [70–72].
В механизмах динамики экспрессии этой пары генов обсуждаются также аутоактивация гена кодируемым им ТФ. Больше всего экспериментальных данных в поддержку аутоактивации накоплено для гена hb, меньше для Kr [13]. Возможные механизмы ауторегуляции выявлены, в большой степени, благодаря результатам моделирования сети туловищных генов gap [13, 35].
Гены pdm-1 и pdm-2. Границы экспрессии гена pdm-1 устанавливаются терминальной системой генов torso и tailless, а также репрессивным градиентом фактора Hb [14, 62]. А именно, передняя граница широкого домена pdm-1 ограничена зиготическим hb, что указывает на то, что белок Hb прямо или опосредованно репрессирует экспрессию pdm [62]. Разделение домена pdm-1 на две полосы контролируется gap-геном kni. Экспериментально показано (паттерн экспрессии pdm в эмбрионах, мутантных по гену Kr), что фактор Kr, напрямую или опосредованно, репрессирует pdm [14].
Пока нет публикаций по регуляторным связям генов cas и grh с другими генами ансамбля на этой стадии сегментации эмбриона, так что выявить или опровергнуть их существование – одна из предстоящих задач.
Моделирование поведения ансамбля в нейрогенезе и сегментации
Имеется обширная научная литература по моделированию поведения генов семейства gap в эмбриональной сегментации [13, 35, 71, 73, 74], но только Nakajima и соавт. [64] провели моделирование поведения ансамбля временнóй спецификации в эмбриональном нейрогенезе [64].
Регуляторная организация ансамбля в нейрогенезе. Nakajima и соавт. [64] перебрали в компьютерных экспериментах все возможные функциональные организации сети из генов hb–Kr–pdm–cas и численно исследовали все соответствующие им временны́е паттерны экспрессии. В результате этого масштабного анализа выявлены ключевые регуляторные связи между генами. Это позволило получить паттерны их экспрессии в нейрогенезе, как для состояния нормы (дикого типа), так и для мутантных фенотипов (потеря функции или постоянная экспрессия для каждого из генов).
По мнению авторов [64], для более ясного понимания функциональной организации ГРС необходимо сконцентрировать внимание на ее подсетях (такие функционально и структурно обособляемые части сети называют сетевыми мотивами или модулями [75–77]). Сравнивая все выявленные моделированием функциональные сети, авторы обнаружили, что минимальная генная структура, обеспечивающая требуемый временнóй паттернинг, содержит две последовательные регуляторные петли (рис. 4а), одна из которых состоит из hb, Kr и pdm, а другая из Kr, pdm и cas.
Рис. 4.
Функциональная организация ансамбля генов hb–Kr–pdm–cas в нейрогенезе (а) и сегментации (б). Стрелки – активация, тупые концы – репрессия.
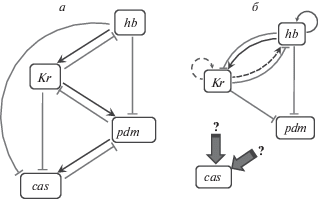
Как видно из рисунков 3 и 4, в каждой петле один ген активирует следующий за ним и подавляет предыдущий: hb репрессирует pdm, а Kr репрессирует cas. Также подавляется ген, следующий за активируемым: pdm репрессирует Kr, а cas репрессирует pdm. Эти взаимодействия и составляют минимальную сеть для последовательной экспрессии (рис. 4а). Nakajima и соавт. [64] показали, что идентифицированных взаимодействий достаточно для воспроизведения последовательной экспрессии в соответствующих условиях, хотя профили экспрессии могут быть легко возмущены при изменении параметров или увеличении уровня шума.
Важное заметить, что в результате численных экспериментов с огромным массивом решений модели Nakajima и соавт. пришли к заключению, что в сети временнóй спецификации должен действовать, как минимум, еще один (все еще не охарактеризованный) ТФ – только тогда модель объясняет устойчивость временнóго паттернинга, как к возмущениям, так и к нокаутам генов ансамбля. Но это умозаключение до сих пор остается гипотетическим – экспериментального подтверждения пока нет.
Регуляторная организация ансамбля в сегментации. Резюмируя все, что известно о роли рассматриваемого генного ансамбля в сегментации (см. ссылки в подразделе “Эмбриональная сегментация”), можно заключить, что первый регуляторный контур (гены hb, Kr и pdm) явно виден и в механизмах сегментации, отличаясь существенно от нейрогенеза только тем, что ген Kr (по-видимому) не активирует ген pdm, а репрессирует его (рис. 4б).
Ранее нами разработана детальная модель динамики паттерна экспрессии гена hb в эмбриональной сегментации в норме (дикий тип) и на фоне двух мутаций (нокаутов) – по генам Kr и kni [74] – и показано, что гены из группы gap-подобных не только служат мишенями для регуляторных действий основных четырех генов gap, но и сами регулируют эти основные гены, в частности, ген hb.
На сегодняшний день нет сведений о регуляции экспрессии гена cas в процессах сегментации.
Таким образом, результаты моделирования временнóй регуляции процессов нейрогенеза и сегментации согласуются с экспериментальные данными, что и в сегментации, и в нейрогенезе пара генов hb–Kr образует функциональное ядро ансамбля, в котором также участвует ген pdm.
На основании системно-биологического анализа ансамбля ГРС в сегментации и нейрогенезе можно предположить, что некоторые механизмы регуляции, присущие временны́м факторам, например, зависящее от концентрации действие (возможно, связанное со способностями ТФ к димеризации [45]) на гены-мишени, реализуются в обоих этих процессах. Кроме того, наличие регуляторных петель аутоактивации, возможно, способствует устойчивости функционирования сетей временнóй спецификации.
Эволюция регуляторных механизмов на примере Hb/Ikaros
Способностью к эволюции обладают не только ГРС, но также и ТФ, причем некоторые из характеристик гомологичных факторов изменяются в эволюции, а другие остаются консервативными.
Самый исследованный представитель семейства Ikaros, Hb дрозофилы, имеет несколько консервативных доменов, функции которых частично охарактеризованы. Этот ТФ репрессирует и активирует гены-мишени по нескольким механизмам [78, 79], что схематично представлено на рис. 5а. В одном из механизмов репрессии участвуют ДНК-связывающий домен (DBD, DNA-binding domain) Hb, который специфически связывает определенный сайт ДНК, и домен димеризации (DMZ, dimerization domain), который отвечает за специфическую димеризацию двух молекул Hb. В другой механизм репрессии вовлечен еще один домен Hb, специфически связывающий фактор Mi-2 и поэтому получивший название Mi2-связывающий [79].
Рис. 5.
Молекулярные механизмы действия фактора Hb/Ikaros на гены-мишени. а – Варианты молекулярных механизмов регуляции генов-мишеней фактором Hunchback (Hb) в процессах сегментации и в нейробластах дрозофилы: активация (или коактивация) мономером и репрессия (корепрессия) димером. A – активатор или коактиватор, R – репрессор или корепрессор. Рисунок из работы [78] приведен с разрешения издательства. б – Возможные варианты (изо)димеров изоформ фактора Ikaros и его гетеродимеров с гомологами (например, с Helios и Aiolos), которые специфически связываются с определенными последовательностями ДНК регуляторных областей генов. (См. детали в тексте.)
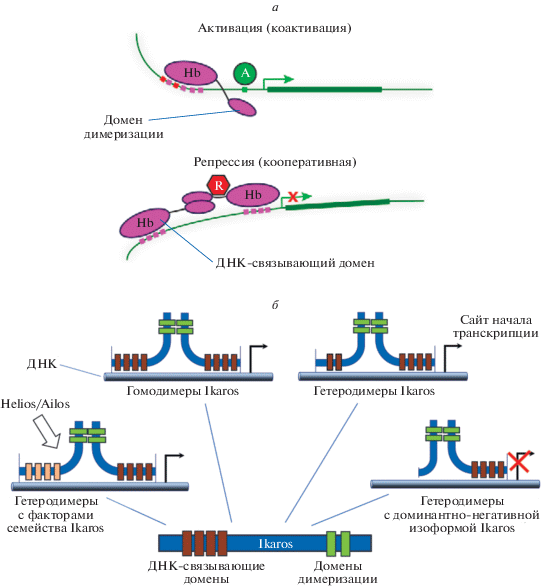
Из эволюционно консервативных особенностей факторов Ikaros, прежде всего, стоит обратить внимание на наличие доменов димеризации в их структуре [80] и взаимодействие с фактором Mi-2 [81] (рис. 5). Вовлеченность Hb/Ikaros в регуляцию гомеозисных генов посредством модуляции хроматина в ансамбле с кофакторами (включая Mi-2) эволюционно консервативна и прослеживается от дрозофилы до человека. Однако однозначного ответа на вопрос о вовлечении этих механизмов в контроль временнóй спецификации у дрозофилы и у млекопитающих пока нет.
Важно отметить, что способность белка Hb формировать димеры и этим модулировать регуляторное действие существенно расширена у млекопитающих. Во-первых, у млекопитающих ген Ikzf (аналог hb дрозофилы) за счет альтернативного сплайсинга кодирует не единственный ТФ, а серию близкородственных факторов [80], что существенно расширяет возможные сценарии и механизмы действия факторов Ikaros (рис. 5). Возможно, одни изоформы действуют как активаторы, а другие как репрессоры [80]. Во-вторых, ТФ семейства Ikaros формируют димеры (и даже мультимеры) не только между альтернативными факторами, кодируемыми данным геном, но и факторами, кодируемыми его гомологами (рис. 5б). Таким образом, факторы семейства Ikaros можно рассматривать как пример консерватизма и эволюции молекулярной машинерии регуляторики сайтспецифическими ТФ их генов-мишеней.
ЭВОЛЮЦИЯ ГЕННОЙ РЕГУЛЯТОРНОЙ СЕТИ ВРЕМЕННÓЙ СПЕЦИФИКАЦИИ НЕЙРОБЛАСТОВ
Принято считать, что на ранней стадии эволюции метазоа генные сети, контролирующие конкретные процессы онтогенеза, могли состоять не более чем из двух или трех взаимодействующих генов. Со временем эти небольшие генные сети дополнялись включением новых генов [82, 83]. Событие, когда некоторый ген, задействованный в каком-то конкретном процессе или механизме в предковом организме, оказывается вовлеченным у вида-потомка еще и в другой процесс или механизм, в литературе определяют термином “кооптация” (cooptation), который и будет использован далее.
В ходе эволюции процесса сегментации у беспозвоночных происходило неоднократное и независимое кооптирование генных регуляторных модулей, которое сохраняло эволюционно самые древние функции этих генов в детерминации осей тела и в удлинении передне-задней оси [84, 85]. Специфически характерные свойства некоторых из этих ГРС делали их легко кооптируемыми в тех ситуациях, когда требовалось создание повторяющихся паттернов экспрессии (для закладки периодических структур – сегментов, или метамеров).
Например, гены семейства gap уже не одно десятилетие находятся в фокусе исследований по эволюционной эмбриологии (область evo-devo). Авторы сходятся во мнении, что сегментация не относится к первичной, эволюционно древней, функции генов gap – скорее всего, произошла кооптация этих генов в процессы эмбриональной сегментации [86–89]. Наиболее консервативная и поэтому, наверное, наиболее древняя роль генов gap заключается, как полагают, в нейрогенезе и в паттернинге головного отдела эмбриона [10, 90–94].
Обнаруженные и наиболее интенсивно изученные у дрозофилы, эти гены начали участвовать в формировании периодических пространственных паттернов экспрессии уже на уровне эволюции членистоногих. Некоторые гены, такие как engrailed и even-skipped, играли эволюционно древнюю роль в формировании нейрального паттерна (тоже сегментного) и, по-видимому, позднее были кооптированы в механизм сегментации передне-задней оси тела (у дрозофилы). Ряд генов сегментации функционируют как в нейрогенезе, так и в сегментации [95, 96], включая gap-гены hb и Kr [13, 73, 97].
Ключевые гены ансамбля, Kr и hb, вовлечены во многие эмбриональные процессы у беспозвоночных и позвоночных [1, 6, 8, 50, 80, 90–94] и, в ряде наиболее исследованных случаев [1, 90, 91, 93], именно как пара взаимосвязанных генов.
Возможно, именно ГРС нервной системы обеспечивают важный эволюционный источник полезных генных компонентов (как отдельных генов, так и целых модулей генных сетей), которые функционируют в регуляторных процессах онтогенеза [10, 86–94].
Как обсуждалось, эмбриональная сегментация определяется эмбриональным паттернингом генов сегментации и одним из ее первых проявлений считают закладку парасегментных борозд [98]. Существенно, что ВНС закладывается согласно парасегментной организации, так что каждый эмбриональный ганглий формируется между соседними парасегментными бороздами. Когда позднее формируется сегментная организация имаго (границы сегментов смещены относительно границ парасегментов), ганглии ВНС остаются локализованными согласно исходной парасегментной организации [98, 99]. Тот факт, что гены семейства gap участвуют не только в сегментации, но и в нейрогенезе, дает новые аргументы в пользу первичности в эволюции ГРС нейрональной метамеризации, кооптированой позднее в механизмы сегментации.
Эволюционная консервативность механизмов временнóй спецификации
Нейронные клетки-предшественники в развивающейся ЦНС млекопитающих также, как и у дрозофилы, производят нейронное потомство в заданном воспроизводимом порядке. В исследованиях ЦНС позвоночных, особенно коры головного мозга и сетчатки, показано, что временнóй порядок появления дочерних клеток коррелирует с определенной специфичностью дифференцирующихся из них нейронов/глиальных клеток (обзоры [22, 100–103]). Хотя для правильной временнóй спецификации требуются как внешние, так и внутренние факторы, изолированные нейрональные стволовые клетки, культивируемые in vitro, могут повторять последовательную генерацию различных типов нейронов, что акцентирует наше внимание на внутриклеточном временнóм контроле этих стволовых клеток [104–106].
В экспериментах in vivo обнаружено, что нейрональные клетки-предшественники в сетчатке позвоночных мультипотентны и генерируют предетерминированные типы клеток в характерном порядке [107–109]. При анализе транскриптомов единичных клеток-предшественников сетчатки на разных стадиях развития выявлена последовательная экспрессия ТФ, гомологичных факторам временнóй спецификации у дрозофилы [110].
Так, фактор Ikaros экспрессируется самыми ранними клетками, генерируемыми клетками-предшественниками сетчатки, и необходим для спецификации самых ранних типов клеток нейронов/глии. Ортолог Ikaros у дрозофилы, Hb, выполняет сходную функцию [111]. Более того, недавно в клетках-предшественниках сетчатки мыши идентифицирован белок zinc finger protein castor homolog 1, Casz1 (гомолог Cas), как поздний фактор временной спецификации [112]. Ген Casz1, в свою очередь, регулируется ранним фактором Ikaros – Ikzf1.
На основании этих данных – что пара регуляторно связанных генов hb/Ikzf1–cas/Casz1 сохраняется в ЦНС дрозофилы и в сетчатке мыши – можно говорить об их эволюционной значимости [6]. Эти ГРС могут быть консервативным модулем, функционирующим в нейрогенезе эволюционно далеких видов.
В результате сравнительного эволюционного анализа выявлено [105, 113], что и некоторые другие ТФ сохраняют в эволюции свою роль в обеспечении спецификации нейронов. Так, для временнóй спецификации нейральных стволовых клеток и нервных клеток-предшественников у позвоночных оказались необходимы факторы COUP-TFI и COUP-TFII (Сhicken Оvalbumin Upstream Promoter-Transcription Factor I/II). COUP-TFI и COUP-TFII позвоночных – гомологи фактора Svp, вовлеченного во временну́ю спецификацию дрозофилы. У эмбрионов мыши нокдаун генов, кодирующих COUP-TFI и COUP-TFII в развивающемся кортикальной слое приводит к образованию дополнительных ранних нейронов за счет утраты поздних нейронов; причем эти ранние нейроны экспрессируют ТФ BRN2 [105]. Белки BRN1 и BRN2 человека гомологичны факторам Pdm1 и Pdm2 дрозофилы, которые необходимы для генерации поздних нейронов [113].
Таким образом, накапливается все больше данных по эволюционно консервативным генным регуляторным модулям в нейрогенезе – от членистоногих до человека; прежде всего, это пары hb/IKZF1–cas/CASZ1 и svp/COUP-TF–pdm/BRN.
ЗАКЛЮЧЕНИЕ
Ключевые гены, hb и Kr, ядра рассматриваемого генного регуляторного ансамбля двукрылых насекомых в функциях контроля нейрогенеза описаны уже на уровне беспозвоночных, а именно, у членистоногих, что свидетельствует об удачно найденной регуляторной архитектуре.
При сравнительном эволюционном анализе выявлено, что ансамбль генов, ядро которого составляют гены hb и Kr, был кооптирован в ходе эволюции в процессы сегментации, по крайней мере, у некоторых таксонов насекомых (скорее всего, независимо и неоднократно). Кооптированию, по-видимому, способствовало то, что эволюционно первичные функции этих генов связаны с метамерной организацией (сегментированностью) нервной системы у членистоногих.
Функциональная, регуляторная организация ансамбля (особенно в ядре из генов hb, Kr и/или pdm или cas), найденная в эволюции еще у предковых форм членистоногих, оказалась настолько удачной, что прослеживается в эволюции вплоть до млекопитающих и человека в роли контроля временной спецификации нейробластов.
К эволюционно консервативным чертам регуляторной организации ансамбля следует отнести, прежде всего, взаимную перекрестную репрессию генов в ансамбле. Она особенно наглядна и хорошо охарактеризована для ядра hb–Kr–pdm у дрозофилы в обеих его функциях (сегментация и нейрогенез).
Из конкретных молекулярных механизмов контроля генов в ансамбле важным представляется, в первую очередь, консерватизм домена для димеризации факторов семейства Ikaros и роли димеризации в контроле активности генов-мишеней IKZF/hb.
Плотная сеть перекрестных репрессивных связей в рассматриваемом ансамбле, как показано в системно-биологических исследованиях, обеспечивает устойчивость функционирования такой ГРС как к внешним возмущениям, так и к внутренним шумам.
Логично ожидать, что охарактеризованные высоко консервативные свойства регуляторной организации рассматриваемого ансамбля могут воспроизводиться в других ГРС временнóй спецификации. Прежде всего, это генные каскады hth–klu–ey–slp–D-tll нейробластов медуллы (отдел зрительной доли мозга) и D-grh–ey нейробластов типа 2 личинки дрозофилы (см. рис. 3а). Таким образом, необходимо дальнейшее системно-биологическое исследование этих модельных объектов.
Знание механизмов формирования паттернов экспрессии во времени важно для понимания процессов нейрогенеза. Это позволит выяснить, как пространственные и временны́е сигналы интегрируются для генерации специфических типов клеток и как созревающие клетки-предшественники изменяют компетентность для генерации определенного типа клеток в определенный момент времени. А значит, будут детализированы наши представления о процессах дифференцировки нейрональных клеток, что в дальнейшем будет использовано в прикладной сфере – нейробиологии и медицине.
Работа проводилась в рамках государственного задания ФАНО России (проект № 01201351572) и поддержана грантом № 16.8549.2017/8.9 Министерства образования и науки Российской Федерации.
Список литературы
Pinnell J., Lindeman P.S., Colavito S., Lowe C., Savage R.M. (2006) The divergent roles of the segmentation gene hunchback. Integr. Comp. Biol. 46, 519–532.
John L.B., Yoong S., Ward A.C. (2009) Evolution of the Ikaros gene family: implications for the origins of adaptive immunity. J. Immunol. 182(8), 4792–4799. doi 10.4049/jimmunol.0802372
Georgopoulos K., Moore D.D., Derfler B. (1992) Ikaros, an early lymphoid-specific transcription factor and a putative mediator for T cell commitment. Science. 258(5083), 808–812.
Hahm K., Ernst P., Lo K., Kim G.S., Turck C., Smale S.T. (1994) The lymphoid transcription factor LyF-1 is encoded by specific, alternatively spliced mRNAs derived from the Ikaros gene. Mol. Cell. Biol. 14(11), 7111–7123.
Kastner P., Chan S. (2011) Role of Ikaros in T-cell acute lymphoblastic leukemia. World J. Biol. Chem. 2(6), 108–114.
Konstantinides N., Rossi A.M., Desplan C. (2015) Common temporal identity factors regulate neuronal diversity in fly ventral nerve cord and mouse retina. Neuron. 85(3), 447–449.
Virden R.A., Thiele C.J., Liu Z. (2012) Characterization of critical domains within the tumor suppressor CASZ1 required for transcriptional regulation and growth suppression. Mol. Cell. Biol. 32(8), 1518–1528.
McConnell B.B., Yang V.W. (2010) Mammalian Krüppel-like factors in health and diseases. Physiol. Rev. 90(4), 1337–1381. doi 10.1152/physrev.00058.2009
Welstead G.G., Brambrink T., Jaenisch R. (2008) Generating iPS cells from MEFS through forced expression of Sox-2, Oct-4, c-Myc, and Klf4. J. Vis. Exp. pii: 734. doi 10.3791/734
Isshiki T., Pearson B., Holbrook S., Doe C.Q. (2001) Drosophila neuroblasts sequentially express transcription factors which specify the temporal identity of their neuronal progeny. Cell. 106, 511–521.
Dominguez M.H., Ayoub A.E., Rakic P. (2013) POU-III transcription factors (Brn1, Brn2, and Oct6) influence neurogenesis, molecular identity, and migratory destination of upper-layer cells of the cerebral cortex. Cereb. Cortex. 23(11), 2632–2643.
Gold D.A., Gates R.D., Jacobs D.K. (2014) The early expansion and evolutionary dynamics of POU class genes. Mol. Biol. Evol. 31, 3136–3147.
Jaeger J. (2011) The gap gene network. Cell. Mol. Life Sci. 68(2), 243–274.
Cockerill K.A., Billin A.N., Poole S.J. (1993) Regulation of expression domains and effects of ectopic expression reveal gap gene-like properties of the linked pdm genes of Drosophila. Mech. Dev. 41,139–153.
Li X., Chen Z., Desplan C. (2013) Temporal patterning of neural progenitors in Drosophila. Curr. Topics Dev. Biol. 105, 69–96. http://dx.doi.org/10.1016/ B978-0-12-396968-2.00003-8.
Rübel O., Weber G.H., Keränen S.V.E., Fowlkes C.C., Luengo Hendriks C.L.L., Simirenko L., Shah N.Y., Eisen M.B., Biggin M.D., Hagen H., Sudar D., Malik J., Knowles D.W., Hamann B. (2006) PointCloudXplore: a visualization tool for 3D gene expression data. In: Visualization of Large and Unstructured Data Sets, GI Lecture Notes in Informatics. Eds Hagen H., Kerren A., Dannenmann P. vol. S-4. Bonn, Germany: Gesellschaftfuer Informatik (GI), pp. 107–117.
Bhat K.M. (1999). Segment polarity genes in neuroblast formation and identity specification during Drosophila neurogenesis. Bioessays. 21, 472–485.
Dessaud E., McMahon A.P., Briscoe J. (2008) Pattern formation in the vertebrate neural tube: a sonic hedgehog morphogen-regulated transcriptional network. Development. 135, 2489–2503.
Doe C.Q. (2008) Neural stem cells: balancing self-renewal with differentiation. Development. 135, 1575–1587.
Knoblich J.A. (2010) Asymmetric cell division: recent developments and their implications for tumour biology. Nat. Rev. Mol. Cell Biol. 11, 849–860.
Lin S., Lee T. (2012) Generating neuronal diversity in the Drosophila central nervous system. Dev. Dyn. 241, 57–68.
Pearson B.J., Doe C.Q. (2004) Specification of temporal identity in the developing nervous system. Annu. Rev. Cell Dev. Biol. 20, 619–647.
Kao C.F., Lee T. (2010) Birth time/order-dependent neuron type specification. Curr. Opin. Neurobiol. 20, 14–21.
Brand A.H., Livesey F.J. (2011) Neural stem cell biology in vertebrates and invertebrates: more alike than different? Neuron. 70, 719–729.
Maurange C. (2012) Temporal specification of neural stem cells: insights from Drosophila neuroblasts. Curr. Top. Dev. Biol. 98, 199–228.
Baek M., Mann R.S. (2009) Lineage and birth date specify motor neuron targeting and dendritic architecture in adult Drosophila. J. Neurosci. 29, 6904–6916.
Bossing T., Udolph G., Doe C.Q., Technau G.M. (1996) The embryonic central nervous system lineages of Drosophila melanogaster. I. Neuroblast lineages derived from the ventral half of the neuroectoderm. Dev. Biol. 179, 41–64.
Jefferis G.S., Marin E.C., Stocker R.F., Luo L. (2001) Target neuron prespecification in the olfactory map of Drosophila. Nature. 414, 204–208.
Karcavich R., Doe C.Q. (2005) Drosophila neuroblast 7-3 cell lineage: a model system for studying programmed cell death, Notch/Numb signaling, and sequential specification of ganglion mother cell identity. J. Comp. Neurol. 481, 240–251.
Pearson B.J., Doe C.Q. (2003) Regulation of neuroblast competence in Drosophila. Nature. 425, 624–628.
Schmid A., Chiba A., Doe, C.Q. (1999) Clonal analysis of Drosophila embryonic neuroblasts: neural cell types, axon projections and muscle targets. Development. 126, 4653–4689.
Schmidt H., Rickert C., Bossing T., Vef O., Urban J., Technau G.M. (1997). The embryonic central nervous system lineages of Drosophila melanogaster. II. Neuroblast lineages derived from the dorsal part of the neuroectoderm. Dev. Biol. 189, 186–204.
Skeath J.B., Thor S. (2003) Genetic control of Drosophila nerve cord development. Curr. Opin. Neurobiol. 13, 8–15.
Yu H.H., Kao C.F., He Y., Ding P., Kao J.C., Lee T. (2010) A complete developmental sequence of a Drosophila neuronal lineage as revealed by twin-spot MARCM. PLoS Biol. 8, e1000461.
Jaeger J., Manu, Reinitz J. (2012) Drosophila blastoderm patterning. Curr. Opin. Genet. Dev. 22, 533–541.
Skeath, J.B. (1999) At the nexus between pattern formation and cell-type specification: the generation of individual neuroblast fates in the Drosophila embryonic central nervous system. Bioessays. 21, 922–931.
Martin-Bermudo M.D., Martinez C., Rodriguez A., Jimenez F. (1991) Distribution and function of the lethal of scute gene product during early neurogenesis in Drosophila. Development. 113, 445–454.
Skeath J.B., Carroll S.B. (1992). Regulation of proneural gene expression and cell fate during neuroblast segregation in the Drosophila embryo. Development. 114, 939–946.
Technau G.M., Berger C., Urbach R. (2006) Generation of cell diversity and segmental pattern in the embryonic central nervous system of Drosophila. Dev. Dyn. 235, 861–869.
Hwang H.L., Rulifson E. (2011) Serial specification of diverse neuroblast identities from acneurogenic placode by Notch and Egfr signaling. Development. 138, 2883–2893.
Kohwi M., Doe C.Q. (2013) Temporal fate specification and neural progenitor competence during development. Nat. Rev. Neurosci. 14, 823–838.
Broadus J., Skeath J.B., Spana E.P., Bossing T., Technau G., Doe C.Q. (1995) New neuroblast markers and the origin of the aCC/pCC neurons in the Drosophila central nervous system. Mech. Dev. 53, 393–402.
Doe C.Q., Technau G.M. (1993) Identification and cell lineage of individual neural precursors in the Drosophila CNS. Trends Neurosci. 16, 510–514.
Egger B., Chell J.M., Brand A.H. (2008) Insights into neural stem cell biology from flies. Phil. Trans. R. Soc. B. 363, 39–56.
Tran K.D., Doe C.Q. (2008) Pdm and Castor close successive temporal identity windows in the NB3‑1 lineage. Development. 135, 3491–3499.
Kambadur R., Koizumi K., Stivers C., Nagle J., Poole S.J., Odenwald W.F. (1998) Regulation of POU genes by castor and hunchback establishes layered compartments in the Drosophila CNS. Genes Dev. 12, 246–260.
Baumgardt M., Karlsson D., Terriente J., Diaz-Benjumea F.J., Thor S. (2009) Neuronal subtype specification within a lineage by opposing temporal feed-forward loops. Cell. 139, 969–982.
Brody T., Odenwald W.F. (2000) Programmed transformations in neuroblast gene expression during Drosophila CNS lineage development. Dev. Biol. 226, 34–44.
Grosskortenhaus R., Pearson B.J., Marusich A., Doe C.Q. (2005) Regulation of temporal identity transitions in Drosophila neuroblasts. Dev. Cell. 8, 193–202.
Romani S., Jimenez F., Hoch M., Patel N.H., Taubert H., Jackle H. (1996) Kruppel, a Drosophila segmentation gene, participates in the specification of neurons and glial cells. Mech. Dev. 60, 95–107.
Benito-Sipos J., Estacio-Gomez A., Moris-Sanz M., Baumgardt M., Thor S., Diaz-Benjumea F. J. (2010). A genetic cascade involving klumpfuss, nab and castor specifies the abdominal leucokinergic neurons in the Drosophila CNS. Development. 137, 3327–3336.
Cleary M.D., Doe C.Q. (2006) Regulation of neuroblast competence: multiple temporal identity factors specify distinct neuronal fates within a single early competence window. Genes Dev. 20, 429–434.
Grosskortenhaus R., Robinson K.J., Doe C.Q. (2006) Pdm and Castor specify lateborn motor neuron identity in the NB7-1 lineage. Genes Dev. 20, 2618–2627.
Novotny T., Eiselt R., Urban J. (2002) Hunchback is required for the specification of the early sublineage of neuroblast 7-3 in the Drosophila central nervous system. Development. 129, 1027–1036.
Cenci C., Gould A.P. (2005) Drosophila Grainyhead specifies late programmes of neural proliferation by regulating the mitotic activity and Hox-dependent apoptosis of neuroblasts. Development. 132, 3835–3845.
Maurange C., Cheng L., Gould A.P. (2008) Temporal transcription factors and their targets schedule the end of neural proliferation in Drosophila. Cell. 133, 891–902.
Bayraktar O.A., Doe C.Q. (2013) Combinatorial temporal patterning in progenitors expands neural diversity. Nature. 498, 449–455.
Tsuji T., Hasegawa E., Isshiki T. (2008) Neuroblast entry into quiescence is regulated intrinsically by the combined action of spatial Hox proteins and temporal identity factors. Development. 135, 3859–3869.
Kao C.F., Yu H.H., He Y., Kao J.C., Lee T. (2012) Hierarchical deployment of factors regulating temporal fate in a diverse neuronal lineage of the Drosophila central brain. Neuron. 73, 677–684.
Huang J.D., Dubnicoff T., Liaw G.J., Bai Y., Valentine S.A., Shirokawa J.M., Lengyel J.A., Courey A.J. (1995) Binding sites for transcription factor NTF-1/Elf-1 contribute to the ventral repression of decapentaplegic. Genes Dev. 9, 3177–3189.
Liaw G.J., Rudolph K.M., Huang J.D., Dubnicoff T., Courey A.J., Lengyel J.A. (1995) The torso response element binds GAGA and NTF-1/Elf-1, and regulates tailless by relief of repression. Genes Dev. 9, 3163–3176.
Lloyd A., Sakonju S. (1991) Characterization of two Drosophila POU domain genes, related to Oct-1 and Oct-2, and the regulation of their expression patterns. Mech. Dev. 36, 87–102.
Billin A.N., Cockerill K.A., Poole S.J. (1991) Isolation of a family of Drosophila POU domain genes expressed in early development. Mech. Dev. 34, 75–84.
Nakajima A., Isshiki T., Kaneko K., Ishihara S. (2010) Robustness under functional constraint: the genetic network for temporal expression in Drosophila neurogenesis. PLoS Comput. Biol. 6(4), e1000760.
Kanai M.I., Okabe M., Hiromi Y. (2005) seven-up controls switching of transcription factors that specify temporal identities of Drosophila neuroblasts. Dev. Cell. 8, 203–213.
Mettler U., Vogler G., Urban J. (2006) Timing of identity: spatiotemporal regulation of hunchback in neuroblast lineages of Drosophila by Seven‑up and Prospero. Development. 133, 429–437.
Zuo P., Stanojević D., Colgan J., Han K., Levine M., Manley J.L. (1991) Activation and repression of transcription by the gap proteins hunchback and Krüppel in cultured Drosophila cells. Genes Dev. 5(2), 254–264.
McCarty A.S., Kleiger G., Eisenberg D., Smale S.T. (2003) Selective dimerization of a C2H2 zinc finger subfamily. Mol. Cell. 11(2), 459–470.
Sauer F., Jäckle H. (1991) Concentration-dependent transcriptional activation or repression by Krüppel from a single binding site. Nature. 353(6344), 563–566.
Perry M.W., Bothma J.P., Luu R.D., Levine M. (2012) Precision of Hunchback expression in the Drosophila embryo. Curr. Biol. 22, 1–6.
Kozlov K., Surkova S., Myasnikova E., Reinitz J., Samsonova M. (2012) Modeling of gap gene expression in Drosophila Krüppel mutants. PLoS Comp. Biol. 8, e1002635.
Surkova S., Golubkova E., Manu, Panok L., Mamon L., Reinitz J., Samsonova M. (2013) Quantitative dynamics and increased variability of segmentation gene expression in the Drosophila Krüppel and knirps mutants. Dev. Biol. 376, 99–112.
Jaeger J., Surkova S., Blagov M., Janssens H., Kosman D., Myasnikova E., Vanario-Alonso C.E., Samsonova M., Sharp D.H., Reinitz J. (2004) Dynamic control of positional information in the early Drosophila blastoderm. Nature. 430, 368–371.
Spirov A.V., Myasnikova E.M., Holloway D. (2016) Sequential construction of a model for modular gene expression control, applied to spatial patterning of the Drosophila gene hunchback. J. Bioinform. Comput. Biol. 14, 1641005.
Ishihara S., Fujimoto K., Shibata T. (2005) Cross talking of network motifs in gene regulation that generates temporal pulses and spatial stripes. Genes Cells. 10, 1025–1038.
Fujimoto K., Ishihara S., Kaneko K. (2008) Network evolution of body plans. PLoS One. 3, e2772
Ishihara S., Shibata T. (2008) Mutual interaction in network motifs robustly sharpens gene expression in developmental processes. J. Theor. Biol. 252, 131–144.
Tran K.D., Miller M.R., Doe C.Q. (2010) Recombineering Hunchback identifies two conserved domains required to maintain neuroblast competence and specify early-born neuronal identity. Development. 137, 1421–1430.
Kehle J., Beuchle D., Treuheit S., Christen B., Kennison J.A., Bienz M., Muller J. (1998) dMi-2, a hunchback-interacting protein that functions in polycomb repression. Science. 282, 1897–1900.
Li Z., Perez-Casellas L.A., Savic A., Song C., Dovat S. (2011) Ikaros isoforms: the saga continues. World J. Biol. Chem. 2(6), 140–145.
Arenzana T.A., Schjerven H., Smale S.T. (2015) Regulation of gene expression dynamics during developmental transitions by the Ikaros transcription factor. Genes Dev. 29, 1801–1816.
Peterson K.J., Davidson E.H. (2000) Regulatory evolution and the origin of the bilaterians. Proc. Natl. Acad. Sci. USA. 97, 4430–4433.
Levine M., Tjian R. (2003) Transcription regulation and animal diversity. Nature. 424,147–151.
Arthur W., Jowett T., Panchen A. (1999) Segments, limbs, homology, and co-option. Evol. Dev. 1, 74–76.
Chipman, AD. (2010) Parallel evolution of segmentation by cooption of ancestral gene regulatory networks. Bioessays. 32, 60–70.
Peel A. (2004) The evolution of arthropod segmentation mechanisms. Bioessays. 26, 1108–1116.
Peel A., Chipman A.D., Akam M. (2005) Arthropod segmentation: beyond the Drosophila paradigm. Nat. Rev. Genet. 6, 905–916.
Peel A.D. (2008) The evolution of developmental gene networks: lessons from comparative studies on holometabolous insects. Phil. Trans. Roy Soc. Lond B. 363, 1539–1547.
Chipman A.D. (2008) Thoughts and speculations on the ancestral arthropod segmentation pathway. In: Evolving Pathways. Eds Minelli A., Fusco G. Cambridge, UK: Cambridge University Press, pp. 343–358.
Patel N.H., Hayward D.C., Lall S., Pirkl N.R., DiPietro D., Ball E.E. (2001) Grasshopper hunchback expression reveals conserved and novel aspects of axis formation and segmentation. Development. 128, 3459–3472.
Liu P.Z., Kaufman T.C. (2004) Kruppel is a gap gene in the intermediate germband insect Oncopeltus fasciatus and is required for development of both blastoderm and germband-derived segment. Development. 131, 4567–4579.
Mito T., Sarashina I., Zhang H., Iwahashi A., Okamoto H., Miyawaki K., Shinmyo Y., Ohuchi H., Noji S. (2005) Noncanonical functions of hunchback in segment patterning of the intermediate germ cricket Gryllus bimaculatus. Development. 132, 2069–2079.
Chipman A.D., Stollewerk A. (2006) Specification of neural precursor identity in the geophilomorph centipede Strigamia maritima. Dev. Biol. 290, 337–350.
Schwager E.E., Pechmann M., Feitosa N.M., McGregor A.P., Damen W.G.M. (2009) hunchback functions as a segmentation gene in the spider Achaearanea tepidariorum. Curr. Biol. 19, 1333–1340.
Patel N.H., Ball E.E., Goodman C.S. (1992) Changing role of even-skipped during the evolution of insect pattern formation. Nature. 357, 339–342.
Patel N.H. (1994) Developmental evolution: insights from studies of insect segmentation. Science. 266, 581–590.
Clyde D.E., Corado M.S.G., Wu X., Pare A., Papatsenko D., Small S. (2003) A self-organizing system of repressor gradients establishes segmental complexity in Drosophila. Nature. 426(6968), 849–853.
Deutsch J.S. (2004) Segments and parasegments in arthropods: a functional perspective. Bioessays. 26(10), 1117–1125.
Damen W.G. (2007) Evolutionary conservation and divergence of the segmentation process in arthropods. Dev. Dyn. 236(6), 1379–1391.
Livesey F.J., Cepko C.L. (2001) Vertebrate neural cell-fate determination: lessons from the retina. Nat. Rev. Neurosci. 2, 109–118.
Molyneaux B.J., Arlotta P., Menezes J.R., Macklis J.D. (2007) Neuronal subtype specification in the cerebral cortex. Nat. Rev. Neurosci. 8, 427–437.
Jacob J., Maurange C., Gould A.P. (2008). Temporal control of neuronal diversity: common regulatory principles in insects and vertebrates? Development. 135, 3481–3489.
Okano H., Temple S. (2009) Cell types to order: temporal specification of CNS stem cells. Curr. Opin. Neurobiol. 19, 112–119.
Gaspard N., Bouschet T., Hourez R., Dimidschstein J., Naeije G., van den Ameele J., Espuny-Camacho I., Herpoel A., Passante L., Schiffmann S.N., Gaillard A., Vanderhaeghen P. (2008) An intrinsic mechanism of corticogenesis from embryonic stem cells. Nature. 455, 351–357.
Naka H., Nakamura S., Shimazaki T., Okano H. (2008) Requirement for COUP-TFI and II in the temporal specification of neural stem cells in CNS development. Nat. Neurosci. 11, 1014–1023.
Shen Q., Wang Y., Dimos J.T., Fasano C.A., Phoenix T.N., Lemischka I.R., Ivanova N.B., Stifani S., Morrisey E.E., Temple S. (2006) The timing of cortical neurogenesis is encoded within lineages of individual progenitor cells. Nat. Neurosci. 9, 743–751.
Shitamukai A., Konno D., Matsuzaki F. (2011) Oblique radial glial divisions in the developing mouse neocortex induce self-renewing progenitors outside the germinal zone that resemble primate outer subventricular zone progenitors. J. Neurosci. 31, 3683–3695.
Conduit P.T., Raff J.W. (2010) Cnn dynamics drive centrosome size asymmetry to ensure daughter centriole retention in Drosophila neuroblasts. Curr. Biol. 20, 2187–2192.
Januschke J., Llamazares S., Reina J., Gonzalez C. (2011) Drosophila neuroblasts retain the daughter centrosome. Nat. Commun. 2, 243.
Wang X.,Tsai J.W., Imai J.H., Lian W.N., Vallee R.B., Shi S.H. (2009) Asymmetric centrosome inheritance maintains neural progenitors in the neocortex. Nature. 461, 947–955.
Gao P., Postiglione M.P., Krieger T.G., Hernandez L., Wang C., Han Z., Streicher C., Papusheva E., Insolera R., Chugh K., Kodish O., Huang K., Simons B.D., Luo L., Hippenmeyer S., Shi S.H. (2014) Deterministic progenitor behavior and unitary production of neurons in the neocortex. Cell. 159, 775–788.
Mattar P., Ericson J., Blackshaw S., Cayouette M. (2015) A conserved regulatory logic controls temporal identity in mouse neural progenitors. Neuron. 85, 497–504.
Homem C.C.F., Repic M., Knoblich J.A. (2015) Proliferation control in neural stem and progenitor cells. Nat. Rev. Neurosci. 16(11), 647–659.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


