Молекулярная биология, 2019, T. 53, № 3, стр. 476-484
Дифференциально экспрессирующиеся длинные некодирующие РНК в промоторной области гена fork head Drosophila melanogaster, выявляемые с помощью Нозерн-блот-гибридизации
А. И. Бурлин a, С. В. Тиллиб a, *
a Институт биологии гена Российской академии наук
119334 Москва, Россия
* E-mail: tillib@genebiology.ru
Поступила в редакцию 03.12.2018
После доработки 13.12.2018
Принята к публикации 13.12.2018
Аннотация
Известно, что длинные (200‒300 н. и более) не кодирующие белок РНК (нкРНК), тканеспецифически экспрессирующиеся с промоторов, расположенных в регуляторных областях генов, связанных с развитием, могут регулировать транскрипцию мРНК этих генов. Предпринята попытка выявить дифференциально экспрессирующиеся нкРНК в расширенной промоторной области гена fork head (fkh) Drosophila melanogaster. Анализировали четыре препарата суммарных РНК: из эмбрионов и взрослых мух (отдельно из самок и самцов), а также из линии культивируемых клеток S2 дрозофилы. Как известно, эмбрионы и мухи содержат клетки с очень разным уровнем экспрессии гена fkh. Нами показано, что в клетках S2 экспрессия этого гена не детектируется. Мы использовали классический метод Нозерн-блот-анализа РНК, разделенных в геле, с помощью радиоактивно меченных ДНК-фрагментов, соответствующих соседним районам промоторной области гена fkh и частично перекрывающихся. В исследуемой области выявлено несколько ранее не известных дифференциально экспрессирующихся нкРНК, в том числе в районах обнаруженных ранее регуляторных элементов и в участке собственно начала транскрипции гена fkh. Полученные данные дополняют и уточняют для исследуемого участка генома результаты ранее проведенных “RNA-seq”-экспериментов, в частности, длину детектируемых РНК. Эти результаты могут стать основой дальнейших исследований механизмов тканеспецифической регуляции экспрессии гена fkh, в которых могут принимать участие выявленные нкРНК.
ВВЕДЕНИЕ
В мировой науке продолжается рост интереса к миру РНК, связанный с новыми важными результатами, указывающими на существование разнообразных не кодирующих белки РНК (нкРНК) и их участие в регуляции структуры хроматина, экспрессии генов высших эукариот на разных этапах транскрипции, трансляции, процессинга и стабильности мРНК. Для нашей лаборатории особый интерес представляют ранее опубликованные данные о возможной связи тканеспецифической транскрипции длинных нкРНК в определенных регуляторных участках генов развития и эпигенетической регуляции активности этих генов. Показана связь функционирования так называемых “модулей клеточной памяти”, особых хромосомных регуляторных элементов, через взаимодействие с которыми проявляется активность белков групп Триторакса (Trithorax, TrxG) и Поликомба (Polycomb, PcG), и экспрессии нкРНК в районах локализации этих “модулей”. Предложены различные механизмы такой нкРНК-зависимой регуляции экспрессии тканеспецифических генов (транскрипционная интерференция, изменение структуры локального хроматина, модификация гистонового кода). На этих вопросах мы остановимся несколько более подробно.
Белки TrxG/PcG представляют собой эпигенетическую регуляторную систему, впервые описанную у Drosophila. Эта система поддержания тканеспецифической активности генов высококонсервативна у многоклеточных, включая растения, червей, мух и позвоночных [1, 2]. Принято считать, что эти две группы белков конкурентно регулируют несколько сотен генов-мишеней, поддерживая активное (TrxG) или репрессированное (PcG) состояние их экспрессии. Генетические исследования, проведенные на мухах и растениях с использованием репортерных генов, показали, что эти регуляторные белки поддерживают эпигенетическую память о тканеспецифической активности генов-мишеней [3, 4]. В пионерских работах 1990-х годов идентифицированы и изучены с помощью трансгенных конструкций несколько ассоциированных с белками TrxG/PcG хромосомных элементов дрозофилы (PRE/TRE) в гомеотических генах комплексов Bithorax и Antennapedia [1]. Эти cis-регуляторные элементы, несмотря на обогащение определенными короткими последовательностями ДНК, мотивами/сайтами связывания, имеют в целом сложную и в значительной мере уникальную для конкретного регулируемого гена структуру. Они предположительно состоят, как показано в подробно изученных случаях, из кооперативно работающих модулей, каждый из которых способен взаимодействовать как с положительными, так и с отрицательными регуляторами хроматина [5], поддерживая активное или репрессированное состояние гена соответственно. Модули PRE/TRE необходимы для поддержания исходно детерминированных правильных паттернов тканеспецифической экспрессии гомеотических и иных генов-мишеней в течение многих клеточных делений. У мух, растений и позвоночных гены-мишени белков PcG и TrxG были идентифицированы сначала генетически, а затем и с помощью новых методов полногеномного анализа. Они включают гены, участвующие в спецификации клеточной идентичности, в передаче сигналов и пролиферации, а также в ответе на стресс [6, 7]. Аберрантная экспрессия белков PcG и TrxG связана с нарушениями развития и метаболизма, а также с раком [8].
Белки PcG и TrxG регулируют свои гены-мишени сложным и динамичным образом. Эти белки сталкиваются с несколькими различными задачами. Во-первых, они должны найти свои целевые гены и связаться с соответствующими регуляторными элементами этих генов. Во-вторых, они должны “чувствовать” состояние промотора и модифицировать местный хроматин в ответ на это состояние. В-третьих, они должны сохранять память об этом состоянии при прохождении через репликацию и митоз. И, наконец, в особых случаях они должны быть способны отказаться от функции памяти и позволить гену переключаться в ответ на сигналы развития или окружающей среды [1, 9‒11].
На протяжении последних двух десятилетий периодически появлялись данные о том, что транскрипция некодирующих последовательностей и некодирующая РНК могут участвовать в “решении” некоторых из этих задач, при этом разные модели приобретали и теряли популярность с течением времени [12‒14]. Еще в первых работах, выполненных на дрозофиле, сравнили локализацию ряда PRE/TRE с местами экспрессии ранних нкРНК и показали, что эти регуляторные “модули памяти” транскрибируются в течение того самого временного окна, когда происходит детерминация активности регулируемого гена [15]. Появляется все больше данных о длинных нкРНК как о важных факторах, вовлеченных в биологические процессы, лежащие в основе патофизиологии заболеваний человека [16]. Эпигенетическую регуляцию активности генов с участием белков групп PcG и TrxG предлагается рассматривать как динамичную и обратимую “бистабильную систему” [17]. Бистабильная система имеет три состояния: два устойчивых экстремальных и промежуточное нестабильное переходное состояние, через которое система должна пройти, чтобы достигнуть любого из крайних состояний, подобно выключателю электрического света. Анализ экспрессии трансгенов у мух, растений и в клеточных культурах млекопитающих позволил получить достаточные генетические доказательства того, что гены, регулируемые белками PcG/TrxG, могут в определенных случаях переключаться с включенного состояния на выключенное и наоборот. В этой связи нкРНК можно рассматривать как инструменты, весьма подходящие для нарушения генспецифической стабильности, потому что в отличие от таких глобальных процессов, как репликация и митоз, транскрипция генов нкРНК потенциально может быть локально, временно и количественно регулируемой.
Опубликованы сообщения о конкретных нкРНК в генах-мишенях PcG/TrxG, которые взаимодействуют с белками TrxG и вызывают активацию гена [17]. К сожалению, влияние нкРНК на ферментативную активность белков TrxG не изучено. В этой связи интересно предположить существование механизма, при котором одна и та же нкРНК может, с одной стороны, ингибировать репрессорный комплекс PRC2, а с другой, стимулировать активность гистон-метилтрансферазы активирующих белков группы TrxG. Транскрибирующаяся нкРНК также может быть ингибитором ферментативных активностей модификаторов хроматина обеих групп белков, следствием чего может быть промежуточное переходное состояние системы, в котором вся локальная ферментативная активность снижается, позволяя формированию нового статуса при поступлении соответствующих сигналов. В целом следует признать, что связь нкРНК с PcG/TrxG-зависимой регуляцией активности генов остается одним из активно изучаемых, но противоречивых и все еще плохо понятных вопросов эпигенетики [18].
В фокусе нашего внимания находилась расширенная промоторная область регионального гомеотического гена fork head (fkh) D. melanogaster, которая на протяжении ряда лет служила модельной системой для изучения нового регуляторного элемента, ассоциированного с TRX. По-видимому, этот элемент является принципиально важной частью “модуля клеточной памяти” гена fkh, необходимого, в частности, для развития слюнных желез [19‒21].
Наблюдаемые в последние годы технологические прорывы в секвенировании геномных ДНК и изучении почти полных транскриптомов с помощью разного рода микрочипов пока не могут полностью заменить традиционное исследование тканеспецифической экспрессии РНК в конкретном участке генома. К нашему удивлению, нам не удалось найти данных даже о реальных размерах зрелой мРНК гена fkh, например, результатов Нозерн-гибридизации РНК дрозофилы с фрагментами ДНК этого гена. Довольно надежно установлены лишь начало ее транскрипции и кодирующая белок последовательность. Белок FKH состоит из 510 аминокислотных остатков и кодируется последовательностью (открытая рамка считывания) длиной 1530 п.н. В базах данных приведены последовательности различающихся сплайсированных вариантов мРНК fkh длиной от 3280 до 4289 н., при этом нет сведений о длине их 3'-нетранслируемого участка. Из базовых работ по регуляции гена fkh дрозофилы [22, 23] известно также о существовании в промоторной области этого гена совсем слабо изученной длинной РНК (dfk, distal fork head, длиной в несколько тысяч нуклеотидов), транскрибирующейся в противоположном направлении относительно мРНК гена fkh и, по-видимому, не кодирующей белок. Начало транскрипции нкРНК dfk отстоит лишь на 424 н. от начала транскрипции мРНК fkh. Во всех доступных базах данных мы провели поиск других возможных транскриптов в интересующей нас области, и нашли в базе EST (Expressed Sequence Tags) данные о трех неполных последовательностях кДНК. Этим последовательностям соответствуют участки РНК, локализующихся, соответственно, в положениях 800–1500, 2500–3200 и 4200–5000 н. от начала транскрипции мРНК fkh. Возможно, это просто три последовательных фрагмента одной и той же транскрибирующейся в ту же сторону нкРНК dfk. Данные о других РНК, локализованных в более удаленной от промотора области мРНК fkh, отсутствуют. Описанный нами новый регуляторный элемент (TRE1), ассоциированный с регуляторным белком TRX, был локализован в 7000 н. перед началом транскрипции мРНК fkh [20], примерно на 2000 н. дальше от промотора, чем предполагаемый 3'-конец нкРНК dfk. Для нас особенно важно было выяснить, существуют ли в этой области дифференциально экспрессирующиеся длинные нкРНК, которые могут быть связаны с функционированием данного TRE. Мы решили использовать метод Нозерн-гибридизации для предварительного гибридизационного анализа и идентификации потенциальных нкРНК в расширенной промоторной области гена fkh. Выбор метода продиктован следующими соображениями. Во-первых, в других подобных регуляторных элементах выявлены достаточно протяженные нкРНК (300 н. и более), что вполне соответствует широкому диапазону РНК, исследуемых методом Нозерн-блот-гибридизации. Во-вторых, мы решили анализировать суммарные препараты РНК (а не поли(А)-содержащие РНК), так как нкРНК вполне может быть неполиаденилированной. В-третьих, пока не выявлено однозначного соответствия нкРНК активному или репрессированному состоянию гена-мишени, поэтому для полноты картины нужно искать нкРНК в разных тканях и на разных стадиях развития организма.
Несмотря на предварительный характер результатов Нозерн-блот-гибридизации, нами получены принципиально новые важные данные. Впервые показано, что ген fkh репрессирован в культивируемых клетках S2 дрозофилы. Эти клетки могут быть удобным негативным контролем в исследованиях механизмов регуляции экспрессии гена fkh в других клетках и тканях дрозофилы. В промоторной области гена fkh выявлено несколько гипотетических дифференциально экспрессирующихся нкРНК, в том числе в районах обнаруженных ранее регуляторных элементов (TRE1 и энхансера sgE, специфичных для клеток слюнных желез), а также в районе собственно начала транскрипции гена fkh. Полученные данные, несмотря на их предварительный характер, заметно дополняют полученные для исследуемого участка генома результаты ранее проведенных “RNA-seq”-экспериментов, в частности, по выявлению длины детектируемых РНК. Эти результаты могут стать основой дальнейших исследований механизмов тканеспецифической регуляции экспрессии гена fkh, в которых могут принимать участие выявленные нкРНК.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Выделение, фракционирование, гибридизация РНК. В качестве исходного материала для выделения РНК использовали эмбрионы (в возрасте 2–24 ч) и взрослых мух дикого типа Орегон, слюнные железы, отобранные под бинокулярным микроскопом из личинок третьей стадии, а также клетки линии S2 D. melanogaster. Клетки линии S2 выращивали в суспензии в среде Schneider’s Insect Medium (“Sigma”, США) с добавлением 10% фетальной бычьей сыворотки (“Sigma”, “HyClone”) при 25°С. Клетки пересевали 1 раз в 3‒4 дня в разведении 1: 6. При необходимости смены среды клетки осаждали центрифугированием при 200 g в течение 10 мин и ресусепендировали в новой среде.
РНК выделяли с помощью реактива TRIzol (“Invitrogen”, США) согласно протоколу фирмы. Выделение поли(А)-РНК на колонке с олиго(dT)-целлюлозой, электрофорез РНК в 1.5%-ном агарозном геле с формальдегидом, капиллярный перенос РНК из геля на мембрану (Hybond-XL, “GE Healthcare”, США) и Нозерн-гибридизацию проводили в основном согласно протоколам, описанным ранее [24], или руководствам фирм, выпускающих используемые материалы. Мечение фрагментов ДНК проводили с помощью набора DecaLabel DNA labeling kit фирмы “Fermentas” (США) и [α-32Р]dАТP российского производства. Фильтры после гибридизации отмывали в жестких условиях, чтобы минимизировать фон лишь частично комплементарных РНК. Экспозицию отмытых фильтров проводили в течение 2‒14 дней при –70°С с использованием рентгеновской пленки и усиливающих экранов Hyperfilm MP, Hyperscreen фирмы “Amersham” (Великобритания).
Получение фрагментов ДНК для мечения. Пары праймеров для ПЦР-амплификации фрагментов 1‒8 ДНК в промоторной области гена fkh (координата 5'-конца указана согласно последовательности AC009213 из базы данных GenBank®):
Фрагмент 1: 124412F 5'-ctatcactgcttcaatcggt-3' и 125353R 5'-acatggcgtcgctcagca-3';
Фрагмент 2: 125018F 5'-gcaccagctccgactcgt-3' и 126154R 5'-gagaaacgagacaatcgtcct-3';
Фрагмент 3: 125981F 5'-cataaaccatagacgcct-3' и 127584R 5'-gcgatcatctgttgctgct-3';
Фрагмент 4: 127291F 5'-gatgacgtccgcagctgt-3' и 129389R 5'-caatcggccaaagtggtac-3';
Фрагмент 6: 129763F 5'-gacaacgagactgctgcca-3' и 131421R 5'-caaccagatggcgacaagca-3';
Фрагмент 7: 131184F 5'-gctgcgactggagtccca-3' и 132782R 5'-gctttgttgccaattgaca-3';
Фрагмент 7-1: 131184F 5'-gctgcgactggagtccca-3' и 131829R 5'-ctgtgctgcccgttatgcgt-3';
Фрагмент 7-2: 132083F 5'-gctgcgactggagtccca-3' и 132782R 5'-gctttgttgccaattgacag-3';
Фрагмент 8: 132572F 5'-gtgtccaccagcagagttc-3' и 133477R 5'-cctcgctggccaaacagca-3'.
ПЦР проводили в стандартных условиях согласно протоколу фирмы “Силекс” (Россия) с некоторыми модификациями. В ряде случаев в реакционный буфер добавляли 0.75–1 M бетаина, а в качестве фермента использовали смесь полимераз Taq и Pfu (“Силекс”) в соотношении 50 : 1. Температуру плавления подбирали для каждой пары праймеров так, чтобы на матрице геномной ДНК дрозофилы амплифицировался только заданный фрагмент ДНК. Продукты ПЦР фракционировали в агарозном геле, вырезали амплифицированный фрагмент, очищали ДНК с помощью набора для экстракции из геля QIAquick (“QIAGEN”, Нидерланды).
ОТ-ПЦР. Поли(А)-РНК получали параллельно из двух препаратов суммарной РНК (из слюнных желез (sg) личинок третьей стадии и из клеток S2), а затем использовали в качестве матриц для синтеза кДНК с помощью обратной транскриптазы (ОТ) и олиго(dТ)18 (“Thermo Fisher Scientific”, США). Процедуру ОТ-ПЦР проводили, используя три пары праймеров для параллельных амплификаций трех ДНК-фрагментов, соответствующих белоккодирующим последовательностям: мРНК fkh (FKH), мРНК рибосомного белка 49 (rp49) и мРНК глицерол-3-фосфат-дегидрогеназы (GPDH). Последние две мРНК часто используют в качестве нормирующего контроля при изучении количественных изменений транскрипции других генов. Пары праймеров для контрольных образцов подбирали таким образом, чтобы размер получаемого продукта соответствовал именно сплайсированной мРНК, а не возможному загрязнению геномной ДНК (в последнем случае получаемый продукт имеет больший размер, так как амплифицируемые участки содержат микроинтроны в пре-мРНК и геномной ДНК). Использовали следующие пары праймеров:
для мРНК fkh (координаты приведены по последовательности AC009213 из базы данных GenBank®):
4534F 5'-gtttgtaaacacacaccgtggt-3' и 4694R 5'-cagctagatattcacgttcgtg-3';
для мРНК GPDH (NM_001273183 в GenBank®):
1372F 5'-cgactgccgaggaggtgaacta-3' и 1512R 5'-gctcagggtgattgcgtatgca-3';
для мРНК rp49 (U92431 в GenBank®):
1F 5'-atgaccatccgcccagca-3' и 394R 5'-cttgagaacgcaggcgaccgt-3'.
Все ПЦР-продукты по размеру соответствовали сплайсированным мРНК.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Ген fork head не экспрессируется в линии клеток S2 дрозофилы
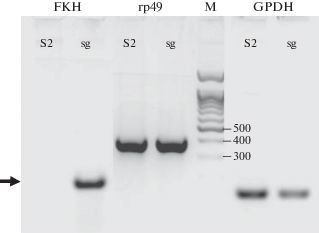
Мы решили параллельно проанализировать РНК из четырех образцов: из эмбрионов (смешали эмбрионы всех стадий), самцов (чтобы учесть как транскрипты в тканях взрослой мухи, так и сперматогенез-специфичные транскрипты), самок (оогенез-специфичные и раннеэмбриональные транскрипты) и из культивируемых клеток линии S2 D. melanogaster. Клетки S2 использовали в качестве негативного контроля. В самом начале работы мы установили, что мРНК fkh в этих клетках не детектируется (рис. 1), т.е. ген fkh в них, по-видимому, репрессирован. В качестве положительного контроля в этом первом эксперименте мы использовали РНК, выделенную из клеток слюнных желез (sg) личинок третьей стадии. Из рис. 1 видно, что в клетках S2 мРНК fkh не детектируется, в отличие от клеток слюнных желез, тогда как референсные РНК (rp49 и GPDH) экспрессируются в количестве не меньшем, чем в контроле.
Рис. 1.
Ген fork head не экспрессируется в линии клеток S2 дрозофилы. Количественный сравнительный OT-ПЦР-анализ мРНК (FKH, rp49, GPDH) в препаратах РНК/кДНК из клеток S2 (S2) и клеток слюнных желез личинок третьей стадии (sg). Стрелкой указано отсутствие продукта, соответствующего мРНК fkh, в клетках S2. М – маркер размеров ДНК-фрагментов.
Нозерн-блот-анализ гибридизующихся РНК в промоторной области гена fkh
В качестве зондов для детекции последовательно использовали радиоактивно меченные слегка перекрывающиеся фрагменты ДНК (восемь фрагментов) расширенной промоторной области гена fkh общей протяженностью 9000 п.н. (схема на рис. 2а).
Рис. 2.
Выявление потенциальных РНК в промоторной области гена fkh с помощью Нозерн-блот-гибридизации суммарной РНК из клеток S2 (S2), эмбрионов (Е), самок (F) и самцов (М) дрозофилы с радиоактивно меченными ДНК-зондами, соответствующими частично перекрывающимся фрагментам геномной ДНК указанной области. а ‒ Схема промоторной области гена fkh. Стрелкой влево указано начало и направление транскрипции мРНК fkh. Сверху обозначены положения сайтов рестрикции. Ниже стрелками показано расположение двух ранее выявленных нкРНК. Черными прямоугольниками выделены два регуляторных участка (TRE1, TRX-ассоциированный элемент и sgE, энхансер, специфичный для клеток слюнных желез). Отрезками снизу обозначены зонды для гибридизации. б – Результаты Нозерн-блот-гибридизации с зондами, от которых отходят длинные стрелки. Маленькими стрелками указано положение детектируемых РНК. Под каждым изображением экспонированных фильтров указаны примерные размеры детектируемых РНК. Для примерной оценки относительных количеств РНК в каждом из четырех образцов в нижней левой части представлена фотография разделенных в геле суммарных РНК с маркерными фрагментами и контрольная гибридизация с мРНК rp49.
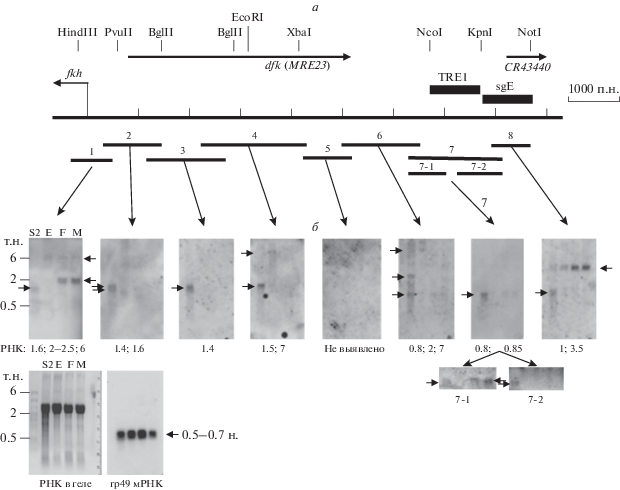
В результате проведенных экспериментов получен значительный объем новых данных (рис. 2б), суммированных на схеме, представленной на рис. 3. В частности, в близлежащем районе регуляторного элемента TRE1 (7000‒9000 н. выше начала транскрипции мРНК fkh) обнаружены две РНК длиной примерно 800 и 3500 н. Более короткая РНК в значительной мере перекрывается с районом TRE1 и экспрессируется в особенно большом количестве (относительно используемой для нормирования мРНК rp49) в клетках S2, в едва детектируемом количестве ‒ во взрослых мухах и совсем не детектируется в препарате эмбриональных РНК. Более длинная РНК локализуется сразу после TRE (дальше от промотора) и перекрывается в значительной мере с участком энхансера слюнных желез (sgE). При этом более длинная РНК, в отличие от более короткой, в наибольших относительных количествах экспрессируется во взрослых мухах, в меньшей степени – в эмбрионах и практически не детектируется в клетках S2, в которых в этом районе обнаружен альтернативный транскрипт длиной примерно 1000 н. (он не найден в трех других препаратах РНК). Подобная (“альтернативная”) ситуация выявлена и в районе нкРНК dfk с использованием трех независимых зондов, покрывающих район в 6000 н. непосредственно выше начала транскрипции мРНК fkh. Эта нкРНК dfk обнаружена только в препарате эмбриональных РНК. Любопытно, что только в клетках S2 в этой промоторной области представлены несколько других транскриптов: размером примерно 1600 н. – в непосредственной близости (возможно, с перекрыванием) к началу транскрипции мРНК fkh; размером 1400 н. – в 1000–2000 н. выше промотора; размером 1500 н. – в 3500 н. выше промотора; 2000 н. – в 6000 н. выше промотора. Интересно, что некоторые из этих новых нкРНК (в этом промоторном участке не выявлено протяженных потенциальных белоккодирующих последовательностей) колокализуются с потенциальными TRE и энхансерами, активными в других тканях (отличных от слюнных желез). В клетках S2 все эти энхансерные элементы не работают, так как ген репрессирован. Возможно, это множество относительно небольших нкРНК размером 800‒2000 н. отражает репрессию гена. С другой стороны, отсутствующие в клетках S2, но обнаруживаемые в более сложных препаратах РНК из многих различных клеток (в одних ген может быть активен, в других нет), относительно длинные РНК, перекрывающиеся с протяженными регуляторными/энхансерными областями, могут быть связаны с активацией транскрипции гена fkh.
Рис. 3.
Схематическое изображение результатов детекции нкРНК в протяженной промоторной области гена fkh. От зондов, соответствующих указанным в середине рисунка участкам промоторной области гена fkh, проведены стрелки к секторам, в которых указаны размеры и потенциальное расположение детектируемых РНК. В этих секторах, разделенных на четыре части сверху вниз, представлены данные для РНК, выделенных из клеток S2, эмбрионов, взрослых самок и самцов.
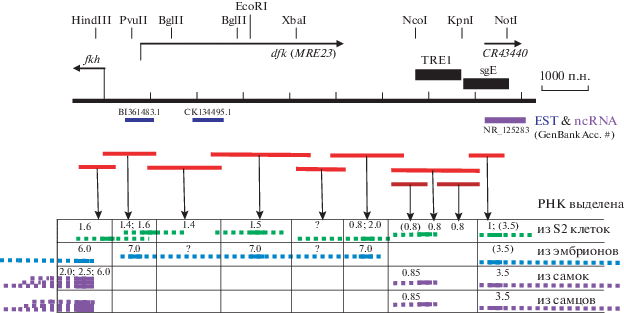
В эмбрионах и взрослых мухах обнаружены новые РНК (по местонахождению и размеру), локализующиеся в собственно промоторной области гена fkh. Эти РНК размером примерно 2000, 2500 и 6000 н. вполне могут соответствовать мРНК, кодирующим белок FKH, они детектируются зондом, узнающим 5'-нетранслируемую часть, которая входит в состав всех известных мРНК fkh. РНК размером примерно 6000 н. выявлена как в эмбрионах, так и во взрослых мухах, тогда как транскрипты длиной примерно 2000 и 2500 н. найдены только во взрослых мухах. Как сказано во введении, белок FKH состоит из 510 аминокислотных остатков и кодируется открытой рамкой считывания из 1530 н. Насколько нам известно, локализация 3'-конца мРНК fkh еще не определена, так что все эти три детектируемые РНК могут быть результатом альтернативного сплайсинга и кодировать белок FKH.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Нами показано, что ген fkh не экспрессируется в линии культивируемых клеток S2 дрозофилы, поэтому клетки S2 могут быть негативным контролем при изучении регуляции активности этого гена в различных модельных экспериментах. Мы использовали этот факт для сравнительного изучения РНК-транскриптов в протяженной области промотора гена fkh в препаратах РНК, выделенных также из эмбрионов и взрослых мух (отдельно из самок и самцов).
Классический метод Нозерн-блот-анализа позволил выявить несколько ранее неизвестных дифференциально экспрессирующихся РНК, в том числе в районах регуляторных элементов (TRE1, энхансера слюнных желез sgE) и в области начала транскрипции гена fkh.
В клетках S2, в которых ген неактивен, в области начала транскрипции гена fkh выявляется РНК размером около 1600 н. Три других РНК найдены в эмбрионах и взрослых мухах. Они могут отражать экспрессионную активность этого гена. РНК размером 6000 н. больше указанных во FlyBase (http://flybase.org) fkhРНК (от 3280 до 4289 н.), но 3'-нетранслируемая область упомянутых РНК не изучена, поэтому точный размер РНК не известен. Две более коротких РНК длиной примерно 2000 и 2500 н. детектируются в этом районе только у взрослых мух и уровень их экспрессии, по-видимому, выше, чем у РНК длиной 6000 н. Предполагается, что эти более короткие РНК, выявляемые зондом в районе начала транскрипции гена fkh у взрослых мух, могут быть продуктами альтернативного сплайсинга и также кодировать собственно белок FKH. В противном случае они могут быть регуляторными РНК и отражать пока неизвестный тип регуляции экспрессии этого гена.
В близлежащем районе потенциального регуляторного элемента TRE1 (6600‒7600 н. выше начала транскрипции мРНК fkh) обнаружены две нкРНК длиной примерно 800 и 850 н. Более длинная РНК (850 н.) экспрессируется преимущественно во взрослых мухах и локализуется в проксимальной области TRE1 (детектируется зондами 7, 7-1 и, по-видимому, 6, но не 7-2). Более короткая (800 н.) РНК в значительной мере перекрывается с дистальной частью TRE1 (выявляется зондами 7, 7-2) и экспрессируется в особенно большом количестве в клетках S2. Таким образом, если РНК длиной 800 н., скорее всего, отражает молчащее состояние гена в этом участке, то РНК длиной 850 н. потенциально может регулировать активность TRE1 во взрослых мухах.
Особое внимание привлекла РНК длиной 3500 н., выявляемая зондом 8 (но не зондом 7), которая перекрывается в значительной мере с участком энхансера слюнных желез sgE. Эта РНК в наибольших количествах экспрессируется во взрослых мухах, в меньшей степени – в эмбрионах и едва детектируется в клетках S2 (отметим, что в клетках S2 в этом районе обнаружен дополнительный транскрипт длиной примерно 1000 н., не найденный в трех других препаратах РНК). При дополнительном анализе баз данных (FlyBase и GenBank) мы нашли информацию о транскрипте D. melanogaster длиной 796 н. (последовательность NR_125283 из базы данных GenBank) в районе зонда 8, обозначенном как длинная нкРНК CR43440 на рис. 2 и 3. Эта РНК, направленная в противоположную сторону от промотора гена fkh относительно мРНК fkh, с высокой вероятностью является частью обнаруженной нами РНК длиной 3500 н. (или, что менее вероятно, соответствует РНК длиной 1000 н., выявленной в наших опытах только в клетках S2). 5'-Конец РНК CR43440 локализуется в середине sgE в 440 н. от сайта расщепления рестриктазой NotI в эволюционно очень консервативном участке генома [25]. Любопытно, что делеция окружающего участка 361–506 п.н. (от сайта NotI) приводит к заметному усилению экспрессии трансгена, которая обеспечивается энхансером sgE в слюнных железах личинок. Сделан вывод о присутствии в этом районе сильного репрессорного элемента [25]. Можно предположить, что и транскрипция нкРНК, соответствующей РНК CR43440 (потенциально нашей РНК длиной 3500 н.), связана с этой репрессорной активностью.
Недавно проведенные количественные эксперименты in vitro показали, что репрессорный комплекс PRC2, состоящий из белков PcG, связывает РНК не совсем случайно [26]. Например, поли(А)-мотивы имеют низкое сродство к PRC2 (Kd ≥ 10 мкМ), тогда как поли(G) имеет наибольшее сродство (8 нМ). Другие последовательности РНК также обладают различным сродством к PRC2 в наномолярном диапазоне (30–280 нМ). Особый интерес представляет достаточно высокое сродство к структурам G4 (80 нМ) и повторам GU (30 нМ). Многие PRE/TRE дрозофилы и сайты связывания PcG позвоночных содержат повторяющиеся GT-участки, способные продуцировать РНК с GU-богатыми последовательностями при транскрипции с соответствующей цепи. Кроме того, с использованием репортерных трансгенов показано, что GT-повторы необходимы для функции PRE/TRE, но как действуют эти мотивы в контексте РНК еще предстоит выяснить [8, 27]. Отметим в этой связи, что в состав РНК CR43440 входят участки, обогащенные GU.
Предполагается, что РНК размером 3500 н. (в эмбрионах и взрослых мухах) и 1000 н. (в клетках S2), обнаруженные нами в районе sgE, как и РНК размером 800 и 850 н. (в районе TRE1), могут участвовать в разных регуляторных процессах. Например, показано, что некодирующая транскрипция с противоположных цепей одного и того же PRE/TRE дрозофилы может приводить к противоположным эффектам in vivo [29].
Конечно же, данные о новых нкРНК требуют дальнейшего уточнения и подтверждения. Полученные результаты могут стать основой дальнейших исследований механизмов тканеспецифической регуляции экспрессии гена fkh, в которых могут принимать участие нкРНК.
Работа выполнена в рамках Программы фундаментальных исследований Государственных академий наук на 2013–2020 годы (тема № АААА-А17-117020110105-5) и при частичной поддержке Российского фонда фундаментальных исследований (проект № 08-04-01395).
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Geisler S.J., Paro R. (2015) Trithorax and Polycomb group-dependent regulation: a tale of opposing activities. Development. 142, 2876–2887.
Grossniklaus U., Paro R. (2014) Transcriptional silencing by Polycomb-group proteins. Cold Spring Harb. Perspect. Biol. 6, a019331.
Berry S., Hartley M., Olsson T.S., Dean C., Howard M. (2015) Local chromatin environment of a Polycomb target gene instructs its own epigenetic inheritance. eLife. 4, e07205.
Steffen P.A., Ringrose L. (2014) What are memories made of? How Polycomb and Trithorax proteins mediate epigenetic memory. Nat. Rev. Mol. Cell Biol. 15, 340–356.
Tillib S., Petruk S., Sedkov Y., Kuzin A., Fujioka M., Goto T., Mazo A. (1999) Trithorax and Polycomb group response elements within a Ultrabithorax transcription maintenance unit consist of closely situated but separable sequences. Mol. Cell. Biol. 19, 5289‒5202.
Ringrose L. (2007) Polycomb comes of age: genome-wide profiling of target sites. Curr. Opin. Cell Biol. 19, 290–297.
Di Croce L., Helin K. (2013) Transcriptional regulation by Polycomb group proteins. Nat. Struct. Mol. Biol. 20, 1147–1155.
Comet I., Riising E.M., Leblanc B., Helin K. (2016) Maintaining cell identity: PRC2-mediated regulation of transcription and cancer. Nat. Rev. Cancer. 16, 803–810.
Bauer M., Trupke J., Ringrose L. (2016) The quest for mammalian Polycomb response elements: Are we there yet? Chromosoma. 125, 471–496.
Kassis J.A., Brown J.L. (2013) Polycomb group response elements in Drosophila and vertebrates. Adv. Genet. 81, 83–118.
Ringrose L., Paro R. (2004) Epigenetic regulation of cellular memory by the Polycomb and Trithorax group proteins. Annu. Rev. Genet. 38, 413–443.
Cech T.R., Steitz J.A. (2014) The noncoding RNA revolution-trashing old rules to forge new ones. Cell. 157, 77–94.
Davidovich C., Cech T.R. (2015) The recruitment of chromatin modifiers by long noncoding RNAs: lessons from PRC2. RNA. 21, 2007–2022.
Hekimoglu B., Ringrose L. (2009) Non-coding RNAs in polycomb/trithorax regulation. RNA Biol. 6, 129–137.
Lempradl A., Ringrose L. (2008) How does noncoding transcription regulate Hox genes? BioEssays 30, 110–121.
DiStefano J.K. (2018) The emerging role of long noncoding RNAs in human disease. Methods Mol. Biol. 1706, 91–110.
Wang K.C., Yang Y.W., Liu B., Sanyal A., Corces-Zimmerman R., Chen Y., Lajoie B.R., Protacio A., Flynn R.A., Gupta R.A., Wysocka J., Lei M., Dekker J., Helms J.A., Chang H.Y. (2011) A long noncoding RNA maintains active chromatin to coordinate homeotic gene expression. Nature. 472, 120–124.
Ringrose L. (2017) Noncoding RNAs in Polycomb and Trithorax regulation: a quantitative perspective. Annu. Rev. Genet. 51, 385–411.
Kuzin B., Tillib S., Sedkov I., Mizrokhi L., Mazo A. (1994) The Drosophila trithorax gene encodes a chromosomal protein and directly regulates the region-specific homeotic gene fork head. Genes Dev. 8, 2478‒2490.
Ряховский А.А., Тиллиб С.В. (2007) Иммунопреципитационное картирование TRX-ассоциированных участков хромосом в промоторе гена fkh в клетках слюнных желез Drosophila melanogaster. Генетика. 43, 1181‒1189.
Ряховский А.А., Тиллиб С.В. (2007) Колокализация S/MAR и TRE в регуляторных участках хромосом тканеспецифически экспрессирующихся генов Drosophila melanogaster. Докл. Акад. Наук. 416, 416‒419.
Weigel D., Jiirgens G., Kuttner F., Seifert E., Jackie H. (1989) The homeotic gene fork head encodes a nuclear protein and is expressed in the terminal regions of the Drosophila embryo. Cell. 57, 645‒658.
Weigel D., Seifert E., Renter D., Jackie H. (1990) Regulatory elements controlling expression of the Drosophila homeotic gene fork head. EMBO J. 9, 1199‒1207.
Molecular Cloning a Laboratory Manual. Eds Sambrook J., Russell D.W. 3rd edn. (2001) 2, New York: Cold Spring Harbor Lab. Press.
Zhou B., Bagri A., Beckendorf S.K. (2001) Salivary gland determination in Drosophila: a salivary-specific, fork head enhancer integrates spatial pattern and allows fork head autoregulation. Developmental Biol. 237, 54–67.
Wang X., Goodrich K.J., Gooding A.R., Naeem H., Archer S., Paucek R.D., Youmans D.T., Cech T.R., Davidovich C. (2017) Targeting of Polycomb repressive complex 2 to RNA by short repeats of consecutive guanines. Mol. Cell. 65, 1056–1067.
Okulski H., Druck B., Bhalerao S., Ringrose L. (2011) Quantitative analysis of Polycomb response elements (PREs) at identical genomic locations distinguishes contributions of PRE sequence and genomic environment. Epigenet. Chromatin. 4, 4.
Ray P., De S., Mitra A., Bezstarosti K., Demmers J.A., Pfeifer K., Kassis J.A. (2016) Combgap contributes to recruitment of Polycomb group proteins in Drosophila. Proc. Natl. Acad. Sci. USA. 113, 3826–3831.
Herzog V.A., Lempradl A., Trupke J., Okulski H., Altmutter C., Ruge F., Boidol B., Kubicek S., Schmauss G., Aumayr K., Ruf M., Pospisilik A., Dimond A., Senergin H.B., Vargas M.L., Simon J.A., Ringrose L. (2014) A strand-specific switch in noncoding transcription switches the function of a Polycomb/Trithorax response element. Nat. Genet. 46, 973–981.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


