Молекулярная биология, 2019, T. 53, № 3, стр. 524-528
Существуют ли сайты сплайсинга в пре-мРНК, приходящиеся на гомоповторы в белках человека?
О. В. Галзитская a, *, Г. С. Новиков b
a Институт белка Российской академии наук
Московская обл142290 Пущино, Россия
b Санкт-Петербургский национальный исследовательский академический университет
194021 Санкт-Петербург, Россия
* E-mail: ogalzit@vega.protres.ru
Поступила в редакцию 31.08.2018
После доработки 25.12.2018
Принята к публикации 28.12.2018
Аннотация
С целью ответить на вопрос, существуют ли сайты сплайсинга в пре-мРНК, приходящиеся на гомоповторы в белках человека, мы исследовали белки с гомоповторами длиной больше 4 аминокислотных остатков. Впервые показано, что в протеоме человека есть 404 белка с гомоповторами, на которые приходится хотя бы один сайт сплайсинга в пре-мРНК. Выявлено, что участки сплайсинга в пре-мРНК чаще локализуются в С-концевой части гомоповтора (67%) и гораздо реже в центральной или N-концевой области. Идентифицировано 10 гомоповторов с двумя сайтами сплайсинга, причем в 9 случаях это гомоповторы лизина.
ВВЕДЕНИЕ
Гены высших организмов состоят из кодирующих и некодирующих участков ДНК, которые не участвуют в экспрессии генов, но могут содержать регуляторные элементы. При сплайсинге происходит вырезание некодирующих участков пре-мРНК. Благодаря альтернативному сплайсингу возникает разное количество изоформ одного и того же белка [1]. Существующий механизм альтернативного сплайсинга увеличивает функциональное разнообразие белков. Известно, что альтернативный сплайсинг мРНК в местах, соответствующих неупорядоченным участкам в белке, может способствовать появлению новых признаков в белках с течением эволюции, развития организмов и болезней. Включение таких неупорядоченных участков может опосредовать новые белковые взаимодействия и, следовательно, изменить контекст, в котором биохимические или молекулярные функции выполняет данный белок [2]. Среди альтернативно сплайсированных экзонов тканеспецифические экзоны играют ведущую роль в поддержании идентичности тканей. Недавно исследованы структурные, функциональные и эволюционные свойства тканеспецифических и других альтернативных экзонов у человека [3].
В особую группу следует выделить участки аминокислотной последовательности, соответствующие неоднократному повтору одной и той же аминокислоты, так называемые гомоповторы. При построении библиотеки неструктурированных шаблонов оказалось, что бóльшая часть шаблонов имеет мотивы с простой сложностью. Вопрос о влиянии гомоповторов в белках на увеличение или уменьшение доли неупорядоченных аминокислотных остатков (а.о.) недавно рассмотрен в нескольких публикациях [4–8]. Показано, что появление гомоповторов с гидрофобными аминокислотами приводит к уменьшению доли неупорядоченных остатков в белке, в то время как для заряженных, полярных и малых а.о. это приводит к росту неупорядоченности в белках. Максимальная доля неупорядоченных остатков получена для белков с гомоповторами лизина и аргинина, а минимальное значение соответствует гомоповторам валина и лейцина [8].
Известно, что развитие ряда тяжелых заболеваний человека ассоциировано с патологической экспансией повторяющихся последовательностей [9, 10]. Бóльшая часть гомоповторов, которая коррелирует с болезнями, найдена в экзонах [9, 11]; при этом митотическая нестабильность или соматический мозаицизм таких гомоповторов определяет их изменение размеров в тканях больного человека [12].
Недавно нами показано, что минимальный размер гомоповторов зависит от типа аминокислоты и протеома [13, 14]. Так, обнаружено, что гомоповторы, содержащие аминокислоты E, S, Q, G, L, P, D, A или H, с большой вероятностью ассоциированы с заболеваниями человека, согласно базе данных OMIM [13].
В представленной работе рассмотрены гомоповторы аминокислот, которые для полярных, заряженных и малых а.о. соответствуют в бóльшей части неструктурированным участкам в белковой структуре. Особый интерес для нас будет представлять вопрос, существуют ли участки сплайсинга в пре-мРНК, приходящиеся на участки гомоповторов в белках (рис. 1). Следует отметить, что аномальное повторение кодона CAG при болезни (глутаминовый гомоповтор, поли-Q) может влиять на сплайсинг [15].
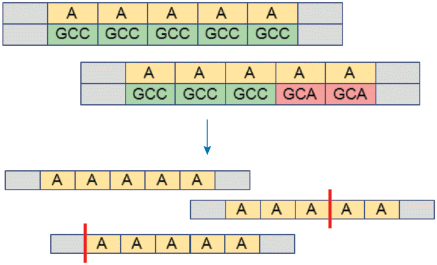
Рис. 1.
Схематичное изображение кодирования гомоповтора из пяти остатков аланина. Показан пример, когда данный гомоповтор кодирует как одинаковый кодон, так и разные. Внизу рисунка показаны возможные варианты расположения сайта сплайсинга для гомоповтора: его может и не быть, может приходиться на середину участка или на N- и C-концевые участки.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Источники данных. Данные по гомоповторам белков человека брали из базы данных Института белка Российской академии наук (Россия) HRaP (http://bioinfo.protres.ru/hrap/), которая включает информацию для 1 449 683 белков из 122 эукариотических и бактериальных протеомов [16]. Информация по белкам, связанными с болезнями, согласно базе данных OMIM (http://www.omim.org/), получена из базы HRaDis (http://bioinfo.protres.ru/ hradis/) того же Института, в ней рассмотрены только гомоповторы длиной больше 4 а.о. [13].
С целью найти кодоны, соответствующие областям гомоповторов, мы проанализировали все кодирующие последовательности белоккодирующих генов с использованием программы Ensembl/Biomart версии 90 [17]. Для получения информации о сайтах сплайсинга для каждого гена проводили выравнивание нуклеотидной последовательности и соответствующей ей аминокислотной последовательности, используя версию 2.2 Exonerate (https://www.ebi.ac.uk/about/vertebrate-genomics/software/exonerate).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Каким гомоповторам в белках чаще всего соответствуют сайты сплайсинга в РНК?
Так как неструктурированные участки, на которые приходятся сайты сплайсинга в пре-мРНК, могут отвечать за появление новых функций у белка и способствовать появлению новых партнеров по взаимодействию, нас интересовали гомоповторы в белках человека, которые для большинства аминокислот являются неструктурированными участками, на которые приходятся сайты сплайсинга. Исходные данные получены из базы данных HRaP для протеома человека, при этом рассматривали только гомоповторы длиной больше 4 а.о. Одновременно получена информация для гомоповторсодержащих белков, ассоциированных с тем или иным заболеванием (из базы данных HRaDis). Далее для белков с гомоповторами искали соответствующий ген, проводили выравнивание, чтобы идентифицировать последовательности, кодирующие эти гомоповторы. Одновременно получали информацию о сайтах сплайсинга рассматриваемых белков (рис. 2).
Рис. 2.
Схема поиска последовательностей, кодирующих участки гомоповторов, и сайтов сплайсинга в пре-мРНК для этих участков. DATA – cписок белков человека из базы данных HRaP, у которых длина гомоповторов больше 4. Для каждого гена при помощи базы данных Ensembl получены их положения в геноме, после чего при помощи программы exonerate гены выровнены на нужные последовательности. Разрывы в выравнивании считали сайтами сплайсинга (ANSWER).
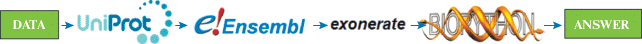
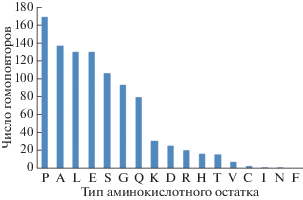
Наибольшее число гомоповторов длиной больше 4 а.о. соответствовали глутаминовой кислоте и пролину (рис. 3). Следует отметить, что 8145 из 59 053 белков человека содержат гомоповторы длиной более 4 а.о., что составляет 14% от протеома человека. Если рассмотреть гомоповторы длиной более 4 а.о. в белках, ассоциированных с болезнями (758 из 2501 записей), то доля таких белков возрастает до 32%, что указывает на связь гомоповторов с тем или иным заболеванием. Для этой базы белков число гомоповторов уменьшилось уже на порядок; причем в лидерах пролиновый гомоповтор (рис. 4).
Рис. 3.
Число гомоповторов длиной более 4 а.о. по базе данных белков HRaP для протеома человека. Данные расположены в порядке убывания.
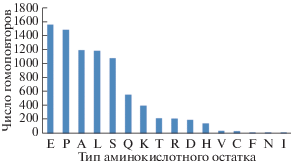
Рис. 4.
Число гомоповторов длиной более 4 а.о., встречающихся в белках человека, ассоциированных с болезнями, для 17 аминокислот. Данные расположены в порядке убывания.
В рассматриваемых гомоповторсодержащих белках идентифицировано 404 гомоповтора, на которые пришелся хотя бы один сайт сплайсинга. Оказалось, что в процентном соотношении сайтов сплайсинга больше для гомоповторов фенилаланина и изолейцина, а также для лизина и аспарагиновой кислоты (не менее 10% от общего числа гомоповторов для данной аминокислоты, рис. 5). Но в численном выражении фенилаланиновых и изолейциновых гомоповторов мало (18 и 16 соответственно), а лизина и аспарагиновой кислоты много (394 и 198 соответственно) (см. табл. 1).
Рис. 5.
Процентное содержание сайтов сплайсинга для гомоповтора конкретной аминокислоты. Данные расположены в порядке, который представлен на рис. 3.
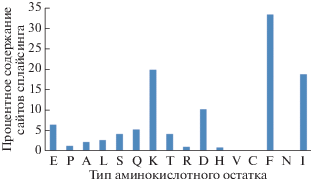
Таблица 1.
Число гомоповторов, которые приходятся на сайты сплайсинга в пре-мРНК, и средняя длина гомоповторов для разных аминокислот
| Гомо-повторы | F | I | L | A | G | T | S | Q | E | D | H | R | K | P |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| N1 | 6 | 3 | 30 | 25 | 41 | 9 | 43 | 29 | 99 | 20 | 1 | 2 | 78 | 18 |
| N2 | 18 | 16 | 1183 | 1194 | 837 | 219 | 1077 | 557 | 1557 | 198 | 142 | 209 | 394 | 1484 |
| L | 6 | 6 | 5.7 | 6.6 | 5.8 | 5.7 | 5.9 | 10.2 | 6.8 | 5.5 | 6 | 6 | 7.1 | 5.3 |
Если оценить среднюю длину гомоповторов, на которые приходятся сайты сплайсинга в пре-мРНК, то она составляет 6 а.о. (табл. 1). Самые длинные участки, которые приходятся на сайты сплайсинга в пре-мРНК, соответствуют глутаминовым повторам: максимальная длина составляет 26 а.о., а средняя 10 а.о. Следует отметить также гомоповторы лизина и глутаминовой кислоты – их средняя длина составила 7 остатков.
Каким областям гомоповторов соответствуют сайты сплайсинга в пре-мРНК?
После идентификации последовательностей, кодирующих гомоповторы, и сайтов сплайсинга, которые попадают в эти гомоповторы, возник вопрос: каким областям гомоповторов соответствуют сайты сплайсинга в пре-мРНК (рис. 1). В результате проведенного анализа обнаружено, что чаще всего сайты сплайсинга расположены на С-концевом участке гомоповтора (67%) и гораздо реже в области N-конца (18%). Следует отметить, что сайты сплайсинга в пре-мРНК, приходящиеся на лейциновые и изолейциновые гомоповторы, попадают только в середину и на С-конец гомоповтора.
Существует ли несколько сайтов сплайсинга в пре-мРНК, приходящихся на гомоповтор в белке?
Наличие двух сайтов сплайсинга в пре-мРНК, приходящихся на участок гомоповтора в белке, может явно указывать на альтернативный сплайсинг. В данной работе мы попытались ответить на этот вопрос о существовании альтернативного сплайсинга в составе гомоповторов. Среди 404 гомоповторов, в которых найден хотя бы один сайт сплайсинга, обнаружено 10 гомоповторов, на которые приходятся два сайта сплайсинга в пре-мРНК. Оказалось, что 9 гомоповторов состоят из остатков лизина и один представлен остатками глутамина (табл. 2), все лизиновые гомоповторы расположены на С-концевом участке белка; из чего можно предположить важность этой области белка для выполнения его функции.
Таблица 2.
Белки человека, для которых найдено два сайта сплайсинга в пре-мРНК, приходящихся на участок гомоповтора в белке
| Название белка (Uniprot) | Используемые кодоны для гомоповторов, на которые приходится 2 сайта сплайсинга. В квадратных скобках [] обозначены участки РНК, находящиеся между сайтами сплайсинга. | Длина гомоповтора |
|---|---|---|
| Лизин | ||
| Q05CP0 | [AAGAAA][AAAAAAAAAAAAAAAAAAAAA][] | 9 |
| Q3B797 | [AGA][AAGAAAAAAAAAAAGAAAAAG][] | 8 |
| Q05CI3 | [GAAAAGAAA][TAAAAAAAAAAAAAAAAAAAA][] | 10 |
| Q6PIS3 | [CAAAAAAAAAAAAAAAAA][AAAAAAAAAAAAAAAAAAAAA][] | 13 |
| Q6PG47 | [AAA][AAAAAAAAAAAAAAAAAAAAAAAAAAA][] | 10 |
| A0PJ62 | [AAGAAAATG][AAATCAAAAAAAAAAAAA][] | 9 |
| Q0VGL2 | [CAATTAGAG][AAAAAAAAAAAAAAA][] | 8 |
| Q05CP4_ | [AAAAAA][AAAAAAAAAAAAAAAAAAAAA][] | 9 |
| Q0VGM1 | [AAA][AAAAAAAAAAAAAAAAAAAATCAA][] | 9 |
| Глутамин | ||
| A8MTU2 | [CAACAACAGCAG][CAACAACAACAAAAC][CAGCAGCAACAGCAA] | 14 |
Далее предстояло дать ответ на следующий вопрос: сколько белков, в которых найдены сайты сплайсинга в пре-мРНК, приходящиеся на гомоповторы, ассоциировано с тем или иным заболеванием по базе данных OMIM. Доля таких белков составила около 9% (36/404 = 0.089). Это вдвое больше, чем доля белков в протеоме человека, для которых к настоящему времени выявлена ассоциация с болезнями, а именно: из общего числа белков в протеоме человека (59 053) с болезнями связано около 4% (2 501).
ЗАКЛЮЧЕНИЕ
Таким образом, в результате проведенного исследования впервые показано, что в протеоме человека есть несколько сотен белков, содержащих гомоповторы аминокислот, которые приходятся на сайты сплайсинга в соответствующих пре-мРНК. Обнаружено, что чаще всего участки сплайсинга в пре-мРНК расположены на С-концевых областях гомоповторов (67%). Два сайта сплайсинга в пре-мРНК, приходящиеся на участок гомоповтора, и, скорее всего, связанные с альтернативным сплайсингом, обнаружены только для гомоповторов лизина и глутамина (9 и 1 случай соответственно). Полученные результаты представляют большой интерес и послужат основой для изучения возможных функций гомоповторов в протеоме человека. В связи с этим интересно сравнить статистику и паттерны сплайсинга в гомоповторах белков человека с другими организмами и проанализировать пре-мРНК онкологических больных.
Авторы выражают признательность Лобанову М.Ю., Глякиной А.В. и Довидченко Н.В. за помощь в оформлении данной рукописи. Мы благодарны сотрудникам Института биоинформатики и компании EPAM за организацию хакатона BioHack2017, который проходил в Санкт-Петербурге в ИТМО, и на котором выполнена часть работы.
Работа выполнена при финансовой поддержке Российского научного фонда (грант № 18-14-00321).
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Blencowe B.J. (2006) Alternative splicing: new insights from global analyses. Cell. 126(1), 37–47.
Buljan M., Chalancon G., Dunker A. K., Bateman A., Balaji S., Fuxreiter M., Babu M.M. (2013) Alternative splicing of intrinsically disordered regions and rewiring of protein interactions. Curr. Opin. Struct. Biol. 23(3), 443–450.
Buljan M., Chalancon G., Eustermann S., Wagner G.P., Fuxreiter M., Bateman A., Babu M.M. (2012) Tissue-specific splicing of disordered segments that embed binding motifs rewires protein interaction networks. Mol. Cell. 46(6), 871–883.
Jorda J., Xue B., Uversky V.N., Kajava A.V. (2010) Protein tandem repeats – the more perfect, the less structured. FEBS J. 277(12), 2673–2682.
Lobanov M.Y., Furletova E.I., Bogatyreva N.S., Roytberg M.A., Galzitskaya O V. (2010) Library of disordered patterns in 3D protein structures. PLoS Comput. Biol. 6(10), e1000958.
Lobanov M.Y., Galzitskaya O.V. (2012) Occurrence of disordered patterns and homorepeats in eukaryotic and bacterial proteomes. Mol. Biosyst. 8(1), 327–337.
Lobanov M.Y., Galzitskaya O.V. (2011) Disordered patterns in clustered Protein Data Bank and in eukaryotic and bacterial proteomes. PLoS One. 6(11), e27142.
Lobanov M.Y., Galzitskaya O.V. (2015) How common is disorder? Occurrence of disordered residues in four domains of life. Int. J. Mol. Sci. 16(8), 19490–19507.
Gatchel J.R., Zoghbi H.Y. (2005) Diseases of unstable repeat expansion: mechanisms and common principles. Nat. Rev. Genet. 6(10), 743–755.
La Spada A.R., Taylor J.P. (2010) Repeat expansion disease: progress and puzzles in disease pathogenesis. Nat. Rev. Genet. 11(4), 247–258.
Usdin K. (2008) The biological effects of simple tandem repeats: lessons from the repeat expansion diseases. Genome Res. 18(7), 1011–1019.
La Spada A.R. (1997) Trinucleotide repeat instability: genetic features and molecular mechanisms. Brain Pathol. Zurich Switz. 7(3), 943–963.
Lobanov M.Y., Klus P., Sokolovsky I.V., Tartaglia G.G., Galzitskaya O.V. (2016) Non-random distribution of homo-repeats: links with biological functions and human diseases. Sci. Rep. 6, 26941.
Лобанов М.Ю., Богатырева Н.С., Галзитская О.В. (2012) Встречаемость мотивов из шести аминокислотных остатков в трех эукариотических протеомах. Молекуляр. биология. 46, 184–190.
Neueder A., Landles C., Ghosh R., Howland D., Myers R.H., Faull R.L.M., Tabrizi S.J., Bates G.P. (2017) The pathogenic exon 1 HTT protein is produced by incomplete splicing in Huntington’s disease patients. Sci. Rep. 7(1), 1307.
Lobanov M.Y., Sokolovskiy I.V., Galzitskaya O.V. (2014) HRaP: database of occurrence of HomoRepeats and patterns in proteomes. Nucleic Acids Res. 42(Database issue), D273–D278.
Yates A., Akanni W., Amode M. R., Barrell D., Billis K., Carvalho-Silva D., Cummins C., Clapham P., Fitzgerald S., Gil L., Girón C. G., Gordon L., Hourlier T., Hunt S.E., Janacek S.H., Johnson N., Juettemann T., Keenan S., Lavidas I., Martin F.J., Maurel T., McLaren W., Murphy D.N., Nag R., Nuhn M., Parker A., Patricio M., Pignatelli M., Rahtz M., Riat H.S., Sheppard D., Taylor K., Thormann A., Vullo A., Wilder S.P., Zadissa A., Birney E., Harrow J., Muffato M., Perry E., Ruffier M., Spudich G., Trevanion S.J., Cunningham F., Aken B.L., Zerbino D.R., Flicek P. (2016) Ensembl 2016. Nucleic Acids Res. 44(D1), D710–D716.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


