Молекулярная биология, 2019, T. 53, № 4, стр. 692-704
Взаимодействие miR-5p и miR-3p с мРНК ортологичных генов
О. Ю. Юрикова a, Д. Е. Айсина a, Р. Е. Ниязова a, Ш. А. Атамбаева a, S. Labeit b, А. Т. Иващенко a, *
a Научно-исследовательский институт проблем биологии и биотехнологии, аль-Фараби
Казахский национальный университет
050040 Алматы, Казахстан
b Institute for Anaesthesiology and Intensive Operative Care Medical Faculty Mannheim
68135 Mannheim, Germany
* E-mail: a_ivashchenko@mail.ru
Поступила в редакцию 07.11.2018
После доработки 21.02.2019
Принята к публикации 21.02.2019
Аннотация
МиРНК (miR) регулируют экспрессию многих генов и участвуют в развитии ряда заболеваний. Изучены miR, частично или полностью комплементарные 5'- и 3'-нетранслируемым последовательностям (UTR) и кодирующим последовательностям мРНК генов-мишеней. Программа MirTarget, используемая в работе, позволяет обнаруживать сайты связывания (СС) miR в полной нуклеотидной последовательности мРНК и определять характеристики взаимодействия miR с мРНК. В кодирующих последовательностях мРНК гена RTL1 человека и животных выявлены пять пар СС, полностью комплементарных miR-127-5p и miR-127-3p, miR-136-5p и miR-136-3p, miR-431-5p и miR-431-3p, miR-432-5p и miR-432-3p, miR-433-5p и miR-433-3p. СС, полностью комплементарные miR-6720-5p и miR-6720-3p, обнаружены в кодирующих последовательностях гена FOXF2; miR-3187-5p, miR-3187-3p – в кодирующей области гена PLPPR3; miR-4665-5p, miR-4665-3p – в 5'-UTR гена KIAA2026; miR-135a-5p, miR-135a-3p – в 3'-UTR гена GLYCTK; miR-7106-5p, miR-7106-3p – в 3'-UTR гена CCDC42B. СС miR-5p и miR-3p, ассоциированных с геном RTL1, найдены в мРНК 32 генов-мишеней человека. СС miR-5p и miR-3p, ассоциированных с генами FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B, найдены в мРНК 27 генов-мишеней, вовлеченных в развитие ряда заболеваний. Нуклеотидные последовательности miR-5p, miR-3p и СС сохраняются в течение десятков миллионов лет дивергенции видов животных. Определены характеристики связывания miR-3120-3p и miR-3120-5p, miR-196b-3p и miR-196b-5p, miR-125a-3p и miR-125a-3p, let-7e-3p и let-7e-5p, miR-99b-3p с полностью комплементарными им СС в некодирующих генах DMN3OS, HOXA10-AS, SPACA6P-AS.
ВВЕДЕНИЕ
МиРНК (miR), подавляющие экспрессию генов на посттрансляционном уровне [1], состоят из 17–27 н., имеют длину от 5 до 9 нм и наноразмерные пропорции. Сайты связывания (СС) miR в 5'-UTR и кодирующих последовательностях (CDS) мРНК еще недостаточно изучены. В связи с этим разработана программа MirTarget, позволяющая поводить поиск СС во всей нуклеотидной последовательности мРНК. Показано, что мРНК содержат СС miR в 5'-UTR, CDS и 3'-UTR [2–5].
В качестве основных объектов исследования был выбран ген RTL1 и несколько других генов, которые проявляют уникальные свойства при взаимодействии с miR. Ген RTL1 участвует во многих важных биологических процессах. Делеция гена Rtl1 у мыши приводит к развитию таких нарушений, как ожирение, блефарофимоз, дисплазия скелета, изменение липидного обмена и гипоплазия плаценты. Примечательно, что ген Rtl1, регулирующий развитие плаценты, кодирует по меньшей мере семь miR, а уровень его экспрессии контролируется механизмом РНК-интерференции [6–8].
Нуклеотидные последовательности некоторых miR полностью комплементарны своим мРНК-мишеням [9, 10]. Такие miR и их мишени представляют особую важность для изучения эволюции взаимодействия miR с мРНК. Обнаружено, что семь miR комплементарно связываются с мРНК гена RTL1 [11]. Эти miR могут подавлять экспрессию гена RTL1, действуя, возможно, как siРНК. Кроме того, ген RTL1, miR-136, miR-127 и другие miR экспрессируются в плаценте [12].
Необходимо выяснить особенности взаимодействий между этими miR и мРНК гена RTL1 и установить, существуют ли другие гены-мишени miR, ассоциированных с геном RTL1. Гены RTL1 и miR-127-5p, miR-127-3p, miR-135a-5p, miR-135a-3p, miR-136-5p, miR-136-3p, miR-431-5p, miR-431-3p, miR-432-5p, miR-432-3p, miR-433-5p и miR-433-3p кодируются в одном локусе антипараллельных цепей ДНК, и поэтому мы называем их miR, ассоциированными с геном RTL1 [11, 12]. Необходимо изучить характеристики взаимодействия пар miR-5p и miR-3p (miR-5p/miR-3p) с 5'‑UTR, CDS и 3'-UTR мРНК других генов. Важно определить, представлены ли в геноме человека другие гены, кодирующие miR, способные ингибировать экспрессию ассоциированных генов. Присутствие таких СС в мРНК ортологичных генов служит доказательством регуляции экспрессии генов miR на ранних этапах эволюции животных. Наличие такой регуляции может служить основанием для использования модельных животных при изучении экспрессии генов с помощью miR.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Нуклеотидные последовательности мРНК генов человека и ортологичных генов получены из базы данных NCBI (http://www.ncbi.nlm.nih.gov) [13]. Значения RPKM (Reads Per Kilobase Million) для FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B взяты из Human Protein Atlas (https:// www.proteinatlas.org). Список видов животных приведен в табл. 1S (см. приложение на сайте http://www.molecbio.ru/downloads/2019/4/supp_Yurikova_rus.pdf). Нуклеотидные последовательности miR человека и животных получены из базы данных miRBase (http://mirbase.org). Последовательности miR-127-5p, miR-127-3p, miR-136-5p, miR-136-3p, miR-431 -5p, miR-431-3p, miR-432-5p, miR-432-3p, miR-433-5p и miR-433-3p, идентичные у человека и животных, приведены в табл. 2S приложения. Нуклеотидные последовательности miR-135a-3p и miR-135a-5p идентичны у Homo sapiens и животных (табл. 3S приложения). У некоторых видов животных miR еще не выявлены, поэтому мы использовали miR человека.
СС miR в 5'-UTR, CDS и 3'-UTR мРНК нескольких генов предсказаны с помощью программы MirTarget [14]. Эта программа определяет следующие характеристики связывания: а) начало связывания miR с мРНК; б) локализацию СС miR в 5'-UTR, CDS и 3'-UTR мРНК; в) свободную энергию взаимодействия (ΔG, кДж/моль), оцениваемую для всей нуклеотидной последовательности miR; г) схемы взаимодействия между нуклеотидами miR и мРНК. Отношение ΔG/ΔGm (%) определяли для каждого сайта, где ΔGm – свободная энергия связывания miR с полностью комплементарной ей нуклеотидной последовательностью. Отбирали только СС miR с отношениями ΔG/ΔGm 90% или более. Программа определяет положение СС в мРНК, начиная с первого нуклеотида 5'-UTR мРНК. Программа MirTarget учитывает водородные связи между аденином (A) и урацилом (U), гуанином (G) и цитозином (C), G и U, A и C. Расстояние между A и C равно 1.04 нм, между G и C, A и U ‒ 1.03 нм, а между G и U – 1.02 нм [15, 16]. Следовательно, из всех возможных пар нуклеотидов только G-C, A-U, G-U и A-C способствуют образованию двухцепочечной структуры РНК без нарушения стэкинг-взаимодействий. Число водородных связей при взаимодействии G-C, A-U, G-U и A-C равно 3, 2, 1 и 1 соответственно. Программа MirTarget не работает непосредственно с базами данных miRBase или NCBI. Поиск генов-мишеней известных miR, представленных в miRBase, в выборке из приведенных в специальный формат 17508 генов, взятых из NCBI, доступен по запросу на mirtarget8@gmail.com.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
С помощью программы MirTarget определены СС 2565 miR с мРНК 17508 генов человека. Выявлено несколько десятков генов, в 5'-UTR, CDS или 3'-UTR мРНК которых содержались СС, комплементарные miR. Из 17508 белоккодирующих генов, только шесть служили мишенями для miR-5p/miR-3p. Мишенями этих miR были также три некодирующих гена.
Сайты связывания miR-5p и miR-3p в мРНК RTL1
мРНК генов RTL1, FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B человека и ассоциированных miR-5p/miR-3p транскрибируются с противоположных комплементарных цепей ДНК [12, 17]. Их транскрипция и дальнейший процессинг пре-мРНК и при-miR происходят независимо друг от друга. Следовательно, концентрации мРНК, miR-5p/miR-3p могут значительно различаться, что отразится на вероятности их взаимодействия.
Мы установили, что CDS мРНК гена RTL1 содержит пять пар СС miR-5p/miR-3p. Представлены характеристики полностью комплементарного взаимодействия этих miR и мРНК генов RTL1, CCDC42B, FOXF2, GLYCTK, KIAA2026, PLPPR3 (табл. 1). Семь из этих 10 miR в мРНК RTL1 были идентифицированы ранее [11, 18]. Ген RTL1 – единственный из 17508 генов человека, содержит пять пар СС, полностью комплементарных miR-5p/miR-3p в CDS.
Таблица 1.
Характеристики взаимодействий miR-5p/miR-3p с мРНК генов RTL1, CCDC42B, FOXF2, GLYCTK, KIAA2026, PLPPR3 человека
| Ген | miR | Начало сайта, н. | ΔG, кДж/моль | ΔG/ΔGm, % | Длина, н. |
|---|---|---|---|---|---|
| RTL1 | miR-127-3p | 1791 | –121 | 100 | 22 |
| То же | miR-127-5p | 1825 | –119 | 100 | 22 |
| » | miR-136-3p | 76 | –113 | 100 | 22 |
| » | miR-136-5p | 109 | –115 | 100 | 23 |
| » | miR-431-3p | 3757 | –121 | 100 | 22 |
| » | miR-431-5p | 3801 | –115 | 100 | 21 |
| » | miR-432-3p | 283 | –115 | 100 | 21 |
| » | miR-432-5p | 329 | –123 | 100 | 23 |
| » | miR-433-3p | 2877 | –119 | 100 | 22 |
| » | miR-433-5p | 2929 | –115 | 100 | 22 |
| » | miR-1296-5p | 3992 | –113 | 91 | 22 |
| » | miR-762 | 3405 | –127 | 94 | 22 |
| CCDC42B | miR-7106-3p | 1350* | –127 | 100 | 23 |
| CCDC42B | miR-7106-5p | 1390* | –113 | 100 | 20 |
| FOXF2 | miR-6720-3p | 491 | –134 | 100 | 22 |
| FOXF2 | miR-6720-5p | 526 | –123 | 100 | 23 |
| GLYCTK | miR-135a-3p | 2773* | –121 | 100 | 22 |
| GLYCTK | miR-135a-5p | 2811* | –113 | 100 | 23 |
| KIAA2026 | miR-4665-3p | 107** | –159 | 100 | 26 |
| KIAA2026 | miR-4665-5p | 146** | –136 | 100 | 23 |
| PLPPR3 | miR-3187-3p | 1229 | –117 | 100 | 20 |
| PLPPR3 | miR-3187-5p | 1263 | –132 | 100 | 23 |
| PPP2CA | miR-3661 | 462 | –123 | 100 | 22 |
| То же | miR-3960 | 125** | –113 | 90 | 20 |
| » | miR-4459 | 126** | –115 | 90 | 22 |
Один из способов определения статистической значимости СС miR в мРНК – подтверждение присутствия таких сайтов в мРНК ортологичных генов. Мы предположили, что ортологичные miR гомологичны miR человека, и использовали hsa-miR для обнаружения СС в мРНК ортологичных генов, у которых не найдены соответствующие miR. Многие miR высококонсервативны в ходе эволюции. СС miR-127-3p и miR-127-5p обнаружены в мРНК ортологичных генов 42 видов животных (табл. 4S (см. приложение на сайте http://www.molecbio.ru/downloads/2019/4/supp_ Yurikova_rus.pdf). СС miR-127-3р и miR-127-5р кодируют, соответственно, олигопептиды QPSSDGSD и SEPSELQ во всех белках RTL1 этих видов (табл. 4S). Нуклеотидные последовательности СС miR-127-3p и miR-127-5p комплементарны этим miR, значение ΔG/ΔGm равно 100%, а значения ΔG равны –121 и –119 кДж/моль соответственно (табл. 1). Олигопептиды QPSSDGSD и SEPSELQ, кодируемые СС miR-127-3p и miR-127-5p, и аминокислотные остатки между ними идентичны в белках всех видов. Вариабельность аминокислотных остатков вне олигопептидов указывает, что за время дивергенции видов Ailuropoda melanoleuca и H. sapiens консервативные олигопептиды QPSSDGSD и SEPSELQ остаются функционально более важными, чем фланкирующие олигопептиды.
Нуклеотидные последовательности miR-136-3p и miR-136-5p комплементарны мРНК гена RTL1 39 видов, ΔG/ΔGm равна 100%, а значения ΔG составляют –113 и –115 кДж/моль соответственно (табл. 1). Олигопептиды DSFETMM и PSSKQME, кодируемые СС miR-136-3p и miR-136-5p, идентичны в белках RTL1 всех видов (табл. 4S). Диаграмма Logo показывает вариабельность аминокислотных остатков белка RTL1, расположенных между DSFETMM и PSSKQME, и в областях, фланкирующих эти олигопептиды (табл. 4S).
miR-431-3p и miR-431-5p имеют комплементарные СС в мРНК RTL1, т.е. ΔG/ΔGm равна 100%, а значения ΔG равны –121 и –115 кДж/моль соответственно (табл. 1). СС miR-431-3p и miR-431-5p кодируют консервативные олигопептиды EALQDDL и HDGLQD во всех белках RTL1 34 видов животных (табл. 4S).
Нуклеотидные последовательности СС miR-432-3p и miR-432-5p комплементарны мРНК 38 видов животных (табл. 4S). Таким образом, ΔG/ΔGm равна 100%, а значения ΔG составляют –115 и ‒123 кДж/моль соответственно (табл. 1). Соответствующие СС кодируют консервативные олигопептиды DMEEPSS и PPNDLLQ во всех белках RTL1 этих видов (табл. 4S). Между олигопептидами DMEEPSS и PPNDLLQ белка RTL1 и в областях, фланкирующих эти олигопептиды, расположены вариабельные аминокислотные остатки.
Нуклеотидные последовательности СС miR-433-3p и miR-433-5p комплементарны мРНК 40 видов животных (табл. 4S). ΔG/ΔGm равна 100%, а значения ΔG составляют –119 и –115 кДж/моль (табл. 1). Эти сайты кодируют олигопептиды НТEEPIMI и NNDRLTV (табл. 4S).
Нуклеотидные последовательности между СС пяти пар miR-5p/miR-3p кодируют олигопептиды различных белков RTL1 у животных разных видов. Все СС пяти пар miR в гене RTL1 распределены приблизительно равномерно по длине мРНК. Диаграмма Logo показывает консервативность олигопептидов, кодируемых СС пар miR-3p и miR-5p. Аминокислоты вне этих олигопептидов и между ними характеризуются вариабельностью (табл. 4S). Следовательно, СС miR-5p/miR-3p в мРНК RTL1 эволюционно древние и сохраняются в течение десятков миллионов лет, что указывает на функциональную важность как их нуклеотидных последовательностей, так и кодируемых ими олигопептидов.
В дополнение к 10 miR, ассоциированным с геном RTL1, miR-1296-5p и miR-762 могут связываться с мРНК RTL1. СС miR-1296-5р и miR-762 расположены в CDS и кодируют олигопептиды EQAARAL и ARPQPQR в белке RTL1. СС этих miR обнаружены в мРНК генов-ортологов RTL1 только у гоминидов и обезьян. Свободная энергия взаимодействия miR-762 с мРНК гена RTL1 составляет –127 кДж/моль и превышает свободную энергию взаимодействия каждой из 10 miR, ассоциированных с геном RTL1 (табл. 1). При сопоставимых концентрациях каждая из 12 miR может подавлять синтез белка RTL1, поскольку их СС не перекрываются и распределены по всей длине мРНК. Поскольку 10 miR могут экспрессироваться с антисмысловой цепи ДНК RTL1 независимо от транскрипции и процессинга мРНК RTL1, вполне вероятным представляется предположение о равной возможности связывания 12 miR с мРНК. Экспериментально показано, что метилирование ДНК отрицательно коррелирует с экспрессией гена RTL1, но положительно – с экспрессией miR-136 в плаценте человека [12]. Т.е. изменения в экспрессии гена и miR-136 соответствуют предполагаемой взаимосвязи miR с геном-мишенью. В качестве аналогичного примера мы проверили экспериментально установленную связь между miR-3661 и геном-мишенью PPP2CA [17]. Программа MirTarget подтвердила полную комплементарность их взаимодействия в CDS и выявила более двух альтернативных miR, которые могут связываться в 5'-UTR мРНК гена PPP2CA. Количественные характеристики взаимодействия мРНК гена с miR-3661, miR-3960 и miR-4459 приведены в табл. 1 и указывают на их сходство.
Необходимо было установить, существуют ли другие гены-мишени miR, ассоциированных с геном RTL1. Наши результаты показали, что miR, ассоциированные с RTL1, имеют более 30 генов-мишеней (табл. 2), которые участвуют в развитии онкологических, сердечно-сосудистых, нейродегенеративных и других заболеваний [19–25]. Следовательно, miR-5p/miR-3p не только подавляют экспрессию ассоциированного гена RTL1, но и могут регулировать трансляцию мРНК генов, вовлеченных в развитие различных заболеваний, что указывает на высокую биологическую значимость miR, ассоциированных с RTL1.
Таблица 2.
Характеристики взаимодействия miR с мРНК их генов-мишеней
| miR | Ген-мишень | Начало сайта, н. | ΔG, кДж/моль | ΔG/ΔGm, % |
Заболевание или функция гена | PMID |
|---|---|---|---|---|---|---|
| miR-127-3p | BDP1 | 673 | –113 | 93 | Транскрипция | 11 040 218 |
| miR-127-3p | EIF3B | 438 | –110 | 91 | Трансляция | 8 995 410 |
| miR-127-5p | CPNE2 | 1915 | –108 | 91 | Кальций-опосредованные процессы | 9 430 674 |
| miR-127-5p | RNF181 | 144** | –110 | 93 | Опухолевый супрессор | 21 391 225 |
| miR-127-5p | URM1 | 3950* | –108 | 91 | Цитокинез | 19 017 811 |
| miR-136-3p | TAF4 | 2475 | –102 | 91 | Нейродегенеративные заболевания | 10 973 244 |
| miR-136-3p | TTN | 71469 | –102 | 91 | Кардиомиопатия | 11 846 417 |
| miR-136-5p | CAP1 | 1454 | –102 | 89 | Клеточная пролиферация | 27 432 289 |
| miR-136-5p | CLDN15 | 37** | –104 | 91 | Опухолевая инвазия | 19 420 749 |
| miR-136-5p | ENAH | 6968* | –102 | 89 | Опухолевая инвазия | 26 112 005 |
| miR-431-3p | AKAP17A | 696 | –110 | 91 | Протеинкиназа | 16 982 639 |
| miR-431-3p | BCL11A | 1433 | –110 | 91 | Лейкоз | 26 707 798 |
| miR-431-3p | COG6 | 856 | –117 | 96 | Нарушения гликозилирования | 20 605 848 |
| miR-431-5p | SPTB | 1546 | –104 | 91 | Анемия | 9 005 995 |
| miR-432-3p | CCDC135 | 2050 | –104 | 91 | Пептидаза | 22 983 010 |
| То же | CDK5RAP2 | 5810 | –106 | 93 | Болезнь Альцгеймера | 21 297 427 |
| » | CPNE2 | 943 | –108 | 94 | Кальций-опосредованные процессы | 9 430 674 |
| » | CYP2A6 | 450 | –106 | 93 | Онкогенез | 16 176 798 |
| » | FHDC1 | 2139 | –104 | 91 | Формирование комплекса Гольджи | 26 564 798 |
| » | FOXB1 | 108** | –106 | 93 | Транскрипционный фактор | 28 725 186 |
| » | IL2RB | 1013 | –106 | 93 | Рак молочной железы | 14 680 494 |
| » | KCTD19 | 1849 | –104 | 91 | Калиевый канал | 26 350 515 |
| » | PPP1R26 | 942 | –106 | 93 | Онкогенез | 26 442 585 |
| » | PRKCSH | 1491 | –104 | 91 | Поликистоз печени | 11 047 756 |
| » | RBP3 | 252 | –104 | 91 | Ретинол-связывающий белок | 2 542 268 |
| » | SH2D5 | 2222* | –104 | 91 | Синаптическая пластичность | 28 103 696 |
| » | SLC22A12 | 2172 | –104 | 91 | Регуляция уровня солей мочевой кислоты в крови | 12 024 214 |
| miR-432-5p | CD177 | 1058 | –110 | 90 | Активация нейтрофилов | 17 580 308 |
| То же | EPB41L1 | 339 | –110 | 90 | Опухолевый супрессор | 26 575 790 |
| » | НТN1 | 4801 | –110 | 90 | Кардиопротектор | 20 036 673 |
| » | PRSS57 | 373 | –113 | 91 | Нейтрофильный ответ | 26 172 376 |
| » | RBP7 | 329 | –110 | 96 | Метаболизм витамина А | 12 177 003 |
Свободная энергия взаимодействия miR-127-5p и miR-127-3p варьирует в диапазоне от –108 до –113 кДж/моль (табл. 2), что составляет 91–93% свободной энергии взаимодействия этих miR с мРНК гена RTL1 (табл. 1). Эти результаты позволяют предположить, что 10 miR, ассоциированных с геном RTL1, участвуют в важных процессах в норме и при патологии. miR-3p и miR-5p могут иметь различное количество генов-мишеней. Например, miR-432-3p и miR-432-5p имеют 13 и четыре гена-мишени соответственно (табл. 2). Однако важно, чтобы обе miR обладали одинаковой консервативностью нуклеотидных последовательностей. Ассоциированные с геном RTL1 miR связываются с CDS мРНК, а их СС кодируют олигопептиды, приведенные в Дополнительном файле 4. Значения свободной энергии взаимодействия пяти пар miR-5p/miR-3p с мРНК гена RTL1 примерно равны (табл. 1). Свободная энергия взаимодействия этих пар miR-5p/miR-3p с мРНК других генов (табл. 2) варьирует от –102 до –117 кДж/моль.
СС в мРНК ортологичных генов мы определяли с использованием hsa-miR, предполагая существование гомологии между miR ортологичных генов и miR человека (табл. 2S). Как правило, многие miR эволюционно высококонсервативны (табл. 2S и 3S) [26]. Следовательно, все млекопитающие, включая лабораторных животных, таких как крысы, мыши и собаки, могут служить модельными объектами для изучения взаимодействия miR с мРНК RTL1 человека.
Взаимодействие miR-6720-5p и miR-6720-3p с мРНК FOXF2
В результате анализа 17508 генов человека мы выявили (помимо RTL1) пять мРНК, которые содержат СС пар miR-3p и miR-5p, полученных из одной пре-miR. СС пар miR-3p и miR-5p обнаружены в 5'-UTR, 3'-UTR и CDS мРНК генов FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B.
СС miR-6720-3p и miR-6720-5p найдены в CDS мРНК FOXF2. Свободная энергия взаимодействия miR-6720-5p и miR-6720-3p с мРНК FOXF2 человека составляет –134 и –123 кДж/моль соответственно (табл. 1). СС miR-6720-3p и miR-6720-5p кодируют три пары олигопептидов, консервативных в каждой из трех групп мРНК 34 видов животных (табл. 5S). Данные, представленные в дополнительном файле 5, показывают, что аминокислотные остатки, входящие в состав олигопептидов IYQFLQAR и RGAYQGWK белка FOXF2 консервативны у 15 видов животных. В другой группе животных СС miR-6720-3p и miR-6720-5p кодируют олигопептиды IYQFLQSR и RGSYQGWK (табл. 5S, В). У животных третьей группы СС miR-6720-3p и miR-6720-5p кодируют олигопептиды IYQSLQАR и RGAYQGWK (табл. 5S, С). Таким образом, взаимодействие между miR и мРНК 34 генов возникло более 10 миллионов лет назад и сохранялось при дивергенции исследуемых видов животных. Нуклеотидные последовательности, локализованные между СС miR-6720-3p и miR-6720-5p, кодируют как консервативный тетрапептид FPFF, так и прилегающий олигопептид NSVRHNLSLNECF.
Определены характеристики СС miR-6720-5p и miR-6720-3p в семи мРНК генов-мишеней, связанных с канцерогенезом молочной железы, толстой кишки, поджелудочной железы и щитовидной железы. СС этих miR расположены только в CDS, а свободная энергия связывания составляет от –110 до –121 кДж/моль (табл. 3).
Таблица 3.
Характеристики взаимодействия miR-5p/miR-3p (ассоциированных с генами CCDC42B, FOXF2, GLYCTK, KIAA2026, PLPPR3) с мРНК генов-мишеней
| miR (ассоциированный ген) | Ген-мишень | Начало сайта, н. | Участок мРНК | ΔG, кДж/моль | ΔG/ΔGm, % | Длина, н. |
|---|---|---|---|---|---|---|
| miR-7106-5p(CCDC42B) | DDX20 | 304 | 5'-UTR | –104 | 92 | 20 |
| То же | KIRREL | 314 | 5'-UTR | –104 | 92 | 20 |
| » | MAML1 | 4045 | 3'-UTR | –106 | 94 | 20 |
| » | MAPKAPK2 | 2892 | 3'-UTR | –104 | 92 | 20 |
| » | MAPT | 1007 | CDS | –106 | 94 | 20 |
| » | SBK1 | 2495 | 3'-UTR | –106 | 94 | 20 |
| » | TEAD2 | 1460 | 3'-UTR | –104 | 92 | 20 |
| miR-7106-3p(CCDC42B) | FAM213B | 86 | CDS | –115 | 90 | 23 |
| miR-7106-3p(CCDC42B) | FANCG | 831 | CDS | –115 | 90 | 23 |
| miR-7106-3p(CCDC42B) | GMEB2 | 2965 | 3'-UTR | –115 | 90 | 23 |
| miR-6720-5p(FOXF2) | CPAMD8 | 148 | CDS | –121 | 90 | 23 |
| miR-6720-5p(FOXF2) | FOXF1 | 298 | То же | –121 | 90 | 23 |
| miR-6720-3p(FOXF2) | ACTN2 | 958 | » | –110 | 90 | 22 |
| То же | B3GНТ1 | 176 | » | –110 | 90 | 22 |
| » | EVPL | 2656 | » | –113 | 91 | 22 |
| » | SLC25A29 | 568 | » | –115 | 93 | 22 |
| » | ZNF653 | 1821 | » | –110 | 90 | 22 |
| miR-135a-5p (GLYCTK) | FHL5 | 2659 | 3'-UTR | –104 | 92 | 23 |
| miR-135a-3p(GLYCTK) | ILDR1 | 1688 | CDS | –110 | 91 | 22 |
| miR-4665-5p(KIAA2026) | EFEMP1 | 266 | 5'-UTR | –123 | 91 | 23 |
| miR-4665-5p(KIAA2026) | ZFP36L2 | 1610 | CDS | –123 | 91 | 23 |
| miR-3187-5p(PLPPR3) | ITGA1 | 57 | CDS | –119 | 90 | 23 |
| miR-3187-5p(PLPPR3) | OGDH | 3408 | 3'-UTR | –125 | 95 | 23 |
| miR-3187-5p (PLPPR3) | PELO | 419 | 5'-UTR | –119 | 90 | 23 |
| miR-3187-3p(PLPPR3) | CNGB1 | 1717 | CDS | –106 | 91 | 20 |
| miR-3187-3p(PLPPR3) | DENND4B | 3667 | CDS | –108 | 93 | 20 |
| miR-3187-3p(PLPPR3) | SMARCA4 | 684 | CDS | –108 | 93 | 20 |
Взаимодействие miR-3187-5p и miR-3187-3p с мРНК PLPPR3
СС miR-3187-5p и miR-3187-3p обнаружены в CDS мРНК PLPPR3. Энергия взаимодействия miR-3187-5p длиной 23 н. и miR-3187-3p длиной 20 н. с мРНК составляет –132 и –117 кДж/моль соответственно. Олигопептиды, кодируемые СС miR-3187-5p и miR-3187-3p, консервативны в мРНК PLPPR3 37 видов животных (табл. 6S). Ортологичные белки PLPPR3 содержат последовательности PRSPMAK и TFSHTLPR, кодируемые СС miR-3187-5p и miR-3187-3p. Однако аминокислоты, фланкирующие эти последовательности, были вариабельными (табл. 6S, C). Шесть СС miR-3187-5p и miR-3187-3p, ассоциированных с геном PLPPR3, обнаружены в генах-мишенях, участвующих в развитии рака молочной железы, желудка, яичника, колоректального рака, спорадической аденомы гипофиза (табл. 3). СС miR расположены в 5'-UTR, 3'-UTR и CDS, свободная энергия их взаимодействия c мРНК изменялась от –106 до –125 кДж/моль.
Взаимодействие miR-4665-5p и miR-4665-3p с мРНК KIAA2026
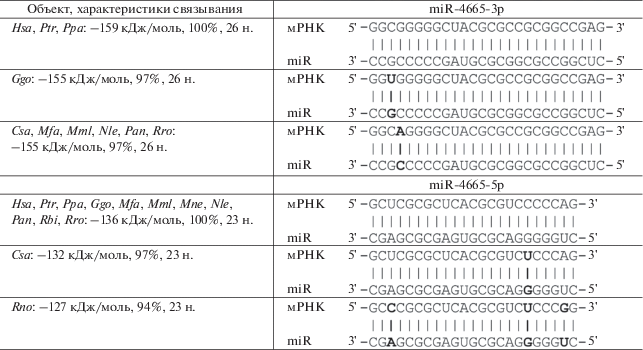
СС miR-4665-5p и miR-4665-3p обнаружены в 5'-UTR мРНК KIAA2026 (табл. 4). Энергия взаимодействия miR-4665-3p (26 н.) с мРНК гена KIAA2026 у H. sapiens, Pan troglodytes и Pan paniscus составляет –159 кДж/моль, а у miR-4665-5p (23 н.) –136 кДж/моль (табл. 4).
Энергия взаимодействия miR-4665-3p с мРНК гена KIAA2026 Gorilla gorilla, Chlorocebus sabaeus, Macaca fascicularis, M. mulatta, Nomascus leucogenys, Papio anubis и Rhinopithecus roxellana составляет –155 кДж/моль благодаря взаимодействию неканонических пар U и G, A и C. miR-4665-5p. Свободные энергии взаимодействия с мРНК гена KIAA2026 C. sabaeus и Rattus norvegicus составляют –132 и –127 кДж/моль, соответственно, так как неканонические пары U-G и A-C также взаимодействуют между собой. miR-4665-5p связывается в 5'-UTR и CDS мРНК EFEMP1 и ZFP36L2, ассоциированных с развитием рака молочной и поджелудочной железы. Они имеют CC с одинаковой степенью комплементарности, равной 91%, и одинаковой свободной энергией взаимодействия, равной –123 кДж/моль (табл. 3).
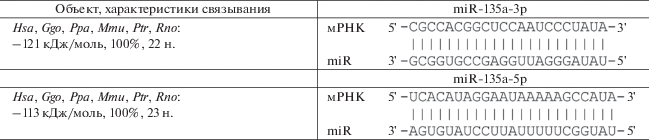
Взаимодействие miR-135a-5p и miR-135a-3p с мРНК GLYCTK
СС miR-135a-5p и miR-135a-3p обнаружены в 3'-UTR мРНК GLYCTK (табл. 5). Свободные энергии взаимодействия miR-135a-5p (23 н.) и miR-135a-3p (22 н.) с мРНК составляют –113 и –121 кДж/моль соответственно (табл. 5). Консервативность нуклеотидных последовательностей СС miR-135a-5p и miR-135a-3p предполагает, что взаимодействие между ними и их геном-мишенью GLYCTK возникло и сохранялось в ходе эволюции. Определены характеристики СС miR-135a-3p и miR-135a-5p. Свободная энергия взаимодействия miR-135a-3p, ассоциированного с геном GLYCTK, с CDS мРНК гена ILDR1, вовлеченного в развитие лимфомы, равна –110 кДж/моль (табл. 3).
Взаимодействие miR-7106-5p и miR-7106-3p с мРНК CCDC42B
СС miR-7106-5p и miR-7106-3p найдены в 3'‑UTR мРНК CCDC42B (табл. 6). Свободная энергия взаимодействия miR-7106-3p длиной 23 н. с мРНК гена CCDC42B H. sapiens, M. mulatta, R. bieti и R. roxellana равна –127 кДж/моль, а свободная энергия взаимодействия miR-7106-5p длиной 20 sн. с мРНК гена CCDC42B H. sapiens, M. mulatta, R. bieti, R. roxellana и Alteromonas naphthalenivorans составляет –113 кДж/моль (табл. 6).
Свободная энергия взаимодействия miR-7106-3p с мРНК гена CCDC42B Pongo abelii и A. naphthalenivorans составляет –125 и –121 кДж/моль соответственно. Свободная энергия взаимодействия miR-7106-5p с мРНК гена CCDC42B P. abelii составляет –108 кДж/моль. Обнаружены 10 СС miR-7106-5p и miR-7106-3p, ассоциированных с геном CCDC42B, в 5'-UTR, 3'-UTR и CDS мРНК генов-кандидатов рака молочной железы, толстой кишки, плоскоклеточного рака пищевода, поджелудочной железы, рака предстательной железы, яичников, шейки матки, желудка, нейробластомы, остеосаркомы, колоректального рака, гепатоцеллюлярной карциномы. Эти miR взаимодействовали с мРНК со свободной энергией от –104 до –115 кДж/моль (табл. 3).
Таким образом, показано, что miR, ассоциированные с генами CCDC42B, FOXF2, GLYCTK, KIAA2026 и PLPPR3, влияют на различные гены-мишени, которые участвуют патогенезе различных онкологических заболеваний. Примечательно, что все нуклеотиды miR и их СС взаимодействовали без образования “пузырей” (табл. 4–6). Взаимодействия нуклеотидов, соединенных водородными связями, дополнительно стабилизировались стэкинг-взаимодействиями всех нуклеотидов. Среднее и стандартное отклонение свободной энергии взаимодействий СС пар miR-5p/miR-3p в мРНК генов FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B варьировало от –144 ± –11 кДж/моль в 5'-UTR, –137 ± –13 кДж/моль в CDS и –118 ± –6 кДж/моль в 3'-UTR. Следовательно, связывание miR в начале мРНК будет более эффективно ингибировать трансляцию, что уменьшит потерю энергии в случае прекращения трансляции.
Взаимодействие miR-5p/miR-3p с некодирующими генами
Показано, что некодирующие белок гены DMN3OS, HOXA10-AS и SPACA6P-AS служат мишенями для miR-5p/miR-3p, кодируемых антипараллельной ДНК (антисмысловой) [12]. В табл. 7 представлены характеристики связывания нуклеотидных последовательностей РНК DMN3OS, HOXA10-AS и SPACA6P-AS (длинные некодирующие РНК, днРНК) с соответствующими miR-5p/miR-3p. В дополнение к miR-3120-3p и miR-3120-5p с днРНК гена DMN3OS могут связываться такие miR, как miR-4506 и miR-6764-3p, предсказанные с помощью анализа 2565 miR из miRBase программой MirTarget. СС miR-3120-3p и miR-3120-5p в днРНК гена DMN3OS разделены участком из 18 н., длина которого соответствует длине участков между CC в гене, кодирующем белок RTL1. Свободная энергия взаимодействия miR-196b-3p и miR-196b-5p с днРНК гена HOXA10-AS составляет –121 и –115 кДж/моль соответственно. miR-3620-5p и miR-3960 взаимодействуют с днРНК гена HOXA10-AS с одинаковой свободной энергией (табл. 7). Т.е., miR-3620-5p и miR-3960 могут одновременно с miR-196b-3p и miR-196b-5p связываться с днРНК гена HOXA10-AS. Расстояние между СС miR-196b-3p и miR-196b-5p в днРНК гена HOXA10-AS составляет 14 н. днРНК гена SPACA6P-AS может связывать три пары miR-5p/miR-3p [12]. Характеристики такого взаимодействия приведены в табл. 7.
Таблица 7.
Характеристики взаимодействий miR-5p/miR-3p с днРНК некодирующих генов DMN3OS, HOXA10-AS, SPACA6P-AS
| Ген | miR | Начало сайта, н. | ΔG, кДж/моль | ΔG/ΔGm, % | Длина, н. |
|---|---|---|---|---|---|
| DMN3OS | miR-3120-3p | 5958 | –113 | 100 | 21 |
| То же | miR-3120-5p | 5996 | –115 | 100 | 21 |
| » | miR-4506 | 3149 | –100 | 94 | 20 |
| » | miR-6764-3p | 450 | –100 | 90 | 21 |
| HOXA10-AS | miR-196b-3p | 282 | –121 | 100 | 22 |
| То же | miR-196b-5p | 317 | –115 | 100 | 22 |
| » | miR-3960-3p | 380 | –115 | 92 | 20 |
| » | miR-3620-5p | 447 | –121 | 92 | 22 |
| SPACA6P-AS | miR-99b-3p | 780 | –123 | 100 | 22 |
| То же | miR-125a-3p | 130 | –121 | 100 | 22 |
| » | miR-125a-5p | 166 | –127 | 100 | 24 |
| » | let-7e-3p | 598 | –119 | 100 | 22 |
| » | let-7e-5p | 643 | –113 | 100 | 22 |
| » | miR-1273g-3p | 2086 | –113 | 96 | 21 |
| » | miR-1285-5p | 3597 | –106 | 94 | 21 |
| » | miR-6741-3p | 724 | –115 | 93 | 22 |
| » | miR-1224-3p | 1365 | –108 | 91 | 21 |
Свободная энергия взаимодействия парных miR-125a-3p и miR-125a-5p с днРНК гена SPACA6P-AS выше, чем у пар let-7e-5p и let-7e-3p, поэтому эти miR могут более эффективно связываться с днРНК SPACA6P-AS. Из 2565 miR, представленных в базе данных miRBase, программа MirTarget предсказала возможность эффективного связывания miR-1273g-3p и miR-1285-5p, miR-6741-3p и miR-1224-3p с днРНК SPACA6P-AS. Расстояние между СС miR-125a-3p и miR-125a-5p в днРНК гена SPACA6P-AS составляло 15 н., а между СС let-7e-5p и let-7e-3p – 24 н. Расстояния между СС четырех пар miR-5p/miR-3p указывают на их сравнимость с расстояниями между miR-5p и miR-3p в большинстве пре-miR. Следовательно, существует сходство во взаимодействии некодирующих и кодирующих генов с miR.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Впервые биоинформатические методы были использованы для идентификации СС пар miR-5p/miR-3p в мРНК генов-мишеней. Обнаружено, что из 17508 генов человека только белоккодирующие гены RTL1, FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B служат мишенями для ассоциированных с ними miR. Ген RTL1 уникален тем, что ассоциированная и ним miR содержит 10 СС, полностью комплементарных пяти парам miR-5p/miR-3p. Нуклеотидные последовательности miR, ассоциированных с другими пятью генами, также полностью комплементарны СС в мРНК генов FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B. Показано, что наряду с изученными белоккодирующими генами RTL1, CCDC42B, FOXF2, GLYCTK, KIAA2026, PLPPR3, некодирующие белок гены DMN3OS, HOXA10-AS и SPACA6P-AS служат мишенями miR-5p/miR-3p, кодируемых антипараллельной ДНК.
Установлено, что miR-127 подавляет экспрессию ассоциированного гена RTL1, возможно, действуя как малая интерферирующая РНК [27]. Маловероятно, что их биологическая роль заключается исключительно в деградации мРНК многих генов-мишеней. Нами обнаружены 10 miR, которые вряд ли будут подавлять синтез белка RTL1. Среди 2565 miR только miR-1296-5p и miR-772 могут связываться с мРНК RTL1. Мы предположили, что СС пар miR-5p/miR-3p возникли в предшественнике (пре-miR), чтобы усилить зависимость экспрессии мРНК-мишени от miR. Кроме того, статистическая значимость этой зависимости увеличивается, когда происходят мутации в СС miR-5p/miR-3p. Биологическая роль RTL1 активно изучается [6–8, 28]. Эта роль весьма значительна, поскольку RTL1 – это единственный известный ген человека, который взаимодействует с пятью парами miR-5p/miR-3p. Нами показано, что пары miR-5p/miR-3p комплементарны СС в CDS мРНК. Аналогичные результаты получены для miR растений [29]. Эти СС в мРНК комплементарны нуклеотидной последовательности соответствующей пре-miR. Аналогичные результаты получены нами для некоторых miR, связывающихся с 3'-UTR мРНК [9].
Полная комплементарность взаимодействия между miR и мРНК соответствует взаимодействию малых интерферирующих РНК с мРНК, но не все они приводят к разрушению мРНК. Косвенно это утверждение подтверждется существованием более 200 генов, мРНК которых содержат комплементарные СС miR-619-5p [9]. В этом случае miR-619-5p должна подавлять экспрессию генов путем деградации мРНК; однако биологическое значение этого феномена трудно объяснить. RTL1 это ассоциированный ген для пяти пар miR-5p/miR-3p. miR-127-5p и miR-127-3p, miR-136-5p и miR-136-3p, miR-431-5p и miR-431-3p, miR-432-5p, miR-432-3p, miR-433-5p и miR-433-3p и ген RTL1 расположены на антипараллельных цепях ДНК и транскрибируются независимо [11]. Спаренные полностью комплементарные СС miR-5p/miR-3p в генах соответствуют пре-miR, кодируемой противоположной цепью ДНК. Повышенный уровень консервативности полностью спаренных СС miR-5p/miR-3p по сравнению с окружающими районами объясняется особенностями профиля консервативности пре-miR.
Эволюция нуклеотидных последовательностей в miR и мРНК протекает совместно (табл. 2S, 3S). Таким образом, если miR существует, то существуют и соответствующие CC в мРНК. Высокая консервативность СС miR в генах RTL1, FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B, которая сохраняется в течение десятков миллионов лет эволюции изучаемых видов, подтверждает, что опосредованная miR регуляция экспрессии этих генов появилась на ранних этапах эволюции животных.
Показано, что FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B играют важную роль в развитии рака молочной железы [30, 31]. Уровень экспрессии генов FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B в молочной железе варьирует от 0.1 до 5.5 RPKM. Forkhead box F2 (FOXF2) функционирует как фактор транскрипции и играет решающую роль в программировании органогенеза и регуляции эпителиально-мезенхимального перехода (EMT) и пролиферации клеток. Данные недавних исследований выявили участие FOXF2 в патогенезе рака молочной железы [32]. Снижение экспрессии мРНК FOXF2 связано с ранним началом метастазирования и плохим прогнозом при раке молочной железы [33]. Изучение гена RTL1 представляет большой интерес, поскольку 10 ассоциированных с ним miR регулируют экспрессию 32 генов (табл. 2), участвующих в развитии сердечно-сосудистых, онкологических, нейродегенеративных и других заболеваний человека. Следовательно, важно знать, у каких экспериментальных животных экспрессия генов RTL1, FOXF2, PLPPR3, KIAA2026, GLYCTK, CCDC42B регулируется с помощью miR, т.е. эти животные могут служить модельными объектами. Полученные результаты указывают на возможность использования животных для анализа влияния miR RTL1. Программа MirTarget позволяет анализировать взаимодействие miR с мРНК ортологичных генов и определять возможность взаимодействия между miR и мРНК генов человека и их ортологов. Полученные результаты показывают, что в качестве адекватных модельных объектов для изучения miR, ассоциированных с генами RTL1, FOXF2, PLPPR3, KIAA2026, GLYCTK, CCDC42B, могут использоваться мыши и крысы. В большинстве работ изучены только некоторые из 10 miR, ассоциированных с RTL1, часто без указания, являются ли они miR-5p/miR-3p [20–24]. Важно контролировать экспрессию всех miR, ассоциированных с генами. Наши результаты показывают, что пары miR-5p/miR-3p могут регулировать экспрессию многих генов и участвовать таким образом в развитии многих заболеваний (табл. 2 и 3). При разработке методов диагностики заболеваний с использованием miR, ассоциированных с генами RTL1, FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B, необходимо учитывать экспрессию miR. Такой контроль будет отражать роль этих участников в патологических процессах. мРНК генов RTL1, FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B имеют полностью комплементарные СС пар miR-5p/miR-3p, консервативных на протяжении десятков миллионов лет дивергенции исследуемых видов. Определены количественные характеристики взаимодействий miR-5p/miR-3p в 5'-UTR, CDS и 3'-UTR мРНК генов-мишеней.
ЗАКЛЮЧЕНИЕ
Определены характеристики связывания пяти пар miR-5p/miR-3p с мРНК гена RTL1, а также взаимодействия пяти пар miR-5p/miR-3p с ассоциированными мРНК генов FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B. днРНК некодирующих генов DMN3OS, HOXA10-AS, SPACA6P-AS имеют СС ассоциированных пар miR-5p/miR-3p. СС miR, ассоциированных с генами RTL1, FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B, найдены в мРНК генов, участвующих в развитии различных заболеваний. Показана консервативность miR и соответствующих СС в ортологичных генах-мишенях. Определены характеристики взаимодействия miR-5p/miR-3p с днРНК генов DMN3OS, HOXA10-AS и SPACA6P-AS. Эти результаты показывают, что некодирующие гены сохраняют некоторые свойства кодирующих.
Мы благодарим студентов А. Акимниязова и Д. Байжигитова за помощь в сборе данных.
Это исследование поддержано грантом (AP05132460) Министерства образования и науки Республики Казахстан, НИИ проблем биологии и биотехнологии, КазНУ им. Аль-Фараби.
ВКЛАД АВТОРОВ
Иващенко Анатолий и Зигфрид Лабейт разработали исследование, подготовили рукопись и дали окончательное согласие на публикацию.
Ниязова Райгуль и Атамбаева Шара разработали исследование и составили рукопись.
Юрикова Оксана получила данные по гену RTL1 для анализа, редактировала рукопись.
Айсина Дана получила данные по генам FOXF2, PLPPR3, KIAA2026, GLYCTK и CCDC42B для анализа, редактировала рукопись. Юрикова О. и Айсина Д. внесли существенный вклад в сбор, обработку и интерпретацию данных.
Все авторы были вовлечены в последующие этапы рецензирования, прочитали и одобрили окончательную рукопись.
В работе не использовали биоматериалов от животных и человека в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов
Список литературы
Bartel D.P. (2018) Metazoan microRNAs. Cell. 173, 20–51.
Lytle J.R., Yario T.A., Steitz J.A. (2007) Target mRNAs are repressed as efficiently by microRNA-binding sites in the 5′ UTR as in the 3′ UTR. Proc. Natl. Acad. Sci. USA. 104, 9667–9672.
Tay Y., Zhang J., Thomson A.M., Lim B., Rigoutsos I. (2008) MicroRNAs to Nanog, Oct4 and Sox2 coding regions modulate embryonic stem cell differentiation. Nature. 455, 1124–1128.
Zhou X., Duan X., Qian J., Li F. (2009) Abundant conserved microRNA target sites in the 5'-untranslated region and coding sequence. Genetica. 137, 159–164.
Ivashchenko A., Berillo O., Pyrkova A., Niyazova R., Atambayeva S. (2014) MiR-3960 binding sites with mRNA of human genes. Bioinformation. 10, 423–427.
Kumamoto S., Takahashi N., Nomura K., Fujiwara M., Kijioka M., Uno Y., Matsuda Y., Sotomaru Y., Kono T. (2017) Overexpression of microRNAs from the Gtl2-Rian locus contributes to postnatal death in mice. Hum. Mol. Genet. 26, 3653–3662.
Ito M., Sferruzzi-Perri A.N., Edwards C.A., Adalsteinsson B.T., Allen S.E., Loo T.H., Kitazawa M., Kaneko-Ishino T., Ishino F., Stewart C.L., Ferguson-Smith A.C. (2015) A trans-homologue interaction between reciprocally imprinted miR-127 and Rtl1 regulates placenta development. Development. 142, 2425–2430.
Sekita Y., Wagatsuma H., Nakamura K., Ono R., Kaga-mi M., Wakisaka N., Hino T., Suzuki-Migishima R., Kohda T., Ogura A., Ogata T., Yokoyama M., Kaneko-Ishino T., Ishino F. (2008) Role of retrotransposon-derived imprinted gene, Rtl1, in the feto-maternal interface of mouse placenta. Nat. Genet. 40, 243–248.
Atambayeva Sh., Niyazova R., Ivashchenko A., Pyrkova A., Pinsky I., Akimniyazova A., Labeit S. (2017) The binding sites of miR-619-5p in the mRNAs of human and orthologous genes. BMC Genomics.18, 428.
Zeng Y., Yi R., Cullen B.R. (2003) MicroRNAs and small interfering RNAs can inhibit mRNA expression by similar mechanisms. Proc. Natl. Acad. Sci. USA. 100, 9779–9784.
Davis E., Caiment F., Tordoir X., Cavaillé J., Ferguson-Smith A., Cockett N., Georges M., Charlier C. (2005) RNAi-mediated allelic trans-interaction at the imprinted Rtl1/Peg11 locus. Curr. Biol. 15, 743–749.
Belot M.P., Nadéri K., Mille C., Boëlle P.Y., Benachi A., Bougnères P., Fradin D. (2017) Role of DNA methylation at the placental RTL1 gene locus in type 1 diabetes. Pediatr. Diabetes. 18, 178–187.
National Center for Biotechnology Information. http://www.ncbi.nlm.nih.gov
Ivashchenko A.T., Pyrkova A.Y., Niyazova R.Y., Alybayeva A., Baskakov K. (2016) Prediction of miRNA binding sites in mRNA. Bioinformation. 12, 237–240.
Kool E.T. (2001) Hydrogen bonding, base stacking, and steric effects in DNA replication. Annu. Rev. Biophys. Biomol. Structure. 30, 1–22.
Leontis N.B., Stombaugh J., Westhof E. (2002) The non-Watson–Crick base pairs and their associated isostericity matrices. Nucl. Acids Res. 30, 3497–3531.
Wang J., Li Z., Liu B., Chen G., Shao N., Ying X., Wang Y. (2016) Systematic study of cis-antisense miRNAs in animal species reveals miR-3661 to target PPP2CA in human cells. RNA. 22, 87–95.
Li S.C., Chan W.C., Hu L.Y., Lai C.H., Hsu C.N., Lin W.C. (2010) Identification of homologous microRNAs in 56 animal genomes. Genomics. 96, 1–9.
Shi N., Deng L., Chen W., Zhang X., Luo R., Jin T., Ma Y., Du C., Lin Z., Jiang K., Guo J., Yang X., Xia Q. (2017) Is microRNA-127 a novel biomarker for acute pancreatitis with lung injury? Dis. Markers. 2017, 1204295.
Shi L., Wang Y., Lu Z., Zhang H., Zhuang N., Wang B., Song Z., Chen G., Huang C., Xu D., Zhang Y., Zhang W., Gao Y. (2017) MiR-127 promotes EMT and stem-like traits in lung cancer through a feed-forward regulatory loop. Oncogene. 36, 1631–1643.
Ying H., Kang Y., Zhang H., Zhao D., Xia J., Lu Z., Wang H., Xu F., Shi L. (2015) MiR-127 modulates macrophage polarization and promotes lung inflammation and injury by activating the JNK pathway. J. Immunol. 194, 1239–1251.
Huang C., Xiao X., Chintagari N.R., Breshears M., Wang Y., Liu L. (2014) MicroRNA and mRNA expression profiling in rat acute respiratory distress syndrome. BMC Med. Genomics. 7, 46.
Ito M., Sferruzzi-Perri A.N., Edwards C.A., Adalsteinsson B.T., Allen S.E., Loo T.H., Kitazawa M., Kaneko-Ishino T., Ishino F., Stewart C.L., Ferguson-Smith A.C. (2015) A trans-homologue interaction between reciprocally imprinted miR-127 and Rtl1 regulates placenta development. Development. 142, 2425–2430.
Bhaskaran M., Wang Y., Zhang H., Weng T., Baviskar P., Guo Y., Gou D., Liu L. (2009) MicroRNA-127 modulates fetal lung development. Physiol. Genomics. 37, 268–278.
Xie T., Liang J., Liu N., Wang Q., Li Y., Noble P.W., Jiang D. (2012) MicroRNA-127 inhibits lung inflammation by targeting IgG Fcγ receptor I. J. Immunol. 188, 2437–2444.
Ivashchenko A., Berillo O., Pyrkova A., Niyazova R., Atambayeva S. (2014) The properties of binding sites of miR-619-5p, miR-5095, miR-5096, and miR-5585-3p in the mRNAs of human genes. BioMed. Res. Int. 2014, 1–8.
Girardot M., Cavaillé J., Feil R. (2012) Small regulatory RNAs controlled by genomic imprinting and their contribution to human disease. Epigenetics. 7, 1341–1348.
Fan G., Ye D., Zhu S., et al. (2017) RTL1 promotes melanoma proliferation by regulating Wnt/β-catenin signalling. Oncotarget. 8, 106026–106037.
Bari A., Orazova S., Ivashchenko A. (2013) miR156- and miR171-binding sites in the protein-coding sequences of several plant genes. Biomed. Res. Int. 2013, 307145.
Lo P.K. (2017) The controversial role of forkhead box F2 (FOXF2) transcription factor in breast cancer. PRAS Open. 1, 9.
Lo P.K., Lee J.S., Liang X., Sukumar S. (2016) The dual role of FOXF2 in regulation of DNA replication and the epithelial-mesenchymal transition in breast cancer progression. Cell Signal. 28, 1502–1519.
Wang Q.S., Kong P.Z., Li X.Q., Yang F., Feng Y.M. (2015) FOXF2 deficiency promotes epithelial-mesenchymal transition and metastasis of basal-like breast cancer. Breast Cancer Res. 17, 30.
Kong P.Z., Yang F., Li L., Li X.Q., Feng Y.M. (2013) Decreased FOXF2 mRNA expression indicates early-onset metastasis and poor prognosis for breast cancer patients with histological grade II tumor. PLoS One. 8, e61591.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология