Молекулярная биология, 2020, T. 54, № 1, стр. 95-102
Модифицированный лентивирусный репортер для магнитной сепарации раковых стволовых клеток
А. Е. Иванова a, Д. С. Кравченко a, С. П. Чумаков a, b, *
a Институт биоорганической химии имени академиков М.М. Шемякина и Ю.А. Овчинникова
Российской академии наук
117997 Москва, Россия
b Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
* E-mail: hathkul@gmail.com
Поступила в редакцию 27.02.2019
После доработки 27.02.2019
Принята к публикации 13.03.2019
Аннотация
Раковые стволовые клетки составляют популяцию наиболее злокачественных клеток опухоли, обладающую повышенным опухоль-инициирующим потенциалом и устойчивостью к химиотерапии. Подобные свойства раковых стволовых клеток делают их перспективными мишенями для разработки направленной терапии, особенно востребованной в контексте высокоагрессивных опухолей. Вместе с тем, проблемой остается точная идентификация раковых клеток, обладающих стволовыми свойствами. Разработанный недавно на основе лентивируса репортер SORE6 позволяет идентифицировать такие клетки по экспрессии генов эмбриональных стволовых факторов SOX2 и OCT4. Репортер SORE6 модифицирован нами для удобства селекции популяции клеток SOX2+/OCT4+ методом иммуномагнитной сепарации и использован для трансдукции клеточных линий HCC1806 и MDA-MB-453 трижды негативного рака молочной железы. Для валидации работы репортера отобраны клетки SOX2+/OCT4+ и проанализировано содержание в них NANOG ‒ ключевого транскрипционного фактора плюропотентности, экспрессия которого регулируется SOX2/OCT4. Определено содержание популяции SOX2+/OCT4+ в каждой линии. Повышенный уровень белка NANOG в отобранных клетках SOX2+/OCT4+ указывает на возможность использования модифицированного репортера для дальнейшего изучения ракового стволового компартмента.
ВВЕДЕНИЕ
Опухоль представляет собой гетерогенную клеточную популяцию, в которой выделяют более дифференцированные клетки, формирующие основную массу, и некоторое количество клеток, обладающих свойствами стволовых [1]. Раковые стволовые клетки (РСК) имеют более высокий опухоль-инициирующий потенциал и большую устойчивость к химиотерапевтическим препаратам, чем остальные клетки опухоли [2‒4]. Попадание РСК в кровеносную систему обуславливает возможность формирования новых опухолевых очагов – метастазирования [5‒7], поэтому сохранение небольшого количества РСК в организме пациента после эрадикации основной опухолевой массы может быть связано с последующим рецидивом онкологического заболевания. Изучение характеристик РСК и расшифровка регуляторных механизмов, лежащих в основе злокачественного фенотипа, вызывает большой интерес [8, 9]. Подобные исследования особенно необходимы в случае высокоагрессивных онкопатологий с низкой общей выживаемостью пациентов. К таким заболеваниям относится трижды негативный рак молочной железы (ТНРМЖ), который, в отличие от других подтипов рака молочной железы (РМЖ), характеризуется неблагоприятным прогнозом и отсутствием эффективной направленной терапии [10, 11]. Предполагаемое активное участие РСК в агрессивном течении ТНРМЖ и низкой выживаемости пациентов подкреплено рядом наблюдений [12‒14]. Изучение вклада РСК при ТНРМЖ затруднено тем, что традиционные методы идентификации РСК основаны на выделении этих клеток по косвенным признакам [15]. В 2003 году Al-Hajj и соавт. впервые идентифицировали в резекционном материале РМЖ популяцию клеток, которые имели фенотип CD44+/CD24−/low и обладали выраженными опухоль-инициирующими свойствами [16]. Трансплантация всего нескольких сотен таких клеток мышам с иммунодефицитом вызывала развитие опухолей с восстановлением гетерогенности, присущей исходной материнской опухоли. В то же время, введение животным нескольких десятков тысяч опухолевых клеток, не подвергшихся фракционированию, не приводило к формированию новообразований. Чуть позже было установлено, что раковые клетки с повышенной экспрессией гена внутриклеточной альдегид-дегидрогеназы 1 (ALDH1) также обладают способностью к инициации опухолей [17]. В настоящее время повышенный уровень ALDH1 и фенотип CD44+/CD24−/low активно используют в качестве признаков, по которым выделяют популяции РСК при РМЖ. Однако пока нет достаточно убедительных данных о предпочтительности одного из этих методов детекции. При этом по результатам ряда исследований пересечение субпопуляций CD44+/CD24–/low и ALDH1+ оказывается довольно низким, что диктует необходимость поиска более точных способов селекции РСК [18‒20]. Кроме того, оба метода позволяют получить лишь обогащенную фракцию РСК, но не чистую популяцию, что также влияет на качество и воспроизводимость результатов исследований [21].
В 2015 году была разработана лентивирусная репортерная система SORE6, позволяющая идентифицировать клетки, в которых активны OCT4 и SOX2 – основные факторы транскрипции, характерные для эмбриональных стволовых клеток [15]. В данной системе шесть повторов элемента, чувствительного к SOX2/OCT4 (SORE6), клонированного из промоторной области NANOG человека, соединены с минимальным цитомегаловирусным промотором (minCMV), контролирующим экспрессию репортерных генов. Последовательность, кодирующая дестабилизированный флуоресцентный белок mCherry (dsmCherry), экспрессируется под контролем OCT4- и SOX2-зависимого промотора, что позволяет визуализировать непосредственно каждую клетку SOX2+/OCT4+ и наблюдать динамику изменений во времени. С помощью этого метода удалось выделить фракцию клеток SOX2+/OCT4+ из нескольких линий гормончувствительного РМЖ и первичных культур, полученных из образцов ткани РМЖ. Высокий потенциал образования опухолей и устойчивость к химиотерапевтическим препаратам показан в экспериментах in vitro и in vivo [15]. На сегодняшний день нет данных об использовании репортерной системы SORE6 для выделения и характеристики РСК в линиях ТНРМЖ, что, по-видимому, обусловлено ее относительной новизной.
В предварительном исследовании мы сравнили содержание клеток SORE6+ и ALDH1 в линиях ТНРМЖ и обнаружили малую перекрываемость количественных значений этих популяций [22]. В данной работе мы модифицировали репортер для удобства селекции РСК на магнитных микросферах и охарактеризовали отобранные клетки по содержанию NANOG, ключевого фактора транскрипции эмбриональных стволовых клеток, экспрессия которого регулируется активностью OCT4 и SOX2.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Клеточные линии. В работе использованы клеточные линии Американской коллекции типовых культур (ATCC®) HEK293T и линии ТНРМЖ HCC1806 и MDA-MB-453. Клетки культивировали при 37°С и 5% CO2 в среде RPMI1640 или DMEM/F12 с добавлением 10% фетальной сыворотки крупного рогатого скота, глутамина и антибиотиков пенициллина и стрептомицина.
Сборка и клонирование репортера SOX2/OCT4. Последовательность SORE6, слитую с minCMV, и PEST-сигнал для дестабилизации mCherry синтезировали de novo с использованием последовательностей, приведенных в [15]. Олигонуклеотиды, использованные для сборки фрагментов SORE6 и dsmCherry, приведены в табл. 1. Конструкцию FLAG-hinge-CD28TМ амплифицировали с плазмиды pLEF1C@CD20-119R, а mCherry – с плазмиды pLCMV-mCherry (табл. 1). Нуклеотидные последовательности праймеров SORE6 CD8 OE rev, FLAG Myc OE dir и CD28 T2A rev кодировали дополнительно сигнал секреции гликопротеина CD8 (CD8s), эпитоп с-Myc и регуляторный пептид Т2А соответственно. Амплифицированные фрагменты разделяли в 1%-ном агарозном геле, вырезали и очищали на колонках Евроген по протоколу CleanUp mini для выделения из гелей. Далее для формирования единой конструкции SORE6-minCMV-MycFLAG-hinge-dsmCherry проводили перекрывающуюся ПЦР. Эту конструкцию клонировали в новую, предварительно расщепленную лентивирусную плазмиду pLSF-RFP, и нарабатывали с использованием набора QIAGEN Plasmid Midi Kit (“QIAGEN”, ФРГ). Последовательность вставки верифицировали секвенированием по Сэнгеру. Олигонуклеотиды, использованные для сборки SORE6 и PEST, подбирали с помощью программы DNAWorks (https://hpcwebapps.cit. nih.gov/dnaworks). Для подбора праймеров и разработки схемы клонирования фрагментов в единую конструкцию использовали программу SnapGene.
Таблица 1.
Нуклеотидные последовательности использованных праймеров
| ПЦР-продукт | Праймер, номер и название | Праймер, нуклеотидная последовательность |
|---|---|---|
| SORE6 | SORE6-1 | GCCAAATTACAAAATTCAAAATTTTATCGATCAGCTACTTTTGCATTACAATG |
| SORE6-2 | AGGCCATTGTAATGCAAAAGTAGCTGCACCAAGGCCATTGTAATGCAAAAGTAGCTGATC | |
| SORE6-3 | ACTTTTGCATTACAATGGCCTTGGTGCAGCTACTTTTGCATTACAATGGCCTTGGTGGAA | |
| SORE6-4 | AGCTGCACCAAGGCCATTGTAATGCAAAAGTAGCTGGAATTCCACCAAGGCCATTGTAAT | |
| SORE6-5 | ATGGCCTTGGTGCAGCTACTTTTGCATTACAATGGCCTTGGTGCAGCTACTTTTGCATTA | |
| SORE6-6 | GTTGGCCCGTACACGCCTAACTAGTCACCAAGGCCATTGTAATGCAAAAGTAGCTGCACC | |
| SORE6-7 | GCGTGTACGGGCCAACTTTTGTATACAAAGTGGTAGGCGTGTACGGTGGGAGGCCTATAT | |
| SORE6-8 | TCTCCAGGCGATCTGACGGTTCACTAAACGAGCTCTGCTTATATAGGCCTCCCACCGTAC | |
| SORE6-9 | CGTCAGATCGCCTGGAGACGCCATCCACGCTGTTTTGACCTCCATAGAAGACACCGGGAC | |
| SORE6-10 | GCTGGATCGGTCCCGGTGTCTTCTATGGA | |
| SORE6 Cla dir | GCCAAATTACAAAATTCAAAATTTTATCGATC | |
| SORE6 CD8 OE rev | GCGGCGTGGAGCAGCAAGGCCAGCGGCAGGAGCAAGGCGGTCACTGG-TAAGGCCATGGTGGCGCTGGATCGGTCCCGG | |
| PEST | PEST-1 | CATGGACGAGCTGTACAAGCT |
| PEST-2 | CATCATCCTGCGCCGCCACCGCCGGCGGGAAGCCATGGCTAAGCTTGTACAGCTCGTCCA | |
| PEST-3 | GCGGCGCAGGATGATGGCACGCTGCCCATGTCTTGTGCCCAGGAGAGCGGGATGGACCGT | |
| PEST-4 | ACACATTGATCCTAGCAGAAGCACAGGCTGCAGGGTGACGGTCCATCCCGCTC | |
| PEST Nhe rev | AGAGAGGCTAGCTCACACATTGATCCTAGCAGAAGC | |
| mCherry | mCherry T2A dir | GTCTTCTAACATGCGGTGACGTGGAGGAGAATCCCGGCCCTATGGTGAGCAAGGGCG |
| mCherry rev | CGGCATGGACGAGCTGTACAAG | |
| Hinge | FLAG Myc OE dir | CTTGCTGCTCCACGCCGCCAGGCCGGAACAAAAACTCATCTCAGAAGAGGATCTGGGATCCGATTACAAGGATGATGATG |
| CD28 T2A rev | GTCACCGCATGTTAGAAGACTTCCTCTGCCCTCTCCGCTTCCGTCGACCACCCAGAAAATAATAAAGGCC |
Сборка лентивирусных частиц. Сборку лентивируса проводили в культуре клеток HEK293T. В день перед трансфекцией клетки HEK293T рассевали в шестилуночные планшеты в плотности 106 клеток на лунку. Непосредственно перед трансфекцией клетки переводили в среду Opti-MEM (“Gibco”, США) без добавок. В качестве упаковочной плазмиды и плазмиды, кодирующей белки оболочки вируса везикулярного стоматита, использовали psPAX2, безвозмездно предоставленную Didier Trono (Addgene plasmid #12260; http://n2t.net/addgene:12260; RRID:Addgene_12260), и pMD2.G, также безвозмездно предоставленную Didier Trono (Addgene plasmid #12259; http://n2t.net/addgene:12259; RRID:Addgene_12259). Вспомогательные плазмиды и модифицированный вектор SORE6 смешивали в эквимолярном соотношении в среде Opti-MEM так, чтобы на лунку шестилуночного планшета приходилось 3 мкг ДНК. Затем к смеси добавляли раствор полиэтиленимина (PEI 25кДа, “Polysciences”, США) 1 мг/мл, из расчета 3 мкг PEI на 1 мкг ДНК, инкубировали в течение 20 мин и раскапывали на клетки. Клетки инкубировали с плазмидами в течение 3‒4 ч, затем среду меняли на полную DMEM/F12 c 10%-ной сывороткой и антибиотиком. Через 48 ч собирали обогащенную вирусом среду, которую использовали для дальнейшего заражения.
Трансдукция линий ТНРМЖ. Клеточные линии HCC1806 и MDA-MB-453 рассаживали в шестилуночные планшеты с плотностью 50%, вносили среду с лентивирусом и 10 мкг/мл Polybrene (“Sigma”, США). Среду с вирусом перед внесением очищали от дебриса при помощи шприца с одноразовым фильтром с диаметром пор 0.45 мкм (“TPP”, Швейцария). Клетки инкубировали с вирусом в течение 3 ч, затем среду меняли на полную RPMI1640 или DMEM/F12. Для контроля сборки лентивируса и определения чувствительности клеток к трансдукции, каждую клеточную линию инфицировали лентивирусом, кодирующим красный флуоресцентный белок. Результат трансдукции оценивали с использованием флуоресцентного микроскопа ZOE (“Bio-Rad”, США). Количество (%) клеток SORE6+ определяли путем подсчета mCherry-флуоресцентных клеток относительно общего количества клеток в поле с помощью программы ImageJ (Wayne Rasband). Анализировали по три поля для каждой линии ТНРМЖ.
Отбор популяции SOX2+/OCT4+ методом магнитной сепарации. Клетки SOX2+/OCT4+ отбирали из трансдуцированных линий ТНРМЖ методом позитивной селекции на магнитных частицах. Селекцию проводили с использованием набора EasySep™ Human “Do-It-Yourself” Selection Kit (“STEMCELL™ Technologies”, Канада), предназначенного для отбора клеток человека с помощью моноклональных антител мыши против белка-маркера. Поскольку клетки SOX2+/OCT4+ экспонировали на своей поверхности пептид FLAG (DYKDDDDK), для обогащения использовали моноклональные антитела против FLAG (“ProteinTech”, 66008-2-Ig, США). Селекцию осуществляли в соответствии с протоколом производителя набора.
Вестерн-блотинг. Клетки, отобранные на магнитных микросферах, лизировали в буфере RIPA (150 мМ NaCl, 1.0% NP-40, 0.1% SDS, 50 мМ Tрис, pH 8.0). Концентрацию белка измеряли при помощи реактива Брэдфорда (“Bio-Rad”, США). Белки разделяли с помощью электрофореза в 10%-ном полиакриламидном геле и переносили на нитроцеллюлозную мембрану. Мембрану блокировали в течение 1 ч в 5%-ном обезжиренном молоке, а затем инкубировали в течение ночи с первичными антителами против NANOG (“PeproTech”, 500-P236, США), разведенными до конечной концентрации 0.1 мкг/мл. На следующий день мембрану отмывали 3 раза от несвязавшихся антител в фосфатно-солевом буфере с добавлением Tween-20 (0.1%) и инкубировали в течение 1 ч со вторичными антителами в разведении 1 : 3000 (“Santa-Cruz”, США). Мембрану проявляли при помощи набора FemtoLUCENT PLUS-HRP (“G-Bioscienses”). С целью контроля нагрузки блот окрашивали антителами к β-актину (AC-15, “Santa-Cruz”).
Анализ результатов. Результаты исследования обрабатывали с использованием программы GraphPad Prism 5 для Windows.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Строение модифицированного лентивирусного репортера SORE6
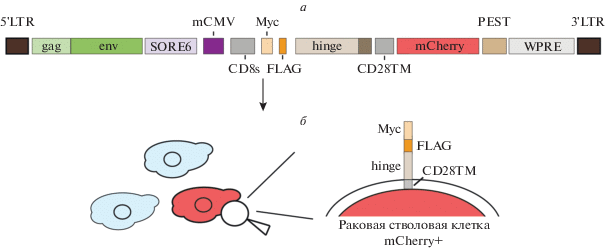
С применением модифицированного лентивирусного репортера SORE6 нами проведена селекция раковых клеток, обладающих стволовым фенотипом, в линиях ТНРМЖ. Для получения модифицированной репортерной конструкции был использован оригинальный элемент SORE6, содержащий шесть повторов чувствительного к SOX2/OCT4 элемента SORE, клонированного с промотора NANOG человека, а также последовательность, кодирующая дестабилизированный флуоресцентный белок mCherry (dsmCherry) [15]. Для удобства селекции клеток на магнитных частицах в конструкцию, содержащую элемент SORE6 и dsmCherry, дополнительно были введены последовательности, кодирующие пептид FLAG (DYKDDDDK), эпитоп с-Myc (EQKLISEEDL), сигнал секреции гликопротеина CD8 (CD8s), трансмембранный домен гликопротеина CD28 (CD28 TM) и шарнирную последовательность (hinge), представляющую собой фрагмент CH2-CH3 иммуноглобулина IgG4 (рис. 1а). Наличие в клетке стволовых факторов SOX2/OCT4 ведет к активации SORE6 и транскрипции последовательностей под контролем minCMV. CD8s-MycFLAG-hinge-CD28 TM транслируется в виде единого продукта. За счет лидерного сигнала CD8s, который впоследствии отщепляется, полипептид направляется на секрецию к мембране клетки. Встраивание трансмембранного домена CD28 в фосфолипидный бислой обеспечивает заякоривание Myc-FLAG-hinge на поверхности мембраны (рис. 1б). За счет регуляторного элемента Т2А mCherry транслируется в виде отдельного белка. При этом не происходит критической для жизнедеятельности РСК аккумуляции флуоресцентного белка, поскольку сигнал PEST направляет mCherry на путь протеасомной деградации.
Рис. 1.
а – Схема модифицированной лентивирусной конструкции, созданной по итогам клонирования. б – Схематическое расположение белковых продуктов, полученных в результате транскрипции-трансляции лентивирусной репортерной конструкции, активной в раковой стволовой клетке, в области клеточной мембраны. Накопление и деградация белка mCherry позволяет визуализировать в общей популяции клетку, в которой экспрессируются гены стволовых факторов SOX2/OCT4. Заякоривание пептидной последовательности Myc-FLAG-hinge на поверхности мембраны такой клетки за счет трансмембранного домена CD28 позволяет отобрать ее методом магнитной сепарации. LTR (Long Terminal Repeat) – длинный концевой повтор, gag (group antigens) – группоспецифические антигены, env (envelope) – белок оболочки, WPRE (Woodchuck Hepatitis Virus Posttranscriptional Regulatory Element) – посттранскрипционный регуляторный элемент вируса гепатита североамериканского лесного сурка.
Линии MDA-MB-453 и HCC1806 содержат популяции клеток, которые экспрессируют SOX2/OCT4 и характеризуются повышенным уровнем NANOG
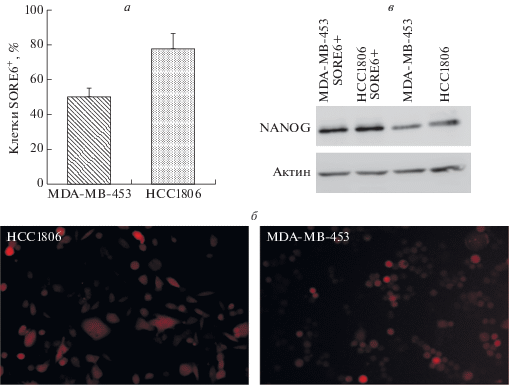
Содержание популяций SORE6+ (%) в линиях ТНРМЖ вычисляли как отношение числа флуоресцентных клеток к общему количеству клеток в поле на микрофотографиях, полученных с помощью флуоресцентного микроскопа. Анализировали по три поля (микрофотографии) каждой линии клеток. В линии MDA-MB-453 содержание клеток SOX2+/OCT4+ составило около 48% от общей популяции, в то время как в линии HCC1806 превалировала фракция SOX2+/OCT4+, которая составила около 78% от общего числа клеток в культуре (рис. 2а, б). Методом вестерн-блотинга показано, что уровень эмбрионального транскрипционного фактора NANOG в селектированных клетках выше, чем в общей популяции (рис. 2в).
Рис. 2.
а – Содержание клеток, экспрессирующих SOX2/OCT4, в линиях MDA-MB-453 и HCC1806, трансдуцированных модифицированным SORE6-репортером. б – Микрофотографии трансдуцированных SORE6-репортером линий HCC1806 и MDA-MB-453, полученные с использованием флуоресцентного микроскопа ZOE (“Bio-Rad”). в – В SORE6+-клетках, отобранных с использованием репортера, уровень NANOG повышен по сравнению с неселектированной популяцией.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Концепция существования в опухоли особых клеток, обладающих свойствами стволовых, имеет фундаментальное значение для понимания процессов канцерогенеза, метастазирования и устойчивости к химиотерапии [7, 23]. В последние годы появляется все больше исследований, направленных на поиск и характеристику РСК как в установленных опухолевых клеточных линиях, так и в резекционном материале, полученном от пациентов с различными онкопатологиями [10, 24‒26]. Раковые клетки, обладающие свойствами стволовых, идентифицируют по различным косвенным признакам, например, по экспрессии особых поверхностных антигенов или некоторых внутриклеточных ферментов [6, 13, 18‒20, 24]. Подобные методы не позволяют получить чистую популяцию клеток с повышенным опухоль-инициирующим потенциалом, что ставит под сомнение их точность и может влиять на воспроизводимость результатов исследований. Для идентификации РСК мы использовали сравнительно недавно разработанную лентивирусную конструкцию SORE6, которая позволяет выявлять клетки со стволовыми свойствами по прямым признакам стволовости – активности факторов транскрипции OCT4 и SOX2 [15]. Для удобства обогащения фракции РСК методом иммуномагнитной сепарации репортерную конструкцию модифицировали таким образом, чтобы клетки SOX2+/OCT4+ с введенным вектором дополнительно экспонировали на своей поверхности пептид FLAG. Модифицированную репортерную конструкцию испытывали на двух линиях ТНРМЖ ‒ HCC1806 и MDA-MB-453. ТНРМЖ характеризуется агрессивностью течения и повышенным метастазированием, что может быть обусловлено наличием выраженной фракции РСК [5, 10, 11, 14, 27, 28]. С использованием модифицированного репортера в обеих линиях удалось визуализировать и отобрать фракцию клеток, экспрессирующих SOX2/OCT4 и экспонирующих на поверхности пептид FLAG. Содержание клеток, обладающих фенотипом SOX2+/OCT4+, в обеих линиях оказалось весьма высоким и составило около 78% в линии HCC1806 и около 48% – в MDA-MB-453. Подобные значения согласуются с современной тенденцией в области исследования РСК, в рамках которой представления о РСК, как о минорной группе, подвергаются переосмыслению. Согласно последним данным, РСК в общей популяции могут быть представлены в изобилии и активно пролиферировать [29]. Вместе с тем установлено, что введение клеток линии MDA-MB-453 иммуносупрессивным мышам не приводит к формированию опухолей [30]. Таким образом, высокое содержание популяции SOX2+/OCT4+ в MDA-MB-453 не ассоциировано с туморогенностью этой клеточной линии. Подобное обстоятельство может объясняться как необходимостью вовлечения иных механизмов и факторов в формирование новых опухолевых очагов, так и приобретенными в результате культивирования специфическими особенностями данной клеточной линии [30]. Линия клеток HCC1806 является туморогенной и используется для получения ксенотрансплантатов in vivo, в связи с чем высокое содержание популяции SOX2+/OCT4+ не вступает в противоречие со свойствами клеточной линии [31].
Транскрипционный фактор NANOG необходим для поддержания плюропотентности эмбриональных стволовых клеток, его ген обычно супрессирован в большинстве тканей взрослого организма. Однако во многих типах раковых клеток, в том числе РМЖ, NANOG гиперэкспрессируется, что приводит к дерегуляции экспрессии многих генов, ассоциированных с онкогенезом и метастазированием [32]. Анализ отобранных клеток SORE6+ показал повышение уровня NANOG, экспрессия гена которого регулируется SOX2/OCT4 [33]. В то же время, достаточно высокий уровень NANOG в общей массе клеток в каждой из двух проанализированных клеточных линий согласовывался с высоким содержанием РСК в популяции.
Таким образом, использование модифицированного репортера на SOX2/OCT4 позволяет идентифицировать и отбирать клетки с эмбриональным стволовым фенотипом. Данная модифицированная репортерная конструкция может служить удобным инструментом изучения подобных клеточных популяций.
Исследование выполнено при финансовой поддержке Российского фонда фундаментальных исследований (грант 18-34-00981/18).
Все процедуры, выполненные в данной работе, соответствуют этическим стандартам институционального комитета по исследовательской этике и Хельсинкской декларации 1964 года и ее последующим изменениям или сопоставимым нормам этики.
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Shackleton M., Quintana E., Fearon E.R., Morrison S.J. (2009) Heterogeneity in cancer: cancer stem cells versus clonal evolution. Cell. 138, 822–829.
Li X., Lewis M.T., Huang J., Gutierrez C., Osborne C.K., Wu M.F., Hilsenbeck S.G., Pavlick A., Zhang X., Chamness G.C., Wong H., Rosen J., Chang J.C. (2008) Intrinsic resistance of tumorigenic breast cancer cells to chemotherapy. J. Natl. Cancer Inst. 100, 672–679.
Meacham C.E., Morrison S.J. (2013) Tumour heterogeneity and cancer cell plasticity. Nature. 501, 328–337.
Chuthapisith S., Eremin J., El-Sheemey M., Eremin O. (2010) Breast cancer chemoresistance: emerging importance of cancer stem cells. Surg. Oncol. 19, 27–32.
Charafe-Jauffret E., Ginestier C., Iovino F., Wicinski J., Cervera N., Finetti P., Hur M.H., Diebel M.E., Monville F., Dutcher J., Brown M., Viens P., Xerri L., Bertucci F., Stassi G., Dontu G., Birnbaum D., Wicha M.S. (2009) Breast cancer cell lines contain functional cancer stem cells with metastatic capacity and a distinct molecular signature. Cancer Res. 69, 1302–1313.
Zhong Y., Shen S., Zhou Y., Mao F., Guan J., Lin Y., Xu Y., Sun Q. (2014) ALDH1 is a better clinical indicator for relapse of invasive ductal breast cancer than the CD44+/CD24-phenotype. Med. Oncol. 31, 864.
Marcato P., Dean C.A., Pan D., Araslanova R., Gillis M., Joshi M., Helyer L., Pan L., Leidal A., Gujar S., Giacomantonio C.A., Lee P.W. (2011) Aldehyde dehydrogenase activity of breast cancer stem cells is primarily due to isoform ALDH1A3 and its expression is predictive of metastasis. Stem Cells. 29, 32–45.
Easwaran H., Tsai H.C., Baylin S.B. (2014) Cancer epigenetics: tumor heterogeneity, plasticity of stem-like states, and drug resistance. Mol. Cell. 54, 716–727.
Thiagarajan P.S., Hitomi M., Hale J.S., Alvarado A.G., Otvos B., Sinyuk M., Stoltz K., Wiechert A., Mulkearns-Hubert E., Jarrar A., Zheng Q., Thomas D., Egelhoff T., Rich J.N., Liu H., Lathia J.D., Reizes O. (2015) Development of a fluorescent reporter system to delineate cancer stem cells in triple-negative breast cancer. Stem Cells. 33, 2114–2125.
Collina F., Di Bonito M., Li Bergolis V., De Laurentiis M., Vitagliano C., Cerrone M., Nuzzo F., Cantile M., Botti G. (2015) Prognostic value of cancer stem cells markers in triple-negative breast cancer. Biomed. Res. Int. 2015, 158682.
Ahn S.G., Kim S.J., Kim C., Jeong J. (2016) Molecular classification of triple-negative breast cancer. J. Breast Cancer. 19, 223–230.
Minn A.J., Kang Y., Serganova I., Gupta G.P., Giri D.D., Doubrovin M., Ponomarev V., Gerald W.L., Blasberg R., Massague J. (2005) Distinct organ-specific metastatic potential of individual breast cancer cells and primary tumors. J. Clin. Invest. 115, 44–55.
Charafe-Jauffret E., Ginestier C., Iovino F., Tarpin C., Diebel M., Esterni B., Houvenaeghel G., Extra J.M., Bertucci F., Jacquemier J., Xerri L., Dontu G., Stassi G., Xiao Y., Barsky S.H., Birnbaum D., Viens P., Wicha M.S. (2010) Aldehyde dehydrogenase 1-positive cancer stem cells mediate metastasis and poor clinical outcome in inflammatory breast cancer. Clin. Cancer Res. 16, 45–55.
Velasco-Velazquez M.A., Popov V.M., Lisanti M.P., Pestell R.G. (2011) The role of breast cancer stem cells in metastasis and therapeutic implications. Am. J. Pathol. 179, 2–11.
Tang B., Raviv A., Esposito D., Flanders K.C., Daniel C., Nghiem B.T., Garfield S., Lim L., Mannan P., Robles A.I., Smith W.I., Jr., Zimmerberg J., Ravin R., Wakefield L.M. (2015) A flexible reporter system for direct observation and isolation of cancer stem cells. Stem Cell Repts. 4, 155–169.
Al-Hajj M., Wicha M.S., Benito-Hernandez A., Morrison S.J., Clarke M.F. (2003) Prospective identification of tumorigenic breast cancer cells. Proc. Natl. Acad. Sci. USA. 100, 3983–3988.
Ginestier C., Hur M.H., Charafe-Jauffret E., Monville F., Dutcher J., Brown M., Jacquemier J., Viens P., Kle-er C.G., Liu S., Schott A., Hayes D., Birnbaum D., Wicha M.S., Dontu G. (2007) ALDH1 is a marker of normal and malignant human mammary stem cells and a predictor of poor clinical outcome. Cell Stem Cell. 1, 555–567.
Ricardo S., Vieira A.F., Gerhard R., Leitao D., Pinto R., Cameselle-Teijeiro J.F., Milanezi F., Schmitt F., Paredes J. (2011) Breast cancer stem cell markers CD44, CD24 and ALDH1: expression distribution within intrinsic molecular subtype. J. Clin. Pathol. 64, 937–946.
Liu Y., Nenutil R., Appleyard M.V., Murray K., Boy-lan M., Thompson A.M., Coates P.J. (2014) Lack of correlation of stem cell markers in breast cancer stem cells. Br. J. Cancer. 110, 2063–2071.
Horimoto Y., Arakawa A., Sasahara N., Tanabe M., Sai S., Himuro T., Saito M. (2016) Combination of cancer stem cell markers CD44 and CD24 is superior to ALDH1 as a prognostic indicator in breast cancer patients with distant metastases. PLoS One. 11, e0165253.
Sajithlal G.B., Rothermund K., Zhang F., Dabbs D.J., Latimer J.J., Grant S.G., Prochownik E.V. (2010) Permanently blocked stem cells derived from breast cancer cell lines. Stem Cells. 28, 1008–1018.
Ivanova A.E.K.D., Lezhnin Y.N., Chumakov S.P., Frolova E.I. (2017) The content analysis of ALDH + and SORE6 + populations of cells in cellular lines of thriple negative breast cancer. J. Pharm. Sci. Res. 9, 354–358.
Li X., Lewis M.T., Huang J., Gutierrez C., Osborne C.K., Wu M.F., Hilsenbeck S.G., Pavlick A., Zhang X., Chamness G.C., Wong H., Rosen J., Chang J.C. (2008) Intrinsic resistance of tumorigenic breast cancer cells to chemotherapy. J. Natl. Cancer Inst. 100, 672–679.
Shao J., Fan W., Ma B., Wu Y. (2016) Breast cancer stem cells expressing different stem cell markers exhibit distinct biological characteristics. Mol. Med. Rep. 14, 4991–4998.
Thomas M.L., de Antueno R., Coyle K.M., Sultan M., Cruickshank B.M., Giacomantonio M.A., Giacomantonio C.A., Duncan R., Marcato P. (2016) Citral reduces breast tumor growth by inhibiting the cancer stem cell marker ALDH1A3. Mol. Oncol. 10, 1485–1496.
Kaur S., Elkahloun A.G., Singh S.P., Chen Q.R., Meerzaman D.M., Song T., Manu N., Wu W., Mannan P., Garfield S.H., Roberts D.D. (2016) A function-blocking CD47 antibody suppresses stem cell and EGF signaling in triple-negative breast cancer. Oncotarget. 7, 10133–10152.
Ginestier C., Wicha M.S. (2007) Mammary stem cell number as a determinate of breast cancer risk. Breast Cancer Res. 9, 109.
Lehmann B.D., Bauer J.A., Chen X., Sanders M.E., Chakravarthy A.B., Shyr Y., Pietenpol J.A. (2011) Identification of human triple-negative breast cancer subtypes and preclinical models for selection of targeted therapies. J. Clin. Invest. 121, 2750–2767.
Batlle E., Clevers H. (2017) Cancer stem cells revisited. Nat. Med. 23, 1124–1134.
Gruber A.D., Pauli B.U. (1999) Tumorigenicity of human breast cancer is associated with loss of the Ca2+-activated chloride channel CLCA2. Cancer Res. 59, 5488–5491.
Sevignani C., Calin G.A., Cesari R., Sarti M., Ishii H., Yendamuri S., Vecchione A., Trapasso F., Croce C.M. (2003) Restoration of fragile histidine triad (FHIT) expression induces apoptosis and suppresses tumorigenicity in breast cancer cell lines. Cancer Res. 63, 1183–1187.
Lu X., Mazur S.J., Lin T., Appella E., Xu Y. (2014) The pluripotency factor nanog promotes breast cancer tumorigenesis and metastasis. Oncogene. 33, 2655–2664.
Rodda D.J., Chew J.L., Lim L.H., Loh Y.H., Wang B., Ng H.H., Robson P. (2005) Transcriptional regulation of NANOG by OCT4 and SOX2. J. Biol. Chem. 280, 24731–24737.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


