Молекулярная биология, 2020, T. 54, № 1, стр. 78-86
Полиаденилирование транскриптов, считываемых РНК-полимеразой III c SINE, значительно удлиняет время их жизни в клетке
И. Г. Устьянцев a, К. А. Татосян a, Д. В. Стасенко a, Н. Ю. Кочанова a, О. Р. Бородулина a, Д. А. Крамеров a, *
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
* E-mail: kramerov@eimb.ru
Поступила в редакцию 05.03.2019
После доработки 05.03.2019
Принята к публикации 07.03.2019
Аннотация
SINE (Short Interspersed Elements) – мобильные генетические элементы высших эукариот, возникавшие в ходе эволюции из различных тРНК, а также из 5S рРНК и 7SL РНК. Подобно генам этих РНК, SINE транскрибируются РНК-полимеразой III. Транскрипты некоторых SINE млекопитающих обладают уникальной для транскриптов РНК-полимеразы III способностью подвергаться AAUAAA-зависимому полиаденилированию. Известно, что полиаденилирование транскриптов РНК-полимеразы II (мРНК) увеличивает время их жизни в клетке. Цель этой работы – проверить, возрастает ли стабильность транскриптов SINE в клетке в результате AAUAAA-зависимого поли-аденилирования. Клетки HeLa трансфицировали ДНК SINE с сигналом полиаденилирования (AATAAA) или без него; через сутки ингибировали транскрипцию актиномицином D и отслеживали снижение уровней транскриптов SINE с помощью нозерн-гибридизации. Для всех восьми исследованных SINE время полужизни неполиаденилированных транскриптов составляло 20–30 мин, а поли-аденилированных – превышало 3 ч. Интересно, что введение дополнительного фрагмента ДНК длиной 80 нуклеотидов в среднюю часть SINE B2 существенно не снижало стабильности полиаденилированных транскриптов. Скорее всего, увеличение времени жизни полиаденилированных транскриптов SINE связано с тем, что поли(A)-хвост, взаимодействуя с поли(A)-связывающим белком (PABP), защищает РНК от деградации экзонуклеазами, действующими с 3'-конца. Основываясь на полученных результатах, мы предполагаем сконструировать векторы на базе SINE, предназначенные для экспрессии коротких некодирующих РНК, которые благодаря полиаденилированию будут стабильны в клетке.
ВВЕДЕНИЕ
Стабильность РНК в клетке варьирует в широких пределах. Одни РНК очень стабильны – их время жизни составляет десятки часов. К таким относятся рибосомные, транспортные и большинство малых ядерных РНК, а также многие мРНК [1]. Другие РНК распадаются значительно быстрее – иногда за время порядка нескольких минут. Среди таких РНК можно назвать некодирующие РНК, транскрибирующиеся с промоторных или энхансерных областей генов (Cryptic Unstable Transcripts – CUTs, Promoter upstream transcripts – PROMPTs) [2]. Некоторые мРНК также очень нестабильны, например мРНК отдельных онкогенов (c-Fos, c-Jun, c-Myc) и цитокинов (TNF, GM-CSF). Короткое время жизни таких мРНК обусловлено наличием AU-богатых элементов в 3'-нетранслируемых областях (3'-UTR) [3]. Напротив, некоторые другие элементы в составе множества мРНК обусловливают их длительное существование в клетке [4]. Как элементы “стабильности”, так и элементы “ускоренного распада” мРНК функционируют, связываясь со специфическим белками. Часто в регуляции распада мРНК помимо белков участвуют длинные и короткие некодирующие РНК (lnc- и miRNA) [5, 6]. Изменение стабильности РНК относится к важным механизмам, позволяющим клетке регулировать экспрессию многих ключевых генов.
Основным путем распада РНК в клетке является деградация экзонуклеазами с 5'- или с 3'-конца. Нуклеазы Xrn1 и Xrn2 отщепляют нуклеотиды с 5'-конца РНК [7], тогда как нуклеазы Dis3 и PM-Scl 100 (у дрожжей – Rrp44 и Rrp6 соответственно), входящие в состав белкового комплекса, называемого экзосомой, осуществляют деградацию РНК с 3'-конца (Rrp44 также обладает эндонуклеазной активностью) [8]. Матричные РНК и некоторые длинные некодирующие РНК защищены от действия 3'-концевых экзонуклеаз поли(A)-последовательностью длиной до 250 нуклеотидов. Такой “хвост” молекул мРНК синтезируется нематрично канонической поли(А)-полимеразой по завершении формирования 3'-конца транскрипта, генерируемого РНК-полимеразой II [9, 10]. C “хвостом” взаимодействуют поли(A)-связывающие белки (PABPs), которые защищают поли(A) от действия деаденилирующих ферментов (Pan2-Pan3), поэтому его деградация замедляется. По мере укорочения поли(A) до ~110 н. другие деаденилазы (Ccr4-Caf1) начинают действовать активнее, и, в конце концов, такая мРНК становится доступной для действия экзосомы, что приводит к полному разрушению молекулы РНК [11].
У дрожжей хорошо изучена деградация дефектных (с нарушенной вторичной структурой или лишенных некоторых модифицированных оснований) молекул некодирующих РНК. Такие молекулы распознаются комплексом TRAMP, состоящим из трех белков: РНК-связывающего белка Air2p, хеликазы Mtr4p и неканонической поли(А)-полимеразы Trf4p. Этот фермент синтезирует олиго(А)-хвост на 3'-конце РНК, благодаря чему она направляется в экзосому, где претерпевает полную деградацию. Таким образом, в данном случае хвост РНК, состоящий из одних остатков аденозина, не увеличивает время жизни РНК, а напротив, служит сигналом для ее деградации [12–14]. В клетках человека есть два белковых комплекса, схожих с TRAMP, которые направляют РНК в экзосомы, но один из них синтезирует олиго(А)-хвост у РНК, а другой – нет [15].
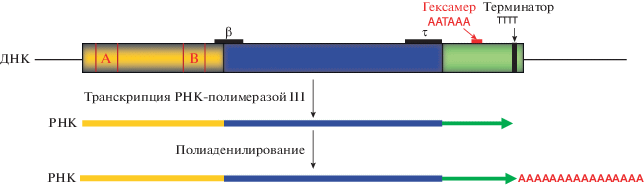
К малоизученным типам некодирующих РНК относятся транскрипты SINE (short interspersed elements). Сотни тысяч копий этих коротких (100–500 п.н.) мобильных генетических элементов рассеяно по геномам млекопитающих и других высших эукариот [16]. Обычно распространение каждого семейства SINE ограничено одним отрядом или несколькими семействами млекопитающих. Большинство семейств SINE произошло от различных видов тРНК. Много копий SINE находится в интронах генов, а также в UTR, поэтому их последовательности оказываются в составе молекул пре-мРНК, синтезируемых РНК-полимеразой II. Однако автономную транскрипцию SINE осуществляет другой фермент – РНК-полимераза III, которая, как известно, транскрибирует гены тРНК, 5S рРНК и ряда других некодирующих РНК. Небольшие РНК, образующиеся в результате такой транскрипции SINE, могут служить матрицами при обратной транскрипции, приводящей к возникновению в геноме новых копий SINE. Транскрипция SINE РНК-полимеразой III инициируется благодаря наличию в их составе внутреннего промотора этой РНК-полимеразы. Промотор состоит из двух участков (боксы А и В), расположенных в головной части SINE на расстоянии 30–40 п.н. друг от друга (рис. 1). Замечено [17], что в 3'-концевой части многих SINE млекопитающих содержится один или несколько гексамеров ААТААА и терминатор транскрипции (ТТТТ) РНК-полимеразы III (рис. 1). Семейства таких SINE были объединены в класс Т+, тогда как семейства SINE, для которых нехарактерно наличие последовательностей ААТААА и ТТТТ, отнесены к классу Т– [17]. Хорошо известно, что гексамер ААТААА в генах, транскрибируемых РНК-полимеразой II, необходим для формирования 3'-конца мРНК, а именно, разрезания транскрипта и синтеза поли(А) на его конце [10]. Несмотря на то, что свойства РНК-полимераз II и III очень различаются, предполагали [17, 18], а затем экспериментально доказали [19, 20], что РНК, образующиеся при транскрипции РНК-полимеразой III SINE класса Т+, полиаденилируются в клетке. Транскрипция таких SINE останавливается на терминаторе, а на 3'-конце образовавшейся РНК синтезируется поли(А) длиной до 250 н. Для такого полиаденилирования необходим гексамер ААТААА, а также два района в SINE, один из которых (β) расположен сразу за промотором (бокс В), тогда как другой (τ) – рядом с хвостовой частью SINE (рис. 1) [20].
Рис. 1.
Схема строения SINE класса T+. Головная часть SINE, происходящая от тРНК, содержит боксы A и B, образующие промотор РНК-полимеразы III. Транскрипция завершается на терминаторе в хвостовой части SINE. Образовавшаяся РНК несет сигнал полиаденилирования AAUAAA, который, вместе с последовательностями в районах бета (β) и тау (τ) обеспечивает синтез поли(A) на 3'-конце транскрипта.
Ранее нами показано [19], что полиаденилированные РНК SINE B2 мыши, синтезированные РНК-полимеразой III, значительно более стабильны в клетке, чем неполиаденилированные транскрипты. Теперь мы исследовали, обладают ли такими же свойствами полиаденилированные транскрипты других SINE. A priori нельзя исключить, что ключевую роль в стабильности таких РНК в клетке играет их первичная или вторичная структура, но альтернативой обеспечения их стабильности может быть строение 3'-конца, в частности наличие поли(A)-хвоста.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Плазмидные конструкции. Использованные в работе плазмидные конструкции, несущие SINE c функциональным или инактивированным мутацией сигналом полиаденилирования, описаны ранее: SINE В2, B2_ins80, Dip, Ves, Can, Ere, Tal, Rhin-1B [19, 20] и Rhin-1A [21]. Rhin-1A и Rhin-1B представляют собой сильно отличающиеся варианты одного SINE из генома большого подковоноса (сравнение их нуклеотидных последовательностей приведено на рис. S1 , см. Приложение на сайте http://www.molecbio.ru/downloads/2019/4/ supp_Ustyantsev_rus.pdf). Плазмида, несущая мышиный ген 4.5SI РНК с 5'-фланкирующей последовательностью длиной 32 нуклеотида, описана ранее [22]. Плазмидные ДНК для трансфекции клеток выделяли с помощью набора NucleoBond PC 100 (“Macherey-Nagel”, Германия).
Культивирование клеток, трансфекция, ингибирование транскрипции и выделение РНК. Клетки HeLa растили на чашках Петри (d = 60 мм) в 5 мл среды DMEM с 10% эмбриональной сыворотки крупного рогатого скота (FBS) и трансфицировали ДНК плазмид с помощью реактива TurboFect, согласно протоколу производителя (“Thermo Fisher Scientific”, США). Для трансфекции клеток на одной чашке использовали 5 мкг плазмиды, несущей SINE, 0.5 мкг контрольной плазмиды с геном 4.5SI РНК и 10 мкл TurboFect. Для ингибирования транскрипции через 20 ч после трансфекции к клеткам добавляли актиномицин D до концентрации 5 мкг/мл. Затем, используя гуанидинтиоцианатный метод [23], выделяли суммарную клеточную РНК через разные временные промежутки (от 0 до 180 мин после добавления актиномицина D). После переосаждения этанолом РНК ресуспендировали в буфере: 10 мМ Трис-HCl (pH 7.5), 2.5 мМ MgCl2, 0.1 мМ CaCl2, – содержащем ДНКазу I (100 мкг/мл), в течение 1 ч при 37°C, с последующей экстракцией хлороформом. Переосажденную этанолом РНК растворяли в 60 мкл воды и измеряли концентрацию на спектрофотометре NanoDrop 1000 (“Termo Fisher Scientific”).
Фракционирование клеток и РНК. Клетки HeLa, трансфицированные Rhin-1A или Rhin-1B, снимали с 4 чашек Петри, суспендировали в 2.5 мл буфера HTM-C: 0.1 М NaCl, 50 мM Трис-HCl (pH 8.0), 5 мM MgCl2, 0.25 M сахароза, – содержащего 1 мМ PMSF. Суспензию разбавляли 2.5 мл буфера HTM-C, содержащего 1% NP-40, инкубировали в течение 5 мин во льду и центрифугировали (10 мин при 1900 × g). Осадок, содержащий ядра клеток, отделяли от цитоплазматической фракции (супернатанта). РНК из ядер выделяли так же, как из целых клеток; цитоплазму экстрагировали смесью фенол (pH 8.0)–хлороформ (1 : 1), после чего РНК осаждали этанолом и обрабатывали ДНКазой I, как описано выше. Для разделения ядерной и цитоплазматической РНК на полиаденилированную, поли(А)+, и неполиаденилированную, поли(А)–, раствор РНК пропускали через колонки с олиго(dT)-целлюлозой в растворе, содержащем 0.5 M LiCl, 10 мM Трис-HCl (pH7.5), 1 мM EDTA, 0.1% SDS. Не связавшийся с колонкой материал представлял собой поли(А)– РНК. Затем колонку промывали таким же раствором, но с 0.15 M LiCl и, наконец, элюировали поли(А)+ РНК, пропуская через колонку H2O. Полученные фракции РНК анализировали с помощью нозерн-гибридизации.
Гибридизационный анализ РНК. Суммарную РНК (10 мкг), выделенную из клеток на каждой чашке, подвергали элетрофорезу в 6%-ном ПААГ с 6 М мочевиной и переносили на мембрану Hybond-XL с помощью полусухого электроблотинга. Мечение гибридизационных зондов для детекции транскриптов SINE и 4.5SI РНК, описанных ранее [20, 22], проводили, осуществляя ПЦР в присутствии α[32P]-dATP. Мембрану с иммобилизованной РНК инкубировали с зондом в гибридизационном растворе (50% формамид, 5× раствор Денхардта, 4× SSC, 1% SDS, 0.1 мг ДНК лосося) при 42°C в течение ночи, после чего отмывали в 0.1× SSC с 0.1% SDS при 42°C в течение 1 ч и экспонировали на рентгеновской пленке. Для количественной оценки радиоактивности гибридизационных сигналов мембрану сканировали в фосфоимеджере Cyclone. Величину сигналов транскриптов SINE нормировали по сигналам контрольной 4.5SI РНК. Такое нормирование проводили только для временного промежутка 0–60 мин с момента добавления актиномицина. Для точек 120 и 180 мин к нормированию не прибегали, так как за это время происходил заметный (до 65% от начального уровня) распад 4.5SI РНК (рис. S2 , см. Приложение).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ И ИХ ОБСУЖДЕНИЕ
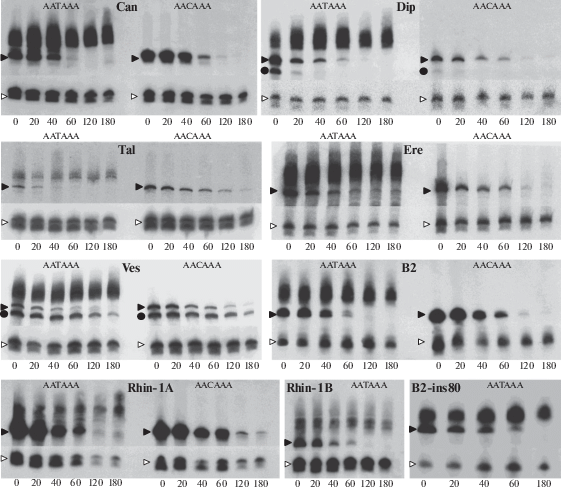
В работе использовали ранее клонированные в плазмидах копии SINE из геномов различных млекопитающих. Кроме того, были получены те же SINE, но с заменами T на C в сигнале полиаденилирования AATAAA, что приводит к его инактивации. Этими плазмидами, содержащими SINE, а также ген 4.5SI РНК в качестве контроля, трансфицировали выращенные в культуре клетки карциномы человека HeLa. Затем в среду добавляли ингибитор транскрипции актиномицин D и оценивали скорость распада изучаемой РНК, измеряя ее содержание в клетке. РНК, выделенную через определенные промежутки времени после обработки актиномицином D, анализировали электрофорезом в ПААГ с последующим нозерн-блотингом с радиоактивно меченным зондом. Радиавтографы отмытых мембран показаны на рис. 2. Транскрипты SINE с инактивированным сигналом полиаденилирования (C-конструкции) представлены, как правило, одной полосой РНК, соответствующей по длине SINE. РНК SINE с функциональным сигналом полиаденилирования (T-конструкции), помимо первичного транскрипта SINE, представлена полиаденилированной РНК – гетерогенным материалом большей длины. 4.5 SI РНК длиной 98 н., характеризующаяся высокой стабильностью [24], использована как контроль эффективности трансфекции и потерь при выделении РНК; эту РНК также выявляли с помощью радиоавтографии. Как видно из рис. 2, яркость полос, соответствующих первичным транскриптам, быстро снижалась со временем и через 3 ч падала почти до нуля. В то же время интенсивность сигнала от полиаденилированной РНК незначительно снижалась за 3 ч. Это свидетельствует о том, что полиаденилированая РНК SINE значительно стабильней, чем неполиаденилированная.
Рис. 2.
Нозерн-гибридизация РНК из клеток HeLa, трансфицированных различными SINE млекопитающих и обработанных актиномицином D для ингибирования транскрипции. Цифры под дорожками указывают время (мин) после добавления в среду актиномицина D. Для трансфекции использовали SINE c функциональным сигналом полиаденилирования (AATAAA) – Т-конструкции – или без него (ААСААА) – С-конструкции. Полноразмерные первичные транскрипты SINE отмечены черными треугольниками; гетерогенный материал с меньшей подвижностью – полиаденилированные транскрипты SINE; черными кружками отмечены укороченные транскрипты SINE Dip и Ves, не подвергающиеся полиаденилированию [20]. При трансфекции в качестве контроля добавляли ген долгоживущей 4.5SI РНК; его транскрипты отмечены светлыми треугольниками. Исследованные SINE (здесь и на других рисунках): Can – собака, Dip – большой тушканчик Allactaga major, Tal – крот Mogera robusta, Ere – лошадь, Ves – водяная ночница Myotis daubentonii, B2 – домовая мышь, Rhin-1A и Rhin-1B – большой подковонос Rhinolophus ferrumequinum, B2-ins80 – B2 со вставкой длиной 80 п.н.
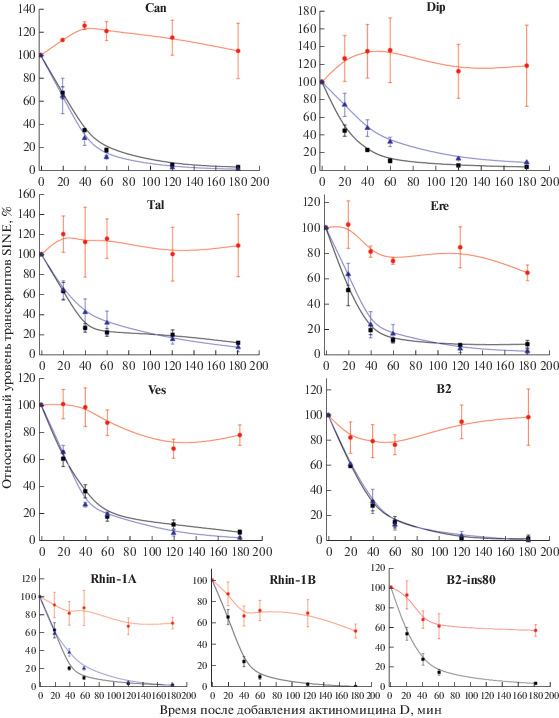
Для количественной оценки с помощью фосфоимеджера измеряли радиоактивность гибридизационных сигналов и строили графики распада неполиаденилированных и полиаденилированных транскриптов SINE. Они представлены на рис. 3. Из этих графиков видно, что время полужизни (t1/2) первичных транскриптов C-конструкций большинства SINE составляет 30 мин. Учитывая, что полиаденилирования этих транскриптов не происходит, логично предположить, что снижение их количества со временем – результат деградации. Что касается первичных транскриптов T-конструкций, то их уровень снижается не только вследствие деградации, но и за счет полиаденилирования, поэтому оценка времени полужизни этих РНК, сделанная на основе полученных графиков, на первый взгляд кажется некорректной. Это относится и к полиаденилированным РНК SINE: с одной стороны, они подвергаются деградации (хотя и с малой скоростью, судя по рис. 3); с другой, их количество пополняется за счет полиаденилирования первичного транскрипта.
Рис. 3.
Кинетика деградации транскриптов SINE. Кривые, отображающие распад первичных транскриптов SINE c функциональным сигналом полиаденилирования или без него, обозначены соответственно треугольниками и квадратами, а отображающие распад полиаденилированных транскриптов SINE – кружками. По оси X отложено время в минутах после добавления актиномицина D, по оси Y – содержание транскрипта SINE в процентах от его уровня до добавления актиномицина D. Графики получены на основании трех опытов по трансфекции; показано стандартное отклонение.
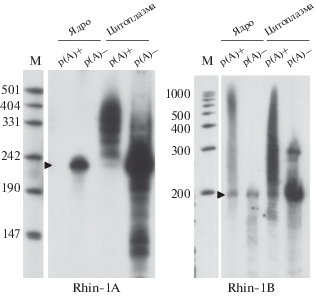
Однако возможно, что большинство молекул первичных транскриптов SINE (избежавших полиаденилирования в ядре) выходит в цитоплазму, где они не могут полиаденилироваться и тем самым пополнять фракцию поли(A)-содержащих транскриптов SINE. С целью проверить это мы проанализировали ядерно-цитоплазматическую локализацию транскриптов SINE Rhin-1A и Rhin-1B. Оказалось, что как первичные, так и полиденилированные транскрипты SINE Rhin быстро выходят из ядра в цитоплазму и накапливаются в ней (рис. 4), что согласуется с вышеупомянутым предположением. Таким образом, преимущественно цитоплазматическая локализация транскриптов SINE позволяет не учитывать вклад полиаденилирования в убыль первичных транскриптов Т-конструкций или пополнение пула полиаденилированых РНК SINE. Вследствие этого, по крайней мере в первом приближении, можно определять время полужизни первичных и полиаденилированных транскриптов Т-конструкций по графикам, приведенным на рис. 3.
Рис. 4.
Полиаденилированные и неполиаденилированные транскрипты SINE Rhin-1A и Rhin-1B преимущественно локализуются в цитоплазме. РНК, связывающаяся с олиго(dT)-целлюлозой, – p(A)+, не связывающаяся – p(A)–. Первичные транскрипты SINE отмечены черными треугольниками. М – маркеры длины РНК (число нуклеотидов).
Действительно, в большинстве случаев время полужизни первичных транскриптов T-конструкций и C-конструкций было сходным и составляло около 30 мин. Заметная разница наблюдалась только для SINE Dip: t1/2 для первичных транскриптов T-конструкций была ~20 мин, в то время как для первичных транскриптов C-конструкций – ~40 мин. Возможно, в данном случае происходит активное полиаденилирование первичного транскрипта T-конструкции Dip, поэтому его уровень быстро снижается (рис. 3). Уровни полиаденилированных транскриптов SINE B2, Can, Dip и Tal практически не изменялись за 3 ч, тогда как транскриптов SINE Ere, Rhin-1A, Rhin-1B и Ves снижались со временем, хотя и медленно – за 3 ч не более чем на 40% от начального.
Из полученных результатов можно сделать следующие выводы. Во-первых, первичные транскрипты всех исследованных SINE характеризуются коротким временем жизни. Ранее мы наблюдали [24] подобное для 4.5SH РНК (t1/2 = 20 мин), также синтезируемой РНК-полимеразой III, тогда как другая малая ядерная РНК (4.5SI) оказалась долгоживущей. Такие различия обусловлены тем, что в 4.5SI РНК 5'- и 3'-концевые районы образуют двуспиральную структуру, отсутствующую у 4.5SH РНК [24]. Искусственная замена 3'-концевого района в SINE, ведущая к образованию аналогичной двуспиральной структуре в транскрипте, также многократно увеличивала время жизни РНК [21]. Вероятно, те РНК, которые лишены особых структурных элементов, “защищающих” от действия нуклеаз, быстро деградируют в клетке. По-видимому, это в полной мере относится и к первичным транскриптам SINE.
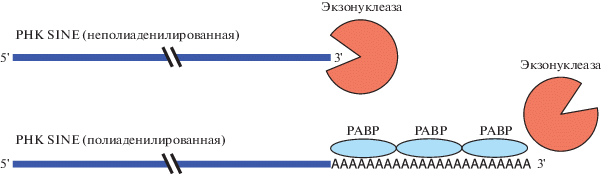
Во-вторых, полиаденилированные транскрипты SINE проявляют значительно большую стабильность в клетке; при этом от первичных транскриптов они отличаются только наличием длинного поли(A)-хвоста. Такой хвост сам по себе, видимо, не защищает РНК от деградации экзонуклеазами с 3'‑конца. Однако известно, что поли(A)-хвост в мРНК увеличивает время ее жизни в клетке благодаря тому, что он связывается с PABPs, защищающими РНК от действия экзонуклеаз [25]. По-видимому, с поли(A) транскриптов SINE также ассоциированы PABPs, что и приводит к увеличению времени жизни этих РНК (рис. 5).
Рис. 5.
Схема, иллюстрирующая возможный механизм повышения стабильности транскриптов SINE в результате их полиаденилирования. 3'-конец неполиаденилированных транскриптов подвержен деградации экзонуклеазами. Поли(A)-хвост полиаденилированных транскриптов взаимодействует с белком PABP, что защищает его от действия экзонуклеаз.
С целью проверить, будет ли сохраняться замедленная деградация полиаденилированного транскрипта SINE, в состав которого введена дополнительная нуклеотидная последовательность, в SINE B2 между районами β и τ вставили синтетический фрагмент ДНК длиной 80 п.н. Плазмидой, несущей удлиненный В2-элемент, трансфицировали клетки HeLa и оценили стабильность образующегося транскрипта. Надо отметить, что эта вставка сильно изменяла вторичную структуру транскрипта В2 в его центральной части и практически не влияла на укладку РНК в концевых областях (рис. S3 , см. Приложение). Первичные транскрипты SINE со вставкой характеризовались низкой стабильностью, сравнимой с SINE B2 “дикого типа”; в то время как его полиаденилированные транскрипты оказались долгоживущими, хотя и уступали в стабильности нативным (рис. 2 и 3). Таким образом, на время жизни транскриптов SINE мало влияют их длина, первичная и, по-видимому, вторичная структура, тогда как наличие поли(А)-хвоста повышает их стабильность. По всей видимости, в клетке транскрипты SINE в большей степени подвержены действию экзонуклеаз, гидролизующих РНК с 3'-конца, и в меньшей – эндонуклеаз; при этом поли(А)-хвост, ассоциированный с PABP, защищает РНК SINE именно от деградации с 3'-конца.
Основываясь на полученных результатах, можно разработать векторы, содержащие последовательности SINE (или их части). Такие векторы могут быть использованы для экспрессии в клетках млекопитающих искусственных некодирующих регуляторных РНК. Можно ожидать, что в клетках млекопитающих конструкции на основе этих векторов будут эффективно транскрибироваться РНК-полимеразой III, а образующиеся транскрипты подвергаться полиаденилированию. Благодаря этому время жизни, а значит, и клеточный уровень такой РНК окажется значительно выше, чем аналогичной РНК, не имеющей поли(А)-хвоста. В результате эффекты, индуцируемые этой искусственной некодирующей РНК, могут быть выражены гораздо сильнее, чем для РНК, не содержащей структурных элементов, препятствующих ее быстрой деградации.
Работа выполнена при поддержке РФФИ (проекты 18-34-00247мол_а и 17-04-00686).
Настоящая статья не содержит каких-либо исследований с использованием животных в качестве объектов.
Настоящая статья не содержит каких-либо исследований с участием людей в качестве объектов исследований.
Конфликт интересов: Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Schoenberg D.R., Maquat L.E. (2012) Regulation of cytoplasmic mRNA decay. Nat. Rev. Genet. 13, 246–259.
Ntini E., Jarvelin A.I., Bornholdt J., Chen Y., Boyd M., Jorgensen M., Andersson R., Hoof I., Schein A., Andersen P.R., Andersen P.K., Preker P., Valen E., Zhao X., Pelechano V., Steinmetz L.M., Sandelin A., Jensen T.H. (2013) Polyadenylation site-induced decay of upstream transcripts enforces promoter directionality. Nat. Struct. Mol. Biol. 20, 923–928.
Barreau C., Paillard L., Osborne H.B. (2005) AU-rich elements and associated factors: are there unifying principles? Nucleic Acids Res. 33, 7138–7150.
Goodarzi H., Najafabadi H.S., Oikonomou P., Greco T.M., Fish L., Salavati R., Cristea I.M., Tavazoie S. (2012) Systematic discovery of structural elements governing stability of mammalian messenger RNAs. Nature. 485, 264–268.
Rashid F., Shah A., Shan G. (2016) Long non-coding RNAs in the cytoplasm. Genomics Proteomics Bioinformatics. 14, 73–80.
Iwakawa H.O., Tomari Y. (2015) The functions of microRNAs: mRNA decay and translational repression. Trends Cell Biol. 25, 651–665.
Nagarajan V.K., Jones C.I., Newbury S.F., Green P.J. (2013) XRN 5' → 3' exoribonucleases: structure, mechanisms and functions. Biochim. Biophys. Acta. 1829, 590–603.
Chlebowski A., Lubas M., Jensen T.H., Dziembowski A. (2013) RNA decay machines: the exosome. Biochim. Biophys. Acta. 1829, 552–560.
Eckmann C.R., Rammelt C., Wahle E. (2011) Control of poly(A) tail length. Wiley Interdiscip. Rev. RNA. 2, 348–361.
Proudfoot N.J. (2011) Ending the message: poly(A) signals then and now. Genes Dev. 25, 1770–1782.
Zheng D., Ezzeddine N., Chen C.Y., Zhu W., He X., Shyu A.B. (2008) Deadenylation is prerequisite for P-body formation and mRNA decay in mammalian cells. J. Cell Biol. 182, 89–101.
Устьянцев И.Г., Голубчикова Ю.С., Бородулина О.Р., Крамеров Д.А. (2017) Каноническое и неканоническое полиаденилирование РНК. Молекуляр. биология. 51, 262–273.
Falk S., Weir J.R., Hentschel J., Reichelt P., Bonneau F., Conti E. (2014) The molecular architecture of the TRAMP complex reveals the organization and interplay of its two catalytic activities. Mol. Cell. 55, 856–867.
Schmidt K., Butler J.S. (2013) Nuclear RNA surveillance: role of TRAMP in controlling exosome specificity. Wiley Interdiscip. Rev. RNA. 4, 217–231.
Lubas M., Andersen P.R., Schein A., Dziembowski A., Kudla G., Jensen T.H. (2015) The human nuclear exosome targeting complex is loaded onto newly synthesized RNA to direct early ribonucleolysis. Cell. Rep. 10, 178–192.
Kramerov D.A., Vassetzky N.S. (2011) SINEs. Wiley Interdiscip. Rev. RNA. 2, 772–786.
Borodulina O.R., Kramerov D.A. (2001) Short interspersed elements (SINEs) from insectivores. Two classes of mammalian SINEs distinguished by A-rich tail structure. Mamm. Genome. 12, 779–786.
Kramerov D.A., Tillib S.V., Shumyatsky G.P., Georgiev G.P. (1990) The most abundant nascent poly(A)+ RNAs are transcribed by RNA polymerase III in murine tumor cells. Nucleic Acids Res. 18, 4499–4506.
Borodulina O.R., Kramerov D.A. (2008) Transcripts synthesized by RNA polymerase III can be polyadenylated in an AAUAAA-dependent manner. RNA. 14, 1865–1873.
Borodulina O.R., Golubchikova J.S., Ustyantsev I.G., Kramerov D.A. (2016) Polyadenylation of RNA transcribed from mammalian SINEs by RNA polymerase III: complex requirements for nucleotide sequences. Biochim. Biophys. Acta. 1859, 355–365.
Koval A.P., Gogolevskaya I.K., Tatosyan K.A., Kra-merov D.A. (2015) A 5'-3' terminal stem in small non-coding RNAs extends their lifetime. Gene. 555, 464–468.
Gogolevskaya I.K., Kramerov D.A. (2010) 4.5SI RNA genes and the role of their 5'-flanking sequences in the gene transcription. Gene. 451, 32–37.
Chomczynski P., Sacchi N. (2006) The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nat. Protoc. 1, 581–585.
Koval A.P., Gogolevskaya I.K., Tatosyan K.A., Kramerov D.A. (2012) Complementarity of end regions increases the lifetime of small RNAs in mammalian cells. PLoS One. 7, e44157.
Goss D.J., Kleiman F.E. (2013) Poly(A) binding proteins: are they all created equal? Wiley Interdiscip. Rev. RNA. 4, 167–179.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


