Молекулярная биология, 2020, T. 54, № 1, стр. 153-163
In silico СКРИНИНГ ФЛАВОНОВ И ИХ ПРОИЗВОДНЫХ КАК ПОТЕНЦИАЛЬНЫХ ИНГИБИТОРОВ КВОРУМ-СЕНСИНГ РЕГУЛЯТОРА LasR Pseudomonas aeruginosa
Н. Абелян a, *, О. Грабский b, С. Тирацуян a, b
a Институт биомедицины и фармации Российско-Армянского университета
0051 Ереван, Армения
b Факультет биологии Ереванского государственного университета
0025 Ереван, Армения
* E-mail: narek.abelyan@rau.am
Поступила в редакцию 20.04.2019
После доработки 20.04.2019
Принята к публикации 06.05.2019
Аннотация
Резистентность к антибиотикам − глобальная проблема общественного здравоохранения. В 2017 г. ВОЗ опубликовала список супербактерий, среди которых критически приоритетна Pseudomonas aeruginosa. Классические методы борьбы против бактериальных сообществ неэффективны, потому что они создают биопленки, придающие повышенную устойчивость к антибиотикам и иммунным реакциям хозяина. Сущность новых стратегий состоит в том, чтобы “не убить, а разоружить” патоген, предполагая, что устойчивость к таким антивирулентным соединениям будет вырабатываться медленнее. Известно, что в системе кворум-сенсинга P. aeruginosa система LasI/LasR основная. Рецепторный белок LasR активирует связывание с нативным аутоиндуктором. Многие флавоноиды и их производные используют как антибактериальные компоненты лекарств. Цель нашей работы − выявление соединений, которые будут ингибировать LasR. Это приведет к подавлению синтеза факторов вирулентности, тем самым лишая бактерии вирулентности. Чтобы получить согласованные прогнозы, в работе проведен высокопроизводительный виртуальный скрининг с использованием программных пакетов AutoDock Vina, rDock, LeDock. Результаты виртуального скрининга позволяют предположить, что бензамиды, синтетические производные флавонов, потенциально способны ингибировать регулятор транскрипции LasR. Это согласуется с недавно опубликованными экспериментальными данными, которые показывают высокую антибактериальную активность бензамидов. Выбранные соединения взаимодействуют с лигандсвязывающим доменом LasR с более высокой аффинностью, чем с ДНК-связывающим доменом. С помощью конформационного анализа среди отобранных соединений обнаружено, что существуют такие, которые связываются с теми же аминокислотами лигандсвязывающего домена, что и нативный аутоиндуктор. Это указывает на возможность конкурентного взаимодействия этих соединений. Также установлен ряд соединений, которые связываются с другими консервативными аминокислотами лигандсвязывающего домена, что интересно для дальнейшего их анализа. Отобранные соединения соответствуют критериям, необходимым для их рассмотрения в качестве лекарственных средств, и могут служить основой для проведения дальнейших экспериментов in vitro/in vivo и разработки современной противоинфекционной терапии, основанной на QS-системе P. aeruginosa.
ВВЕДЕНИЕ22
Резистентность к антибиотикам − глобальная проблема современности, каждый год в США, как минимум 2 миллиона человек получают резистентную к антибиотикам инфекцию, и, по меньшей мере, 23 000 человек умирают [1]. В 2017 г. ВОЗ публикует список бактерий, для которых срочно необходимы новые антибиотики, и критической приоритетностью обладает синегнойная палочка (Pseudomonas aeruginosa) [2]. Эти бактерии размножаются в организме хозяина, а при достижении определенной концентрации, именуемой QS, объединяются в сообщества (биопленки) [3, 4]. Секретируя молекулярные сигналы, ответственные за клеточную коммуникацию, бактерии могут совместно принимать решения для приспособления к особенностям среды и защиты. Биопленки способствуют экспрессии генов вирулентности, включая гены резистентности к антибиотикам и множественной лекарственной резистентности, а также представляют собой физический барьер, предотвращающий проникновение вредных веществ, в том числе и антибиотиков [5, 6].
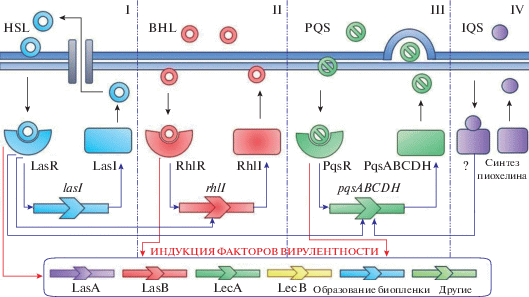
У P. aeruginosa обнаружены четыре типа сигнальных систем кворум-сенсинга. Две из них основаны на ацилгомосеринлактонах HSLs: OdDHL и BHL. Данные аутоиндукторы распознают две разные сигнальные системы, каждая из которых включает рецептор типа LuxR и синтазу типа LuxI. LasI синтезирует OdDHL, который связывается с рецептором LasR [7, 8]. RhlI синтезирует BHL, который взаимодействует с рецепторным белком RhlR [9, 10]. Эти две системы интегрированы с третьей, в которой использованы химически отличные от HSLs сигнальные молекулы: 2‑гептил-3-гидрокси-4(1Н)-хинолон. Третья система названа PQS [11]. Четвертая система IQS недостаточно изучена, а рецептор IQS не идентифицирован [12]. В регуляции кворум-сенсинга P. aeruginosa существует иерархия, согласно которой активация LasR ведет за собой дальнейшую активацию систем RhlR и PQS. Именно по этой причине LasR представляет больший интерес в плане дальнейших исследований [4] (рис. 1).
В присутствии зрелых биопленок P. aeruginosa фагоциты не могут осуществлять фагоцитоз из-за наличия экзополисахаридной поверхности биопленок. С другой стороны, бактерии могут продолжать агрессивно реагировать на присутствие лейкоцитов, регулируя производство ряда QS-контролируемых факторов вирулентности, включая рамнолипиды [13], цианиды, антибиотик пиоцианин [14], и экзополисахариды [15], которые затем окружают биопленку и влияют на проникновение иммунных клеток.
Подход, направленный на исправление этой ситуации, предполагает разработку соединений, не убивающих патогенные бактерии, но ингибирующих или инактивирующих их факторы вирулентности. Иначе говоря, стратегия состоит в том, чтобы “не убить, а разоружить” патоген. Предполагаем, что устойчивость к таким антипатогенным соединениям будет вырабатываться медленнее, поскольку они не будут влиять непосредственно на жизнеспособность бактерий, а будут действовать только на их способность инфицировать человека.
Цель этой работы ‒ найти соединения, которые будут препятствовать функционированию LasR. Это приведет к подавлению образования биопленок и синтеза факторов вирулентности. Высокопроизводительный in vitro скрининг – один из наиболее эффективных и хорошо зарекомендовавших себя методов, используемых в области разработки лекарственных препаратов. Однако на сегодняшний день широко признано предварительное использование in silico методов, что уменьшает количество тестируемых соединений для дальнейших экспериментов in vitro и in vivo, тем самым снижая их высокую стоимость. Также это сокращает время исследований и увеличивает вероятность обнаружения биологически активных соединений.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
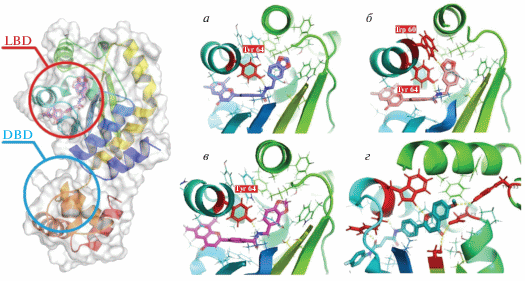
Подготовка структуры белка и идентификация сайтов связывания. Из-за плохой растворимости белка LasR отсутствуют кристаллографические данные о полной структуре рецептора. В связи с этим в качестве мишени впервые взята виртуально реконструированная полная структура рецептора LasR [16]. Сайты связывания также взяты из работы [16] и проверены с помощью программы MetaPocket [17]. В результате идентифицировано 2 основных связывающих кармана, один из которых расположен в лигандсвязывающем домене (LBD), а другой – в ДНК-связывающем домене (DBD).
Создание библиотеки химических соединений. Флавоноиды − группа вторичных метаболитов, которые проявляют широкий спектр фармакологических активностей, таких как противомикробные, противовоспалительные, антивирусные, антинеопластические, антиатерогенные и др. [18]. В некоторых исследованиях показана способность флавоноидов ингибировать образование биопленок [19]. Исходя из этого, мы собрали библиотеку флавонов и их производных в качестве потенциальных соединений, способных ингибировать систему кворум-сенсинга P. aeruginosa, коррелирующую с образованием биопленок.
Чтобы создать библиотеку химических соединений, использовали базу данных PubChem [20], из которой методом Substructure отобрали 51 675 флавонов и их производных. Далее на основании правила пяти Lipinski [21] мы исключили те соединения, для которых спрогнозирована плохая адсорбция или проникающая способность. В результате число соединений уменьшили до 21 232. Вдобавок, чтобы удалить соединения, не имеющие химических производных, использован фильтр Chemical Vendors, что привело к сокращению числа соединений до 10 532. Чтобы подготовить и оптимизировать лиганды, рассчитали конформационную подвижность (определение степеней свободы), добавили полярные атомы водорода и рассчитали заряды всех атомов. Структуры обработаны с использованием программного пакета OpenBabel v2.4.0 [22].
Молекулярный докинг осуществляли с использованием трех программных пакетов: AutoDock Vina [23], rDock [24] и LeDock [25], – которые показали высокую производительность в недавней комплексной оценке программ докинга [26].
Программы молекулярного докинга основаны на поиске конформации лиганда с минимальной энергией. При ранжировании возможных конформаций лиганда использовали аффинность связывания ΔG [27]. Последнюю рассчитывают на основе полной энергии межмолекулярных сил, включающих вандерваальсовы, электростатические, гидрофобные взаймодействия и водородные связи. Низкие значения ΔG соответствуют сильному, тогда как высокие – слабому связыванию лиганда [28].
На начальном этапе определяли пространственно-энергетические показатели взаимодействия исследуемых соединений с двумя активными центрами с использованием программного пакета AutoDock Vina [23]. При настройке, запуске и анализе симуляции использован бесплатный графический интерфейс AutoDock Tools [29]. С помощью этой программы осуществляли выбор двух боксoв докинга, размеры которых не превышали 27 000 Å3, и далее использовали значение глубины поиска, равное 10, которое рекомендуют для быстрого виртуального скрининга малых боксов [30]. Результаты докинг-анализа 10 соединений, показавших самую высокую энергию связывания с обоими сайтами, приведены в табл. 1 .
Таблица 1.
Результаты докинг-анализа соединений, показавших самую высокую энергию связывания с LBD и DBD LasR
| № | PubChem CID | Аффинность связывания LBD, ккал/моль | PubChem CID | Аффинность связывания DBD, ккал/моль |
|---|---|---|---|---|
| 1 | 108 786 995 | –12.6 | 60 617 261 | –10.9 |
| 2 | 108 754 324 | –12.3 | 46 684 079 | –10.5 |
| 3 | 108 787 038 | –12.2 | 108 773 117 | –10.2 |
| 4 | 108 800 047 | –12.2 | 16 297 521 | –10.2 |
| 5 | 1695 677 | –12.0 | 24 335 408 | –10.1 |
| 6 | 108 802 999 | –12.0 | 16 505 442 | –10.0 |
| 7 | 108 790 351 | –11.9 | 108 773 119 | –10.0 |
| 8 | 108 803 145 | –11.9 | 31 836 827 | –10.0 |
| 9 | 108 800 021 | –11.8 | 41 418 450 | –10.0 |
| 10 | 108 786 994 | –11.8 | 52 655 891 | –9.9 |
Важно отметить, что связывание нативного лиганда OdDHL с LBD происходит с энергией, равной –5.7 ккал. Полученные результаты свидетельствуют о том, что связывание выбранных соединений происходит интенсивно как с LBD, так и с DBD. Однако большинство химических соединений проявляло более высокую аффинность к LBD, поэтому дальнейшие исследования проводили именно с этим сайтом взаимодействия. Так как программы докинга несовершенны, для своих расчетов они используют упрощенные модели квантово-химических взаимодействий, а также менее ресурсозатратные алгоритмы, то для получения более достоверных результатов проведен молекулярный докинг тех же 10 532 соединений другими программами докинга, а именно rDock [24] и LeDock [25]. Молекулярный докинг с помощью rDock [24] включает три основных шага: первый – определение системы, второй – генерация полости, третий – докинг. Во время докинга проводили 50 запусков для каждого лиганда, согласно рекомендации разработчиков rDock [24]. При проведении молекулярного докинга с помощью программы LeDock [25] генерацию входных файлов осуществляли по программе LePro [25].
В связи с использованием трех различных программ возникла необходимость обработки огромного количества информации по молекулярным докингам, поэтому мы разработали методику обработки данных.
Методика обработки данных. На первом этапе проводили нормализацию полученных результатов и создание общей оценочной системы. Поскольку разные программные пакеты рассчитывают энергию взаимодействия в разных единицах, то возникла необходимость перевода их в единую оценочную систему, где 100 единиц получает соединение, имеющее максимальную величину энергии взаимодействия, a 1 – соединение с минимальной энергией. Энергию взаимодействия всех соединений для каждой программы переводили в общую оценочную систему. В табл. 2 представлены нормализованные оценки связывания с LBD LasR 10 соединений из первоначального списка, включающего 10 532 наименований.
Таблица 2.
Нормализованная оценка связывания с LBD LasR соединений, проявивших высокую аффинность по программам AutoDock Vina [23], rDock [24] и LeDock [25]
| AutoDock | rDock | LeDock | |||||
|---|---|---|---|---|---|---|---|
| PubChem CID | Нормализованная оценка | PubChem CID | Нормализованная оценка | PubChem CID | Нормализованная оценка | ||
| 108 786 995 | 100 | 16 801 690 | 100 | 24 280 119 | 100 | ||
| 108 754 324 | 96.04 | 2 817 219 | 98.74 | 53 486 694 | 97.52 | ||
| 108 787 038 | 94.72 | 18 568 736 | 95.66 | 16 408 601 | 97.68 | ||
| 108 800 047 | 94.72 | 82 046 503 | 95.45 | 6 336 429 | 97.26 | ||
| 1 695 677 | 92.08 | 25 555 952 | 94.66 | 108 809 778 | 96.84 | ||
| 108 802 999 | 92.08 | 93 713 806 | 94.57 | 396 728 | 96.84 | ||
| 108 790 351 | 90.76 | 21 000 649 | 94.51 | 16 408 064 | 96.63 | ||
| 108 803 145 | 90.76 | 18 568 755 | 94.48 | 108 787 045 | 95.58 | ||
| 108 800 021 | 89.44 | 93 713 804 | 93.28 | 5 449 792 | 95.37 | ||
| 108 786 994 | 89.44 | 108 811 376 | 92.98 | 16 407 858 | 94.95 | ||
Вторым шагом осуществляли суммирование нормализованных оценок, полученных тремя программами для каждого химического соединения. Затем, третьим шагом, проводили ранжирование соединений по их суммарной оценке, в результате чего наверху списка оказались соединения, получившие хорошую предсказательную оценку тремя программами (табл. 3). Таким образом, у тех соединений, которые получали ложные положительные результаты одной программой, понижался ранг в списке. Не исключено при этом, что все три программы предскажут ложные положительные или отрицательные результаты, но вероятность этого крайне мала, так как каждая из этих программ использует своеобразные методы предсказания. В свою очередь, вероятность ложного предсказания программами мала по сравнению с вероятностью истинного предсказания. Поскольку цель виртуального скрининга – поиск компромисса между долей действительно активных соединений в верхней части списка и их общим числом в списке, мы считаем использование этого подхода вполне обоснованным.
Таблица 3.
Соединения, получившие хорошие оценки всеми программами
| Соединение по IUPAC-номенклатуре | PubChem | AutoDock | rDock | LeDock | Sum |
|---|---|---|---|---|---|
| N-(1,3-benzodioxol-5-ylmethyl)-4-(6,8-dimethyl-4-oxochromen-2-yl)benzamide | 108 786 995 | 100 | 81.12 | 72.46 | 253.59 |
| N-[2-(3,4-dihydroxyphenyl)ethyl]-2-(5-hydroxy-4-oxo-2-phenyl-chromen-7-yl)oxyacetamide | 16 408 424 | 68.32 | 86.34 | 92.85 | 247.52 |
| N-(3-acetamidophenyl)-2-(5-hydroxy-4-oxo-2-phenylchromen-7-yl)oxyacetamide | 16 407 791 | 70.96 | 86.03 | 88.64 | 245.64 |
| 4-(6-methyl-4-oxochromen-2-yl)-N-[5-(2-methylpropyl)-1,3,4-thiadiazol-2-yl]benzamide | 108 803 142 | 85.48 | 76.67 | 82.34 | 244.50 |
| 4-(6-methyl-4-oxochromen-2-yl)-N-[2-(4-sulfamoylphenyl)ethyl]benzamide | 108 790 338 | 78.88 | 85.36 | 85.36 | 243.01 |
| 2-(5-hydroxy-4-oxo-2-phenylchromen-7-yl)oxy-N-(2-oxochromen-6-yl)acetamide | 16 408 830 | 65.68 | 83.23 | 92.01 | 240.92 |
| (2S)-2-[[2-(5-hydroxy-4-oxo-2-phenylchromen-7-yl)oxyacetyl]amino]-3-(1H-indol-3-yl)propanoic acid | 29 147 486 | 73.6 | 78.45 | 88.22 | 240.28 |
| 3-[[[4-(6,8-dimethyl-4-oxochromen-2-yl)benzoyl]ami-no]methyl]benzoic acid | 108 800 059 | 88.12 | 71.76 | 79.61 | 239.49 |
| 2-(5-hydroxy-4-oxo-2-phenylchromen-7-yl)oxy-N-(pyridin-3-ylmethyl)acetamide | 16 408 601 | 67.0 | 74.34 | 97.68 | 239.03 |
| N'-(4-fluorophenyl)-N-[4-(6-methoxy-4-oxochromen-2-yl)phenyl]butanediamide | 108 754 330 | 81.52 | 85.79 | 70.57 | 237.89 |
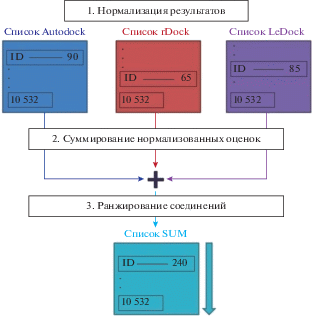
Чтобы автоматизировать весь процесс обработки данных, написали скрипт. Схема работы алгоритма представлена на рис. 2.
Рис. 2.
Схема работы алгоритма, ID – идентификационный номер соединения, напротив – значение нормализованной оценки.
Анализ сходства с лекарственными препаратами. Расчет физико-химических дескрипторов, а также предсказание параметров ADMEТ (абсорбции (Absorption), распределения по тканям (Distribution), метаболизма (Metabolism), выделений из организма (Excretion) и токсичности (Toxicity)) проводили с помощью трех различных программ, в частности SwissADME [31], admetSAR [32] и PASS online [33], которые удачно используют при открытии лекарств. Прогноз токсичности, проведенный в PASS online, включает в себя оценку вероятности наличия (Pa) и отсутствия каждой активности (Pi) со значениями в диапазоне от 0 до 1.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
В исходной библиотеке мы отобрали 10 соединений, которые получили высокие оценки по результатам трех программ (рис. S1 , см. Приложение на сайте http://www.molecbio.ru/downloads/2020/1/ supp_Абелян_rus.pdf).
В структуре нативного лиганда (OdDHL) есть амидная связь [–CO–NH–], ответственная за связывание с Asp73 и Tyr56 LBD LasR. Такая же химическая связь присутствует в некоторых уже известных соединениях, взаимодействующих с LasR [34–37]. Примечательно, что во всех 10 выявленных нами соединениях присутствовала амидная связь. Это может говорить о сопоставимости полученных результатов и важности этой связи в структуре соединений, обеспечивающих сильное взаимодействие с LBD LasR.
Выявленные нами лидирующие соединения имеют некоторое структурное сходство с соединениями, для которых уже выявлен ингибирующий эффект системы LasR/LasI.
Из 10 соединений, содержащих амидную связь, в семи встречается бензамидная группа. В трех из них бензольное кольцо связано с атомом азота аминогруппы (CID 16407791, CID 16 408 830, CID 108 754 330) а в четырех − с углеродом карбонильной группы (CID 108 786 995, CID 108 803 142, CID 108790338, CID 108 800 059). Из бензамидов CID 108786995 получил максимальную оценку по результатам трех программ (табл. 3, рис. 3). Согласно некоторым экспериментальным данным, соединения, содержащие бензамидную группу, обладали анти-вирулентными свойствами в in vitro/ in vivo моделях. В работе [38] выявлено соединение с бензамидной группой – нифуроксазид, которое оказывало ингибирующее действие на систему QS, с непосредственным ингибированием регулятора транскрипции белка LasR. В работе [37] обнаружены трифенилмиметики OdDHL, взаимодействующие с LasR, которые также содержали бензамидную группу. В работе [36] в результате скрининга найден ряд соединений, содержащих бензамидную группу с выраженными анти-вирулентными свойствами. Эти соединения снижали продукцию HAQ, которая продуцируется системой PqsR. Наиболее мощные ингибиторы (M51, M34, M62, M50 и M64) не ингибировали белки, участвующие в непосредственном синтезе HAQ (экспрессируемые генами pqsABCD), что позволяет предположить, что эти ингибиторы нацелены на MvfR или другой восходящий регуляторный компонент. Показано, что соединение M64 взаимодействует с MvfR, что может привести к снижению продукции HAQ. Однако следует отметить, что снижение продукции HAQ и активация каскада системы PqsR (MvfR) происходит также через систему LasI/LasR. И поскольку в большинстве выявленных нами потенциальных кандидатов, ингибирующих LasR, есть бензамидная группа, то можно предположить, что для остальных соединений снижение продукции HAQ также обусловлено именно ингибированием системы LasR/LasI. Согласно статье [38], выявленные 3 анти-вирулентные соединения, включая нифуроксазид, кроме непосредственного ингибирования LasR, обладали также прямым ингибирующим действием на системы кворум-сенсинга Rhl и PqsR.
Рис. 3.
Взаимодействие CID 108786995 с LasR LBD: а − конформация лиганда, предсказанная программой AutoDock; б − конформация лиганда, предсказання rDock; в − конформация лиганда, предсказання LeDock; г − связывание CID 108754330 с LBD LasR, предсказання rDock. При визуализации результатов использовали программы MarvinSketch [41], PyMOL [42] и VMD [43].
CID29147486 содержит бензол, связанный с пирролом (индол). Такая же химическая структура встречается и у индол HSL – соединения, которое ингибирует белок LasR [39].
CID 108800059 содержит бензойную кислоту, которая входит в состав салициловой кислоты и также обладает ингибирующим действием на LasR [38].
CID 16408830 содержит кумариновую группу (Coumarin(1,2-Benzopyrone)). Согласно исследованиям, кумарин обладает антибиопленочной активностью против патогена человека P. aeruginosa [40].
Конформационный анализ
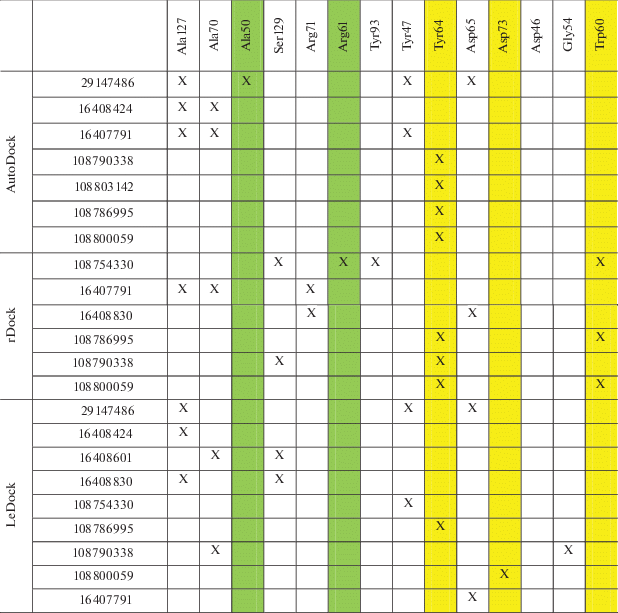
Проведен конформационный анализ с визуализацией программой LigPlot+ для 10 наилучших соединений, входящих в ранжированный список [44]. Он показал, что некоторые из отобранных нами соединений связываются с теми же аминокислотами LBD, что и нативный лиганд. На основании кристаллографических данных установлено, что лактон OdDHL образует водородные связи с Asp73, Trp60, Tyr56, Ser129 [34] кармана LBD (рис. 4). Это может указывать на возможность конкурентного взаимодействия этих соединений. Следует отметить, что также обнаружены лиганды, которые могут прочно связываться с другими консервативными аминокислотами с образованием водородных связей или за счет ван-дер-ваальсовых сил и электростатических взаимодействий (рис. S2 –S4, см. Приложение на сайте http://www.molecbio.ru/ downloads/2020/1/supp_Абелян_rus.pdf).
Рис. 4.
Кристаллографическая структура нативного лиганда OdDHL с LBD LasR (а); связывание CID 108754330 с LBD LasR, предсказанное rDock (б).
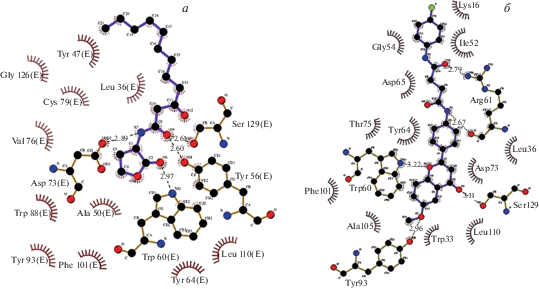
Анализ конформаций, полученных с использованием AutoDock Vina [23], показал, что соединения CID 29147486, 16408424, 16407791, 108790338, 108803142, 108786995 и 108800059 образуют водородные связи с LBD LasR. Из упомянутых соединений только соединения CID 108790338, 108803142, 108786995, 108800059 формируют водородную связь с консервативным аминокислотным остатком Tyr64 LBD LasR, тогда как CID 29147486 − с полуконсервативным Ala50 (табл. 4).
Анализ конформаций, установленных с помощью молекулярного докинга rDock [24], показал, что CID 16407791, 16408424, 16408830, 108754330, 108786995, 108790338, 108800059 образуют водородные связи с LBD LasR, соединения CID 108754330, 108786995, 108800059 − с консервативным аминокислотным остатком Trp60, тогда как CID 108786995, 108790338, 108800059 − с Tyr64 и CID 108754330 – с Arg61.
Результаты анализа конформаций докинга LeDock [25] свидетельствуют об образовании водородных связей с LBD LasR CID 29 147 486, 16 408 424, 16 408 601, 16 408 830, 108 754 330, 108 786 995, 108 790 338, 108 800 059, 16 407 791. Среди указанных соединений CID 108 800 059 образует водородные связи с консервативным Asp73, а CID 108786995 − с Tyr64.
Анализ сходства с лекарственными препаратами
По расчетным параметрам соединение CID 108 786 995 обладало потенциальными свойствами для использования в качестве лекарственного средства в биологических системах (рис. S5 , см. Приложение на сайте http://www.molecbio.ru/ downloads/2020/1/supp_Абелян_rus.pdf). В admetSAR [32] предсказана высокая вероятность адсорбции желудочно-кишечным трактом (HIA+) и проникновения через гематоэнцефалический барьер (BBB+). По программе SwissADME [31] также предсказана активность HIA+, но не установлена BBB–. Прогноз токсичности выполнен с использованием PASS онлайн [33]. Согласно прогнозируемым результатам CID 108 786 995 не токсичен. Первые десять соединений, входящие в список, удовлетворяют критериям сходства с лекарственными средствами, как и CID 108 786 995, поэтому они заслуживают дальнейшего изучения как в тестах in vitro, так и в экспериментах in vivo.
ВЫВОДЫ
Большинство флавонов и их производных можно рассматривать как потенциальные антибактериальные лекарственные соединения, которые взаимодействуют с LBD LasR с более высокой аффинностью связывания, чем с DBD. Среди выбранных на основании конформационного анализа соединений обнаружено, что существуют такие, которые связываются с теми же аминокислотами LBD, что и нативный лиганд. Это может указывать на возможность их конкурентного взаимодействия. Выявлен также ряд соединений, которые связываются с другими консервативными аминокислотами LBD и представляют интерес для дальнейшего изучения. Результаты виртуального скрининга согласуются с экспериментальными данными, которые показывают высокую антибактериальную активность бензамидов [35–37]. Сравнивая имеющиеся литературные данные, можно предположить, что бензамидсодержащие соединения способны ингибировать различные пути регуляторной сети системы QS. Таким образом, идентифицированные нами соединения могут потенциально ингибировать одновременно две системы QS. Выбранные соединения соответствуют критериям, необходимым для их рассмотрения в качестве лекарственных средств, и могут служить основой для проведения дальнейших экспериментов in vitro/in vivo при разработке современной противоинфекционной терапии на основе системы кворум-сенсинга P. aeruginosa.
Мы благодарны за финансовую поддержку Министерству образования и науки Республики Армения (грант 10-2/I-1).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Hampton T. (2013) Report reveals scope of US antibiotic resistance threat. JAMA. 310, 1661–1663. https://doi.org/10.1001/jama.2013.280695
Tacconelli E., Carrara E., Savoldi A., Harbarth S., Mendelson M., Monnet D.L., Pulcini C., Kahlmeter G., Kluytmans J., Carmeli Y., Ouellette M., Outterson K., Patel J., Cavaleri M., Cox E.M., Houchens CR., Grayson M.L., Hansen P., Singh N., Theuretzbacher U., Magrini N. (2018) Discovery, research, and development of new antibiotics: the WHO priority list of antibiotic-resistant bacteria and tuberculosis. Lancet Infect. Dis. 18, 318–327. https://doi.org/10.1016/S1473-3099(17)30753-3
Lee J., Zhang L. (2015) The hierarchy quorum sensing network in Pseudomonas aeruginosa. Protein Cell. 6, 26–41. https://doi.org/10.1007/s13238-014-0100-x
Pérez-Pérez M., Jorge P., Pérez Rodríguez G., Pereira M.O., Lourenço A. (2017) Quorum sensing inhibition in Pseudomonas aeruginosa biofilms: new insights through network mining. Biofouling. 33, 128–142. https://doi.org/10.1080/08927014.2016.1272104
Williams P., Cámara M. (2009) Quorum sensing and environmental adaptation in Pseudomonas aeruginosa: a tale of regulatory networks and multifunctional signal molecules. Curr. Opin. Microbiol. 12, 182–191. https://doi.org/10.1016/j.mib.2009.01.005
Rutherford S.T., Bassler B.L. (2012) Bacterial quorum sensing: its role in virulence and possibilities for its control. Cold Spring Harb. Perspect. Med. 2. https://doi.org/10.1101/cshperspect.a012427
Moré M.I., Finger L.D., Stryker J.L., Fuqua C., Eberhard A., Winans S.C. (1996) Enzymatic synthesis of a quorum-sensing autoinducer through use of defined substrates. Science. 272, 1655–1658. https://doi.org/10.1126/science.272.5268.1655
Parsek M.R., Val D.L., Hanzelka B.L., Cronan J.E., Greenberg E.P. (1999) Acyl homoserine-lactone quorum-sensing signal generation. Proc. Natl. Acad. Sci. USA. 96, 4360–4365. https://doi.org/10.1073/pnas.96.8.4360
Ochsner U.A., Fiechter A., Reiser J. (1994) Isolation, characterization, and expression in Escherichia coli of the Pseudomonas aeruginosa rhlAB genes encoding a rhamnosyltransferase involved in rhamnolipid biosurfactant synthesis. J. Biol. Chem. 269, 19787–19795.
Pearson J.P., Passador L., Iglewski B.H., Greenberg E.P. (1995) A second N-acylhomoserine lactone signal produced by Pseudomonas aeruginosa. Proc. Natl. Acad. Sci. USA. 92, 1490–1494. https://doi.org/10.1073/pnas.92.5.1490
McGrath S., Wade D.S., Pesci E.C. (2004) Dueling quorum sensing systems in Pseudomonas aeruginosa control the production of the Pseudomonas quinolone signal (PQS). FEMS Microbiol. Lett. 230, 27–34. https://doi.org/10.1016/S0378-1097(03)00849-8
Moradali M.F., Ghods S., Rehm B.H. (2017) Pseudomonas aeruginosa lifestyle: a paradigm for adaptation, survival, and persistence. Front Cell. Infect. Microbiol. 7, 39. https://doi.org/10.3389/fcimb.2017.00039
Jensen P.Ø., Bjarnsholt T., Phipps R., Rasmussen T.B., Calum H., Christoffersen L., Moser C., Williams P., Pressler T., Givskov M., Høiby N. (2007) Rapid necrotic killing of polymorphonuclear leukocytes is caused by quorum-sensing-controlled production of rhamnolipid by Pseudomonas aeruginosa. Microbiology. 153, 1329–1338. https://doi.org/10.1099/mic.0.2006/003863-0
Allen L., Dockrell D.H., Pattery T., Lee D.G., Cornelis P., Hellewell P.G., Whyte M.K. (2005) Pyocyanin production by Pseudomonas aeruginosa induces neutrophil apoptosis and impairs neutrophil-mediated host defenses in vivo. J. Immunol. 174, 3643–3649. https://doi.org/10.4049/jimmunol.174.6.3643
Bylund J., Burgess L.A., Cescutti P., Ernst R.K., Speert D.P. (2006) Exopolysaccharides from Burkholderia cenocepacia inhibit neutrophil chemotaxis and scavenge reactive oxygen species. J. Biol. Chem. 281, 2526–2532. https://doi.org/10.1074/jbc.M510692200
Grabski H., Hunanyan L., Tiratsuyan S., Vardapetyan H. (2019) Interaction of N-3-oxododecanoyl homoserine lactone with transcriptional regulator LasR of Pseudomonas aeruginosa: Insights from molecular docking and dynamics simulations. F1000Research. 8, 324. https://doi.org/10.12688/f1000research.18435.1
Huang B. (2009) MetaPocket: a meta approach to improve protein ligand binding site prediction. Omics. 13, 325–330. https://doi.org/10.1089/omi.2009.0045
Kumar S., Pandey A.K. (2013) Chemistry and biological activities of flavonoids: an overview. Sci. World J. 2013, 162750. https://doi.org/10.1155/2013/162750
Paczkowski J.E., Mukherjee S., McCready A.R., Cong J.P., Aquino C.J., Kim H., Henke‖ B.R., Smith Ch.D., Bassler B.L. (2017) Flavonoids suppress Pseudomonas aeruginosa virulence through allosteric inhibition of quorum-sensing receptors. J. Biol. Chem. 292, 4064–4076.https://doi.org/10.1074/jbc.M116.770552
Pawson A.J., Sharman J.L., Benson H.E., Faccenda E., Alexander S.P., Buneman O.P., Davenport A.P., McGrath J.C., Peters J.A., Southan Ch., Spedding M., Yu W., Harmar A.J., NC-IUPHAR. (2014) The IUPHAR/BPS Guide to PHARMACOLOGY: an expert-driven knowledgebase of drug targets and their ligands. Nucleic Acids Res. 42, D1098–106. https://doi.org/10.1093/nar/gkt1143
Lipinski C.A. (2004) Lead- and drug-like compounds: the rule-of-five revolution. Drug Discov. Today Technol. 1, 337–341. https://doi.org/10.1016/j.ddtec.2004.11.007
O’Boyle N.M., Banck M., James C.A., Morley C., Vandermeersch T., Hutchison G.R. (2011) Open Babel: an open chemical toolbox. J. Cheminform. 3, 33. https://doi.org/10.1186/1758-2946-3-33
Trott O., Olson A.J. (2010) AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading. J. Comput. Chem. 31, 455–461. https://doi.org/10.1002/jcc.21334
Li L., Chen R., Weng Z. (2003) RDOCK: refinement of rigid-body protein docking predictions. Proteins. 53, 693–707. https://doi.org/10.1002/prot.10460
Kolb P., Huang D., Dey F., Caflisch A. (2008) Discovery of kinase inhibitors by high-throughput docking and scoring based on a transferable linear interaction energy model. J. Med. Chem. 51, 1179–1188. https://doi.org/10.1021/jm070654j
Wang Z., Sun H., Yao X., Li D., Xu L., Li Y., Tian Sh., Hou T. (2016) Comprehensive evaluation of ten docking programs on a diverse set of protein-ligand complexes: the prediction accuracy of sampling power and scoring power. Phys. Chem. Chem. Phys. 18, 12964–12975. https://doi.org/10.1039/c6cp01555g
Pagadala N.S., Syed K., Tuszynski J. (2017) Software for molecular docking: a review. Biophys. Rev. 9, 91–102. https://doi.org/10.1007/s12551-016-0247-1
Jacob R.B., Andersen T., McDougal O.M. (2012) Accessible high-throughput virtual screening molecular docking software for students and educators. PLoS Comput. Biol. 8, e1002499. https://doi.org/10.1371/journal.pcbi.1002499
Morris G.M., Huey R., Lindstrom W., Sanner M.F., Belew R.K., Goodsell D.S., Olson A.J. (2009) AutoDock4 and AutoDockTools4: Automated docking with selective receptor flexibility. J. Comput. Chem. 30, 2785–2791. https://doi.org/10.1002/jcc.21256
Jaghoori M.M., Bleijlevens B., Olabarriaga S.D. (2016) 1001 Ways to run AutoDock Vina for virtual screening. J. Comput. Aided Mol. Des. 30, 237–249. https://doi.org/10.1007/s10822-016-9900-9
Daina A., Michielin O., Zoete V. (2017) SwissADME: a free web tool to evaluate pharmacokinetics, drug-likeness and medicinal chemistry friendliness of small molecules. Sci. Rep. 7, 42717. https://doi.org/10.1038/srep42717
Cheng F., Li W., Zhou Y., Shen J., Wu Z., Liu G., Lee P.W., Tang Y. (2012) admetSAR: a comprehensive source and free tool for assessment of chemical ADMET properties. J. Chem. Inf. Model. 52, 3099–3105. https://doi.org/10.1021/ci300367a
Filimonov D.A., Lagunin A.A., Gloriozova T.A., Rudik A.V., Druzhilovskii D.S., Pogodin P.V., Poroikov V.V. (2014) Prediction of the biological activity spectra of organic compounds using the pass online web resource. Chem. Heterocycl. Comp. 50, 444–457. https://doi.org/10.1007/s10593-014-1496-1
Müh U., Hare B.J., Duerkop B.A., Schuster M., Hanzelka B.L., Heim R., Olson E.R., Greenberg E.P. (2006) A structurally unrelated mimic of a Pseudomonas aeruginosa acyl-homoserine lactone quorum-sensing signal. Proc. Natl. Acad. Sci. USA. 103, 16948–16952. https://doi.org/10.1073/pnas.0608348103
O’Loughlin C.T., Miller L.C., Siryaporn A., Drescher K., Semmelhack M.F., Bassler B.L. (2013) A quorum-sensing inhibitor blocks Pseudomonas aeruginosa virulence and biofilm formation. Proc. Natl. Acad. Sci. USA. 110, 17981–17986. https://doi.org/10.1073/pnas.1316981110
Smith K.M., Bu Y., Suga H. (2003) Induction and inhibition of Pseudomonas aeruginosa quorum sensing by synthetic autoinducer analogs. Chem. Biol. 10, 81–89. https://doi.org/10.1016/S1074-5521(03)00002-4
Starkey M., Lepine F., Maura D., Bandyopadhaya A., Lesic B., He J., Kitao T., Righi V., Milot S., Tzika A., Rahme L. (2014) Identification of anti-virulence compounds that disrupt quorum-sensing regulated acute and persistent pathogenicity. PLoS Pathogens. 10, e1004321. https://doi.org/10.1371/journal.ppat.1004321
Yang L., Rybtke M.T., Jakobsen T.H., Hentzer M., Bjarnsholt T., Givskov M., Tolker-Nielsen T. (2009) Computer-aided identification of recognized drugs as Pseudomonas aeruginosa quorum-sensing inhibitors. Antimicrob. Agents Chemother. 53, 2432–2443. https://doi.org/10.1128/AAC.01283-08
Geske G.D., Wezeman R.J., Siegel A.P., Blackwell H.E. (2005) Small molecule inhibitors of bacterial quorum sensing and biofilm formation. J. Am. Chem. Soc. 127, 12762–12763. https://doi.org/10.1021/ja0530321
D’Almeida R.E., Molina R.D.I., Viola C.M., Luciardi M.C., Nieto Peñalver C., Bardón A., Arena M.E. (2017) Comparison of seven structurally related coumarins on the inhibition of Quorum sensing of Pseudomonas aeruginosa and Chromobacterium violaceum. Bioorg. Chem. 73, 37–42. https://doi.org/10.1016/j.bioorg.2017.05.011
Csizmadia F. (2000) JChem: Java applets and modules supporting chemical database handling from web browsers. J. Chem. Inf. Comput. Sci. 40, 323–324. https://doi.org/10.1021/ci9902696
Yuan S., Chan H.C.S., Hu Z. (2017) Using PyMOL as a platform for computational drug design. Wiley Interdisciplinary Reviews: Comput. Mol. Sci. 7, e1298. https://doi.org/10.1002/wcms.1298
Humphrey W., Dalke A., Schulten K. (1996) VMD: visual molecular dynamics. J. Mol. Graph. 14, 33–8, 27. https://doi.org/10.1016/0263-7855(96)00018-5
Laskowski R.A., Swindells M.B. (2011) LigPlot+: multiple ligand-protein interaction diagrams for drug discovery. J. Chem. Inf. Mod. 51, 2778–2786. https://doi.org/10.1021/ci200227u
Дополнительные материалы
- скачать ESM_1.tiff
- Fig. S1: 10 compounds that received high scores based on the three programs results
- скачать ESM_2.tiff
- Fig. S2: Hydrogen and hydrophobic interactions of the selected compounds with LBD of LasR obtained using Autodock Vina
- скачать ESM_3.tiff
- Fig. S3: Hydrogen and hydrophobic interactions of the selected compounds with LBD of LasR obtained using LeDock
- скачать ESM_4.tiff
- Fig. S4: Hydrogen and hydrophobic interactions of the selected compounds with LBD of LasR obtained using rDock
- скачать ESM_5.tiff
- Fig. S5: Similarity analysis of drugs
Инструменты
Молекулярная биология