Молекулярная биология, 2020, T. 54, № 1, стр. 29-50
Дизайн РНК-гидов для CRISPR/Cas редактирования геномов растений
Г. А. Геращенков a, *, Н. А. Рожнова a, Б. Р. Кулуев a, О. Ю. Кирьянова a, b, Г. Р. Гумерова a, А. В. Князев a, З. Р. Вершинина a, Е. В. Михайлова a, Д. А. Чемерис a, Р. Т. Матниязов a, Ан. Х. Баймиев a, И. М. Губайдуллин c, Ал. Х. Баймиев a, А. В. Чемерис a
a Институт биохимии и генетики Российской академии наук
450054 Уфа, Россия
b Уфимский государственный нефтяной технический университет
450062 Уфа, Россия
c Институт нефтехимии и катализа Российской академии наук
450075 Уфа, Россия
* E-mail: apomixis@anrb.ru
Поступила в редакцию 31.05.2019
После доработки 31.05.2019
Принята к публикации 08.07.2019
Аннотация
CRISPR/Cas-технология геномного редактирования ‒ мощный инструмент для внесения направленных изменений в ДНК различных организмов, включая растения. К важной составляющей этой технологии относится выбор мишеней в виде конкретных нуклеотидных последовательностей (протоспейсеров) в намеченных для редактирования генах, который сопряжен с дизайном РНК-гидов (gРНК) с использованием специализированных компьютерных программ. В представленном обзоре в общей сложности рассмотрено и упомянуто около 100 программ дизайна gРНК. Подробно рассмотрены программы дизайна gРНК, позволяющие вести поиск нецелевых сайтов редактирования в геномах организмов из царства растений. Приведены также перечни web-ресурсов и off-line программных продуктов, с помощью которых можно проводить дизайн gРНК разных организмов, но не позволяющих контролировать возникновение потенциальных нецелевых сайтов редактирования в геномах растений. Проведено сравнение используемых для геномного редактирования нуклеаз Cas9 и Cas12a и выявлены и аргументированы преимущества последней. Определенное внимание уделено набирающему силу редактированию отдельных азотистых оснований путем транзиций C→T и G→A без образования двухцепочечных разрывов. Указаны основные требования, предъявляемые к эффективным и высокоспецифичным gРНК. Отмечены перспективные направления совершенствования программ их дизайна. Обсуждается меньшая опасность нецелевого редактирования геномов растений в отличие от животных организмов. Рассмотрены особенности редактирования растительных геномов, в том числе не приводящие к созданию трансгенных растений.
ВВЕДЕНИЕ
CRISPR/Cas-технология геномного редактирования ‒ это мощный инструмент для внесения направленных изменений в ДНК различных организмов, включая растения. Эта технология состоит из нескольких этапов и каждый из них в той или иной степени определяет общий успех в достижении конечного результата. Среди этапов редактирования растительных геномов важная роль отводится способу доставки CRISPR/Cas-компонентов к месту редактирования, от чего зависит в том числе и генетический (генномодифицированный) статус создаваемого растения [1]. Также к ключевым моментам относится выбор мишеней ‒ конкретных нуклеотидных последовательностей (протоспейсеров) внутри намеченных для редактирования генов. Эта стадия сопряжена с дизайном РНК-гидов (gРНК), для чего прибегают к помощи специализированных компьютерных программ. На сегодняшний день разработано множество таких программ и именно они рассмотрены в представленном обзоре.
В ряде обзорных статей обсуждаются программы дизайна gРНК [2‒9]. Мы тоже опубликовали обзор на эту тему [10], где рассмотрели не только программы для дизайна gРНК, но и базы данных по этим компонентам CRISPR/Cas-редактирования, а также программы анализа редактированных геномов. Однако, благодаря бурному развитию генно-инженерных и биоинформационных технологий, эти обзоры уже не в полной мере отражают современные подходы к дизайну gРНК. Так, в последнее время в некоторые ранее разработанные программы внесены обновления, появилось немало новых ресурсов, так что назрела необходимость вновь обратить внимание на этот крайне важный этап геномного редактирования. В отличие от других обзорных публикаций, нами рассмотрены практически все существующие программы дизайна gРНК, нацеленные именно на редактирование геномов растений. В связи с тем, что эта статья рассчитана, в первую очередь, на конечных пользователей таких программных продуктов, алгоритмы, используемые разными разработчиками при написании самих программ, обсуждаются очень кратко. Программы анализа результатов геномного редактирования также останутся вне рассмотрения, так как это тема для отдельной статьи.
Что касается программ дизайна gРНК, предназначенных для редактирования геномов организмов не из царства растений, необходимо отметить, что большинство из них подходит для дизайна gРНК для любых нуклеотидных последовательностей ‒ независимо от принадлежности к тем или иным формам Жизни. Однако такие программы не позволяют оценивать наличие в геномах конкретных растений нецелевых мест редактирования. Так, например, программный продукт CRISPR-Cas9 guide RNA design checker (https://eu.idtdna.com/ site/order/designtool/index/crispr_sequence) известной фирмы “Integrated DNA Technologies” позволяет осуществлять дизайн gРНК и проверку их эффективности на предмет отсутствия нецелевых сайтов в геномах человека, мыши, крысы, аквариумной рыбки Danio rerio и нематоды. Для других видов можно выставить опцию “Other species”, где в скобках указывается “no off-target ana-lysis”. В этом случае у экспериментатора в качестве альтернативы есть возможность проверить как полное, так и неполное совпадение спейсерных последовательностей gРНК, подобранных этой или аналогичными программами, по базе данных GenBank с использованием сервиса Nucleotide BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi). Также заметим, что до сих пор известно не так много полных геномов растений, причем в основном это модельные объекты или возделываемые в промышленных масштабах культуры. В случае редактирования еще не секвенированного генома можно использовать любые программы для дизайна gРНК (их список приведен ниже с указанием URL-адресов), в том числе не предназначенные для выявления нецелевых мишеней в известных полных геномах растений.
Прежде чем перейти к изложению основного материала, необходимо коснуться вопроса обозначения применяемых для геномного редактирования Cas-нуклеаз и их мутантных форм, созданных генно-инженерным путем и иногда обозначаемых 2‒3-буквенными аббревиатурами латинских названий бактерий (табл. 1). В этой статье мы обозначаем их так же, как разработчики описываемых программ. Заметим, что под обозначением Cas9, где не оговорено происхождение, подразумевается конкретный фермент ‒ по умолчанию это нуклеаза дикого типа wtSpCas9 из Streptococcus pyogenes.
Таблица 1.
Нуклеазы системы CRISPR-Cas, наиболее часто используемые в геномном редактировании
| Полное название нуклеазы (организм) | Аббревиатура |
| CRISPR-associated protein 9 (Streptococcus pyogenes M1) | Cas9 |
| Дикий тип Cas9 (Streptococcus pyogenes M1) | wtSpCas9 |
| Модифицированный вариант VRER Cas9 (Streptococcus pyogenes) | VRER SpCas9 |
| Модифицированный вариант VQR Cas9 (Streptococcus pyogenes) | VQR SpCas9 |
| Один из мельчайших ортологов Cas9 (Campylobacter jejuni) | CjCas9 |
| Один из мельчайших ортологов Cas9 (Neisseria meningitidis) | NmCas9 |
| Cas9-нуклеаза с модифицированным кодоном для риса (Streptococcuspyogenes М1) | MmCas9 |
| CRISPR-associated protein 9 (Streptococcus thermophilus) | StCas9 |
| CRISPR-associated protein 9 (Staphylococcus aureus) | SaCas9 |
| Белок CRISPR системы из семейства Cas12a (Prevotella и Francisella 1) | Cpf1 (также известен как Cas12a) |
| As ‒ член семейства Cpf1 | AsCpf1 |
| Lb ‒ член семейства Cpf1 | LbCpf1 |
| Fn ‒ член семейства Cpf1 | FnCpf1 |
| Семейство нуклеаз Cas12b (Bacillus hisashii) | BhCas12b |
| Двудоменный искусственный белок класса 2 системы CRISPR-Cas с уникальными свойствами | C2c2 (Cas13a) |
| Рекомбинантный белок на основе нуклеазного домена FokI и неактивной Cas9 | FokI-dCas9 |
| Рекомбинантные белки на основе цитидин- или адениндезаминаз и мутантной формы Cas9 для однонуклеотидных замен в ДНК без образования двухцепочечных разрывов | Например, cytidine deaminase-Cas9 |
НУКЛЕАЗЫ CAS9/12A, ИСПОЛЬЗУЕМЫЕ В ГЕНОМНОМ РЕДАКТИРОВАНИИ
Подробное рассмотрение CRISPR/Cas-технологии геномного редактирования, включая детальный механизм действия используемых нуклеаз и различные достижения по улучшению хозяйственно ценных признаков растений, в задачи данной статьи не входят, тем более, что этим вопросам посвящено множество обзоров [11‒22]. Однако следует коротко остановиться на самих нуклеазах Cas9 и Cas12a (Cpf1). Основные различия между ними, как можно видеть из рис. 1, заключаются в расположении узнаваемой первой последовательности, соседствующей с протоспейсером (Protospacer Adjacent Motif, PAM), а также в сайтах расщепления цепей ДНК и структурной организации остова gРНК ‒ области CRISPR РНК (crРНК) и транс-активированной CRISPR РНК (tracrРНК), образующей с тем или иным апоферментом соответствующий рибонуклеопротеин (РНП).
Рис. 1.
Схемы взаимодействий рибонуклеопротеиновых комплексов, состоящих из полноразмерных РНК-гидов и нуклеаз Cas9 (а) или Cas12a (б), с редактируемой ДНК (см. пояснения в тексте). Ножницами показаны сайты расщепления нуклеазами.
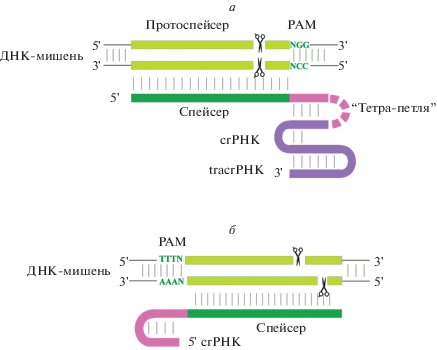
Классический вариант нуклеазы Cas9 узнает PAM‑последовательность NGG‑3′, а Cpf1-нуклеаза ‒ расположенную на противоположном конце протоспейсера AT‑богатую PAM‑последовательность 5′‑TTTN. Протяженность спейсерных последовательностей gРНК этих нуклеаз составляют 20 и 23 н. соответственно. Наиболее важную для узнавания протоспейсера часть спейсерной последовательности, прилегающую к PAM-участку, называют “seed sequence” (в данной статье она будет упоминаться как “якорная последовательность”, или “якорный участок”). Ее длина для нуклеаз из группы Cas9 составляет 8–12 н., а у Cas12a-нуклеаз она короче – всего 5–6 н. Cpf1-нуклеаза образует на некотором удалении от PAM-участка двухцепочечный разрыв с выступающими 4–5 н. на 5′‑концах против обычно образуемого нуклеазой Cas9 разрыва с тупыми концами в якорном участке, поблизости от PAM-последовательности. У Cpf1-нуклеазы имеется один каталитический домен RuvC, вносящий разрыв в обе цепи ДНК, тогда как Cas9-нуклеаза для расщепления комплементарных цепей ДНК использует два каталитических домена: RuvC и HNH, единичные мутации в которых (H840A в RuvC или D10A в HNH) превращают Cas9-нуклеазу в соответствующие nCas9-никазы, так как оставшийся неизмененным домен разрезает только одну цепь. При одновременном нарушении обоих каталитических доменов Cas9-нуклеаза, называемая уже dCas9-нуклеазой, теряет способность к разрезанию цепей ДНК. Однако, если подобную неактивную нуклеазу сшить с мономером рестрикционной эндонуклеазы FokI, образуется нуклеаза FokI-dCas9, сокращенно обозначаемая также как fCas9. При использовании двух таких химерных ферментов, с разными сайтами узнавания, и связывании их с редактируемой ДНК на нужном расстоянии возникает димер FokI, обладающий каталитической активностью. FokI-dCas9, наряду с nCas9-никазами, также находят применение в геномном редактировании, теоретически снижая число сайтов нецелевого редактирования за счет уменьшения вероятности нахождения двух протяженных последовательностей разных протоспейсеров на близком расстоянии.
Еще один тип химерных Cas-нуклеаз ‒ лишенные каталитической активности нуклеазы dCas или никазы nCas, несущие пришитые к ним цитидин- или адениндезаминазы (а также некоторые другие ферменты), позволяющие производить геномное редактирование отдельных азотистых оснований путем транзиций C→T и A→G без образования двухцепочечных разрывов молекул ДНК. Здесь необходимо заметить, что редактирование отдельных азотистых оснований позволяет осуществлять и нокаутное редактирование за счет формирования терминирующих кодонов (TAA, TAG, TGA) из кодонов, кодирующих глутамин (CAA и CAG), аргинин (CGA) и триптофан (TGG). Впрочем, возможно и обратное восстановление кодонов этих аминокислот из терминаторов транскрипции, что немаловажно для геномного редактирования в виде удаления ненужных мутаций.
У разных видов бактерий найдены Cas9/12a-нуклеазы, узнающие заметно отличающиеся PAM-последовательности. Немало создано подобных генетически модифированных Cas-нуклеаз, также узнающих разные PAM-участки, что, с одной стороны, расширяет возможности экспериментаторов при поиске в геномах подходящих протоспейсеров, а с другой – снижает риск возникновения нецелевых сайтов редактирования при использовании нуклеаз, узнающих более протяженные PAM-последовательности, которых априори меньше в редактируемых геномах.
Серьезное отличие нуклеазы Cpf1 от Cas9-нуклеаз заключается в том, что для ее функционирования не требуется tracrРНК, поэтому длина crРНК, достаточная для формирования активного комплекса с Cpf1-апоферментом, ограничена 42‒44 н. против обычно используемой для Cas9-нуклеазы полноразмерной gРНК длиной около 100 н., в состав которой входят crРНК, tracrРНК и искусственная тетрануклеотидная петля GAAA. Благодаря укороченному размеру остова gРНК для нуклеазы Cpf1 этот компонент РНП удобнее синтезировать химическим способом [23], что, правда, применимо и для нуклеазы Cas9 [24]. Это заметно упрощает задачу геномного редактирования, тем более что готовые ферменты уже выпускает ряд фирм. Обнаружено, что химически синтезированная gРНК лучше синтезированной энзиматически, так как последняя при геномном редактировании с использованием Cpf1-содержащих РНП приводила к необъяснимым инсерциям фрагментов ДНК [25]. Показано также, что специфичность действия Cas12a-нуклеазы более выражена, чем Cas9 [26], так что, в принципе, ее использование более предпочтительно при редактировании геномов, хотя пока по масштабу применения она заметно уступает Cas9-нуклеазам. Можно предположить, что высокая специфичность Cas12a-нуклеазы связана с тем, что она расщепляет ДНК относительно далеко от PAM-участка, поэтому следующие за этим процессы “плавления” цепей ДНК, а также продвижения так называемой R-петли, чувствительные к возможным неспариваниям и идущие постепенно, могут в какой-то момент, из-за наличия некомплементарных нуклеотидов, застопориться, что приведет к разрушению комплекса РНП с участком ДНК из-за неполностью совпадающего нецелевого сайта. В случае Cas9-нуклеазы разрезание обеих цепей ДНК происходит вблизи PAM-участка и теоретически оно может произойти до того, как сформируется полноценная R-петля в нецелевом сайте редактирования.
ОСНОВНЫЕ ТРЕБОВАНИЯ, ПРЕДЪЯВЛЯЕМЫЕ К ДИЗАЙНУ РНК-ГИДА
Для успешного геномного редактирования и исключения нецелевых мутаций при эффективном произведении целевых требуется выполнить качественный дизайн gРНК. Для этого необходим анализ всего генома конкретного организма (в тех случаях, когда его последовательность известна) на предмет наличия в нем сходных с редактируемым участком нуклеотидных последовательностей. В связи с тем, что неспаривание отдельных нуклеотидов и даже наличие небольших вставок/делеций ‒ инделов (как в ДНК, так и в РНК-цепях) ‒ теоретически может не быть препятствием для нецелевого редактирования, во внимание следует принимать не только 100%-ное совпадение нуклеотидных последовательностей gРНК с потенциальными протоспейсерами. Таким образом, в выбранных gРНК не должно быть полностью совпадающих нецелевых сайтов в редактируемом геноме, а наличие частично гомологичных должно быть сведено к минимуму. Особое внимание следует уделять якорной части, где замены нуклеотидов наиболее критичны. Важна и локализация потенциальных нецелевых сайтов: в кодирующих областях, интронах, промоторах, межгенных участках, – так как от этого зависит степень риска нежелательного редактирования. Для более эффективного нокаутирования конкретных генов предпочтительнее выбирать расположение редактируемого участка в первом экзоне (при его наличии) и на расстоянии 150–500 п.н. от стартового кодона. Для небольших белков этот диапазон составляет от 100 до 300 п.н. Стремясь исключить появление усеченного по C-концу, но остающегося функциональным белка и располагая место редактирования ближе к NH2-концу, нельзя забывать о возможном лидерном пептиде, нарушения в котором не затронут сам белок, но изменят его компартментализацию.
Еще в 2013 году анализ более 700 gРНК позволил выработать ряд рекомендаций по их подбору, снижающему возможность нецелевого редактирования и написать одну из первых программ для дизайна gРНК [27]. Главным требованием было исключение нахождения рядом с нецелевым участком, имеющим гомологию с целевым, PAM-последовательности, так как именно на нее первоначально ориентируется нуклеаза. Далее уже для участков с PAM-последовательностью было рекомендовано исключить полную гомологию нецелевого сайта с целевым и допускать наличие в нем не менее 3 нуклеотидных замен, причем минимум две из них должны находиться в области, прилегающей к PAM-последовательности. Высказывалось пожелание, чтобы такими неспариваниями характеризовались соседние нуклеотиды или отстоящие бы друг от друга не более чем на 4 позиции. При анализе 1841 варианта gРНК Doench и др. [28] продемонстрировали предпочтительное нахождение тех или иных азотистых оснований в спейсерной последовательности, что позволило создать предиктивную модель их активности и выработать определенные правила, названные позже как Rule Set 1. Так, было обнаружено, что Cas9-нуклеаза предпочитает, чтобы вариабельный нуклеотид PAM-последовательности был цитозином, но он крайне нежелателен в соседствующем с ней участке ДНК, который взаимодействует с gРНК ‒ там предпочтителен гуанин. Продолжив изучение целевого и нецелевого редактирования gРНК, Doench и др. [29] разработали усовершенствованные правила, Rule Set 2, послужившие основой для целого ряда программ дизайна gРНК. В дальнейшем, при усовершенствовании программных продуктов дизайна gРНК и создании новых, стали активно применять алгоритмы, основанные на технологиях глубокого машинного обучения, позволяющие вести улучшенный поиск уникальных протоспейсеров с уменьшенной вероятностью возникновения нецелевых сайтов редактирования [30‒36].
Как будет видно из дальнейшего изложения, программы дизайна gРНК, предназначенные для редактирования геномов растительных организмов, заметно отличаются друг от друга, однако ряд моментов носят общий характер. К ним относится протяженность спейсерных областей gРНК, которая, хотя и варьирует при дизайне в пределах от 15 до 25 н., характеризуется предустановленными во многих программах длинами в 20 и 23 н. (по умолчанию) для нуклеаз Cas9 и Cas12a (Cpf1) соответственно. Считается, что использование gРНК с укороченным спейсером несколько снижает неспецифичное редактирование, однако чрезмерное укорочение может обернуться потерей каталитической активности Cas-нуклеаз вообще. Немаловажное значение имеет и нуклеотидный состав протоспейсеров. Не рекомендуется выбирать подобные участки как с высоким, так и с низким GC-содержанием ‒ желательно укладываться в границы от 40 до 60%, хотя интервал 20‒80% считается приемлемыми. Необходимо исключать гомополимерные участки из четырех и более одинаковых нуклеотидов, а присутствие в протоспейсере четырех тиминов подряд (или даже трех) необходимо исключить в случае наработки gРНК путем транскрипции, так как эта последовательность служит терминатором этого процесса. Также необходимо учитывать системы экспрессии, применяемые для наработки gРНК и требующие для повышения эффективности транскрипции наличия на 5′-конце двух гуанинов в случае использования РНК-полимеразы под контролем Т7-промотора или одного гуанина для промотора U6. Нуклеотидная последовательность выбранного участка не должна вызывать возникновение неправильной пространственной структуры всей молекулы gРНК. При подборе протоспейсеров в месте двухцепочечного разрыва и последующей репарации желательно предусматривать присутствие сайтов эндонуклеаз рестрикции, которые при расщеплении ампликонов, образованных с помощью ПЦР с фланкирующими праймерами, служат контролем произошедшего геномного редактирования, предшествуя секвенированию данного участка генома. При проведении экспериментов по CRISPR/Cas-редактированию в нокин-вариантах подобраннные протоспейсеры, в том числе с небольшим числом замен, должны отсутствовать в донорной ДНК.
Недавно при анализе большого числа “неуспешных” (не самых эффективных) gРНК показано, что большинство из них содержат в проксимальной части спейсерных последовательностей среди четырех непосредственно прилегающих к PAM-участку нуклеотидов мотивы TT и GCC, что снижает эффективность редактирования на порядок [37]. Еще ранее разработчики программы WU-CRISPR для дизайна gРНК сообщали [38], что, если 4 н. спейсерной последовательности, прилегающие к PAM-участку, обогащены пиримидинами, эффективность геномного редактирования с участием таких gРНК резко снижается. Причина состоит в том, что эти основания могут спариваться с определенными нуклеотидами остова gРНК, удлиняя тем самым одну из ее петлевых структур и исключая тем самым важную часть якорной последовательности из взаимодействия с сайтом редактирования.
ОСОБЕННОСТИ РЕДАКТИРОВАНИЯ ГЕНОМОВ ОРГАНИЗМОВ ИЗ ЦАРСТВА РАСТЕНИЙ
При редактировании (в нокаутном варианте) растительных геномов появление помимо целевых мутаций иных мест, подвергнувшихся редактированию, не так катастрофично, как для животных организмов. Причина тому ‒ крупные размеры геномов многих растений. Они содержат значительную долю некодирующих участков, часто носят полиплоидный характер, включая и так называемые палеополиплоиды (теперь считающиеся диплоидными формами), копийность многих генов увеличена, нередко встречается эндополиплоидное состояние отдельных клеток и тканей, вызванное эндоредупликацией. Еще одна причина сниженной опасности нецелевого редактирования геномов растений заключается в возможности для большинства видов относительно легкой сегрегации нежелательных признаков в ряду поколений, а также проведение возвратных и насыщающих скрещиваний. Наконец, известны последствия для растений и спонтанного мутагенеза, происходящего под воздействием радиации или химических агентов, в результате чего происходит множество мутаций в непредсказуемых местах генома; при этом отдельные растения не только выживают, но иногда приобретают и полезные признаки.
Фактически нецелевое геномное CRISPR/Cas-редактирование, хотя и не совсем случайное, можно считать мало отличающимся от химического или радиационного мутагенеза. Теоретически можно представить внесение многочисленных мутаций в различные позиции генома растений, например, с помощью неких вырожденных gРНК (аналогично вырожденным праймерам, отжигающимся на множестве мест [39]), способным произвести редактирование такого же множества протоспейсеров в ДНК растения. Это позволит получать растения с улучшенными полезными признаками. Возможно, вместо обычных Cas-нуклеаз, приводящих к образованию инделов, для такой цели более удобным окажется относительно “мягкое” редактирование отдельных азотистых оснований с помощью дезаминаз, сшитых с неактивными Cas-нуклеазами. Такие растения теоретически не будут относиться к генетически модифицированным ввиду нецелевого воздействия ‒ аналогично подвергшимся мутагенезу при обработке химическими агентами или ионизирующим излучением. Однако трудоемкость, длительность, да и стоимость такого подхода для множественного и практически случайного редактирования значительно превышают таковые при обработке семян испытанными мутагенами, поэтому его применение для получения мутантных форм растений с улучшенными характеристиками нерентабельно.
Показательно название одной из недавно опубликованных статей “CRISPR/Cas precision: do we need to worry about off-targeting in plants?”, в котором Hahn & Nekrasov прямо задаются вопросом, надо ли вообще беспокоиться о нецелевых местах CRISPR/Cas-редактирования в геномах растений [40]. Они ссылаются на крупномасштабное исследование других авторов [41], не обнаруживших массовых мутаций после геномного редактирования. Так, было проведено полногеномное секвенирование 69 растений риса, из которых 34 редактировали нуклеазой Cas9, 15 ‒ нуклеазой Cpf1 и 20 использовали в качестве контроля [41]. В результате обнаружили, что практически все мутации (от 102 до 148 однонуклеотидных замен и от 32 до 83 инделов на растение) возникли естественным путем и только одна мутация оказалась следствием нецелевого редактирования Cas9-нуклеазой, для которой было использовано 12 gРНК. Кроме того, три gРНК были предназначены для Cpf1-нуклеазы, нецелевого редактирования при использовании которой не обнаружили вообще. Это свидетельствует, в первую очередь, о хорошо подобранных gРНК, а также о том, что нецелевое редактирование растительных геномов происходит реже, чем появляются естественные мутации. Например, для арабидопсиса это преимущественно транзиции и небольшие инделы, встречающиеся в среднем с частотой около 10–9 на сайт за одно поколение [42]. Wolt и соавт. [43] проанализировали данные по нескольким видам растений и пришли к выводу, что в большинстве случаев редактирование с помощью CRISPR/Cas-систем, как работающих транзиентно, так и после их внедрения в геномы растений, было целевым. Wong и др. [38] также отмечали, что нецелевое редактирование, на самом деле, имеет гораздо меньший масштаб, чем предполагалось раньше, причем для разных организмов [38].
Здесь необходимо заметить, что эффективность (полнота) геномного редактирования и его специфичность (воздействие преимущественно или исключительно на целевые сайты) фактически противостоят друг другу. Так, эффективность редактирования намеченных участков генома будет тем выше, чем дольше в клетке работает РНП на основе той или иной Cas-нуклеазы и gРНК, но при этом увеличивается вероятность нецелевого редактирования. Продолжительность работы РНП зависит от выбранной процедуры доставки, которая для растений может как обеспечить встраивание в геном всех или только части CRISPR/Cas-компонентов (постоянное воздействие) или привести к их транзиентной экспрессии (пролонгированное воздействие), так и произвести только кратковременное воздействие ‒ когда доставляется непросредственно готовый РНП-комплекс. В последнем случае имеет место так называемая бесплазмидная доставка, дополнительным преимуществом которой, помимо технической быстроты процедуры редактирования, служит невозможность встраивания чужеродной ДНК (при нокаутном редактировании) в геном растения, которое принципиально не может стать в этом случае трансгенным. И подобные подходы в последние годы получают свое развитие [44‒47].
С учетом увеличенной копийности отдельных генов, вариаций нуклеотидных последовательностей, а также полиплоидной природы у многих видов растений для более эффективного нокаутного редактирования следует подбирать несколько gРНК на каждый ген ‒ в расчете на то, что, по крайней мере, в разных копиях гена в редактировании будет задействована не одна, так другая из этих gРНК.
КОМПЬЮТЕРНЫЕ ПРОГРАММЫ И WEB-РЕСУРСЫ ДЛЯ ДИЗАЙНА РНК-ГИДА
При дизайне gРНК к важным моментам относится прогнозирование наличия нецелевых сайтов при CRISPR/Cas-редактировании геномов. Для этого программы дизайна gРНК должны иметь доступ к соответствующим геномным последовательностям. В табл. 2 представлена краткая информация по некоторым программным продуктам для дизайна gРНК, содержащая также данные по геномам, в том числе из царства растений, используемым для анализа. Следует заметить, что для отдельных программ по просьбе пользователей разработчики готовы включить дополнительные геномы.
Таблица 2.
Программы дизайна РНК-гидов с анализом потенциальных нецелевых сайтов в геномах растений
a Для многих программ периодически производится их обновление и при этом, как правило, увеличивается список доступных для анализа геномов, поэтому к моменту выхода данной статьи указанные здесь числа, актуальные на начало апреля 2019 года, могут служить лишь приблизительным ориентиром. b Число доступных геномов соответствует таковым, находящимся в различных разделах базы данных Ensembl, например в http://plants.ensembl.org/index.html, для растений. c Есть возможность добавить геном, связавшись с разработчиками. d Не путать с базой данных CrisprGE (http://crdd.osdd.net/servers/crisprge/) и программой ge-CRISPR (http://bioinfo.imtech.res.in/manojk/gecrispr/index.php).
В табл. 3 суммированы основные характеристики (более 30) описанных ниже программ дизайна gРНК, сгруппированные в блоки (оттененные разным фоном), касающиеся Cas-нуклеаз (A–F), протоспейсеров (G–K), вариантов ввода предназначенных для редактирования последовательностей ДНК/РНК (L–N), а также особенностей выдаваемых результатов (O–W) и несущие дополнительную информацию (X–Z). Помимо этой информации в табл. 3 представлены сведения по возможности работы некоторых программ в автономном режиме и валидации ранее подобранных gРНК, в том числе иными программами дизайна. Некоторые программы обеспечивают еще больше вариаций анализа подобранных gРНК, чем отражено в этой таблице, на них мы остановимся дальше, при детальном описании.
Таблица 3.
Основные характеристики некоторых программ дизайна РНК-гидов для CRISPR/Cas-редактирования геномов организмов из царства растений*
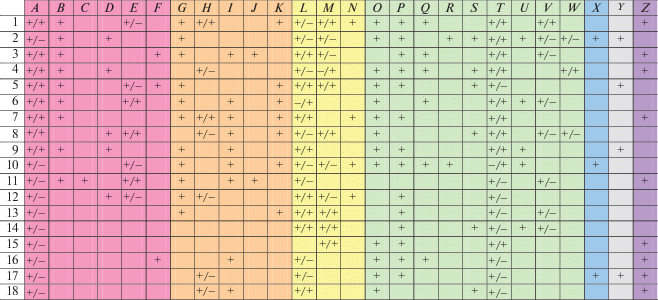
* Обозначения: 1 ‒ CRISPR‑P 2.0\CRISPR Local; 2 ‒ CRISPR‑GE; 3 ‒ CRISPR RGEN Tools; 4 ‒ CRISPOR; 5 ‒ Benchling; 6 ‒ DESKGEN; 7 ‒ CCTop; 8 ‒ CHOPCHOP; 9 ‒ Breaking Cas; 10 ‒ E‑CRISP; 11 ‒ CT‑Finder\CRISPR‑DT\CRISPR‑RT; 12 ‒ CRISPR MultiTargeter; 13 ‒ CGAT; 14 ‒ CRISPRdirect; 15 ‒ CRISPR‑PLANT; 16 ‒ GT‑Scan; 17 ‒ CRISTA; 18 ‒ PhytoCRISP‑Ex. A – wtSpCas9 нуклеаза/ортологи и мутанты Cas9; B – Cpf1 (Cas12a); C – C2c2 (Cas13a); D – Custom PAM; E – никазы/FokI-Cas9; F – нуклеаза-деаминаза; G – длина (прото)спейсера; H – 5′‑конец gРНК/промотор для транскрипции in vivo; I – неспаривания; J – инделы в спейсерах и протоспейсерах; K – GC-состав протоспейсеров; L – ввод ДНК через буфер обмена/в виде файла; M – ввод конкретных генов с помощью идентификатора (наименование гена, Accession number)/ввод ДНК с помощью геномных координат; N – множественные последовательности; O – ранжирование gРНК; P – нецелевые сайты; Q – микрогомология; R ‒ фланкирующие участки; S – сайты рестрикции; T – обе цепи ДНК/область редактирования (экзон, интрон, межгенный участок); U – присутствие TTT(T); V – GC-состав протоспейсеров/вторичная структура gРНК (константной и вариабельной частей); W – олигонуклеотиды и праймеры для клонирования/ПЦР детекции; X ‒ валидация sgRNA; Y – демоверсии; Z – автономное использование.
Общий порядок дизайна gРНК для большинства программ в целом схож. Для тех программных продуктов, для которых возможны определенные опции, поиск протоспейсеров заключается в выборе конкретных Cas-нуклеаз с указанием особенностей спейсерной области gРНК (длины, неспариваний, инделов, GC-состава и др.). Потенциальные протоспейсеры выявляют в намечаемой для редактирования последовательности генома, которую берут в анализ разными способами: ввод нуклеотидной последовательности через буфер обмена в FASTA-формате или в текстовом формате; загрузка файла; указание конкретных генов с помощью идентификатора (наименование гена, Accession number), а также с использованием геномных координат. Ряд программ допускает ввод множественных последовательностей для дизайна gРНК, пригодных для редактирования одинаковых консервативных участков геномов разных (эволюционно близких) видов. После завершения ввода редактируемой последовательности, выбора Cas-нуклеаз и прочих параметров, включая опциональные, запускают поиск, по завершении которого программы выдают все потенциальные протоспейсеры, ранжированные или не ранжированные по их пригодности, и другую информацию.
Некоторые из колонок табл. 3, несущих схожую или сопряженную информацию, дублируют друг друга. Так, GC-составу протоспейсеров отведены две колонки (K и V), так как в одних программах он задается на стадии установки параметров поиска, а в других – лишь сопровождает уже выбранные последовательности протоспейсеров. Таким образом, эти сведения относятся к разным блокам, и объединять их в одной колонке (даже в двойной) было нелогично. Мы не стали перегружать таблицу сведениями о протяженности анализируемых разными программами нуклеотидных последовательностей на предмет поиска в них протоспейсеров; но, ввиду важности этого параметра, сочли необходимым привести эти сведения вне таблицы. Так, разброс длин таких участков ДНК для разных программ варьирует в широких пределах, указанных здесь как число нуклеотидов по нарастающей: CCTop – 500; E‑CRISP – 500; CRISPR RGEN Tools – 1000; CRISPOR – 2000; CRISPR‑P 2.0 – 5000; CRISPR‑GE – 10 000; CRISPRdirect – 10 000; DESKGEN – 10 000; CHOPCHOP – 20 000; Breaking Cas – 20 000; CRISPR‑PLANT – 30 000; CRISPR MultiTargeter – 50 000; PhytoCRISP‑Ex – 2 000 000. Для ряда программ (Benchling, CGAT, GT‑Scan, CRISTA, CT‑Finder/CRISPR‑DT/CRISPR‑RT) такая информация, к сожалению, отсутствует. Следует признать, что для большинства случаев направленного нокаутного редактирования какого-либо гена для выбора конкретного места (протоспейсера) вполне достаточным может оказаться анализ 2000 и даже 1000 н., тогда как 500 н. иногда может оказаться мало ввиду возможного отсутствия в этом фрагменте гена наиболее эффективных мест для связывания gРНК. В таком случае потребуется повторный анализ еще одного участка такой же протяженности.
Как можно видеть из табл. 3, верхние строки занимают программы, которые обладают большим функционалом и дают пользователям больше информации для улучшенного дизайна gРНК. В таком порядке они и будут рассмотрены далее более подробно.
Web-ресурс CRISPR-P стал первой специализированной программой для дизайна gРНК, рассчитанной на редактирование геномов растений [60]. Позже, сохранив интерфейс прежней версии, эту программу усовершенствовали, обозначив как CRISPR-P 2.0 [61]. Оценку целевых и нецелевых сайтов в геномах стали проводить, исходя из последних воззрений на их взаимодействия с SpCas9 и другими Cas-нуклеазами, число которых заметно возросло. Так, программа CRISPR-P 2.0 предполагает подбор протоспейсеров для их расщепления, помимо нуклеазы SpCas9 (сайты узнавания NGG и NRG), следующими ферментами (полные названия приведены в табл. 1): StCas9 (NNAGAAW), MmCas9 (NNNNGMTT), SaCas9 (NNGRRT), ‒ а также нуклеазами Cas12a: AsCpf1 (TTTV), LbCpf1 (TTTV), FnCpf1 (TTN) ‒ и другими. Это расширяет возможности экспериментаторов по подбору оптимальных мишеней в редактируемых геномах растений. Результаты поиска эта программа выдает в виде расположения на линейном графике для обеих цепей ДНК мест взаимодействия спейсеров с комплементарными цепями протоспейсеров, окрашеными в три разных цвета в соответствии с их релевантностью. Табличная форма с результатами работы программы помимо последовательностей протоспейсеров включает информацию по их GC-составу, положению, оценочным характеристикам подобранных gРНК для целевых сайтов, которые могут быть ранжированы по любому из этих параметров. Например, можно выбрать варианты, указав тип первого нуклеотида на 5′-конце – G или A. При наведении курсора мыши на тот или иной протоспейсер в левой части таблицы можно просмотреть информацию о числе присущих ему нецелевых сайтов в редактируемом геноме, ранжированных по их специфичности (приводятся первые 20), где красным шрифтом показаны неспаривающиеся нуклеотиды. Предусмотрена возможность получения большей информации о протоспейсерах в виде сведений о микрогомологии соседних участков с местом разреза ДНК Cas-нуклеазой, а также в отдельном окне можно увидеть изображение предполагаемой вторичной структуры gРНК. Все это позволяет считать программу CRISPR-P 2.0, обладающую широкими возможностями, одной из наиболее перспективных для пользователей. Определенным развитием программы CRISPR-P 2.0 стала разработка автономно работающей версии CRISPR-Local [62], в которой уменьшен ассортимент нуклеаз и одновременно до 71 расширена база референсных геномов ‒ для их редактирования с помощью нуклеаз Cas9 (NGG), Cpf1 (TTTV и TTV) уже подобраны все возможные gРНК, доступные для скачивания в архивированном виде.
Пакет прикладных программ CRISPR-GE (Genome Editing) [57], состоит из следующих инструментов: targetDesign, offTarget, primerDesign-V, primerDesign-A и др. Дизайн gРНК с помощью программы targetDesign начинают с выбора нуклеазы и характерной для нее PAM-последовательности, среди которых SpCas9 (NGG), FnCpf1 (TTN) и AsCpf1 (TTTN), при этом можно указать иной тип PAM-участка. Затем следует определиться с длиной протоспейсера и выбрать конкретный геном из имеющегося списка, при этом допускается не указывать никакого генома, выбрав вариант “None”. Результаты поиска программа выдает в табличной форме, уведомляя о возможности демонстрации сайтов рестрикционных эндонуклеаз, но можно и скрыть эту информацию, занимающую, как правило, довольно много места. В первой колонке таблицы можно отметить соответствующие ячейки и для этих gРНК будет в дальнейшем подобран комплект праймеров для конструирования нужной последовательности ДНК для ее клонирования в подходящем векторе. В последующих столбцах сообщается о неудачных gРНК ‒ они отмечены разным числом восклицательных знаков. Оценку вероятности нецелевого редактирования проводят с помощью инструмента offTarget, а для дизайна праймеров используют программу primerDesign и две ее подпрограммы: primerDesign-V и primerDesign-A. Первая отвечает за дизайн олигонуклеотидов для клонирования, а вторая – за амплификацию целевых участков, что необходимо для подтверждения факта редактирования, но это уже другой этап геномного редактирования, который остается за пределами рассмотрения этой статьи. Программа offTarget может работать независимо от программы targetDesign и анализировать введенные в нее ранее найденные последовательности gРНК на предмет возможного редактирования геномов с их участием в нецелевых местах. Для такого анализа необходимо выбрать тип нуклеазы и конкретный геном, после чего следует ввести спейсерную последовательность gРНК, применяя для последующих gРНК опцию “Insert”.
Создан специализированный портал, также содержащий пакет прикладных программ CRISPR RGEN Tools, где представлены несколько программных продуктов, используемых в экспериментах по CRISPR/Cas-редактированию: Cas-OFFinder [65], Microhomology-Predictor [66], Cas-Designer [67], BE-Designer [69], – а также некоторые другие программы, использующиеся при анализе произошедшего редактирования геномов и поэтому не рассматриваемые здесь. Кроме того, этот web-портал содержит две базы данных: Cas-Database [67] и Cpf1-Database [70], ‒ в которых содержится информация по нескольким десяткам тысяч ранее подобранных gРНК для пяти видов растений (Arabidopsis thaliana, Musa acuminata, Vitis vinifera, Solanum lycopersicum, Glycine max).
Программа Cas-Designer в анализируемой последовательности находит все возможные протоспейсеры. Предлагается широкий выбор нуклеаз, в том числе их мутантных форм с отличающимися PAM-последовательностями: SpCas9 (NGG для целевых сайтов и NRG для нецелевых сайтов), StCas9 (NNAGAAW), NmCas9 (NNNNGMTT), SaCas9 (NNGRRT), CjCas9 (NNNVRYAC), CjCas9 (NNNNRYAC), wtSpCas9 (NNGTGA), VRER SpCas9 (NGCG), VQR SpCas9 (NGA), AsCpf1 (TTTN), AsCpf1 (TTTV), FnCpf1 (TTN), FnCpf1 (KYTV), BhCas12b (TTN) и др. При поиске протоспейсеров можно отметить, нужно ли допускать вероятность однонуклеотидных инделов, при этом будет предупреждение об увеличении времени поиска, хотя результаты поиска без учета этой опции и даже с ее установкой выдаются независимо и быстро. Остальные параметры задаются по умолчанию. Для поиска необходимо выбрать тот или иной геном из разделенного на группы предлагаемого списка, включающего и группу “Plant”. Самостоятельно менять дополнительные опции программы Cas-Designer можно, установив ее на персональный компьютер.
Программа Microhomology-Predictor анализирует микрогомологию нуклеотидных последовательностей в месте образования двуцепочных разрывов, что способствует прогнозированию типа объединения концов при репарации по типу негомологичного соединения концов (nonhomo-logous end-joining, NHEJ) и опосредованного микрогомологией соединения концов (microhomology-mediated end-joining, MMEJ). Результаты поиска отображаются в виде таблицы, содержащей различную информацию, которую можно сужать, меняя ряд параметров.
Для обнаружения возможных нецелевых мест в исследуемых геномах предназначена программа Cas-OFFinder, которая может работать независимо, ведя поиск нецелевых мест в геномах разных организмов для ранее уже подобранных gРНК; при этом у экспериментатора появляется больше возможностей для такого анализа. Необходимо ввести последовательности подобранных gРНК и задать допустимое число неспариваний нуклеотидов, число инделов (как на цепи ДНК, так и РНК), после чего выбрать анализируемый геном из предлагаемого сгруппированного списка, как и при использовании Cas-Designer.
Программа BE-Designer позволяет осуществлять дизайн gРНК для редактирования отдельных азотистых оснований. Работа этой программы в целом схожа с дизайном gРНК с помощью программы Cas-Designer, но есть и отличия. Так, выбор PAM-участков и соответствующим им нуклеаз уже несколько сужен и необходимо выбрать размер “окна” для поиска в нем мест редактирования и желаемый тип изменения конкретного нуклеотида (C→T или A→G). Результаты поиска отображаются в таблице, где изменяемый нуклеотид выделен цветом.
Программа CRISPOR [54, 55] также представляет собой довольно удобный в плане использования продукт, содержащий подробное руководство для пользователя. С января 2019 года функционирует обновленная версия CRISPOR V4.7, с помощью которой в три шага предлагается выбрать оптимальные протоспейсеры. На первом шаге вводится анализируемая последовательность. На втором предлагается выбрать конкретный геном, в котором будет проводиться поиск нецелевых сайтов, при этом какой-либо геном можно вообще не выбирать (вариант “No Genome”). На третьем шаге необходимо выбрать нуклеазу Cas9 из предлагаемых 17 вариантов, включающих кроме стандартной SpCas9 ее различные мутантные формы с измененными PAM-участками, а также другие ортологичные нуклеазы этого типа и их мутантные формы. В дополнении к ним можно выбрать нуклеазу Cas12a ‒ AsCpf1 с двумя вариантами PAM-участков. Результаты поиска выдаются в графическом и табличном форматах. Приводится анализируемая последовательность, под которой указаны потенциальные места редактирования, выделенные разными цветами для высокой, средней и низкой специфичности. В итоговой таблице содержится информация о последовательностях протоспейсеров и их позициях с указанием цепей ДНК, а также ссылка “Cloning/ PCR primers”, позволяющая ознакомиться с необходимой информацией о клонировании и экспрессии конкретной gРНК и праймерах для детекции. Предлагаемые gРНК ранжированы по специфичности и приведена оценка их эффективности по двум разным алгоритмам. Отдельное внимание уделено микрогомологии. Нецелевые сайты перечислены с учетом числа неспариваний. Следует заметить, что ссылка на результаты поиска может быть отправлена коллегам для ознакомления, а разработчики обещают, что смогут хранить все выполненные анализы в течение, по крайней мере, одного года.
Облачный сервис Benchling бесплатен для академических организаций (https://benchling.com/academic), но необходима регистрация. Данный ресурс содержит целый ряд биоинформатических программ, одна из которых позволяет планировать эксперименты по CRISPR/Cas-редактированию геномов и осуществлять дизайн gРНК под относительно широкий спектр нуклеаз Cas9 и Cas12a, узнающих следующие PAM-участки: NGG, NAG, NNNNGATT, NNAGAAW, NAAAAC, NNGRR, NNGRRT и TTTN. Необходимо выбрать тип редактирования – с помощью обычной одиночной Cas-нуклеазы, никаз или редактирование отдельных азотистых оснований без образования двухцепочечных разрывов за счет неактивной Cas-нуклеазы, сшитой с соответствующей дезаминазой. Есть ряд дополнительных опций, среди которых параметры для демонстрации целевых и нецелевых сайтов, нуклеотидный состав gРНК. Результаты поиска отображаются в графическом виде, где приводится вся анализируемая нуклеотидная последовательность с сайтами эндонуклеаз рестрикции, а также табличные данные по каждому протоспейсеру с указанием мест локализации, цепей ДНК и оценок для эффективности и специфичности целевого и нецелевого редактирования. Отличительной особенностью выдаваемой информации при редактировании отдельных азотистых оснований служит оценка эффективности этого процесса и выделение цветом изменяемых нуклеотидов в протоспейсерах.
Облачный сервис DESKGEN фирмы “Desktop Genetics” бесплатно доступен для некоммерческих организаций, при этом требуется регистрация, после которой становится возможна работа с такими программами, как Knockout, Knockin, Guide Picker [73] и Genome Editor. В настоящее время для анализа доступно 26 геномов, включая шесть растительных, при этом программа Guide Picker работает только с геномами человека и мыши. Схема действий довольно стандартна: сначала предстоит выбрать референсный геном, затем конкретный ген, тип нуклеазы и длину вариабельного участка gРНК. Не рекомендуется анализировать последовательность ДНК длиной более 10 т.п.н. из-за замедления работы программы. Возможен дизайн gРНК для образования комплексов с 10 различными нуклеазами, включая три из группы Cpf1, а также каталитически неактивную нуклеазу SpdCas9-FokI, хотя программа настроена на использование классической Cas9-нуклеазы и выдаваемые результаты по остальным ферментам могут быть не совсем корректны. В отличие от некоторых других программ, сервис DESKGEN отмечает последовательности, представленные даже не четырьмя тиминами, а только тремя (TTT), как нежелательные.
Программа CCTop (CRISPR/Cas9 Target online predictor) [49] позволяет осуществлять дизайн gРНК и искать для них целевые и нецелевые сайты. В программе предоставлен широкий выбор нуклеаз Cas9 из разных видов бактерий (Streptococcus pyogenes, Staphylococcus aureus, Neisseria meningitidis, Streptococcus thermophilus, Treponema denticola, Campylobacter jejuni), включая мутантные варианты, а также Cpf1 из Acadaminoccus/Lachnospiraceae и Francisella novicida. Особенность этой программы ‒ возможность добавления дополнительных нуклеотидов к спейсерной области в виде одного или двух гуанинов на 5′-конце для обеспечения улучшенной транскрипции in vitro. Также можно указывать желательные два нуклеотида, прилегающие к PAM-участку, ‒ во избежание их спаривания с остовом gРНК. Результаты дизайна gРНК выдаются в графическом и табличном виде, причем ранжирование сейчас осуществляется с помощью алгоритма прогнозирования CRISPRater [32]. РНК-гиды в экзонах, интронах и межгенных участках выделены разным цветом.
Web-ресурс CHOPCHOP [51, 52] предназначен для поиска gРНК, работающих в комплексе с Cas9-нуклеазой, ее никазными вариантами и с нуклеазой Cpf1. Имеется возможность задать поиск только в конкретной кодирующей области или во всех экзонах, в сайтах сплайсинга, 5'- или 3'-некодирующих областях, а также в промоторной последовательности, длину которой в широких пределах задают отдельно. Также можно вести поиск сайтов рестрикции в редактируемом участке, для чего соответствующие ферменты выбирают через указание производителя (предлагается несколько фирм). После завершения поиска выдается масштабируемое цветное графическое изображение и интерактивная табличная форма с ранжированными последовательностями мишеней для gРНК, включая их GC-состав, указание цепей ДНК и некоторую другую информацию.
Web-сервер Breaking Cas [48], наряду с некоторыми другими программами дизайна gРНК, позволяет искать нецелевые сайты во всех известных к настоящему времени эукариотических геномах, доступных из базы данных Ensembl (http://ensemblgenomes.org), содержащей по состоянию на январь 2019 года 1335 геномов. Дизайн gРНК с помощью web-сервера Breaking Cas начинают с выбора конкретного генома из выпадающего алфавитного списка или в строке ввода набирают несколько первых букв тривиального названия организма или его латинского обозначения; причем неважно, принадлежат они роду или виду. Необходимо указать тип нуклеазы из четырех вариантов Cas9 и трех вариантов Cpf1 с предустановленными значениями длин спейсерных последовательностей и числа неспариваний. Кроме этих ферментов допустим выбор некоей нуклеазы “Custom”, сопровождаемый введением с клавиатуры соответствующих ей PAM-последовательности и длины спейсерной последовательности gРНК, а также выбором числа допустимых неспариваний, что в целом дает широкие возможности целевого редактирования конкретных мест в выбранном геноме. Позволяется менять значимость каждого неспаривания в спейсерной последовательности в зависимости от его расстояния от PAM-участка для ранжирования предлагаемых gРНК.
Написанная в 2014 году программа E-CRISP [74] претерпела ряд усовершенствований, включая увеличение числа анализируемых геномов с 12 до 55, из которых более десятка ‒ растительные. В модуле Design задается уровень поиска, исходя из трех режимов: смягченный, средний или строгий, ‒ причем в этих случаях во внимание можно приниматья или не принимать интроны, CpG-островки, 5′-нетранслируемые последовательности. Необходимо указать, требуется ли единичная gРНК или ее парные варианты для использования никазы или для нокин-экспериментов. Для уточнения поиска дозволяется указывать конкретные опции из большого их числа. В модуле MultiCRISP предполагается выравнивание нескольких родственных последовательностей. В модуле Evaluation можно подвергнуть ревизии ранее найденные gРНК на предмет нахождения для них в тех же геномах нецелевых сайтов, причем позволяется задавать допускаемое число неспариваний и их локализацию. Программа E-CRISP способна осуществлять дизайн gРНК для вариантов CRISPR/Cas-технологии в виде репрессии и активации генов ‒ CRISPRi (CRISPR inerference) и CRISPRa CRISPR activation) соответственно. Подобная возможность реализована также в программах CLD [53] и CRISPR-ERA (http://crispr-era.stanford.edu) [77], однако в последней нет анализа растительных геномов.
Web-ресурс CT-Finder (CRISPR Target Finder) [71] позволяет вести поиск gРНК для геномного редактирования с помощью трех ферментов: Cas9-нуклеазы, nCas9-никазы и RFN-системы (RNA-guided FokI Nuclease) в виде fCas9-нуклеазы. Для обычной Cas9-нуклеазы программа находит единичные gРНК, при использовании по отдельности никазы nCas9 (D10A) и нуклеазы fCas9 в результате поиска предлагаются парные gРНК; причем в случае последних задают расстояния между ними. Также указывают число анализируемых цепей ДНК – одна или две ‒ и устанавливают ряд других опций.
В дальнейшем Zhu и Liang [36] усовершенствовали программный продукт CT-Finder, в котором описан дизайн gРНК с помощью программы CRISPR-DT (DNA Targeting), ‒ первой программы, рассчитанной на дизайн gРНК с использованием нуклеазы Cpf1. Для этого авторы предварительно провели анализ большого числа gРНК, классифицируя данные методом опорных векторов, что позволило обеспечить максимальную эффективность и специфичность дизайна gРНК в программе CRISPR-DT.
Но это относится только к семи видам организмов, среди которых три растительных: арабидопсис, рис и соя. Экспериментаторам предоставлены две возможности: осуществить дизайн gРНК de novo или проверить эффективность редактирования уже ранее подобранных gРНК; при этом по умолчанию для дизайна gРНК используют PAM-последовательность TTTV, тогда как при поиске нецелевых мест редактирования программа использует иной PAM-участок – TTTN. Дизайн de novo состоит из нескольких шагов, первый из которых ‒ ввод намечаемой к редактированию нуклеотидной последовательности, после чего указывают организм из тех же, что и в программе CT-Finder, с которой совпадают все дальнейшие процедуры. Необходимо выбрать чувствительность поиска из четырех градаций: низкая, средняя, высокая и крайне высокая. Результаты поиска отображаются в виде таблицы. Приводится GC-состав подобранных протоспейсеров как для всей последовательности, так и отдельно для шести нуклеотидов, прилегающих к PAM-участку.
Программа CRISPR-RT (RNA Targeting) этой же группы авторов [72] ‒ первая программа дизайна gРНК, где в качестве нуклеазы использован фермент C2c2 (Cas13a). После ввода анализируемой последовательности следует выбрать один из 10 предзагруженных транскриптомов, из которых три – растительные: Oryza sativa, Zea mays и Arabidopsis thaliana; причем для последнего вида доступны два транскриптома из разных баз данных. Имеется ряд опций, позволяющих сузить разброс выдаваемых результатов.
Программа CRISPR MultiTargeter [58, 59] интересна тем, что помимо дизайна sgRNA для одиночных последовательностей позволяет проводить множественное выравнивание гомологичных последовательностей для последующего выбора в качестве протоспейсера одинаковых мест. PAM-участок может быть введен экспериментатором или NGG, причем только в последнем случае в выбранном геноме будет производиться поиск нецелевых сайтов с помощью сопряженных программ.
Работа в программе CRISPR Genome Analysis Tool – CGAT [50] состоит из двух этапов. На первом для редактирования с помощью Cas9 выбирают конкретный ген или последовательность нуклеотидов, которую вводят через буфер обмена, После установления ряда параметров: длина gРНК, желательный GC-состав (от 40 до 60%) и еще одного параметра в виде допустимого числа повторяющихся нуклеотидов в гомополимерных участках ‒ запускается поиск, результаты которого отображаются в виде таблицы с ранжированием пригодности gРНК для эффективного геномного редактирования. Опционально можно определить потенциальные места нецелевого редактирования геномов, выбранных из тех же видов растений.
Программа CRISPRdirect [56] ‒ простой и удобный web-ресурс, обеспечивающий дизайн gРНК для нуклеазы Cas9. Для выбора протоспейсеров в анализируемой последовательности требуется указать PAM-участок, хотя выбор небольшой: NGG (по умолчанию), NRG или NAG. Доступных геномов достаточно много и среди них 98 геномов представителей царства растений, включающих как основные сельскохозяйственные культуры и модельные объекты, так и виды из дикорастущей флоры. Вместо поиска новых gРНК можно вести анализ подобранных ранее. Результаты оформляются в графическом виде и в табличной форме, в которой приводятся позиции протоспейсеров с указанием конкретных цепей ДНК, непосредственно сама последовательность с PAM-участком, ее GC-состав и температура плавления (без PAM-участка), наличие сайтов рестрикции и TTTT-последовательности, а также число нецелевых сайтов для подобранных gРНК для 20-, 12- и 8-звенных последовательностей спейсера по отдельности. Поставив галочку в соответствующее окошко, можно ограничить вывод результатов только для высокоспецифичных вариантов.
Web-ресурс CRISPR-PLANT рассчитан на дизайн gРНК и отслеживание нецелевых сайтов в 8 растительных геномах [35, 63]. Для новой версии программы, v2, разработан используемый при поиске нецелевых сайтов новый алгоритм, заметно повышающий достоверность такого анализа, что должно улучшить эффективность геномного редактирования с помощью нуклеазы Cas9. В качестве PAM-участков представлены только последовательности NGG, так как варианты NAG в большей степени предрасположены к нецелевому редактированию. Изменений каких-либо опций не предусмотрено. Результаты поиска протоспейсеров выдаются в табличной форме отдельно для gРНК классов 0.0 и 1.0, предполагающих соответственно отсутствие нецелевых сайтов в геноме редактируемого вида растений и присутствие таковых, содержащих четыре или большее число неспариваний во всей мишени или три неспаривания в области, близкой к PAM-участку. Первая группа – хорошие gРНК с минимальной возможностью нецелевого спаривания, а вторая – менее хорошие и их лучше не использовать. Приводятся также сведения о цепи ДНК и характеристики мест редактирования: экзон, интрон, межгенный участок.
Программа GT-Scan [75] позволяет искать gРНК для нуклеазы Cas9 с двумя вариациями PAM-участка, устанавливая уровень специфичности от 0 до 3. Результаты оформляются графически и в виде перечня подходящих последовательностей протоспейсеров с выделенными разными цветами якорной областью и остальным участком, сопровождаемые информацией о локализации этих последовательностей, принадлежности к той или иной цепи ДНК и о числе потенциальных нецелевых участков редактирования в зависимости от строгости поиска. Для каждой подобранной последовательности можно получить более подробную информацию о расположении нецелевых сайтов в анализируемом геноме. На данном сайте содержатся также и другие программные продукты, среди которых можно упомянуть программу CUNE (Computational Universal Nucleotide Editor). Несмотря на то, что CUNE рассчитана на работу только с геномом мыши, она относится к тем немногим, которые позволяют проектировать избирательное редактирование азотистых оснований [78]. Программа CUNE в целом похожа на программу BE-Designer из пакета CRISPR RGEN Tools, отличаясь тем, что в ней для дизайна gРНК используется только одна нуклеаза Cas9 с канонической PAM-последовательностью NGG.
Web-ресурс CRISTA (CRISPR Target Assessment) [30] позволяет проводить анализ ранее выбранных gРНК и их ранжирование для редактирования конкретного гена, а также обнаружение нецелевых сайтов. Для дизайна gРНК необходимо запустить программу RANK TARGETS IN GENE, рассчитанную на поиск протоспейсеров, узнаваемых нуклеазой Cas9 с PAM-участком в виде NGG, введя через буфер обмена анализируемую последовательность ДНК. В программе не предусмотрен выбор дополнительных параметров и опций. После завершения поиска формируется таблица с ранжированными по эффективности редактирования gРНК, для которых в дальнейшем с помощью подключаемой программы FIND OFF-TARGETS можно выявить нецелевые сайты редактирования в одном из имеющихся на web-ресуре CRISTA геномов. Заметим, что программу FIND OFF-TARGETS можно использовать непосредственно для анализа специфичности ранее подобранных gРНК. Программа sgRNA:DNA SCORE также способна вести анализ готовых gРНК на предмет их эффективности.
Программа PhytoCRISP-Ex позволяет осуществлять дизайн gРНК для редактирования с помощью нуклеазы Cas9 геномов 13 видов фитопланктона [76]. Применяются два параллельно работающих типа фильтров, отсекающих разные варианты gРНК с потенциальными нецелевыми сайтами в геноме редактируемого вида. После завершения поиска программа выдает информацию о наличии в подобранных протоспейсерах сайтов рестрикционных эндонуклеаз для подтверждения произошедшего редактирования с помощью ПЦР-ПДРФ (ПЦР-полиморфизм длин рестрикционных фрагментов) с фланкирующими праймерами. При установке программы PhytoCRISP-Ex на компьютер пользователя ее функциональные возможности будут несколько расширены.
Программа WheatCrispr ориентирована на дизайн gРНК, используемых исключительно для редактирования генома мягкой хлебной пшеницы Triticum aestivum. Для поиска протоспейсеров необходимо выбрать конкретный редактируемый ген из созданной усилиями International Wheat Genome Sequencing Consortium базы данных IWGSC RefSeq assembly v1.0 (https://wheat-urgi. versailles.inra.fr/Seq-Repository/Assemblies) или через буфер обмена ввести последовательность. Максимально допустимая длина последовательности не оговорена; нам удалось провести дизайн gРНК для 1 815 н., при этом было найдено 350 потенцильных протоспейсеров. Результаты ранжируются по общей эффективности редактирования с учетом возможных нецелевых сайтов. Учитывая, что мягкая пшеница относится к гексаплоидам, можно искать протоспейсеры в гомологичных последовательностях всех трех субгеномов: B, A и D, ‒ выбрав соответствующую опцию. Еще одна опция этой программы позволяет выбрать участок редактирования – кодирующие области или промоторы.
Помимо web-ресурсов, позволяющих проводить дизайн gРНК в режиме on-line, существуют подобные программы, работающие автономно, среди которых есть рассчитанные и на геномное редактирование растительных объектов. Одна из них ‒ уже упоминавшаяся программа CLD (CRISPR Library Designer) [53], написанная теми же авторами, что и программа E-CRISPR [74], что объясняет совпадение у них ряда опций. Дизайн gРНК с помощью программы CLD заключается в выборе из базы данных Ensembl конкретного генома, в котором будет вестись обнаружение нецелевых мест редактирования; формирования перечня генов и установки для нуклеазы Cas9 параметров поиска потенциальных протоспейсеров (длины, GC-состава, неспариваний в якорной и остальной частях). Для приемлемых вариантов gРНК проводят аннотирование мест их связывания, сопровождаемое информацией о цепи ДНК, сайтах рестрикции, рекомендуемыми олиогонуклеотидами для клонирования. К особенностям программы CLD относится дизайн gРНК для вариантов CRISPR/Cas-технологии в виде CRISPRi и CRISPRa.
Еще один программный продукт, работающий автономно и нацеленный на растения, ‒ программа CRISPR Primer Designer [64]. Поиск протоспейсеров для нуклеазы Cas9 ведут в загружаемой через буфер обмена последовательности ДНК (протяженность может превышать 5 000 н.) после установки ряда параметров: длины якорной последовательности, GC-состава, указания 5′-нуклеотида в виде “G” и др. Возможное появление нецелевых сайтов редактирования с помощью CRISPR Primer Designer в геномах растений отслеживается только для риса и арабидопсиса, для чего программа автоматически обращается к web-ресурсу BLAST, генерируя ранжированные протоспейсеры для дизайна gРНК. Второй режим работы этой программы направлен на дизайн gРНК, служащих для маркирования (визуализации) хромосомных участков in vivo, и в этом случае предусмотрены свои особенности параметров поиска. Ни одна другая программа дизайна gРНК такой возможности не предоставляет.
Недавно написана программа GRIBCG (Guide RNA Identifier for Balancer Chromosome Generation) [79], рассчитанная на поиск gРНК в пределах одной хромосомы, которые фланкируют опредленный хромосомный участок с целью его инверсии после образования двухцепочечных разрывов ДНК, генерируемых под действием SpCas9-нуклеазы. Эта программа работает в режиме off-line и доступна для скачивания по ссылке https://sourceforge.net/projects/gribcg/. Поиск, включая обнаружение нецелевых сайтов редактирования, осуществляется на хромосомах организмов 6 видов, среди которых два растительных – арабидопсис и рис.
В последние годы стали появляться web-ресурсы, позволяющие экспериментаторам не просто осуществлять дизайн gРНК, но и за определенную плату тут же заказывать или химический синтез соответствующих молекул gРНК, или создание генно-инженерных конструкций, непосредственно готовых для проведения геномного CRISPR/Cas-редактирования конкретных генов. Ниже рассмотрены именно такие программы.
Web-ресурс Dharmacon CRISPR Design Tool фирмы “Dharmacon, Inc.” позволяет осуществлять дизайн gРНК, используя для введения последовательности ДНК длиной до 10 т.н., буфер обмена, ID конкретного гена. Затем требуется выбрать организм из предлагаемого относительно небольшого списка, в котором есть пять растительных (кукуруза, соя, рис, яблоня, хлопчатник). Результаты поиска отображаются в графическом и табличном виде, несущем различные сведения о предлагаемых последовательностях протоспейсеров, включая информацию по специфичности, локализации, возможности просмотра окружающих участков ДНК. На следующем этапе, выбрав ту или иную последовательность, можно перейти к заказу химического синтеза соответствующей gРНК, указав также его масштаб. Такой инструмент как CRISPR Specificity Analysis Tool позволяет выявить нецелевые сайты редактирования в тех же геномах, для чего надо ввести последовательность протоспейсеров и выбрать параметры поиска: один из двух вариантов PAM-участка (NGG или NAG), длину якорной последовательности и число неспариваний и инделов с указанием цепи (ДНК и/или РНК).
Другой подобный web-ресурс Synthego CRISPR Design Tool фирмы “Syntego” позволяет для нокаутного редактирования с помощью нуклеазы SpCas9 осуществлять дизайн gРНК, анализируя на предмет наличия нецелевых сайтов геномы, в том числе растительные, доступные из базы данных Ensembl. Необходимо ввести название организма или ID генома, затем название гена или его ID и запустить поиск, в результате которого программа выдаст рекомендуемые последовательности gРНК, химический синтез которых можно на следующем шаге заказать за соответствующую плату с тем, чтобы вести затем геномное редактирование с помощью РНП.
Web-ресурс CRISPR gRNA Design tool фирмы “Atum” дает возможность осуществить дизайн gРНК для редактирования геномов с помощью нуклеазы Cas9 дикого типа или никаз на ее основе, для чего требуется задать диапазон в нуклеотидах, на котором должна располагаться пара никаз. Также, выбрав вариант “I have my own gRNA”, можно проводить анализ имеющихся gРНК на предмет наличия для них нецелевых сайтов в редактируемых геномах пяти видов организмов, один из которых растительный (Arabidopsis thaliana), введя соответствующую нуклеотидную последовательность. Для дизайна gРНК de novo необходимо ввести фрагмент ДНК, намеченный для редактирования. Это можно сделать, указав название гена или его координаты в геноме или путем ввода нуклеотидной последовательности длиной не более 10 т.н. через буфер обмена. Результаты выводятся в графическом и текстовом виде с ранжированием последовательности по степени их пригодности для редактирования. Завершить процедуру поиска можно путем выбора вектора для платного клонирования подобранных gРНК.
Широкий спектр продуктов для CRISPR/Cas9-технологии представлен на сайте известной фирмы “Sigma-Aldrich” на странице MISSION CRISPR/ Cas9 Products and Service (https://www.sigmaaldrich. com/catalog/product/sigma/crispr), где, заполнив соответствующую форму, можно заказать в том числе и gРНК для использования с SpCas9-нуклеазой или с парными никазами nCas9 (D10A) для редактирования в нокаутном или иных режимах практически любого генома, полная нуклеотидная последовательность которого известна.
НЕ НАЦЕЛЕННЫЕ НА РЕДАКТИРОВАНИЕ ГЕНОМОВ РАСТЕНИЙ ПРОГРАММЫ И WEB-РЕСУРСЫ ДЛЯ ДИЗАЙНА РНК-ГИДОВ
Помимо вышеописанных программных продуктов, предназначенных или исключительно, или в том числе для редактирования геномов из царства растений, есть ряд программ для дизайна gРНК, которые можно использовать при редактировании геномов растительных объектов, если для них нет возможности выявления нецелевых сайтов или наличие последних непринципиально. Ниже приведены перечни таких программ на основе характера их работы – в сети интернет или только в автономном режиме.
Программы дизайна gРНК, работающие on-line (по алфавиту): CasBLASTR (http://www.casblastr.org); CasOT (http://casot.cbi.pku.edu.cn); COSMID (https://crispr.bme.gatech.edu); CREATE Designer (http://www.thebioverse.org); CRISPcut (http://web.iitd.ac.in/crispcut/webserver/index.html); CRISPETa (http://crispeta.crg.eu); CRISPR4P (http://bahlerweb.cs.ucl.ac.uk/cgi-bin/crispr4p/webapp.py); CRISPR-Cas9 guide RNA design checker (https://eu.idtdna.com/site/order/designtool/index/ crispr_sequence); CRISPR-DO (http://cistrome.org/ crispr/); CRISPR Efficiency Predictor (http://www. flyrnai.org/evaluateCrispr/); CRISPR-ERA (http://crispr-era.stanford.edu); CRISPR-FOCUS (http://cistrome.org/crispr-focus/); CrispRGold (http://crisprgold.mdc-berlin.de); CRISPR Mapper (http://crdd.osdd.net/servers/crisprge/mapper.php); CRISPR.ML (https://crispr.ml); CRISPRoff (https://rth.dk/resources/crispr/crisproff/); CRISPR Optimal Target Finder (http://targetfinder.flycrispr. neuro.brown.edu); CRISPR-PN (http://www.crispr-pn.net); CRISPRScan (http://www.crisprscan.org); CRISPR sgRNA Design Tool (http://www.genscript. com/gRNA-design-tool.html); CRISPR-SKIP (http:// song.igb.illinois.edu/crispr-skip); CRISPy CHO (http://staff.biosustain.dtu.dk/laeb/crispy); CRISPys (http://multicrispr.tau.ac.il); CRISPy-web (https:// crispy.secondarymetabolites.org); CROP-IT (http:// www.adlilab.org/CROP-IT/cas9tool.html); DeepCRISPR (http://www.deepcrispr.net); EuPaGDT (http://grna.ctegd.uga.edu); FORECasT (https:// partslab.sanger.ac.uk/FORECasT); GB CRISPR Tools (https://gbcloning.upv.es/tools/crisprs); ge-CRISPR (http://bioinfo.imtech.res.in/manojk/gecrispr/index.php); GPP Web Portal (https://portals. broadinstitute.org/gpp/public); Green Listed (http:// greenlisted.cmm.ki.se); grID (http://crispr.technology); Guide RNA Generator (http://penchovsky.atwebpages.com/applications.php?page=48); GUIDES (http://guides.sanjanalab.org/#/); GuideScan (http://guidescan.com); inDelphi (https:// www.crisprindelphi.design/about); Mojo Hand (http://www. talendesign.org); Off-Spotter (https://cm.jefferson.edu/ Off-Spotter/); PAVOOC (https://pavooc.me); sgRNA Designer (CRISPRko) (https://portals.broadinstitute.org/ gpp/public/analysis-tools/sgrna-design); Stupar Lab’s CRISPR Design (http://stuparcrispr.cfans.umn.edu/ CRISPR/); ZiFiT (http://zifit.partners.org/ZiFiT).
Программы дизайна gРНК, работающие автономно (по алфавиту): Azimuth (https://github.com/ MicrosoftResearch/Azimuth); CasFinder (http:// arep.med.harvard.edu/CasFinder); CASPER (https:// github.com/trinhlab/casper); Crisflash (https:// github.com/ crisflash/crisflash); CRISPR-Analyser (https://github.com/htgt/CRISPR-Analyser); CRISPRer (http://jstacs.de/index.php/CRISPRer); CRISPRO (https://gitlab.com/bauerlab/crispro); CRISPR-offinder (https://sourceforge.net/projects/crispr-offinder-v1-2/); CRISSPRpred (https:// github.com/khaled-rahman/CRISPRpred); CRISPRseek (http://bioconductor.org/packages/release/bioc/html/CRISPRseek. html); DeepCas9 (https://github.com/lje00006/ DeepCas9); FlashFry (https://github.com/aaronmck/ FlashFry); GRIBCG (https://sourceforge.net/projects/ gribcg/); MENTHU (http:// genesculpt.org/menthu); pgRNAFinder (https://github.com/xiexiaowei/ pgRNAFinder); predictSGRNA (http://www.ams. sunysb.edu/~pfkuan/softwares.html#predictsgrn); sgRNAcas9 (https://sourceforge.net/projects/sgrnacas9); WU-CRISPR (http://crispr.wustl.edu).
Среди этих программ есть как довольно простые инструменты с ограниченными возможностями, так и характеризующиеся широким спектром таковых в виде большого числа используемых нуклеаз, различных опций, способствующих улучшенному дизайну gРНК с предсказанием нецелевых сайтов в связанных с данными программами геномах, относящихся к разным группам организмов. Часть этих программ написана довольно давно и хорошо себя зарекомендовала, но есть и появившиеся относительно недавно, при этом обладающие неплохим потенциалом.
Следует сказать, что иногда имеет смысл произвести дизайн gРНК для редактирования геномов растений одной из этих “нерастительных” программ, позволяющих сделать это без указания конкретного генома (режимы “None”, “No Genome” и им подобные), после чего осуществить поиск нецелевых мест редактирования с использованием некоторых программ (CRISPR RGEN Tools, CRISPR-GE, E-CRISP, CRISTA, CRISPR gRNA Design tool), в которых есть возможность провести валидацию подобранных ранее gРНК, анализируя конкретные растительные геномы (при их наличии в этих программах).
Отдельный интерес для исследователей, занятых редактированием геномов растительных организмов, могут представлять программы, рассчитанные на дизайн gРНК не только с использованием классического варианта нуклеазы Cas9, но и ее ортологов, а также нуклеаз Cas12a (Cpf1), характеризующихся AT-богатыми PAM-участками, что, как уже отмечалось выше, дает для растений заметно больше возможностей выбора мест редактирования, да и сам этот фермент характеризуется повышенной специфичностью действия. Так, например, web-ресурс CRISPRScan [80], помимо классического варианта нуклеазы Cas9, позволяет использовать нуклеазы Cas12a: LbCpf1 (TTTV), AsCpf1 (TTTV), LbCpf1 (TTTN) и AsCpf1 (TTTN). Этот ресурс настроен на поиск нецелевых сайтов только в 14 геномах животных организмов и дрожжей, но для дизайна gРНК для иных объектов, включая растения, можно выбрать вариант “No search” и получить подобранные последовательности протоспейсеров.
ЗАКЛЮЧЕНИЕ
В представленном обзоре достаточно подробно рассмотрены или просто упомянуты около 100 программных продуктов, позволяющих осуществлять дизайн gРНК для CRISPR/Cas-технологии геномного редактирования. В настоящее время это самая полная подборка таких инструментов. Около четверти из них позволяют вести поиск целевых и нецелевых сайтов в геномах организмов из царства растений. Хотя последствий возникновения нецелевых мутаций при геномном редактировании растительных геномов можно сильно и не опасаться, тем не менее, безусловно, надо пытаться максимально избегать нецелевого редактирования и поэтому дизайну gРНК, когда известны полные последовательности геномов, необходимо уделять повышенное внимание. В этой связи неудивительно, что написано уже множество соответствующих компьютерных программ для дизайна gРНК, в том числе ориентированных на геномы растений, которые, однако, не в полной мере отвечают бурному развитию технологий геномного редактирования с помощью CRISPR/Cas-систем. Так, подбирать gРНК для Cas12a-нуклеаз позволяет только часть программ, но при этом для большинства из них оговаривается, что полноценного ранжирования и поиска нецелевых сайтов для этой нуклеазы не производится. Учитывая перспективность редактирования геномов растений именно с помощью Cas12a (CpfI), желательно разрабатывать для нее специальные алгоритмы поиска мест редактирования и анализа gРНК, включая нецелевые сайты. И этот процесс уже идет [36]. При все более частом использовании для редактирования РНП с химически синтезированными молекулами gРНК также представляется актуальным при подборе потенциальных протоспейсеров в самом начале работы предлагать исследователю альтернативу в виде выбора вариантов редактирования геномов с использованием или ферментативно или химически синтезированной РНК и соответственно подстраивать под них требования при поиске протоспейсеров.
Перспективным выглядит редактирование отдельных азотистых оснований, в том числе по причине того, что используемые в этом процесе химерные нуклеазы позволяют без образования двухцепочечных разрывов и инделов вести нокаутное редактирование. К сожалению, пока не так много программ дизайна gРНК, предоставляющих такую возможность, но можно прогнозировать, что их число будет расти. Процесс этот также идет. Фактически уже после завершения написания этого обзора появилась статья [81], в которой сообщается о новой такой программе beditor (https://github.com/rraadd88/beditor), позволяющей осуществлять дизайн gРНК для редактирования отдельных азотистых оснований и вести поиск нецелевых сайтов в 125 геномах разных организмов, включая растения.
Еще одному важному моменту в последнее время стали уделять серьезное внимание. Это локализация редактируемых мест генома в нуклеосомах, в эу- или гетерохроматиновых участках, так как от этого, как показано, зависит эффективность процесса редактирования [82‒85]. Правда, в литературе есть и другая информация. Например, у кукурузы не обнаружено различий в эффективности редактирования геномной ДНК, расположенной как в эухроматиновых, так и в гетерохроматиновых регионах [86]. Сейчас фактически есть только один web-ресурс, CROP-IT (CRISPR/Cas9 Off-target Prediction and Identification Tool) [87] (http://www.adlilab.org/CROP-IT/ cas9tool.html), который рассчитан на дизайн gРНК и выявление потенциальных нецелевых сайтов редактирования с учетом состояния хроматина в этих местах. Однако такой анализ с помощью этой программы доступен всего для двух геномов: человека и мыши (на сайте программы сообщается, что вскоре может быть добавлено большее число геномов) ‒ и только для нуклеазы Cas9 с двумя вариантами PAM-последовательностей. Можно предположить, что при написании новых программ дизайна gРНК и совершенствовании существующих, в том числе для редактирования геномов из царства растений, будет по возможности учитываться состояние хроматина, что позволит повысить эффективность CRISPR/Cas-технологии. В недавно опубликованной работе Zhang S. и соавт. [88] как раз обсуждают синергетический эффект предсказания мест геномного редактирования с учетом принятия во внимание состояния хроматина.
Также для прогнозирования эффективности редактирования целевых сайтов и активности по нецелевым необходимо учитывать термодинамику взаимодействия Cas-нуклеаз с мишенями ДНК. Это недавно показано Zhang D. и др. [89] на примере Cas9-нуклеазы, для которой была применена модель “ближайших соседей” при подсчете свободной энергии перекрывающихся динуклеотидов. Разработанная этими авторами модель uCRISPR (http://rna.physics.missouri.edu/ uCRISPR/index.html) позволила улучшить предсказания нецелевых сайтов редактирования.
Мы сознательно не стали здесь указывать, какие из программ дизайна gРНК больше подходят для начинающих пользователей, а какие для продвинутых, так как это, во-первых, в какой-то степени субъективно и, во-вторых, стоящие перед разными экспериментаторами задачи и определяют выбор того или иного конкретного инструмента. Именно поэтому главная цель этой статьи ‒ составить перечень таких программ и кратко охарактеризовать их возможности. Заметим, что в связи с продолжающимся обнаружением новых закономерностей и нюансов функционирования Cas-нуклеаз и выяснением влияния на взаимодействие с ними определенных нуклеотидных последовательностей в целевых и нецелевых сайтах геномов алгоритмы программ для дизайна gРНК постоянно совершенствуются и для достижения наилучших результатов при проведении геномного редактирования следует постоянно следить за их обновлениями. И все же, основываясь на собственном опыте, мы можем порекомендовать для дизайна gРНК в рамках одного эксперимента использовать сразу несколько программ, сопоставляя выдаваемые результаты.
Работа выполнена при финансовой поддержке РФФИ в рамках научных проектов №№ 18-04-00118 А, 19-016-00117 А, 19-016-00139 А, а также в рамках Госзадания по темам №№ АААА-А16-116020350028-4, AAAA-A19-119021190011-0.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Кулуев Б.Р., Гумерова Г.Р., Михайлова Е.В., Геращенков Г.А., Рожнова Н.А., Вершинина З.Р., Князев А.В., Матниязов Р.Т., Баймиев Ан.Х., Бай-миев Ал.Х., Чемерис А.В. (2019) Доставка CRISPR/ CAS-компонентов в клетки высших растений для редактирования их геномов. Физиология растений. 66, 339‒353. https://doi.org/10.1134/S0015330319050117
Graham D.B., Root D.E. (2015) Resources for the design of CRISPR gene editing experiments. Genome Biol. 16, 260. https://doi.org/10.1186/s13059-015-0823-x
Kanchiswamy C.N., Maffei M., Malnoy M., Velasco R., Kim J.S. (2016) Fine-tuning next-generation genome editing tools. Trends Biotechnol. 34, 562‒574. https://doi.org/10.1016/j.tibtech.2016.03.007
Chuai G.H., Wang Q.L., Liu Q. (2017) In silico meets in vivo: towards computational CRISPR-based sgRNA design. Trends Biotechnol. 35, 12‒21. https://doi.org/10.1016/j.tibtech.2016.06.008
Periwal V. (2017) A comprehensive overview of computational resources to aid in precision genome editing with engineered nucleases. Brief Bioinform. 18, 698‒711. https://doi.org/10.1093/bib/bbw052
Yennmalli R.M., Kalra S., Srivastava P.A., Garlapati V.K. (2017) Computational tools and resources for CRISPR/ Cas 9 genome editing method. MOJ Proteomics Bioinform. 5, 00164.
Cui Y., Xu J., Cheng M., Liao X., Peng S. (2018) Review of CRISPR/Cas9 sgRNA design tools. Interdiscip. Sci. 10, 455‒465. https://doi.org/10.1007/s12539-018-0298-z
Demirci Y., Zhang B., Unver T. (2018) CRISPR/Cas9: an RNA-guided highly precise synthetic tool for plant genome editing. J. Cell Physiol. 233, 1844‒1859. https://doi.org/10.1002/jcp.25970
Yan J., Chuai G., Zhou C., Zhu C., Yang J., Zhang C., Gu F., Xu H., Wei J., Liu Q. (2018) Benchmarking CRISPR on-target sgRNA design. Brief Bioinform. 19, 721‒724. https://doi.org/10.1093/bib/bbx001
Чемерис Д.А., Кирьянова О.Ю., Геращенков Г.А., Кулуев Б.Р., Рожнова Н.А., Матниязов Р.Т., Баймиев Ан.Х., Баймиев Ал.Х., Губайдуллин И.М., Чемерис А.В. (2017) Биоинформатические ресурсы для CRISPR/Cas редактирования геномов. Биомика. 9, 203‒208.
Чугунова А.А., Донцова О.А., Сергиев П.В. (2016) Методы изменения геномов: новая эра в молекулярной биологии. Биохимия. 81, 881‒898.
Jiang F., Doudna J.A. (2017) CRISPR-Cas9 structures and mechanisms. Annu. Rev. Biophys. 46, 505‒529. https://doi.org/10.1146/annurev-biophys-062215-010822
Банников А.В., Лавров А.В. (2017) CRISPR/CAS9 – король геномного редактирования. Молекуляр. биология. 51, 582‒594. https://doi.org/10.7868/S0026898417040036
Кулуев Б.Р., Геращенков Г.А., Рожнова Н.А., Баймиев Ан.Х., Вершинина З.Р., Князев А.В., Матниязов Р.Т., Гумерова Г.Р., Михайлова Е.В., Никоноров Ю.М., Чемерис Д.А., Баймиев Ал.Х., Чемерис А.В. (2017) CRISPR/Cas редактирование геномов растений. Биомика. 9, 155‒182.
Карагяур М.Н., Рубцов Ю.П., Васильев П.А., Ткачук В.А. (2018) Практические рекомендации по повышению эффективности и точности системы редактирования генома CRISPR/Cas9. Биохимия. 83, 800‒815.
Ahmad H.I., Ahmad M.J., Asif A.R., Adnan M., Iqbal M.K., Mehmood K., Muhammad S.A., Bhuiyan A.A., Elokil A., Du X., Zhao C., Liu X., Xie S. (2018) A review of CRISPR-based genome editing: Survival, evolution and challenges. Curr. Issues Mol. Biol. 28, 47‒68. https://doi.org/10.21775/cimb.028.047
Makarova S.S., Khromov A.V., Spechenkova N.A., Taliansky M.E., Kalinina N.O. (2018) Application of the CRISPR/Cas system for generation of pathogen-resistant plants. Biochemistry (Moscow). 83, 1552‒1562. https://doi.org/10.1134/S0006297918120131
(2018) Редактирование генов и геномов. Отв. ред. Закиян С.М., Медведев С.П., Дементьева Е.В., Покушалов Е.А., Власов В.В. 2-е изд., т. 3. Новосибирск: Изд-во СО РАН, 301 с.
Злобин Н.Е., Лебедева М.В., Таранов В.В., Харченко П.Н., Бабаков А.В. (2018) Редактирование генома растений путем направленной замены азотистых оснований. Биотехнология. 34, 59–68. https://doi.org/10.21519/0234-2758-2018-34-6-59-68
Korotkova A.M., Gerasimova S.V., Khlestkina E.K. (2019) Current achievements in modifying crop genes using CRISPR/Cas system. Вавиловский журнал генетики и селекции. 23, 29‒37. https://doi.org/10.18699/VJ19.458
Molla K.A., Yang Y. (2019) CRISPR/Cas-mediated base editing: technical considerations and practical applications. Trends Biotechnol. pii: S0167-7799(19)30053-8. https://doi.org/10.1016/j.tibtech.2019.03.008
Wolter F., Schindele P., Puchta H. (2019) Plant bree-ding at the speed of light: the power of CRISPR/Cas to generate directed genetic diversity at multiple sites. BMC Plant Biol. 19, 176. https://doi.org/10.1186/s12870-019-1775-1
Swarts D.C., Jinek M. (2018) Cas9 versus Cas12a/Cpf1: structure-function comparisons and implications for genome editing. Wiley Interdiscip. Rev. RNA. e1481. https://doi.org/10.1002/wrna.1481
Kelley M.L., Strezoska Z., He K., Vermeulen A., Smith A. (2016) Versatility of chemically synthesized guide RNAs for CRISPR-Cas9 genome editing. J. Biotechnol. 233, 74–83. https://doi.org/10.1016/j.jbiotec.2016.06.011
Andersson M., Turesson H., Olsson N., Fält A.S., Ohlsson P., Gonzalez M.N., Samuelsson M., Hofvander P. (2018) Genome editing in potato via CRISPR-Cas9 ribonucleoprotein delivery. Physiol. Plant. 164, 378‒384. https://doi.org/10.1111/ppl.12731
Strohkendl I., Saifuddin F.A., Rybarski J.R., Finkelstein I.J., Russell R. (2018) Kinetic basis for DNA target specificity of CRISPR-Cas12a. Mol. Cell. 71, 816‒824.e3. https://doi.org/10.1016/j.molcel.2018.06.043
Hsu P.D., Scott D.A., Weinstein J.A., Ran F.A., Konermann S., Agarwala V., Li Y., Fine E.J., Wu X., Shalem O., Cradick T.J., Marraffini L.A., Bao G., Zhang F. (2013) DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol. 31, 827‒832. https://doi.org/10.1038/nbt.2647
Doench J.G., Hartenian E., Graham D.B., Tothova Z., Hegde M., Smith I., Sullender M., Ebert B.L., Xavier R.J., Root D.E. (2014) Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nat. Biotechnol. 2014. 32, 1262‒1267. https://doi.org/10.1038/nbt.3026
Doench J.G., Fusi N., Sullender M., Hegde M., Vaimberg E.W., Donovan K.F., Smith I., Tothova Z., Wilen C., Orchard R., Virgin H.W., Listgarten J., Root D.E. (2016) Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nat. Biotechnol. 34, 184‒191. https://doi.org/10.1038/nbt.3437
Abadi S., Yan W.X., Amar D., Mayrose I. (2017) A machine learning approach for predicting CRISPR-Cas9 cleavage efficiencies and patterns underlying its mechanism of action. PLoS Comput. Biol. 13, e1005807. https://doi.org/10.1371/journal.pcbi.1005807
Kim H.K., Min S., Song M., Jung S., Choi J.W., Kim Y., Lee S., Yoon S., Kim H.H. (2018) Deep learning improves prediction of CRISPR-Cpf1 guide RNA activity. Nat. Biotechnol. 36, 239‒241. https://doi.org/10.1038/nbt.4061
Labuhn M., Adams F.F., Ng M., Knoess S., Schambach A., Charpentier E.M., Schwarzer A., Mateo J.L., Klusmann J.H., Heckl D. (2018) Refined sgRNA efficacy prediction improves large- and small-scale CRISPR-Cas9 applications. Nucleic Acids Res. 46, 1375‒1385. https://doi.org/10.1093/nar/gkx1268
Listgarten J., Weinstein M., Kleinstiver B.P., Sousa A.A., Joung J.K., Crawford J., Gao K., Hoang L., Elibol M., Doench J.G., Fusi N. (2018) Prediction of off-target activities for the end-to-end design of CRISPR guide RNAs. Nat. Biomed. Eng. 2, 38‒47. https://doi.org/10.1038/s41551-017-0178-6
Xue L., Tang B., Chen W., Luo J. (2019) Prediction of CRISPR sgRNA activity using a deep convolutional neural network. J. Chem. Inf. Model. 59, 615‒624. https://doi.org/10.1021/acs.jcim.8b00368
Minkenberg B., Zhang J., Xie K., Yang Y. (2019) CRISPR-PLANT v2: an online resource for highly specific guide RNA spacers based on improved off-target analysis. Plant Biotechnol. J. 17, 5‒8. https://doi.org/10.1111/pbi.13025
Zhu H., Liang C. (2019) CRISPR-DT: designing gRNAs for the CRISPR-Cpf1 system with improved target efficiency and specificity. Bioinformatics. 35, 2783–2789. https://doi.org/10.1093/bioinformatics/bty1061
Graf R., Li X., Chu V.T., Rajewsky K. (2019) sgRNA sequence motifs blocking efficient CRISPR/Cas9-mediated gene editing. Cell Rep. 26, 1098‒1103.e3. https://doi.org/10.1016/j.celrep.2019.01.024
Wong N., Liu W., Wang X. (2015) WU-CRISPR: characteristics of functional guide RNAs for the CRISPR/ Cas9 system. Genome Biol. 16, 218. https://doi.org/10.1186/s13059-015-0784-0
Гарафутдинов Р.Р., Баймиев Ан.Х., Малеев Г.В., Алексеев Я.И., Зубов В.В., Чемерис Д.А., Кирьянова О.Ю., Губайдуллин И.М., Матниязов Р.Т., Сахабутдинова А.Р., Никоноров Ю.М., Кулуев Б.Р., Баймиев Ал.Х., Чемерис А.В. (2019) Разнообразие праймеров для ПЦР и принципы их подбора. Биомика. 11, 23–70. https://doi.org/10.31301/2221-6197.bmcs.2019-04
Hahn F., Nekrasov V. (2019) CRISPR/Cas precision: do we need to worry about off-targeting in plants? Plant Cell Rep. 38, 437‒441. https://doi.org/10.1007/s00299-018-2355-9
Tang X., Liu G., Zhou J., Ren Q., You Q., Tian L., Xin X., Zhong Z., Liu B., Zheng X., Zhang D., Malzahn A., Gong Z., Qi Y., Zhang T., Zhang Y. (2018) A large-scale whole-genome sequencing analysis reveals highly specific genome editing by both Cas9 and Cpf1 (Cas12a) nucleases in rice. Genome Biol. 19, 84. https://doi.org/10.1186/s13059-018-1458-5
Ossowski S., Schneeberger K., Lucas-Lledó J.I., Warthmann N., Clark R.M., Shaw R.G., Weigel D., Lynch M. (2010) The rate and molecular spectrum of spontaneous mutations in Arabidopsis thaliana. Science. 327, 92‒94. https://doi.org/10.1126/science.1180677
Wolt J.D., Wang K., Sashital D., Lawrence-Dill C.J. (2016) Achieving plant CRISPR targeting that limits off-target effects. Plant Genome. 9(3). https://doi.org/10.3835/plantgenome2016.05.0047
Svitashev S., Schwartz C., Lenderts B., Young J.K., Mark Cigan A. (2016) Genome editing in maize directed by CRISPR-Cas9 ribonucleoprotein complexes. Nat. Commun. 7, 13274. https://doi.org/10.1038/ncomms13274
Liang Z., Chen K., Li T., Zhang Y., Wang Y., Zhao Q., Liu J., Zhang H., Liu C., Ran Y., Gao C. (2017) Efficient DNA-free genome editing of bread wheat using CRISPR/Cas9 ribonucleoprotein complexes. Nat. Commun. 8, 14261. https://doi.org/10.1038/ncomms14261
Хромов А.В., Махотенко А.В., Снигирь Е.В., Макарова С.С., Макаров В.В., Супрунова Т.П., Мирошниченко Д.Н., Калинина Н.О., Долгов С.В., Тальянский М.Э. (2018) Доставка рибонуклеопротеидного комплекса CRISPR/Cas9 в клетки апикальной меристемы для бесплазмидного редактирования генома картофеля Solanum tuberosum. Биотехнология. 34, 51‒58.
Мирошниченко Д.Н., Шульга О.А., Тимербаев В.Р., Долгов С.В. (2019) Достижения, проблемы и перспективы получения нетрансгенных растений с отредактированным геномом. Биотехнология. 35, 3‒26. https://doi.org/10.21519/0234-2758-2019-35-1-3-26
Oliveros J.C., Franch M., Tabas-Madrid D., San-León D., Montoliu L., Cubas P., Pazos F. (2016) Breaking-Cas-interactive design of guide RNAs for CRISPR-Cas experiments for ENSEMBL genomes. Nucleic Acids Res. 44, 267‒271. https://doi.org/10.1093/nar/gkw407
Stemmer M., Thumberger T., Del Sol Keyer M., Wittbrodt J., Mateo J.L. (2015) CCTop: an intuitive, flexible and reliable CRISPR/Cas9 target prediction tool. PLoS One. 24, e0124633.
Brazelton V.A. Jr., Zarecor S., Wright D.A., Wang Y., Liu J., Chen K., Yang B., Lawrence-Dill C.J. (2015) A quick guide to CRISPR sgRNA design tools. GM Crops Food. 6, 266–276. https://doi.org/10.1080/21645698.2015.1137690
Montague T.G., Cruz J.M., Gagnon J.A., Church G.M., Valen E. (2014) CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Res. 42, 401–407. https://doi.org/10.1093/nar/gku410
Labun K., Montague T.G., Gagnon J.A., Thyme S.B., Valen E. (2016) CHOPCHOP v2: a web tool for the next generation of CRISPR genome engineering. Nucleic Acids Res. 44, 272‒276. https://doi.org/10.1093/nar/gkw398
Heigwer F., Zhan T., Breinig M., Winter J., Brugemann D., Leible S., Boutros M. (2016) CRISPR library designer (CLD): software for multispecies design of single guide RNA libraries. Genome Biol. 17, 55. https://doi.org/10.1186/s13059-016-0915-2
Haeussler M., Schönig K., Eckert H., Eschstruth A., Mianné J., Renaud J.B., Schneider-Maunoury S., Shkumatava A., Teboul L., Kent J., Joly J.S.,Concor-det J.P. (2016) Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biol. 17, 148. https://doi.org/10.1186/s13059-016-1012-2
Concordet J.P., Haeussler M. (2018) CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46, 242‒245. https://doi.org/10.1093/nar/gky354
Naito Y., Hino K., Bono H., Ui-Tei K. (2015) CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31, 1120–1123. https://doi.org/10.1093/bioinformatics/btu743
Xie X., Ma X., Zhu Q., Zeng D., Li G., Liu Y-G. (2017) CRISPR-GE: A convenient software toolkit for CRISPR-based genome editing. Mol. Plant. 10, 1246‒1249. https://doi.org/10.1016/j.molp.2017.06.004
Prykhozhij S.V., Rajan V., Gaston D., Berman J.N. (2015) CRISPR Multitargeter: a web tool to find common and unique CRISPR single guide RNA targets in a set of similar sequences. PLoS One. 10, e0119372. https://doi.org/10.1371/journal.pone.0119372
Prykhozhij S.V., Rajan V., Gaston D., Berman J.N. (2015) Correction: CRISPR MultiTargeter: a web tool to find common and unique CRISPR single guide RNA targets in a set of similar sequences. PLoS One. 10, e0138634. https://doi.org/10.1371/journal.pone.0138634
Lei Y., Lu L., Liu H.Y., Li S., Xing F., Chen L.L. (2014) CRISPR-P: a web tool for synthetic single-guide RNA design of CRISPR-system in plants. Mol. Plant. 7, 1494–1496. https://doi.org/10.1093/mp/ssu044
Liu H., Ding Y., Zhou Y., Jin W., Xie K., Chen L.L. (2017) CRISPR-P 2.0: an improved CRISPR-Cas9 tool for genome editing in plants. Mol. Plant. 10, 530–532. https://doi.org/10.1016/j.molp.2017.01.003
Sun J., Liu H., Liu J., Cheng S., Peng Y., Zhang Q., Yan J., Liu H.J., Chen L.L. (2019) CRISPR-Local: a local single-guide RNA (sgRNA) design tool for non-reference plant genomes. Bioinformatics. 35, 2501–2503. https://doi.org/10.1093/bioinformatics/bty970
Xie K., Zhang J., Yang Y. (2014) Genome-wide prediction of highly specific guide RNA spacers for the CRISPR-Cas9 mediated genome editing in model plants and major crops. Mol. Plant. 7, 923‒926. https://doi.org/10.1093/mp/ssu009
Yan M., Zhou S.R., Xue H.W. (2015) CRISPR Primer Designer: design primers for knockout and chromosome imaging CRISPR-Cas system. J. Integr. Plant Biol. 57, 613‒617. https://doi.org/10.1111/jipb.12295
Bae S., Park J., Kim J.S. (2014) Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30, 1473–1475. https://doi.org/10.1093/bioinformatics/btu048
Bae S., Kweon J., Kim H.S., Kim J.S. (2014) Microhomology-based choice of Cas9 nuclease target sites. Nat. Methods. 11, 705–706. https://doi.org/10.1038/nmeth.3015
Park J., Bae S., Kim J.S. (2015) Cas-Designer: a web-based tool for choice of CRISPR-Cas9 target sites. Bioinformatics. 31, 4014–4016. https://doi.org/10.1093/bioinformatics/btv537
Park J., Kim J.S., Bae S. (2016) Cas-Database: web-based genome-wide guide RNA library design for gene knockout screens using CRISPR-Cas9. Bioinformatics. 32, 2017–2023. https://doi.org/10.1093/bioinformatics/btw103
Hwang G.H., Park J., Lim K., Kim S., Yu J., Yu E., Kim S.T., Eils R., Kim J.S., Bae S. (2018) Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19, 542. https://doi.org/10.1186/s12859-018-2585-4
Park J., Bae S. (2018) Cpf1-Database: web-based genome-wide guide RNA library design for gene knockout screens using CRISPR-Cpf1. Bioinformatics. 34, 1077‒1079. https://doi.org/10.1093/bioinformatics/btx695
Zhu H., Misel L., Graham M., Robinson M.L., Liang C. (2016) CT-Finder: a web service for CRISPR optimal target prediction and visualization. Sci. Rep. 23, 25516. https://doi.org/10.1038/srep25516
Zhu H., Richmond E., Liang C. (2018) CRISPR-RT: a web service for designing CRISPR-C2c2 crRNA with improved target specificity. Bioinformatics. 34, 117‒119. https://doi.org/10.1093/bioinformatics/btx580
Hough S.H., Kancleris K., Brody L., Humphryes-Kirilov N., Wolanski J., Dunaway K., Ajetunmobi A., Dillard V. (2017) Guide Picker is a comprehensive design tool for visualizing and selecting guides for CRISPR experiments. BMC Bioinformatics. 18, 167. https://doi.org/10.1186/s12859-017-1581-4
Heigwer F., Kerr G., Boutros M. (2014) E-CRISP: fast CRISPR target site identification. Nat. Methods. 11, 122–123. https://doi.org/10.1038/nmeth.2812
O’Brien A., Bailey T.L. (2014) GT-Scan: identifying unique genomic targets. Bioinformatics. 30, 2673–2675. https://doi.org/10.1093/bioinformatics/btu354
Rastogi A., Murik O., Bowler C., Tirichine L. (2016) PhytoCRISP-Ex: a web-based and stand-alone application to find specific target sequences for CRISPR/ CAS editing. BMC Bioinformatics. 17, 261. https://doi.org/10.1186/s12859-016-1143-1
Liu H., Wei Z., Dominguez A., Li Y., Wang X., Qi L.S. (2015) CRISPR-ERA: a comprehensive design tool for CRISPR-mediated gene editing, repression and activation. Bioinformatics. 31, 3676–3678. https://doi.org/10.1093/bioinformatics/btv423
O'Brien A.R., Wilson L.O.W., Burgio G., Bauer D.C. (2019) Unlocking HDR-mediated nucleotide editing by identifying high-efficiency target sites using machine learning. Sci. Rep. 9, 2788. https://doi.org/10.1038/s41598-019-39142-0
Merritt B.B., Cheung L.C. (2019) GRIBCG: a software for selection of sgRNAs in the design of balancer chromosomes. BMC Bioinformatics. 20, 122. https://doi.org/10.1186/s12859-019-2712-x
Moreno-Mateos M.A., Vejnar C.E., Beaudoin J.D., Fernandez J.P., Mis E.K., Khokha M.K., Giraldez A.J. (2015) CRISPRscan: designing highly efficient sgRNAs for CRISPR-Cas9 targeting in vivo. Nat. Methods. 12, 982‒988. https://doi.org/10.1038/nmeth.3543
Dandage R., Després P.C., Yachie N., Landry C.R. (2019) beditor: a computational workflow for designing libraries of guide RNAs for CRISPR-mediated base editing. Genetics. 212, 377‒385, https://doi.org/10.1534/genetics.119.302089
Chari R., Mali P., Moosburner M., Church G.M. (2015) Unraveling CRISPR-Cas9 genome engineering parameters via a library-on-library approach. Nat. Methods. 12, 823‒826. https://doi.org/10.1038/nmeth.3473
Horlbeck M.A., Witkowsky L.B., Guglielmi B., Replogle J.M., Gilbert L.A., Villalta J.E., Torigoe S.E., Tjian R., Weissman J.S. (2016) Nucleosomes impede Cas9 access to DNA in vivo and in vitro. Elife. 5, pii: e12677. https://doi.org/10.7554/eLife.12677
Jensen K.T., Fløe L., Petersen T.S., Huang J., Xu F., Bolund L., Luo Y., Lin L. (2017) Chromatin accessibility and guide sequence secondary structure affect CRISPR-Cas9 gene editing efficiency. FEBS Lett. 591, 1892‒1901. https://doi.org/10.1002/1873-3468.12707
Yarrington R.M., Verma S., Schwartz S., Trautman J.K., Carroll D. (2018) Nucleosomes inhibit target cleavage by CRISPR-Cas9 in vivo. Proc. Natl. Acad. Sci. USA. 115, 9351‒9358. https://doi.org/10.1073/pnas.1810062115
Feng C., Yuan J., Wang R., Liu Y., Birchler J.A., Han F. (2016) Efficient targeted genome modification in maize using CRISPR/Cas9 system. J. Genet. Genomics. 43, 37‒43. https://doi.org/10.1016/j.jgg.2015.10.002
Singh R., Kuscu C., Quinlan A., Qi Y., Adli M. (2015) Cas9-chromatin binding information enables more accurate CRISPR off-target prediction. Nucleic Acids Res. 43, e118. https://doi.org/10.1093/nar/gkv575
Zhang S., Li X., Lin Q., Wong K.C. (2019) Synergizing CRISPR/Cas9 off-target predictions for ensemble insights and practical applications. Bioinformatics. 35, 1108‒1115. https://doi.org/10.1093/bioinformatics/bty748
Zhang D., Hurst T., Duan D., Chen S.J. (2019) Unified energetics analysis unravels SpCas9 cleavage acti-vity for optimal gRNA design. Proc. Natl. Acad. Sci. USA. 116, 8693‒8698. https://doi.org/10.1073/pnas.1820523116
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


