Молекулярная биология, 2020, T. 54, № 2, стр. 212-223
Изменчивость фрагментов экзона 11 ядерного гена Brca1 и митохондриального гена Cox1 у домовых мышей Mus musculus
А. С. Богданов a, *, А. Н. Мальцев b, Е. В. Котенкова b, В. Г. Маликов c, А. А. Лисовский d, В. В. Стахеев e, J. Darvish f, R. Castiglia g
a Институт биологии развития им. Н.К. Кольцова Российской академии наук
119334 Москва, Россия
b Институт проблем экологии и эволюции им. А.Н. Северцова Российской академии наук
119071 Москва, Россия
c Зоологический институт Российской академии наук
199034 Санкт-Петербург, Россия
d Зоологический музей Московского государственного университета им. М.В. Ломоносова
125009 Москва, Россия
e Южный научный центр Российской академии наук
344006 Ростов-на-Дону, Россия
f Ferdowsi University of Mashhad
9177948974 Mashhad, Iran
g Università degli Studi di Roma “La Sapienza”
00161 Rome, Italy
* E-mail: bogdalst@yahoo.com
Поступила в редакцию 06.06.2019
После доработки 27.09.2019
Принята к публикации 07.10.2019
Аннотация
С целью уточнения генетических различий между подвидами Mus musculus, их распространения и гибридизации впервые проведен сравнительный анализ изменчивости нуклеотидных последовательностей фрагментов экзона 11 ядерного гена Brca1 (2331 п.н.) и митохондриального гена Cox1 (1260 п.н.) у 40 домовых мышей из Западной и Восточной Европы, Закавказья, Сибири, Средней и Южной Азии. Генотипы гена Brca1 разделились на пять основных групп, отличающихся друг от друга целым рядом фиксированных замен. Прослеживается отчетливая приуроченность генотипов каждой из этих групп к определенным географическим регионам и их специфичность для подвидов M. m. musculus, M. m. domesticus, M. m. castaneus и M. m. wagneri вместе с M. m. gansuensis; пятая группа соответствует неидентифицированному подвиду или отдельной генетической форме из индийского штата Сикким. Помимо гомозиготных экземпляров обнаружены также особи, гетерозиготные одновременно по всем диагностическим локусам и определенные нами как гибридные. Они выявлены в основном в зонах контактов подвидов, но в нескольких случаях – на большом расстоянии от них, вероятнее всего, вследствие завоза с транспортом. У двух гибридных мышей – из провинции Бахтиария (Иран), и из Забайкальского края (Россия) – выявлены уникальные гаплотипы Brca1. Не исключено, что они могут быть хотя бы отчасти свойственны подвидам M. m. bactrianus и M. m. gansuensis соответственно. Таким образом, наши результаты указывают на ядерный ген Brca1 как на весьма перспективный молекулярно-генетический маркер для изучения изменчивости, дифференциации и гибридизации домовых мышей, а также для подвидовой идентификации особей M. musculus. Ген Cox1, эволюционирующий быстрее гена Brca1, мало пригоден для дискриминации особей M. m. musculus, M. m. wagneri, M. m. gansuensis и закавказских представителей M. m. domesticus вследствие интрогрессии и длительного сохранения в популяциях чужеродной митохондриальной ДНК. Однако анализ гена Cox1 (наряду с диагностикой по ядерной ДНК) может быть полезным при оценке популяционных различий в пределах подвидов M. m. castaneus и M. m. domesticus.
ВВЕДЕНИЕ
Домовые мыши (род Mus) – гетерогенная группа, которая включает в себя множество форм, в разной степени отличающихся друг от друга как по морфологическим, экологическим, этологическим, так и по генетическим характеристикам. Вследствие высокого разнообразия, несинхронной изменчивости морфологических и генетических признаков, а также из-за гибридизации синантропных форм, весьма осложняющей выделение дискретных популяционных группировок и определение их специфических особенностей, таксономическая система рода многократно пересматривалась и в настоящее время не является общепринятой. Бесспорным можно признать лишь видовой уровень различий “дикоживущих” домовых мышей по отношению к синантропным. “Дикоживущие” формы (македонская мышь M. macedonicus, курганчиковая мышь M. spicilegus, западно-средиземноморская мышь M. spretus) также определяются в настоящее время большинством исследователей [1–3] как алловиды, т.е. фактически их статус приравнивается к видовому.
Множество парапатричных синантропных форм объединяется в составе вида домовая мышь M. musculus (или надвида, с иной точки зрения). Их систематика остается дискуссионной несмотря на то, что M. musculus – один из основных лабораторных объектов генетических и молекулярно-биологических исследований, а его полный геном был расшифрован вторым после человека [4]. Обычно синантропные формы домовой мыши рассматривают в качестве подвидов [2, 3, 5–8], хотя высказывались предложения о повышении таксономического ранга некоторых из них (M. m. domesticus, M. m. castaneus, M. m. musculus) до видового [9–13]. Столь же противоречивые сведения опубликованы о признаваемом до недавнего времени среднеазиатском подвиде M. m. wagneri и забайкальском M. m. gansuensis, которые считались наиболее близкими морфологически и генетически к M. m. musculus. Так, согласно Л.Н. Спиридоновой и соавт. [14], по комплексу RAPD-PCR-маркеров возможно определение всех подвидов M. musculus, причем уровень различий между некоторыми из них (M. m. wagneri и M. m. musculus) сопоставим с межвидовым. Также сообщалось об успешной идентификации особей M. m. wagneri, M. m. gansuensis и M. m. musculus по кариотипическим признакам [15]. Однако анализ изменчивости второго интрона ядерного гена b1-цепи гемоглобина (Hbb-b1) выявил у домовых мышей Средней Азии и Забайкалья только два генотипа, один из которых определен как характерный для M. m. musculus, а другой – как общий у M. m. wagneri и M. m. gansuensis [16]. Аналогично, при изучении полиморфизма восьми сцепленных генов восьмой аутосомы в Азии обнаружены две филогруппы домовых мышей (“musculus” и “wagneri”) [17]. Результаты недавних исследований фрагментов мтДНК – контрольного региона (D-loop) и гена цитохрома b (Cytb) – показали полную, примерно одинаковую, но заметно уступающую алловидам “дикоживущих” мышей, генетическую обособленность подвидов M. m. domesticus, M. m. musculus, M. m. castaneus, M. m. gentilulus, M. m. bactrianus, нового подвида M. m. isatissus и неклассифицированной формы из Непала [7, 8, 16, 17]. В то же время, несмотря на довольно высокую изменчивость митохондриальных генов, подвиды M. m. musculus, M. m. wagneri и M. m. gansuensis не сформировали отдельных клад на дендрограммах [7, 16–18]. Выделение субкластеров, которые зачастую включали мышей, происходящих из весьма удаленных регионов и относящихся к разным подвидам, имело, по-видимому, случайный характер. Предполагалось замещение мтДНК у подвидов M. m. wagneri и M. m. gansuensis мтДНК M. m. musculus вследствие поглотительной гибридизации [16]. Ввиду ограниченности генетических данных, свидетельствующих в пользу обособленности M. m. wagneri и M. m. gansuensis как по отношению друг к другу, так и к M. m. musculus, в сводке по млекопитающим мировой фауны [3] первые два подвида признаны синонимичными последнему.
Изучение разнообразия, филогенетических отношений и гибридизации домовых мышей по-прежнему основано на использовании, главным образом митохондриальных генов, эволюционирующих гораздо быстрее, чем ядерные [19–21]. По-видимому, по этой причине ядерные гены исследовали (за исключением нескольких упомянутых работ) при сравнении видов домовых мышей и наиболее отличающихся подвидов M. musculus на ограниченном количестве животных [13, 22–24]. Однако анализ мтДНК, имеющей иной тип наследования (передающейся потомкам через цитоплазму яйцеклеток, т.е. по материнской линии) и не рекомбинирующей ни с другими молекулами мтДНК, ни с яДНК, способен выявить смешанный состав популяции, но не позволяет без привлечения ядерных маркеров определить гибридное происхождение отдельных особей. Таким образом, несовпадение результатов их идентификации по кариотипическим, ядерным маркерам, с одной стороны, и митохондриальным, с другой, не удивительно и даже ожидаемо, особенно в гибридных зонах. В этом отношении необходимым представляется анализ наиболее быстро эволюционирующих ядерных аутосомных генов, и первостепенное значение приобретает подбор подходящего ядерного маркера, обладающего специфичностью у всех или хотя бы у большинства внутривидовых форм M. musculus (что необходимо для изучения их гибридизации), и в то же время достаточной популяционной изменчивостью для анализа генетического полиморфизма.
Основная цель настоящей работы – исследование генетического разнообразия домовых мышей из нескольких популяций Европы, Закавказья, Средней Азии и Сибири по нуклеотидной последовательности фрагмента экзона 11 аутосомного ядерного гена Brca1 (Breast Cancer 1) и оценка пригодности данного маркера для анализа филогенетических отношений и гибридизации внутривидовых форм M. musculus. Помимо гена Brca1, у части особей нами проведено секвенирование фрагмента митохондриального гена первой субъединицы цитохромоксидазы (Cox1) с целью определения уровня полиморфизма данного маркера в группе домовых мышей, уточнения генетических различий, выявленных при анализе гена Brca1, и дополнительной проверки гибридного происхождения некоторых экземпляров.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
В общей сложности нами исследовано 40 особей M. musculus, принадлежащих, по крайней мере, пяти подвидам – M. m. musculus, M. m. wagneri, M. m. gansuensis, M. m. domesticus и M. m. castaneus. Еще две македонские мыши (M. macedonicus) и одна курганчиковая (M. spicilegus) использованы в качестве аутгрупп. Пункты отлова мышей, их коллекционные номера, состав выборок, в которых изучены гены Brca1 и Cox1, номера нуклеотидных последовательностей, депонированных в базу GenBank, представлены в табл. 1. Помимо собственного материала, в анализ включены ранее опубликованные [25–28] последовательности гена Brca1 лабораторных мышей линий C57BL/6 и C57BL/6j (GenBank accession numbers: XM_017314233, BC068303, NM_009764, U35641, U31625), которые происходят от западноевропейских домовых мышей M. m. domesticus и содержат в геноме около 80% генов данного подвида [29, 30].
Таблица 1.
Материал из собственных коллекций, использованный в работе
| Вид | Коллекционный номер | Пункт отлова | GenBank accession number | |
|---|---|---|---|---|
| Brca1 | Cox1 | |||
| M. musculus | SBP1 | 1. Италия, Granica | MN228628 | MN228588 |
| SBP14 | 2. Италия, Collebaccaro | MN228629 | MN228589 | |
| 26258 | 3. Украина, окр. г. Киев | MN228603 | MN228581 | |
| 26637 | 4. Белоруссия, Минская обл., Воложинский р-н, окр. пос. Саковщина и Калдыки | MN228622 | – | |
| 26643 | MN228616 | – | ||
| 26920 | 5. Россия, Московская обл., Ногинский р-н, окр. пос. Ивановское | MN228604 | – | |
| 26921 | MN228617 | – | ||
| 24133 | 6. Россия, Курская обл., окр. г. Курск, пос. Заповедный | MN228598 | MN228578 | |
| 26071 | 7. Россия, Краснодарский край, Темрюкский р-н, окр. пос. Приморский | MN228615 | – | |
| 26072 | MN228602 | – | ||
| 3 | 8. Россия, Астраханская обл., 45 км к югу от г. Астрахань, дельта р. Волга | MN228605 | – | |
| 5 | MN228609 | – | ||
| 100 | 9. Армения, г. Ереван | MN228630 | – | |
| 126 | MN228631 | MN228590 | ||
| 240 | MN228632 | MN228591 | ||
| 24980 | 10. Азербайджан, Ленкоранский р-н, окр. пос. Даштатюк (бывш. Алексеевка) | MN228634 | – | |
| 24981 | MN228614 | – | ||
| 406 | 11. Казахстан, Западно-Казахстанская обл., пойма р. Есенанкаты | MN228606 | – | |
| 413 | 12. Казахстан, Западно-Казахстанская обл., Акжаикский р-н, Тайпакский сельский округ, зимовка Шауеш | MN228607 | – | |
| 24634 | 13. Россия, Тюменская обл., Ямало-Ненецкий АО, г. Лабытнанги | MN228600 | MN228579 | |
| 24635 | MN228601 | MN228580 | ||
| 16 | 14. Россия, Тюменская обл., г. Ишим | MN228633 | – | |
| 19 | MN228623 | – | ||
| 24223 | 15. Россия, Новосибирская обл., окр. г. Новосибирск в районе Академгородка, 1 км к северу от дер. Кирово | MN228610 | – | |
| 24227 | 16. Россия, Алтайский край, Солтонский р-н, пос. Солтон | MN228611 | – | |
| 24228 | MN228599 | – | ||
| 418 | 17. Россия, Забайкальский край, г. Чита | MN228624 | – | |
| 424 | 18. Россия, Забайкальский край, Ононский р-н, пос. Большевик. Даурский заповедник | MN228625 | MN228586 | |
| 425 | MN228626 | MN228587 | ||
| 437 | 19. Россия, Забайкальский край, Ононский р-н, окр. пос. Нижний Цасучей, Цасучейский бор | MN228627 | – | |
| 447 | 20. Россия, Забайкальский край, Ононский р-н, пос. Усть-Ималка | MN228608 | MN228582 | |
| 24245 | 21. Казахстан, Восточно-Казахстанская обл., Тарбагатайский р-н, западное побережье оз. Зайсан | MN228619 | – | |
| 24279 | MN228612 | – | ||
| 24295 | 22. Казахстан, Алматинская обл., Балхашский р-н, 8 км от пос. Баканас в сторону пос. Акколь | MN228613 | – | |
| 24330 | 23. Казахстан, Кызылординская обл., Чиилийский р-н, левый берег р. Сырдарья в 1.5 км ниже по течению от пос. Тартогай | MN228620 | MN228584 | |
| 24383 | 24. Туркменистан, Балканский велаят, Сюнт-Хасардагский заповедник. Хребет Копетдаг | MN228621 | MN228585 | |
| 25559 | 25. Таджикистан, Рогунский р-н, окраина пос. Оби Гарм | MN228618 | MN228583 | |
| M. musculus | 1214 | 26. Индия, штат Химачал-Прадеш, долина Кулу, пос. Наггар | MN228637 | MN228594 |
| 1140 | 27. Индия, штат Сикким, Maenam Hill | MN228636 | MN228593 | |
| 26925 | 28. Иран, остан Бахтиария, Kohne Pist | MN228635 | MN228592 | |
| M. macedonicus | 26922 | 29. Иран, остан Казвин, окр. г. Буин-Захра, пос. Фейзабад | MN228639 | MN228596 |
| 26923 | MN228640 | MN228597 | ||
| M. spicilegus | K-14 | 30. Молдавия, окр. г. Кишинёв, г. Крикова | MN228638 | MN228595 |
Суммарную ДНК выделяли фенол-хлороформным методом после обработки измельченных тканей протеиназой К [31] или при использовании набора реагентов GeneJET™ (“Fermentas”, США). Нуклеотидную последовательность фрагмента экзона 11 гена Brca1 определяли путем секвенирования трех перекрывающихся участков по отдельности. При анализе гена Cox1 секвенировали два перекрывающихся участка. Предварительное клонирование фрагментов обоих генов не проводили. Праймеры для полимеразной цепной реакции (ПЦР) и секвенирования перечислены в табл. 2. ПЦР проводили в смеси, содержащей 25–35 нг ДНК, 2 мкл 10-кратного Taq-буфера, 1.6 мкл 2.5 мM раствора dNTP, 4 пM каждого праймера, 1 ед. Taq-полимеразы и деионизированную воду до конечного объема 20 мкл. Амплификация выполнена на приборе ТЕРЦИК (Россия) по следующей программе: предварительный прогрев при 94°С (3 мин), далее 35 циклов в последовательном режиме 30 с – 94°С, 1 мин – 55–57°С (при амплификации фрагментов II и III гена Brca1, а также фрагмента II гена Cox1), 60°С (при амплификации фрагмента I гена Brca1) или 63°С (при амплификации фрагмента I гена Cox1), 1 мин – 72°С; в завершение реакции проводили однократную элонгацию ПЦР-продуктов при 72°С (6 мин). Автоматическое секвенирование осуществлено с использованием набора ABI PRISM®BigDyeTM Terminator v. 3.1 (“ABI”, США) в Институте биологии развития им. Н.К. Кольцова РАН на генетическом анализаторе AB 3500 (“ABI”, США).
Таблица 2.
Праймеры, использованные для проведения ПЦР и секвенирования экзона 11 ядерного гена Brca1 и митохондриального гена Cox1
| Ген | Фрагмент | Последовательности прямого (верхняя строка) и обратного (нижняя строка) праймеров, 5'–3' | Источник |
|---|---|---|---|
| Brca1, экзон 11 | I | BRCA1-1F (GGCATAAGAAACATTGAACATCATCA) | [32] |
| BRCA1-M1R (TTTCTTCACTGCTACCACAACTAT) | Подобран нами | ||
| II | BRCA1-M2F (ACGTCCACAGTTCAAAAGCACCTA) | ||
| BRCA1-M2R (TGAAAATAAAGCAAATGACTGAC) | |||
| III | BRCA1-M3F (GTGTATGACTCAGTTTATCGC) | ||
| BRCA1-3R (TTAGGCCCTCTGTTTCTACCTAG) | [32] | ||
| Cox1 | I | L5310 (CCTACTCAGCCATTTTACCTATG) | [33] |
| R6036 (ACTTCTGGGTGTCCAAAGAATCA) | |||
| II | L5850-Mus (TAACACAATATCAAACTCCA) | Подобран нами | |
| R6560-Mus (GAAGAATGTTATGTTTACTCCTA) |
После сложения и выравнивания нуклеотидных последовательностей получены и проанализированы фрагменты гена Brca1 размером 2331 и 2328 п.н. (с делецией). Начало исследованного участка приходится на первый нуклеотид кодона 254 транслируемой части гена. Это белоккодирующий на всем протяжении фрагмент, соответствующий последовательности из 776–777 аминокислотных остатков у разных экземпляров. Исследованный участок гена Cox1 состоял из 1260 п.н., начиная с его первого кодона.
Статистическую обработку данных выполняли с помощью компьютерной программы Mega 6.06 [34]. Выбор подходящей модели нуклеотидных замен производили по показателю BIC (Bayesian Information Criterion). При анализе фрагмента гена Brca1 использовали модель TN93 (Tamura-Nei model) для построения дендрограммы по методу Maximum Likelihood (ML) и расчета генетических дистанций (D), а опцию “partial deletion” – для учета нуклеотидов, утраченных в результате делеций, как отдельных признаков, аналогично заменам. При анализе фрагмента гена Cox1 применяли модель T92 + G (Tamura 3-parameter model with gamma distributed). Значения бутстреп-индекса определены по 1000 репликаций.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Уровень изменчивости исследованного нами участка гена Brca1 оказался, в целом, высоким. Существенные различия выявлены не только между видами домовых мышей, особенно между “дикоживущими” M. macedonicus, M. spicilegus и синантропным M. musculus, но и между разными популяциями последнего вида, а в ряде случаев – даже между особями одной и той же популяции и линии, что неожиданно для ядерного гена. Таким образом, у M. musculus дифференциация по гену Brca1 имеет сложный, многоуровневый характер.
На первом, более “глубоком”, уровне наблюдается отчетливое разделение генотипов M. musculus на пять групп (I–V) по целому ряду фиксированных замен. Например, группы I и II отличаются друг от друга по шести транзициям и двум трансверсиям, еще по двум транзициям ясно прослеживается “сортировка” генотипов, но она не является полной. Группа III в сравнении с группой II характеризуется тремя фиксированными транзициями, еще по двум трансверсиям наблюдается неполная “сортировка” генотипов. Общая особенность генотипов, относящихся к группам I и II – делеция трех нуклеотидов подряд в последней четверти исследованного фрагмента гена Brca1, что ведет к элиминации из полипептидной цепи одного аминокислотного остатка (глутаминовой кислоты) и замене предыдущего (фенилаланина на лейцин).
Помимо особей, гомозиготных по фиксированным в разных группах заменам, обнаружены и гетерозиготные особи, причем по всем маркерным локусам одновременно. Этот результат указывает на существование в пределах вида M. musculus нескольких генетически разобщенных группировок, а экземпляры, гетерозиготные по всем маркерным локусам, могут быть определены как гибридные. У большей их части (№№ 5, 16, 24223, 24227, 24279, 24295, 24980, 24981, 26071, 26643, 26921) выявлено сочетание гаплотипов, обычных для групп I, II и III, однако у нескольких гибридных особей найдены и уникальные. Наиболее пристального внимания заслуживают две мыши. Мышь № 26925 из остана Бахтиария (Иран) была наиболее близка к животным группы III, однако в последней четверти исследованного фрагмента гена Brca1 у нее обнаружено в гетерозиготном состоянии пять замен, уникальных для всей выборки M. musculus. У экземпляра № 447 из пос. Усть-Ималка Забайкальского края, как и у животных из соседних пунктов, прослеживаются все маркерные замены, свойственные мышам группы II; однако у этой особи выявлено в гетерозиготном состоянии семь из восьми замен, характеризующих группу I, а также четыре уникальных замены и трехнуклеотидная вставка, свойственная мышам групп III–V. Уникальные гаплотипы нескольких гетерозиготных экземпляров, гипотетически определенные при удалении типичного для группы II или III гаплотипа из комбинированной “гибридной” последовательности, были использованы нами для последующего анализа.
Дендрограмма, представленная на рис. 1, показывает различия и филогенетические отношения между видами домовых мышей и популяционными группировками M. musculus по гену Brca1 с учетом уникальных гаплотипов, обнаруженных у нескольких гибридов. Последние сформировали отдельные ветви, примыкающие к группам I (I-A, экз. № 447) и III (III-A, экз. № 26925). С высокой вероятностью можно допустить, что добавление в анализ подобных последовательностей приведет к формированию на дендрограмме дополнительных кластеров. Дендрограмма, как и значения генетических дистанций (D = 0.002–0.009, в среднем 0.005) (табл. 3), наглядно показывает высокий уровень изменчивости гена Brca1 в пределах вида M. musculus и дифференциацию его популяционных группировок. Примечательно, что средняя генетическая дистанция между ними сопоставима с дистанцией между македонскими и курганчиковой мышами (0.005), а максимальная – почти в 2 раза превышает уровень различий между M. macedonicus и M. spicilegus.
Второй уровень изменчивости и дифференциации M. musculus представлен популяционными различиями, особенно выраженными внутри кластера III: средняя генетическая дистанция в данной группе достигает значения 0.002. Разные генотипы гена Brca1 обнаружены даже у мышей одной и той же линии (C57BL/6 или C57BL/6j), о чем уже упоминалось. Кластеры I и II имеют более компактный вид, хотя и здесь в одном случае отмечена внутрипопуляционная изменчивость (№№ 24634, 24635 из г. Лабытнанги, группа I).
Рис. 1.
ML-дендрограмма, построенная по результатам сравнения фрагмента экзона 11 гена Brca1 (2331 п.н.) отдельных особей M. musculus, M. spicilegus, M. macedonicus из природных популяций, а также лабораторных мышей линий C57BL/6 и C57BL/6j. Для филогенетической реконструкции использованы все гомозиготные и гетерозиготные по единичным, недиагностическим заменам особи; во втором случае из двух нуклеотидов гетерозиготного локуса выбран наиболее часто встречающийся в общей выборке мышей. Гетерозиготные по ряду нуклеотидных замен (гибридные) экземпляры исключены из анализа, кроме № 447 и № 26925 (при построении дендрограммы использованы их гипотетически определенные уникальные гаплотипы), а также № 16 и № 24980 (использован гаплотип группы III); гибридные мыши отмечены звездочкой. В узлах ветвления дендрограммы указаны значения бутстреп-индекса, превышающие 70%, а справа от ветвей – коллекционные номера животных.
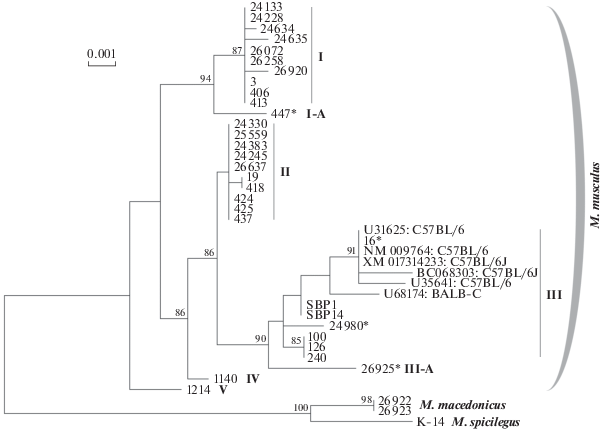
Таблица 3.
Значения средних генетических дистанций (D), рассчитанных при сравнении нуклеотидных последовательностей фрагмента (2331 п.н.) ядерного гена Brca1 домовых мышей пяти популяционных групп, а также курганчиковых и македонских мышей
| Вид | Группа | Значения генетических дистанций | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| M. musculus | M. spicilegus | |||||||||
| I (0.000) |
I-A | II (0.000) |
III (0.002) |
III-A | IV | V | Вид в целом (0.005) |
|||
| M. musculus | I | — | ||||||||
| I-A | 0.003 | — | ||||||||
| II | 0.005 | 0.005 | — | |||||||
| III | 0.008 | 0.009 | 0.004 | — | ||||||
| III-A | 0.008 | 0.009 | 0.005 | 0.005 | — | |||||
| IV | 0.004 | 0.005 | 0.002 | 0.005 | 0.006 | — | ||||
| V | 0.005 | 0.006 | 0.005 | 0.007 | 0.007 | 0.004 | — | |||
| M. spicilegus | 0.022 | 0.023 | 0.023 | 0.025 | 0.025 | 0.021 | 0.021 | 0.023 | — | |
| M. macedonicus | 0.021 | 0.021 | 0.021 | 0.023 | 0.023 | 0.020 | 0.019 | 0.022 | 0.005 | |
Примечание. В скобках указаны средние внутригрупповые или внутривидовые дистанции (для выборок, представленных более чем одним экземпляром). При расчете дистанций использованы те же выборки, генотипы и гаплотипы, что и при построении ML-дендрограммы (см. рис. 1).
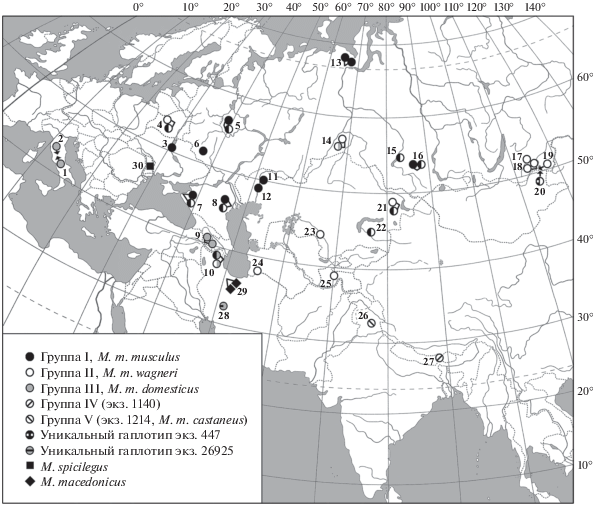
На рис. 2 представлено распространение в природных популяциях домовых мышей выявленных генотипов Brca1. В целом у M. musculus прослеживается их отчетливая приуроченность к определенным регионам: например, генотипы группы I преимущественно отмечены у особей из популяций Восточной Европы и Северного Зауралья, группы II – у мышей Средней Азии и Сибири, группы III – у животных Западной Европы и Закавказья, групп IV и V – у экземпляров из Индии. Тем не менее, в ряде случаев животные с тем или иным генотипом или гаплотипом были отловлены на слишком большом расстоянии от области его основного распространения. Так, гаплотип группы II обнаружен у мышей (главным образом гибридных) в отдельных пунктах Белоруссии и Московской обл., группы III – у одной мыши из г. Ишим Тюменской обл., группы I – у мышей, отловленных в окрестностях оз. Зайсан и Балхаш (Восточный Казахстан), т.е. наблюдается значительное взаимопроникновение популяционных группировок M. musculus, выявленных при анализе гена Brca1.
Рис. 2.
Распространение домовых мышей с разными генотипами Brca1 в исследованных природных популяциях. Каждый значок соответствует одному экземпляру; комбинированные значки обозначают гетерозиготных (гибридных) мышей с разными гаплотипами. Нумерацию пунктов отлова животных см. в табл. 1.
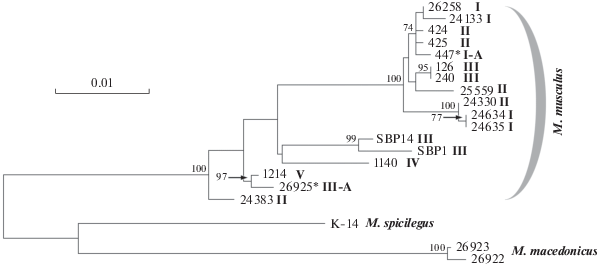
Это могло быть причиной не вполне успешного определения подвидовой принадлежности домовых мышей по митохондриальным генам [7, 16–18]. Для проверки данной гипотезы мы проанализировали изменчивость фрагмента митохондриального гена Cox1 у особей M. musculus, определенных как гомозиготные (“чистые”) представители групп I–V по ядерному гену Brca1 и происходящих из регионов, удаленных от зон гибридизации. В анализ были добавлены лишь гибридные особи № 447 и № 26925 с уникальными гаплотипами яДНК. Несмотря на это, дендрограмма, построенная при сравнении нуклеотидных последовательностей фрагмента гена Cox1 домовых мышей (рис. 3), примерно соответствовала дереву, полученному при исследовании гена Brca1, лишь в плане межвидовой дифференциации. В пределах же вида M. musculus топология деревьев, состав кластеров, а также масштабы различий между ними были кардинально иными. Прежде всего, у бóльшей части особей, по генотипам гена Brca1 принадлежащих к разным внутривидовым группировкам (I, II и частично III), выявлены достаточно близкие митотипы гена Cox1, объединившиеся на дереве в один кластер, без отчетливой упорядоченности. Кроме того, очевидны существенные различия в характере филогенетических отношений домовых мышей M. musculus, особенно тех, которые находятся в основании дендрограмм. Если по гену Brca1 базальное положение занимали особи №№ 1140 и 1214 (относящиеся к группам IV и V соответственно), то по гену Cox1 наиболее древними являются митотипы мышей № 26925 (группа III-A) и № 24383 (группа II). И наконец, уровень различий между некоторыми особями по генам Cox1 и Brca1 был прямо противоположным. Так, особи №№ 24634 и 24635 из г. Лабытнанги, как уже упоминалось, отличаются по генотипам гена Brca1, но имеют одинаковый митотип гена Cox1; обратная ситуация наблюдается у мышей SBP1 и SBP14 из Италии, принадлежащих к разным хромосомным формам (2n = 40 и 2n = 22 соответственно).
Рис. 3.
ML-дендрограмма, построенная по результатам сравнения нуклеотидных последовательностей фрагмента митохондриального гена Cox1 (1260 п.н.) отдельных особей M. musculus, M. spicilegus, M. macedonicus из природных популяций. Из анализа исключены гетерозиготные (гибридные) особи, выявленные при исследовании изменчивости гена Brca1, кроме № 447 и № 26925 (обозначены звездочкой). В узлах ветвления дендрограммы указаны значения бутстреп-индекса, превышающие 70%, а справа от ветвей – коллекционные номера животных и группа, к которой они относились по гену Brca1 (римская цифра).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Секвенирование участков протяженного экзона 11 ядерного гена Brca1 активно проводится в последнее время с целью анализа филогенетических связей видов и родов в разных группах млекопитающих [32, 35, 36]. Согласно полученным нами данным, в группе домовых мышей ген Brca1 проявляет не только высокий межвидовой, но и внутривидовой и даже внутриподвидовой полиморфизм. Характер изменчивости и распространения генотипов гена Brca1 указывает на наличие в пределах вида M. musculus нескольких дискретных генетических группировок, которые сформировались в условиях длительной изоляции, накопив за это время ряд характерных для каждой из них мутаций. Однако впоследствии эти группировки широко расселились (возможно, при участии человека) и вступили в гибридизацию в зонах контакта.
Генотипы группы I выявлены, главным образом, у домовых мышей из популяций Восточной Европы и Северного Зауралья, т.е. свойственны подвиду M. m. musculus в самом узком его понимании. Группа III включает генотипы мышей из Италии, несомненно принадлежащих подвиду M. m. domesticus, особей линий C57BL/6 и C57BL/6j, также происходящих от последнего подвида, и мышей из Армении. Принимая во внимание сведения о наличии в Закавказье широкой гибридной зоны между M. m. domesticus и M. m. musculus [12, 37–41], представляется неудивительным объединение генотипов мышей линий C57BL/6 и C57BL/6j вместе с мышами из Италии и Армении в один кластер. Очевидно, что генотипы группы III, несмотря на ее гетерогенность, имеют общие корни и характерны для подвида M. m. domesticus. Генотипы, составляющие кластер II, выявлены преимущественно у мышей Средней Азии (подвид M. m. wagneri) и юга Сибири, включая Забайкалье, откуда происходили животные №№ 424, 425, 437, 447, сходные по внешним признакам с представителями M. m. gansuensis. Таким образом, можно предполагать, что генотипы группы II свойственны этим двум подвидам. То обстоятельство, что группа II связана более тесным родством с группой III (с M. m. domesticus), а не с группой I (M. m. musculus), противоречит мнению о синонимичности M. m. wagneri, M. m. gansuensis и M. m. musculus [3].
Мыши №№ 1140 и 1214, которые сформировали на дендрограмме отдельные базальные ветви IV и V соответственно, происходят из Индии, входящей в область распространения подвида M. m. castaneus. Однако принадлежность к этому подвиду обеих указанных особей сомнительна, поскольку генетическая дистанция между ними относительно высока (D = 0.004, см. табл. 3), а ветви IV и V исходят из разных узлов дерева. При использовании функции BLAST установлено, что ген Cox1 мыши № 1140 менее сходен (98.5%) с депонированными в GenBank последовательностями мтДНК представителей подвида M. m. castaneus (KF781663, KF781664), чем Cox1 экземпляра № 1214 (99.29%). Таким образом, особь № 1214 предварительно можно рассматривать как принадлежащую к указанному подвиду, а мышь № 1140 – как представителя некого неидентифицированного подвида или генетической формы. Северная Индия, где установлен высокий анцестральный полиморфизм популяций синантропных домовых мышей, считается их родиной [5, 38]. Полученные нами сведения подтверждают это предположение.
Гибридные экземпляры обнаружены, в основном, в зонах контактов подвидов и содержат соответствующие им гаплотипы. Так, гибриды M. m. wagneri и M. m. musculus отмечены в нескольких пунктах Прикаспия и южных областей Западной Сибири, гибриды M. m. wagneri и M. m. domesticus, а также M. m. musculus и M. m. domesticus – в Закавказье, т.е. как раз там, где их следовало ожидать (рис. 2). Несколько случаев выявления особей, преимущественно гибридных, с нехарактерным для популяций данной территории гаплотипом можно объяснить завозом животных с транспортом или инвазией в природные популяции лабораторных мышей, судя по идентичности одного из гаплотипов гибридного экземпляра № 16 из г. Ишим и варианта гена Brca1 нескольких особей линий C57BL/6 и C57BL/6j (рис. 1).
Как уже отмечалось, гипотетически определенный уникальный гаплотип гибридного экземпляра № 447 из Забайкальского края (ветвь I-A) имеет четыре замены, не обнаруженные более ни у одной другой исследованной мыши. По-видимому, эти мутации появились в недавнем прошлом, так как они специфичны лишь для животных некой отдельной генетической формы. С другой стороны, этот же гаплотип содержит три добавочных нуклеотида, которые характерны для более древних групп III–V, но отсутствуют в вариантах гена Brca1, составляющих группы I и II, соседние и самые близкие по отношению к ветви I-A. Данная особенность позволяет охарактеризовать уникальный гаплотип особи № 447 как “пра-musculus”. Не исключено, что он является характерным, хотя и не абсолютным, признаком подвида M. m. gansuensis и впоследствии будет обнаружен у части других его представителей. Принадлежность к какому-либо подвиду или генетически дискретной форме M. musculus уникального гаплотипа особи № 26925 (ветвь III-A), который отличается пятью заменами, не обнаруженными более ни у одной другой мыши, можно определить двояко – либо как один из уклоняющихся вариантов гена Brca1 в пределах подвида M. m. domesticus (принимая во внимание его высокую генетическую гетерогенность), либо как гаплотип, характеризующий какой-нибудь другой, близкий к M. m. domesticus подвид, например M. m. bactrianus. Для разрешения этой дилеммы необходимо исследование дополнительного материала из Южной Азии.
Наибольшим разнообразием генотипов Brca1 отличаются популяции домовых мышей Каспийского региона, что согласуется с гипотезой о его близости к центру происхождения рода Mus и расположении на пути древних миграций предшественников современных подвидов [38].
5'-Концевая область митохондриального гена Cox1 (около 700 п.н. от его начала) считается хорошим видоспецифичным маркером и активно используется при “ДНК-паспортизации” видов и построении филогенетических реконструкций [42]. При исследовании близкой группы мышей (лесные мыши рода Sylvaemus) анализ полиморфизма гена Cox1 позволил успешно дискриминировать не только их виды, но и генетически близкие внутривидовые группировки [43]. Однако даже достаточно протяженный фрагмент Cox1, составляющий более трех четвертей полного гена, оказался малопригодным для идентификации особей подвидов M. m. musculus, M. m. wagneri, M. m. gansuensis, а также M. m. domesticus из Закавказья, хотя они и были “чистыми” по ядерному гену Brca1. Таким образом, наши результаты анализа гена Cox1 соответствуют данным других авторов, изучавших митохондриальные гены D-loop, Cytb [7, 16–18], и могут объясняться либо стабилизирующим отбором мтДНК у домовых мышей подвидов M. m. musculus, M. m. wagneri и M. m. gansuensis, либо ее масштабной интрогрессией от какого-либо одного подвида в генофонд других вследствие гибридизации [16]. Следует отметить, что присутствие чужеродной мтДНК прослеживается в нашей выборке у некоторых особей с хорошо различающимися митотипами гена Cox1 и наилучшим образом объясняет различия в топологии дендрограмм, построенных по ядерному и митохондриальному генам. Так, экземпляр № 24383, “чистый” представитель подвида M. m. wagneri по гену Brca1 (группа II), имеет митотип Cox1, весьма близкий к митотипу мыши № 1214 и подвида M. m. castaneus. Сходный митотип Cox1 имеет и экземпляр № 26925, показавший большее сходство с особями подвида M. m. domesticus по яДНК.
Таким образом, результаты проведенного нами исследования показывают, что ядерный ген Brca1 является весьма перспективным молекулярно-генетическим маркером для изучения изменчивости, дифференциации и гибридизации домовых мышей, а также для определения их подвидовой принадлежности. Ген Cox1, эволюционирующий быстрее гена Brca1, оказался, тем не менее, малопригодным для дискриминации особей M. m. musculus, M. m. wagneri, M. m. gansuensis и закавказских представителей M. m. domesticus вследствие интрогрессии и длительного сохранения в популяциях чужеродной мтДНК. Однако анализ гена Cox1 (наряду с диагностикой животных по яДНК) может быть полезным при оценке популяционных различий в пределах подвидов M. m. castaneus и M. m. domesticus.
Авторы выражают искреннюю признательность О.В. Брандлеру (Институт биологии развития РАН), А.Ю. Пузаченко (Институт географии РАН), С.Б. Поле (Казахский научный центр карантинных и зоонозных инфекций), А.А. Власову (Центрально-Чернозeмный заповедник), Е.И. Жолнеровской (Институт систематики и экологии животных СО РАН), В.И. Ермолаеву (Институт цитологии и генетики СО РАН), В.Г. Штро (Стационар Института экологии растений и животных УрО РАН, г. Лабытнанги) и А.С. Саидову (Институт зоологии и паразитологии Академии наук Таджикистана) за содействие, оказанное при отлове мышей и лабораторной обработке материала, а также Д.М. Щепетову и В.Г. Тамбовцевой (Институт биологии развития РАН) за помощь при проведении молекулярно-генетического анализа. Часть животных любезно передана нам В.С. Лукаревским и Т.А. Мироновой (Институт проблем экологии и эволюции РАН), А.Ю. Левых (Ишимский государственный педагогический институт), Ю.A. Баженовым (Заповедник “Даурский”), А.А. Банниковой (Московский государственный университет, биологический факультет), А.Е. Зыкову (г. Киев), Н.Е. Бурко, И.С. Садовничей (г. Минск).
Работа выполнена в рамках Государственных заданий Института биологии развития им. Н.К. Кольцова РАН (№ 0108-2019-0007, “Молекулярно-генетические и экологические механизмы видообразования и ранних этапов эволюции. Разработка подходов для оценки гомеостаза развития биологических систем (методология популяционной биологии развития)”), Института проблем экологии и эволюции им. А.Н. Северцова РАН (№ 0109-2018-0073, “Эволюционная морфология и микроэволюция позвоночных животных”) и Зоологического института РАН (№ AAAA-A17-117022810195-3, “Филогения, морфология и систематика плацентарных млекопитающих”).
Все процедуры, проведенные с участием животных, соответствовали этическим стандартам учреждений или принятой практике таких исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Bonhomme F., Catalan J., Britton-Davidian J., Chapman V.M., Moriwaki K., Nevo E., Thaler L. (1984) Biochemical diversity and evolution in the genus Mus. Biochem. Genet. 22, 275–303.
Boursot P., Auffray J.-C., Britton-Davidian J., Bonhomme F. (1993) The evolution of house mice. Annu. Rev. Ecol. Systematics. 24, 119–152.
Musser G.G., Carleton M.D. (2005) Superfamily Muroidea. Mammal species of the World: a taxonomic and geographic reference, third ed. Eds Wilson D.E., Reeder D.M. Baltimore: Johns Hopkins Univ. Press, pp. 894–1531.
Mouse Genome Sequencing Consortium (2002) Initial sequencing and comparative analysis of the mouse genome. Nature. 420, 520–562.
Din W., Anand R., Boursot P., Darviche D., Dad B., Jouvin-Marche E., Orth A., Talwar G.P., Cazenave P.-A., Bonhomme F. (1996) Origin and radiation of the house mouse: clues from nuclear genes. J. Evol. Biol. 9, 519–539.
Guénet J.-L., Bonhomme F. (2003) Wild mice: An ever-increasing contribution to a popular mammalian model. Trends Genet. 19, 24–31.
Suzuki H., Nunome M., Kinoshita G., Aplin K.P., Vogel P., Kryukov A.P., Jin M.-L., Han S.-H., Maryanto I., Tsuchiya K., Ikeda H., Shiroishi T., Yonekawa H., Moriwaki K. (2013) Evolutionary and dispersal history of Eurasian house mice Mus musculus clarified by more extensive geographic sampling of mitochondrial DNA. Heredity. 111, 375–390.
Hamid H.Sh., Darvish J., Rastegar-Pouyani E., Mahmoudi A. (2017) Subspecies differentiation of the house mouse Mus musculus Linnaeus, 1758 in the center and east of the Iranian plateau and Afghanistan. Mammalia. 81, 147–168.
Marshall J.T., Jr., Sage R.D. (1981) Taxonomy of the house mouse. Symp. Zool. Soc. London. 47, 15–25.
Sage R.D., Atchley W.R., Capanna E. (1993) House mice as models in systematic biology. Systematic Biology. 42, 523–561.
Marshall J.T., Jr. (1998) Identification and scientific names of Eurasian house mice and their European allies, subgenus Mus (Rodentia: Muridae). Springfield, Virginia: National Museum of Natural History, pp. 1–80.
Prager E.M., Orrego C., Sage R.D. (1998) Genetic variation and phylogeography of Central Asian and other house mice, including a major new mitochondrial lineage in Yemen. Genetics. 150, 835–861.
Geraldes A., Basset P., Gibson B., Smith K.L., Harr B., Yu H.-T., Bulatova N., Ziv Y., Nachman M.W. (2008) Inferring the history of speciation in house mice from autosomal, X-linked, Y-linked and mitochondrial genes. Mol. Ecol. 17, 5349–5363.
Спиридонова Л.Н., Челомина Г.Н., Мориваки К., Ионекава Х., Богданов А.С. (2004) Генетическое и таксономическое разнообразие домовых мышей азиатской части бывшего СССР. Генетика. 40, 1378–1388.
Якименко Л.В., Коробицына К.В., Фрисман Л.В., Мориваки К., Йонекава Х. (2003) Цитогенетика и систематика домовых мышей России и прилежащих стран. Проблемы эволюции. Т. V. Владивосток: Дальнаука, 62–89.
Спиридонова Л.Н. (2014) Интрогрессия маркеров ядерной и митохондриальной ДНК Mus musculus musculus в аборигенные популяции мышей Средней Азии (M. m. wagneri) и юга Сибири (M. m. gansuensis). Молекуляр. биология. 48, 89–98.
Suzuki H., Yakimenko L.V., Usuda D., Frisman L.V. (2015) Tracing the eastward dispersal of the house mouse, Mus musculus. Genes Environment. 37, 20.
Мальцев А.Н., Стахеев В.В., Богданов A.C., Фомина Е.С., Котенкова Е.В. (2015) Филогенетические взаимоотношения внутривидовых форм домовой мыши Mus musculus: анализ изменчивости контрольного региона (D-петли) митохондриальной ДНК. Докл. Акад. Наук. 465, 380–383.
Brown W.M., George M., Wilson A.C. (1979) Rapid evolution of animal mitochondrial DNA. Proc. Natl. Acad. Sci. USA. 76, 1967–1971.
Wilson A.C., Cann R.L., Carr S.M., George M., Gyllensten U.B., Helm-Bychowski K.M., Higuchi R.G., Palumbi S.R., Prager E.M., Sage R.D., Stoneking M. (1985) Mitochondrial DNA and two perspectives on evolutionary genetics. Biol. J. Linnean Soc. 26, 375–400.
Банникова А.А. (2004) Молекулярные маркеры и современная филогенетика млекопитающих. Журн. общей биологии. 65, 278–305.
Lundrigan B.L., Jansa S.A., Tucker P.K. (2002) Phylogenetic relationships in the genus Mus, based on paternally, maternally, and biparentally inherited characters. Syst. Biol. 51, 410–431.
Suzuki H., Shimada T., Terashima M., Tsuchiya K., Aplin K. (2004) Temporal, spatial, and ecological modes of evolution of Eurasian Mus based on mitochondrial and nuclear gene sequences. Mol. Phyl. Evol. 33, 626–646.
Tucker P.K., Sandstedt S.A., Lundrigan B.L. (2005) Phylogenetic relationships in the subgenus Mus (genus Mus, family Muridae, subfamily Murinae): examining gene trees and species trees. Biol. J. Linnean Society. 84, 653–662.
Abel K.J., Xu J., Yin G.-Y., Lyons R.H., Meisler M.H., Weber B.L. (1995) Mouse Brca1: localization sequence analysis and identification of evolutionarily conserved domains. Hum. Mol. Genet. 4, 2265–2273.
Sharan S.K., Wims M., Bradley A. (1995) Murine Brca1: sequence and significance for human missense mutations. Hum. Mol. Genet. 4, 2275–2278.
Mammalian Gene Collection (MGC) Program Team (2002) Generation and initial analysis of more than 15,000 full-length human and mouse cDNA sequences. Proc. Natl. Acad. Sci. USA. 99, 16899–16903.
Juvekar A., Hu H., Yadegarynia S., Lyssiotis C.A., Ullas S., Lien E.C., Bellinger G., Son J., Hok R.C., Seth P., Daly M.B., Kim B., Scully R., Asara J.M., Cantley L.C., Wulf G.M. (2016) Phosphoinositide 3-kinase inhibitors induce DNA damage through nucleoside depletion. Proc. Natl. Acad. Sci. USA. 113, E4338–E4347.
Yang H., Bell T.A., Churchill G.A., de Villena F.P.-M. (2007) On the subspecific origin of the laboratory mouse. Nat. Genet. 39, 1100–1107.
Yang H., Wang J.R., Didion J.P., Buus R.J., Bell T.A., Welsh C.E., Bonhomme F., Yu A.H.-T., Nachman M.W., Pialek J., Tucker P., Boursot P., McMillan L., Churchill G.A., de Villena F.P.-M. (2011) Subspecific origin and haplotype diversity in the laboratory mouse. Nat. Genet. 43, 648–655.
Sambrook J., Fritsch E.F., Maniatis T. (1989) Molecular cloning: a laboratory manual. N.Y.: Cold Spring Harbor Lab. Press, 1–398.
Adkins R.M., Gelke E.L., Rowe D., Honeycutt R.L. (2001) Molecular phylogeny and divergence time estimates for major rodent groups: evidence from multiple genes. Mol. Biol. Evol. 18, 777–791.
Nakamura I., Ohnuma A., Ichihashi T. (2008) Genbank Database (http://www.ncbi.nlm.nih.gov/nuccore/ 193248428)
Tamura K., Stecher G., Peterson D., Filipski A., Kumar S. (2013) MEGA6: Molecular Evolutionary Genetics Analysis version 6.0. Mol. Biol. Evol. 30, 2725–2729.
Банникова А.А., Сижажева А.М., Маликов В.Г., Голенищев Ф.Н., Дзуев Р.И. (2013) Генетическое разнообразие рода Chionomys (Mammalia, Arvicolinae) и сравнительная филогеография трех видов снеговых полевок. Генетика. 49, 649–664.
Rusin M., Lebedev V., Matrosova V., Zemlemerova E., Lopatina N., Bannikova A. (2018) Hidden diversity in the Caucasian mountains: an example of birch mice (Rodentia, Sminthidae, Sicista). Hystrix, Italian J. Mammalogy. 29, 61–66.
Межжерин С.В., Котенкова Е.В., Михайленко А.Г. (1994) Гибридные зоны. Домовая мышь. Происхождение, распространение, систематика, поведение. М.: Наука, 37–50.
Boursot P., Din W., Anand R., Darvich D., Dad B., Von Deimling F., Talwar G.P., Bonhomme F. (1996) Origin and radiation of the house mouse: mitochondrial DNA phylogeny. J. Evol. Biol. 9, 391–415.
Mezhzherin S.V., Kotenkova E.V., Mikhailenko A.G. (1998) The house mice, Mus musculus s.l., hybrid zone of Transcaucasus. Zeitshrift fur Zaugetierkunde. 63, 154–168.
Котенкова Е.В. (2002) Гибридизация синантропных видов домовых мышей и ее роль в эволюции. Успехи совр. биологии. 122, 580–593.
Милишников А.Н., Лавренченко Л.А., Лебедев В.С. (2004) Происхождение домовых мышей Закавказья (надвидовой комплекс Mus musculus). Новый взгляд на пути их расселения и эволюцию. Генетика. 40, 1234–1250.
Ratnasingham S., Hebert P.D.N. (2007) BOLD: The Barcode of Life Data System (www.barcodinglife.org). Mol. Ecol. Notes. 7, 355–364.
Богданов А.С., Стахеев В.В., Зыков А.Е., Якименко В.В., Малькова М.Г. (2012) Анализ генетической изменчивости и дифференциации лесных мышей рода Sylvaemus по результатам секвенирования фрагмента гена первой субъединицы цитохромоксидазы. Генетика. 48, 211–224.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


