Молекулярная биология, 2020, T. 54, № 3, стр. 435-444
Сложность обнаружения гомологичной рекомбинации, опосредованной CRISPR/Cas9, у Danio rerio
Y. Pi a, b, K. Z. He a, W. Q. Zhang a, Z. Q. Dong b, e, F. G. Jiang c, K. J. Jiang d, *, S. Guo a, b, **
a State Key Laboratory of Genetic Engineering, School of Life Sciences, Fudan University
200433 Shanghai, China
b Department of Bioengineering and Therapeutic Sciences, Programs in Human Genetics and Biological Sciences,
University of California
CA 94143-2811 San, Francisco, USA
c Department of Molecular and Cell Biology, University of California
CA 94720 Berkeley, USA
d East China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences
Shanghai, 200090China
e Huazhong Agricultural University
430070 Wuhan, China
* E-mail: jiangkj@ecsf.ac.cn
** E-mail: suguo@fudan.edu.cnsu.guo@ucsf.edu
Поступила в редакцию 28.02.2019
После доработки 30.09.2019
Принята к публикации 05.11.2019
Аннотация
Методы модификации генома, основанные на использовании гомологичных последовательностей, позволяют вносить направленные изменения в ДНК. Несмотря на множество успешных примеров подобного редактирования, эффективность модификации многих геномных локусов in vivo оставляет желать лучшего, а редактирование этих участков представляет собой сложную задачу. Осуществлена попытка встроить при помощи системы CRISPR/Cas9 репортерный ген GFP в геномный локус gad полосатого данио (Danio rerio). При отборе событий гомологичной рекомбинации мы детектировали артефакт ПЦР-амплификации, однако эта проблема была решена после оптимизации условий ПЦР и снижения количества вводимой в эмбрионы плазмиды. В этих оптимизированных условиях выявлено уменьшение в ходе эмбриогенеза количества эмбрионов, содержащих нужную вставку. Сигналы GFP также ослабевали на поздних стадиях развития, что соответствовало данным ПЦР-анализа. Как снижение сигналов GFP, так и выявление вставки с помощью ПЦР подтвердило потерю репортерного гена на поздних стадиях эмбриогенеза. Потеря вставки может объяснить, почему до сих пор не удалось получить трансгенные зародышевые линии D. rerio, содержащие GFP в локусе gad. Полученные нами результаты позволяют предположить, что низкая эффективность гомологичной рекомбинации, наблюдаемая в экспериментах по CRISPR-опосредованному встраиванию последовательностей в локус gad, частично может объясняться потерей вставки после интеграции в геном.
ВВЕДЕНИЕ
Аквариумная рыбка Danio rerio – модельный организм, на котором можно изучать функции генов in vivo, создавать модели болезней человека, а также проводить широкомасштабные генетические и химические скрининги [1, 2]. Разработаны методы химического и инсерционного мутагенеза, которые широко используются для нарушения функций генов [3, 4], однако эффективные методы обратной генетики для этого организма отсутствовали в течение долгого времени.
В последние годы для введения двухцепочечных разрывов ДНК в целевые геномные локусы D. rerio успешно использовали нуклеазы с мотивом цинковых пальцев (ZFN ‒ Zinc-finger nucleases) и нуклеазы TALE (TALEN ‒ Transcription activator-like effector nucleases). Эта манипуляция приводит к появлению делеции или инсерции в нужном локусе за счет репарации разрыва по механизму негомологичного соединения концов (NHEJ) [5‒7]. Более того, TALE-нуклеазы использовали для индукции репарации по механизму гомологичной рекомбинации в присутствии коротких олигонуклеотидов, гомологичных целевому локусу, либо плазмид, содержащих длинные участки гомологии [8, 9]. Однако использование нуклеаз обоих типов затруднено необходимостью каждый раз создавать ДНК-связывающий домен, специфичный к конкретной нуклеотидной последовательности. Фактически для каждой новой мишени нужно “собрать” новый уникальный белок.
CRISPR в последнее время зарекомендовал себя как простой и эффективный метод геномного редактирования [10, 11]. Для специфичного к последовательности расщепления ДНК система CRISPR типа II из бактерии Streptococcus pyogenes была модифицирована в двухкомпонентный инструмент, состоящий из нуклеазы Cas9 и так назывемой единой (химерной) гидовой РНК (single guide RNA, sgРНК). sgРНК содержит специфичную часть (любые 20 н., расположенные сразу перед мотивом PAM) и константную структурную часть [11, 12]. Таким образом, систему CRISPR можно легко нацелить на любой ген за счет подбора и синтеза sgРНК. Метод CRISPR широко используется для модификации генома модельных организмов посредством как NHEJ, так и направленного встраивания последовательностей по механизму гомологичной рекомбинации (HD knock-in) [13‒15].
Для мечения клеток определенного типа D. rerio обычно подбирают промоторные или энхансерные элементы, специфичные для клеток этого типа, и используют эти элементы для регуляции экспрессии репортерных генов в ходе трансгенеза, опосредованного Tol2. Этот подход имеет ряд недостатков. Во-первых, идентифицировать регуляторные элементы, способные управлять экспрессией репортерных генов с сохранением тканеспецифичного профиля экспрессии генов, достаточно сложно и требует много времени. Во-вторых, трансген встраивается в геном случайным образом, поэтому экспрессия трансгена во многом зависит от позиционного эффекта. Направленное встраивание последовательностей по механизму HD knock-in в этом смысле представляет собой идеальный метод мечения различных типов клеток. Тем не менее, суммарная эффективность этого метода остается очень низкой, несмотря на примеры успешного применения у данио [16, 17].
В представленной работе мы попытались встроить репортерный ген GFP в локус gad, который кодирует ключевой фермент, необходимый для синтеза ингибиторного нейромедиатора ГАМК. В противоположность NHEJ-опосредованному нарушению последовательности гена, при котором введение белка Cas9 было более эффективным, чем мРНК Cas9 [18], мы обнаружили, что при опосредованной Cas9 гомологичной вставке с последующей детекцией событий рекомбинации, результаты могут искажаться потенциальными артефактами ПЦР и потерей вставки с течением времени, что делает невозможным получение трансгенной зародышевой линии.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Животные. В работе использовали линию AB полосатого данио (D. rerio). Рыб разводили и содержали согласно стандартным протоколам [19]. Все манипуляции проводили в строгом согласии с правилами Центра животных ресурсов Фуданьского университета и университетского комитета по использованию животных. Исследование одобрено ведомственными протоколами. Все операции проводили с использованием анастезии трикаинметансульфонатом (E10521, “Aldrich”, Германия).
Получение sgРНК. Таргетную последовательность ДНК локуса gad для получения sgРНК подбирали при помощи доступных интернет-ресурсов [20, 21]. Изначально выбирали по две последовательности для каждого целевого сайта. ДНК-матрицу амплифицировали с плазмиды, содержащей структурную часть sgРНК. Использовали прямой праймер с Т7-промотором (5'-TAATACGACTCACTATAGGGAG-3') и универсальный обратный праймер, гомологичный 3'-концу структурной части (5'-AAAAAAAGCACCGACTCGGTGCCAC-3').
sgРНК против локуса tyr получали с использованием плазмиды pTyr-gRNA, предоставленной Wenbiao Chen [22]. Для приготовления sgРНК плазмиду pTyr-gRNA расщепляли BamHI. РНК получали с использованием набора MEGAshortscript T7 kit (“Life”) с последующей фенол‒хлороформной экстракцией и переосаждением sgРНК этиловым спиртом.
Tyr sgРНК (GGACTGGAGGACTTCTGGGG (AGG)), gad sgРНК-1 (TGAGATCGAGCGGCTCGGTC (AGG)) и gad sgРН-К2 (TTGGTCGCCTACGCGTTTAA (CGG)) получены при помощи транскрипции in vitro непосредственно с синтезированных олигонуклеотидов (набор MEGA short transcript synthesis kit, “Ambion”, США). Для использования в этой работе подобраны две sgРНК против локуса gad.
Получение мРНК Cas9 и белка Cas9. Рамка считывания Cas9, оптимизированная по кодонам для D. rerio, предоставлена Wenbiao Chen [22]. Для получения мРНК Cas9 ДНК-матрицу линеаризовали по XbaI и очищали на колонке QIAprep (“Qiagen”, США). РНК синтезировали при помощи транскрипции in vitro с использованием набора T3 mMESSAGE mMACHINE Kit (“Ambion”). Белок Cas9 предоставлен лабораторией J. Doudna (Калифорнийский университет в Беркли). Рекомбинантный белок Cas9 Streptococcus pyogenes, содержащий на С-конце два сигнала ядерной локализации SV40 (2×NLS) и N-концевой тэг His-MBP, нарабатывали в клетках Escherichia coli Bl21 (DE3) (“EMD Millipore”, Германия). His-MBP в дальнейшем удаляли с помощью TEV-протеазы, и полученный белок очищали по протоколу, описанному ранее [23]. Cas9 высокой степени очистки хранили при −80°С в буфере, содержащем 30 мM HEPES pH 7.5, 150 мM KCl, 10% глицерина, 0.5 мM Tрис-(2-хлорэтил)-фосфат (TCEP).
Получение векторов для встраивания по механизму гомологичной рекомбинации (HD knock-in). Основой для векторов, нацеленных на gad, послужил вектор pGEM-T easy. Плечо гомологии 5'-gad длиной 424 п.н. и плечо гомологии 3'-gad (1023 п.н.) амплифицировали методом ПЦР и клонировали в вектор pGEM-T easy вместе с последовательностью, кодирующей GFP, путем ПЦР-сборки. Последовательность PAM в составе итоговой плазмиды мутировали таким образом, чтобы она не распознавалась sgРНК. Праймеры для получения плазмиды приведены ниже:
Плечо гомологии 5'-gad F: GAATGATGAA-TCTTCATTTGCATAAAG,
Плечо гомологии 5'-gad R: CTCCGCTTCCCAGATCCTGACCGAGCCGCT;
Центр gad F: TCAGGATCTGGGAAGCGGAGCTACTAACTT,
Центр gad R: TATAGATGTTAAAAAACCTCCCACACCTCC;
Плечо гомологии 3'-gad F: gaggttttttAACATCTATAATAGAGGCTCATTGGTCG,
Плечо гомологии 3'-gad R: CATCGTTCCAAACACAACAGCT.
Микроинъекция. В одноклеточные эмбрионы D. rerio микроинъецировали смесь белка Cas9 с sgРНК или мРНК Cas9 с sgРНК (табл. 1). Для оценки эффективности выбранную sgРНК в концентрации от 30 до 200 нг/мкл и РНК Cas9 (1.1 мкг/мкл) смешивали с буфером для микроинъекции (рабочая концентрация sgРНК 30 нг/мкл). Для HD knock-in sgРНК и Cas9 вводили в виде мРНК или белка вместе с донорной плазмидой. Концентрация белка Cas9 ‒ 1.1 мкг/мкл, итоговая рабочая концентрация sgРНК ‒ 200 нг/мкл. В каждый эмбрион вводили 1‒2 нл cмеси. Для микроинъекции белка Cas9 использовали 20 мM HEPES-KOH-буфер, содержащий 30% глицерина, 0.1% NP40, 300 мM KCl, 0.5 мM дитиотреитол, 0.5 мM фенилметансульфонилфторид, pH 7.9. Рабочая концентрация белка приведена в табл. 1. В качестве контроля вводили донорную плазмиду. После инъекции эмбрионы инкубировали при температуре 28.5°C до определенных указанных временных точек развития, после чего определяли их генотипы и фенотипы.
Таблица 1.
Концентрации комплексов, инъецированных в одноклеточные эмбрионы, для выбора оптимальной концентрации sgРНК tyr
| Белок Cas9 | ||
|---|---|---|
| 1 | 2 | 3 |
| Белок Cas9, 1.1 мкг/мкл | Белок Cas9, 1.1 мкг/мкл | Белок Cas9, 1.1 мкг/мкл |
| sgРНК tyr, 30 нг/мкл | sgРНК tyr, 100 нг/мкл | sgРНК tyr, 200 нг/мкл |
| мРНК Cas9 | ||
| 1 | 2 | 3 |
| мРНК Cas9, 1.1 мкг/мкл | мРНК Cas9, 1.1 мкг/мкл | мРНК Cas9, 1.1 мкг/мкл |
| sgРНК tyr, 30 нг/мкл | sgРНК tyr, 100 нг/мкл | РНК tyr, 200 нг/мкл |
Оценка эффективности индукции NHEJ с помощью sgРНК. Способность sgРНК индуцировать двухцепочечные разрывы с последующей NHEJ в локусе gad оценивали при помощи нуклеазы Surveyor (Surveyor Mutation Detection Kit, “Transgenomics”, CША). Геномную ДНК выделяли из развивающихся эмбрионов полосатого данио с использованием NaOH. Нужный участок амплифицировали при помощи следующих пар праймеров:
gad SA 5'F: CATAGACATAGTCGTACAGAGGTCG,
gad SA 3'R: ACAAGACCTACGACAAGGGAAG.
Эффективность нокаута локуса тирозиназы (tyr) оценивали визуально по нарушению пигментации.
Поиск событий рекомбинации. Эффективность целевого встраивания определяли по флуоресцентному сигналу в формирующихся ГАМКергических нейронах и при помощи ПЦР с геномной ДНК со следующими праймерами:
GFP F: CAAGCCATGACAAGGATTACAAC,
HR gad 3'R: AAGACGACCAACCAACAGAAC
(отжигается на последовательность вне 3'-участка гомологии).
Геномную ДНК выделяли по стандартному протоколу. ПЦР проводили с Phusion High-Fidelity DNA Polymerase (“Thermo Scientific”, США). Полученные ПЦР-фрагменты секвенировали, чтобы подтвердить сайт инсерции (“Genewiz”, США). Из-за большого количества повторов, локализованных проксимально к 5'-плечу gad-гомологии, мы не смогли получить ПЦР-фрагменты, поэтому точность вставки с этой стороны подтвердить не удалось.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Введение белка Cas9 в одноклеточные эмбрионы Danio rerio более эффективно индуцирует NHEJ, чем мРНК Cas9
Ранее сообщалось, что белок Cas9 индуцирует разрывы в ДНК с последующим NHEJ более эффективно, чем мРНК Cas9 [18]. Чтобы убедиться в этом, мы проверили эффективность выключения гена tyr (рис. 1a), который отвечает за превращение тирозина в пигмент меланин. Нарушение функций этого гена удобно наблюдать по дефектам пигментации у мальков [22]. Были проверены разные концентрации sgРНК tyr в комбинации с разными концентрациями белка или мРНК Cas9 (табл. 1). В лучшем случае примерно из половины (59.6%) эмбрионов, инъецированных белком Cas9, развились рыбки, практически лишенные пигментации (что означает разрушение обоих аллелей гена на ранней стадии развития). При этом только 4% эмбрионов с мРНК Cas9 имели такой фенотип (табл. 2, рис. 1б). Однако в эксперименте с мРНК Cas9 наблюдали высокий уровень мозаицизма (рис. 1б). Это закономерно, так как мРНК Cas9 обеспечивает более длительный эффект редактирования и объясняет также повышенный уровень гибели эмбрионов в данном случае. Таким образом, наглядно показано, что введение белка Cas9 более эффективно и при этом более безопасно по сравнению с введением мРНК Cas9.
Рис. 1.
Оценка эффективности и токсичности доставки белка и мРНК Cas9 в эксперименте по нарушению функции гена в ранних эмбрионах рыб. а – Схема расположения целевого сайта для sgРНК гена tyr. б – Нарушение функции тирозиназы (tyr) путем введения мРНК Cas9 (средняя панель) и белка Cas9 (правая панель) приводит к появлению фенотипа, мозаичного по пигментации.
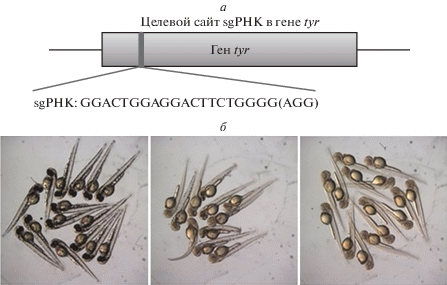
Таблица 2.
Количественная оценка эффективности инъекции белка и мРНК Cas9 в однодневных эмбрионах
| sgРНК, нг/мкл | Белок, % | мРНК, % | Ctl, % | |
|---|---|---|---|---|
| Нокаут | ||||
| Погибло, % | 11.8 | 38.0 | 4.0 | |
| 30 | Дефект, % | 5.9 | 35.3 | 2.1 |
| Эффективность, % | 39.2 | 4.0 | – | |
| Погибло, % | 10.5 | 46.3 | 0 | |
| 100 | Дефект, % | 10.5 | 40.7 | 0 |
| Эффективность, % | 50.9 | 7.4 | – | |
| Погибло, % | 15.4 | 65.4 | 0 | |
| 200 | Дефект, % | 19.2 | 30.9 | 0 |
| Эффективность, % | 59.6 | 1.8 | – | |
| Вставка | ||||
| Погибло, % | 17.3 | 26.2 | 12.3 | |
| 50 | Дефект, % | 5.3 | 10.7 | 1.4 |
| Эффективность, % | 0 | 1.0 | – | |
| Погибло, % | 44.6 | 40.8 | 10.0 | |
| 200 | Дефект, % | 5.4 | 12.2 | 0 |
| Эффективность, % | 0 | 12.2 | – | |
| Погибло, % | 38.7 | 43.5 | 0 | |
| 600 | Дефект, % | 37.3 | 13.0 | 0 |
| Эффективность, % | 0 | 1.8 | – | |
Обнаружение событий гомологичной рекомбинации в Danio rerio
Поставленная нами задача подразумевала маркирование различных подтипов нейронов при помощи направленного встраивания репортеров в эндогенные локусы. Эту задачу мы решили с использованием CRISPR для введения гена GFP в последний экзон непосредственно перед стоп-кодоном гена gad (рис. 2a). Ген gad экспрессируется в большинстве ГАМКергических нейронов, составляющих ингибиторную систему нейромедиаторов [24, 25].
Рис. 2.
Оценка эффективности и токсичности белка и мРНК Cas9 в эксперименте по Cas9-индуцированной
гомологичной встройке в ранних эмбрионах полосатого данио. а – Схема расположения целевых сайтов для sgРНК в гене gad. б – Оценка эффективности редактирования шести случайно отобранных эмбрионов при помощи
нуклеазы Surveyor. Показано, что sgРНК-2 gad индуцирует более высокий уровень мутагенеза
в целевом локусе, чем sgРНК-1. Стрелки указывают на фрагменты, образованные после
расщепления Surveyor. в – Изображения мозга эмбриона, синтезирующего GFP в ГАМКергических нейронах (1 дпо
‒ однодневный эмбрион). г ‒ In vitro ПЦР-проверка событий направленного встраивания в трех эмбрионах, синтезирующих GFP.
д ‒ ПЦР-проверка вставки в локус gad в 10 эмбрионах без сигнала GFP и в 10 GFP-положительных
(N1~N10) через 1 сутки после инъекции (1 дпо), а также 10 эмбрионов (№ 4‒6 GFP-положительные)
через 2 суток после инъекции (2 дпо). Звездочкой (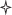 ) обозначены ПЦР-положительные клоны. N – отрицательный контроль (дикий тип). S ‒
эмбрион GFP+ LA и RA ‒ левое и правое плечо вектора соответственно.
) обозначены ПЦР-положительные клоны. N – отрицательный контроль (дикий тип). S ‒
эмбрион GFP+ LA и RA ‒ левое и правое плечо вектора соответственно.
Были подобраны две sgРНК, нацеленные на последовательности вблизи нужного сайта инсерции. sgРНК включала оптимизированную структурную часть, которая улучшала связывание Cas9 [12, 22] (рис. 2a). Расстояние последовательность-специфичной части sgРНК от сайта вставки составляло 11 и 38 н. соответственно. При помощи нуклеазы Surveyor оценили эффективность sgРНК на эмбрионах рыб, которым ввели in vitro полученные sgРНК и мРНК Cas9. Показано, что sgРНК-2 более эффективно индуцирует двухцепочечный разрыв в целевом сайте (рис. 2б).
Мы сконструировали донорный вектор для рекомбинации, содержащий два участка гомологии с 5'- и 3'-концами гена gad (423 и 1023 п.н.), соответственно, между которыми располагался ген GFP. Последовательность PAM рядом с целевыми sgРНК была изменена, чтобы избежать распознавания и расщепления плазмидной ДНК. Мы провели коинъекцию мРНК Cas9, sgРНК-2 и донорного вектора, концентрация которого варьировала от 50 до 600 нг/мкл (табл. 2S, см. Приложение). Сигналы GFP детектировали в инъецированных эмбрионах всех групп, однако наибольший процент встраивания наблюдали при концентрации донорной плазмиды 200 нг/мкл (12 из 98 эмбрионов, 12.2%) (табл. 2, рис. 2в).
Введение белка Cas9 более эффективно для индукции событий негомологичного соединения концов, поэтому мы решили проверить, будет ли введение белка более эффективно индуцировать сигналы GFP при гомологичной встройке. Смертность эмбрионов, которым инъецировали комплексы, содержащие белок Cas9, sgРНК gad и донорную плазмиду, была ниже, чем у эмбрионов, которым ввели комплексы с мРНК Cas9 (табл. 2). Тем не менее, мы не увидели сигнала GFP даже на третьи сутки после инъекции в одноклеточные эмбрионы комплексов с белком Cas9 в различных концентрациях (табл. 2S, табл. 1S, см. Приложение). Однако в противоположность этому инъекция мРНК Cas9 приводила к более эффективной индукции встройки, чем инъекция белка.
В дополнение к оценке эффективности встройки по уровню сигнала GFP мы верифицировали встройку с помощью ПЦР-анализа ДНК эмбрионов. С этой целью из каждой группы отбирали по три эмбриона и выделяли из них геномную ДНК, на которой затем проводили ПЦР с праймером, комплементарным последовательностям, не входящим в 3'-участок гомологии gad. Амплифицированные фрагменты ДНК имели ожидаемую длину (рис. 2г).
Ранее сообщалось, что при ПЦР-анализе ДНК D. rerio, направленном на поиск встройки, можно наблюдать артефакты [26]. Поначалу мы также детектировали этот артефакт после анализа эмбрионов, которым инъецировали донорную плазмиду вместе с Cas9 и sgРНК (рис. 1S, см. Приложение). Через 24 ч после оплодотворения (чпо) фрагмент ожидаемой длины присутствовал в 11 GFP-положительных эмбрионах и в 17 из 18 GFP-отрицательных. Чтобы избавиться от артефакта, мы уменьшили концентрацию плазмиды и использовали высокоточную полимеразу. Кроме того, мы ввели дополнительный контроль ‒ эмбрионы, инъецированные только донорной плазмидой. В результате ожидаемый ПЦР-фрагмент не детектировался в эмбрионах (2, 4, 6, 8 чпо), которым ввели донорную плазмиду в концентрации 50 нг/мкл, но детектировался в GFP-положительных 24 чпо эмбрионах после инъекции (рис. 2S, см. Приложение). В итоге через 1 сутки после инъекции фрагмент нужного размера не обнаружили ни в одном из 10 GFP-отрицательных эмбрионов, но выявили его в 7 из 10 GFP-положительных эмбрионов (рис. 2д).
С использованием секвенирования подтвердили амплификацию нужной области с помощью ПЦР-анализа (рис. 3). Из-за повторов, расположенных вблизи 5'-области гена gad, мы не смогли амплифицировать эту область и подтвердить наличие вставки.
Повременной анализ встройки
Чтобы объяснить различную эффективность белка и мРНК Cas9 в индукции NHEJ и вставки по механизму гомологичной рекомбинации, мы проанализировали эти события, детектируя их на разных стадиях развития, начиная с 2 ч после оплодотворения (инъекции). Следы репарации по механизму NHEJ обнаружены в первой же точке, через 2 ч после инъекции комплекса белок Cas9:sgРНК, т.е. на 4 ч раньше, чем в случае комбинации мРНК Cas9:sgРНК (рис. 4а).
Рис. 4.
Повременной анализ событий редактирования. а – Анализ ранних стадий развития и появления нокаута гена gad после инъекции белка либо мРНК Cas9 в одноклеточные эмбрионы. б ‒ Анализ ранних стадий развития и встраивания GFP в ген gad после инъекции белка либо мРНК Cas9 в одноклеточные эмбрионы. Стрелками отмечены продукты расщепления фрагмента нуклеазой Surveyor. N – отрицательный контроль (дикий тип).
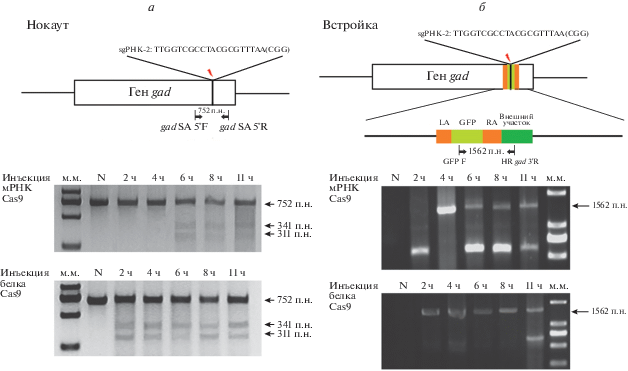
Геномную ДНК для проведения ПЦР выделяли из трех эмбрионов каждой группы. По-видимому, опосредованное белком Cas9 событие вставки могло произойти на 2-й час после инъекции, так как оно обнаружено в одной из четырех групп в точке 2 ч и детектировалось также в точках 4, 6, 8, 11 ч после инъекции (рис. 4б). Тем не менее, проверка эмбрионов из 10 групп через 1, 2 и 3 суток не выявила в них вставок (табл. 1S, Приложение). При помощи ПЦР вставка обнаружена в эмбрионах ранних стадий, в которые ввели мРНК Cas9 (рис. 4б). ПЦР-продукт ожидаемой длины не найден ни в одной из четырех групп в точке 2 ч; в точках 4 и 6 ч найден во всех четырех группах; в трех из четырех групп ‒ в точках 8 и 11 ч. Таким образом, число ПЦР-положительных эмбрионов со временем уменьшалось.
Мы также детектировали события встройки при помощи флуоресцентного микроскопа. Сигнал GFP можно было наблюдать в течение 1‒3 суток с момента инъекции комплексов в одноклеточные эмбрионы. Через сутки после инъекции белка Cas9 флуоресцентный сигнал отсутствовал, что соответствовало результатам ПЦР-анализа (табл. 2). К нашему удивлению, в группах, которым вводили мРНК Cas9, флуоресцентный сигнал со временем исчезал. Сигнал GFP детектировался через сутки после инъекции в 44 из 351 эмбрионов. Через 2 суток половина GFP-положительных эмбрионов потеряла сигнал, а через 3 суток GFP-положительных эмбрионов не осталось (табл. 3). В контрольных эмбрионах, которым вводили только донорную плазмиду, флуоресцентный сигнал также отсутствовал (табл. 3). Для проверки места вставки мы взяли по 10 эмбрионов через 24 и 48 ч после инъекции и провели ПЦР-анализ. Фрагмент нужной длины обнаружен в 7 из 10 GFP-положительных эмбрионов через 24 ч после инъекции и только в двух через 48 ч (рис. 2д).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В данной работе мы сравнили эффективность индукции NHEJ путем введения белка Cas9 и мРНК Cas9. Согласно предыдущим исследованиям [18, 27], белок Cas9 более эффективен для получения нокаута, чем мРНК Cas9. Учитывая время, необходимое для трансляции мРНК в функциональный белок, закономерно предположить, что доставка белка Cas9 должна приводить к редактированию в более ранней временнóй точке. Это помогает избежать мозаицизма и увеличить общую эффективность нокаута при помощи NHEJ.
Тем не менее, когда дело дошло до гомологичной вставки (HD knock-in), мы получили менее однозначные результаты. Несмотря на известный ПЦР-артефакт, появляющийся при анализе событий гомологичной рекомбинации у D. rerio [26], после оптимизации условий ПЦР (снижение концентрации вводимой донорной плазмиды и использование высокоточной полимеразы) мы смогли избавиться от ложноположительного сигнала ПЦР в контрольных эмбрионах, в которые ввели только донорную плазмиду. Показано, что количество ПЦР-положительных эмбрионов постепенно снижается (между точками 2 и 24 ч). Более того, к третьим суткам после инъекции сигнал GFP становился более слабым (табл. 3). Потеря флуоресцентного сигнала от нейронов со временем совпадает с потерей вставки, детектируемой при помощи ПЦР. Нестабильность вставки может быть основной причиной, по которой до сих пор не удалось получить зародышевые трансгенные линии со вставкой GFP в локус gad. Другое возможное объяснение может состоять в том, что потеря гена GFP происходит при “разбавлении” отредактированных клеток. Механизм Cas9-опосредованного гомологичного встраивания до сих пор не полностью ясен, особенно у D. rerio. Кроме того, маловероятно, что наше наблюдение касается только локуса gad, поэтому изучение других локусов могло бы способствовать пониманию механизма этого события.
Нами показано, что нокаут локуса gad при помощи NHEJ с большей эффективностью происходит при введении белка Cas9, чем мРНК Cas9 в одноклеточные эмбрионы рыб, что согласуется с полученными ранее результатами. Кроме того, мы получили сложно интерпретируемые результаты. Показано снижение количества эмбрионов, содержащих вставку репортера, в ходе эмбриогенеза, которое можно объяснить потерей вставки на поздних стадиях эмбриогенеза. Потеря вставки может лежать в основе низкой эффективности точного геномного редактирования у D. rerio, что препятствует проведению исследований с участием этого модельного организма.
Мы благодарим Wenbiao Chen из Vanderbilt University за предоставленную плазмиду pTyr-gRNA. Мы также благодарим Jennifer Doudna за ценную дискуссию.
Работа поддержана National Natural Science Foundation of China (№ 31310103032) (K.J.) и National Institute of Health (NIH) США (DA035680 и NS095734) (S.G.).
Все процедуры, проведенные с участием животных, соответствовали этическим стандартам учреждений или принятой практике таких исследований.
Y.P. и S.G. планировали дизайн исследования; Y.P., K.H., и F.J. предоставили реактивы и материалы; Y.P., K.H., K.J., W.Z., Z.D. и S.G. провели эксперименты и анализ данных; Y.P., K.H., W.Z., K.J. и S.G. вычитывали рукопись; все авторы участвовали в обсуждении результатов и внесли вклад в окончательный вариант статьи.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Asada H., Kawamura Y., Maruyama K., Kume H., Ding R.G., Kanbara N., Kuzume H., Sanbo M., Yagi T., Obata K. (1997) Cleft palate and decreased brain gamma-aminobutyric acid in mice lacking the 67-kDa isoform of glutamic acid decarboxylase. Proc. Natl. Acad. Sci. USA. 94, 6496‒6499.
Auer T.O., Del Bene F. (2014) CRISPR/Cas9 and TALEN-mediated knock-in approaches in zebrafish. Methods. 69, 142‒150.
Auer T.O., Duroure K., De Cian A., Concordet J.P., Del Bene F. (2014) Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Res. 24, 142‒153.
Bedell V.M., Wang Y., Campbell J.M., Poshusta T.L., Starker C.G., Krug R.G., Tan W.F., Penheiter S.G., Ma A.C., Leung A.Y.H., Fahrenkrug S.C., Carlson D.F., Voytas D.F., Clark K.J., Essner J.J., Ekker S.C. (2012) In vivo genome editing using a high-efficiency TALEN system. Nature. 491, 114‒118.
Bowman T.V., Zon L.I. (2010) Swimming into the future of drug discovery: in vivo chemical screens in zebrafish. Acs Chem. Biol. 5, 159‒161.
Feng Z., Zhang B., Ding W., Liu X., Yang D.L., Wei P., Cao F., Zhu S., Zhang F., Mao Y., Zhu J.K. (2013) Efficient genome editing in plants using a CRISPR/Cas system. Cell Res. 23, 1229‒1232.
Filippi A., Mueller T., Driever W. (2014) vglut2 and gad expression reveal distinct patterns of dual GABAergic versus glutamatergic cotransmitter phenotypes of dopaminergic and noradrenergic neurons in the zebrafish brain. J. Comp. Neurol. 522, 2019‒2037.
Gagnon J.A., Valen E., Thyme S.B., Huang P., Akhmetova L., Pauli A., Montague T.G., Zimmerman S., Richter C., Schier A.F. (2014) Efficient mutagenesis by Cas9 protein-mediated oligonucleotide insertion and large-scale assessment of single-guide RNAs. PLoS One. 9, e98186.
Hoshijima K., Jurynec M.J., Grunwald D.J. (2016) Precise editing of the zebrafish genome made simple and efficient. Dev. Cell. 36, 654‒567.
Hwang W.Y., Fu Y., Reyon D., Maeder M.L., Tsai S.Q., Sander J.D., Peterson R.T., Yeh J.R., Joung J.K. (2013) Efficient genome editing in zebrafish using a CRISPR-Cas system. Nat. Biotechnol. 31, 227‒229.
Irion U., Krauss J., Nusslein-Volhard C. (2014) Precise and efficient genome editing in zebrafish using the CRISPR/Cas9 system. Development. 141, 4827‒4830.
Jao L.E., Wente S.R., Chen W. (2013) Efficient multiplex biallelic zebrafish genome editing using a CRISPR nuclease system. Proc. Natl. Acad. Sci. USA. 110, 13904‒13909.
Jinek M., Chylinski K., Fonfara I., Hauer M., Dou-dna J.A., Charpentier E. (2012) A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816‒821.
Jinek M., Jiang F.G., Taylor D.W., Sternberg S.H., Kaya E., Ma E.B., Anders C., Hauer M., Zhou K.H., Lin S., Kaplan M., Iavarone A.T., Charpentier E., Nogales E., Doudna J.A. (2014) Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. Science. 343, 1247997.
Kimmel C.B., Ballard W.W., Kimmel S.R., Ullmann B., Schilling T.F. (1995) Stages of embryonic-development of the zebrafish. Dev. Dynamics. 203, 253‒310.
Lieschke G.J., Currie P.D. (2007) Animal models of human disease: zebrafish swim into view. Nat. Rev. Genetics. 8, 353‒367.
Meng X.D., Noyes M.B., Zhu L.H.J., Lawson N.D., Wolfe S.A. (2008) Targeted gene inactivation in zebrafish using engineered zinc-finger nucleases. Nat. Biotechnol. 26, 695‒701.
Patton E.E., Zon L.I. (2001) The art and design of genetic screens: zebrafish. Nat. Rev. Genetics. 2, 956‒966.
Platt R.J., Chen S.D., Zhou Y., Yim M.J., Swiech L., Kempton H.R., Dahlman J.E., Parnas O., Eisenhaure T.M., Jovanovic M., Graham D.B., Jhunjhunwala S., Heidenreich M., Xavier R.J., Langer R., Anderson D.G., Hacohen N., Regev A., Feng G.P., Sharp P.A., Zhang F. (2014) CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159, 440‒455.
Ran F.A., Hsu P.D., Wright J., Agarwala V., Scott D.A., Zhang F. (2013) Genome engineering using the CRISPR-Cas9 system. Nat. Protoc. 8, 2281‒2308.
Sander J.D., Cade L., Khayter C., Reyon D., Peterson R.T., Joung J.K., Yeh J.R. (2011) Targeted gene disruption in somatic zebrafish cells using engineered TALENs. Nat. Biotechnol. 29, 697‒698.
Shin J., Chen J.K., Solnica-Krezel L. (2014) Efficient homologous recombination-mediated genome engineering in zebrafish using TALE nucleases. Development. 141, 3807‒3818.
Stemmer M., Thumberger T., Keyer M.D., Wittbrodt J., Mateo J.L. (2015) CCTop: An intuitive, flexible and reliable CRISPR/Cas9 target prediction tool. PLoS One. 10, e0124633.
Sung Y.H., Kim J.M., Kim H.T., Lee J., Jeon J., Jin Y., Choi J.H., Ban Y.H., Ha S.J., Kim C.H., Lee H.W., Kim J.S. (2014) Highly efficient gene knockout in mice and zebrafish with RNA-guided endonucleases. Genome Res. 24, 125‒131.
Won M., Dawid I.B. (2017) PCR artifact in testing for homologous recombination in genomic editing in zebrafish. PLoS One. 12, e0172802.
Woods I.G., Schier A.F. (2008) Targeted mutagenesis in zebrafish. Nat. Biotechnol. 26, 650–651.
Zu Y., Tong X.J., Wang Z.X., Liu D., Pan R.C., Li Z., Hu Y.Y., Luo Z., Huang P., Wu Q., Zhu Z.Y., Zhang B., Lin S. (2013) TALEN-mediated precise genome modification by homologous recombination in zebrafish. Nat. Methods. 10, 329‒331.
Дополнительные материалы
- скачать ESM_1.docx
- Additional file
Инструменты
Молекулярная биология



