Молекулярная биология, 2020, T. 54, № 3, стр. 374-388
Функциональная характеристика промотора гена ZmHAK1 кукурузы и регулирующих его факторов транскрипции
H. Sheng a, D. L. Cong b, H. Y. Ju a, *
a College of Plant Science, Jilin University
130062 Changchun, P. R. China
b College of Pharmacy, Jilin University
130021 Changchun, P. R. China
* E-mail: juhy@jlu.edu.cn
Поступила в редакцию 07.01.2019
После доработки 07.03.2019
Принята к публикации 14.03.2019
Аннотация
Дефицит калия (K+) в почве может существенно влиять на урожайность и развитие растений, которые обычно удовлетворяют потребность в этом элементе, используя транспортеры K+ и/или К+-каналы. Белки семейства высокоаффинных переносчиков калия (ZmHAK) играют решающую роль в поглощении и распределении K+ в растениях кукурузы (Zea mays L.). В настоящей работе проведен функциональный анализ промотора гена ZmHAK1 кукурузы и факторов транскрипции, которые регулируют экспрессию этого гена. Семейство генов HAK кукурузы включает 34 члена, и анализ их филогенетического дерева показывает как эволюционную консервативность, так и разнообразие. В промоторе гена ZmHAK1 выявлено множество функциональных элементов, связанных с абиотическим стрессом. С помощью репортерной конструкции pCambia1301:ProZmHAK1:GUS активность промотора гена ZmHAK1 выявлена в корнях, стеблях и листьях кукурузы. В дрожжевой одногибридной системе показано, что промотор ZmHAK1 взаимодействует с факторами транскрипции ZmRAP2.11 и ZmARF2, и эти взаимодействия картированы в разных частях промотора. Фактор транскрипции ZmRAP2.11 локализуется в ядре, тогда как ZmARF2 обнаружен как в ядре, так и в цитоплазме клетки. Полученные данные указывают на важную роль гена ZmHAK1 в процессе поглощения ионов калия и, возможно, в ответе растений кукурузы на абиотический стресс.
ВВЕДЕНИЕ
Калий (K+) ‒ важный элемент, на долю которого приходится до 10% сухой массы растений [1]. К+ играет важную роль не только в росте и развитии сельскохозяйственных культур, но и в их урожайности и качестве [2, 3]. K+ участвует во многих физиологических процессах, таких как активация ферментов, поддержание тургора клеток, рост растений и движение органов [4]. Клеточная вакуоль представляет собой камеру хранения, в которой концентрация K+ составляет от 100 до 200 мМ. Концентрация K+ в растениях существенно выше, чем в почве (всего около 0.1‒1 мМ) [5]. Поэтому для поглощения K+ из почвы корни растений должны использовать транспортеры K+ и K+-каналы. Кроме того, растения выработали различные механизмы поглощения K+ в соответствии с содержанием K+ в среде. Ранее показали, что при высокой концентрации K+ (>0.3 мМ) в среде растения поглощают K+ пассивно через канальные белки с низким сродством к K+, в то время как при низкой концентрации K+ (<0.2 мМ) в окружающей среде K+ активно транспортируется с помощью высокоаффинных переносчиков [6, 7]. За последние годы у высших растений обнаружены различные транспортеры K+ и K+-каналы [1, 8, 9]. Влияние этих двух транспортных систем на поглощение K+ соответствует концентрациям K во внешней среде. Транспортеры становятся активными при низкой концентрация K+, а K+-каналы активны при высокой концентрация K+ [6, 10]. Семейство KUP/HAK/KT ‒ наиболее изученное и наиболее распространенное семейство высокоаффинных переносчиков K+ в растениях [11]. Например, HAK5 Arabidopsis является переносчиком K+ [11, 12]. В это семейство входит также HAK1, который играет важную роль в K+-опосредованной регуляции роста и солеустойчивости растений риса при низких концентрациях K+ [13].
Применение трансгенной технологии требует понимания роли генов и связанных с ними регуляторных механизмов, а также их места и уровня экспрессии в растениях. Поскольку экспрессия чужеродного гена контролируется факторами транскрипции, особенно важно определить регуляторные элементы в промоторе чужеродного гена [14]. Показано, что промоторы многих генов индуцируются различными факторами [15, 16]. Например, промотор гена AlHAK1 индуцируется абсцизовой кислотой (ABA), засухой и высокой температурой [17]. Передача сигнала в растениях, находящихся в условиях стресса, а также связанная с этим регуляция межгенных сетей изучены довольно подробно [18, 19]. В довольно сложную транскрипционную программу у растений в условиях стресса вовлечены сотни факторов транскрипции [20]. По-видимому, экспрессия генов подвергается сложной регуляции в ответ на калиевый стресс [21‒24]. Транскрипционный фактор RAP2.11 может отвечать на низкий уровень К+, связываясь непосредственно с элементами ERE и GCC-box в промоторе гена HAK5 in vitro и in vivo [25]. Более того, фактор ауксинового ответа ARF2 (отрицательный регулятор) связывается с промотором HAK5 и регулирует его транскрипцию, а остаток серина в положении 689 ARF2 может служить сайтом фосфорилирования в ответ на низкое содержание K+ [26].
Кукуруза – одна из основных продовольственных культур, обладающая уникальными характеристиками, на урожайность которой сильно влияет дефицит калия. Чтобы увеличить поглощение калия из почвы и обеспечить растение необходимым количеством K+ проводится поиск высокоаффинных переносчиков калия. Однако изучению высокоаффинных переносчиков K+ в растениях кукурузы посвящено лишь небольшое число публикаций [26, 27]. Ген ZmHAK1 кукурузы, кодирующий высокоаффинный переносчик калия, ранее был клонирован в нашей лаборатории. В настоящей работе проведен полногеномный поиск генов семейства HAK кукурузы (ZmHAK). Кроме того, проведен анализ промотора гена HAK1 и изучено взаимодействие ZmHAK1 с факторами транскрипции ZmRAP2.11 и ZmARF2. Полученные результаты способствуют пониманию механизмов регуляции ZmHAK1.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Множественное выравнивание последовательностей, филогенетический анализ, определение координат генов в хромосомах. Последовательности генов и белков семейства HAK арабидопсиса, кукурузы и риса получены из базы данных геномов растений Phytozome (доступна на сайте: https:// phytozome.jgi.doe.gov). Эти последовательности выравнивали с использованием ClustalW, а полученное выравнивание анализировали с помощью ресурса MEGA7 для построения филогенетических деревьев [28]. Филогенетические деревья строили с использованием методов соединения соседних усредненных точек и p-расстояния. Значение начальной загрузки оценивали после 1000 итераций. Филогенетические деревья визуализировали с помощью онлайн-программы Evolview (доступна онлайн: http://www.evolgenius.info/ evolview/).
Геномные координаты каждого из генов HAK кукурузы получали из базы данных Phytozome 12 Zea mays Ensembl-18 (доступна на сайте: https:// phytozome.jgi.doe.gov). Информацию о длине каждой хромосомы получали из базы данных по сравнительной функциональной геномике “Грамена” (доступна на сайте: http://www.gramene.org). Расположение генов HAK кукурузы на хромосоме изображали с помощью программного обеспечения Circos.
Растительный материал. Семена Nicotiana tabacum получены из Лаборатории питания растений Университета Цзилинь. Растения выращивали в горшках в тепличных условиях: 16 ч освещения при 25°C и 8 ч темноты при 20°C, относительная влажность 70–80%.
Анализ in silico. Предполагаемые TATA-бокс и сайт инициации транскрипции предсказывали с использованием программы распознавания промотора растений TSSP [29]. Распределение различных факторов транскрипции и регуляторных элементов, включая полноразмерные и короткие версии промоторов, определяли по базе данных PLANTCARE (доступна онлайн: http://bioinformatics.psb.ugent.be/webtools/plantcare/html/).
Клонирование промотора гена ZmHAK1. Полноразмерная область промотора 2246 п.н. гена HAK1 кукурузы и четыре укороченных с 5'-конца фрагмента получены с помощью ПЦР-амплификации [P1 (–1217), P2 (–735), P3 (–323), P4 (–6)]. С этой целью разработаны пять различных наборов прямых праймеров (табл. S1 , см. Приложение 1), и использован один и тот же обратный праймер. Затем полноразмерный и укороченные промоторы клонировали в вектор pMD-18T. Наличие вставок проверяли с помощью ПЦР-амплификации и секвенирования. Фрагменты промотора гена HAK1, клонированные в вектор pMD-18T, амплифицировали с помощью праймеров, указанных в табл. S1 (см. Приложение 1), и клонировали в плазмиду pCambia1301 по сайтам рестриктаз PstI и NcoI (“Takara”, КНР) перед открытой рамкой считывания репортерного гена GUS.
Трансформация растений с помощью агроинфильтрации. Культуры Agrobacterium, трансформированные рекомбинантными плазмидами pCambia1301: GUS, растили в 10 мл жидкой среды LB (1% пептона, 0.5% дрожжевого экстракта, 0.5% NaCl), содержащей 50 мкг/мл канамицина и 25 мкг/мл рифампицина, при 28°С и перемешивании (200 об./мин) в течение 16 ч. Использовали два способа трансформации растений с помощью агробактерий. При первом способе после достижения культуры OD600 = 0.5–0.6 клетки агробактерий собирали центрифугированием при 5000 g в течение 10 мин и суспендировали в инфильтрационной среде (2 мМ Na3PO4 ⋅ 12H2O, 10 мМ CaCl2, 50 мМ MES, pH 5.6, 10 г/л D-фруктозы, 0.1 мМ ацетилсирингон). Суспензию агробактерий вводили в хорошо развитые листья растения табака с помощью шприца объемом 1 мл и анализировали через 2–3 дня. При другом способе различные органы табака разрезали на маленькие кусочки и предварительно культивировали на среде MS в течение 2 дней для формирования каллуса. Полученный каллус инкубировали с суспензией агробактерий в течение 30 мин под вакуумом. Избыток суспензии агробактерий удаляли на стерильной бумаге, помещали каллус в среду MS и культивировали при слабом освещении и 28°С в течение 2 дней. Последующие эксперименты проводили на выращенном органе.
Гистохимическая окраска на активность β-глюкоуронидазы (GUS). Органы растения погружали в окрашивающий раствор GUS (50 мМ натрий-фосфатный буфер, рН 7.0, 2 мМ 5-бром-4-хлор-3-индолил-β-D-глюкуроновая кислота, 10 мМ EDТА, 0.1% Тритон Х-100), выдерживали в вакууме в течение 30 мин, а затем инкубировали в течение 12‒16 ч в темноте при 37°С. Обработанные органы растений несколько раз промывали 70%-ным спиртом, чтобы полностью удалить хлорофилл и другие пигменты. Листья табака растирали в порошок в жидком азоте. Измельченные образцы помещали в предварительно охлажденную пробирку объемом 1.5 мл, а затем добавляли 600 мкл буфера для экстракции GUS (100 мМ натрий-фосфатный буфер, pH 7.0, 10% SDS, 500 мМ EDTA, pH 8.0, 0.1% Тритон X-100, 25 мМ β-меркаптоэтанол) и тщательно перемешивали. Гомогенат центрифугировали при 12 000 об/мин в течение 15 мин при 4°С и собирали супернатант. Концентрацию белка определяли по методу Брэдфорда [30], калибровочную кривую строили с использованием бычьего сывороточного альбумина (BSA). Измеряли поглощение при OD595, концентрацию белка рассчитывали по калибровочной кривой. Всего 250 мкл образца добавляли в 450 мкл предварительно нагретого реакционного буфера при 37°С (конечная концентрация 4-метилумбеллиферил -D-глюкуронида (MUG) составляла 2 мМ), тщательно перемешивали. В точках 0, 10, 20, 30 и 40 мин отбирали по 50 мкл образца, немедленно добавляли 950 мкл раствора для остановки реакции (0.2 М Na2CO3) и инкубировали в темноте в водяной бане при 37°С. Интенсивность флуоресценции образца измеряли с помощью спекторофлуориметра (RF-5301pc) при длине волны возбуждения 365 нм и длине волны излучения 455 нм. Количество 4-метилумбеллиферона (4-МЕ), образующегося в результате реакции, рассчитывали по калибровочной кривой. Активность GUS рассчитывали по следующей формуле:
Дрожжевая одногибридная система (Y1H). Для создания системы Y1H полноразмерные кДНК генов ZmRAP2.11 и ZmARF2, полноразмерный промотор ZmHAK1 и его фрагменты [PZmHAK1 (–1669…–573 п.н.), ΔP1 (–1669…–1223 п.н.), ΔP2 (–1243…–883 п.н.), ∆P3 (–903…–471 п.н.), ΔP4 (–499…–109 п.н.), ΔP5 (–151…–139 п.н.), ΔP6 (+121…‒573 п.н.)] амплифицировали со специфичными праймерами (табл. S1 , Приложение 1) и клонировали в векторы pGAD424 (AD; мишень) и pLacZi (AC; приманка) бесшовным методом с использованием ферментов из набора TransGen Biotch:Peasy-Uni. В итоге получили плазмиды AD-ZmARF2, AD-ZmRAP2.11, AC-PZmHAK1, AC-ΔP1, AC-ΔP2, AC-ΔP3, AC-ΔP4, AC-ΔP5 и AC-ΔP6. Различными комбинациями плазмид (плазмида с “приманкой” в линеаризованном виде) трансформировали клетки штамма дрожжей YM4271 и выращивали в течение 2 дней при 30°С на чашках с агаром SD-Leu/-Ura.
Определение активности β-галактозидазы. В качестве субстрата β-галактозидазы использовали CPRG (хлорфенол красный-β-D-галактопиранозид). Минимум три независимых колонии растили при 30°С в течение 12‒16 ч в жидкой среде SD-Leu/-Ura, переносили в YPD и растили в течение 3‒5 ч до плотности OD600 = 0.5‒0.8. Затем клетки осаждали центрифугированием и ресуспендировали в буфере 1 (HEPES 23.8 г/л, NaCl 9 г/л, L-Asp 0.65 г/л, BSA 10 г/л, Твин-20 500 мкл/л, pH 7.25‒7.3). Суспензию клеток помещали в жидкий азот на 1 мин, затем переносили в водяную баню (37°С, 1 мин) и повторяли этот цикл 3 раза. Далее добавляли буфер 2 (CPRG 1.355 г/л, растворенный в буфере 1) и инкубировали при температуре 30°С. После окрашивания опытной реакционной смеси в желтый цвет в опытные образцы и в отрицательный контроль добавляли 3 мМ ZnCl2, время реакции записывали в минутах. Пробирки центрифугировали (13 000 об./мин, 1 мин), супернатант осторожно переносили в чистые кюветы, чтобы определить значение OD578. Активность β-галактозидазы рассчитывали по формуле: активность (у.е.) β-галактозидазы = = 1000 × OD578/(t × V × OD600), t = время инкубации, V = 0.1 мл × коэффициент концентрации (коэффициент концентрации 15).
Субклеточная локализация. Полноразмерную кДНК ZmRAP2.11 и ZmARF2 получали с помощью ПЦР-амплификации с использованием ген-специфичных праймеров (табл. S1 , Приложения 1) и клонировали в донорный вектор Gateway pDONOR207 путем гомологичной рекомбинации. Донорные векторы рекомбинировали с репортерными векторами pEarleyGate101. Полученными плазмидами ZmARF2-GFP и ZmRAP2.11-GFP трансформировали агробактерии и вводили в листья табака. Флуоресценцию GFP в клетках эпидермиса листьев табака наблюдали с использованием флуоресцентного микроскопа (Nikon DS-Ri2) через 4‒5 дней.
Статистический анализ. Данные представлены в виде среднего, разброс данных – в виде стандартной ошибки (mean ± SEM). Сравнение групп данных выполняли с помощью теста ANOVA в составе программного обеспечения SPSS (версия 19.0). Значение P < 0.05 рассматривали как статистически значимое (** соответствует P < 0.01, * P < 0.05).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Белки HAK кукурузы и их филогенетические связи с ортологами из Arabidopsis и риса
С целью поиска других членов семейства HAK в растениях кукурузы проведен поиск последовательностей, обладающих наибольшим сходством с ZmHAK1, в базе данных Phytozome. Из полученных последовательностей отобрали 34, которые рассматривали как члены семейства генов ZmHAK. Все 34 последовательности приведены в табл. S2 (см. Приложение 2 ). Предсказанные белки семейства HAK состоят из 336‒921 аминокислотных остатков, имеют молекулярную массу 37.3–100.8 кДа и изоэлектрическую точку 5.85‒9.29. Все белки HAK содержат домен транспорта K+. Кроме того, с помощью WOLF pSORT предсказана субклеточная локализация этих белков. Согласно полученным ранее результатам [31], белки HAK предположительно локализованы на клеточной мембране, а также могут находиться в цитоплазме, ядре, митохондриях и эндоплазматической сети (табл. S2 , Приложение 2).
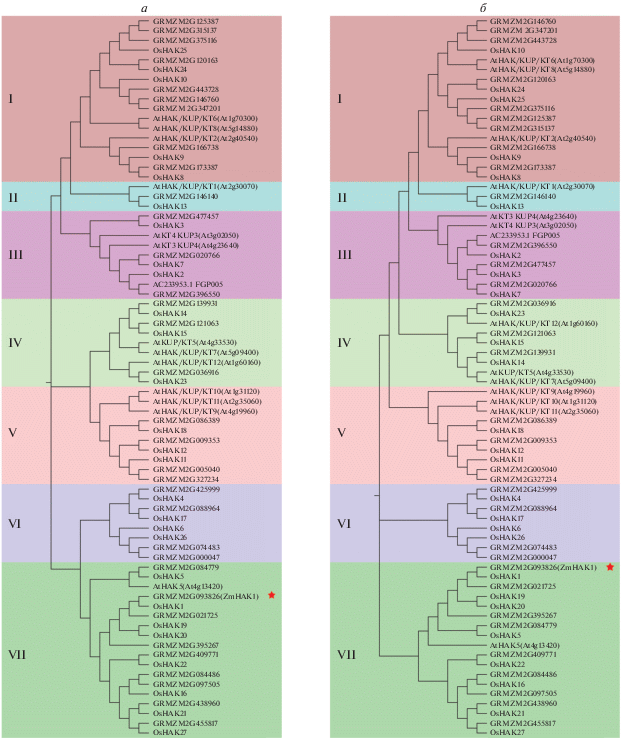
Чтобы изучить эволюционные взаимоотношения между белками семейства HAK кукурузы и других видов, мы использовали последовательности генов и белков кукурузы (34 гена), A. thaliana (13 генов) и риса (27 генов) и построили филогенетические деревья (рис. 1). Полученные филогенетические деревья имеют сходную структуру. Мы разделили эти гены и соответствующие белки на семь кластеров. Кластер I содержал 17 членов (9, 5, 3 у кукурузы, риса и арабидопсиса соответственно). Кластер II включал три члена (по одному у кукурузы, риса и арабидопсиса). Кластер III состоял из 9 членов (4, 3 и 2 у кукурузы, риса и арабидопсиса соответственно), как и кластер IV (по три у каждого вида). Кластер V включает 10 членов (4, 3, 3 у кукурузы, риса и арабидопсиса соответственно). Кластер VI содержит 8 членов (по 4 у кукурузы и риса соответственно), а кластер VII ‒ 18 (9, 8, 1 у кукурузы, риса и арабидопсиса соответственно). Согласно результатам филогенетического анализа члены семейства HAK кукурузы обычно группируются с семейством HAK риса и арабидопсиса. Например, в кластере II GRMZM2G146140 образует одну ветвь с OsHAK13 и At2g30070, что указывает на консервативность членов семейства HAK у разных видов растений. Напротив, GRMZM2G125387 и GRMZM2G315137, GRMZM2G005040 и GRMZM2G327234, GRMZM2G084486 и GRMZM2G097505 формируют отдельные ветви на филогенетическом дереве, предполагая, что HAK может быть специфичным для видов растений.
Рис. 1.
Филогенетический анализ семейства HAK у растений кукурузы, риса и Arabidopsis. Филогенетические деревья получены с использованием матрицы CDS (кодирующие последовательности) (а) или белковой матрицы (б). Римскими цифрами показаны номера групп (I‒VII). Красной звездочкой указан ZmHAK1 (GRMZM2G093826).
Положение генов семейства HAK на хромосомах кукурузы
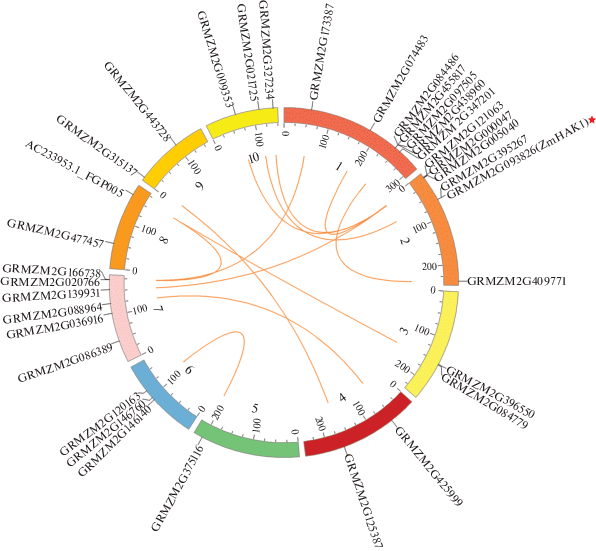
Положение генов HAK на хромосомах кукурузы представлено на рис. 2. Все 34 гена HAK неравномерно распределены по 10 хромосомам, наибольшее количество генов HAK7 находится на хромосоме 1, тогда как на хромосоме 5 локализован только один ген. ZmHAK1 расположен на хромосоме 2. Можно отметить, что ряд генов HAK дуплицирован.
Рис. 2.
Положение и дупликация генов HAK на хромосомах кукурузы. Каждая хромосома окрашена своим цветом и имеет свой номер. Положение каждого гена HAK на хромосоме отмечено короткой черной линией, а два гена с фрагментами дупликации связаны между собой оранжевой линией. Красной звездочкой обозначен ZmHAK1 (GRMZM2G093826).
Анализ промотора гена ZmHAK1 in silico
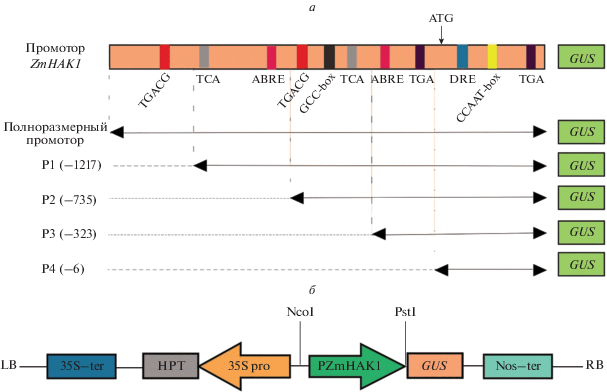
Промотор гена ZmHAK1 анализировали in silico с помощью PlantCARE. Показано, что промотор содержит несколько основных регуляторных элементов (табл. 1), в том числе 11 CAAT-боксов и четыре TATA-бокса. Промотор содержит также элементы, связанные с индукцией гормонами, такие как ABRE (контролируемый абсцизовой кислотой), CGTCA- и TGACG-мотивы (контролируемые MeJA), p-бокс (контролируемый гиббереллином), и TGA-элемент (контролируемый ауксином) (рис. 3). Кроме того, обнаружены элементы, реагирующие на изменение освещенности и засуху.
Таблица 1.
Цис-регуляторые элементы промотора ZmHAK1, обнаруженные с помощью PlantCARE
| Цис-регулятор-ный элемент | Положение в промоторе гена ZmHAK1 | Описание | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | ||
| A-бокс | –1518 | Цис-регуляторный элемент | ||||||||||
| ABRE | –1050 | –398 | –1047 | +382 | –1048 | –401 | Цис-регуляторный элемент, участвующие в ответе на абсцизовую кислоту | |||||
| ACE | +122 | Цис-регуляторный элемент, контролируемый освещенностью | ||||||||||
| AE-бокс | –1228 | Часть составного модуля, реагирующего на свет | ||||||||||
| ARE | –504 | Цис-регуляторный элемент, активируемый в анаэробных условиях | ||||||||||
| Бокс 4 | –755 | –436 | Часть составного модуля, реагирующего на свет | |||||||||
| CAAT-бокс | –1186 | –513 | –922 | –197 | –997 | –480 | –819 | –482 | –1173 | –466 | –996 | Базовый цис-регуляторный элемент промоторов и энхасеров |
| CAT-бокс | –1713 | Цис-регуляторный элемент, функционирующий в меристеме | ||||||||||
| CCAAT-бокс | +271 | Сайт связывания MYBHv1 | ||||||||||
| CGTCA мотив | –1571 | –714 | –1370 | –593 | –1460 | –717 | Цис-регуляторный элемент, вовлеченный в ответ на MeJA (метилжасмонат) | |||||
| G-бокс | –1048 | +382 | –1 | –399 | –881 | +251 | Цис-регуляторный элемент, вовлеченный в реакцию на свет | |||||
| GC мотив | –1336 | Энхансероподобный элемент, участвующий в ответе на недостаток кислорода | ||||||||||
| I-бокс | +22 | +419 | Часть элемента, вовлеченного в реакцию на свет | |||||||||
| MBS | –337 | Сайт связывания MYB, вовлеченный в ответ на засуху | ||||||||||
| O2-сайт | –1243 | +217 | –694 | Цис-регуляторный элемент, вовлеченный в регуляцию метаболизма зеина | ||||||||
| p-бокс | –917 | Гиббереллин-чувствительный регуляторный элемент | ||||||||||
| Sp1 | –1522 | –676 | Светочувствительный регуляторный элемент | |||||||||
| TATA-бокс | –930 | –724 | –855 | +292 | Центральная часть промотора, находящаяся около –30 старта транскрипции | |||||||
| TGA-элемент | –270 | +320 | +176 | –143 | +266 | Ауксин-чувствительный регуляторный элемент | ||||||
| TGACG-мотив | –1571 | –714 | –1369 | –593 | –1460 | –717 | Цис-регуляторный элемент, вовлеченный в ответ на MeJA | |||||
Рис. 3.
Нуклеотидная последовательность и положение предполагаемых сайтов связывания факторов транскрипции в промоторной области ZmHAK1, предсказанных с помощью PlantCARE. Последовательности предсказанных цис-регуляторных элементов в промоторе ZmHAK1 выделены прямоугольниками определенных цветов, рядом с которыми написана функция или название соответствующего элемента. Стрелки указывают направление цис-регуляторного элемента, стартовый кодон ATG обведен овалом.

Клонирование промотора ZmHAK1 и получение репортерных векторов
В целях изучения активности промотора и описания экспрессии ZmHAK1, полноразмерную последовательность промотора этого гена и четырех его укороченных вариантов (P1, P2, P3 и P4) длиной 2246, 1782, 1300, 888 и 571 п.н., соответственно (рис. 4а), клонировали из генома кукурузы сорта JiDan27 (а именно 4–144) и встраивали в вектор экспрессии pCambia1301:GUS с удаленным промотором 35S (рис. 4б). Экспрессионные конструкции, содержащие пять вариантов промотора гена ZmHAK1, соединенного с репортерным геном GUS, переносили в Agrobacterium (штамм EHA105). Для ПЦР-валидации бактериальных трансформантов использовали специфичные праймеры.
Рис. 4.
Схемы репортерных экспрессионных конструкций с вариантами промотора ZmHAK1, соединенного с геном GUS. а ‒ Расположение цис-регуляторных элементов в промоторе гена ZmHAK1 обозначено прямоугольниками разных цветов. Схема химерных репортерных генов с полноразмерным промотором ZmHAK1 и его укороченными вариантами (P1, P2, P3 и P4). б ‒ Общая схема экспрессионной конструкции pCambia1301 с промотором ZmHAK1, клонированным по сайтам NcoI и PstI.
Кратковременная экспрессия репортерной конструкции под контролем промотора ZmHAK1 в растениях табака
Химерные гены ProZmHAK1:GUS экспрессировали в различных органах табака, включая корни, стебли и листья, используя агроинфильтрацию. Активность вариантов промотора ZmHAK1 оценивали по активности GUS, выявленной с помощью гистохимического окрашивания. Полученные результаты указывают на то, что ген GUS, контролируемый полноразмерным промотором ZmHAK1, экспрессируется в корнях, стеблях и листьях табака (рис. 5, б1‒б4). В отрицательном контроле (нетрансформированный материал) активность GUS не выявлена (рис. 5, в1‒в4); в положительном контроле (pCambia1301:ProCaMV35S: GUS) высокую активность GUS наблюдали в корнях, стеблях и листьях (рис. 5, а1‒а4). Важно отметить, что активность GUS, экспрессируемой под контролем полноразмерного промотора ZmHAK1, близка к активности в положительном контроле.
Рис. 5.
Гистохимический анализ активности GUS под контролем вариантов промотора гена ZmHAK1 при временной трансформации растений табака. а‒в ‒ Определение активности GUS с помощью X-Gluc в различных органах (корень (1); стебель (2); поперечное сечение среза стебля (3); листья (4)). а1‒а4 ‒ Положительный контроль ‒ органы, трансформированные CaMV35S:GUS. б1‒б4 ‒ Окрашивание органов, трансформированных GUS под контролем полноразмерного промотора ZmHAK1. в1‒в4 ‒ нетрансформированные органы. г‒и ‒ Активность GUS в листьях под контролем промотора CaMV35S (г), полноразмерного промотора ZmHAK1 и его делеционных вариантов (д‒и). к ‒ Активность GUS через 48 ч после инфильтрации листьев табака Agrobacterium, содержащей конструкции ProCaMV35S: GUS (положительный контроль) или промотор ZmHAK1 (полноразмерный или делеционные варианты P1, P2, P3 и P4). Данные представлены как среднее значение, вычисленное из четырех независимых экспериментов.
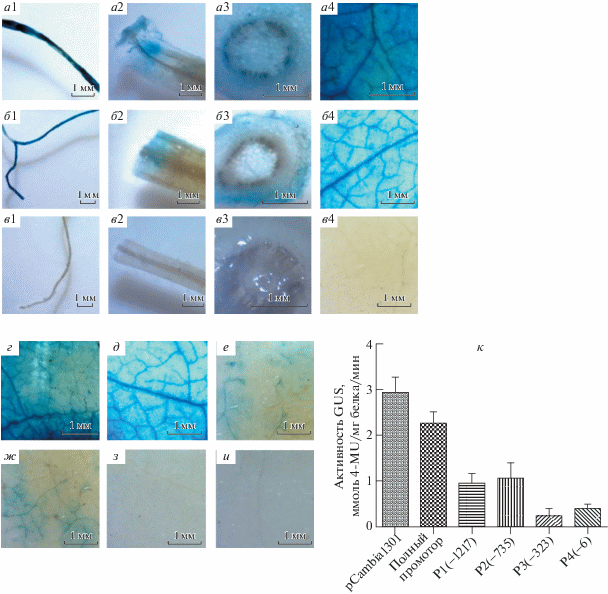
Мы также анализировали активность делеционных вариантов промотора ZmHAK1. Показано, что полноразмерный промотор более эффективно повышает экспрессию GUS (рис. 5д), чем положительный контроль (рис. 5г), его варианты P1 (рис. 5е) и P2 (рис. 5ж) проявляют средний уровень активности, в то время как P3 (рис. 5з) и P4 (рис. 5и) не способны определять экспрессию GUS. Эти результаты подтверждаются также данными количественного анализа активности GUS (рис. 5к). Таким образом, полноразмерный промотор ZmHAK1 обладает высокой активностью и может использоваться в дальнейших экспериментах.
Промотор ZmHAK1 взаимодействует с факторами транскрипции ZmRAP2.11 и ZmARF2
Факторы транскрипции, которые связываются с промотором ZmHAK1, идентифицировали с использованием дрожжевой одногибридной системы. Гены ZmARF2 и ZmRAP2.11 клонировали в вектор pGAD424 и трансформировали им штамм дрожжей YM4271 одновременно с вектором placZi, экспрессирующим ген lacZ под контролем промотора ZmHAK1 (рис. 6а). В качестве отрицательноно контроля использовали следующие три группы котрансформированных плазмид: пустой вектор pGAD424 и пустой вектор placZi (AD/AC), пустой вектор pGAD424 и вектор placZi с промотором ZmHAK1 (AD/AC-ZmHAK1), вектор pGAD424, экспрессирующий ZmRAP2.11 или ZmARF2, и пустой вектор placZi (AD-ZmRAP2.11/AC; AD-ZmARF2/AC). Колонии с положительным сигналом высевали и культивировали для количественных экспериментов с CPRG. Результаты определения активности репортерного гена в присутствии полноразмерного промотора ZmHAK1 и гена ZmRAP2.11 представлены на рис. 6б. Показано, что активность β-галактозидазы в опыте значительно выше, чем в отрицательном контроле. Аналогичные результаты получены и в случае полноразмерного промотора ZmHAK1 и гена ZmARF2 (рис. 6в). Это указывает на существование взаимодействий между факторами транскрипции ZmRAP2.11 и ZmARF2 и полноразмерным промотором ZmHAK1.
Рис. 6.
Взаимодействие промотора ZmHAK1 с факторами транскрипции ZmRAP2.11 и ZmARF2 в одногибридной дрожжевой системе. а ‒ Общая схема генетических конструкций, использованных в дрожжевой одногибридной системе. Гены ZmRAP2.11 и ZmARF2 клонировали в вектор pGAD424 (эффектор), а полноразмерный промотор ZmHAK1 и шесть его вариантов ‒ в вектор placZi (репортер). б, в ‒ Результаты количественной оценки активности β-галактозидазы (у.е.) указывают на то, что оба фактора транскрипции ‒ ZmRAP2.11 и ZmARF2 ‒ взаимодействуют с полноразмерным промотором ZmHAK1. г ‒ ZmRAP2.11 взаимодействует также с вариантами ΔP3 и ΔP6 промотора ZmHAK1. д ‒ ZmARF2 взаимодействует с вариантами ΔP1 и ΔP6 промотора ZmHAK1.
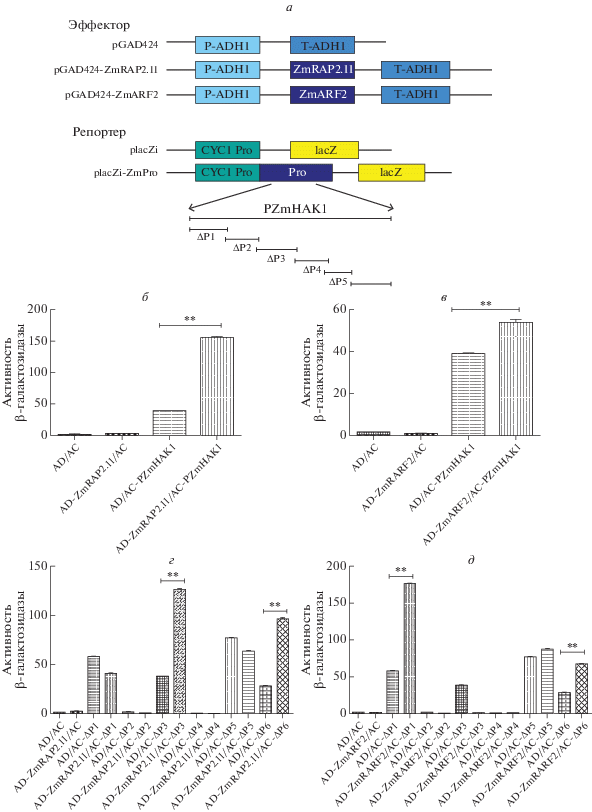
Чтобы определить специфическую последовательность промотора ZmHAK1, участвующую в связывании факторов транскрипции, мы разделили промотор на шесть перекрывающихся фрагментов с сегментами между каждым фрагментом (рис. 6a). Результаты количественного анализа активности β‑галактозидазы с помощью CPRG указывают на то, что ZmRAP2.11 взаимодействует с фрагментами ΔP3 (–903…–471 п.н.) и ΔP6 (+121…+573 п.н.) промотора ZmHAK1 (рис. 6г), а ZmARF2 взаимодействует с ΔP1 (–1669…–1223 п.н.) и ΔP6 (+121…+573 п.н.) промотора ZmHAK1 (рис. 6д).
Субклеточная локализация факторов транскрипции ZmRAP2.11 и ZmARF2
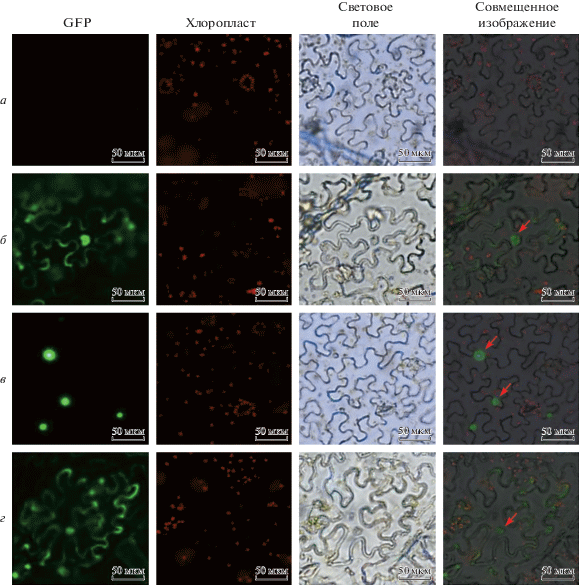
Чтобы определить внутриклеточную локализацию белков ZmARF2 и ZmRAP2.11, полноразмерные кДНК соответствующих генов без стоп-кодонов лигировали в модифицированный экспрессионный вектор pEarleyGate101 по технологии Gateway. Полученные плазмиды, несущие химерные репортерные гены ZmARF2-GFP и ZmRAP2.11-GFP, инъецировали в листья табака с помощью агроинфильтрации. После трансформации регистрировали флуоресцентные сигналы GFP. В качестве отрицательного контроля использовали неинъецированные листья табака (рис. 7а). Листья табака, трансформированные “пустым” вектором pEarleyGate101, служили положительным контролем. Сигналы зеленой флуоресценции наблюдали в ядре и мембране клеток табака (рис. 7б). После трансформации конструкцией, кодирующей ZmRAP2.11, сигнал флуоресценции обнаружен в ядре листьев табака, что указывает на локализацию ZmRAP2.11 в ядре (рис. 7в). После трансформации конструкцией, кодирующей ZmARF2, флуоресценцию обнаруживали в ядре и мембране клеток эпидермиса листьев табака, которая не перекрывалась с автофлуоресценцией хлоропластов, что указывает на локализацию ZmARF2 как в мембране, так и в ядре (рис. 7г).
Рис. 7.
Субклеточная локализация ZmRAP2.11 и ZmARF2 в листьях табака. Флуоресцентные сигналы визуализировали с помощью сканирующей микроскопии. а ‒ Листья табака дикого типа. б ‒ Листья после инъекции пустым вектором pEarleyGate 101. в ‒ Сигнал конструкции, кодирующей ZmRAP2.11-GFP. г ‒ Сигнал конструкции, кодирующей ZmARF2-GFP. Слева направо: зеленая флуоресценция, автофлуоресценция хлоропластов, световое поле и совмещенное изображение. Красные стрелки указывают на ядро.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Большинство растительных KT/KUP/HAK-транспортеров играют значительную роль в высокоаффинном транспорте K+ [18, 32–37], например, ген AtHAK5 может быстро и значительно индуцироваться при низкой концентрации калия [17, 18, 38‒40]. Нами ранее обнаружен и клонирован высокоаффинный переносчик калия ZmHAK1 кукурузы. Мы обнаружили, что последовательность гена ZmHAK1 имеет высокое сходство с последовательностью гена OsHAK1 риса. Его филогенетический анализ показал, что эти два гена принадлежат к одному кластеру, что указывает на то, что они могут иметь сходную функцию [41]. Мы также показали, что ZmHAK1 хорошо экспрессируется в корнях и стеблях кукурузы, что может повысить адаптацию растений к низкому содержанию калия и действию высоких концентраций солей. Однако проведено мало исследований функции промотора ZmHAK1 и его регуляции. В настоящей работе мы обнаружили, что полноразмерный промотор ZmHAK1 обеспечивает высокую активность GUS в корнях, стеблях и листьях табака в экспериментах с временной экспрессией репортерных конструкций. Промотор ZmHAK1 взаимодействует с факторами транскрипции ZmRAP2.11 и ZmARF2. С помощью делеционного анализа показано, что транскрипционный фактор ZmRAP2.11 взаимодействует с фрагментами ΔP3 и ΔP6 промотора ZmHAK, а фактор ZmARF2 ‒ с фрагментами ΔP1 и ΔP6 промотора ZmHAK1. В экспериментах по субклеточной локализации установлено, что ZmRAP2.11 находится в основном в ядре, тогда как ZmARF2 обнаруживается как в цитоплазме, так и в ядре. Широко распространено мнение, что тандемная экспансия и удвоение сегментов хромосом являются важным механизмом эволюции семейств генов [40]. В настоящей работе мы обнаружили 34 гена кукурузы, очень похожих на ZmHAK1 и неравномерно распределенных по 10 хромосомам. Мы также изучили эволюционную связь между семейством генов HAK кукурузы (34 гена) и других видов, включая Arabidopsis thaliana (13 генов) и рис (27 генов). Анализ филогенетического дерева показал, что гены HAK консервативны среди разных видов растений.
Растительные гормоны, такие как салициловая кислота и MeJA, участвуют в реакции растений на биотические и абиотические стрессы [42]. Промотор ZmHAK1 содержит несколько цис-действующих элементов, потенциально способных участвовать в клеточном ответе на биотические и абиотические стрессы. Мы обнаружили, что полноразмерный промотор ZmHAK1 активен в корнях, стеблях и листьях табака, и активность полноразмерного промотора выше, чем у четырех 5'-укороченных фрагментов. Ранее показали, что ген HAK кукурузы кодирует высокоаффинный переносчик калия, уровень транскрипции которого чувствителен к изменениям внутриклеточной и внеклеточной концентрации калия. В случае высокой концентрации K+ ген HAK1 экспрессируется на низком уровне. При снижении концентрации ионов калия наблюдается быстрая индукция гена HAK1 [27, 43]. Поэтому регуляция гена HAK1 на уровне транскрипции представляется важной. Тем не менее, транскрипционной регуляции семейства HAK1 посвящено мало исследований. В 2012 году сообщено, что транскрипционный фактор RAP2.11 может положительно регулировать уровень транскрипции гена HAK5, и ген HAK5 может также реагировать на этилен и активные формы кислорода [25]. Протеинкиназа CIPK23 может регулировать активность белка HAK5 путем его фосфорилирования [44]. В последние годы также сообщалось, что фактор ARF2 может негативно регулировать экспрессию HAK5 и реагировать на изменение концентрации калия посредством изменения статуса аутофосфорилирования [26]. Нами обнаружено, что промотор гена ZmHAK1 взаимодействует с факторами транскрипции ZmRAP2.11 и ZmARF2. Эти результаты согласуются с полученными ранее данными, указывающими на то, что фрагмент ΔP3 промотора содержит GCC-box, который специфически взаимодействует с RAP2.11 [19], а ΔP1 имеет три последовательности TGTC, которые специфически связывают ARF2 [45]. ΔP6 может содержать элементы, специфичные для обоих факторов транскрипции, что мы предполагаем проверить в дальнейшем.
Для дальнейшего изучения взаимодействия промотора ZmHAK1 с факторами транскрипции ZmRAP2.11 и ZmARF2 мы проанализировали характеристики этих двух факторов. ZmRAP2.11 относится к семейству AP2/ERF факторов транскрипции. Факторы транскрипции этого семейства связаны в основном с ростом и развитием растений, а также с абиотическим стрессом [46–51]. Сообщалось, что фактор ответа на ауксин ‒ ARF2, обеспечивает связь между сигнальными путями, активируемыми абсцизовой кислотой и ауксином [52]. Ауксин участвует во многих процессах и играет важную роль в нормальном росте и развитии растений. Многие абиотические стрессы могут влиять на содержание или распределение ауксина в растениях [53]. Мы обнаружили, что ZmARF2 локализуется как в мембране, так и в ядре и может играть важную роль в регуляции ZmHAK1.
Список литературы
Véry A., Sentenac H. (2003) Molecular mechanisms and regulation of K+ transport in higher plants. Annu. Rev. Plant Biol. 54, 575‒603.
Wang Y., Wu W.H. (2015) Genetic approaches for improvement of the crop potassium acquisition and utilization efficiency. Curr. Opin. Plant Biol. 25, 46‒52.
Pettigrew W.T. (2008) Potassium influences on yield and quality production for maize, wheat, soybean and cotton. Physiol. Plant. 133, 670‒681.
Nieves-Cordones M., Aleman F., Martinez V., Rubio F. (2014) K+ uptake in plant roots. The systems involved, their regulation and parallels in other organisms. J. Plant. Physiol. 171, 688‒695.
Maathuis F.J. (2009) Physiological functions of mineral macronutrients. Curr. Opin. Plant. Biol. 12, 250‒258.
Epstein E., Rains D.W., Elzam O.E. (1963) Resolution of dual mechanisms of potassium absorption by barley roots. Proc. Natl. Acad. Sci. USA. 49, 684‒692.
Maathuis F.J., Sanders D. (1994) Mechanism of high-affinity potassium uptake in roots of Arabidopsis thaliana. Proc. Natl. Acad. Sci. USA. 91, 9272‒9276.
Maser P., Thomine S., Schroeder J.I., Ward J.M., Hirschi K., Sze H., Talke I.N., Amtmann A., Maathuis F.J., Sanders D., Harper J.F., Tchieu J., Gribskov M., Persans M.W., Salt D.E., Kim S.A., Guerinot M.L. (2001) Phylogenetic relationships within cation transporter families of Arabidopsis. Plant Physiol. 126, 1646‒1667.
Ward J.M., Mäser P., Schroeder J.I. (2009) Plant ion channels: gene families, physiology, and functional genomics analyses. Annu. Rev. Physiol. 71, 59‒82.
Kochian L.V., Xin-Zhi J., Lucas W.J. (1985) Potassium transport in corn roots: IV. Characterization of the linear component. Plant Physiol. 79, 771‒776.
Ahn S J., Shin R., Schachtman D P. (2004) Expression of KT/KUP genes in Arabidopsis and the role of root hairs in K+ uptake. Plant Physiol. 134, 1135‒1145.
Gierth M., Maser P., Schroeder J.I. (2005) The potassium transporter AtHAK5 functions in K+ deprivation-induced high-affinity K+ uptake and AKT1 K+ channel contribution to K+ uptake kinetics in Arabidopsis roots. Plant Physiol. 137, 1105‒1114.
Chen G., Hu Q., Luo L., Yang T., Zhang S., Hu Y., Yu L., Xu G. (2015) Rice potassium transporter OsHAK1 is essential for maintaining potassium-mediated growth and functions in salt tolerance over low and high potassium concentration ranges. Plant Cell Environ. 38, 2747‒2765.
Wang F., Liu J., Li J., Zhang S., Pan H. (2014) Functional analyses of the maize CKS2 gene promoter in response to abiotic stresses and hormones. Acta Physiol. Plantarum. 36, 1867‒1878.
Delatorre C.A., Cohen Y., Liu L., Delatorre C.A., Cohen Y., Liu L., Peleg Z., Blumwald E. (2012) The regulation of the SARK promoter activity by hormones and environmental signals. Plant Sci. 193, 39‒47.
Tao Y., Wang F., Jia D., Li J., Zhang Y., Jia C., Wang D., Pan H. (2015) Cloning and functional analysis of the promoter of a stress-inducible gene (ZmRXO1) in maize. Plant Mol. Biol. Reporter. 33, 200‒208.
Zhang G.H., Wang H., Wang X.D., Feng M., Li H.M., Li S.Y. (2012) Isolation of the promoter region of HAK gene from Aeluropus littoralis and functional analysis in rice. Yi Chuan. 34, 742‒748.
Chen W.J., Zhu T. (2004) Networks of transcription factors with roles in environmental stress response. Trends Plant Sci. 9, 591‒596.
Golldack D., Lüking I., Yang O. (2011) Plant tolerance to drought and salinity: stress regulating transcription factors and their functional significance in the cellular transcriptional network. Plant Cell Repts. 30, 1383‒1391.
Amtmann A., Blatt M.R. (2009) Regulation of macronutrient transport. New Phytol. 181, 35‒52.
Ashley M.K., Grant M., Grabov A. (2006) Plant responses to potassium deficiencies: a role for potassium transport proteins. J. Exp. Bot.57, 425‒436.
Wang Y., Wu W. (2017) Regulation of potassium transport and signaling in plants. Curr. Opin. Plant Biol. 39, 123‒128.
Wang Y., Wu W. (2013) Potassium transport and signaling in higher plants. Annu. Rev. Plant Biol. 64, 451‒476.
Cherel I., Lefoulon C., Boeglin M., Sentenac H. (2014) Molecular mechanisms involved in plant adaptation to low K+ availability. J. Exp. Bot. 65, 833‒848.
Kim M.J., Ruzicka D., Shin R., Schachtman D.P. (2012) The Arabidopsis AP2/ERF transcription factor RAP2.11 modulates plant response to low-potassium conditions. Mol. Plant. 5, 1042‒1057.
Zhao S., Zhang M., Ma T., Wang Y. (2017) Phosphorylation of ARF2 relieves its repression of transcription of the K+ transporter gene HAK5 in response to low potassium stress. Plant Cell. 28, 3005‒3019.
Zhang Z., Zhang J., Chen Y., Li R., Wang H., Wei J. (2012) Genome-wide analysis and identification of HAK potassium transporter gene family in maize (Zea mays L.). Mol. Biol. Rep. 39, 8465‒8473.
Kumar S., Stecher G., Tamura K. (2016) MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Mol. Biol. Evolution. 3 3, 1870‒1874.
Shahmuradov I.A. (2003) PlantProm: a database of plant promoter sequences. Nucl. Acids Res. 31, 114‒117.
Kruger N.J. (1994) The Bradford method for protein quantitation. Methods Mol. Biol. 32, 9‒15.
Shen Y., Shen L., Shen Z., Jing W., Ge H., Zhao J., Zhang W. (2015). The potassium transporter OsHAK21 functions in the maintenance of ion homeostasis and tolerance to salt stress in rice. Plant Cell Environ. 38, 2766‒2779.
Banuelos M.A., Garciadeblas B., Cubero B., Rodriguez-Navarro A. (2002) Inventory and functional characterization of the HAK potassium transporters of rice. Plant Physiol. 130, 784‒795.
Martinez-Cordero M.A., Martinez V., Rubio F. (2004) Cloning and functional characterization of the high-affinity K+ transporter HAK1 of pepper. Plant Mol. Biol. 56, 413‒421.
Nieves-Cordones M., Miller A J., Aleman F., Martinez V., Rubio F. (2008) A putative role for the plasma membrane potential in the control of the expression of the gene encoding the tomato high-affinity potassium transporter HAK5. Plant Mol. Biol. 68, 521‒532.
Santa-Maria G.E., Rubio F., Dubcovsky J., Rodriguez-Navarro A. (1997) The HAK1 gene of barley is a member of a large gene family and encodes a high-affinity potassium transporter. Plant Cell. 9, 2281‒2289.
Su Q., Feng S., An L., Zhang G. (2007) Cloning and functional expression in Saccharomyces cereviae of a K+ transporter, AlHAK, from the graminaceous halophyte, Aeluropus littoralis. Biotechnol. Lett. 29, 1959‒1963.
Yang T., Zhang S., Hu Y., Wu F., Hu Q., Chen G., Cai J., Wu T., Moran N., Yu L., Xu G. (2014) The role of a potassium transporter OsHAK5 in potassium acquisition and transport from roots to shoots in rice at low potassium supply levels. Plant Physiol. 166, 945‒959.
Armengaud P., Breitling R., Amtmann A. (2004) The potassium-dependent transcriptome of Arabidopsis reveals a prominent role of jasmonic acid in nutrient signaling. Plant Physiol. 136, 2556‒2576.
Shin R., Schachtman D.P. (2004) Hydrogen peroxide mediates plant root cell response to nutrient deprivation. Proc. Natl. Acad. Sci. USA. 101, 8827‒8832.
Leister D. (2004) Tandem and segmental gene duplication and recombination in the evolution of plant disease resistance gene. Trends Genet. 20, 116‒122.
Zhang Zh.B., Zhang J.W., Chen Y.J., Li R.F., Wang H.Zh., Wei J.H. (2012) Genome-wide analysis and identification of HAK potassium transporter gene family in maize (Zea mays L.). Mol. Biol. Rep. 39, 8465–8473.
Ohshima M., Itoh H., Matsuoka M., Murakami T., Ohashi Y. (1990) Analysis of stress-induced or salicylic acid-induced expression of the pathogenesis-related 1a protein gene in transgenic tobacco. Plant Cell. 2, 95‒106.
Qin Y.J., Wu W.H., Wang Y. (2019) ZmHAK5 and ZmHAK1 function in K+ uptake and distribution in maize under low K+ conditions. J. Integr. Plant. Biol. 61(6), 691‒705. https://doi.org/10.1111/jipb.12756
Ragel P., Rodenas R., Garcia-Martin E., Andres Z., Villalta I., Nieves-Cordones M., Rivero R.M., Martinez V., Pardo J.M., Quintero F.J., Rubio F. (2015) The CBL-interacting protein kinase CIPK23 regulates HAK5-mediated high-affinity K+ uptake in Arabidopsis roots. Plant Physiol. 169, 2863‒2873.
Cherenkov P., Novikova D., Omelyanchuk N., Levitsky V., Grosse I., Weijers D., Mironova V. (2018) Diversity of cis-regulatory elements associated with auxin response in Arabidopsis thaliana. J. Exp. Bot. 69, 329‒339.
Liu C., Zhang T. (2017) Expansion and stress responses of the AP2/EREBP superfamily in cotton. BMC Genomics. 18, 118.
Li H., Wang Y., Wu M., Li L., Li C., Han Z., Yuan J., Chen C., Song W., Wang C. (2017) Genome-wide identification of AP2/ERF transcription factors in cauliflower and expression profiling of the ERF family under salt and drought stresses. Front. Plant Sci. 8, 946.
Cui L., Feng K., Wang M., Wang M., Deng P., Song W., Nie X. (2016) Genome-wide identification, phylogeny and expression analysis of AP2/ERF transcription factors family in Brachypodium distachyon. BMC Genomics. 17, 636.
Li X., Gao B., Zhang D., Liang Y., Liu X., Zhao J., Zhang J., Wood A J. (2018) Identification, classification, and functional analysis of AP2/ERF family genes in the desert moss Bryum argenteum. Int. J. Mol. Sci. 19, pii: E3637.
Li X., Tao S., Wei S., Ming M., Huang X., Zhang S., Wu J. (2018) The mining and evolutionary investigation of AP2/ERF genes in pear (Pyrus). BMC Plant Biol. 18, 46.
Ahn H., Jung I., Shin S.J., Park J., Rhee S., Kim J.K., Jung W., Kwon H.B., Kim S. (2017) Transcriptional network analysis reveals drought resistance mechanisms of AP2/ERF transgenic rice. Front. Plant Sci. 8, 1044.
Wang L., Hua D., He J., Duan Y., Chen Z., Hong X., Gong Z. (2011) Auxin Response Factor 2 (ARF2) and its regulated homeodomain gene HB33 mediate abscisic acid response in Arabidopsis. PLoS Genet. 7, e1002172.
Vanneste S., Friml J. (2009) Auxin: a trigger for change in plant development. Cell. 136, 1005‒1016.
Дополнительные материалы
- скачать ESM_1.docx
- Supplemental Table 1. Primer sequences and enzyme sites used in this study.
- скачать ESM_2.xlsx
- Supplemental Table 2. Characterization of HAK proteins in maize.
Инструменты
Молекулярная биология


